Method Article
Quantification de l’agrégation de protéines insolubles chez Caenorhabditis elegans au cours du vieillissement avec un nouveau flux de travail d’acquisition indépendant des données
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce nouveau flux de travail permet d’extraire et d’isoler efficacement les protéines insolubles dans le SDS (insolublome) de Caenorhabditis elegans avec un minimum de matériel de départ pour l’analyse protéomique différentielle quantitative. Le protocole utilise une analyse complète par spectrométrie de masse à acquisition indépendante des données pour quantifier l’insolublome et une analyse bioinformatique pour obtenir des informations biologiques sur les mécanismes et les pathologies du vieillissement.
Résumé
Nous et d’autres avons montré que le processus de vieillissement entraîne une accumulation de protéines insolubles à l’échelle du protéome. L’élimination des gènes codant pour les protéines insolubles plus de 40 % du temps entraîne une prolongation de la durée de vie de C. elegans, ce qui suggère que bon nombre de ces protéines sont des déterminants clés du processus de vieillissement. L’isolement et l’identification quantitative de ces protéines insolubles sont essentiels pour comprendre les principaux processus biologiques qui se produisent au cours du vieillissement. Ici, nous présentons un protocole modifié et amélioré qui détaille comment extraire et isoler plus efficacement les protéines insolubles SDS (insolublome) de C. elegans afin de rationaliser les flux de travail de spectrométrie de masse via une nouvelle analyse protéomique quantitative sans marquage. Ce protocole amélioré utilise un sonicateur très efficace pour la lyse des vers qui augmente considérablement l’efficacité de l’extraction des protéines et nous permet d’utiliser beaucoup moins de matériel de départ (environ 3 000 vers) que dans les protocoles précédents (utilisant généralement au moins 40 000 vers). L’analyse protéomique quantitative ultérieure de l’insolublome a été réalisée à l’aide de l’acquisition dépendante des données (DDA) pour la découverte et l’identification des protéines et de l’acquisition indépendante des données (DIA) pour une quantification complète et plus précise des protéines. L’analyse bioinformatique des protéines quantifiées fournit des candidats potentiels qui peuvent être facilement suivis avec d’autres méthodes moléculaires chez C. elegans. Grâce à ce flux de travail, nous identifions régulièrement plus de 1000 protéines et quantifions plus de 500 protéines. Ce nouveau protocole permet un criblage efficace des composés avec C. elegans. Ici, nous avons validé et appliqué ce protocole amélioré à la souche N2-Bristol de C. elegans de type sauvage et confirmé que les vers N2 âgés du jour 10 présentaient une plus grande accumulation de l’insolublome que les jeunes vers du jour 2.
Introduction
L’homéostasie des protéines diminue progressivement avec l’âge et entraîne une augmentation de l’agrégation des protéines 1,2,3. L’agrégation des protéines est associée à plusieurs maladies neurodégénératives, notamment la maladie d’Alzheimer, la maladie de Parkinson, la maladie de Huntington et la sclérose latérale amyotrophique4. Le vieillissement est considéré comme l’un des principaux facteurs de risque d’apparition de troubles neurodégénératifs associés à l’agrégation de protéines. Les protéines qui ont tendance à former des agrégats insolubles sont souvent liées à une toxicité cellulaire et à un dysfonctionnement tissulaire, ce qui pourrait accélérer l’agrégation d’autres protéines 5,6,7. Alternativement, les agrégats de protéines insolubles peuvent activer les mécanismes de défense cellulaire pour éliminer les formes oligomères toxiques de la protéine du système. L’inhibition de gènes sélectionnés codant pour des protéines insolubles module la durée de vie de Caenorhabditis elegans (C. elegans) dans le contexte des maladies liées à l’âge et du vieillissement normal 5,8,9. Ainsi, l’étude des mécanismes cellulaires et moléculaires de l’agrégation des protéines est cruciale pour comprendre le vieillissement et, en fin de compte, pourrait conduire à des approches pour traiter les maladies neurodégénératives.
Le nématode C. elegans est devenu l’un des organismes modèles les plus utilisés pour étudier l’agrégation des protéines dans le vieillissement et les maladies liées à l’âge en raison de ses caractéristiques uniques, telles qu’une durée de vie relativement courte (environ 2 semaines), la facilité de culture et la manipulation génétique.
La capacité d’extraire et de caractériser des protéines insolubles a joué un rôle essentiel dans la détermination des changements liés à l’âge associés à l’agrégation des protéines dans les modèles de C. elegans. Pour étudier la contribution de l’agrégation des protéines aux processus de vieillissement normaux, nous5 et d’autres2 avons préalablement extrait et digéré protéolytiquement l’insolublome de C. elegans jeunes par rapport à des C. elegans âgés, marqués chimiquement à l’aide de réactifs iTRAQ (« marquage isobare pour une quantification relative et absolue »), puis quantifiés à l’aide de méthodes basées sur la MS. En utilisant une méthode de marquage isobare et 120 mg de vers humides (environ 40 000 vers), nous avons pu obtenir une profondeur et une couverture insolublomes protéiques significatives5. L’analyse quantitative a démontré que 203 des 1200 protéines identifiées étaient significativement enrichies dans l’insolublome de C. elegans âgés par rapport à des fractions insolublomes similaires de jeunes vers5. Indépendamment, David et al. ont également utilisé un flux de travail iTRAQ LC-MS/MS pour examiner les altérations des agrégats de protéines avec le vieillissement normal2. En commençant avec environ 300 mg de vers, ils ont identifié ~1000 protéines insolubles à l’aide de deux réplicats biologiques et ont déterminé que ~700 protéines sur environ 1000 se sont accumulées 1,5 fois ou plus avec l’âge par rapport aux jeunes vers2. Dans l’ensemble, ces résultats indépendants indiquent que l’insolubilité et l’agrégation généralisées des protéines font partie intégrante du vieillissement normal et peuvent affecter à la fois la durée de vie et l’incidence des maladies neurodégénératives 2,5.
L’étude de l’insolublome nous a permis de déterminer comment les influences environnementales peuvent accélérer ou ralentir le processus de vieillissement. Klang et al. ont établi des flux de travail protéomiques sans marquage chez C. elegans pour étudier le rôle de la métallostase dans la longévité10. Dans cette étude, au moins 40 000 vers ont été utilisés pour extraire l’insolublome10. Les données ont montré que les niveaux de fer, de cuivre, de calcium et de manganèse augmentent avec l’âge et que l’alimentation des vers avec un régime riche en fer accélérait considérablement l’accumulation de protéines insolubles liée à l’âge10. En utilisant le même flux de travail pour examiner les effets de la vitamine D sur l’insolublome de C. elegans, 38 protéines ont été quantifiées chez de jeunes vers (jour 2) et 721 protéines chez des vers âgés (jour 8). L’alimentation en vitamine D a considérablement réduit l’insolublme des vers âgés de 721 à 371 protéines11. Des recherches plus approfondies ont révélé que l’alimentation en vitamine D supprimait l’insolubilité des protéines avec l’âge, favorisait l’homéostasie des protéines et prolongeait la durée de vie chez les vers sauvages C . elegans N211. Ainsi, l’étude de l’insolublome peut aider à identifier de nouveaux modulateurs du vieillissement et des maladies liées à l’âge.
Bien que l’étude de l’insolublome ait été inestimable pour faire progresser la compréhension du processus de vieillissement, elle a été entravée par la nécessité de collecter de grandes quantités d’échantillons de départ. Groh et al. ont récemment introduit un flux de travail de quantification protéomique sans marquage pour étudier les changements inhérents à l’agrégation des protéines chez C. elegans avec l’âge ; Cependant, il nécessitait de grandes quantités de matière première (350 mg de vers moulus)12. Dans le présent rapport, nous avons établi un nouveau protocole d’extraction et d’isolement amélioré (figure 1). L’utilisation du sonicateur très efficace pendant la lyse des vers a considérablement amélioré l’efficacité de l’extraction et a par conséquent réduit la quantité de matériau de départ nécessaire, de 40 000 à 3 000 vers. La combinaison de ce nouveau protocole d’isolement insolublome avec un flux de travail de spectrométrie de masse DIA (Data-Independent Acquisition) sans marquage a considérablement amélioré la profondeur et la couverture des protéines. Le protocole présenté ici est rentable et facilement modifiable pour permettre la réalisation d’analyses insolublomes dans d’autres systèmes modèles.
Protocole
REMARQUE : Pour une meilleure compréhension de la procédure expérimentale, voir la figure 1 pour un schéma du flux de travail.
1. Culture de masse de C. elegans vieillissants synchronisés
- Préparation des plaques
- Verser 30 mL de gélose NGM avec ou sans 50 mM de fluorodésoxyuridine (FUdR) dans chaque plaque de 90 mm et laisser sécher les plaques pendant 2 jours dans des hottes de culture.
- La veille de l’ensemencement de la gélose NGM, inoculer une aliquote de 50 μL de glycérol congelé de la souche bactérienne OP50 dans 1 L de milieu LB pré-autoclavé dans une fiole de 2 L.
- Faites pousser les bactéries à 37 °C dans un incubateur à agitation à 250 tr/min pendant 16 h.
- Après 16 h, centrifuger les cultures bactériennes à une vitesse de 5 000 x g pendant 10 min.
- Jeter le surnageant et remettre complètement en suspension la pastille bactérienne dans 40 mL de solution S-basale autoclavée (contenant 50 mM K3PO4, 100 mM de NaCl dans l’eau) pour obtenir la culture d’E. coli OP50 concentrée 25x.
- Versez 2 ml de la culture bactérienne concentrée 25x sur chaque plaque de gélose NGM et étalez-la uniformément de sorte que la culture bactérienne couvre toute la surface de la gélose.
- Retirez les couvercles des plaques dans une hotte de culture bactérienne pendant 2 à 3 h jusqu’à ce que les plaques soient correctement sèches.
REMARQUE : Assurez-vous de répartir uniformément les bactéries en faisant tourner les plaques toutes les 15 minutes. Ceci est important car les bactéries ont tendance à se concentrer sur un côté de la plaque, ce qui entraîne des régions à faible densité bactérienne. - Une fois les plaques sèches, fermez les couvercles et laissez-les à température ambiante pendant 48 h pour assurer un séchage complet de la source alimentaire OP50 E . coli .
REMARQUE : Les plaques peuvent être préparées 2 semaines à l’avance et stockées à 4 °C avant utilisation.
- Maintenance et préparation de cohortes synchronisées de C. elegans
- Pour obtenir l’apport requis de lysat de protéine (1,0 à 1,5 mg) pour l’isolement de la fraction insoluble dans le SDS, utilisez environ 3 000 vers synchronisés par échantillon.
- Transférez ~100 vers adultes gravides de type sauvage ~100 N2 dans une plaque ensemencée par E. coli NGM 25x OP50 et maintenez les vers à 20 °C pendant ~72 h jusqu’à ce que la plaque soit pleine de vers adultes gravides.
- Pipeter ~10 mL de solution S-basale sur chaque plaque et recueillir tous les vers dans un tube de 15 mL.
REMARQUE : Il est important d’utiliser des pointes à faible rétention ou des pipettes Pasteur pendant cette étape et toutes les étapes futures impliquant la collecte de vers pour éviter toute perte de vers due à la fixation sur les surfaces des pointes. - Faites tourner l’échantillon à 520 x g pendant 30 s. Retirez le surnageant après l’essorage. Pour éliminer toute E. coli OP50, ajoutez 10 ml de solution S-basale à la pastille et essorez à nouveau, puis retirez le surnageant. Répétez cette étape.
- Ajouter 10 ml de solution de blanchiment à l’hypochlorite de sodium (contenant 0,5 M de KOH et 0,48 % d’hypochlorite de sodium dans l’eau) à la pastille de vers dans un tube conique de 15 ml et agiter vigoureusement pendant 2 minutes à température ambiante.
- Centrifuger à 520 x g pendant 30 s à température ambiante pour éliminer les corps de vers et les œufs incomplètement dissous. Retirez ensuite le surnageant.
- Répéter les étapes 1.2.5 et 1.2.6 pour s’assurer de l’élimination complète de tous les vers et de la libération des œufs (voir le tube au microscope pour s’en assurer).
- Lavez la pastille d’œuf au moins 4 fois avec 10 ml de solution S-basale pour assurer l’élimination complète de la solution d’eau de Javel.
REMARQUE : Il est important d’enlever complètement l’eau de Javel, car l’eau de Javel restante peut entraver l’éclosion des œufs. - Laisser les œufs en suspension dans 5 mL de milieu S-basal dans un tube de 15 mL et les placer dans le rotor à 20 °C pendant 24 h pour permettre aux œufs d’éclore.
- Après 24 h, prélever 10 μL de solution de larves S-basale pour compter le nombre de vers qu’elle contient. Utiliser au moins trois aliquotes distinctes de 10 μL de solution de larves S-basales pour la réplication du nombre de vers. Calculez la moyenne des trois concentrations larvaires. Sur la base de la concentration moyenne, diluer la solution de vers de terre pour obtenir ~3 000 larves par 200 μL de solution.
REMARQUE : Assurez-vous de secouer doucement le tube en le retournant car les larves ont tendance à se déposer si elles ne sont pas dérangées, ce qui peut affecter le nombre de larves. - Bien mélanger et ajouter 200 μL de solution de larves S-basale sur chacune des plaques de 90 mm ensemencées par des bactéries NGM.
REMARQUE : Ajouter des gouttes de solution de larves S-basales dans toute la plaque pour une répartition uniforme des larves afin d’éviter l’épuisement bactérien de certains endroits. - Laisser sécher la solution S-basale, puis placer les plaques inversées dans un incubateur à 20 °C pendant 48 h.
- Après 48 h, recueillir les vers de la plaque en solution S-basale et les transférer dans une plaque fraîche ensemencée de NGM contenant 50 mM de FUDR et incuber des plaques à 20 °C.
REMARQUE : Il est important de transférer les vers tôt dans l’étape L4 pour assurer l’efficacité du FUDR. - Prélever des échantillons de jeunes adultes au jour 2 après 48 h en ajoutant 10 mL de solution S-basale sur la plaque et en transférant la solution dans un tube de 15 mL. Laissez les vers se déposer par gravité et retirez la solution S-basale une fois qu’elle devient claire.
- Lavez la pastille de ver 2 à 3 fois dans 5 mL de solution S-basale pour éliminer toutes les bactéries attachées. Prélever autant de solution S-basale que possible et congeler la pastille de vers (en tubes de 15 ml) dans un bain de glace sèche ou d’éthanol. Les échantillons congelés sont conservés à -80 °C.
- Transférez les vers de l’échantillon dans une plaque fraîche NGM OP50 E . coli contenant du FUDR tous les deux jours jusqu’au 10e jour. Collectez et congelez les vers comme décrit aux étapes 1.2.14 et 1.2.15.
REMARQUE : Commencez avec un nombre plus élevé de vers pour les échantillons du jour 10 afin de tenir compte de toute perte de vers due au transfert et à l’enfouissement.
2. Extraction de la fraction insoluble dans les SDS des vers
- Décongeler la pastille de ver congelée sur de la glace en présence de 900 μL de cocktail tampon de lyse de vers glacé contenant 20 mM de base Tris, un pH de 7,4, 100 mM de NaCl, 1 mM de MgCl2 et un inhibiteur de protéase sans EDTA. Échantillonner brièvement des vortex pour assurer la remise en suspension complète de la pastille de ver dans le tampon de lyse.
- Placez les tubes de 15 ml dans le bain de sonication et réglez pour 10 cycles de sonication (30 s allumés et 30 s éteints pour chaque cycle) à haute intensité. Répétez l’opération jusqu’à cinq cycles (assurez-vous que les vers sont complètement lysés).
- Faites tourner le lysat à 3 000 x g pendant 4 min dans la chambre froide. Après la centrifugation, jeter la pastille contenant les débris de vers et transférer soigneusement le surnageant dans des tubes à centrifuger de 1,5 ml pré-refroidis sur de la glace.
- Quantifier la concentration en protéines à l’aide du dosage de l’ACB. Transférez les aliquotes de lysat, contenant généralement environ 1 mg de protéines, dans des tubes à centrifuger frais et prérefroidis de 1,5 ml.
- Centrifuger le lysat de protéines pendant 15 min à 20 000 x g dans la chambre froide.
- Sans déranger la pastille, transférez le surnageant dans un tube à centrifuger frais de 1,5 mL et conservez-le sous forme de fraction protéique soluble dans l’eau.
- Lavez la pastille de l’étape précédente dans 500 μL de tampon de lyse sans fin contenant 1 % de SDS à température ambiante et centrifugez-la à 20 000 x g pendant 15 min à température ambiante. Retirez le surnageant et enregistrez-le en tant que fraction soluble dans la SDS. Répétez cette étape de lavage deux fois pour éliminer toute fraction soluble dans le SDS. La pastille restante après le troisième cycle de lavage est définie comme la fraction protéique insoluble à 1 % de SDS.
- Remettez en suspension la pastille de protéine insoluble dans 60 μL d’acide formique à 70 % et agitez vigoureusement pour dissoudre les protéines. Répétez le vortex vigoureux autant de fois que nécessaire pour dissoudre la pastille.
- Sonicez la pastille pendant 30 min dans un bain-marie à ultrasons à température ambiante.
REMARQUE : La pastille insoluble SDS est généralement difficile à dissoudre dans le tampon. Cependant, à la fin, la totalité de la pastille doit être dissoute dans cette étape. - Sécher les échantillons dans un concentrateur sous vide pendant 1 h pour éliminer complètement la solution d’acide formique.
- Ajoutez 40 μL de 1 tampon de gel d’échantillon LDS dans la pastille séchée et chauffez l’échantillon à 95 °C pendant 10 min. Vortex brièvement et faites tourner les échantillons. Chargez 13 μL sur un gel NUPAGE Bis-Tris à 4-12 % et faites couler le gel. Teindre le gel avec un colorant protéique fluorescent pour l’imagerie. Conservez le reste de l’échantillon pour l’analyse par spectrométrie de masse (MS).
- Chargez l’échantillon restant dans un gel de Bis-Tris à 4-12 % et laissez fonctionner pendant environ 20 minutes pour la digestion MS dans le gel.
3. Digestion en gel avec de la trypsine protéase pour isoler les protéines pour l’analyse MS
- Préparez les solutions suivantes (fraîches) : 25 mM de NH4HCO3 (pH 7-8), 25 mM de NH4HCO3 dans de l’acétonitrile à 50 % (ACN, pH 7-8) et 50 % d’ACN dans de l’acide formique à 5 %.
- Coupez chaque tranche de gel en petits morceaux (généralement <1 mm2) et placez-les dans des tubes siliconés de 0,65 ml.
- Ajouter environ 100 μL (ou assez pour couvrir) d’une solution de 25 mM de NH4HCO3/50 % d’ACN et agiter à température ambiante pendant 10 min. Extraire le surnageant et le transférer dans un tube séparé (à jeter). Répétez cette étape deux fois de plus.
- Séchez complètement les morceaux de gel dans un concentrateur sous vide (~20 min).
- Préparez des solutions fraîches et ajoutez ~100 μL (ou assez pour couvrir) de 10 mM de DTT dans 25 mM de NH4HCO3 aux morceaux de gel séchés (utilisez du NH4HCO3 fraîchement préparé). Vortex et tournez brièvement. Laisser la réaction se dérouler à 56 °C pendant 1 h et en agitant à 1400 tr/min sur un mélangeur.
- Retirer le surnageant et ajouter 100 μL de 55 mM d’iodoacétamide (IAA) dans 25 mM de NH4HCO3 aux morceaux de gel. Vortex et tournez brièvement. Laisser la réaction se dérouler dans l’obscurité pendant 45 minutes à RT.
- Retirer le surnageant et le jeter. Lavez les morceaux de gel en ajoutant ~100 μL de 25 mM NH4HCO3 et en tourbillonnant pendant 10 min. Essorez brièvement et retirez le surnageant, en jetant ce dernier.
- Déshydratez les morceaux de gel en ajoutant ~100 μL (ou assez pour couvrir) de 25 mM de NH4HCO3 dans 50 % d’ACN aux morceaux de gel et vortex pendant 10 min. Ensuite, essorez brièvement pour retirer et jeter le surnageant. Répétez cette étape deux ou même une troisième fois si les morceaux de gel ne sont pas tout à fait secs, comme l’indiquent la taille plus petite des morceaux de gel et la couleur blanche trouble.
- Séchez complètement les morceaux de gel dans un concentrateur sous vide (~20 min).
- Ajoutez d’abord 15 μL de solution de trypsine (250 ng de trypsine) à chaque échantillon, puis ajoutez suffisamment de 25 mM de NH4HCO3 pour couvrir les morceaux de gel (~100 μL). Vortex pendant 10 min. Ensuite, essorez brièvement et incubez à 4 °C dans la chambre froide pendant 30 min sans mélanger.
- Ajouter 25 mM de NH4HCO3 au besoin pour couvrir complètement les morceaux de gel. Essorer et incuber à 37 °C pendant une nuit de 16 à 20 h à 1400 tr/min sur un mélangeur.
- Le lendemain, faites un bref vortex et faites tourner la digestion. Ajouter environ 100 μL d’eau de qualité HPLC, essorer, parafilmer le tube et sonicer en continu pendant 10 min. Tourner brièvement après la sonication.
- Transférez la solution de digestion, représentant l’extraction aqueuse, dans un tube siliconé propre de 0,65 mL.
- Ajouter ~100 μL d’acide 50 % ACN/5 % d’acide formique aux morceaux de gel (assez pour les couvrir), agiter 10 min à température ambiante, tourner brièvement et recueillir la solution, et répéter une fois. Regroupez toutes les solutions contenant les peptides extraits de cette étape et de l’étape précédente 3.13 dans un seul tube.
- Vortex les digestions extraites. Sécher complètement les peptides dans un concentrateur sous vide (~2 h).
- Ajouter 30 μL d’acide formique à 0,2 % pour remettre les peptides en suspension sur un mélangeur dans la chambre froide pendant 10 min.
- Faites tourner les échantillons à 1850 x g à température ambiante pendant 5 min. Aspirez la solution peptidique et placez-la dans un nouveau tube siliconé propre de 0,65 ml. Dessalez ensuite la solution de peptides à l’aide d’embouts de dessalage C18 (voir ci-dessous).
4. Dessalage des peptides digérés avec une pointe de dessalage C18
- Réglez la pipette à 10 μL et fixez l’embout de dessalage C18. Mouillez l’embout de dessalage en pipetant 10 μL de 100 % d’ACN, puis en le jetant. Répétez cette étape 2 fois.
- Lavez l’embout de dessalage C18 en pipetant 10 μL de 50 % d’ACN, 49,8 % d’eau et 0,2 % d’AF, puis jetez-le. Répétez cette étape 2 fois.
- Équilibrez l’embout de dessalage en C18 en pipetant 10 μL de 0,2 % de FA dans de l’eau, puis en le jetant. Répétez cette étape 2 fois.
- Réglez la pipette à 10 μL et chargez les peptides de la solution dans la résine en pipetant les peptides digérés à travers la résine de haut en bas 15x.
REMARQUE : L’action répétée assurera la liaison de tous les peptides à la résine des pointes de dessalage. - Dessalez les peptides digérés liés à la résine en pipetant 10 μL de 0,2 % de FA dans de l’eau, puis en les jetant. Répétez cette étape 4 fois pour terminer l’étape de dessalage.
- Dans un nouveau tube, éluez les peptides avec 10 μL de 50 % d’ACN, 49,8 % d’eau et 0,2 % d’AF en pipetant de haut en bas 10x. Répétez cette étape une fois pour éluer la digestion des protéines une deuxième fois dans le même tube.
- Séchez complètement les peptides dessalés dans un concentrateur sous vide (~20 min).
- Remettre en suspension les peptides dans 15 μL de peptides à 0,2 % d’AF + 1 μL d’iRT (temps de rétention indexé). Voxtex pendant 10 min, centrifuger à 12 000 x g pendant 2 min, puis transférer dans un flacon d’échantillonneur automatique pour l’analyse MS (voir ci-dessous).
5. Analyse par spectrométrie de masse de peptides digérés à l’aide de DDA et DIA
REMARQUE : Les échantillons peuvent être analysés à l’aide des méthodes DDA ou DIA LC-MS/MS. Dans cette étude, les échantillons ont été analysés à l’aide d’un système HPLC nano-LC 2D couplé à un spectromètre de masse à haute résolution.
- Utilisez un système HPLC combiné à un système HPLC basé sur une puce directement connecté à un spectromètre de masse quadripolaire à temps de vol (d’autres configurations et systèmes LC-MS peuvent également être utilisés).
- Analysez des échantillons à l’aide de HPLC-ESI-MS/MS en phase inverse.
- Construisez le protocole de chromatographie pour charger un échantillon de peptides sur une puce de pré-colonne C18. Laver et dessaler les peptides chargés avec un solvant de charge (acide formique à 0,1 %) pendant 10 min à un débit de 2 μL/min.
- Transférez les peptides dans une puce C18 de colonne analytique et éluez-les à un débit de 300 nL/min avec un gradient de 3 heures en utilisant les phases mobiles A (2 % d’acétonitrile, 0,1 % d’acide formique) et B (98 % d’acétonitrile, 0,1 % d’acide formique). La première étape d’élution consiste en un gradient linéaire de 5 % B à 35 % B sur une période de 80 min.
- Augmentez la phase mobile B à 80 % pendant 5 min, puis maintenez la phase mobile B à 80 % pendant 8 min avant de passer à 5 % B pour rééquilibrer la colonne pendant 25 min.
- Configurez une méthode d’instrument MS pour DDA et définissez les paramètres de l’instrument comme suit.
- Utilisez les paramètres suivants pour le balayage des ions précurseurs de l’expérience 1 :MS1 de m/z 400 à 1500 (temps d’accumulation de 250 ms) ; régler le seuil d’intensité pour déclencher les balayages MS/MS pour les ions des états de charge de 2 à 5 à 200 comptes ; Réglez l’exclusion dynamique des ions précurseurs sur 60 s).
- Utilisez les paramètres suivants pour l’expérience 2 : balayages d’ions de produit MS/MS avec une plage de balayage MS2 de m/z 100 à 1500 (temps d’accumulation de 100 ms pour chacun des 30 balayages d’ions de produit par cycle) ; réglez la propagation de l’énergie de collision sur CES=5 et cochez la case 'Mode de balayage ionique du produit haute sensibilité'.
REMARQUE : La méthode d’acquisition DDA est utilisée pour construire des bibliothèques spectrales comme décrit à l’étape 6.3 (voir ci-dessous). Ici, il acquerra des spectres MS/MS pour les 30 ions précurseurs les plus abondants après chaque balayage MS1 par cycle ; La durée totale du cycle est d’environ 3,3 s.
- Configurez une méthode d’instrument MS pour DIA et définissez les paramètres instrumentaux comme suit.
- Utilisez les paramètres suivants pour l’expérience 1 : balayage des ions précurseurs MS1 de m/z 400 à 1250 (temps d’accumulation de 250 ms).
- Utiliser les paramètres suivants pour l’expérience 2 : balayages d’ions de produit MS/MS pour 64 segments SWATH variables avec une plage de balayage MS2 de m/z 100-1500 (temps d’accumulation de 45 ms pour chacun des 64 balayages d’ions de produit par cycle) ; réglez l’étalement de l’énergie de collision sur CES=10, cochez la case 'Mode de balayage ionique du produit haute sensibilité'.
REMARQUE : Utiliser la méthode d’acquisition DIA/SWATH à 64 variables décrite par Schilling et al.13 pour effectuer une quantification sans marquage avec une durée de cycle totale d’environ 3,2 s. Pour l’acquisition DDA, au lieu d’utiliser le quadripôle Q1 pour transmettre l’ion précurseur d’une plage de masse étroite à la cellule de collision, une plage de largeur de fenêtre variable (par exemple m/z 5–90) est utilisée pour franchir progressivement la gamme complète m/z (m/z 400–1250) avec 64 segments SWATH, 45 ms de temps d’accumulation pour chaque segment, ce qui permet d’obtenir un temps de cycle de 3,2 secondes, qui comprend un balayage MS1 avec un temps d’accumulation de 250 ms. La largeur de la fenêtre variable est ajustée en fonction de la complexité du courant ionique MS1 typique observé dans une certaine plage m/z à l’aide d’un algorithme SCIEX « calculateur de fenêtre variable» 13 (les fenêtres plus étroites sont appliquées aux plages m/z « occupées », les fenêtres plus larges sont appliquées aux plages m/z avec peu d’ions précurseurs éluants). Sur d’autres plates-formes MS, d’autres stratégies de sélection de fenêtre DIA peuvent également être mises en œuvre.
6. Analyse des données
REMARQUE : Certains paramètres d’analyse des données doivent être adaptés à des conditions expérimentales spécifiques. Par exemple, la base de données de protéines (fichier fasta) sélectionnée dépendra de l’espèce à partir de laquelle l’échantillon a été préparé (dans ce protocole, nous avons utilisé C. elegans).
- Utilisez un moteur de recherche de base de données MS pour analyser les acquisitions de DDA et identifier les protéines. Générez une méthode de moteur de recherche darabase comme suit :
- Pour Paramètres de description de l’échantillon, sélectionnez Identification ; sous Type d’échantillon, sélectionnez Acide iodoacétique ; sous Alkylation de cystéine, sélectionnez Trypsine ; sous Digestion (en supposant un clivage C-terminal au niveau de la lysine et de l’arginine), sélectionnez le nom du spectromètre de masse sous Instrument ; et sélectionnez Caenorhabditis elegans sous Espèces.
- Pour les paramètres de traitement spécifiques, cochez Modifications biologiques ; sous ID Focus, sélectionnez SwissProt ; sous Base de données, cochez Identification approfondie ; sous Activités de recherche, sélectionnez 0,05 (10 %) sous Seuil de protéines détectées ; et cochez Exécuter l’analyse du taux de découverte erronée sous Qualité des résultats. Enregistrez la méthode du moteur de recherche et soumettez les fichiers bruts MS au moteur de recherche de la base de données pour traitement, à l’aide de la méthode générée.
REMARQUE : Dans un processus itératif, tous les scans MS et MS/MS ont été automatiquement recalibrés par le moteur de recherche en fonction des annotations et des résultats initiaux.
- Cliquez sur Exporter le résumé des peptides à la fin de la recherche et filtrez tous les résultats d’identification des peptides par le seuil de confiance de 99 dans Excel (taux de fausses découvertes (FDR) = 1 %).
- Créez des bibliothèques spectrales MS/MS pour le traitement ultérieur du fichier de données brutes DIA et pour la quantification ultérieure des données relatives.
- Ouvrez le logiciel d’analyse quantitative DIA. Sélectionnez l’onglet Bibliothèque , puis (en bas de la page) cliquez sur Générer une bibliothèque spectrale à partir du « Moteur de recherche de base de données » et ouvrez un rapport FDR « Moteur de recherche de base de données » (le fichier de feuille de calcul *FDR.xlsx) qui a été généré automatiquement dans le cadre du processus de recherche dans la base de données du fichier de données brutes DDA.
- Cliquez sur Suivant, sélectionnez le schéma des paramètres de la bibliothèque , puis cliquez sur Suivant. Sélectionnez Uniprot_Caenorhabditis elegans _proteome comme base de données puis cliquez sur Terminer, la bibliothèque spectrale sera générée.
- Utilisez le logiciel d’analyse quantitative DIA pour analyser les acquisitions DIA afin d’obtenir une quantification complète des protéines relatives. Le pipeline d’analyse de données DIA est décrit comme suit.
- Pour analyser et quantifier des peptides, ouvrez le logiciel d’analyse quantitative DIA pour configurer un schéma d’analyse à l’aide du schéma de modèle. Le schéma du modèle est disponible dans le logiciel en cliquant sur Paramètres, puis sur Analyse DIA, et enfin sur Paramètres d’usine BGS.
- Configurez les paramètres dans le schéma de modèle comme suit :
1) sous identification , sélectionnez Localisation PTM (seuil de probabilité = 0,75).
2) sous Quantification, sélectionnez Quantité du groupe principal comme Quantité peptidique totale, configurez le groupe principal N comme Max 7 et Min 1, sélectionnez Quantité du groupe secondaire comme Quantité de précurseur de somme, et configurez le groupe secondaire N comme Max 10 et Min 1, et sélectionnez Filtrage des données comme Qvalue clairsemée et ne sélectionnez pas Normalisation entre les exécutions.
- Effectuez une analyse de quantification relative.
- Sélectionnez l’onglet Pipeline , cliquez sur Configurer une analyse DIA à partir d’un fichier, ouvrez les fichiers bruts MS DIA qui vous intéressent (par exemple : échantillon et contrôle), cliquez sur Attribuer une bibliothèque spectrale, sélectionnez la bibliothèque générée dans la version 6.3, cliquez sur Charger, puis enfin sur Suivant.
- Sélectionnez le schéma d’analyse configuré dans la version 6.5.1 et cliquez sur Suivant. Sélectionnez le fichier fasta de base de données approprié (dans ce protocole : Uniprot_Caenorhabditis elegans_proteome) et cliquez sur Suivant. Définissez la configuration des conditions et attribuez les différentes conditions en fonction des échantillons, puis cliquez sur Suivant.
- Passez en revue la vue d’ensemble de l’analyse (résumé de la configuration de l’expérience), accédez au répertoire de sortie , puis cliquez sur Terminer. Enfin, cliquez sur Exécuter le pipeline pour effectuer l’analyse quantitative sans étiquette.
REMARQUE : Les modules statistiques du logiciel d’analyse quantitative DIA effectuent automatiquement l’analyse FDR, génèrent des cartes thermiques et des graphiques volcaniques comparant les différentes conditions, génèrent des listes de peptides et de protéines identifiés et quantifiés et fournissent des valeurs Q ainsi que des changements de pliage relatifs comparant différentes conditions. Ici, les conditions d’échantillonnage mises en place comprenaient des vers âgés du jour 10 avec deux répétitions biologiques et des jeunes vers du jour 2 avec deux réplicats biologiques comme témoin.
Résultats
Les méthodes traditionnelles de lyse par vis sans fin présentent divers inconvénients. Par exemple, les méthodes de sonication et de battage de billes basées sur des sondes produisent une chaleur excessive en permettant le contact de la pointe métallique ou des billes directement avec les échantillons, ce qui entraîne des récupérations variables des protéines et une dénaturation des protéines. Le broyage à l’azote liquide suivi d’une sonication dans un tampon de lyse, peut prendre beaucoup de temps et nécessite un grand nombre de vers. En raison des limites des méthodes traditionnelles de lyse des vers, les flux de travail MS précédents, tels que les méthodes d’étiquetage iTRAQ ou les méthodes sans marquage qui ont été historiquement utilisées dans le système de modèle C . elegans pour obtenir des informations quantitatives sur l’insolublome, nécessitent un apport important de matériel de départ (au moins 40 000 vers). Un travail laborieux d’élevage de vers est nécessaire pour obtenir ce nombre de vers. De plus, les méthodes d’étiquetage nécessitent des étiquettes chimiques isobares coûteuses. Les méthodes de quantification sans marquage sont rentables et offrent des méthodes de préparation et de marquage des échantillons plus faciles et plus simples, mais nécessitent un nombre considérablement élevé de vers pour obtenir une couverture d’analyse MS suffisante.
Le sonicateur que nous avons utilisé augmente considérablement l’efficacité et la reproductibilité de la lyse des vers en lysant simultanément plusieurs échantillons de vers dans un sonicateur à bain d’eau à température contrôlée sans contamination croisée14, réduisant ainsi considérablement la quantité de matériau de vis sans fin de départ nécessaire. En combinant la méthode de sonication très efficace et l’approche quantitative de la MS sans marquage DIA, nous avons pu quantifier de manière robuste l’insolublme des vers âgés et jeunes en utilisant ~3 000 vers. Ici, nous avons testé et validé l’efficacité du protocole et comparé l’insolublome de vers âgés et jeunes d’une souche de ver de type sauvage, N2-Bristol C. elegans. Nous avons appliqué ce protocole pour extraire et isoler l’insolublome de ~3 000 N2 C. elegans âgés et jeunes (deux réplicats biologiques pour chaque condition), suivi d’une analyse MS avec un spectromètre de masse quadripolaire à temps de vol ou d’autres systèmes MS utilisant une combinaison d’acquisition dépendante des données (DDA) et d’acquisitions indépendantes des données (DIA/SWATH) pour l’identification et la quantification des protéines. Les protéines insolubles ont d’abord été analysées sur un gel Bis-Tris à gradient de 4 à 12 % afin de déterminer la quantité de protéines dans chaque échantillon insolublome. Comme le montre la figure 2, l’échantillon d’insolublome des vers âgés N2 (voies 2 et 3, expériences de réplication biologique) contient significativement plus de protéines que les échantillons de jeunes vers N2 (voies 1 et 4, expériences de réplication biologique).
Après la digestion dans le gel, les profils protéiques de l’insolublome ont été analysés par HPLC-MS. À l’aide de ce flux de travail, nous pouvons généralement identifier 1000 à 1500 protéines et quantifier 500 à 1 000 protéines de la fraction insoluble SDS avec une reproductibilité élevée (données non publiées). Ici, nous avons pu quantifier 989 protéines de l’insolublome de N2-Bristol C. elegans en analysant les données DIA et en supprimant la redondance : 768 protéines ont été significativement enrichies et 27 protéines ont été significativement diminuées dans l’insolublome du ver N2 âgé (jour 10) par rapport au jeune (jour 2) en utilisant un changement de pli d’au moins 1,5 et une valeur Q inférieure à 0,01 (Figure 3A). Comme le montre le graphique de l’histogramme (figure 3B), le changement de pliage des protéines significativement modifiées montre une distribution normale. Il a été démontré que les vers âgés étaient significativement enrichis pour l’insolublome : le plus grand changement observé a montré que l’abondance relative des protéines chez l’insolublome était 592 fois plus élevée chez les vers âgés que chez les jeunes vers ; Et pour 32 protéines, l’abondance relative des protéines dans l’insolublome était >250 fois plus élevée chez les vers âgés que chez les jeunes vers, indiquant des changements spectaculaires de l’insolublome avec l’âge.
Après avoir extrait la liste des protéines insolubles qui sont significativement augmentées chez les vers âgés et identifiées par la base de vers (WS271), l’analyse de la voie KEGG et de l’ontologie génétique (GO) a été effectuée pour déterminer les voies qui sont enrichies dans l’insolublome âgé afin d’obtenir des informations biologiques sur la façon dont elles sont liées au vieillissement. L’analyse de la voie KEGG des protéines identifiées dans cette étude montre un enrichissement de plusieurs voies impliquant les ribosomes, les mitochondries, le protéasome et l’épissage (Figure 4A). L’analyse de l’ontologie génétique montre que l’insolublome des vers âgés comprend de nombreuses protéines dans des catégories particulières, notamment les protéines mitochondriales, développementales, déterminantes de la durée de vie adulte et ribosomales (figure 4B et tableau supplémentaire 1A). Nous avons ensuite comparé la liste des protéines identifiées dans cette étude avec les travaux précédemment publiés de David et al.2 et Mark et al.11 , comme le démontrent les diagrammes de Venn (Figure 5, A, 5B). La comparaison a montré un chevauchement significatif des protéines identifiées 394/721 et 444/721 avec l’étude de David et al (Figure 5A) et de Mark et al. (Figure 5B), respectivement. Les voies biologiques révélées par l’analyse KEGG de l’insolublome à partir de cette étude ont également été identifiées dans le passé, validant ainsi notre méthodologie (tableau supplémentaire 1B). L’identification de ces voies et protéines suggère qu’elles pourraient servir de candidats pour une étude biologique plus approfondie en ce qui concerne leur fonction dans le contexte du vieillissement.
En résumé, l’utilisation de la méthode de sonication efficace permet la lyse de plusieurs échantillons de vers en même temps dans un environnement avec des températures bien contrôlées et une contamination croisée réduite pour obtenir une couverture protéique élevée avec beaucoup moins de matière de vers de départ. La combinaison de la méthode de sonication efficace avec un flux de travail de quantification des protéines sans marquage DIA a permis d’obtenir des résultats fiables et reproductibles pour la quantification des protéines de ver insolubles.

Graphique 1. Flux de travail expérimental du protocole. C. elegans a été cultivé et récolté à des jours différents. Après lyse par lombricompost avec un sonicateur, la fraction protéique insoluble à 1 % du SDS (insolublome) a été extraite et isolée du lysat. L’insolublome a ensuite été digéré par digestion à la trypsine dans le gel et quantifié par spectrométrie de masse DIA, suivi d’une analyse bioinformatique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
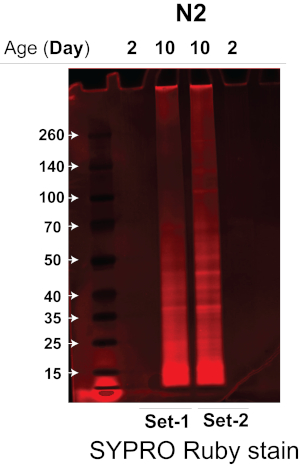
Graphique 2. Le gel SDS-PAGE de l’insolublome isole les jeunes vers par rapport aux vieux vers de la souche N2-Bristol. Les insolublomes des jeunes vers par rapport aux vers âgés de la souche N2-Bristol ont été analysés par SDS-PAGE pour déterminer la quantité de protéines présentes. Le gel SDS-PAGE a été coloré avec une coloration protéique fluorescente pour visualiser les bandes de protéines. Couloirs 1 et 4 : Insolublome issu de deux expériences de réplication biologique de jeunes vers N2 (Jour 2). Couloirs 2 et 3 : Insolublome issu de deux expériences de réplication biologique de vers N2 âgés (Jour 10). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 3. Les protéines candidates identifiées comme montrant une altération significative chez l’insolublome âgé par rapport au jeune et leurs distributions de changement de plissement. (A) Graphique volcanique pour la quantification de l’insolublème des vers N2-Bristol âgés par rapport aux jeunes vers. Les candidats avec un changement de pli absolu >=1,5 et une valeur Q <0,01 sont représentés par des points rouges. (B) Graphique d’histogramme pour la distribution des changements de pli des protéines insolubles SDS significativement enrichies dans des échantillons de vers âgés par rapport à de jeunes vers. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 4. Analyse du parcours KEGG et de l’ontologie génétique (GO). (A) Analyse de la voie KEGG de l’insolublome du jour 10 disposé en fonction de la valeur p avec une voie très significative indiquée en haut. (B) L’analyse de l’ontologie génétique montre que l’insolublome des vers âgés est enrichi pour de nombreuses protéines dans des catégories particulières, notamment les protéines mitochondriales, développementales, déterminantes de la durée de vie adulte et ribosomiales. La vue en nuage de points visualise les termes GO dans un « espace sémantique » où les termes les plus similaires sont positionnés plus près les uns des autres. La couleur de la bulle reflète la valeur p obtenue dans l’analyse STRING, tandis que sa taille reflète la généralité du terme GO dans la base de données UniProt-GOA. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 5. Le chevauchement de la protéine insolublome identifié dans l’insolublome du jour 10 compare cette étude avec (A) David et al.2 et (B) les études Mark et al.11 . Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau supplémentaire 1 (en lien avec les figures 4 et 5). (A) Ontologie génétique (processus biologique) analysée avec la base de données STRING. (B) Liste détaillée des protéines et des voies KEGG identifiées dans cette étude avec des codes de couleur décrivant leur chevauchement avec les travaux publiés. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Dans ce protocole, nous rapportons une méthode améliorée de préparation d’échantillons pour l’extraction de protéines insolubles de C. elegans. En remplaçant la lyse par vis sans fin traditionnelle (par exemple, les techniques de sonication par sonde ou de batteur de billes) par un sonicateur efficace, nous avons augmenté le rendement de l’extraction des protéines insolubles et réduit le nombre de vers nécessaires pour l’analyse MS sans marquage de 40 000 vers à 3 000 vers. Un moteur de recherche dans les bases de données a été utilisé pour l’identification des protéines à partir des données DDA et la bibliothèque spectrale de C. elegans a été construite à l’aide du « logiciel d’analyse quantitative DIA » et des résultats de recherche correspondants dans les bases de données DDA (importation des rapports de fichiers FDR générés par le moteur de recherche dans les bases de données). La quantification relative des données insolubles de C. elegans âgés et jeunes a été réalisée à l’aide du « logiciel d’analyse quantitative DIA » pour traiter le nouvel ensemble de données DIA et la bibliothèque spectrale générée.
Plusieurs étapes sont essentielles dans le protocole. La courte durée de vie de C. elegans en fait un système idéal pour étudier le vieillissement, par rapport à d’autres eucaryotes tels que les cellules de mammifères, mais il est crucial d’isoler une population homogène de vers lors de l’étude des phénomènes liés au vieillissement. FUDR a été utilisé dans ce protocole pour obtenir des vers vieillissants synchronisés. Il est important de transférer les vers au début de leur stade L4 sur la plaque ensemencée par NGM contenant le FUDR pour garantir son efficacité. Lors de la lyse à vis à l’aide du sonicateur efficace, la température du bain-marie doit être réglée à 4 °C et la sonication à 30 s ON et 30 s OFF pour éviter la surchauffe des échantillons. Après la première série de sonications pendant 10 cycles (10 min), il est important de vérifier au microscope pour s’assurer que tous les vers ont été lysés efficacement. Si ce n’est pas le cas, d’autres cycles de sonication sont nécessaires. Au cours du processus de digestion dans le gel, chaque tranche de gel doit être coupée en morceaux de la bonne taille (<1 mm2) - si elle est trop petite, elle peut être perdue dans le processus de préparation de l’échantillon, et si elle est trop grande, la digestion peut être inadéquate.
Le besoin de beaucoup moins de matériel de départ réduit considérablement le travail laborieux associé à la culture des vers pour obtenir des échantillons pour les analyses d’insolubolome. Cependant, l’extraction et l’isolement de la fraction protéique insoluble à 1 % de SDS impliquent plusieurs étapes de lavage et une manipulation soigneuse de l’échantillon est nécessaire pour éviter la perte d’échantillon et garantir des résultats reproductibles. La quantité de matériel générée pour l’analyse MS est suffisante pour ~3 injections pour les analyses ultérieures DDA et DIA, mais pas pour les expériences futures. De plus, malgré ses effets potentiellement confondants15, nous avons utilisé la plus faible concentration possible de FUdR pour stériliser les vers pendant le processus de vieillissement. Des études futures pourraient contourner l’utilisation du FUdR en utilisant des mutants stériles ou en transférant et en collectant manuellement des vers.
L’utilisation du sonicateur très efficace pour la lyse des vers permet une extraction efficace de l’insolublome, ce qui permet une bonne couverture protéique et une analyse DIA MS sans marquage rentable pour quantifier l’insolublome à l’aide d’un nombre considérablement réduit de vers. Il réduit considérablement la charge de travail, ce qui permet de dépister davantage de conditions par expérience. De plus, le flux de travail MS DIA sans marquage est rentable et offre une profondeur et une couverture protéiques à des niveaux comparables à ceux des méthodes de marquage telles que iTRAQ, TMT ou SILAC. Le modèle C. elegans est un système de dépistage rapide pour la recherche sur le vieillissement. Le flux de travail peut être facilement modifié et appliqué pour étudier le vieillissement et la recherche sur les maladies liées à l’âge dans cet organisme et dans d’autres. Par exemple, dans le cadre d’études en cours, nous appliquons ce flux de travail pour étudier les profils protéiques de l’insolublome et de la protéostasie dans divers modèles de C. elegans de la maladie d’Alzheimer (MA), y compris les vers Abeta, tau et les vers doubles Abeta/tau avec ou sans différentes interventions médicamenteuses pour le dépistage futur de médicaments à haut débit.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par une subvention d’instrumentation partagée des NIH pour un système TripleTOF (1S10 OD016281, Buck Institute), une subvention NIH, RF1 AG057358 (GJL, JKA) et une subvention NIH U01AG045844 (GJL). XX est soutenu par une bourse postdoctorale T32 (subvention NIH 5T32AG000266, PI : Judith Campisi et Lisa Ellerby). MC est soutenu par une bourse postdoctorale de la Fondation Larry L. Hillblom.
matériels
| Name | Company | Catalog Number | Comments |
| Strains used | |||
| Esherichia coli OP50 | Caenorhabditis Genetics Center (CGC) | ||
| N2 (Bristol) | Caenorhabditis Genetics Center (CGC) | ||
| Buffer/Solution | |||
| NGM (Nematode Growth Media) | Recipe: 3 g/L NaCl, 23 g/L agar; 2.5 g/L peptone; 1 mM CaCl2, 5 mg/L cholesterol, 1 mM MgSO4, 25 mM KH2PO4 | ||
| S-basal solution | Recipe: 5.85 g/L NaCl, 1g/L K2HPO4, 6 g/L KH2PO4, H2O to 1 L | ||
| Sodium hypochlorite bleach solution | Recipe: Mix 0.5 mL 5 N NaOH with 1 ml Sodium hypochlorite (5%) and make volume to 5 mL with H20. | ||
| Material/ Equipment | |||
| Agar | Difco Granulated Agar, BD Biosciences | 90000-782 | |
| Bioruptor Plus sonication device | Diagenode, USA | B01020001 | |
| Cholesterol | Sigma | c8503 | |
| 2'-deoxy-5-fluorouridine | VWR | TCD2235 | |
| Glycerol | Millipore Sigma | 356350-1000ML | |
| LB broth, Miller | Millipore Sigma | 60801-450 | |
| Sodium dodecyl sulfate (SDS? | Sigma | L4509-250G | |
| Sodium chloride | Sigma | 59888 | |
| M880 Ultrasonic bath, 117 V, holds 5.5 gallons | VWR, USA | 89375-458 | |
| Magnesium sulphate | Sigma | M506 | |
| Magnesium chloride | Sigma | 208337 | |
| NGM agar plate | VWR Disposable Petri Dishes | 25384-342 | |
| NuPAGE LDS Sample Buffer (4X) | Thermo Fisher Scientific | NP0007 | |
| NuPAGE protein gels, 4-12% | Invitrogen | NP 0335BOX | |
| Protease inhibiotr cocktail (PIC) | Roche | 11836170001 | |
| Pierce BCA Assay | Thermo Fisher Scientific | 23225 | |
| Sodium hypochlorite 5% | VWR | JT9416-1 | |
| SYPRO Ruby Protein Gel Stain | Thermo Fisher Scientific | S12000 | |
| MS Section | |||
| Acetonitrile, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 36XL66 | |
| Agilent Zorbax 300Extend C18 column | Agilent Technologies Inc., Santa Clara, CA, USA | 770995-902 | |
| Ammonium bicarbonate | Sigma Aldrich, St. Louis, MO, USA | 9830 (1 kg) | |
| Dithiothreitol (DTT) | Sigma Aldrich, St. Louis, MO, USA | D9779-5G | |
| Eppendorf Thermomixer Compact | Eppendorf AG, Hamburg, Germany | T1317-1EA | |
| Formic acid | Sigma Aldrich, St. Louis, MO, USA | F0507-500ML | |
| Indexed retention time (iRT) normalization peptide standard | Biognosys AG, Schlieren, Zurich, Switzerland | Ki-3002-2 | |
| Iodoacetamide (IAA) | Sigma Aldrich, St. Louis, MO, USA | I1149-25G | |
| Methanol, HPLC Grade | Honeywell International Inc., Charlotte, NC, USA | 34885 | |
| Nano cHiPLC Trap ChromXP C18-CL, 200 um x 6 mm, 3 um, 120A. (pre-column chip) (200 um x 6 mm ChromXP C18-CL chip, 3 um, 300 A) | Sciex LLC, Framingham, MA, USA | 804-00006 | |
| Nano cHiPLC ChromXP 75 um by 15cm, C18-CL, 3 um, 120 A (analytical column chip) | Sciex LLC, Framingham, MA, USA | 804-00001 | |
| Orthoganol quadrupole time-of-flight (QqTOF) TripleTOP 6600 mass spectrometer | Sciex LLC, Framingham, MA, USA | Per quote | |
| ProteinPilot 5.0 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Savant SPD131DDA Speedvac Concentrator | Thermo Fisher Scientific, Waltham, MA, USA | SPD131DDA-115 | |
| Sequencing-grade lyophilized trypsin | Life Technologies | 23225 | |
| Spectronaut | Biognosys AG, Schlieren, Zurich, Switzerland | Sw-3001 | |
| SWATH 2.0 plugin into PeakView 2.2 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Ultra Plus nano-LC 2D HPLC system | Sciex LLC, Eksigent Division, Framingham, MA, USA | Model # 845 | |
| Water, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 600-30-76 | |
| Waters 1525 binary HPLC pump system | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters 2487 Dual Wavelength UV detector | Waters Corp., Milford, MA, USA | WAT081110 | |
| Waters 717plus Autosampler | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters Fraction Collector III | Waters Corp., Milford, MA, USA | 186001878 |
Références
- Walther, D. M., et al. Widespread Proteome Remodeling and Aggregation in Aging C. elegans. Cell. , 919-932 (2015).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Labbadia, J., Morimoto, R. I. The Biology of Proteostasis in Aging and Disease. Annual Review of Biochemistry. (1), 435-464 (2013).
- Ross, C. A., Poirier, M. A. Protein aggregation and neurodegenerative disease. Nature Medicine. , 10-17 (2004).
- Reis-Rodrigues, P., et al. Proteomic analysis of age-dependent changes in protein solubility identifies genes that modulate lifespan. Aging Cell. 11 (1), 120-127 (2012).
- Ayyadevara, S., et al. Proteins that accumulate with age in human skeletal-muscle aggregates contribute to declines in muscle mass and function in Caenorhabditis elegans. Aging. 8 (12), 3486-3497 (2016).
- Huang, C., et al. Intrinsically aggregation-prone proteins form amyloid-like aggregates and contribute to tissue aging in Caenorhabditis elegans. eLife. 8, (2019).
- Morimoto, R. I. Proteotoxic stress and inducible chaperone networks in neurodegenerative disease and aging. Genes and Development. 22 (11), 1427-1438 (2008).
- Morimoto, R. I., Cuervo, A. M. Protein homeostasis and aging: Taking care of proteins from the cradle to the grave. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 64 (2), 167-170 (2009).
- Klang, I. M., et al. Iron promotes protein insolubility and aging in C. elegans. Aging. 6 (11), 975-991 (2014).
- Mark, K. A., et al. Vitamin D Promotes Protein Homeostasis and Longevity via the Stress Response Pathway Genes skn-1, ire-1, and xbp-1. Cell Reports. 17 (5), 1227-1237 (2016).
- Groh, N., et al. Methods to study changes in inherent protein aggregation with age in caenorhabditis elegans. Journal of Visualized Experiments. (129), 1-12 (2017).
- Schilling, B., Gibson, B. W., Hunter, C. L. Generation of High-Quality SWATH((R)) Acquisition Data for Label-free Quantitative Proteomics Studies Using TripleTOF((R)) Mass Spectrometers. Proteomics: Methods and Protocols, Methods in Molecular Biology. 1550, 223-233 (2017).
- Walther, D. M., et al. Widespread proteome remodeling and aggregation in aging C. elegans. Cell. 161 (4), 919-932 (2015).
- Angeli, S., et al. A DNA synthesis inhibitor is protective against proteotoxic stressors via modulation of fertility pathways in Caenorhabditis elegans. Aging. 5 (10), 759-769 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon