Method Article
Quantification of Insoluble Protein Aggregation in Caenorhabditis elegans during Aging with a Novel Data-Independent Acquisition Workflow (새로운 데이터 독립적 수집 워크플로우를 통한 노화 중 Caenorhabditis elegans의 불용성 단백질 응집의 정량화)
요약
이 새로운 워크플로우는 정량적 차등 단백질체학 분석을 위해 최소한의 출발 물질로 Caenorhabditis elegans 에서 SDS-불용성 단백질(불용성)을 효율적으로 추출하고 분리합니다. 이 프로토콜은 포괄적인 데이터 독립적 수집 질량 분석 분석을 사용하여 불용성 및 생물정보학 분석을 정량화하여 노화 메커니즘 및 병리학에 대한 생물학적 통찰력을 얻습니다.
초록
우리와 다른 사람들은 노화 과정이 단백질 전체에 불용성 단백질을 축적하는 결과를 낳는다는 것을 보여주었습니다. 불용성 단백질을 암호화하는 유전자를 40% 이상 제거하면 예쁜꼬마선충의 수명이 연장되며, 이는 이러한 단백질 중 많은 수가 노화 과정의 주요 결정 요인임을 시사합니다. 이러한 불용성 단백질의 분리 및 정량적 식별은 노화 중에 발생하는 주요 생물학적 과정을 이해하는 데 중요합니다. 여기에서는 예쁜꼬마선충에서 SDS-불용성 단백질(불용성)을 보다 효율적으로 추출하고 분리하여 새로운 label-free 정량적 단백질체학 분석을 통해 질량 분석 워크플로우를 간소화하는 방법을 자세히 설명하는 수정 및 개선된 프로토콜을 제시합니다. 이 개선된 프로토콜은 웜 용해를 위한 고효율 초음파 발생기를 사용하여 단백질 추출의 효율성을 크게 높이고 이전 프로토콜(일반적으로 최소 40,000개의 웜 사용)보다 훨씬 적은 시작 물질(약 3,000개의 웜)을 사용할 수 있습니다. 단백질 발견 및 식별을 위한 데이터 의존적 수집(DDA)과 포괄적이고 정확한 단백질 정량화를 위한 데이터 독립적 수집(DIA)을 사용하여 불용용체에 대한 후속 정량적 단백질체학 분석을 수행했습니다. 정량화된 단백질의 생물정보학적 분석은 예쁜꼬마선충의 다른 분자 방법으로 쉽게 추적할 수 있는 잠재적 후보를 제공합니다. 이 워크플로우를 통해 당사는 일상적으로 1,000개 이상의 단백질을 식별하고 500개 이상의 단백질을 정량화합니다. 이 새로운 프로토콜은 예쁜꼬마선충(C. elegans)을 이용한 효율적인 화합물 스크리닝을 가능하게 합니다. 여기서, 우리는 이 개선된 프로토콜을 야생형 예쁜꼬마선충 N2-Bristol 균주에 검증하고 적용했으며, 10일째 N2 기생충이 2일째 어린 기생충보다 불용성체가 더 많이 축적됨을 확인했습니다.
서문
단백질 항상성은 노화에 따라 점진적으로 감소하며 단백질 응집이 증가합니다 1,2,3. 단백질 응집은 알츠하이머병, 파킨슨병, 헌팅턴병, 근위축성 측삭 경화증을 포함한 여러 신경퇴행성 질환과 관련이 있다4. 노화는 단백질 응집과 관련된 신경퇴행성 질환 발병의 주요 위험 요인으로 간주됩니다. 불용성 응집체를 형성하기 쉬운 단백질은 종종 세포 독성 및 조직 기능 장애와 관련이 있으며, 이는 다른 단백질의 응집을 더욱 가속화 할 수 있습니다 5,6,7. 또는 불용성 단백질 응집체는 세포 방어 메커니즘을 활성화하여 단백질의 독성 올리고머 형태를 시스템에서 제거할 수 있습니다. 불용성 단백질을 암호화하는 선별된 유전자를 knockdown하면 노화 관련 질병과 정상적인 노화의 맥락에서 Caenorhabditis elegans(C. elegans)의 수명이 조절됩니다 5,8,9. 따라서 단백질 응집의 세포 및 분자 메커니즘을 연구하는 것은 노화를 이해하는 데 중요하며 궁극적으로 신경 퇴행성 질환을 치료하기 위한 접근 방식으로 이어질 수 있습니다.
예쁜꼬마선충(C. elegans)은 상대적으로 짧은 수명(약 2주), 재배 용이성 및 유전자 조작과 같은 독특한 특성으로 인해 노화 및 노화 관련 질병에서 단백질 응집을 연구하는 데 가장 광범위하게 사용되는 모델 유기체 중 하나가 되었습니다.
불용성 단백질을 추출하고 특성화하는 능력은 예쁜꼬마선충(C. elegans) 모델에서 단백질 응집과 관련된 노화 관련 변화를 결정하는 데 중요한 역할을 했습니다. 정상적인 노화 과정에 대한 단백질 응집의 기여도를 조사하기 위해 우리5 및 기타2는 이전에 어린 예쁜꼬마선충과 늙은 예쁜꼬마선충의 불용성을 추출하고 단백질 분해로 소화한 후 iTRAQ 시약('상대 및 절대 정량을 위한 등압 태깅')을 사용하여 화학적으로 라벨링한 다음 MS 기반 방법을 사용하여 정량화했습니다. 등압 표지 방법과 120mg의 습식 지렁이(약 40,000마리의 지렁이)를 사용하여 상당한 단백질 불용성 깊이와 적용 범위를 얻을 수 있었습니다5. 정량적 분석에 따르면 확인된 1,200개의 단백질 중 203개가 어린 벌레의 유사한 불용성 분획과 비교하여 노화된 예쁜꼬마선충의 불용성체에서 유의하게 농축되어 있음을 보여주었습니다5. 또한 David와 동료들은 독립적으로 iTRAQ LC-MS/MS 워크플로우를 활용하여 정상적인 노화2로 인한 단백질 응집체의 변화를 조사했습니다. 약 300mg의 기생충으로 시작하여 두 가지 생물학적 복제물을 사용하여 ~1000개의 불용성 단백질을 확인하고 약 1000개의 단백질 중 ~700개가 어린 기생충에 비해 나이가 들면서 1.5배 이상 축적된다는 것을 확인했습니다2. 전반적으로, 이러한 독립적인 결과는 광범위한 단백질 불용성 및 응집이 정상적인 노화의 고유한 부분이며 수명과 신경퇴행성 질환의 발병률 모두에 영향을 미칠 수 있음을 나타냅니다 2,5.
불용성체를 연구함으로써 우리는 환경적 영향이 어떻게 노화 과정을 가속화하거나 늦출 수 있는지 결정할 수 있었다. Klang 등은 장수에서 금속로소의 역할을 조사하기 위해 예쁜꼬마선충(C. elegans)에서 무표지 단백질체학 워크플로우를 확립했습니다10. 이 연구에서는 불용성10을 추출하기 위해 최소 40,000마리의 벌레가 사용되었습니다. 데이터에 따르면 철, 구리, 칼슘 및 망간 수치는 노화에 따라 증가하며, 지렁이에게 철분이 많이 함유된 음식을 먹이면 노화와 관련된 불용성 단백질의 축적이 크게 가속화되는 것으로 나타났습니다10. 예쁜꼬마선충의 불용성에 대한 비타민 D의 효과를 조사하기 위해 동일한 워크플로우를 사용하여 어린 벌레에서 38개의 단백질(2일차)과 노화된 벌레에서 721개의 단백질(8일차)을 정량화했습니다. 비타민 D를 섭취하면 노화된 기생충의 불용성체(insolblome)가 721개에서 371개로 현저히 감소했다11. 추가 조사에 따르면 비타민 D를 먹이면 나이가 들면서 단백질 불용성이 억제되고 단백질 항상성이 촉진되며 예쁜꼬마선충 N2 야생형 벌레의 수명이 연장되는 것으로 나타났습니다11. 따라서 불용성체를 연구하면 노화 및 노화 관련 질병의 새로운 조절인자를 식별하는 데 도움이 될 수 있습니다.
불용성체를 연구하는 것은 노화 과정에 대한 이해를 진전시키는 데 매우 중요하지만, 많은 양의 시작 샘플 물질을 수집해야 하는 요구 사항으로 인해 방해를 받았습니다. Groh 등은 최근 노화에 따른 예쁜꼬마선충의 고유한 단백질 응집 변화를 연구하기 위해 표지가 없는 단백질체 정량화 워크플로우를 도입했습니다. 그러나 많은 양의 출발 물질(350mg의 지렁이)이 필요했습니다12. 본 보고서에서는 개선된 새로운 추출 및 분리 프로토콜을 수립했습니다(그림 1). 웜 용해 중 고효율 초음파 발생기를 사용하면 추출 효율성이 크게 향상되어 필요한 시작 물질의 양이 40,000개에서 3,000개로 감소했습니다. 이 새로운 불용성 분리 프로토콜과 무표지 데이터 독립적 수집(DIA) 질량 분석 워크플로우를 결합하여 단백질 깊이와 적용 범위를 크게 개선했습니다. 여기에 제시된 프로토콜은 비용 효율적이며 다른 모델 시스템에서 불용성 분석을 수행할 수 있도록 쉽게 수정할 수 있습니다.
프로토콜
참고: 실험 절차에 대한 더 나은 이해를 위해 그림 1 에서 워크플로우의 개략도를 참조하십시오.
1. 동기화된 노화 C. elegans의 대량 문화
- 접시 준비
- 각 90mm 플레이트에 50mM 플루오로데옥시우리딘(FUdR)이 있거나 없는 NGM 한천 30mL를 붓고 배양 후드에서 플레이트를 2일 동안 건조시킵니다.
- NGM 한천 플레이트를 파종하기 전날, OP50 박테리아 균주의 동결 글리세롤 스톡 50μL를 2L 플라스크에 있는 1L의 사전 오토클레이브 LB 배지에 접종합니다.
- 37°C의 쉐이킹 인큐베이터에서 250rpm으로 16시간 동안 박테리아를 성장시킵니다.
- 16시간 후 10분 동안 5,000 x g 의 속도로 박테리아 배양물을 원심분리합니다.
- 상층액을 버리고 40mL의 오토클레이브 S-염기 용액(물 내 50mMK,PO4, 100mM NaCl 함유)에 박테리아 펠릿을 완전히 재현탁시켜 25배 농축된 OP50 E. coli 배양을 생성합니다.
- 25x 농축된 박테리아 배양액 2mL를 각 NGM 한천 플레이트에 분주하고 박테리아 배양액이 전체 한천 표면을 덮도록 고르게 펴 바릅니다.
- 플레이트가 제대로 건조될 때까지 2-3시간 동안 박테리아 배양 후드에서 플레이트의 뚜껑을 제거합니다.
알림: 15분마다 플레이트를 소용돌이쳐 박테리아가 고르게 분포되도록 하십시오. 박테리아가 플레이트의 한쪽 면에 집중되는 경향이 있어 박테리아 밀도가 낮은 영역이 생성되기 때문에 이는 중요합니다. - 플레이트가 건조된 후 뚜껑을 닫고 플레이트를 실온에서 48시간 동안 그대로 두어 OP50 E. coli 식품 공급원이 완전히 건조되도록 합니다.
참고: 플레이트는 2주 전에 준비하고 사용하기 전에 4°C에서 보관할 수 있습니다.
- C. elegans의 동기화된 코호트의 유지 및 준비
- SDS-불용성 분획의 분리에 필요한 단백질 용해물(1.0–1.5 mg)의 투입을 얻으려면 샘플당 약 3,000개의 동기화된 웜을 사용하십시오.
- ~100 N2 야생형 그레이비드 성충을 NGM 25x OP50 E. coli-seeded 플레이트로 옮기고 플레이트가 그레이비드 성충으로 가득 찰 때까지 20°C에서 ~72시간 동안 유지합니다.
- 각 플레이트에 ~10mL의 S-기저 용액을 피펫팅하고 모든 웜을 15mL 튜브에 모읍니다.
알림: 팁 표면에 부착되어 기생충이 손실되는 것을 방지하기 위해 기생충 수집과 관련된 이 단계 및 향후 모든 단계에서 저유지량 팁 또는 파스퇴르 피펫을 사용하는 것이 중요합니다. - 520 x g 에서 30초 동안 샘플을 회전시킵니다. 회전 후 상등액을 제거하십시오. OP50 E. coli를 제거하려면 펠릿에 10mL의 S-basal 용액을 첨가하고 다시 회전시킨 다음 상층액을 제거합니다. 이 단계를 반복합니다.
- 15mL 원추형 튜브의 웜 펠릿에 차아염소산나트륨 표백액 10mL(물에 0.5M KOH 및 0.48% 차아염소산나트륨 함유)를 넣고 실온에서 2분 동안 격렬하게 소용돌이칩니다.
- 실온에서 520 x g 의 온도에서 30초 동안 원심분리기를 사용하여 불완전하게 용해된 벌레 몸체와 알을 회전시킵니다. 그런 다음 상층액을 제거합니다.
- 1.2.5 및 1.2.6 단계를 반복하여 모든 웜 본체를 완전히 제거하고 알을 방출합니다(확인하기 위해 현미경으로 튜브를 봅니다).
- 표백제 용액이 완전히 제거되도록 난자 펠릿을 10mL의 S-기저 용액으로 최소 4회 세척하십시오.
알림: 남아 있는 표백제는 알의 부화를 방해할 수 있으므로 표백제를 완전히 제거하는 것이 중요합니다. - 15mL 튜브에 5mL의 S-기저 배지에 난자를 재현탁시키고 20°C의 로터에 24시간 동안 넣어 알이 부화할 수 있도록 합니다.
- 24시간 후 10μL의 S-기저 유충 용액을 취하여 그 안에 있는 벌레의 수를 계산합니다. 웜 수의 복제를 위해 10μL의 S-기저 유충 용액의 개별 부분 표본을 3개 이상 사용합니다. 세 가지 유충 농도의 평균을 계산합니다. 평균 농도를 기준으로 웜 용액을 희석하여 용액 200μL당 ~3,000마리의 유충을 얻습니다.
알림: 유충을 방해하지 않고 방치하면 가라앉는 경향이 있고 유충 수에 영향을 줄 수 있으므로 거꾸로 튜브를 부드럽게 흔들어야 합니다. - 잘 섞어 200μL의 S-기저 유충 용액을 90mm NGM 박테리아 파종 플레이트 각각에 추가합니다.
참고: 특정 지점에서 박테리아가 고갈되는 것을 방지하기 위해 유충의 균일한 분포를 위해 플레이트 전체에 S-basal 유충 용액 한 방울을 추가합니다. - S-basal 용액을 건조시킨 다음 플레이트를 거꾸로 20°C 인큐베이터에 48시간 동안 놓습니다.
- 48시간 후, S-basal 용액의 플레이트에서 웜을 채취하여 50mM FUDR을 포함하는 새로운 NGM 시딩 플레이트로 옮기고 20°C에서 플레이트를 배양합니다.
참고: FUDR 효율성을 보장하기 위해 L4 단계 초기에 웜을 전송하는 것이 중요합니다. - 플레이트에 10mL의 S-basal 용액을 추가하고 용액을 15mL 튜브로 옮겨 48시간 후 2일차 성인 샘플을 수집합니다. 벌레가 중력에 의해 정착하도록 하고 S-기저 용액이 투명해진 후 제거합니다.
- 웜 펠릿을 2-3x S-basal 용액 5mL로 세척하여 부착된 박테리아를 제거합니다. S-basal 용액을 최대한 제거하고 웜 펠릿(15mL 튜브에 있음)을 드라이 아이스/에탄올 수조에서 얼립니다. 냉동 샘플은 -80 °C에서 보관됩니다.
- 10일째까지 격일로 FUDR이 포함된 새로운 NGM OP50 E. coli 플레이트로 샘플 웜을 옮깁니다. 1.2.14 및 1.2.15단계에 설명된 대로 웜을 수집하고 동결합니다.
참고: 이동 및 굴 파기로 인한 지렁이 수의 손실을 설명하기 위해 10일째 샘플에 대해 더 많은 수의 지렁이로 시작합니다.
2. 벌레에서 SDS 불용해성 조각의 적출
- 20mM Tris 염기, pH 7.4, 100mM NaCl, 1mM MgCl2 및 EDTA-free 프로테아제 억제제를 함유한 900μL의 얼음-저온 웜 용해 완충액 칵테일이 있는 상태에서 얼음 위에서 동결된 웜 펠릿을 해동합니다. 용해 완충액에서 웜 펠릿의 완전한 재현유를 보장하기 위해 샘플을 간략하게 와류화합니다.
- 15mL 튜브를 초음파 처리 수조에 넣고 고강도로 10 회 초음파 처리 주기 (각 주기에 대해 30초 켜기 및 30초 끄기)로 설정합니다. 최대 5회 동안 반복합니다(웜이 완전히 용해되었는지 확인).
- 차가운 방에서 3,000 x g 에서 4분 동안 용해물을 회전시킵니다. 원심분리 후 벌레 파편이 포함된 펠릿을 버리고 상등액을 얼음 위의 사전 냉각된 새 1.5mL 원심분리 튜브로 조심스럽게 옮깁니다.
- BCA 분석을 사용하여 단백질 농도를 정량화합니다. 일반적으로 약 1mg의 단백질을 함유하는 용해물 분취액을 사전 냉각된 새로운 1.5mL 원심분리 튜브로 옮깁니다.
- 단백질 용해물을 냉장실에서 20,000 x g 으로 15분 동안 원심분리합니다.
- 펠릿을 방해하지 않고 상층액을 새로운 1.5mL 원심분리 튜브로 옮기고 수용성 단백질 분획으로 저장합니다.
- 이전 단계의 펠릿을 실온에서 1% SDS가 포함된 500μL의 웜 용해 완충액으로 세척하고 실온에서 15분 동안 20,000 x g 로 원심분리합니다. 상층액을 제거하고 SDS 용해성 분획으로 저장합니다. 이 세척 단계를 두 번 반복하여 SDS 용해성 분획을 제거합니다. 3차 세척 후 남은 펠릿은 1% SDS-불용성 단백질 분획으로 정의됩니다.
- SDS-불용성 단백질 펠릿을 70% 포름산 60μL에 재현탁하고 격렬하게 소용돌이쳐 단백질을 용해시킵니다. 펠릿을 용해시키는 데 필요한 만큼 격렬한 소용돌이를 반복합니다.
- 펠릿을 실온의 초음파 수조에서 30분 동안 초음파 처리합니다.
참고: SDS- 불용성 펠릿은 일반적으로 완충액에 용해되기 어렵습니다. 그러나 결국 이 단계에서 전체 펠릿을 용해해야 합니다. - 진공 농축기에서 샘플을 1시간 동안 건조시켜 포름산 용액을 완전히 제거합니다.
- 건조된 펠릿에 40μL의 1x LDS 샘플 젤 버퍼를 추가하고 샘플을 95°C에서 10분 동안 가열합니다. 샘플을 간단히 소용돌이치고 스핀 다운합니다. 4-12% NUPAGE Bis-Tris 겔에 13μL를 로드하고 겔을 실행합니다. 이미징을 위해 형광 단백질 염색으로 젤을 염색합니다. 질량분석법(MS) 분석을 위해 샘플의 나머지 부분을 저장합니다.
- 나머지 샘플을 4-12% Bis-Tris 겔에 넣고 MS 인-겔 분해를 위해 약 20분 동안 실행합니다.
3. MS 분석을 위해 단백질을 분리하기 위해 트립신 프로테아제를 사용한 겔 내 소화
- 다음 용액(신선)을 준비합니다: 50% 아세토니트릴(ACN, pH 7–8) 중 25mM NH4HCO3 및 5% 포름산 중 50% ACN.
- 각 젤 슬라이스를 작은 조각(일반적으로 <1mm2)으로 깍둑썰기하고 0.65mL 실리콘 튜브에 넣습니다.
- 약 100μL의 25mM NH4HCO3/50% ACN 용액을 넣고 실온에서 10분 동안 와류를 추가합니다. 상층액을 추출하고 별도의 튜브(폐기)로 옮깁니다. 이 단계를 두 번 더 반복합니다.
- 진공 농축기에서 젤 조각을 완전히 건조시킵니다(~20분).
- 새로운 용액을 준비하고 건조된 겔 조각에 25mM NH4HCO3 에서 10 mM DTT ~100 μL (또는 덮을 수 있을 만큼)를 추가합니다(새로 준비된 NH4HCO3 사용). 소용돌이치고 잠시 회전합니다. 56°C에서 1시간 동안 반응이 진행되고 믹서에서 1400rpm으로 진탕하도록 합니다.
- 상층액을 제거하고 25mM NH4HCO3 중 100μL의 55mM 요오드아세트아미드(IAA)를 겔 조각에 추가합니다. 소용돌이치고 잠시 회전합니다. RT에서 45분 동안 어둠 속에서 반응이 진행되도록 합니다.
- 상등액을 제거하고 버리십시오. ~100μL의 25mM NH4HCO3 를 넣고 10분 동안 볼텍싱하여 젤 조각을 세척합니다. 상층액을 짧게 돌려서 제거하고 후자는 버립니다.
- 50% ACN의 25mM NH4HCO3 의 ~100 μL(또는 덮을 수 있을 만큼)를 젤 조각과 10분 동안 와류에 첨가하여 젤 조각을 탈수합니다. 그런 다음 잠시 회전하여 상층액을 제거하고 버립니다. 이 단계를 두 번 반복하거나 젤 조각이 완전히 건조하지 않은 경우 젤 조각의 크기가 더 작고 흐린 흰색으로 표시됩니다.
- 진공 농축기에서 젤 조각을 완전히 건조시킵니다(~20분).
- 처음에 각 샘플에 15μL의 트립신 용액(250ng 트립신)을 첨가한 다음 겔 조각(~100μL)을 덮기에 충분한 25mM NH4HCO3 를 추가합니다. 10분 동안 소용돌이 그런 다음 간단히 회전시키고 섞지 않고 4 분 동안 차가운 방에서 30 ° C에서 배양합니다.
- 필요에 따라 25mM NH4HCO3 를 추가하여 젤 조각을 완전히 덮습니다. 믹서에서 37 ° C에서 하룻밤 동안 16-20 rpm에서 1400 시간 동안 회전하고 배양합니다.
- 다음날, 잠시 소용돌이를 일으키고 소화를 돌립니다. 약 100μL의 HPLC 등급 물을 첨가하고 튜브를 스핀하고 파라필름으로 촬영한 다음 10분 동안 연속적으로 초음파 처리합니다. 초음파 처리 후 잠시 회전합니다.
- 수성 추출을 나타내는 분해 용액을 깨끗한 0.65mL 실리콘 튜브로 옮깁니다.
- 겔 조각에 ~100μL의 50% ACN/5% 포름산을 추가하고(이를 덮을 수 있을 만큼), 실온에서 10분 동안 소용돌이치고, 짧게 회전하여 용액을 모으고, 한 번 반복합니다. 이 단계와 이전 단계 3.13에서 추출된 펩타이드를 포함하는 모든 용액을 단일 튜브에 함께 모읍니다.
- 추출된 소화물을 소용돌이치십시오. 펩타이드를 진공 농축기(~2시간)에서 완전히 건조시킵니다.
- 30μL의 0.2% 포름산을 첨가하여 펩타이드를 냉장실의 믹서에 10분 동안 재현탁시킵니다.
- 실온에서 1850 x g 에서 5분 동안 샘플을 회전시킵니다. 펩타이드 용액을 흡입하고 깨끗한 새 0.65mL 실리콘 튜브로 이동합니다. 그런 다음 C18 탈염 팁을 사용하여 펩타이드 용액을 탈염합니다(아래 참조).
4. C18 탈염 팁을 가진 소화된 펩티드를 탈염하기
- 피펫을 10 μL로 설정하고 C18 탈염 팁을 부착합니다. 100% ACN 10μL를 피펫팅한 후 폐기하여 탈염 팁을 적십니다. 이 단계를 2회 반복합니다.
- 10μL의 50% ACN, 49.8% 물 및 0.2% FA를 피펫팅하여 C18 탈염 팁을 세척한 다음 폐기합니다. 이 단계를 2회 반복합니다.
- 0.2% FA의 10 μL를 물에 넣고 피펫팅한 다음 폐기하여 C18 탈염 팁을 평형을 이룹니다. 이 단계를 2회 반복합니다.
- 피펫을 10 μL로 설정하고 레진을 통해 분해된 펩타이드를 위아래로 15배 피펫팅하여 용액에서 레진으로 펩타이드를 로드합니다.
참고: 반복적인 작업은 탈염 팁의 수지에 모든 펩타이드의 결합을 보장합니다. - 수지에 결합된 분해된 펩타이드를 물에 10μL의 0.2% FA를 피펫팅한 후 폐기하여 분해합니다. 이 단계를 4번 반복하여 탈염 단계를 완료합니다.
- 새 튜브에서 10μL의 50% ACN, 49.8% 물 및 0.2% FA로 10회 위아래로 피펫팅하여 펩타이드를 용리시킵니다. 이 단계를 한 번 반복하여 단백질 분해물을 동일한 튜브에서 두 번째로 용리시킵니다.
- 탈염된 펩타이드를 진공 농축기에서 완전히 건조시킵니다(~20분).
- 15 μL의 0.2% FA + 1 μL iRT(indexed Retention Time) 펩타이드에 펩타이드를 재현탁합니다. Voxtex를 10분 동안 사용하고 12,000 x g 에서 2분 동안 원심분리한 다음 MS 분석을 위해 자동 시료 주입기 바이알로 옮깁니다(아래 참조).
5. DDA 및 DIA를 사용한 분해된 펩타이드의 질량 분석
참고: DDA 또는 DIA LC-MS/MS 방법을 사용하여 샘플을 분석할 수 있습니다. 이 연구에서는 고해상도 질량 분석기와 결합된 nano-LC 2D HPLC 시스템을 사용하여 샘플을 분석했습니다.
- 사중극자 ToF(Time-of-Flight) 질량분석기에 직접 연결된 칩 기반 HPLC 시스템과 결합된 HPLC 시스템을 사용합니다(다른 LC-MS 구성 및 시스템도 사용할 수 있음).
- 역상 HPLC-ESI-MS/MS를 사용하여 샘플을 분석합니다.
- 크로마토그래피 프로토콜을 구축하여 펩타이드 시료를 C18 프리 컬럼 칩에 로드합니다. 로딩된 펩타이드를 로딩 용매(0.1% 포름산)로 2μL/min의 유속으로 10분 동안 세척 및 탈염합니다.
- 펩타이드를 분석 컬럼 C18 칩으로 옮기고 이동상 A(2% 아세토니트릴, 0.1% 포름산) 및 B(98% 아세토니트릴, 0.1% 포름산)를 사용하여 3시간 그래디언트로 300nL/min의 유속으로 용리합니다. 첫 번째 용리 단계는 80분 동안 5% B에서 35% B까지의 선형 그래디언트로 구성됩니다.
- 5분에 걸쳐 이동상 B를 80%로 증가시킨 다음 이동상 B를 8분 동안 80%로 유지한 후 5% B로 변경하여 25분 동안 컬럼을 다시 평형화합니다.
- DDA에 대한 MS Instrument Method를 설정하고 다음과 같이 Instrument 파라미터를 정의합니다.
- 실험 1에 대해 다음 매개변수를 사용하십시오: m/z 400–1500에서 MS1 전구체 이온 스캔(누적 시간 250ms); 2–5에서 200 카운트의 전하 상태 이온에 대한 MS/MS 스캔을 트리거하도록 강도 임계값을 설정합니다. Dynamic Exclusion of Precursor Ions(전구체 이온의 동적 제외)를 60초로 설정합니다.
- 실험 2에 다음 매개변수를 사용합니다: MS2 스캔 범위가 m/z 100–1500인 MS/MS 생성물 이온 스캔(사이클당 30개의 생성물 이온 스캔 각각당 100ms의 누적 시간); 충돌 에너지 확산을 CES=5로 설정하고 '고감도 제품 이온 스캔 모드'에 체크 표시를 합니다.
참고: DDA 수집 방법은 6.3단계(아래 참조)에 설명된 대로 스펙트럼 라이브러리를 구축하는 데 사용됩니다. 여기에서 사이클당 각 MS1 스캔 후 30개의 가장 풍부한 전구체 이온에 대한 MS/MS 스펙트럼을 획득합니다. 총 사이클 시간은 약 3.3초입니다.
- DIA를 얻습니다. DIA에 대한 MS Instrument Method를 설정하고 다음과 같이 기기 파라미터를 정의합니다.
- 실험 1에 대해 다음 매개변수를 사용하십시오: m/z 400-1250에서 MS1 전구체 이온 스캔(누적 시간 250ms).
- 실험 2에 다음 매개변수를 사용합니다: MS2 스캔 범위가 m/z 100-1500인 64개의 가변 SWATH 세그먼트에 대한 MS/MS 생성물 이온 스캔(사이클당 64개의 생성물 이온 스캔 각각당 45msec의 누적 시간); 충돌 에너지 확산을 CES=10으로 설정하고 'High Sensitivity Product Ion Scan Mode'에 체크 표시를 합니다.
참고: Schilling et al.13에서 설명한 대로 64개 변수 창 DIA/SWATH 수집 방법을 사용하여 약 3.2초의 총 주기 시간으로 무표지 정량화를 수행합니다. DDA 획득의 경우, Q1 사중극자를 사용하여 좁은 질량 범위의 전구 이온을 충돌 셀로 전송하는 대신, 가변 창 너비 범위(예: m/z 5–90)를 사용하여 64개의 SWATH 세그먼트, 각 세그먼트에 대한 45ms 누적 시간, 그 결과 3.2초의 사이클 시간이 발생하며, 여기에는 250ms 누적 시간의 MS1 스캔 1회가 포함됩니다. 가변 윈도우 폭은 SCIEX '가변 윈도우 계산기' 알고리즘13을 사용하여 특정 m/z 범위 내에서 관찰되는 일반적인 MS1 이온 전류의 복잡성에 따라 조정됩니다('바쁜' m/z 범위에는 더 좁은 윈도우가 적용되고, 용리 전구체 이온이 거의 없는 m/z 범위에는 더 넓은 윈도우가 적용됨). 다른 MS 플랫폼에서는 대체 DIA 창 선택 전략도 구현할 수 있습니다.
6. 데이터 분석
참고: 특정 데이터 분석 설정은 특정 실험 조건에 맞게 조정되어야 합니다. 예를 들어, 선택한 단백질 데이터베이스(fasta 파일)는 샘플이 준비된 종에 따라 다릅니다(이 프로토콜에서는 C. elegans를 사용함).
- MS 데이터베이스 검색 엔진을 사용하여 DDA 획득을 분석하고 단백질을 식별할 수 있습니다. 다음과 같이 darabase 검색 엔진 방법을 생성합니다.
- Sample Description Parameters(샘플 설명 매개변수)에서 Identification(식별)을 선택합니다. Sample Type(샘플 유형)에서 Iodoacetic Acid(요오도아세트산)를 선택합니다. Cysteine Alkylation에서 Trypsin을 선택합니다. Digestion(라이신 및 아르기닌에서 C-말단 절단을 가정)에서 Instrument(기기)에서 질량분석기의 이름을 선택합니다. 을 선택하고 Species(종)에서 Caenorhabditis elegans를 선택합니다.
- 특정 처리 매개 변수의 경우 생물학적 변형을 확인하십시오. ID Focus에서 SwissProt을 선택합니다. 데이터베이스에서 철저한 ID를 확인하십시오. Search Effort(검색 노력)의 Detected Protein Threshold(감지된 단백질 임계값)에서 0.05(10%)를 선택합니다. 을 선택하고 Results Quality(결과 품질)에서 Run False Discovery Rate Analysis(거짓 발견 비율 분석 실행)를 선택합니다. 검색 엔진 방법을 저장하고 생성된 방법을 사용하여 처리를 위해 MS 원시 파일을 데이터베이스 검색 엔진에 제출합니다.
참고: 반복적인 프로세스에서 모든 MS 및 MS/MS 스캔은 초기 주석 및 결과를 기반으로 검색 엔진에 의해 자동으로 재보정되었습니다.
- 검색 완료 후 Export Peptide Summary(펩타이드 요약 내보내기 )를 클릭하고 Excel에서 신뢰 임계값 99(FDR(false discovery rate) = 1%)로 모든 펩타이드 식별 결과를 필터링합니다.
- DIA 원시 데이터 파일의 추가 처리 및 추가 상대 데이터 정량화를 위해 MS/MS 스펙트럼 라이브러리를 구축합니다.
- DIA 정량 분석 소프트웨어를 엽니다. 라이브러리 탭을 선택한 다음(페이지 아래쪽에서) "데이터베이스 검색 엔진"에서 스펙트럼 라이브러리 생성을 클릭하고 DDA 원시 데이터 파일 데이터베이스 검색 프로세스의 일부로 자동으로 생성된 "데이터베이스 검색 엔진" FDR 보고서(*FDR.xlsx 스프레드시트 파일)를 엽니다.
- Next(다음)를 클릭하고 Library Settings Schema(라이브러리 설정 스키마)를 선택한 후 Next(다음)를 클릭합니다. 데이터베이스로 Uniprot_Caenorhabditis elegans _proteome 선택한 다음 Finish를 클릭하면 스펙트럼 라이브러리가 생성됩니다.
- DIA 정량 분석 소프트웨어를 사용하여 포괄적인 상대 단백질 정량화를 위한 DIA 획득을 분석할 수 있습니다. DIA 데이터 분석 파이프라인은 다음과 같이 설명됩니다.
- 펩타이드를 분석하고 정량화하려면 DIA 정량 분석 소프트웨어를 열고 템플릿 스키마를 사용하여 분석 스키마를 설정합니다. 템플릿 스키마는 설정, DIA 분석, 마지막으로 BGS 공장 설정을 클릭하여 소프트웨어에서 사용할 수 있습니다.
- template schema의 매개 변수를 다음과 같이 설정합니다.
1) ID 에서 PTM 현지화 를 선택합니다(확률 컷오프 = 0.75).
2) quantification에서 Major Group Quantity 를 Sum peptide quantity로 선택하고, Major Group N 을 Max 7 및 Min 1로 설정하고, Minor Group Quantity 를 Sum precursor quantity로 선택하고, Minor Group N 을 Max 10 및 Min 1로 설정하고, Data Filtering 을 Qvalue sparse 로 선택하고 Cross Run Normalization을 선택하지 않습니다.
- 상대 정량화 분석을 수행합니다.
- 파이프라인 탭을 선택하고, 파일에서 DIA 분석 설정을 클릭하고, 관심 있는 MS DIA 원시 파일(예: 샘플 및 제어)을 열고, 스펙트럼 라이브러리 할당을 클릭하고, 6.3에서 생성된 라이브러리를 선택하고, 로드를 클릭하고, 마지막으로 다음을 클릭합니다.
- 6.5.1에 설정된 분석 스키마를 선택하고 다음을 클릭합니다. 적절한 데이터베이스 fasta 파일(Uniprot_Caenorhabditis elegans_proteome)을 선택하고 다음을 클릭합니다. 조건 설정을 정의하고 샘플에 따라 다른 조건을 할당한 후 Next(다음)를 클릭합니다.
- 분석 개요(실험 설정 요약)를 검토하고 Output Directory(출력 디렉토리 )로 이동하여 Finish(마침)를 클릭합니다. 마지막으로 Run Pipeline(파이프라인 실행 )을 클릭하여 레이블이 없는 정량 분석을 수행합니다.
참고: 'DIA 정량 분석 소프트웨어'의 통계 모듈은 FDR 분석을 자동으로 수행하고, 다양한 조건을 비교하는 히트 맵 및 화산 플롯을 생성하고, 식별 및 정량화된 펩타이드 및 단백질 목록을 생성하고, 서로 다른 조건을 비교하는 상대적 폴드 변화와 함께 Q 값을 제공합니다. 여기에서 샘플 조건 설정에는 두 개의 생물학적 복제물이 있는 10일차 노화된 벌레와 두 개의 생물학적 복제물이 대조군으로 포함된 2일차 어린 벌레가 포함되었습니다.
결과
전통적인 웜 용해 방법에는 여러 가지 단점이 있습니다. 예를 들어, 프로브 기반 초음파 처리 및 비드 비터 방법은 금속 팁 또는 비드가 샘플과 직접 접촉할 수 있도록 하여 과도한 열을 발생시켜 다양한 단백질 회수 및 단백질 변성을 초래합니다. 액체 질소 분쇄 후 용해 완충액에서 초음파 처리는 시간이 많이 소요될 수 있으며 많은 수의 웜이 필요합니다. 기존 웜 용해 방법의 한계로 인해 불용성에 대한 정량적 정보를 얻기 위해 C. elegans 모델 시스템에서 역사적으로 사용되었던 라벨링 방법 iTRAQ 또는 label-free 방법과 같은 이전 MS 워크플로우에는 많은 시작 물질(최소 40,000개의 웜)을 입력해야 합니다. 이 수의 지렁이를 얻기 위해서는 힘든 지렁이 배양 작업이 필요합니다. 또한 라벨링 방법에는 값비싼 동중원소 화학 라벨이 필요합니다. Label-free 정량화 방법은 비용 효율적이며 더 쉽고 간단한 시료 준비 및 라벨링 방법을 가지고 있지만, 충분한 MS 분석 범위를 달성하기 위해 상당히 많은 수의 웜이 필요합니다.
우리가 사용한 초음파 발생기는 교차 오염 없이 온도 조절 수조 초음파 발생기14에서 여러 개의 벌레 샘플을 동시에 용해함으로써 벌레 용해의 효율성과 재현성을 크게 증가시켜 필요한 초기 벌레 물질의 양을 크게 줄였습니다. 고효율 초음파 처리 방법과 정량적 DIA label-free MS 접근 방식을 결합하여 ~ 3,000 개의 웜을 사용하여 늙은 웜과 어린 웜의 불용성을 강력하게 정량화 할 수있었습니다. 여기에서 우리는 프로토콜의 효율성을 테스트하고 검증했으며 야생형 기생충 균주인 N2-Bristol C. elegans의 늙은 기생충과 어린 기생충의 불용성을 비교했습니다. 우리는 이 프로토콜을 적용하여 ~3,000마리의 나이 많고 젊은 N2 C. 예쁜꼬마선충 (각 조건에 대한 2개의 생물학적 복제)에서 불용성을 추출하고 분리한 다음, 단백질 식별 및 정량화를 위해 데이터 의존적 수집(DDA) 및 데이터 독립적 수집(DIA/SWATH)의 조합을 사용하여 사중극자 비행 시간 질량 분석기 또는 기타 MS 시스템으로 MS 분석을 수행했습니다. 불용성 단백질은 먼저 Bis-Tris 4-12% 구배 겔에서 분석하여 각 불용성 샘플의 단백질 양을 측정했습니다. 그림 2에서 볼 수 있듯이, N2 노화된 벌레의 불용성 샘플(2번 및 3번 레인, 생물학적 복제 실험)은 N2 어린 벌레의 샘플(1번 및 4번 레인, 생물학적 복제 실험)보다 훨씬 더 많은 단백질을 함유하고 있습니다.
겔 내 분해 후, 불용성의 단백질 프로파일을 HPLC-MS로 분석하였다. 이 워크플로우를 사용하면 일반적으로 1000-1,500개의 단백질을 식별하고 SDS-불용성 분획에서 500-1,000개의 단백질을 높은 재현성으로 정량화할 수 있습니다(미발표 데이터). 여기서 우리는 DIA 데이터를 분석하고 중복을 제거함으로써 N2-Bristol C. elegans 의 불용성체에서 989개의 단백질을 정량화할 수 있었습니다: 768개의 단백질이 현저히 농축되었고, 27개의 단백질은 1.5 이상의 폴드 변화와 0.01 미만의 Q 값을 사용하여 어린 (2일)에 비해 노화된 N2 벌레(10일)의 불용성체에서 현저히 감소했습니다(그림 3A). 히스토그램 플롯(그림 3B)에서 볼 수 있듯이, 현저하게 변형된 단백질의 접힘 변화는 정규 분포를 보여줍니다. 늙은 벌레는 불용성체에 대해 상당히 풍부하다는 것이 입증되었다: 관찰된 가장 큰 변화는 불용성체의 상대적 단백질 풍부도가 젊은 지렁이에 비해 늙은 지렁이에서 592배 더 높다는 것을 보여주었다. 그리고 32개의 단백질에 대해 불용성 단백질의 상대적 단백질 풍부도는 젊은 지렁이에 비해 늙은 지렁이에서 >250배 더 높았으며, 이는 나이가 들면서 불용성 지렁이가 극적으로 변함을 나타냅니다.
노화된 벌레에서 현저히 증가하고 웜베이스(WS271)에 의해 확인된 불용성 단백질 목록을 추출한 후, KEGG 경로 및 유전자 온톨로지(GO) 분석을 수행하여 노화된 불용성에서 풍부한 경로를 결정하여 이러한 단백질이 노화와 어떤 관련이 있는지에 대한 생물학적 통찰력을 얻었습니다. 이 연구에서 확인된 단백질의 KEGG 경로 분석은 리보솜, 미토콘드리아, 프로테아솜 및 스플라이소솜과 관련된 여러 경로의 농축을 보여줍니다(그림 4A). 유전자 온톨로지 분석은 노화된 기생충의 불용성체가 미토콘드리아, 발달, 성체 수명의 결정 요인 및 리보솜 단백질을 포함한 특정 범주의 많은 단백질로 구성되어 있음을 보여줍니다(그림 4B 및 보충 표 1A). 그런 다음 이 연구에서 확인된 단백질 목록을 벤 다이어그램(그림 5, A, 5B)에서 입증된 바와 같이 David et al.2 및 Mark et al.11의 이전에 발표된 작업과 비교했습니다. 비교 결과, 확인된 단백질 394/721 및 444/721이 David 등(그림 5A) 및 Mark 등(그림 5B) 연구와 유의하게 중복되는 것으로 나타났습니다. 이 연구에서 불용성에 대한 KEGG 분석에 의해 밝혀진 생물학적 경로는 과거에도 확인되어 우리의 방법론을 검증했습니다(보충 표 1B). 이러한 경로와 단백질의 확인은 노화의 맥락에서 그들의 기능과 관련하여 추가 생물학적 조사의 후보로 작용할 수 있음을 시사합니다.
요약하면, 효율적인 초음파 처리 방법을 사용하면 온도가 잘 제어되고 교차 오염이 감소한 환경에서 동시에 여러 웜 샘플을 용해할 수 있어 시작 웜 물질이 현저히 적은 높은 단백질 커버리지를 달성할 수 있습니다. 효율적인 초음파 처리 방법과 DIA label-free 단백질 정량 분석 워크플로우를 결합하면 불용성 웜 단백질의 정량화를 위한 신뢰할 수 있고 재현성 있는 결과를 얻을 수 있습니다.

그림 1. 프로토콜의 실험적 워크플로우. 예쁜꼬마선충(C. elegans)은 서로 다른 날에 양식되고 수집되었습니다. 초음파 처리기로 웜 용해 후 1% SDS-불용성 단백질 분획(불용성)을 추출하여 용해물에서 분리했습니다. 그런 다음 불용성을 겔 내 트립신 분해를 통해 분해하고 DIA 질량 분석법을 통해 정량화한 후 생물정보학적 분석을 수행했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
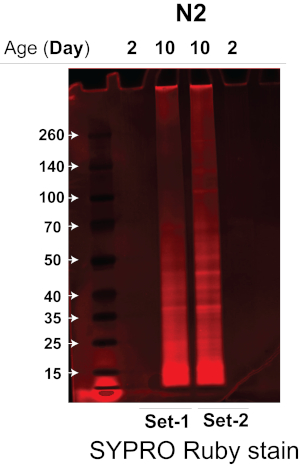
그림 2. 불용성의 SDS-PAGE 젤은 N2-Bristol 균주의 젊은 벌레와 늙은 벌레를 분리했습니다. N2-Bristol 균주의 젊은 벌레와 늙은 벌레의 불용성을 SDS-PAGE로 분석하여 존재하는 단백질의 양을 측정했습니다. SDS-PAGE 겔을 단백질 띠를 시각화하기 위해 형광 단백질 염색으로 염색했습니다. 레인 1 및 4: 어린 N2 벌레에 대한 두 가지 생물학적 복제 실험의 불용성(2일차). 레인 2 및 3: 노화된 N2 벌레에 대한 두 가지 생물학적 복제 실험에서 불용성(10일차). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3. 단백질 후보는 늙은 불용성 유전자와 젊은 불용성 분포 및 그 접힘 변화 분포에서 상당한 변화를 보이는 것으로 확인되었습니다. (A) 늙은 N2-브리스톨 웜과 어린 N2-브리스톨 웜의 불용성 정량화를 위한 화산 플롯. 절대 폴드 변화가 >=1.5이고 Q 값이 <0.01인 후보는 빨간색 점으로 표시됩니다. (B) 노화된 벌레 샘플과 어린 벌레 샘플에서 현저하게 농축된 SDS-불용성 단백질의 접힘 변화 분포에 대한 히스토그램 그래프. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4. KEGG 경로 및 유전자 온톨로지(GO) 분석. (A) p-값에 따라 정렬된 10일차 불용용성의 KEGG 경로 분석이 상단에 표시된 매우 유의한 경로. (B) 유전자 온톨로지 분석은 노화된 벌레의 불용성이 미토콘드리아, 발달, 성체 수명 결정 요인 및 리보솜 단백질을 포함한 특정 범주의 많은 단백질에 대해 풍부하다는 것을 보여줍니다. 산점도 보기는 더 유사한 용어가 서로 더 가깝게 배치되는 "의미론적 공간"에서 GO 용어를 시각화합니다. 거품의 색상은 STRING 분석에서 얻은 p-값을 반영하는 반면, 거품의 크기는 UniProt-GOA 데이터베이스에 있는 GO 항의 일반성을 반영합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5. 10일째 불용성 단백질에서 확인된 불용성 단백질 중복은 이 연구를 (A) David et al.2 및 (B) Mark et al.11 연구와 비교합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 표 1 (그림 4 및 그림 5 관련). (A) STRING 데이터베이스로 분석된 유전자 온톨로지(생물학적 과정). (B) 이 연구에서 확인된 단백질 및 KEGG 경로의 자세한 목록과 함께 발표된 연구와의 중복을 나타내는 색상 코드. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
이 프로토콜에서는 예쁜꼬마선충(C. elegans)에서 불용성 단백질을 추출하기 위한 개선된 시료 전처리 방법을 보고합니다. 기존의 웜 용해(예: 프로브 초음파 처리 또는 비드 비터 기술)를 효율적인 초음파 처리기로 대체함으로써 불용성 단백질 추출의 수율을 높이고 무표지 MS 분석에 필요한 웜 수를 40,000개에서 3,000개로 줄였습니다. DDA 데이터에서 단백질을 식별하기 위해 데이터베이스 검색 엔진이 사용되었으며, 'DIA 정량 분석 소프트웨어'와 해당 DDA 데이터베이스 검색 결과(데이터베이스 검색 엔진에서 생성된 FDR 파일 보고서 가져오기)를 사용하여 C. elegans 스펙트럼 라이브러리가 구축되었습니다. 새로운 DIA 데이터 세트와 생성된 스펙트럼 라이브러리를 처리하기 위해 'DIA 정량 분석 소프트웨어'를 사용하여 노화된 예쁜꼬마선충 불용성 데이터의 상대적 정량화를 수행했습니다.
프로토콜에서 몇 가지 단계가 중요합니다. 예쁜꼬마선충(C. elegans)의 수명이 짧기 때문에 포유류 세포와 같은 다른 진핵생물에 비해 노화를 연구하는 데 이상적인 시스템이지만, 노화 관련 현상을 연구할 때는 균질한 기생충 집단을 분리하는 것이 중요합니다. FUDR은 동기화된 에이징 웜을 얻기 위해 이 프로토콜에서 사용되었습니다. FedR이 포함된 NGM 시딩된 플레이트로 L4 단계 초기에 웜을 옮기는 것이 중요합니다. 효율적인 초음파 발생기를 사용하여 웜 용해하는 동안 수조 온도는 4 ° C로 설정하고 초음파 처리는 30 초 ON 및 30 초 OFF로 설정하여 샘플의 과열을 방지해야합니다. 10 사이클 (10 분) 동안 첫 번째 초음파 처리 후 모든 웜이 효율적으로 용해되었는지 확인하기 위해 현미경 아래에서 확인하는 것이 중요합니다. 그렇지 않은 경우 더 많은 초음파 처리 주기가 필요합니다. 겔 내 분해 과정에서 각 겔 슬라이스는 적절한 크기(<1mm2)의 조각으로 깍둑썰기해야 하며, 너무 작으면 샘플 준비 과정에서 손실될 수 있으며, 너무 크면 분해가 부적절할 수 있습니다.
훨씬 적은 시작 물질의 필요성은 불용성 분석을 위한 샘플을 얻기 위한 지렁이 배양과 관련된 힘든 작업을 크게 줄여줍니다. 그러나 1% SDS-불용성 단백질 분획의 추출 및 분리에는 여러 세척 단계가 포함되며 샘플 손실을 방지하고 재현성 있는 결과를 보장하기 위해 신중한 샘플 취급이 필요합니다. MS 분석을 위해 생성된 물질의 양은 후속 DDA 및 DIA 분석을 위해 ~3회 주입하기에 충분하지만 향후 실험을 위해 저장하기에는 충분하지 않습니다. 또한, 잠재적으로 혼란스러운 영향에도 불구하고15, 우리는 노화 과정에서 벌레를 살균하기 위해 가능한 가장 낮은 농도의 FUdR을 사용했습니다. 향후 연구에서는 무균 돌연변이를 사용하거나 수동으로 웜을 전송 및 수집하여 FUdR의 사용을 우회할 수 있습니다.
웜 용해를 위한 고효율 초음파 발생기를 사용하면 불용성을 효율적으로 추출할 수 있어 단백질 커버리지가 우수하고 비용 효율적인 무표지 DIA MS 분석을 통해 크게 줄어든 웜 수를 사용하여 불용성을 정량화할 수 있습니다. 작업량을 크게 줄여 실험당 더 많은 조건을 스크리닝할 수 있습니다. 또한 label-free MS DIA 워크플로우는 비용 효율적이며 iTRAQ, TMT 또는 SILAC를 포함한 라벨링 방법과 유사한 수준의 단백질 깊이와 커버리지를 제공합니다. 예쁜꼬마선충(C. elegans) 모델은 노화 연구를 위한 빠른 스크리닝 시스템입니다. 워크플로우는 쉽게 수정하고 이 유기체 및 다른 유기체의 노화 및 노화 관련 질병 연구를 연구하는 데 적용할 수 있습니다. 예를 들어, 진행 중인 연구에서는 이 워크플로우를 적용하여 향후 고처리량 약물 스크리닝을 위해 다양한 약물 중재를 사용하거나 사용하지 않는 Abeta, tau 및 이중 Abeta/tau 벌레를 포함한 다양한 알츠하이머병(AD) C. elegans 모델에서 불용성 및 프로테오스타시스의 단백질 프로파일을 조사하고 있습니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
이 작업은 TripleTOF 시스템(1S10 OD016281, Buck Institute), NIH 보조금, RF1 AG057358(GJL, JKA) 및 NIH 보조금 U01AG045844(GJL)에 대한 NIH 공유 계측 보조금의 지원을 받았습니다. XX는 T32 박사후 연구원(NIH 보조금 5T32AG000266, PI: Judith Campisi 및 Lisa Ellerby)의 지원을 받습니다. MC는 Larry L. Hillblom Foundation의 박사후 연구원의 지원을 받습니다.
자료
| Name | Company | Catalog Number | Comments |
| Strains used | |||
| Esherichia coli OP50 | Caenorhabditis Genetics Center (CGC) | ||
| N2 (Bristol) | Caenorhabditis Genetics Center (CGC) | ||
| Buffer/Solution | |||
| NGM (Nematode Growth Media) | Recipe: 3 g/L NaCl, 23 g/L agar; 2.5 g/L peptone; 1 mM CaCl2, 5 mg/L cholesterol, 1 mM MgSO4, 25 mM KH2PO4 | ||
| S-basal solution | Recipe: 5.85 g/L NaCl, 1g/L K2HPO4, 6 g/L KH2PO4, H2O to 1 L | ||
| Sodium hypochlorite bleach solution | Recipe: Mix 0.5 mL 5 N NaOH with 1 ml Sodium hypochlorite (5%) and make volume to 5 mL with H20. | ||
| Material/ Equipment | |||
| Agar | Difco Granulated Agar, BD Biosciences | 90000-782 | |
| Bioruptor Plus sonication device | Diagenode, USA | B01020001 | |
| Cholesterol | Sigma | c8503 | |
| 2'-deoxy-5-fluorouridine | VWR | TCD2235 | |
| Glycerol | Millipore Sigma | 356350-1000ML | |
| LB broth, Miller | Millipore Sigma | 60801-450 | |
| Sodium dodecyl sulfate (SDS? | Sigma | L4509-250G | |
| Sodium chloride | Sigma | 59888 | |
| M880 Ultrasonic bath, 117 V, holds 5.5 gallons | VWR, USA | 89375-458 | |
| Magnesium sulphate | Sigma | M506 | |
| Magnesium chloride | Sigma | 208337 | |
| NGM agar plate | VWR Disposable Petri Dishes | 25384-342 | |
| NuPAGE LDS Sample Buffer (4X) | Thermo Fisher Scientific | NP0007 | |
| NuPAGE protein gels, 4-12% | Invitrogen | NP 0335BOX | |
| Protease inhibiotr cocktail (PIC) | Roche | 11836170001 | |
| Pierce BCA Assay | Thermo Fisher Scientific | 23225 | |
| Sodium hypochlorite 5% | VWR | JT9416-1 | |
| SYPRO Ruby Protein Gel Stain | Thermo Fisher Scientific | S12000 | |
| MS Section | |||
| Acetonitrile, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 36XL66 | |
| Agilent Zorbax 300Extend C18 column | Agilent Technologies Inc., Santa Clara, CA, USA | 770995-902 | |
| Ammonium bicarbonate | Sigma Aldrich, St. Louis, MO, USA | 9830 (1 kg) | |
| Dithiothreitol (DTT) | Sigma Aldrich, St. Louis, MO, USA | D9779-5G | |
| Eppendorf Thermomixer Compact | Eppendorf AG, Hamburg, Germany | T1317-1EA | |
| Formic acid | Sigma Aldrich, St. Louis, MO, USA | F0507-500ML | |
| Indexed retention time (iRT) normalization peptide standard | Biognosys AG, Schlieren, Zurich, Switzerland | Ki-3002-2 | |
| Iodoacetamide (IAA) | Sigma Aldrich, St. Louis, MO, USA | I1149-25G | |
| Methanol, HPLC Grade | Honeywell International Inc., Charlotte, NC, USA | 34885 | |
| Nano cHiPLC Trap ChromXP C18-CL, 200 um x 6 mm, 3 um, 120A. (pre-column chip) (200 um x 6 mm ChromXP C18-CL chip, 3 um, 300 A) | Sciex LLC, Framingham, MA, USA | 804-00006 | |
| Nano cHiPLC ChromXP 75 um by 15cm, C18-CL, 3 um, 120 A (analytical column chip) | Sciex LLC, Framingham, MA, USA | 804-00001 | |
| Orthoganol quadrupole time-of-flight (QqTOF) TripleTOP 6600 mass spectrometer | Sciex LLC, Framingham, MA, USA | Per quote | |
| ProteinPilot 5.0 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Savant SPD131DDA Speedvac Concentrator | Thermo Fisher Scientific, Waltham, MA, USA | SPD131DDA-115 | |
| Sequencing-grade lyophilized trypsin | Life Technologies | 23225 | |
| Spectronaut | Biognosys AG, Schlieren, Zurich, Switzerland | Sw-3001 | |
| SWATH 2.0 plugin into PeakView 2.2 | Sciex LLC, Framingham, MA, USA | software download Sciex | |
| Ultra Plus nano-LC 2D HPLC system | Sciex LLC, Eksigent Division, Framingham, MA, USA | Model # 845 | |
| Water, Burdick and Jackson LC-MS | Honeywell International Inc., Charlotte, NC, USA | 600-30-76 | |
| Waters 1525 binary HPLC pump system | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters 2487 Dual Wavelength UV detector | Waters Corp., Milford, MA, USA | WAT081110 | |
| Waters 717plus Autosampler | Waters Corp., Milford, MA, USA | WAT022939 | |
| Waters Fraction Collector III | Waters Corp., Milford, MA, USA | 186001878 |
참고문헌
- Walther, D. M., et al. Widespread Proteome Remodeling and Aggregation in Aging C. elegans. Cell. , 919-932 (2015).
- David, D. C., et al. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biology. 8 (8), 47-48 (2010).
- Labbadia, J., Morimoto, R. I. The Biology of Proteostasis in Aging and Disease. Annual Review of Biochemistry. (1), 435-464 (2013).
- Ross, C. A., Poirier, M. A. Protein aggregation and neurodegenerative disease. Nature Medicine. , 10-17 (2004).
- Reis-Rodrigues, P., et al. Proteomic analysis of age-dependent changes in protein solubility identifies genes that modulate lifespan. Aging Cell. 11 (1), 120-127 (2012).
- Ayyadevara, S., et al. Proteins that accumulate with age in human skeletal-muscle aggregates contribute to declines in muscle mass and function in Caenorhabditis elegans. Aging. 8 (12), 3486-3497 (2016).
- Huang, C., et al. Intrinsically aggregation-prone proteins form amyloid-like aggregates and contribute to tissue aging in Caenorhabditis elegans. eLife. 8, (2019).
- Morimoto, R. I. Proteotoxic stress and inducible chaperone networks in neurodegenerative disease and aging. Genes and Development. 22 (11), 1427-1438 (2008).
- Morimoto, R. I., Cuervo, A. M. Protein homeostasis and aging: Taking care of proteins from the cradle to the grave. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 64 (2), 167-170 (2009).
- Klang, I. M., et al. Iron promotes protein insolubility and aging in C. elegans. Aging. 6 (11), 975-991 (2014).
- Mark, K. A., et al. Vitamin D Promotes Protein Homeostasis and Longevity via the Stress Response Pathway Genes skn-1, ire-1, and xbp-1. Cell Reports. 17 (5), 1227-1237 (2016).
- Groh, N., et al. Methods to study changes in inherent protein aggregation with age in caenorhabditis elegans. Journal of Visualized Experiments. (129), 1-12 (2017).
- Schilling, B., Gibson, B. W., Hunter, C. L. Generation of High-Quality SWATH((R)) Acquisition Data for Label-free Quantitative Proteomics Studies Using TripleTOF((R)) Mass Spectrometers. Proteomics: Methods and Protocols, Methods in Molecular Biology. 1550, 223-233 (2017).
- Walther, D. M., et al. Widespread proteome remodeling and aggregation in aging C. elegans. Cell. 161 (4), 919-932 (2015).
- Angeli, S., et al. A DNA synthesis inhibitor is protective against proteotoxic stressors via modulation of fertility pathways in Caenorhabditis elegans. Aging. 5 (10), 759-769 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유