Method Article
FLICK Assay를 사용한 약물 반응의 종합적인 분석
요약
이 프로토콜은 약물 반응을 평가하기 위해 FLICK 분석을 사용하는 방법을 설명하며, 이 분석을 사용하여 약물 유도 성장률 및 사멸률을 계산하고 약물 유도 세포 사멸 메커니즘을 평가하기 위한 자세한 지침을 포함합니다.
초록
약물 효능을 이해하기 위해서는 약물에 의한 세포 사멸의 정도를 규명하는 것이 매우 중요합니다. 약물에 의한 세포 사멸의 수준을 정량화하려는 노력은 각각 고유한 활성화 타이밍과 생화학적 특징을 가진 12가지 이상의 분자적으로 구별되는 조절 사멸 형태의 존재로 인해 도전을 받고 있습니다. 또한, 일부 괴사성 사멸 아형의 경우, 특징적인 특징은 일시적으로만 관찰되며 세포 파열로 인해 빠르게 손실됩니다. 따라서 사멸 경로 특이적 분석법의 조합을 사용하는 경우에도 세포 사멸의 총량 또는 각 사멸 아형의 상대적 기여도를 정확하게 정량화하는 것은 어렵습니다. 또 다른 문제는 많은 사망 특이적 분석법이 약물이 세포 증식에 미치는 영향을 무시하기 때문에 약물 치료를 받는 집단이 확장 또는 축소되고 있는지 해석하기 어렵다는 것입니다. FLICK 분석은 사멸에 특이적이지만 활성화된 사멸 유형에 크게 구애받지 않는 방식으로 자극 후 세포 사멸의 총 수준을 정량화할 수 있습니다. 또한 FLICK 분석은 총 개체군 크기 및 세포 증식 속도에 대한 정보를 유지합니다. 이 원고에서는 FLICK 분석의 기본 사용, 다양한 유형의 생물학적 물질을 사용할 때 이 분석의 문제를 해결하는 방법, FLICK 분석을 사용하여 관찰된 약물 반응에 대한 각 유형의 세포 사멸의 기여도를 정량화하는 방법에 대해 설명합니다.
서문
항암제의 경우, 약물 감수성의 전임상 평가에는 일반적으로 약물이 배양1에서 세포의 생존력에 어떤 영향을 미치는지 테스트하는 것이 포함됩니다. 약물 노출 후 세포 생존력은 적어도 두 가지 효과, 즉 약물에 의한 세포 증식 억제와 세포 사멸 활성화의 산물입니다2. 불행히도, 세포 사멸이 지속적인 약물 반응에 필요한 중요한 기능임에도 불구하고, 표준 접근법은 약물이 세포 사멸을 활성화하는 정도를 명확히 하지 못합니다3.
일반적인 약물 반응 분석법에는 세포를 직접 계수하거나(예: Coulter Counter, 유세포 분석 또는 현미경 검사의 일부 사용), 세포의 증식 능력을 정량화하거나(예: 콜로니 형성 분석), 대사 활성을 정량화하는 분석(예: CellTiter-Glo, 테트라졸륨 기반 MTT 또는 MTS 분석)이 포함됩니다. 이러한 분석의 공통된 특징은 생성된 데이터가 살아 있는 세포의 수에 비례한다는 것입니다. 약물은 성장 억제와 세포 사멸을 조정하는 방법이 상당히 다르기 때문에 약물 노출 후 살아있는 세포의 수는 약물 유도 세포 사멸 수준에 대한 신뢰할 수 없는 통찰력을 제공합니다3. 더욱이, 암세포는 일반적으로 세포 배양에서 빠르게 증식하기 때문에 세포 사멸을 유발하지 않고 치료되지 않은 집단에 비해 살아있는 세포의 수를 크게 줄일 수 있습니다4. 따라서 핵심적인 결함은 살아 있는 세포와 죽은 세포의 수를 모두 측정하지 않고는 세포 사멸 정도를 정량화할 수 없다는 것입니다.
약물 노출 후 죽은 세포의 수를 정확하게 정량화하는 것은 여러 가지 이유로 인해 어렵습니다2. 첫째, 12개 이상의 조절된 세포 사멸 경로가 존재합니다 5,6,7,8. 일반적으로 각 유형의 조절된 세포 사멸을 식별하기 위해 생화학적 마커가 존재하지만, 이러한 마커는 특이성이 다양하며 모든 사멸 하위 유형을 동시에 정량화하는 데 단일 분석을 사용할 수 없습니다. 둘째, 각 형태의 세포 사멸에 대한 활성화 시기는 상황에 따라 매우 극적으로 달라질 수 있으므로 시간이 지남에 따라 죽음을 정량화하지 않는 한 완전한 그림이 나타날 수 없습니다 9,10. 세포 사멸을 정량화하기 위한 많은 생화학 분석은 종말점 측정을 생성하므로 역학 데이터를 생성하는 것은 까다롭고 비용에 의해 제한될 수 있습니다. 세 번째 문제는 죽은 세포 자체가 살아있는 세포 상태와 해리된 세포 파편 사이의 일시적인 중간 상태라는 것입니다. 죽은 세포의 안정성은 사멸 아형에 따라 다르며, 세포사멸과 같은 일부 유형은 상대적으로 안정적인 시체를 생성하는 반면 다른 유형의 사멸은 빠른 용해를 유발합니다. 따라서 죽은 세포의 수집 및 계수가 필요한 사멸을 정량화하는 방법도 세포 사멸에 대한 편향된 이해를 초래할 것입니다. 마지막으로, 네 번째 한계는 세포 사멸 정도를 정량화하는 생화학적 분석법이 일반적으로 약물이 증식을 어떻게 변화시키는지에 대한 통찰력을 제공하지 못한다는 것입니다. 따라서 전체 개체군 규모, 그리고 중요한 것은 개체군이 확장되고 있는지 축소되고 있는지 여부를 해석할 수 없습니다.
STACK 및 SPARKL과 같은 일부 현미경 기반 분석은 시간 경과에 따른 살아있는 세포와 죽은 세포를 측정하는 데 효과적이며, 이러한 분석은 약물 유발 세포 사멸에 대한 포괄적인 통찰력을 제공할 수 있습니다10,11. 그러나 이러한 분석에는 Incucyte 현미경과 같은 특수 기기가 필요하므로 처리량과 이러한 접근 방식에 대한 접근에 제한이 있습니다. 또한 현미경 기반 기술은 죽은 세포가 실험 기간 동안 현미경의 초점면에 남아 있어야 하므로 플레이트에서 부착력을 잃거나 시간이 지남에 따라 죽은 세포가 부패할 때 죽은 세포를 정량화하는 능력이 저하됩니다. 마찬가지로, 현미경 기반 분석은 현탁 배양의 맥락에서 적용할 때 세포가 주어진 초점면 안팎으로 표류하기 때문에 문제에 직면합니다.
위에서 강조한 문제를 해결하기 위해 FLICK(Fluorescence-based and lysis-dependent Inference of Cell Death Kinetics)이라는 분석을 생성했습니다12,13. FLICK 분석의 목표는 세포가 어떻게 죽어가는지에 관계없이 약물에 의한 세포 사멸 수준을 측정하는 것입니다. FLICK 방법은 형광이 DNA 결합에 따라 달라지는 세포 침투성 염료를 사용합니다. FLICK의 주요 특징은 이러한 형광단을 사용하여 접근 가능한 DNA를 통해 시간이 지남에 따라 사체 축적을 라벨링한 다음, 기계적 세제 기반 용해를 통해 분석 종료 시 살아있는 세포를 투과화하는 것입니다. 이러한 데이터는 수학적 모델링과 결합되어 죽은 세포를 수집하거나 처리할 필요 없이 지속적인 시간 분해능으로 살아있는 세포와 죽은 세포 집단 모두를 정량화할 수 있습니다. 또한, 플레이트 리더를 사용하여 사세포 형광을 평가하면 사세포가 손상되지 않은 상태로 유지될 필요 없이 사세포를 평가할 수 있으므로 세포 파열을 초래하는 괴사 형태의 죽음에 대한 편견을 완화할 수 있습니다. 마지막으로, FLICK 분석은 최소한의 플레이트 취급이 필요하며 역학 측정을 신속하게 생성할 수 있어 고처리량 약물 스크리닝이 가능합니다. 이 프로토콜에서는 FLICK을 사용하여 약물 유도 성장률, 사멸률 및/또는 세포 사멸 메커니즘을 추론하는 방법을 포함하여 FLICK 분석의 사용에 중점을 둡니다.
프로토콜
1. 관심 세포주별 투과화 시간 최적화
참고: 설명된 부피와 양은 하나의 세포주를 최적화하기 위한 것입니다. 이러한 값은 테스트할 세포주의 수에 따라 확장해야 합니다.
- 96-well, 광학 바닥, 검은색 벽 플레이트의 각 웰에 원하는 수의 셀을 플레이트합니다. 100μL의 완전한 배지를 추가하고 세포가 밤새 플레이트에 부착되도록 합니다.
참고: 도금된 세포 수는 세포의 성장 속도, 최적 밀도 및 분석 길이를 고려하여 최적화해야 합니다. 72시간 동안 측정된 부착성 암 세포주에 대한 일반적인 시작 세포 수는 웰당 1500 - 5000 세포입니다. 예를 들어, U2OS 세포는 10% FBS, 2mM 글루타민 및 1% Pen-Strep을 사용하여 DMEM에서 웰당 2000개의 세포로 도금하고 표준 조건(5% CO2, 37°C, 습도 포함)에서 밤새 배양할 수 있습니다. - 15mL 원뿔형 원심분리 튜브에 1.5%의 Triton-X 용액 1.5mL를 인산염 완충 식염수(PBS)에 넣습니다. 1.5% Triton-X 용액을 최대 속도로 5초 동안 Vortex합니다. 37°C의 수조에 최소 30분 동안 둡니다.
- 용액을 육안으로 검사하여 Triton-X가 몇 초 동안 와류에 의해 완전히 용해되었는지 확인합니다. 용액이 동질적인지 확인합니다.
- 1.5% Triton-X 용액 10μL를 도금된 셀의 각 웰에 추가합니다. 섞지 마십시오.
참고: 이 단계에서는 기포 형성으로 인해 반복적인 피펫팅을 사용하여 혼합하는 것은 바람직하지 않습니다. - 습도 세포 배양 인큐베이터를 사용하여 최소 5시간 동안 세포를 37% CO2, 1°C로 되돌립니다.
참고: Triton-X 용액을 적용한 후 대부분의 세포주는 2-3시간 내에 완전히 용해됩니다. 최대 24시간을 기다리면 SYTOX Green과 같은 염료를 사용할 때 형광 신호에 미치는 영향이 최소화됩니다. Triton-X의 비율을 늘리면 용해가 어려운 세포주의 배양 시간을 단축할 수 있습니다. - 10x 대물렌즈를 사용하여 광학 현미경에서 세포 형태를 관찰합니다. 세포체가 더 이상 보이지 않을 때까지 한 시간에 한 번씩 세포를 검사합니다. 세포 투과화에 필요한 시간을 기록합니다.
참고: 형광 현미경을 사용할 수 있는 경우 SYTOX Green과 같은 세포 불투과성 DNA 염료를 사용하여 용해를 시각화할 수 있습니다. 염료는 Triton-X 용액에 최종 농도의 10배로 첨가할 수 있으며, 형광 현미경으로 투과화를 확인할 수 있습니다.
2. DNA 염색의 선택 및 보정
참고: FLICK 분석의 요구 사항은 DNA 결합 의존적 방식으로 신호를 방출하고, 세포 생존율에 영향을 미치지 않으며, 세포 수에 따라 선형으로 확장되는 신호를 생성하는 세포 투과성 형광단을 사용하는 것입니다. 이 프로토콜은 SYTOX Green을 사용합니다. 유사한 특성을 가진 다른 염료도 FLICK 분석에 적합할 수 있지만 각각 평가 및 보정해야 합니다. 예는 표 1 을 참조하십시오.
- 선택한 DNA 염료의 경우 제조업체의 권장 사항에 따라 테스트할 농도 범위를 결정합니다.
- 테스트할 각 농도에 대해 180μL의 세포 배양 배지에 있는 40,000개의 세포를 96웰의 광학 바닥, 검은색 벽 플레이트의 가장 왼쪽 열을 따라 3중으로 플레이트합니다. 나머지 웰에 90μL의 배지를 추가합니다.
참고: 이것은 플레이트의 행에 걸쳐 선형 세포 적정을 생성하는 데 사용됩니다. 첫 번째 웰의 최종 농도는 20,000 cells/well입니다. 광학 바닥 플레이트는 웰 상단에서 형광을 판독하는 플레이트 리더에는 필요하지 않습니다. 세포 배양 배지는 테스트 중인 세포를 성장시키는 데 사용되는 것과 동일한 배지여야 합니다. 예를 들어, U2OS 셀을 사용하는 경우 40,000개의 세포가 180μL의 DMEM에 재현탁되어야 합니다. - 멀티채널 피펫을 사용하여 가장 왼쪽 컬럼에서 오른쪽 인접 컬럼으로 90 μL를 전달하여 1:2 직렬 희석을 생성합니다. 피펫 15x 혼합.
- 2.3단계를 반복하여 두 번째 열에서 세 번째 열로 이동한 다음 세 번째 열에서 네 번째 열로 이동하는 방식으로 이동합니다. 플레이트의 두 번째에서 마지막 컬럼에서 적정을 마무리합니다. 세포 배양 배지의 배경 신호를 얻을 수 있도록 마지막 열을 세포 없이 둡니다.
- 끝에서 두 번째 컬럼의 경우 90μL를 제거하여 모든 웰이 다양한 수의 세포를 포함하는 90μL의 배지를 갖도록 합니다. 37°C 세포 배양 인큐베이터에서 6시간 동안 세포를 부착합니다.
- 테스트할 염료의 각 농도에 대해 10x DNA 염료 용액을 준비하고, DNA 염료를 희석하여 관심 세포를 배양하는 데 사용되는 완전한 성장 배지로 만듭니다. 1단계에서 설명한 대로 PBS에서 1.5% Triton-X 용액 1.5mL를 준비합니다.
- 96웰 플레이트의 각 웰에 10x DNA 염료 10μL를 추가합니다. 각 웰에 10μL의 1.5% Triton-X 용액을 첨가하여 세포를 투과시킵니다. 1단계에서 결정된 최적의 시간 동안 이 용액에서 세포를 배양합니다.
참고: Triton-X를 첨가한 후 DNA 염료의 최종 농도는 1x보다 약간 낮지만 이는 중요하지 않습니다. 배양 후에는 전체 플레이트를 용해해야 합니다. 수동 세포 적정은 최적의 DNA 염료 농도와 형광 획득을 위한 최적의 설정을 식별하는 데 사용됩니다. - 세포 적정 플레이트에서 형광 강도를 측정합니다. DNA 염료 제조업체가 설명한 다양한 획득 설정에 걸쳐 형광을 정량화합니다. 사용된 플레이트 리더에 따라 여기 및 방출 파장 및/또는 디지털 게인을 수정합니다.
- 셀이 없는 열의 평균 신호를 빼서 각 측정에서 배경 신호를 제거합니다. 이 값은 연속 희석 플레이트에 있는 웰의 가장 오른쪽 열에서 찾을 수 있습니다.
- 각 획득 설정에 대한 각 DNA 염료 농도의 선형성을 측정합니다. 선형성은 형광 신호에 대해 세포 수를 그래프로 표시하고 선형 회귀를 수행하여 결정할 수 있습니다. 측정 계수(r2)는 형광 신호가 세포 수와 선형적으로 관련되는 정도를 보고합니다.
- 선형성과 동적 범위의 최상의 조합을 가진 수집 설정과 DNA 염료 농도를 선택하십시오.
참고: 0에서 20,000 셀까지의 전체 셀 번호 범위를 고려하십시오. 이득 및 DNA 염료 농도가 0에서 1000 세포 사이의 낮은 세포 수에서 선형인지 확인합니다. 여러 기기 설정 또는 DNA 염료 농도는 전체 범위에 걸쳐 강력한 상관 관계를 제공할 수 있습니다. 그러나 죽은 세포의 작은 변화를 정확하게 측정할 수 있도록 로우 엔드 감도가 견고해야 합니다.
3. 약물 처리 플레이트에서 세포 도금, In-well 약물 적용 및 시간 경과에 따른 사체 형광 측정
참고: 약물 희석 플레이트는 실험적 요구 사항에 따라 유연하게 설계할 수 있습니다. 일반적으로 약물 희석 플레이트에는 하나 또는 여러 약물의 로그 또는 반로그 희석 시리즈가 포함됩니다.
- 실험을 위한 최적의 도금 레이아웃을 고려하십시오. 외부 웰은 플레이트의 내부 웰과 비교하여 다른 온도, 산소화 및 증발을 경험하므로 성장 변화와 관련된 소음을 줄이기 위해 96웰 플레이트의 외부 웰에서 측정을 피하십시오.
참고: 플레이트의 가로 전체에 컨트롤을 포함하면 성장 데이터의 견고성이 향상됩니다. 플레이트의 중앙과 가장자리에 제어 웰을 포함하는 것을 고려하십시오. - 실험에 필요한 플레이트의 수를 결정합니다. 약물 투약 시 웰당 평균 세포 수를 설정하기 위해 분석 시작 시 용해할 플레이트를 하나 더 포함합니다(즉, T0 대조 플레이트).
- 완전한 성장 배지에서 세포 현탁액을 준비합니다.
- 실험에 필요한 플레이트의 수(3.2단계에서 계산)에 따라 필요한 세포 현탁액의 부피를 결정합니다. 웰당 90μL에서 플레이트를 파스딩하는 경우 플레이트당 약 10mL가 필요합니다. 필요한 플레이트 수에 10mL를 곱합니다.
참고: reservoir의 dead volume과 pipetting 중 부피 손실을 설명하기 위해 이 부피를 증가시키는 것이 바람직합니다. 예를 들어, 계산된 부피에 1.2를 곱할 수 있습니다. - 세포를 계수하고 웰당 원하는 세포 수에 따라 세포 희석을 준비합니다. 예를 들어, 96웰 플레이트의 웰당 90μL의 배지에 2000개의 세포를 시드하려는 경우 총 3개의 플레이트에 대해 실험에 필요한 총 세포 수를 아래와 같이 계산합니다.

마찬가지로, 계수된 세포 현탁액이 400,000 cells/mL인 경우 실험에 필요한 세포 현탁액의 부피는 다음과 같습니다.

- 추가될 계수된 세포 현탁액의 부피를 빼서 도금에 사용되는 세포 배양 배지의 부피를 수정합니다.
36 mL 배지 - 2 mL 세포 현탁액 = 34 mL 보정된 배지 부피
- 실험에 필요한 플레이트의 수(3.2단계에서 계산)에 따라 필요한 세포 현탁액의 부피를 결정합니다. 웰당 90μL에서 플레이트를 파스딩하는 경우 플레이트당 약 10mL가 필요합니다. 필요한 플레이트 수에 10mL를 곱합니다.
- 혈청학 피펫을 사용하여 계수된 세포 현탁액을 올바른 배지 부피와 혼합합니다. 이 세포 현탁액을 V-bottom 시약 저장소로 옮깁니다.
- 멀티채널 피펫을 사용하여 96웰 플레이트의 각 웰에 90μL의 세포 현탁액을 추가합니다. 혈청학적 피펫과 함께 반복적인 피펫팅을 사용하여 정기적으로 시약 저장소의 세포 현탁액을 혼합하여 부피당 원하는 세포 농도가 유지되도록 합니다.
- 37°C 세포 배양 인큐베이터(일반적으로 12-24시간)에서 밤새 세포를 접착시킵니다.
- 1단계에서 설명한 대로 PBS에서 1.5% Triton-X 용액을 준비합니다. 이 용액의 부피는 모든 플레이트를 용해하기에 충분해야 합니다. PBS에 함유된 약 1.5mL의 1.5% Triton-X는 96웰 플레이트 1개를 용해시키기에 충분합니다.
참고: PBS의 1.5% Triton-X 용액은 일주일 이상 안정적이며 편의를 위해 미리 만들 수 있습니다. - 약물 희석 플레이트를 만드는 데 필요한 세포 배양 배지의 양을 측정합니다. U- 또는 V-바닥 96-well 투명 플레이트에 약물 희석 플레이트를 준비합니다.
- 희석된 약물의 부피가 실험에 충분한지 확인하려면 파이펫팅 중 부피 손실을 고려하여 필요한 최소 부피를 늘리십시오. 예를 들어, 96웰 플레이트의 각 웰에 10μL에 약물을 첨가하고 실험에 3개의 플레이트가 포함된 경우 웰당 30μL의 희석된 약물이 필요합니다. 부피 손실을 설명하기 위해 웰당 40-50μL의 약물 배지를 준비합니다. T0 control plate에 필요한 매체를 고려하는 데 필요한 매체 부피에 1.5mL를 추가합니다.
- 완전한 성장 배지에서 선택한 DNA 염료의 10배 농도를 준비합니다. 이 농도는 2.11단계에서 선택한 농도를 기반으로 합니다. 이 솔루션의 총 부피는 3.8단계에서 계산됩니다.
- 3.9단계의 DNA 염료 + 성장 배지를 사용하여 테스트할 각 약물의 10배 농도를 준비합니다. 약물 희석 플레이트에 각 약물의 최고 용량만 생성하고 멀티채널 피펫으로 약물을 연속적으로 희석하여 각 웰 사이에 15배씩 혼합합니다.
참고: 약물 희석 플레이트에서 주어진 약물에 대해 최고 용량을 만들 때 농도가 정확한지 확인하기 위해 약물의 부피를 고려해야 합니다. - 멀티채널 피펫을 사용하여 3.10단계의 약물 + DNA 염료 용액 10μL를 세포가 포함된 플레이트에 추가합니다.
참고: 연속 희석이 생성된 약물 희석 플레이트의 경우, 약물이 가장 낮은 농도의 약물에서 시작하여 가장 높은 농도까지 세포에 추가되면 다채널 파이펫의 팁을 변경할 필요가 없습니다. 그러나 다채널 피펫의 팁은 각 플레이트 사이와 팁이 다른 약물 또는 더 높은 농도의 약물을 포함하는 우물에서 사용될 때마다 교체해야 합니다. - 약물이 첨가된 모든 플레이트의 형광을 읽습니다. 이 형광 판독값은 T0 판독값(즉, Time = 0 h에서 죽은 세포)입니다.
- T0 형광 판독을 수행한 후 플레이트를 인큐베이터에 다시 넣습니다.
- 3.9단계에서 생성된 10x DNA 염료 용액 10 μL와 3.7단계에서 1.5% Triton-X 용액 10 μL를 T0 control plate에 추가합니다. 1.8단계에서 선택한 시간 동안 이 플레이트를 인큐베이터에 반환합니다.
- 1.6단계에서 설명한 대로 세포가 완전히 용해되면 T0 대조판을 읽습니다.
4. 약물 처리된 플레이트의 시간 경과에 따른 사세포 형광 측정
알림: 플레이트가 인큐베이터에서 나오는 시간을 최소화하십시오. 온도 변화가 장기화되면 세포 생존율에 영향을 미칠 수 있으며, 빛에 노출되면 SYTOX Green과 같은 DNA 형광단이 손상될 수 있습니다.
- 약물 투약 후 3-4시간마다 모든 약물 처리된 플레이트에 대한 형광 판독값을 획득합니다. 플레이트는 매우 정확한 죽음의 역학이 필요하지 않는 한 하룻밤 사이에 읽을 필요가 없습니다.
참고: 고정/표준 간격으로 시점을 취할 필요가 없습니다. 일반적으로 시점은 사망 역학의 다운스트림 분석에서 오류를 일으키지 않고 정상 근무 시간 중에만 독점적으로 사용할 수 있습니다. 역학은 사멸 발병 전, 사멸의 상승 단계 및 포화 / 고원 단계에서 일부 측정이 수행되는 한 추론 할 수 있습니다. - 원하는 최종 시점에서, 형광 판독값을 획득합니다. 그 직후, 3.7단계에서 준비한 1.5% Triton-X 용액 10μL를 첨가하여 세포를 용해시킵니다.
- 플레이트를 인큐베이터로 되돌리고 1단계에서 결정한 시간 동안 세포가 투과되도록 합니다.
- 투과화 후 형광 판독값을 획득합니다. 이 형광 값은 각 웰에 대한 총 세포 수(즉, 살아있는 세포 + 죽은 세포)에 비례합니다.
5. 치사율 역학 계산
참고: 이 프로토콜에 설명된 계산은 모든 형식 또는 소프트웨어로 분석할 수 있습니다. 그러나 MATLAB, R 또는 Python과 같은 프로그래밍 환경을 사용하면 더 빠르고 유연한 분석이 가능합니다.
- 50% 절사 평균을 사용하여 T0 제어판의 평균 형광 값을 계산합니다. 이 값은 약물 투여 전의 총 세포 수에 비례합니다.
- 곡선 피팅과 지수 성장 함수를 사용하여 실험의 모든 우물에 대한 개체군 성장의 역학을 계산합니다. 각 웰의 초기 개체군 크기는 5.1단계에서 계산된 평균 T0 형광 값입니다. 각 웰의 최종 개체군 크기는 4.4단계에서 계산된 투과화 후 값입니다. 분석 기간은 0시간부터 4.4단계에서 선택한 분석 종말점까지입니다.
- 5.2단계에서 결정된 성장 매개변수를 사용하여 모든 웰에 대한 분석에서 측정된 모든 시점의 총 세포 수를 계산합니다. 5.2단계에서 계산된 총 세포에서 dead cell 측정값을 빼서 측정된 각 측정 시점의 살아있는 세포 수를 결정합니다.
참고: 분석에서 소량의 노이즈로 인해 때때로 살아있는 세포 수가 작은 음수일 수 있습니다. 라이브 셀의 수가 음수일 수 없기 때문에 수동으로 0으로 설정할 수 있으며 해당 웰의 모든 셀이 죽었음을 나타낼 가능성이 높습니다. - dead cell 형광 신호를 각 시점의 총 세포 신호로 나누어 각 시점에 대한 치사 분율(LF)을 계산합니다.
- LED(Lag Exponential Death) 방정식을 치사 분수 시간 경과 데이터10에 맞춥니다. 유의미한 치사율을 유발하지 않는 약물 용량에 대한 임의의 동역학 매개변수를 피하려면 기울기가 0인 선형 모델에 맞춥니다. 분석의 잡음 또는 약물이 투여되지 않은 조건에서 관찰된 LF를 기반으로 상당한 수준의 치사율을 결정합니다.
- LED 방정식에서 사망 발병 시간(DO)을 기록합니다. LED 방정식에는 초기 LF(LFi), 안정기에서의 LF(LFp), 초기 사망률(DR) 및 DO의 4가지 매개변수가 있습니다. 비선형 회귀(즉, 곡선 피팅)를 사용하여 데이터에서 이러한 매개변수를 추론합니다.
- 각 용량에서 각 약물에 대한 분석 종말점에서 분획 생존율(FV)을 계산합니다. 1에서 엔드포인트 LF 값을 빼거나 살아있는 셀의 수를 총 셀로 나눕니다.
참고: FV는 분석 종말점뿐만 아니라 언제든지 계산할 수 있습니다. 이러한 데이터는 용량 약리학(예: 약물 IC50 또는 EC50)을 평가하는 데 사용됩니다.
6. GR 값 계산
- 분석 시작 시 각 웰에 대한 살아있는 세포의 평균 수를 측정합니다. 3.15단계에서 계산된 투과화 후 T0 판독값을 사용하여 이 값을 계산하고 3.12단계에서 수집된 각 웰에 대한 T0 판독값을 뺍니다. 아래 GR 방정식(단계 6.3)에서 이 값을x0이라고 합니다.
주: 때때로 약의 과다 복용은 DNA 염료의 형광 독서를 방해할 수 있습니다. 제어 웰을 사용하여 시간 0에서 살아있는 세포의 평균 수를 계산하는 것이 바람직할 수 있습니다. - control wells(xctrl)의 경우, 분석 종말점에서 각 well에 대한 살아있는 세포의 평균 수를 측정합니다. 각 약물 처리 조건에 대한 분석 종말점에서 살아있는 세포의 평균 수를 측정합니다(x(c)).
참고: 플레이트 전체의 세포 성장 변화에 따라 각 웰의 정규화에 다른 xctrl 값을 사용할 수 있습니다. 예를 들어, xctrl 값이 플레이트 전체에 걸쳐 체계적으로 변하는 경우 플레이트의 평균 xctrl 값보다 가장 가까운 xctrl 값을 사용하는 것이 바람직할 수 있습니다. - 각 약물 처리된 웰에 대해 다음 방정식4를 사용하여 정규화된 성장률 억제 값(GR)을 계산합니다.
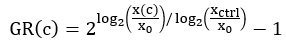
4-모수 로지스틱 회귀 분석을 사용하여 곡선 피팅을 수행합니다. GR은 -1에서 1까지의 척도입니다.
7. GRADE 방법을 사용하여 약물로 인한 성장 및 사망률 계산
참고: GR은 실제 세포 증식률이 아닌 순 개체군 성장률을 나타냅니다. 약물로 인한 인구 증가율과 사망률은 GR과 치사율(LF)의 조합을 사용하여 계산할 수 있습니다.
- T0 대조군 판독값의 평균 살아있는 세포 수(x0), 분석 종말점에서 제어 조건의 평균 살아있는 세포 수(xctrl) 및 시간 단위의 분석 길이(t)를 사용하여 실험 중 세포주의 배가 시간(τ)을 측정하여 다음 방정식을 풉니다.

참고: 세포 배가 시간(τ)을 정확하게 계산하려면 세포가 분석 기간 동안 지속적으로 두 배가 되어야 합니다. 접촉 억제를 경험하는 세포의 경우, 세포는 원하는 분석 시간 동안 합류하지 않는 시작 밀도로 파종되어야 합니다. 예를 들어 U2OS의 평균 배가 시간은 24시간입니다. 72시간 분석에서 웰당 2000개의 세포를 파종하는 것은 성장 속도를 해석하는 데 최적입니다. - 성장률/사망률과 인구 규모 간의 관계를 확인합니다. 이를 위해 출생/사망 모델과 성장률과 사망률의 모든 그럴듯한 쌍별 조합으로 초기화된 시뮬레이션을 사용합니다. 이 시뮬레이션에는 T0 제어판의 평균 살아있는 세포 수(x0), 분석 길이(t) 및 그럴듯한 증식률(p) 및 사멸률(d)의 사용자 정의 범위가 필요합니다.


- 성장률에 대한 그럴듯한 값을 결정하려면 가장 높은 성장률로 1/T(1/T)당 세포 집단 배가의 처리되지 않은 성장률부터 시작하고 가장 낮은 성장률로 0을 시작합니다. 이 범위를 500개의 동일한 간격의 세그먼트로 나눕니다. 사망률에 대해서도 유사한 작업을 적용하여 0에서 1까지의 범위를 테스트할 수 있습니다.
참고: 500 속도의 모든 쌍별 조합은 모든 프로그래밍 환경을 사용하여 수행할 수 있지만 일부 소프트웨어를 사용하는 경우 계산 강도가 너무 높을 수 있습니다. 이를 50 성장률과 사망률의 모든 쌍별 조합으로 줄이면 추론의 정밀도를 줄이면서 이 문제를 완화할 수 있습니다. 스프레드시트 템플릿이 이 프로토콜과 함께 제공되었습니다.
- 성장률에 대한 그럴듯한 값을 결정하려면 가장 높은 성장률로 1/T(1/T)당 세포 집단 배가의 처리되지 않은 성장률부터 시작하고 가장 낮은 성장률로 0을 시작합니다. 이 범위를 500개의 동일한 간격의 세그먼트로 나눕니다. 사망률에 대해서도 유사한 작업을 적용하여 0에서 1까지의 범위를 테스트할 수 있습니다.
- 시뮬레이션된 각 확산(p) 및 사망(d) 속도 쌍에 대한 GR 및 LF 값(위에서 설명)을 결정합니다.
참고: 7.2-7.3단계는 증식 및 사망률의 각 이론적 쌍을 포함하는 테이블을 생성하며, 해당 이론적 쌍에 대해 계산된 GR 및 LF 값을 포함합니다. 이것은 실험적으로 관찰된 GR 및 LF 값 쌍을 한 쌍의 약물 유발 증식 및 사망률과 연관시키는 조회 테이블로 작동합니다. 증식 및 사멸률에 대한 500개 이상의 개별 값을 테스트하면 수치적으로 더 정확한 값을 얻을 수 있지만 이 수준의 정밀도는 분석의 정밀도를 넘어설 수 있습니다. - 룩업 테이블에서 실험적으로 계산된 각 GR/LF 쌍과 각 이론적 GR/LF 쌍 사이의 쌍별 거리를 계산합니다. 실험적으로 관찰된 GR/LF 값 쌍까지의 최소 거리를 가진 이론적 쌍을 실제 약물 유발 증식 및 사망률로 추론합니다.
8. 경로 선택성 화학 억제제를 사용한 사멸 경로 결정
참고: 화학적 억제제만으로는 특정 약물의 사멸 기전을 확정적으로 결정하기에 충분하지 않습니다. 사멸 경로의 화학적 억제제를 사용하여 형태학적 평가, 경로 특이적 생화학적 마커 및 유전적 의존성 평가를 포함할 가능성이 있는 후속 실험에서 어떤 생화학적 또는 표현형 반응을 탐색해야 하는지 결정해야 합니다.
- 관심 있는 세포주에서 각 세포 사멸 억제제의 용량을 최적화하려면 관심 있는 사멸 경로의 표준 활성제와 관련하여 억제제 농도의 용량 범위를 테스트합니다(표 2). 이상적으로는 선택한 억제제 용량이 그 자체로 세포 생존율에 영향을 미치지 않아야 합니다(GR 지표 확인).
참고: 모든 세포주가 모든 사멸 경로를 활성화할 수 있는 것은 아닙니다. 다양한 사멸 경로의 활성제/억제제를 테스트할 때 생화학적 또는 표현형 검증이 필요할 수 있습니다. - 억제제 스크린을 위한 최적의 도금 레이아웃을 고려하십시오. 배치 효과를 최소화하려면 각 약물을 평가 중인 억제제와 동일한 플레이트에 보관하고 복제는 별도의 플레이트에 보관합니다.
- 3.3단계와 유사하게 96웰, 광학 바닥, 검은색 벽 플레이트의 각 웰에 원하는 수의 세포를 파딩합니다. 억제제와 약물의 부피를 고려하여 도금 부피를 80μL로 줄입니다.
- 완전한 배지에서 테스트할 억제제의 10x concertation을 준비하고 각 웰에 10μL를 추가합니다. 사멸 경로 억제제로 세포를 2-4시간 동안 전처리합니다. 그런 다음 관심 약물을 추가하고 3-5단계와 같이 형광 판독값을 얻습니다.
- 억제제의 효과를 평가하기 위해 사망 발병 시간(DO) 및/또는 최대 치사 비율의 변화를 평가합니다. DO는 LED(Lag Exponential Death) 모델에서 추론된 매개변수입니다. 5.6 - 5.7단계를 참조하십시오.
결과
이 프로토콜을 사용하여 HDAC 억제제인 Belinostat에 대한 U2OS 세포의 민감도를 조사했습니다. 이러한 실험은 2μM SYTOX Green을 사용하여 죽은 세포를 표지하여 수행했습니다(그림 1A). 운동 판독은 130 게인 설정에서 형광판 판독기를 사용하여 수행되었습니다(그림 1B). 세포를 PBS의 1.5% Triton-X 용액에서 2시간 동안 용해시켰습니다(그림 1C-D).
FLICK 프로토콜은 개체군 증가율 및 약물 유발 세포 사멸률에 대한 통찰력을 제공합니다. 이러한 인사이트는 개별적으로 또는 약물 GRADE 시각화 및 분석 프레임워크를 사용하여 함께 볼 수 있습니다(그림 1E). GR 값을 사용하여 Belinostat 감도를 평가하면 1μM Belinostat의 GR 값이 약 0임을 알 수 있습니다(그림 2A). GR 척도에서 양수 값은 인구 증가 속도를 보고하고 음수 값은 인구 감소 속도를 보고합니다. 따라서 GR 값이 0이면 개체군이 정지 상태(즉, 세포증식 억제 약물 반응)를 유지하고 있음을 나타냅니다. 세포정지에 대한 기존의 해석을 감안할 때, 1μM Belinostat는 세포 사멸 없이 완전한 성장 억제를 유발한다는 결론을 내릴 수 있습니다. 그러나 약물 유발 치사율을 평가한 결과, 1μM Belinostat가 이 분석 과정에서 약 50%의 치사율을 일으킨 것으로 나타났습니다(그림 2B). 대부분의 경우 이 두 가지 통찰력은 사과 대 사과 방식으로 비교할 수 없는 서로 다른 실험에서 파생됩니다. 중요한 것은 FLICK 분석을 사용하면 두 가지 통찰력이 동일한 실험 데이터에서 파생되지만 집단 성장과 세포 사멸에 대한 Belinostat의 효과를 포착하기 위해 다르게 분석된다는 것입니다.
이 두 가지 통찰력은 GRADE 분석 방법을 사용하여 통합할 수도 있습니다(그림 2C). 약물 GRADE는 평균 사망률에 비례하는 치사율(Lethal Fraction)과 순 인구 증가율인 GR 값을 병치합니다. 이러한 방식으로 데이터를 통합하면 약물이 성장 억제 및 사멸 활성화를 어떻게 조정하는지 시각화할 수 있습니다. GRADE 플롯 내에서: 사멸을 활성화하지 않고 성장을 억제하는 약물(즉, 일반적인 해석을 사용하는 세포증식 억제제)은 상단 경계를 차지하고, 세포 증식 속도를 변경하지 않고 사멸만 하는 약물은 오른쪽 경계를 차지하며, 먼저 성장 억제를 유발한 후 성장이 정지된 세포의 사멸을 유발하는 약물은 왼쪽 경계를 차지합니다(즉, 이위상 약물; 그림 2C). 이러한 경계에 속하는 GR/LF 반응을 유발하는 약물은 세포 사멸을 활성화하는 동시에 증식을 어느 정도 억제하는 것으로 추론할 수 있습니다(즉, 동시 약물)3. 중요한 것은 GR/LF 공간 내에서 각 약물 반응의 위치를 사용하여 각 약물 농도에 대한 실제 증식률(p)과 평균 사망률(d)을 계산할 수 있다는 것입니다(그림 2D-E). GR 값은 실제 세포 증식률이 아니라 순 개체군 성장률(즉, 실제 증식률과 약물 유발 사망률을 결합한 순 효과)입니다. 또한 GRADE 분석에서 생성된 사망률(평균 사망률인 d)은 LED 역학 분석의 사망률 매개변수(DR, 최대 속도 또는 최대 사망률)와 구별됩니다.
FLICK 분석은 약물 유발 치사 분획과 같은 사망 특이적 데이터를 생성할 수 있습니다. 그러나 이러한 데이터는 세포 사멸 메커니즘에 구애받지 않습니다. FLICK을 사용하여 세포 사멸 메커니즘에 대한 통찰력을 얻기 위한 가장 간단한 방법은 사멸 경로 특이적 억제제의 포함으로 인해 치사량 분획 역학이 어떻게 변경되는지 확인하는 것입니다. 관련 사멸 기전을 억제하는 것은 약물에 의한 치사율을 감소시키거나 사멸 발병 시간을 지연시키는 결과를 낳아야 한다. 여기에서는 50μM의 pan-caspase 억제제 z-VAD가 1μM Belinostat에 의해 유도된 치사율의 약 50%를 구출한다는 것을 보여줍니다(그림 2F). 중요한 것은 약물과 마찬가지로 억제제는 특이성이 제한되어 있으며 실수로 다른 사망 메커니즘을 억제하거나 악화시킬 수 있다는 것입니다. 따라서 결정적인 결론을 내리기 전에 이러한 데이터는 세포 형태학, 활성화의 생화학적 마커 측정 및/또는 경로 특이적 유전적 의존성 평가와 같은 다른 통찰력으로 보완되어야 합니다14.
FLICK 기반 평가 및 GRADE 기반 분석에 의해 약물 반응의 해석이 어떻게 바뀔 수 있는지 공식적으로 설명하기 위해 다음으로 다양한 유형의 성장/사멸 조정을 특징으로 하는 세 가지 약물을 탐색했습니다. 팔보시클립(Palbociclib)은 세포증식 반응(cytostatic response, 세포 사멸이 없는 성장 억제)을 유발하는 Cdk4/6 억제제입니다. 캄토테신(Camptothecin)은 토포이소머라제 I 억제제(topoisomerase I inhibitor)로 이위상 반응(growth inhibition, 고용량에서 세포 사멸)을 일으킵니다. Belinostat는 동시 반응(모든 용량에서 부분적인 성장 억제 및 사멸 활성화, 그러나 용량에 따라 다양한 비율)을 유발하는 HDAC 억제제입니다. 이러한 데이터에 대한 기존 분석을 사용하면 세 가지 약물 모두 U2OS 세포의 상대적 생존율을 크게 감소시키는 것을 관찰할 수 있습니다(그림 2G). 팔보시클립의 효능이 눈에 띄게 떨어지긴 하지만, 이러한 데이터로는 팔보시클립이 세포 사멸을 활성화하지 못한다는 것은 명확하지 않습니다. 약물이 순 인구 증가율에 어떤 영향을 미치는지를 보고하는 GR 기반 분석을 사용하면 팔보시클립으로 투여된 집단은 모든 용량에서 계속 확장되는 반면, 고용량의 캄토테신 또는 벨리노스타트는 인구 감소를 초래하므로 이러한 약물의 고용량 투여 후 세포 사멸이 활성화되었음에 틀림없다는 것을 보다 정확하게 해석할 수 있습니다(그림 2H)). 그러나 GR 기반 분석은 성장과 사멸 사이의 다양한 조정을 설명하지 못하며, 이는 주어진 약물에 의해 활성화된 세포 사멸 수준에 대한 잘못된 결론으로 이어질 수 있습니다. 예를 들어, GR 데이터를 기반으로 Camptothecin이 -0.25의 GR 값을 나타내고 Belinostat가 GR 값이 0이라는 점을 감안할 때 1μM Camptothecin이 1μM Belinostat보다 더 큰 정도로 세포 사멸을 활성화한다는 결론을 내릴 수 있습니다. -0.25 GR 값은 처리되지 않은 개체군이 확장되는 속도만큼 개체군이 25% 감소하고 있음을 의미합니다. GRADE 기반 시각화 및 분석을 사용하여 Camptothecin과 Belinostat의 성장/사망 조정이 다르다는 것을 관찰할 수 있습니다. 따라서, 1μM 용량에서 두 Belinostat 모두 Camptothecin보다 더 높은 비율로 사멸을 활성화하지만(Belinostat의 경우 0.8% death/hr, Camptothecin의 경우 0.7% death/hr), Camptothecin 투여군은 이 약물에 의해 유도된 더 뚜렷한 성장 억제로 인해 더 빠르게 감소합니다(그림 2I).

그림 1: FLICK 프로토콜의 흐름도. (A) 세포 희석액을 생성하여 마이크로플레이트 리더에서 형광 측정의 DNA 염료 농도 및 선형성을 최적화합니다. (B) U2OS 세포에서 2μM SYTOX Green에 대한 세포 수에 걸쳐 적정을 얻습니다. 이득 130은 모든 셀 번호에서 가장 선형적인 신호를 제공합니다. (C) 10x 대물렌즈를 사용하여 시간이 지남에 따라 1.5% Triton-X로 용해된 U2OS 세포의 상 이미지. 박스형 영역은 SYTOX Green 형광을 확대한 모습입니다. 배율 막대는 모든 이미지를 대표합니다. (D) 표시된 시간에 용해 후 SYTOX 녹색 신호. 데이터는 3개의 생물학적 복제에 대한 평균 +/- SD입니다. (E) 설정부터 분석까지 분석에 대한 개념적 개요. 주요 단계는 약물 희석 플레이트를 만들고, 다중 웰 플레이트에서 세포를 투여하고, 시간 경과에 따른 형광 측정값을 획득하고, 성장 및 사멸 지표를 계산하는 것입니다. 패널 B 및 D의 경우 데이터는 n = 3개의 독립적인 생물학적 복제물에 대해 평균 ± SD입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: FLICK 분석 및 GRADE 기반 분석을 사용한 약물 반응 특성화. (A) 용량 대비 HDAC 억제제인 Belinostat에 대한 GR 메트릭. 표시된 데이터는 66시간인 분석 종점에서 가져온 것입니다. 1μM에서 Belinostat의 GR 값은 약 0으로 세포증식 반응(cytostatic response)을 나타냅니다. (B) Belinostat에 대한 치사 분획 역학. 1μM에서 Belinostat는 GR 값으로 포착되지 않는 거의 50%의 치사율을 유도합니다. (C) 가능한 모든 성장 및 사망률의 시뮬레이션을 기반으로 한 GRADE 위상 다이어그램. 시뮬레이션은 이 분석에서 U2OS의 배가 시간(27.12시간)을 기반으로 했습니다. (D) 분석 종말점에서 투여량에 걸친 Belinostat에 대한 GRADE 플롯. (E) Belinostat의 투여량에 대한 GRADE 추론 성장(p, 시간당 인구 두 배) 및 사망(d, 시간당 사망하는 인구 비율) 비율의 막대 그래프. 속도는 (C)의 시뮬레이션된 속도와 (D)의 데이터로부터 결정되었습니다. (F) 자가사멸 억제제 z-VAD의 50μM(보라색) 또는 없음(검은색)으로 처리된 1μM Belinostat에 대한 치사 분획 플롯. ΔLF = 0.18은 대조군의 평균 최대 LF에서 억제제 처리 조건의 평균 최대 LF를 빼서 계산했습니다. (G) 72시간 노출 후 3가지 약물에 대한 상대적 생존율(약물 처리 상태의 살아있는 세포의 수를 치료되지 않은 상태의 살아있는 세포의 수로 나눈 값). 모든 약물은 세포 생존력을 감소시키지만 세포 사멸이 반응에 기여하는지는 알려져 있지 않습니다. (H) (G)의 3가지 약물에 대한 GR 지표. 이 데이터는 3가지 약물 중 2가지가 GR < 0으로 표시된 개체군 감소를 초래한다는 것을 보여줍니다. 세포 사멸의 기여도는 아직 불분명하지만, GR 값이 음수일 때 세포 사멸이 활성화되었음에 틀림없습니다. (I) 3가지 약물에 대한 GRADE 추론 요율. 팔보시클립은 치명적이지는 않지만 성장 정지를 일으킵니다. Camptothecin은 biphasic입니다. 더 낮은 용량은 성장 정지를 초래하고 더 높은 용량은 성장 정지를 초래한 후 치사를 초래합니다. 벨리노스타트는 우연의 일치로 투여되는 약물입니다. 용량 범위는 GRADE 플롯의 중간을 가로지르며, 이는 모든 용량이 성장 교란 및 세포 사멸 활성화를 초래함을 나타냅니다. 모든 패널의 데이터는 3개의 독립적인 생물학적 복제 실험에 대한 평균 ± SD입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 염료 | 운동의 | 끝점 |
| 사이톡스 그린 | Y | Y |
| 셀톡스 그린 | Y | Y |
| 넥스팟: 500/515, 594/615, 750/780 | Y | Y |
| 요요-3 | Y | Y |
| 프로피듐 요오드화물 | N | Y |
| 7-AAD | N | Y |
표 1: DNA 염료 및 FLICK에서의 응용. 많은 유형의 염료를 종말점 전용 FLICK 분석에 사용할 수 있습니다. 분석 전반에 걸쳐 지속적으로 배양해야 하는 조건을 충족하는 염료의 수가 더 적습니다. 녹색이 아닌 염료를 포함한 다른 염료는 깊이 특성화되지 않았으므로 사용하기 전에 신중하게 평가해야 합니다.
| 죽음의 경로 | 억제제 | 일반적인 복용량 |
| 내인성 세포사멸(Intrinsic apoptosis) | ZVAD-FMK(마이크로소프트 파이엠케이) | 50 마이크로미터 |
| 외인성 세포사멸(Extrinsic apoptosis) | Z-IETD | 30 μM |
| 페롭토시스 | 페로스타틴-1 | 10 μM |
| 괴사(Necroptosis) | 네크로스타틴-1 | 50 마이크로미터 |
| 파이롭토시스 | VX-765 시리즈 | 50 마이크로미터 |
| 파르타나토스 | 루카파립 | 1 μM |
| 자가포식 의존성 세포 사멸 | 하이드록시클로로퀸(HCQ) | 10 μM |
| 리소좀 의존성 세포 사멸 | E-64D | 10 μM |
| 쿠프로토시스 | 테트라티오몰리브덴트산염(TTM) | 5 μM |
| 옥세이토시스 | N-아세틸-l-시스테인(NAC) | 2 밀리엠 |
| MPT에 의한 괴사 | 사이클로스포린 A(CsA) | 10 μM |
표 2: 세포 사멸 경로 및 그 억제제. 세포 사멸 억제제는 각 사멸 경로에 대한 표준 활성제에 대해 검증되어야 합니다. 선택된 억제제 용량은 이상적으로는 세포 생존율에 영향을 미치지 않아야 합니다.
보충 표 1: GRADE 계산기. FLICK 기반 GR 및 FV 데이터에서 약물로 인한 성장 및 사망률을 계산하기 위한 스프레드시트. 사용자는 GR 및 FV 데이터와 함께 분석 길이(시간), 세포 배가 시간(시간), 시작/종료 집단 크기를 입력해야 합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
FLICK 분석은 세포 집단의 성장과 사멸에 대한 약물의 효과에 대한 포괄적인 평가를 생성하기 위한 강력한 방법입니다. 이 방법은 세포를 직접 계수하지 않기 때문에 프로토콜의 중요한 단계는 트리톤 투과화 단계 동안 분석 선형성을 보장하고 용해를 완료하는 것입니다. 정확한 투과화 시간은 이 프로토콜에서 강조된 바와 같이 시각적으로 식별하거나 시간 경과에 따른 플레이트 형광을 읽고 신호가 안정될 때를 식별하여 정량적으로 식별할 수 있습니다. 그러나 우리의 경험에 비추어 볼 때, 의심스러울 때 더 오래 기다릴 필요가 없는 것으로 보입니다: FLICK 분석에 사용한 DNA 염료의 경우, 신호 강도는 용해 후 며칠 동안 안정적으로 유지됩니다. 우리는 정기적으로 SYTOX Green 핵산 염색을 사용하며, 표 1에 언급된 각 형광단을 검증하지는 않았지만 다른 여러 프로토콜에서 효과적인 사용을 입증했습니다 13,15,16. 다양한 세포 수와 기기 설정에서 주어진 세포 침투성 DNA 염료의 선형성을 평가하면 강력하고 정량적으로 신뢰할 수 있는 데이터를 보장할 수 있습니다.
일부 세포주는 다른 세포주보다 용해가 더 어려우며, 한 세포주에 대한 용해 시간이 다른 세포주와 같다고 가정해서는 안 됩니다13. 일부 세포주의 경우 밀도가 더 높으면 세포 용해로 인해 세포가 시트처럼 웰에서 들어 올려져 플레이트 리더 오류가 발생할 수 있습니다. 이는 시작 파종(seeding) 밀도를 줄이거나 용해 후 및 판독 전에 웰을 피펫 혼합하여 방지할 수 있습니다. 용해 시간 또는 용해 효율을 개선하기 위해 Triton-X 솔루션을 더 높거나 더 낮은 비율로 조정할 수 있지만, 이것이 필요한 시나리오는 아직 발생하지 않았습니다.
실험 설계 및 약물 도금 레이아웃은 강력한 성장 데이터가 필요한 경우 신중하게 고려해야 합니다. FLICK 분석을 위한 약물 희석 플레이트를 만드는 것은 다른 96웰 플레이트 분석과 동일합니다. 그러나 플레이트 랜드스케이프의 여러 부분을 포괄하는 많은 컨트롤을 포함하면 성장 역학에 대한 강력한 통찰력을 생성하는 데 도움이 되며 체계적인 성장 변동이 존재하는지 확인하는 데 도움이 될 수 있습니다. 플레이트의 바깥쪽 웰을 피하면 이러한 웰은 온도, 산소 또는 습도 변동에 더 민감하여 성장에 영향을 미치기 때문에 보다 정확한 성장 데이터를 생성할 수 있습니다. 마지막으로, 복제 플레이트에서 약물의 위치를 전환하는 것과 같은 의사 무작위 약물 복용 패턴을 사용하면 약물 작용에 대한 보다 정확한 평가를 제공할 수 있습니다.
FLICK 분석의 정확한 사용을 위해 몇 가지 제한 사항이 있습니다. 이러한 제한 사항은 엔드포인트 데이터가 실험적으로 관찰된 값이기 때문에 대부분 운동 추론과 관련이 있습니다. 살아있는 세포 증식 역학(live cell proliferation kinetics)과 사멸 역학(death kinetics)은 모두 약물 치료 상태에서 시간 경과에 따른 살아있는 세포의 성장 궤적에 관한 몇 가지 가정에 의존합니다. 이 프로토콜에서는 이러한 성장 궤적을 기하급수적이며 시간이 지남에 따라 균일한 속도로 설명합니다. 이러한 특징은 약물이 없을 때 관찰될 수 있지만 약물이 있는 곳에서는 준수되지 않을 수 있습니다. FLICK 방법은 성장 궤적에 대한 가정이 항상 정확하기 때문이 아니라 약물 투여 집단이 크게 증식하지 않는 경우(즉, 분석 기간 동안 2 또는 3 미만의 집단 배가) 잘못된 가정의 영향을 최소화하기 때문에 정확합니다. FLICK에서 정확하게 프로파일링할 수 없는 약물은 발견되지 않았습니다. 그러나 이 이론은 세포 배가 되는 시간의 몇 배에 걸쳐 사멸이 매우 천천히 발생하는 약물에 대해 FLICK 분석이 더 이상 정확하지 않을 것임을 시사합니다(참고: 이러한 기능을 가진 약물은 확인되지 않았습니다3). 그러나 전체 모집단의 성장 역학에 대한 잘못된 가정의 영향은 경험적으로 측정된 데드 셀 값과 분석 종료 시 실험적으로 결정된 최종 총 세포 값에 의해 제한되기 때문에 LED 적합에서 얻은 사멸 시작 시간 또는 최종 최대 LF 값에 영향을 미치지 않습니다.
이러한 한계에도 불구하고 FLICK 분석은 다른 약물 반응 방법을 사용하여 생성하기 어려운 통찰력을 제공합니다. 대부분의 약물 반응 방법은 살아있는 세포의 수에 비례하는 신호를 생성하며, 이러한 방법은 약물이 순 개체군 증가율(즉, GR 값 또는 이에 상응하는 값)에 미치는 영향을 정량화하는 데 사용할 수 있지만 약물의 세포독성과 세포증식 효과를 정확하게 구별할 수는 없습니다. 또는 살아있는 세포와 죽은 세포를 모두 측정하는 현미경 기반 분석은 확실히 포괄적인 그림을 생성할 수 있습니다. 그러나 현미경 기반 분석은 세포가 파편으로 분해된 후 죽은 세포를 계수하는 데 어려움을 겪을 수 있으며, 이는 많은 비세포사멸 형태의 세포 사멸의 맥락에서 빠르게 발생합니다. FLICK의 주요 특징은 사체 세포를 직접 계수하는 대신 총 사각 세포 형광을 집계하는 플레이트 리더에서 측정이 이루어진다는 것입니다. 따라서 FLICK의 사세포 신호는 비세포사멸(non-apoptotic cell death)의 맥락에서 유일하게 필수적인 사멸세포의 온전한 세포성에 의존하지 않습니다. 또한 FLICK 분석의 고유한 특징은 동일한 시약을 사용하여 살아있는 세포 집단과 죽은 세포 집단을 모두 측정할 수 있다는 것입니다. 따라서 FLICK 분석은 동일한 감도로 살아있는 세포와 죽은 세포를 측정합니다. 이 기능을 사용하면 GRADE 방법을 사용하여 분석할 수 있으며 GRADE의 동시 성장 및 사망률 계산의 정확도를 높일 수 있습니다. 포함된 보충 표 1 에는 약물 GRADE의 시각화에 사용되는 템플릿이 포함되어 있으며, 여기서 사용자는 분석 길이, 7.1단계에서 계산된 성장률, 대조 조건의 시작 및 종료 세포 수 또는 형광 판독값, 5.4 및 6.3단계에서 계산된 LF 및 GR을 입력할 수 있습니다. 이 파일에는 50개의 성장률과 사망률의 모든 쌍별 조합에 대한 시뮬레이션이 포함되어 있으며, 이는 사용자 정의 매개변수를 기반으로 자동으로 업데이트됩니다. 이 템플릿은 약물 반응과 GRADE 추론 성장(p) 및 사망률(d)에 대한 GRADE 기반 시각화를 생성합니다.
향후 연구에서는 3D 세포 배양 시나리오 또는 종양 유래 오가노이드의 약물 반응 평가에서 FLICK 분석의 적용을 모색해야 합니다. FLICK 데이터의 선형성 및 감도는 이러한 맥락에서 심층적으로 조사되지 않았지만 이론적으로 FLICK 분석은 3차원 샘플에 형광을 통합하기 위해 몇 가지 수정을 가하면 효과적이어야 합니다. 또한 세포 특이적 표지를 추가하면 공동 배양에서 두 개 이상의 세포 유형을 구별하는 데 도움이 됩니다. 이러한 발전은 면역 세포와 암세포의 상호 작용을 탐구하는 데 유용할 것입니다. 마지막으로, SPARKL과 같은 분석은 FLICK 분석 형식의 죽음 특이적 리포터의 사용에 영감을 주어 세포 사멸 통찰력의 정확성을 유지하면서 처리량을 향상시킬 수 있습니다11,16.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
약물 반응 평가에 대한 우리 실험실의 관점에 기여한 Lee Lab의 전현직 구성원 모두에게 감사드립니다. 이 작업은 미국 국립보건원(National Institutes of Health)에서 MJL(R21CA294000 및 R35GM152194)에 대한 자금 지원으로 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
참고문헌
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유