Method Article
FLICKアッセイを用いた薬物反応の包括的解析
要約
このプロトコルでは、薬物反応を評価するためにFLICKアッセイを使用する方法について説明しており、このアッセイを使用して薬物誘発増殖率と死亡率を計算し、薬物誘発性細胞死のメカニズムを評価するための詳細な手順が含まれています。
要約
薬効を理解するためには、薬物による細胞死の程度を特徴づけることが非常に重要です。薬物誘発性細胞死のレベルを定量化する取り組みは、それぞれが独自の活性化タイミングと生化学的特徴を持つ、分子的に異なる12種類以上の制御死の存在によって課題となっています。さらに、一部の壊死性死サブタイプでは、特徴的な特徴は一過性にしか観察されず、細胞の破裂により急速に失われます。したがって、死経路特異的アッセイを組み合わせて使用する場合でも、細胞死の総量や各死サブタイプの相対的な寄与を正確に定量することは困難です。もう一つの問題は、多くの死亡特異的アッセイが薬物が細胞増殖にどのように影響するかを無視しているため、薬物治療を受けた集団が拡大しているのか縮小しているのかを解釈するのが難しいことです。FLICKアッセイは、刺激後の細胞死の総レベルを、死に特異的でありながら、活性化された死の種類に大きく依存しない方法で定量化することができます。さらに、FLICKアッセイは、総集団サイズと細胞増殖速度に関する情報を保持します。この原稿では、FLICKアッセイの基本的な使用法、異なるタイプの生物学的材料を使用する場合のこのアッセイのトラブルシューティング方法、および観察された薬物応答に対する各タイプの細胞死の寄与を定量化するためのFLICKアッセイの使用方法について説明します。
概要
抗がん剤の場合、薬剤感受性の前臨床評価には、一般に、薬物が培養中の細胞の生存率にどのように影響するかを調べることが含まれます1。薬物曝露後の細胞生存率は、薬物による細胞増殖の阻害と細胞死の活性化という、少なくとも2つの別々の効果の産物です2。残念ながら、細胞死は持続的な薬物応答に必要な重要な特徴ですが、標準的なアプローチでは、薬物が細胞死を活性化する程度を明確にすることはできません3。
一般的な薬物反応アッセイには、細胞を直接カウントするアッセイ(コールターカウンター、フローサイトメトリーや顕微鏡法の一部の使用)、細胞の増殖能力を定量化するアッセイ(コロニー形成アッセイなど)、代謝活性を定量化するアッセイ(CellTiter-Glo、テトラゾリウムベースのMTTまたはMTSアッセイなど)などがあります。これらのアッセイに共通する特徴は、生成されるデータが生細胞の数に比例することです。薬物は増殖阻害と細胞死をどのように調整するかによって大きく異なるため、薬物曝露後の生細胞の数は、薬物誘発性細胞死のレベルについて信頼性の低い洞察を提供します3。さらに、がん細胞は一般に細胞培養で急速に増殖するため、細胞死を誘発することなく、未処理の集団と比較して生細胞の数を劇的に減らすことができる4。したがって、中心的な欠陥は、生細胞と死細胞の両方の数を測定しなければ、細胞死の程度を定量化できないことです。
薬物曝露後の死細胞の数を定量化することは、いくつかの理由から正確に行うことは困難です2。まず、12を超える制御された細胞死経路が存在します5、6、7、8。一般に、制御された細胞死の各タイプを同定するための生化学的マーカーが存在しますが、これらのマーカーの特異性はさまざまであり、単一のアッセイを使用してすべての細胞死サブタイプを同時に定量することはできません。第二に、細胞死の各形態の活性化のタイミングは、状況によって非常に劇的に異なる可能性があるため、死が経時的に定量化されない限り、全体像は明らかになることができない9,10。細胞死を定量するための多くの生化学的アッセイでは、エンドポイント測定が行われるため、速度論的データの生成は困難であり、コストによって制限される可能性があります。第3の問題は、死細胞自体が生細胞状態と解離細胞破片との間の一時的な中間状態であることである。死んだ細胞の安定性は死のサブタイプによって異なり、アポトーシスなどの一部のタイプでは比較的安定した死体が作成されますが、他のタイプの死では急速な溶解を引き起こします。したがって、死細胞の収集とカウントを必要とする死を定量化する方法も、細胞死の偏った理解を生み出します。最後に、4つ目の限界は、細胞死の程度を定量化する生化学的アッセイでは、通常、薬物が増殖をどのように変化させるかについての洞察が得られないことです。したがって、全体的な人口規模、そして重要なことに、人口が拡大しているのか減少しているのかは解釈できません。
STACKやSPARKLなどの顕微鏡ベースのアッセイの中には、生細胞と死細胞の経時的な測定に効果的であり、これらのアッセイは薬物誘発性細胞死に関する包括的な洞察を生み出すことができます10,11。しかし、これらのアッセイにはIncuCyte顕微鏡などの特殊な機器が必要であり、スループットやこれらのアプローチへのアクセスに限界が生じます。さらに、顕微鏡法に基づく技術では、死細胞が実験期間中ずっと顕微鏡の焦点面に留まる必要があるため、死細胞がプレートからの接着を失った場合や、死んだ細胞が時間の経過とともに崩壊するにつれて、死細胞を定量化する能力が損なわれます。同様に、顕微鏡法に基づくアッセイは、細胞が特定の焦点面に出入りする浮遊培養の文脈で適用されると、課題に直面します。
上記で強調された問題に対処するために、FLICK(Fluorescence-based and Lysis-dependent Inference of Cell Death Kinetics)12,13と呼ばれるアッセイを生成しました。FLICKアッセイの目標は、細胞の死滅方法に関係なく、薬物誘発性細胞死のレベルを決定することです。FLICK法は、細胞不透過性色素を使用し、その蛍光はDNA結合に依存します。FLICKの主な特徴は、これらの蛍光色素を使用して、アクセス可能なDNAにより、死細胞の経時的な蓄積を標識し、その後、アッセイの最後には、メカニカルな界面活性剤ベースの溶解により、任意の生細胞を透過化することです。これらのデータを数学的モデリングと組み合わせることで、生細胞集団と死細胞集団の両方を連続的な時間分解能で定量化し、死細胞の収集や取り扱いを必要とせずに行うことができます。さらに、プレートリーダーを使用して死細胞の蛍光を評価することで、死細胞が無傷のままであることなく死細胞の評価が可能になり、細胞破裂を引き起こす壊死形態の死に対するバイアスが軽減されます。最後に、FLICKアッセイはプレートの取り扱いを最小限に抑え、速度論的測定値を迅速に生成できるため、ハイスループットの薬物スクリーニングが可能になります。このプロトコルでは、FLICKを使用して薬物誘発性増殖速度、死亡率、および/または細胞死のメカニズムを推測する方法など、FLICKアッセイの使用に焦点を当てています。
プロトコル
1. 目的細胞株ごとの透過化時間の最適化
注:記載されている容量と量は、1つの細胞株を最適化するためのものです。これらの値は、試験する細胞株の数に基づいてスケールアップする必要があります。
- 96ウェルの光学底部黒壁プレートの各ウェルに所望の細胞数をプレート化します。100 μLの完全培地を加え、細胞をプレートに一晩接着させます。
注:播種した細胞数は、細胞の増殖速度、最適な密度、およびアッセイの長さを考慮して最適化する必要があります。72時間にわたって測定された接着がん細胞株の典型的な開始細胞数は、ウェルあたり1500〜5000細胞です。例えば、U2OS細胞は、10% FBS、2 mMグルタミン、および1%Pen-Strepを添加したDMEMでウェルあたり2000細胞でプレーティングし、標準条件(5% CO2、37°C、湿度)で一晩培養することができます。 - 15 mLの円錐形遠心チューブで、リン酸緩衝生理食塩水(PBS)に溶解したTriton-Xの1.5%溶液1.5 mLを調製します。1.5% Triton-X 溶液を最高速度で 5 秒間ボルテックスします。37°Cのウォーターバスに30分以上入れます。
- 溶液を目視検査し、Triton-Xが数秒間ボルテックスして完全に溶解したことを確認します。ソリューションが均一であることを確認します。
- 1.5% Triton-X溶液10 μLを、プレーティングしたセルの各ウェルに加えます。混ぜないでください。
注:この段階では、気泡が形成されるため、繰り返しピペッティングを使用して混合することは望ましくありません。 - 細胞を5% CO2, 37°Cに、湿度細胞培養インキュベーターで少なくとも1時間戻します。
注:Triton-X溶液の適用後、ほとんどの細胞株は2〜3時間で完全に溶解されます。SYTOX Greenなどの色素を使用する場合、最大24時間待っても蛍光シグナルへの影響は最小限に抑えられます。Triton-Xの割合を増やすと、溶解が困難な細胞株のインキュベーション時間が短縮される可能性があります。 - 10倍対物レンズを使用して、光学顕微鏡で細胞の形態を観察します。細胞体が見えなくなるまで、1時間に1回細胞を検査します。細胞の透過化に必要な時間を記録します。
注:蛍光顕微鏡が利用可能な場合は、SYTOX Greenなどの細胞不透過性DNA色素を使用して溶解を視覚化できます。この色素は、Triton-X溶液に最終濃度の10倍で添加することができ、透過化は蛍光顕微鏡法で確認することができます。
2. DNA染色剤の選抜とキャリブレーション
注:FLICKアッセイの要件は、DNA結合依存性でシグナルを放出し、細胞生存率に影響を与えず、細胞数に比例するシグナルを生成する細胞不透過性蛍光色素の使用です。このプロトコルはSYTOX Greenを使用します。同様の特性を持つ他の色素もFLICKアッセイに適している場合がありますが、これらはそれぞれ評価およびキャリブレーションする必要があります。例については 、表 1 を参照してください。
- 選択したDNA色素について、メーカーの推奨に基づいて試験する濃度の範囲を決定します。
- 試験する濃度ごとに、180 μLの細胞培養培地に40,000個の細胞を3重にプレート化し、96ウェルの光学底部黒壁プレートの左端のカラムに沿ってプレート化します。残りのウェルに90μLの培地を加えます。
注:これは、プレートの列全体に線形セル滴定を生成するために使用されます。最初のウェルの最終濃度は20,000細胞/ウェルになります。光学ボトムプレートは、ウェルの上部から蛍光を読み取るプレートリーダーには必要ありません。細胞培養培地は、試験対象の細胞の増殖に使用される培地と同じである必要があります。例えば、U2OS細胞を使用する場合、40,000個の細胞を180μLのDMEMに再懸濁する必要があります。 - マルチチャンネルピペットを使用して、左端のカラムから90 μLを右の隣接するカラムに移すことにより、1:2の段階希釈を作成します。ピペット15倍で混ぜます。
- 手順 2.3 を繰り返し、2 列目から 3 列目へ、次に 3 列目から 4 列へ、というように移動します。プレートの最後から2番目の列で滴定を終了します。最後のカラムは細胞を含まないままにして、細胞培養培地のバックグラウンドシグナルを取得できるようにします。
- 最後から2番目のカラムでは、90 μLを取り出して、すべてのウェルにさまざまな数の細胞を含む90 μLの培地を確保します。細胞を37°Cの細胞培養インキュベーターで6時間接着させます。
- 試験する色素の濃度ごとにDNA色素の10倍溶液を調製し、DNA色素を希釈して目的の細胞の培養に使用する完全な増殖培地にします。ステップ1で説明したように、PBS中の1.5% Triton-X溶液1.5 mLを調製します。
- 96ウェルプレートの各ウェルに、10 μLの10x DNA色素を加えます。1.5% Triton-X溶液10 μLを各ウェルに添加して、細胞を透過化します。この溶液で細胞を最適な時間インキュベートします(これはステップ1で決定しました)。
注:Triton-Xの添加後、DNA色素の最終濃度は1倍よりわずかに少なくなりますが、これは重要ではありません。インキュベーション後、プレート全体を溶解する必要があります。手動細胞滴定は、最適なDNA色素濃度と蛍光を取得するための最適な設定を特定するために使用されます。 - 細胞滴定プレート全体の蛍光強度を測定します。DNA色素のメーカーが概説したさまざまな取得設定で蛍光を定量化します。使用するプレートリーダーに応じて、励起波長と発光波長、および/またはデジタルゲインを変更します。
- セルを含まない列の平均信号を差し引くことにより、各測定値からバックグラウンド信号を削除します。これらの値は、段階希釈プレートのウェルの右端の列にあります。
- 各取得設定に対するDNA色素の各濃度の直線性を決定します。線形性は、蛍光シグナルに対して細胞番号をグラフ化し、線形回帰を実行することで決定できます。決定係数(r2)は、蛍光シグナルが細胞数に線形に関連している度合いを報告します。
- 取得設定とDNA色素濃度のうち、直線性とダイナミックレンジの最適な組み合わせを選択します。
注:0から20,000セルまでのセル番号の全範囲を考慮してください。ゲインとDNA色素の濃度が、0〜1000細胞の低細胞数で直線的であることを確認してください。いくつかの機器の設定やDNA色素の濃度によっては、全範囲にわたって強い相関関係が見られる場合があります。ただし、死細胞の小さな変化を正確に測定できるように、ローエンドの感度が堅牢であることが不可欠です。
3. 薬剤処理プレートにおける細胞プレーティング、インウェル薬物適用、および死細胞蛍光の経時的測定
注:薬物希釈プレートは、実験ニーズに基づいて柔軟に設計できます。一般に、薬物希釈プレートには、1つまたは複数の薬物の対数または半対数希釈シリーズが含まれます。
- 実験に最適なめっきレイアウトを検討します。成長変動に関連するノイズを減らすために、96ウェルプレートの外側のウェルからの測定は避けてください。これは、外側のウェルがプレートの内側のウェルと比較して異なる温度、酸素化、および蒸発を経験するためです。
注:プレートのランドスケープ全体にコントロールを含めると、成長データの堅牢性が向上します。プレートの中央と端にコントロールウェルを含めることを検討してください。 - 実験に必要なプレート数を決定します。薬物投与時のウェルあたりの平均細胞数を確立するために、アッセイの開始時に溶解する追加のプレートを1つ含めます(すなわち、T0コントロールプレート)。
- 完全増殖培地に細胞懸濁液を調製します。
- 実験に必要なプレート数(ステップ3.2で計算)に基づいて、必要な細胞懸濁液の量を決定します。プレートをウェルあたり90μLで播種する場合、プレートあたり約10mLが必要です。必要なプレート数に10mLを掛けます。
注:リザーバーのデッドボリュームとピペッティング中のボリューム損失を考慮して、このボリュームを増やすことが望ましいです。たとえば、計算されたボリュームに1.2を掛けることができます。 - 細胞をカウントし、ウェルあたりの細胞数に基づいて細胞希釈液を調製します。たとえば、96ウェルプレートのウェルあたり90 μLの培地に2000個の細胞を播種することを目指す場合、合計3枚のプレートについて、実験に必要な細胞の総数を次のように計算します。

同様に、カウントされた細胞懸濁液が400,000細胞/mLの場合、実験に必要な細胞懸濁液の量は次のようになります。

- プレーティングに使用する細胞培養培地の量を、添加するカウントされた細胞懸濁液の量を差し引いて補正します。
36 mL培地 - 2 mL細胞懸濁液 = 34 mL補正培地容量
- 実験に必要なプレート数(ステップ3.2で計算)に基づいて、必要な細胞懸濁液の量を決定します。プレートをウェルあたり90μLで播種する場合、プレートあたり約10mLが必要です。必要なプレート数に10mLを掛けます。
- セロロジカルピペットを使用して、カウントした細胞懸濁液を正しい培地容量と混合します。この細胞懸濁液をV底試薬リザーバーに移します。
- マルチチャンネルピペットを使用して、96ウェルプレートの各ウェルに90μLの細胞懸濁液を加えます。血清ピペットでピペッティングを繰り返し使用して、試薬リザーバー内の細胞懸濁液を定期的に混合し、容量あたりの細胞の所望の濃度が維持されるようにします。
- 細胞を37°Cの細胞培養インキュベーターで一晩(通常は12〜24時間)接着させます。
- ステップ1で説明したように、PBS中の1.5% Triton-X溶液を調製します。この溶液の容量は、すべてのプレートを溶解するのに十分である必要があります。PBS中の1.5% Triton-Xは約1.5 mLで、96ウェルプレート1枚を溶解するのに十分です。
注:PBS中の1.5%Triton-X溶液は1週間以上安定しており、便宜上事前に製造することができます。 - 薬物希釈プレートの作成に必要な細胞培養培地の量を決定します。U字型またはV型の96ウェルクリアープレートに薬剤希釈プレートを調製します。
- 希釈した薬物の量が実験に十分であることを確認するために、ピペッティング中の容量損失を考慮して、必要な最小容量を増やしてください。例えば、96ウェルプレートの各ウェルに10μLの薬剤を添加し、実験に3枚のプレートが含まれる場合、ウェルごとに30μLの希釈薬剤が必要です。ウェルあたり40〜50μLの薬物製剤を調製し、体積損失を考慮します。T0コントロールプレートに必要な培地を考慮するために必要な培地の量に1.5mLを追加します。
- 選択したDNA色素の10倍濃度を完全増殖培地に調製します。この濃度は、ステップ 2.11 で選択した濃度に基づいています。この溶液の総体積は、ステップ3.8で計算されます。
- ステップ3.9のDNA色素+増殖培地を使用して、試験する各薬物の10倍の濃度を調製します。薬剤希釈プレートに各薬剤の最高用量のみを作成し、マルチチャンネルピペットで薬剤を連続希釈し、各ウェル間で15倍を混合します。
注:薬物希釈プレートで特定の薬物の最高用量を投与する場合、濃度が正確であることを保証するために、薬物の量を考慮する必要があります。. - マルチチャンネルピペットを使用して、ステップ3.10の薬剤+DNA色素溶液10 μLを細胞入りプレートに加えます。
注:段階希釈が作成された薬物希釈プレートの場合、薬物を最低濃度の薬物から始めて最高濃度まで細胞に薬物を添加する場合、マルチチャンネルピペットの先端を変更する必要はありません。ただし、マルチチャンネルピペットのチップは、各プレート間で交換する必要があります。また、チップが異なる薬物や高濃度の薬物を含むウェルで使用された場合は常に交換する必要があります。 - 薬剤を添加したすべてのプレートの蛍光を読み取ります。この蛍光読み取り値がT0読み取り値です(つまり、Time = 0 hの死細胞)。
- T0蛍光測定値を取得した後、プレートをインキュベーターに戻します。
- ステップ3.9で作製した10x DNA色素溶液10 μLとステップ3.7で調製した1.5% Triton-X溶液10 μLをT0コントロールプレートに加えます。このプレートをステップ1.8で選択した時間インキュベーターに戻します。
- ステップ1.6で説明したように、細胞が完全に溶解されたら、T0コントロールプレートを読み取ります。
4. 薬剤処理プレートの死細胞の蛍光を経時的に測定します
注:インキュベーターから出ているタイムプレートを最小限に抑えます。温度変化が長引くと細胞の生存率に影響を与える可能性があり、光にさらされると、SYTOX GreenなどのDNA蛍光色素が損なわれる可能性があります。
- 薬物投与後3〜4時間ごとに、すべての薬物処理プレートの蛍光測定値を取得します。プレートは、非常に正確なデスキネティクスが必要でない限り、一晩で読み取る必要はありません。
注:時点は、固定/標準間隔で取得する必要はありません。一般に、タイムポイントは、死亡動態の下流分析でエラーを引き起こすことなく、通常の勤務時間中にのみ取得できます。動力学は、死が始まる前の期間、死の上昇期、および飽和/プラトー期に何らかの測定が行われる限り、推測できます。 - 目的の最終時点で、蛍光測定値を取得します。その直後に、ステップ3.7で調製した1.5% Triton-X溶液10 μLを加えて細胞を溶解します。
- プレートをインキュベーターに戻し、ステップ1で決定した時間、細胞を透過処理します。
- 透過処理後の蛍光測定値を取得します。この蛍光値は、各ウェルの細胞の総数(すなわち、生細胞+死細胞)に比例します。
5.致死画率の動力学を計算します
注:このプロトコルで説明されている計算は、任意の形式またはソフトウェアで分析できます。ただし、MATLAB、R、Pythonなどのプログラミング環境を使用すると、より高速で柔軟な解析が可能になります。
- T0 コントロールプレートからの平均蛍光値を、50% トリミングされた平均値を使用して計算します。この値は、薬剤投与前の細胞の総数に比例します。
- 曲線近似と指数関数的成長関数を使用して、実験のすべてのウェルの人口増加の動力学を計算します。各ウェルの初期ポピュレーションサイズは、ステップ5.1で計算した平均T0蛍光値です。各ウェルの最終的なポピュレーションサイズは、ステップ 4.4 で計算された透過後の値です。アッセイの持続時間は、ステップ4.4で選択されたアッセイエンドポイントまで0時間です。
- ステップ5.2で決定した増殖パラメータを使用して、アッセイの各測定時点における各ウェルの総細胞数を計算します。ステップ5.2で計算した全細胞からデッドセルの測定値を差し引いて、各測定時点における生細胞の数を決定します。
注:アッセイ中のノイズ量が少ないため、時折、生細胞数が小さな負の数になる場合があります。これは、負の数の生細胞が存在することはなく、そのウェル内のすべての細胞が死んでいることを示している可能性が高いため、手動で 0 に設定できます。 - 死細胞の蛍光シグナルを各時点の総細胞シグナルで割ることにより、各時点の致死率(LF)を計算します。
- Lag Exponential Death(LED)方程式を致死分数の時間経過データ10に適合させます。有意な致死性を誘発しない薬物の用量について任意の動態パラメータを回避するには、傾きが0に等しい線形モデルに適合させます。アッセイのノイズまたは無薬質な条件で観察されたLFに基づいて、有意な致死レベルを決定します。
- LED方程式から死亡開始時間(DO)を記録します。LED方程式には、初期LF(LFi)、プラトーでのLF(LFp)、初期死亡率(DR)、およびDOの4つのパラメータがあります。これらのパラメータは、非線形回帰(つまり、カーブフィッティング)を使用してデータから推論します。
- 各用量で、各薬剤のアッセイエンドポイントでの分画生存率(FV)を計算します。エンドポイントの LF 値を 1 から減算するか、生細胞の数を合計セルで除算します。
注:FVは、アッセイエンドポイントだけでなく、任意の時点で計算できます。これらのデータは、用量薬理学(薬物IC50またはEC50など)の評価に使用されます。
6. GR値を計算する
- アッセイ開始時に各ウェルの生細胞の平均数を決定します。この値を計算するには、ステップ 3.15 で計算した透過処理後の T0 読み取り値を使用し、ステップ 3.12 で収集した各ウェルの T0 読み取り値を差し引きます。以下のGR式(ステップ6.3)では、この値はx0と呼ばれます。
注:時折、薬物の最高用量がDNA色素の蛍光読み取りを妨げることがあります。コントロールウェルを用いて時間0における生細胞の平均数を計算することが望ましい場合がある。 - コントロールウェル(xctrl)について、アッセイエンドポイントにおける各ウェルの生細胞の平均数を決定します。各薬物処理条件(x(c))のアッセイエンドポイントにおける生細胞の平均数を決定します。
注:プレート全体の細胞増殖の変動に応じて、各ウェルの正規化に異なるxctrl値を使用できます。たとえば、xctrl値がプレート全体で系統的に異なる場合、プレートの平均xctrl値ではなく、最も近いxctrl値を使用することが望ましい場合があります。 - 各薬剤で処理されたウェルについて、次の式4 を使用して正規化された成長率阻害値 (GR) を計算します。
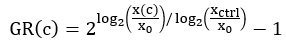
4 パラメーターのロジスティック回帰を使用して曲線近似を実行します。GRは-1から1までのスケールです。
7. GRADE法を使用して薬物誘発性増殖率と死亡率を計算する
注:GRは、真の細胞増殖率ではなく、正味の個体数増加率を表します。薬物誘発性の人口増加率と死亡率は、GRと致死率(LF)の組み合わせを使用して計算できます。
- T0コントロールの読み取り値からの平均生細胞数(x0)、アッセイエンドポイントでのコントロール条件からの平均生細胞数(xctrl)、およびアッセイの長さ(t)を使用して、実験中の細胞株の倍加時間(τ)を決定し、次の式を解きます。

注:細胞倍加時間(τ)を正確に計算するには、アッセイの長さを通じて細胞が連続的に倍増する必要があります。接触阻害を経験する細胞の場合、細胞は、目的のアッセイ時間中にコンフルエントにならない開始密度で播種する必要があります。たとえば、U2OS の平均倍増時間は 24 時間です。ウェルあたり2000個の細胞を72時間アッセイで播種することは、それらの増殖速度を解釈するのに最適です。 - 成長率/死亡率と人口規模の関係を判定します。これを行うには、出生/死亡モデルと、成長率と死亡率のすべての妥当なペアワイズの組み合わせで初期化されたシミュレーションを使用します。このシミュレーションには、T0コントロールプレートの平均生細胞数(x0)、アッセイの長さ(t)、およびユーザー定義の妥当な増殖率(p)と死亡率(d)の範囲が必要です。


- 成長率の妥当な値を決定するには、まず、細胞集団の未処理の成長率を1時間あたりの倍増数(1/T)を最高成長率として、0を最低成長率として開始します。この範囲を等間隔の 500 のセグメントに分割します。死亡率についても同様の操作を適用し、0 から 1 の範囲をテストできます。
注:500レートのすべてのペアワイズの組み合わせは、任意のプログラミング環境を使用して実行できますが、一部のソフトウェアを使用すると計算量が多すぎる可能性があります。これを 50 の成長率と死亡率のすべてのペアワイズの組み合わせに減らすと、推論の精度が低下して、この問題を軽減できます。このプロトコルには、スプレッドシートテンプレートが用意されています。
- 成長率の妥当な値を決定するには、まず、細胞集団の未処理の成長率を1時間あたりの倍増数(1/T)を最高成長率として、0を最低成長率として開始します。この範囲を等間隔の 500 のセグメントに分割します。死亡率についても同様の操作を適用し、0 から 1 の範囲をテストできます。
- シミュレートされた増殖率(p)と死亡率(d)のペアごとに、GR値とLF値(上記)を決定します。
注:手順7.2〜7.3では、増殖率と死亡率の理論上の各ペアと、その理論上のペアの計算されたGR値とLF値を含むテーブルが生成されます。これは、実験的に観察された GR 値と LF 値のペアを、薬物誘発性増殖率と死亡率のペアに関連付けるためのルックアップ テーブルとして機能します。500を超える離散値の増殖率と死亡率を試験すると、数値的にはより正確な値が得られますが、この精度はアッセイの精度を超える可能性があります。 - ルックアップ テーブルで実験的に計算された各 GR/LF ペアと理論上の各 GR/LF ペアの間のペアワイズ ディスタンスを計算します。実験的に観察された GR/LF 値のペアまでの距離が最小の理論ペアを、真の薬物誘発性増殖率と死亡率であると推論します。
8. 経路選択的化学阻害剤を用いた死亡経路の決定
注:化学阻害剤だけでは、特定の薬物の死のメカニズムを明確に決定するには不十分です。.その後の実験でどの生化学的反応または表現型反応を探求すべきかを決定するために、死亡経路の化学的阻害剤を使用すべきであり、これには形態学的評価、経路特異的な生化学的マーカー、および遺伝的依存関係の評価が含まれる可能性が高い。
- 目的の細胞株における各細胞死阻害剤の用量を最適化するには、目的の細胞株の標準的な活性化因子のコンテキストで阻害剤濃度の用量範囲をテストします(表2)。理想的には、選択した用量の阻害剤がそれ自体で細胞の生存率に影響を与えないようにする必要があります(GR指標を確認してください)。
注:すべての細胞株がすべての死経路を活性化できるわけではありません。生化学的または表現型の検証は、さまざまな死経路の活性化因子/阻害剤を試験する際に必要になる場合があります。 - 阻害剤スクリーニングの最適なめっきレイアウトを検討します。バッチ効果を最小限に抑えるには、各薬剤を評価対象の阻害剤と同じプレートに保持し、別々のプレートに複製します。
- ステップ3.3と同様に、96ウェルの光学底の黒壁プレートの各ウェルに所望の数の細胞を播種します。インヒビターと薬剤の量を考慮して、めっき量を80μLに減らします。
- 試験する阻害剤の10倍コンサーテーションを完全培地で調製し、各ウェルに10 μLを添加します。細胞を死細胞経路阻害剤で2〜4時間前処理します。その後、目的の薬物を追加し、ステップ3〜5のように蛍光測定値を取得します。
- インヒビターの有効性を評価するために、死亡開始時間 (DO) および/または最大致死率の変化を評価します。DO は、Lag Exponential Death (LED) モデルから推論されたパラメーターです。手順 5.6 から 5.7 を参照してください。
結果
このプロトコルを使用して、HDAC阻害剤Belinostatに対するU2OS細胞の感受性を調査しました。これらの実験は、2 μM SYTOX Greenを使用して死細胞を標識するために行いました(図1A)。キネティック読み取りは、130ゲイン設定の蛍光プレートリーダーを使用して行われました(図1B)。アッセイの終了時に、PBS中の1.5% Triton-X溶液に細胞を2時間溶解しました(図1C-D)。
FLICKプロトコルは、集団増加率と薬物誘発性細胞死率に関する洞察をもたらします。これらの洞察は、個別に表示することも、ドラッグGRADEの視覚化および分析フレームワークを使用してまとめて表示することもできます(図1E)。GR値を使用してBelinostat感度を評価すると、1μMのBelinostatのGR値が約0になることがわかります(図2A)。GR スケールでは、正の値は人口増加率を報告し、負の値は人口減少率を報告します。したがって、GR値が0の場合、集団はうっ滞状態(すなわち、細胞増殖抑制性薬物反応)のままであることがわかります。細胞うっ血の従来の解釈を考えると、1μMのベリノスタットは細胞を殺さずに完全な増殖阻害を引き起こすと結論付けることができます。しかし、薬物誘発致死画分を評価すると、このアッセイの過程で1μMのベリノスタットが約50%の致死率を引き起こしたことが明らかになります(図2B)。ほとんどの場合、これら 2 つの洞察は、同一条件で比較できない異なる実験から導き出されます。重要なことは、FLICKアッセイを使用すると、両方の洞察が同じ実験データから導き出されますが、Belinostatが集団増加と細胞死に及ぼす影響を捉えるために異なる方法で分析されます。
これら2つの洞察は、GRADE分析法を使用して統合することもできます(図2C)。Drug GRADEは、平均死亡率に比例する致死率と、純人口増加率であるGR値を並置しています。このようにデータを統合することで、薬剤が増殖阻害と死の活性化をどのように調整するかを視覚化できます。GRADEプロット内では、死を活性化せずに成長を阻害する薬物(すなわち、一般的な解釈を用いる細胞増殖抑制剤)が上限を占め、細胞増殖速度を変えずに死滅させるだけの薬物が右境界を占め、最初に成長阻害を引き起こし、続いて増殖停止細胞が死滅する薬物が左境界を占める(すなわち、 二相性薬物;図2C)。これらの境界内にGR/LF応答をもたらす薬物は、増殖をある程度阻害すると同時に細胞死を活性化する(すなわち、一致した薬物)と推測できます3。重要なことは、GR/LF空間内の各薬物反応の位置を使用して、薬物の各濃度の真の増殖率(p)と平均死亡率(d)を計算できることです(図2D-E)。GR値は真の細胞増殖率ではなく、正味の集団増加率(すなわち、真の増殖率と薬物誘発死亡率を組み合わせた正味の影響)であることに注意してください。また、GRADE解析で生成される死亡率(d、平均死亡率)は、LED動態解析の死亡率パラメータ(DR、最大速度または最大死亡率)とは異なります。
FLICKアッセイは、薬物誘発致死画分などの死亡特異的なデータを生成できます。しかし、これらのデータは細胞死のメカニズムにとらわれません。FLICKを使用して細胞死のメカニズムに関する洞察を得るための最も簡単な方法は、死経路特異的阻害剤を含めることによって致死画動態がどのように変化するかを決定することです。関連する死亡メカニズムを阻害すると、薬物誘発致死画分が減少し、および/または死亡発症時間が遅れるはずです。.ここでは、50 μMの汎カスパーゼ阻害剤z-VADが、1 μMのBelinostatによって誘発される致死率の約50%を救うことを示しています(図2F)。重要なことは、薬物と同様に、阻害剤は特異性が限られており、他の死のメカニズムを誤って阻害または悪化させる可能性があることです。したがって、決定的な結論が下される前に、これらのデータは、細胞形態学、活性化の生化学的マーカーの測定、および/または経路特異的な遺伝的依存関係の評価などの他の洞察で補完されるべきである14。
薬物反応の解釈がFLICKベースの評価とGRADEベースの分析によってどのように変化するかを形式的に示すために、次に、異なるタイプの成長/死亡協調を特徴とする3つの薬物を調査しました。パルボシクリブは、細胞増殖抑制反応(細胞死を伴わない増殖阻害)をもたらすCdk4/6阻害剤です。カンプトテシンは、二相性応答(増殖阻害、それに続く高用量での細胞死)を引き起こすトポイソメラーゼI阻害剤です。Belinostatは、同時反応を引き起こすHDAC阻害剤です(すべての用量で部分的な成長阻害と死亡活性化を引き起こしますが、用量によって比率は異なります)。これらのデータの従来の分析を用いると、3種類の薬剤すべてがU2OS細胞の相対的な生存率を大幅に低下させることが観察されます(図2G)。パルボシクリブは明らかに効力が低いですが、これらのデータからパルボシクリブが細胞死を活性化できないかどうかは明らかではありません。薬物が純人口増加率にどのように影響するかを報告するGRベースの分析を使用すると、パルボシクリブで治療された集団はすべての用量で拡大し続けるのに対し、高用量のカンプトテシンまたはベリノスタットは集団の縮小をもたらすため、これらの薬物を高用量で投与すると細胞死が活性化されたに違いないとより正確に解釈できます(図2H)).しかし、GRベースの解析では、増殖と死の間のさまざまな調整を説明できず、特定の薬剤によって活性化される細胞死のレベルについて誤った結論を導く可能性があります。例えば、GRデータに基づくと、カンプトテシンのGR値が-0.25、BelinostatのGR値が0であることを考えると、1μMのカンプトテシンは1μMのBelinostatよりも細胞死を活性化すると結論付けることができます。-0.25 GR の値は、未処理の人口が拡大しているのと同じ速さで人口が 25% 減少していることを意味します。GRADEベースの視覚化と分析を使用すると、カンプトテシンとベリノスタットの成長/死亡の協調性が異なることが観察できます。したがって、1μMの用量では、両方のベリノスタットはカンプトテシンよりも高い割合で死亡を活性化します(ベリノスタットの0.8%死亡/時間、カンプトテシンの0.7%死亡/時間と比較して)が、カンプトテシンで治療された集団は、この薬物によって誘発されるより顕著な成長阻害により、より速く縮小します(図2I)。

図1:FLICKプロトコルのフローチャート (A)マイクロプレートリーダーでの蛍光測定のDNA色素濃度と直線性を最適化するための細胞希釈液を作成します。(B)U2OS細胞中の2 μM SYTOX Greenの細胞数全体でのゲイン滴定。ゲインが 130 の場合、すべてのセル番号で最も線形な信号が得られます。(C)10倍対物レンズを使用して1.5% Triton-Xで経時的に溶解したU2OS細胞のフェーズ画像。四角い領域は、SYTOX Green蛍光の拡大図です。スケールバーは、すべての画像を表しています。(D)指定された時間に溶解した後のSYTOXグリーン信号。データは、3 回の生物学的複製の平均 +/- SD です。(E)セットアップから分析までのアッセイの概念的な概要。主なステップは、薬剤希釈プレートの作成、マルチウェルプレートでの細胞の薬物投与、経時的な蛍光測定値の取得、および増殖と死亡の指標の計算です。パネルBおよびDの場合、データはn = 3回の独立した生物学的反復のSD±平均です。 この図の拡大版を表示するには、ここをクリックしてください。

図2:FLICKアッセイとGRADEベースの分析を使用した薬物反応の特性評価(A)HDAC阻害剤であるBelinostatの用量に対するGR指標。示されているデータは、アッセイエンドポイントである66時間からのものです。1 μMでは、BelinostatのGR値は約0であり、細胞増殖抑制反応を示しています。(B)ベリノスタットの致死率動態。1 μMでは、Belinostatは50%近くの致死性を誘導しますが、これはGR値では捕捉されません。(C)考えられるすべての成長率と死亡率のシミュレーションに基づくGRADEの位相図。シミュレーションは、このアッセイでのU2OSの倍加時間(27.12時間)に基づいていました。(D)アッセイエンドポイントでの用量全体にわたるBelinostatのGRADEプロット。(E)ベリノスタットの投与量について、GRADEが推定する成長率(p、1時間あたりの人口倍増)および死亡率(d、1時間あたりの死亡者数の割合)の棒グラフ。(C)のシミュレーション率と(D)のデータから率を求めた。(F) アポトーシス阻害剤 z-VAD の (紫色) またはなし (黒) 50 μM で処理された 1 μM Belinostat の致死画分プロット。ΔLF = 0.18は、コントロールからの平均最大LFから阻害剤治療条件からの平均最大LFを差し引くことによって計算されました。(G)72時間の曝露後の3つの薬物の相対生存率(薬物治療状態の生細胞の数を未治療状態の生細胞の数で割ったもの)。すべての薬剤は細胞生存率を低下させますが、細胞死が応答に寄与することはわかっていません。(H) (G) の 3 つの薬剤の GR メトリック。これらのデータは、3つの薬剤のうち2つがGR<0で示される人口減少をもたらすことを示しています。細胞死の寄与はまだ不明ですが、GR値が負のときに細胞死が活性化されたはずです。(I)3つの薬剤のGRADE推定率。パルボシクリブは非致死性ですが、成長が停止します。カンプトテシンは二相性です。低用量は成長停止を引き起こし、高用量は成長停止とそれに続く致死性をもたらします。ベリノスタットは偶然の薬です。用量範囲はGRADEプロットの中央を通り抜け、すべての用量が成長摂動と細胞死活性化をもたらすことを示しています。すべてのパネルのデータは、3つの独立した生物学的複製実験の平均±SDです。この図の拡大版を表示するには、ここをクリックしてください。
| 染める | 運動の | 終点 |
| SYTOXグリーン | Y | Y |
| セルトックスグリーン | Y | Y |
| NucSpot 500/515、594/615、750/780 | Y | Y |
| ヨーヨー3 | Y | Y |
| ヨウ化プロピジウム | N | Y |
| 7-AADの | N | Y |
表1:DNA色素とそのFLICKへの応用。 多くの種類の色素をエンドポイントのみのFLICKアッセイに使用できます。アッセイ全体で継続的にインキュベートする条件を満たす色素は少なくなります。緑色でない染料を含む他の染料は、深く特徴付けられていないため、使用前に慎重に評価する必要があります。
| デスパスウェイ | 阻害 剤 | 典型的な線量 |
| 内因性アポトーシス | ZVAD-FMKの | 50 μM |
| 外因性アポトーシス | Z-IETD | 30 μM |
| フェロトーシス | フェロスタチン-1 | 10 μM |
| ネクロプトーシス | ネクロスタチン-1 | 50 μM |
| ピロトーシス | VX-765 | 50 μM |
| パルタナトス | ルカパリブ | 1 μM |
| オートファジー依存性細胞死 | ヒドロキシクロロキン(HCQ) | 10 μM |
| リソソーム依存性細胞死 | E-64D型 | 10 μM |
| クプロプトーシス | テトラチオモリブデン酸(TTM) | 5 μM |
| オキセイプトーシス | N-アセチル-l-システイン(NAC) | 2 mM |
| MPTによる壊死 | シクロスポリンA(CsA) | 10 μM |
表2:細胞死経路とその阻害剤。 細胞死阻害剤は、各死経路について標準的な活性化因子に対して検証する必要があります。選択された阻害剤の用量は、理想的には細胞の生存率に影響を与えないはずです。
補足表1:GRADE計算機。 FLICKベースのGRおよびFVデータから薬物誘発性増殖率と死亡率を計算するためのスプレッドシート。ユーザーは、GRおよびFVデータ、アッセイの長さ(時間単位)、細胞倍加時間(時間単位)、および開始/終了集団サイズを入力する必要があります。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
FLICKアッセイは、細胞集団の成長と死に対する薬物の影響を包括的に評価するための堅牢な方法です。この方法では細胞を直接カウントしないため、プロトコルの重要なステップは、トリトン透過化ステップ中にアッセイの直線性と完全な溶解を確保することです。正しい透過化時間は、このプロトコルで強調されているように視覚的に識別することも、プレートの蛍光を経時的に読み取り、シグナルがプラトー化するタイミングを特定することで定量的に識別することもできます。しかし、私たちの経験では、疑わしい場合、より長く待つことによるコストはないように思われます:FLICKアッセイで使用したDNA色素の場合、シグナル強度は溶解後数日間安定したままです。私たちは定期的にSYTOX Green核酸染色試薬を使用しており、表1で参照されている各蛍光色素はバリデーションしていませんが、他のいくつかのプロトコルはそれらの効果的な使用を示しています13,15,16。特定の細胞非透過性DNA色素の直線性を、さまざまな細胞数と機器設定で評価することで、堅牢で定量的に信頼性の高いデータが保証されます。
一部の細胞株は他の細胞株よりも溶解が難しく、1つの細胞株の溶解時間が別の13細胞株と同じであると仮定すべきではありません。一部の細胞株では、密度が高いと、細胞溶解により細胞がシート状にウェルから浮き上がり、プレートリーダーエラーが発生する可能性があります。これは、開始播種密度を下げるか、溶解後および読み取り前にウェルをピペットで混合することで回避できます。溶解時間や溶解効率を向上させるために、Triton-X溶液をより高いまたは低い割合に調整することができますが、これが必要だったシナリオにはまだ遭遇していません。
実験デザインと薬物プレーティングのレイアウトは、堅牢な成長データが必要な場合は慎重に検討する必要があります。FLICKアッセイ用の薬物希釈プレートの作成は、他の96ウェルプレートアッセイと同じです。ただし、プレートのランドスケープのさまざまな部分をカバーする多くのコントロールを含めると、成長速度論に関する堅牢な洞察を作成するのに役立ち、体系的な成長変動が存在するかどうかを判断するのに役立ちます。プレートの外側のウェルを避けると、これらのウェルは温度、酸素、または湿度の変動により敏感になり、成長に影響を与えるため、より正確な成長データが生成されます。最後に、レプリケートプレート上の薬物の位置を切り替えるなど、疑似ランダム化薬物パターンを使用すると、薬物の挙動をより正確に評価できます。
FLICKアッセイの正確な使用にはいくつかの制限があります。これらの制限は、エンドポイントデータが実験的に観察された値であるため、主に動力学推論に関連しています。生細胞の増殖速度と死の動態はどちらも、薬物治療条件下での生細胞の経時的な成長軌道に関する何らかの仮定に依存します。このプロトコルでは、この成長軌道を指数関数的であり、時間の経過とともに均一な速度で記述します。これらの特徴は、薬物がない場合に観察される可能性がありますが、薬物の存在下では付着しない場合があります。FLICK法が正確であるのは、成長軌道に関する仮定が常に正しいからではなく、薬物治療を受けた集団があまり増殖しない場合(すなわち、アッセイ期間における集団倍増が2〜3倍未満の場合)に、誤った仮定の影響が最小限に抑えられるからです。FLICKで正確にプロファイリングできない薬物には遭遇していません。しかし、この理論では、細胞倍加時間の数倍である長期間にわたって、死が非常にゆっくりと起こる薬物に対しては、FLICKアッセイが正確でなくなることが示唆されます(注:これらの特徴を持つ薬物は特定されていません3)。ただし、全集団の成長速度に関する誤った仮定の影響は、経験的に測定された死細胞値とアッセイの終了時に実験的に決定された最終総細胞値によって制約されるため、LEDフィットから得られる死亡開始時間または最終的な最大LF値に影響を与えません。
これらの制限にもかかわらず、FLICKアッセイは、他の薬物反応法では得られない洞察を可能にします。ほとんどの薬物応答法は、生細胞の数に比例したシグナルを発し、これらの方法は、薬物が正味の集団増加率(すなわち、GR値または同等物)にどのように影響するかを定量化するために使用できますが、薬物の細胞毒性効果と細胞増殖抑制効果を正確に区別することはできません。あるいは、生細胞と死細胞の両方を測定する顕微鏡ベースのアッセイは、確実に包括的な画像を生成することができます。しかし、顕微鏡法によるアッセイでは、細胞が分解して破片になった後に死んだ細胞を数えるのに苦労することがあります。これは、多くの非アポトーシス型の細胞死の状況で急速に起こることです。FLICKの主な特徴は、死細胞を直接カウントするのではなく、死細胞の蛍光を集約するプレートリーダーで測定が行われることです。したがって、FLICKの死細胞シグナルは、死細胞の無傷の細胞性に依存しないため、非アポトーシス細胞死の文脈では、これは一意に不可欠です。さらに、FLICKアッセイのユニークな特徴は、同じ試薬を使用して生細胞集団と死細胞集団の両方を測定できることです。したがって、FLICKアッセイは、生細胞と死細胞の測定値を同等に測定します。この機能により、GRADE法による解析が可能となり、GRADEの成長率と死亡率の同時計算の精度が向上します。付属の 補足表1 には、薬物GRADEの可視化に使用されるテンプレートが含まれており、ユーザーはアッセイの長さ、ステップ7.1から計算された成長率、コントロール条件からの開始および終了細胞数または蛍光測定値、およびステップ5.4および6.3から計算されたLFおよびGRを入力できます。このファイルには、ユーザー定義のパラメータに基づいて自動的に更新される50の増加率と死亡率のすべてのペアワイズの組み合わせのシミュレーションが含まれています。このテンプレートは、薬物反応とGRADEで推定される成長率(p)および死亡率(d)をGRADEベースで視覚化します。
今後の研究では、3D細胞培養シナリオや腫瘍由来オルガノイドの薬物応答の評価におけるFLICKアッセイの応用を探求する必要があります。これらの文脈では、FLICKデータの直線性と感度は深く検討されていませんが、理論的には、FLICKアッセイは、3次元サンプル間で蛍光を統合するためにいくつかの変更を加えることで効果的であるはずです。さらに、細胞特異的な標識を追加することで、共培養で2つ以上の細胞タイプを区別するのに役立ちます。これらの進歩は、免疫細胞とがん細胞との相互作用を探求する上で有益です。最後に、SPARKLのようなアッセイは、FLICKアッセイフォーマットでの死特異的レポーターの使用を促し、細胞死の洞察11,16の精度を維持しながらスループットを向上させる可能性があります。
開示事項
著者には、開示すべき利益相反はありません。
謝辞
私たちは、薬物反応の評価に関する当研究室の視点に貢献してくださったLee Labの過去および現在のすべてのメンバーに感謝します。この研究は、国立衛生研究所からMJL(R21CA294000およびR35GM152194)への資金提供によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
参考文献
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved