Method Article
ניתוח מקיף של התגובה לתרופות באמצעות מבחן FLICK
In This Article
Summary
פרוטוקול זה מתאר כיצד להשתמש במבחן FLICK להערכת תגובות לתרופות, כולל הוראות מפורטות לשימוש בבדיקה זו כדי לחשב את שיעורי הגדילה ושיעורי התמותה הנגרמת על ידי תרופות ולהעריך את מנגנון המוות של תאים הנגרמים על ידי תרופות.
Abstract
להבנת יעילות התרופה, צורך קריטי הוא לאפיין את היקף המוות התאי הנגרם על ידי תרופות. המאמצים לכמת את רמת המוות התאי הנגרם על ידי תרופות מאותגרים על ידי קיומן של יותר מתריסר צורות מובחנות מולקולרית של מוות מווסת, שלכל אחת מהן תזמון הפעלה ותכונות ביוכימיות משלה. יתר על כן, עבור כמה תת-סוגים של מוות נמק, תכונות סימן ההיכר נצפות רק באופן חולף ואובדות במהירות עקב קרע בתאים. לפיכך, גם כאשר משתמשים בשילוב של מבחנים ספציפיים למסלול המוות, זה מאתגר לכמת במדויק את הכמות הכוללת של מוות תאים או את התרומות היחסיות של כל תת-סוג מוות. בעיה נוספת היא שבדיקות ספציפיות למוות רבות מתעלמות מהאופן שבו תרופות משפיעות על התפשטות התאים, מה שמקשה על הפרשנות אם אוכלוסייה שטופלה בתרופות מתרחבת או מתכווצת. בדיקת FLICK מאפשרת לכמת את הרמה הכוללת של מוות תאי לאחר גירוי באופן ספציפי למוות אך גם אגנוסטי במידה רבה לסוג המוות המופעל. בנוסף, בדיקת FLICK שומרת מידע על גודל האוכלוסייה הכולל וקצב התפשטות התאים. בכתב יד זה, אנו מתארים את השימוש הבסיסי במבחן FLICK, כיצד לפתור בעיות בבדיקה זו בעת שימוש בסוגים שונים של חומר ביולוגי, וכיצד להשתמש במבחן FLICK כדי לכמת את התרומות של כל סוג של מוות תאי לתגובה לתרופה שנצפתה.
Introduction
עבור תרופות אנטי-סרטניות, הערכה פרה-קלינית של רגישות לתרופות כוללת בדרך כלל בדיקת האופן שבו תרופות משפיעות על כדאיות התאים בתרבית1. כדאיות התאים לאחר חשיפה לתרופות היא תוצר של לפחות שתי השפעות נפרדות: עיכוב המושרה על ידי תרופות של התפשטות תאים והפעלת מוות תאי2. למרבה הצער, למרות שמוות תאי הוא תכונה קריטית הנדרשת לתגובות תרופתיות עמידות, גישות סטנדרטיות אינן מצליחות להבהיר את המידה שבה תרופה מפעילה מוות תאי3.
מבחני תגובה תרופתיים נפוצים כוללים את אלה שסופרים ישירות תאים (למשל, קולטר קונטר, שימושים מסוימים בזרימה ציטומטרית או מיקרוסקופיה), מכמתים את יכולתם של תאים להתרבות (למשל, בדיקת היווצרות מושבה), או מכמתים פעילות מטבולית (למשל, CellTiter-Glo, מבחני MTT או MTS מבוססי טטרזוליום). מאפיין משותף של מבחנים אלה הוא שהנתונים שנוצרים הם פרופורציונליים למספר התאים החיים. מכיוון שתרופות שונות במידה ניכרת באופן שבו הן מתואמות עיכוב גדילה ומוות תאי, מספר התאים החיים לאחר חשיפה לתרופות מספק תובנה לא אמינה לגבי רמת המוות התאי הנגרם על ידי תרופות3. יתר על כן, מכיוון שתאי סרטן בדרך כלל מתרבים במהירות בתרבית תאים, ניתן להפחית באופן דרמטי את מספר התאים החיים ביחס לאוכלוסייה הלא מטופלת מבלי לגרום למוות תאיםכלשהו. לפיכך, פגם מרכזי הוא שלא ניתן לכמת את מידת המוות של התאים מבלי למדוד את מספר התאים החיים והמתים כאחד.
כימות מספר התאים המתים בעקבות חשיפה לתרופות הוא מאתגר לביצוע מדויק מכמה סיבות2. ראשית, קיימים יותר מתריסר מסלולי מוות תאים מוסדרים 5,6,7,8. למרות שקיימים בדרך כלל סמנים ביוכימיים לזיהוי כל סוג של מוות תאי מוסדר, סמנים אלה משתנים בספציפיות שלהם, ולא ניתן להשתמש בבדיקה אחת כדי לכמת בו זמנית את כל תת-סוגי המוות. שנית, תזמון ההפעלה של כל צורה של מוות תאי יכול להשתנות באופן דרמטי למדי בהתאם להקשר, כך שלא ניתן להופיע תמונה מלאה אלא אם כן מכמתים את המוות לאורך זמן 9,10. בדיקות ביוכימיות רבות לכימות מוות תאים מייצרות מדידה של נקודת קצה, כך שהפקת נתונים קינטיים יכולה להיות מאתגרת ומוגבלת בעלות. סיבוך שלישי הוא שהתא המת עצמו הוא מצב ביניים חולף בין מצב התא החי לפסולת תאים מנותקת. היציבות של תאים מתים משתנה בהתאם לתת-סוג המוות, כאשר סוגים מסוימים, כגון אפופטוזיס, יוצרים גופות יציבות יחסית, בעוד שסוגים אחרים של מוות גורמים לליזה מהירה. לפיכך, שיטות לכימות מוות הדורשות איסוף וספירה של תאים מתים יפיקו גם הבנה מוטה של מוות תאי. לבסוף, מגבלה רביעית היא שמבחנים ביוכימיים המכמתים את מידת מוות התאים בדרך כלל אינם מספקים תובנה כלשהי לגבי האופן שבו תרופה משנה את ההתפשטות. לפיכך, לא ניתן לפרש את גודל האוכלוסייה הכולל - וחשוב מכך, האם האוכלוסייה מתרחבת או מתכווצת.
כמה בדיקות מבוססות מיקרוסקופיה, כגון STACK ו-SPARKL, יעילות במדידת תאים חיים ומתים לאורך זמן, ובדיקות אלה יכולות לייצר תובנות מקיפות על מוות תאים הנגרם על ידי תרופות10,11. עם זאת, בדיקות אלו דורשות מכשירים מיוחדים, כגון מיקרוסקופ Incucyte, מה שיוצר מגבלות בתפוקה ובגישה לגישות אלה. בנוסף, טכניקות מבוססות מיקרוסקופיה דורשות שתאים מתים יישארו במישור המוקד של המיקרוסקופ לאורך כל הניסוי, מה שפוגע ביכולת לכמת תאים מתים כאשר הם מאבדים את ההיצמדות לצלחת או לאורך זמן כאשר תאים מתים מתפרקים. באופן דומה, בדיקות מבוססות מיקרוסקופיה מתמודדות עם אתגרים כאשר הן מיושמות בהקשר של תרביות תרחיף, כאשר תאים נסחפים פנימה והחוצה ממישור מוקד נתון.
כדי לטפל בבעיות שהודגשו לעיל, יצרנו בדיקה הנקראת FLICK (Fluorescence-based and Lysis-dependent Inference of Cell Death Kinetics)12,13. מטרת בדיקת FLICK היא לקבוע את רמת המוות התאי הנגרם על ידי תרופות, ללא קשר לאופן שבו התאים מתים. שיטת FLICK משתמשת בצבעים אימפרמניים של תאים, שהפלואורסצנטיות שלהם תלויה בקשירת ה-DNA. מאפיין מרכזי של FLICK הוא השימוש בפלואורופורים אלה כדי לתייג הצטברות תאים מתים לאורך זמן בזכות ה-DNA הנגיש שלהם, ואחריו ליזה מכנית מבוססת חומר ניקוי כדי לחלחל כל תא חי בסוף הבדיקה. נתונים אלה, בשילוב עם מודלים מתמטיים, מאפשרים לכמת אוכלוסיות תאים חיים ומתים ברזולוציה זמנית רציפה וללא צורך באיסוף או טיפול בתאים מתים. יתר על כן, השימוש בקורא צלחות להערכת פלואורסצנטיות של תאים מתים מאפשר הערכה של תאים מתים מבלי לדרוש שתאים מתים יישארו שלמים, ובכך להקל על הטיה נגד צורות מוות נמקיות הגורמות לקרע בתאים. לבסוף, בדיקת FLICK דורשת טיפול מינימלי בצלחת ויכולה לייצר במהירות מדידות קינטיות, מה שמאפשר בדיקת תרופות בתפוקה גבוהה. בפרוטוקול זה, אנו מתמקדים בשימוש במבחן FLICK, כולל כיצד להשתמש ב-FLICK כדי להסיק את קצב הגדילה המושרה על ידי תרופה, שיעור התמותה ו/או מנגנוני המוות התאי.
Protocol
1. אופטימיזציה של זמן החדירה לכל קו תא מעניין
הערה: אמצעי האחסון והכמויות המתוארים מיועדים למיטוב שורת תא אחת. יש להגדיל ערכים אלה על סמך מספר שורות התאים שיש לבדוק.
- צלחת את מספר התאים הרצוי בכל באר של צלחת בעלת 96 בארות, תחתית אופטית, בעלת דופן שחורה. מוסיפים 100 מיקרוליטר של מדיום שלם ונותנים לתאים להיצמד לצלחת למשך הלילה.
הערה: יש לבצע אופטימיזציה של מספר התאים המצופים תוך התחשבות בקצב הגידול של התאים, הצפיפות האופטימלית ואורך הבדיקה. מספרי תאי התחלה אופייניים לשורות תאים סרטניים דביקים שנמדדו במשך 72 שעות הם 1500 - 5000 תאים לבאר. לדוגמה, תאי U2OS עשויים להיות מצופים ב-2000 תאים לבאר ב-DMEM עם 10% FBS, 2 מ"מ גלוטמין ו-1% Pen-Strep ולתרבת לילה בתנאים סטנדרטיים (5% CO2, 37 מעלות צלזיוס, עם לחות). - בצינור צנטריפוגה חרוטי של 15 מ"ל, הכינו 1.5 מ"ל של תמיסה של 1.5% של Triton-X בתמיסת מלח חוצצת פוספט (PBS). מערבולת הפתרון של 1.5% Triton-X למשך 5 שניות במהירות מקסימלית. מניחים באמבט מים של 37 מעלות צלזיוס למשך 30 דקות לפחות.
- בדוק ויזואלית את התמיסה כדי לוודא ש-Triton-X התמוסס לחלוטין על ידי מערבולת למשך מספר שניות. ודא שהתמיסה הומוגנית.
- הוסף 10 מיקרוליטר מתמיסת 1.5% Triton-X לכל באר של התאים המצופים. אין לערבב.
הערה: בשלב זה, לא רצוי לערבב באמצעות פיפטינג חוזר עקב היווצרות בועות. - החזר תאים ל-5% CO2, 37 מעלות צלזיוס עם חממת תרבית תאים לחות למשך שעה לפחות.
הערה: לאחר יישום תמיסת Triton-X, רוב קווי התאים עוברים ליזה מלאה תוך 2-3 שעות. להמתנה של עד 24 שעות יש השפעה מינימלית על אות הקרינה בעת שימוש בצבעים כגון SYTOX Green. הגדלת אחוז הטריטון-X עשויה לקצר את זמן הדגירה עבור קווי תאים קשים לליזה. - התבונן במורפולוגיה של תאים במיקרוסקופ אור באמצעות מטרה של פי 10. בדוק תאים אחת לשעה עד שגופי התאים כבר לא נראים לעין. רשום את הזמן הנדרש לחדירות תאים.
הערה: אם קיים מיקרוסקופ פלואורסצנטי, ניתן לדמיין ליזה באמצעות צבע DNA אטום לתאים, כגון SYTOX Green. ניתן להוסיף את הצבע בריכוז סופי פי 10 לתמיסת Triton-X, וניתן לאשר חדירות על ידי מיקרוסקופיה פלואורסצנטית.
2. בחירה וכיול של כתם DNA
הערה: דרישה לבדיקת FLICK היא שימוש בפלואורופור תא אימפרנטי הפולט אות באופן תלוי בקשירת DNA, אינו משפיע על כדאיות התא ומייצר אות המשתנה באופן ליניארי עם מספר התא. פרוטוקול זה משתמש ב-SYTOX Green. צבעים אחרים בעלי תכונות דומות עשויים להתאים גם לבדיקת FLICK, אך יש להעריך ולכייל כל אחד מהם. ראה טבלה 1 לדוגמאות.
- עבור צבע ה-DNA שנבחר, קבע את טווח הריכוזים שיש לבדוק על סמך המלצת היצרן.
- עבור כל ריכוז שייבדק, צלחת 40,000 תאים ב-180 מיקרוליטר של מדיום תרבית תאים משולש לאורך העמודה השמאלית ביותר של צלחת בעלת 96 בארות, תחתית אופטית, בעלת דופן שחורה. הוסף 90 מיקרוליטר בינוני לבארות הנותרות.
הערה: זה ישמש ליצירת טיטרציה של תאים ליניאריים על פני שורות הצלחת. הריכוז הסופי של הבארות הראשונות יהיה 20,000 תאים לבאר. לוחות תחתונים אופטיים אינם נדרשים לקוראי צלחות הקוראים פלואורסצנטיות מראש הבאר. מדיום תרבית התאים צריך להיות אותו מדיום המשמש לגידול התאים הנבדקים. לדוגמה, אם משתמשים בתאי U2OS, יש להשעות מחדש 40,000 תאים ב-180 מיקרוליטר של DMEM. - באמצעות פיפטה רב-ערוצית, צור דילול טורי של 1:2 על ידי העברת 90 מיקרוליטר מהעמודה השמאלית ביותר לעמודה הסמוכה מימין. פיפטה 15x לערבב.
- חזור על שלב 2.3, מעבר מהעמודה השנייה לעמודה השלישית, לאחר מכן מהעמודה השלישית לעמודה הרביעית וכן הלאה. סיים את הטיטרציה בעמודה השנייה עד האחרונה של הצלחת. השאר את העמודה האחרונה ללא תאים כך שניתן יהיה לקבל את אות הרקע של מדיה של תרבית תאים.
- עבור העמודה השנייה עד האחרונה, הסר 90 מיקרוליטר כך שלכל הבארות יש 90 מיקרוליטר של מדיום המכיל מספר משתנה של תאים. תן לתאים להיצמד במשך 6 שעות בחממת תרבית תאים של 37 מעלות צלזיוס.
- הכן תמיסה פי 10 של צבע DNA עבור כל ריכוז צבע שייבדק, כאשר צבע ה-DNA מדולל למצע גידול שלם המשמש לתרבית התאים המעניינים. הכן 1.5 מ"ל של תמיסת Triton-X של 1.5% ב-PBS, כמתואר בשלב 1.
- לכל באר של צלחת 96 הבארות, הוסף 10 מיקרוליטר מצבע ה- DNA פי 10. הוסף 10 מיקרוליטר של תמיסת טריטון-X 1.5% לכל באר כדי לחלחל לתאים. דגירה של תאים בתמיסה זו למשך הזמן האופטימלי, שנקבע בשלב 1.
הערה: לאחר הוספת Triton-X, הריכוז הסופי של צבע ה-DNA הוא מעט פחות מ-1x, אך זה חסר חשיבות. לאחר הדגירה יש ליז את כל הצלחת. טיטרציה ידנית של התא תשמש לזיהוי ריכוז צבע ה-DNA האופטימלי וההגדרות האופטימליות לרכישת פלואורסצנטיות. - מדוד את עוצמות הקרינה על פני לוחית הטיטרציה של התא. כמת את הקרינה על פני מגוון הגדרות רכישה כפי שמתואר על ידי יצרן צבע ה-DNA. בהתאם לקורא הלוחות בו נעשה שימוש, שנה את אורכי גל העירור והפליטה ו/או את הרווח הדיגיטלי.
- הסר את אות הרקע מכל מדידה על ידי הפחתת האות הממוצע של העמודה שאינה מכילה תאים. ניתן למצוא ערכים אלה בעמודת הבארות הימנית ביותר בצלחת הדילול הסדרתי.
- קבע את הליניאריות של כל ריכוז של צבע DNA עבור כל הגדרת רכישה. ניתן לקבוע לינאריות על ידי גרף מספר התא כנגד אות הקרינה וביצוע רגרסיה ליניארית. מקדם הקביעה (r2) מדווח על המידה שבה אות הקרינה קשור באופן ליניארי למספר התא.
- בחר את הגדרות הרכישה וריכוז צבע ה-DNA שיש לו את השילוב הטוב ביותר של ליניאריות וטווח דינמי.
הערה: שקול את כל טווח מספרי התאים מ- 0 עד 20,000 תאים. ודא שהרווח וריכוז צבע ה-DNA הם ליניאריים במספרי תאים נמוכים, בין 0 ל-1000 תאים. מספר הגדרות מכשירים או ריכוזי צבע DNA עשויים לספק מתאם חזק על פני כל הטווח. עם זאת, חיוני שהרגישות הנמוכה תהיה חזקה, כך שניתן יהיה למדוד במדויק שינויים קטנים בתאים מתים.
3. ציפוי תאים, יישום תרופות בבאר ומדידת פלואורסצנציה של תאים מתים לאורך זמן בצלחות שטופלו בתרופות
הערה: ניתן לעצב צלחות דילול תרופות בצורה גמישה על סמך צרכי הניסוי. בדרך כלל, לוחות דילול תרופות יכללו סדרת דילול לוג או חצי לוג של תרופה אחת או כמה.
- שקול את פריסת הציפוי האופטימלית לניסוי. הימנע ממדידות מהבארות החיצוניות של צלחת של 96 בארות כדי להפחית את הרעש הקשור לשונות בגדילה, מכיוון שהבארות החיצוניות חוות טמפרטורות, חמצון ואידוי שונים בהשוואה לבארות הפנימיות של הצלחת.
הערה: הכללת בקרות בכל נוף הצלחת תגדיל את החוסן של נתוני הצמיחה. שקול לכלול בארות בקרה במרכז ובקצוות הצלחת. - קבע את מספר הלוחות הנדרשים לניסוי. כלול צלחת אחת נוספת לליזה בתחילת הבדיקה כדי לקבוע את מספר התאים הממוצע לבאר בזמן הסימום (כלומר, לוחית הבקרה T0).
- הכן תרחיף תאים באמצעי גידול שלם.
- בהתבסס על מספר הלוחות הדרושים לניסוי (מחושב בשלב 3.2), קבע את נפח מתלה התאים הנדרש. אם זריעה של צלחות ב 90 מיקרוליטר לבאר, נדרשים כ -10 מ"ל לצלחת. הכפל את מספר הלוחות הנדרשים ב -10 מ"ל.
הערה: רצוי להגדיל נפח זה כדי לקחת בחשבון את הנפח המת של המאגר ואת אובדן הנפח במהלך הפיפטה. לדוגמה, ניתן להכפיל את הנפח המחושב ב-1.2. - ספרו תאים והכינו דילול תאים על סמך מספר התאים הרצוי לבאר. לדוגמה, אם שואפים לזרוע 2000 תאים ב-90 מיקרוליטר מדיה לבאר של צלחת של 96 בארות, עבור 3 לוחות בסך הכל, חשב את המספר הכולל של התאים הדרושים לניסוי כדלקמן:

באופן דומה, אם תרחיף התאים שנספר הוא 400,000 תאים/מ"ל, נפח תרחיף התאים הדרוש לניסוי יהיה:

- תקן את נפח אמצעי תרבית התאים המשמשים לציפוי על ידי הפחתת נפח תרחיף התאים שנספר שיש להוסיף.
מדיה של 36 מ"ל - מתלה תאים של 2 מ"ל = נפח מדיה מתוקן של 34 מ"ל
- בהתבסס על מספר הלוחות הדרושים לניסוי (מחושב בשלב 3.2), קבע את נפח מתלה התאים הנדרש. אם זריעה של צלחות ב 90 מיקרוליטר לבאר, נדרשים כ -10 מ"ל לצלחת. הכפל את מספר הלוחות הנדרשים ב -10 מ"ל.
- מערבבים את תרחיף התאים שנספר עם נפח המדיה הנכון באמצעות פיפטה סרולוגית. העבר את תרחיף התא הזה למאגר ריאגנט תחתון V.
- בעזרת פיפטה רב-ערוצית, הוסף 90 מיקרוליטר של מתלה התא לכל באר מלוחות 96 הבארות. מערבבים את תרחיף התאים במאגר המגיב באופן קבוע באמצעות פיפטינג חוזר עם פיפטה סרולוגית כדי להבטיח שהרצוי להבטיח שהריכוז הרצוי של תאים לנפח נשמר.
- תן לתאים להיצמד למשך הלילה באינקובטור תרבית תאים של 37 מעלות צלזיוס (בדרך כלל 12-24 שעות).
- הכן תמיסת Triton-X של 1.5% ב-PBS, כמתואר בשלב 1. נפח תמיסה זו אמור להספיק בכדי ליז את כל הצלחות. כ-1.5 מ"ל של 1.5% טריטון-X ב-PBS מספיקים כדי לליז צלחת אחת של 96 בארות.
הערה: תמיסת ה-1.5% Triton-X ב-PBS יציבה במשך למעלה משבוע וניתן להכין אותה מראש מטעמי נוחות. - קבע את כמות אמצעי תרבית התאים הנדרשת להכנת צלחת דילול תרופות. הכן צלחות דילול תרופות בצלחות שקופות 96 בארות U או V.
- כדי להבטיח שנפח התרופה המדוללת יספיק לניסוי, הגדל את הנפח המינימלי הנדרש כדי להסביר את אובדן הנפח במהלך הפיפטינג. לדוגמה, אם יש להוסיף תרופות ב-10 מיקרוליטר לכל באר של צלחת של 96 בארות והניסוי כולל שלוש צלחות, נדרשים 30 מיקרוליטר של תרופה מדוללת לכל באר. הכן 40-50 מיקרוליטר של מדיה מסוממת לבאר כדי לקחת בחשבון את אובדן הנפח. הוסף 1.5 מ"ל לנפח המדיה הנדרש כדי לקחת בחשבון את המדיה הדרושה ללוחית הבקרה T0.
- הכן ריכוז פי 10 של צבע ה-DNA שנבחר במצע גידול שלם. ריכוז זה מבוסס על הריכוז שנבחר בשלב 2.11. הנפח הכולל של פתרון זה מחושב בשלב 3.8.
- בעזרת צבע ה- DNA + מדיום הגידול משלב 3.9, הכינו ריכוז פי 10 מכל תרופה לבדיקה. צור רק את המינון הגבוה ביותר של כל תרופה בצלחת דילול התרופות ודלל את התרופות באופן סדרתי עם פיפטה רב-ערוצית, תוך ערבוב פי 15 בין כל באר.
הערה: בעת ביצוע המינון הגבוה ביותר עבור תרופה נתונה בצלחת דילול התרופה, יש לקחת בחשבון את נפח התרופה כדי להבטיח שהריכוז מדויק. - בעזרת פיפטה רב-ערוצית, הוסף 10 מיקרוליטר של תמיסת צבע תרופה + DNA משלב 3.10 לצלחות המכילות תאים.
הערה: עבור צלחות דילול תרופות שבהן נוצר דילול סדרתי, אם מוסיפים תרופות לתאים החל מהריכוז הנמוך ביותר של התרופה ועד לריכוז הגבוה ביותר, אין צורך לשנות את קצות הפיפטה הרב-ערוצית. עם זאת, יש להחליף את הקצוות על הפיפטה הרב-ערוצית בין כל צלחת ובכל פעם שהטיפים שימשו בבאר המכילה תרופה אחרת או ריכוז גבוה יותר של התרופה. - קרא את הקרינה עבור כל הצלחות שבהן נוספו תרופות. קריאת הקרינה הזו היא קריאת T0 (כלומר, תאים מתים בזמן = 0 שעות).
- החזר את הצלחות לחממה לאחר ביצוע קריאת הקרינה T0.
- הוסף 10 μL של תמיסת צבע DNA 10x שנוצרה בשלב 3.9 ו- 10 μL של תמיסת Triton-X 1.5% בשלב 3.7 ללוחית הבקרה T0. החזר צלחת זו לחממה למשך הזמן שנבחר בשלב 1.8.
- קרא את לוחית הבקרה T0 לאחר שהתאים עברו ליזה מלאה, כמתואר בשלב 1.6.
4. מדוד את הקרינה של התאים המתים לאורך זמן עבור צלחות שטופלו בתרופות
הערה: צמצם את הזמן שצלחות הלחות מחוץ לחממה. שינויים ממושכים בטמפרטורה יכולים להשפיע על כדאיות התאים, וחשיפה לאור עלולה לפגוע בפלואורופורים של DNA, כגון SYTOX Green.
- רכוש קריאות פלואורסצנטיות עבור כל הצלחות שטופלו בתרופות כל 3-4 שעות לאחר הסימום. אין צורך לקרוא צלחות בן לילה אלא אם כן נדרשת קינטיקה מדויקת מאוד של מוות.
הערה: אין צורך לצלם נקודות זמן במרווחי זמן קבועים/סטנדרטיים. באופן כללי, ניתן לקחת נקודות זמן אך ורק בשעות העבודה הרגילות מבלי לגרום לטעויות בניתוח במורד הזרם של קינטיקה של מוות. ניתן להסיק את הקינטיקה כל עוד נלקחות מדידות מסוימות בתקופה שלפני הופעת המוות, במהלך שלב העלייה של המוות ובמהלך שלב הרוויה/מישור. - בנקודת הזמן הסופית הרצויה, רכשו קריאת פלואורסצנטיות. מיד לאחר מכן, הוסף 10 מיקרוליטר מתמיסת 1.5% Triton-X שהוכנה בשלב 3.7 כדי ליז את התאים.
- החזירו את הצלחות לחממה ואפשרו לתאים לחלחל למשך הזמן שנקבע בשלב 1.
- רכוש קריאות פלואורסצנטיות לאחר חדירה. ערך פלואורסצנטי זה פרופורציונלי למספר הכולל של תאים (כלומר, תאים חיים + מתים) עבור כל באר.
5. חשב את קינטיקה של השבר הקטלני
הערה: ניתן לנתח את החישובים המתוארים בפרוטוקול זה בכל תבנית או תוכנה. עם זאת, שימוש בסביבת תכנות כגון MATLAB, R או Python יאפשר ניתוח מהיר וגמיש יותר.
- חשב את ערכי הקרינה הממוצעים מלוח הבקרה T0 באמצעות הממוצע החתוך של 50%. ערך זה פרופורציונלי למספר הכולל של התאים לפני הסימום.
- באמצעות התאמת עקומה ופונקציית גידול מעריכית, חשב את הקינטיקה של גידול האוכלוסייה עבור כל הבארות בניסוי. גודל האוכלוסייה ההתחלתי עבור כל באר הוא ערך הקרינה הממוצע של T0 המחושב בשלב 5.1. גודל האוכלוסייה הסופי עבור כל באר הוא הערך שלאחר החדירה המחושב בשלב 4.4. משך הבדיקה הוא מ-0 שעות עד לנקודת הסיום של הבדיקה שנבחרה בשלב 4.4.
- באמצעות פרמטרי הגידול שנקבעו בשלב 5.2, חשב את מספר התאים הכולל בכל נקודת זמן שנמדדה בבדיקה עבור כל באר. קבע את מספר התאים החיים בכל נקודת זמן שנמדדה על ידי הפחתת מדידת התאים המתים מסך התאים המחושבים בשלב 5.2.
הערה: עקב כמויות קטנות של רעש בבדיקה, מדי פעם, מספר התא החי עשוי להיות מספר שלילי קטן. ניתן להגדיר זאת באופן ידני ל-0 מכיוון שלא יכול להיות מספר שלילי של תאים חיים וככל הנראה מציין שכל התאים בבאר זו היו מתים. - חשב שבר קטלני (LF) עבור כל נקודת זמן על ידי חלוקת אות הקרינה של התא המת באות התא הכולל עבור כל נקודת זמן.
- התאם משוואת מוות מעריכי פיגור (LED) לנתוני מהלך זמן השבר הקטלני10. כדי להימנע מפרמטרים קינטיים שרירותיים למינונים של תרופה שאינם גורמים לקטלניות משמעותית, התאימו למודל ליניארי עם שיפוע השווה ל-0. קבע רמות משמעותיות של קטלניות על סמך הרעש בבדיקה או ה-LF שנצפה במצבים לא מסוממים.
- רשום את זמן תחילת המוות (DO) ממשוואת LED. למשוואת LED יש ארבעה פרמטרים: ה-LF הראשוני (LFi), ה-LF במישור (LFp), שיעור התמותה ההתחלתי (DR) וה-DO. הסיק פרמטרים אלה מהנתונים באמצעות רגרסיה לא ליניארית (כלומר, התאמת עקומה).
- חשב את הכדאיות החלקית (FV) בנקודת הקצה של הבדיקה עבור כל תרופה בכל מנה. הפחיתו את ערך LF של נקודת הקצה מ- 1 או חלקו את מספר התאים החיים בסך התאים.
הערה: ניתן לחשב FV בכל נקודת זמן, לא רק בנקודת הקצה של הבדיקה. נתונים אלה משמשים להערכת פרמקולוגיה של מינון (למשל, תרופה IC50 או EC50).
6. חשב את ערך ה-GR
- קבע את המספר הממוצע של תאים חיים עבור כל באר בתחילת הבדיקה. חשב ערך זה באמצעות קריאת T0 לאחר חדירה המחושבת בשלב 3.15 והחסרת קריאת T0 עבור כל באר, שנאספה בשלב 3.12. במשוואת GR למטה (שלב 6.3), ערך זה מכונה x0.
הערה: לפעמים המינון הגבוה ביותר של תרופה יכול להפריע לקריאת הקרינה של צבע ה-DNA. ייתכן שרצוי לחשב את המספר הממוצע של תאים חיים בזמן 0 באמצעות בארות הבקרה. - עבור בארות הבקרה (xctrl), קבע את המספר הממוצע של תאים חיים עבור כל באר בנקודת הקצה של הבדיקה. קבע את המספר הממוצע של תאים חיים בנקודת הקצה של הבדיקה עבור כל מצב שטופל בתרופה (x(c)).
הערה: בהתאם לשונות בצמיחת התאים על פני הצלחת, ניתן להשתמש בערכי xctrl שונים לנורמליזציה של כל באר. לדוגמה, אם ערכי xctrl משתנים באופן שיטתי על פני הלוח, ייתכן שיהיה רצוי להשתמש בערך xctrl הקרוב ביותר ולא בערך xctrl הממוצע של הלוח. - עבור כל באר שטופלה בתרופה, חשב את ערך עיכוב קצב הגדילה המנורמל (GR) באמצעות המשוואה הבאה4:
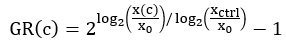
בצע התאמת עקומה באמצעות רגרסיה לוגיסטית של 4 פרמטרים. GR הוא בסולם מ-1 עד 1.
7. לחשב את שיעורי הגדילה והתמותה הנגרמת על ידי תרופות בשיטת GRADE
הערה: GR מייצג את קצב גידול האוכלוסייה נטו, לא את קצב התפשטות התאים האמיתי. ניתן לחשב את גידול האוכלוסייה ושיעורי התמותה הנגרמים על ידי תרופות באמצעות שילוב של GR והחלק הקטלני (LF).
- קבע את זמן ההכפלה (τ) של קו התאים במהלך הניסוי באמצעות המספר הממוצע של תאים חיים מקריאת הבקרה T0 (x0), המספר הממוצע של תאים חיים מתנאי הבקרה בנקודת הסיום של הבדיקה (xctrl), ואורך הבדיקה בשעות (t) כדי לפתור את המשוואה הבאה:

הערה: חישוב מדויק של זמן הכפלת תאים (τ) דורש שהתאים יוכפלו באופן רציף לאורך כל הבדיקה. עבור תאים שחווים עיכוב מגע, יש לזרוע תאים בצפיפות התחלתית שלא תהפוך למתכנסת בזמן הבדיקה הרצוי. לדוגמה, ל-U2OS יש זמן הכפלה ממוצע של 24 שעות. זריעת 2000 תאים לבאר בבדיקה של 72 שעות היא אופטימלית לפירוש קצב הגידול שלהם. - קבע את הקשר בין שיעורי צמיחה/תמותה לגודל האוכלוסייה. כדי לעשות זאת השתמש במודל לידה/מוות ובסימולציה שאותחלה עם כל השילובים הזוגיים הסבירים של שיעורי גדילה ותמותה. סימולציה זו דורשת את המספר הממוצע של תאים חיים מלוח הבקרה T0 (x0), אורך הבדיקה (t) וטווח מוגדר על ידי המשתמש של שיעורי התפשטות סבירים (p) ושיעורי תמותה (d).


- כדי לקבוע ערכים סבירים לשיעורי גדילה, התחל עם קצב הגידול הלא מטופל בהכפלת אוכלוסיית התאים לשעה (1/T) כקצב הגידול הגבוה ביותר ו-0 כקצב הגידול הנמוך ביותר. חלקו טווח זה ל-500 מקטעים במרווחים שווים. ניתן ליישם ניתוח דומה עבור שיעור התמותה, תוך בדיקת טווח בין 0 ל-1.
הערה: ניתן לבצע את כל השילובים הזוגיים של 500 קצבים באמצעות כל סביבת תכנות, אך עשויים להיות אינטנסיביים מדי מבחינה חישובית אם משתמשים בתוכנה כלשהי. צמצום זה לכל השילובים הזוגיים של 50 שיעורי גידול ותמותה יכול להקל על בעיה זו, עם הפחתה ברמת הדיוק של המסקנה. תבנית גיליון אלקטרוני סופקה עם פרוטוקול זה.
- כדי לקבוע ערכים סבירים לשיעורי גדילה, התחל עם קצב הגידול הלא מטופל בהכפלת אוכלוסיית התאים לשעה (1/T) כקצב הגידול הגבוה ביותר ו-0 כקצב הגידול הנמוך ביותר. חלקו טווח זה ל-500 מקטעים במרווחים שווים. ניתן ליישם ניתוח דומה עבור שיעור התמותה, תוך בדיקת טווח בין 0 ל-1.
- קבע את ערכי GR ו-LF (שתוארו לעיל) עבור כל זוג קצב התפשטות מדומה (p) ומוות (d).
הערה: שלבים 7.2-7.3 ייצרו טבלה המכילה כל זוג תיאורטי של שיעורי התפשטות ותמותה, עם ערכי GR ו-LF המחושבים עבור אותו זוג תיאורטי. זה יתפקד כטבלת חיפוש כדי לשייך זוג שנצפה בניסוי של ערכי GR ו-LF עם זוג שיעורי התפשטות ותמותה הנגרמת על ידי תרופות. בדיקת יותר מ-500 ערכים בדידים לשיעורי התפשטות ותמותה תביא לערך מדויק יותר מבחינה מספרית, אך רמת דיוק זו תהיה ככל הנראה מעבר לדיוק הבדיקה. - חשב את המרחק הזוגי בין כל זוג GR/LF מחושב בניסוי לבין כל זוג GR/LF תיאורטי בטבלת החיפוש. הסיק את הצמד התיאורטי עם המרחק המינימלי לזוג ערכי GR/LF שנצפה בניסוי כשיעורי ההתפשטות והתמותה האמיתיים הנגרמים על ידי תרופות.
8. קביעת מסלולי מוות באמצעות מעכבים כימיים סלקטיביים למסלול
הערה: מעכבים כימיים לבדם אינם מספיקים לקביעה סופית של מנגנון המוות של תרופה נתונה. יש להשתמש במעכבים כימיים של מסלולי מוות כדי לקבוע אילו תגובות ביוכימיות או פנוטיפיות יש לחקור בניסויים הבאים, אשר עשויים לכלול הערכה מורפולוגית, סמנים ביוכימיים ספציפיים למסלול והערכת תלות גנטית.
- מטב את המינון של כל מעכב מוות תאים בקו התאים המעניין על ידי בדיקת טווח מינון של ריכוזי מעכבים בהקשר של מפעיל קנוני של מסלולי המוות המעניינים (טבלה 2). באופן אידיאלי, המינון הנבחר של המעכב לא אמור להשפיע על כדאיות התא בפני עצמו (בדוק את מדד ה-GR).
הערה: לא כל קווי התאים יכולים להפעיל את כל מסלולי המוות. ייתכן שיידרש אימות ביוכימי או פנוטיפי בעת בדיקת מפעילים/מעכבים של מסלולי מוות שונים. - שקול את פריסת הציפוי האופטימלית עבור מסך מעכב. כדי למזער את השפעות האצווה, שמור כל תרופה באותה צלחת כמו המעכבים המוערכים, עם שכפולים על צלחות נפרדות.
- זרע את מספר התאים הרצוי בכל באר של צלחת בעלת 96 בארות, תחתית אופטית, בעלת דופן שחורה, בדומה לשלב 3.3. הקטן את נפח הציפוי ל-80 מיקרוליטר כדי להסביר את נפחי המעכב והתרופה.
- הכן קונצרט פי 10 של המעכבים לבדיקה במדיה שלמה והוסף 10 מיקרוליטר לכל באר. יש לטפל בתאים עם מעכבי מסלול מוות למשך 2-4 שעות. לאחר מכן, הוסף את התרופות המעניינות ורכוש קריאת פלואורסצנטיות, כמו בשלבים 3-5.
- העריכו את השינוי בזמן תחילת המוות (DO) ו/או השבר הקטלני המקסימלי כדי להעריך את יעילות המעכבים. ה-DO הוא פרמטר מוסק ממודל Lag Exponential Death (LED). ראה שלבים 5.6 - 5.7.
תוצאות
באמצעות פרוטוקול זה, חקרנו את הרגישות של תאי U2OS למעכב HDAC בלינוסטט. ניסויים אלה בוצעו באמצעות 2 מיקרומטר SYTOX Green כדי לסמן תאים מתים (איור 1A). קריאות קינטיות נעשו באמצעות קורא לוחות פלואורסצנטיים בהגדרת רווח של 130 (איור 1B). התאים עברו ליזה בתמיסת טריטון-X של 1.5% ב-PBS למשך שעתיים בסוף הבדיקה (איור 1C-D).
פרוטוקול FLICK מייצר תובנות לגבי קצב גידול האוכלוסייה ושיעור תמותת התאים הנגרמת על ידי תרופות. ניתן לראות את התובנות האלה בנפרד או באמצעות מסגרת ההדמיה והניתוח של התרופות GRADE, ביחד (איור 1E). הערכת רגישות לבלינוסטט באמצעות ערך GR מגלה ש-1 מיקרומטר בלינוסטט מביא לערך GR של כ-0 (איור 2A). בסולם GR, ערכים חיוביים מדווחים על קצב התרחבות האוכלוסייה, וערכים שליליים מדווחים על קצב התכווצות האוכלוסייה. לפיכך, ערך GR של 0 מגלה שהאוכלוסייה נשארת בקיפאון (כלומר, תגובה ציטוסטטית לתרופה). בהתחשב בפרשנות המקובלת של ציטוסטזיס, ניתן להסיק כי 1 מיקרומטר בלינוסטט גורם לעיכוב גדילה מלא ללא כל הרג תאים. אולם הערכת המקטע הקטלני שנגרם על ידי תרופה מגלה ש-1 מיקרומטר בלינוסטט גרם לקטלניות של כ-50% במהלך הבדיקה הזו (איור 2B). ברוב המקרים, שתי התובנות הללו נגזרות מניסויים שונים שלא ניתן להשוות אותם באופן של תפוח לתפוח. חשוב לציין, באמצעות מבחן FLICK, שתי התובנות נגזרות מאותם נתונים ניסיוניים אך מנותחות באופן שונה כדי ללכוד את ההשפעות של בלינוסטט על גידול האוכלוסייה לעומת מוות תאי.
ניתן לשלב את שתי התובנות הללו גם באמצעות השיטה האנליטית GRADE (איור 2C). Drug GRADE מציב את החלק הקטלני, שהוא פרופורציונלי לשיעור התמותה הממוצע, מול ערך ה-GR, קצב גידול האוכלוסייה נטו. שילוב הנתונים בדרך זו מספק הדמיה של האופן שבו תרופה מתאמת עיכוב גדילה והפעלת מוות. בתוך תרשים ה-GRADE: תרופות המעכבות גדילה מבלי להפעיל מוות (כלומר, תרופות ציטוסטטיות, לפי הפירוש המקובל) יתפסו את הגבול העליון, תרופות שרק הורגות מבלי לשנות את קצב התפשטות התאים יתפסו את הגבול הנכון, ותרופות שגורמות תחילה לעיכוב גדילה, ולאחר מכן מוות של תאים עצורי גדילה יתפסו את הגבול השמאלי (כלומר, תרופות דו-פאזיות; איור 2C). ניתן להסיק כי תרופות הגורמות לתגובות GR/LF הנופלות בגבולות אלה מעכבות בו זמנית התפשטות במידה מסוימת תוך הפעלת מוות תאי (כלומר, תרופות מקריות)3. חשוב לציין שהמיקום של כל תגובה לתרופה במרחב GR/LF יכול לשמש לחישוב קצב ההתפשטות האמיתי (p) ושיעור התמותה הממוצע (d) עבור כל ריכוז של תרופה (איור 2D-E). שימו לב שערך ה-GR אינו קצב התפשטות התאים האמיתי אלא קצב גידול האוכלוסייה נטו (כלומר, ההשפעה נטו המשלבת את קצב ההתפשטות האמיתי ושיעור התמותה הנגרמת על ידי תרופות). כמו כן, שיעור התמותה המופק בניתוח GRADE (d, שהוא שיעור התמותה הממוצע) שונה מפרמטר שיעור התמותה בניתוח הקינטי של LED (DR, המהירות המקסימלית או שיעור התמותה המקסימלי).
בדיקת FLICK יכולה לייצר נתונים ספציפיים למוות, כגון החלק הקטלני המושרה על ידי תרופה; עם זאת, נתונים אלה אינם תלויים במנגנון המוות התאי. כדי לקבל תובנות לגבי מנגנון המוות התאי באמצעות FLICK, השיטה הפשוטה ביותר היא לקבוע כיצד קינטיקה של שבר קטלני משתנה על ידי הכללת מעכבים ספציפיים למסלול המוות. עיכוב מנגנון מוות רלוונטי אמור לגרום להפחתת המקטע הקטלני הנגרם על ידי התרופה ו/או לעכב את זמן תחילת המוות. כאן, אנו מראים ש-50 מיקרומטר של מעכב הפאן-קספאז z-VAD מציל כ-50% מהקטלניות הנגרמת על ידי 1 מיקרומטר בלינוסטט (איור 2F). חשוב לציין, בדומה לתרופות, למעכבים יש ספציפיות מוגבלת והם עלולים לעכב או להחמיר בטעות מנגנוני מוות אחרים. לפיכך, לפני שמגיעים למסקנה סופית, יש להשלים נתונים אלה עם תובנות אחרות, כגון מורפולוגיה של תאים, מדידת סמנים ביוכימיים של הפעלה ו/או הערכה של תלות גנטית ספציפית למסלול14.
כדי להמחיש באופן רשמי כיצד ניתן לשנות את הפרשנות של תגובה לתרופה על ידי הערכה מבוססת FLICK וניתוח מבוסס GRADE, לאחר מכן חקרנו שלוש תרופות הכוללות סוגים שונים של תיאום גדילה/מוות. Palbociclib הוא מעכב Cdk4/6 המביא לתגובה ציטוסטטית (עיכוב גדילה ללא מוות תאי). קמפטותצין הוא מעכב טופואיזומראז I הגורם לתגובה דו-פאזית (עיכוב גדילה, ואחריו מוות תאים במינונים גבוהים). בלינוסטט הוא מעכב HDAC הגורם לתגובה מקרית (עיכוב גדילה חלקי והפעלת מוות בכל מנה, אך עם פרופורציות שונות בין המנות). באמצעות ניתוח קונבנציונלי של נתונים אלה, ניתן לראות שכל שלוש התרופות מפחיתות באופן משמעותי את הכדאיות היחסית של תאי U2OS (איור 2G). למרות ש-Palbociclib הוא פחות חזק באופן ניכר, לא ברור מנתונים אלה ש-Palbociclib לא מצליח להפעיל מוות תאי. באמצעות ניתוח מבוסס GR, המדווח כיצד תרופות משפיעות על קצב גידול האוכלוסייה נטו, ניתן לפרש בצורה מדויקת יותר שאוכלוסיות שטופלו ב-Palbociclib ממשיכות להתרחב בכל המינונים, בעוד שמינונים גבוהים של Camptothecin או Belinostat גורמים להתכווצות האוכלוסייה, ולכן מוות תאי חייב להיות מופעל בעקבות מינונים גבוהים של תרופות אלה (איור 2H). עם זאת, ניתוח מבוסס GR אינו מביא בחשבון תיאום מגוון בין גדילה למוות, מה שעלול להוביל למסקנות שגויות לגבי רמת המוות התאי המופעלת על ידי תרופה נתונה. לדוגמה, בהתבסס על נתוני GR, ניתן להסיק ש-1 מיקרומטר קמפטותצין מפעיל מוות תאי במידה רבה יותר מ-1 מיקרומטר בלינוסטט, בהתחשב בכך שקמפטותצין מביא לערך GR של -0.25 ובלינוסטט מביא לערך GR של 0; ערך של -0.25 GR פירושו שהאוכלוסייה מתכווצת ב-25% במהירות שבה האוכלוסייה הלא מטופלת מתרחבת. באמצעות הדמיה וניתוח מבוססי GRADE, ניתן לראות כי קמפטותצין ובלינוסטט נבדלים זה מזה בתיאום הגדילה/מוות שלהם. לפיכך, במינון של 1 מיקרומטר, גם בלינוסטט מפעיל מוות בשיעור גבוה יותר מאשר קמפטותצין (0.8% מוות לשעה עבור בלינוסטט, בהשוואה ל-0.7% מוות לשעה עבור קמפטותצין), אולם האוכלוסייה שטופלה בקמפטוצין מתכווצת מהר יותר עקב עיכוב גדילה בולט יותר שנגרם על ידי תרופה זו (איור 2I).

איור 1: תרשים זרימה של פרוטוקול FLICK. (A) צור דילול תאים כדי למטב את ריכוז צבע ה- DNA ואת הליניאריות של מדידות הקרינה על קורא מיקרו-לוחות. (B) השג טיטרציה על פני מספר התאים עבור 2 מיקרומטר SYTOX ירוק בתאי U2OS. רווח של 130 מספק את האות הליניארי ביותר בכל מספרי התאים. (C) תמונות פאזה של תאי U2OS שעברו ליזה עם 1.5% Triton-X לאורך זמן באמצעות מטרה של פי 10. האזור הקופסה הוא תצוגה מוגדלת של הקרינה הירוקה של SYTOX. פסי קנה מידה מייצגים את כל התמונות. (ד) אות ירוק של SYTOX לאחר הליזיס בזמנים שצוינו. הנתונים הם ממוצע +/- SD עבור 3 שכפולים ביולוגיים. (ה) סקירה מושגית של הבדיקה, מההגדרה ועד לניתוח. השלבים העיקריים הם יצירת צלחת דילול תרופות, סימום תאים בצלחת מרובת בארות, רכישת מדידות פלואורסצנטיות לאורך זמן וחישוב מדדי גדילה ומוות. עבור לוחות B ו-D, הנתונים הם ממוצע ± SD עבור n = 3 שכפולים ביולוגיים בלתי תלויים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 2: אפיון תגובות לתרופות באמצעות מבחן FLICK וניתוח מבוסס GRADE. (A) מדד GR עבור מעכב HDAC, בלינוסטט, מעל מינון של מנות. הנתונים המוצגים הם מנקודת הקצה של הבדיקה, שהייתה 66 שעות. ב-1 מיקרומטר, לבלינוסטט יש ערך GR של כ-0, המצביע על תגובה ציטוסטטית. (B) קינטיקה קטלנית של שברים עבור בלינוסטט. ב-1 מיקרומטר בלינוסטט גורם לקטלניות של כמעט 50%, שאינה נלכדת על ידי ערך ה-GR. (C) דיאגרמת שלב GRADE המבוססת על סימולציה של כל שיעורי הגדילה והתמותה האפשריים. הסימולציה התבססה על זמן ההכפלה של U2OS בבדיקה זו (27.12 שעות). (D) תרשים GRADE עבור בלינוסטט על פני המינון בנקודת הסיום של הבדיקה. (E) תרשים עמודות של שיעורי גידול מוסק בדרגה (p, הכפלת אוכלוסייה לשעה) ומוות (d, חלק מהאוכלוסייה שמתה בשעה) עבור מנות של Belinostat. השיעורים נקבעו משיעורים מדומים ב-(C) ונתונים מ-(D). (F) תרשים שבר קטלני עבור 1 מיקרומטר בלינוסטט שטופל ב-50 מיקרומטר (סגול) או בלי (שחור) של המעכב האפופטוטי z-VAD. ΔLF = 0.18 חושב על ידי הפחתת ה-LF המקסימלי הממוצע מהמצב שטופל במעכב מה-LF המקסימלי הממוצע מהבקרה. (G) כדאיות יחסית (מספר התאים החיים במצב שטופל בתרופה, חלקי מספר התאים החיים במצב הלא מטופל) עבור שלוש תרופות לאחר חשיפה של 72 שעות. כל התרופות מפחיתות את כדאיות התאים, אך התרומה של מוות תאים לתגובה אינה ידועה. (H) מדד GR עבור שלוש תרופות ב-(G). נתונים אלה מראים כי שתיים מתוך שלוש התרופות גורמות להתכווצות האוכלוסייה, כפי שמצוין על ידי GR < 0. התרומה של מוות תאי עדיין אינה ברורה, אם כי מוות תאי חייב להיות מופעל כאשר ערך ה-GR שלילי. (I) שיעורים מוסקים בדרגה עבור שלוש תרופות. פלבוצ'יקליב אינו קטלני אך גורם לעצירת גדילה. קמפטותצין הוא דו-פאזי; מינונים נמוכים יותר גורמים לעצירת גדילה בעוד מינונים גבוהים יותר גורמים לעצירת גדילה ואחריה קטלניות. בלינוסטט היא תרופה מקרית; טווח המינון חותך את אמצע תרשים ה-GRADE, מה שמצביע על כך שכל המינונים גורמים להפרעה בגדילה ולהפעלת מוות תאי. הנתונים בכל הפאנלים הם הממוצע ± SD עבור שלושה ניסויי שכפול ביולוגיים עצמאיים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| צבע | קינטית | נקודת סיום |
| ירוק SYTOX | Y | Y |
| ירוק CellTox | Y | Y |
| NucSpot 500/515, 594/615, 750/780 | Y | Y |
| יויו-3 | Y | Y |
| פרופידיום יודיד | N | Y |
| 7-AAD | N | Y |
טבלה 1: צבעי DNA ויישומם ב-FLICK. ניתן להשתמש בסוגים רבים של צבעים עבור מבחני FLICK של נקודת קצה בלבד. פחות צבעים עומדים בתנאים שיש לדגור ברציפות לאורך כל הבדיקה. צבעים אחרים, כולל צבעים שאינם ירוקים, לא אופיינו לעומק ויש להעריך אותם בקפידה לפני השימוש.
| נתיב המוות | מעכב | מינון אופייני |
| אפופטוזיס פנימי | ZVAD-FMK | 50 מיקרומטר |
| אפופטוזיס חיצוני | Z-IETD | 30 מיקרומטר |
| פרופטוזיס | פרוסטטין-1 | 10 מיקרומטר |
| נקרופטוזיס | נקרוסטוטין-1 | 50 מיקרומטר |
| פירופטוזיס | VX-765 | 50 מיקרומטר |
| פרטנטוס | רוקפאריב | 1 מיקרומטר |
| מוות תאי תלוי אוטופאגי | הידרוקסיכלורוקווין (HCQ) | 10 מיקרומטר |
| מוות תאי תלוי ליזוזומלי | E-64D | 10 מיקרומטר |
| קופרופטוזיס | טטרתיאומוליבדט (TTM) | 5 מיקרומטר |
| אוקסיפטוזיס | N-אצטיל-L-ציסטאין (NAC) | 2 מ"מ |
| נמק מונע MPT | ציקלוספורין A (CsA) | 10 מיקרומטר |
טבלה 2: מסלולי מוות תאים ומעכביהם. יש לתקף מעכבי מוות תאי כנגד מפעיל קנוני עבור כל מסלול מוות. מינוני מעכבים נבחרים לא אמורים להשפיע באופן אידיאלי על כדאיות התאים.
טבלה משלימה 1: מחשבון GRADE. גיליון אלקטרוני לחישוב שיעורי צמיחה ותמותה הנגרמת על ידי תרופות מנתוני GR ו-FV מבוססי FLICK. על המשתמשים להזין נתוני GR ו-FV, יחד עם אורך הבדיקה בשעות, זמן הכפלת התאים בשעות וגודל אוכלוסייה התחלה/סיום. אנא לחץ כאן להורדת קובץ זה.
Discussion
בדיקת FLICK היא שיטה חזקה ליצירת הערכה מקיפה של השפעת התרופה על צמיחה ומוות של אוכלוסיית תאים. מכיוון ששיטה זו אינה סופרת תאים ישירות, השלבים הקריטיים בפרוטוקול הם להבטיח ליניאריות בדיקה וליזה מלאה במהלך שלבי חדירת הטריטון. ניתן לזהות את זמן החדירות הנכון באופן ויזואלי, כפי שמודגש בפרוטוקול זה, או כמותית על ידי קריאת הקרינה של הצלחת לאורך זמן וזיהוי מתי האות מישור. עם זאת, מניסיוננו, כאשר יש ספק, לא נראה שיש מחיר להמתנה ארוכה יותר: עבור צבעי ה-DNA שבהם השתמשנו בבדיקת FLICK, עוצמת האות נשארת יציבה במשך מספר ימים לאחר הליזיס. אנו משתמשים באופן קבוע בצבע חומצת הגרעין הירוקה של SYTOX, ולמרות שלא אימתנו כל פלואורופור המוזכר בטבלה 1, מספר פרוטוקולים אחרים מדגימים את השימוש היעיל בהם 13,15,16. הערכת הליניאריות של צבע DNA נתון על פני מגוון של מספרי תאים והגדרות מכשירים תבטיח נתונים חזקים ואמינים מבחינה כמותית.
קווי תאים מסוימים קשים יותר לליזה מאחרים, ולעולם אין להניח שזמן הליזה של קו תאים אחד זההל-13 אחר. עבור קווי תאים מסוימים, בצפיפות גבוהה יותר, ליזיס תאים גורם לתאים להתרומם מהבאר כגיליון, מה שעלול לגרום לשגיאת קורא צלחות. ניתן להימנע מכך על ידי הפחתת צפיפות הזריעה ההתחלתית או ערבוב פיפטות הבארות לאחר הליזיס ולפני הקריאה. כדי לשפר את זמן הליזיס או את יעילות הליזיס, ניתן להתאים את תמיסת Triton-X לאחוז גבוה או נמוך יותר, אך עדיין לא נתקלנו בתרחיש שבו זה היה נחוץ.
יש לשקול בזהירות תכנון ניסיוני ופריסת ציפוי תרופות כאשר רוצים נתוני צמיחה חזקים. יצירת צלחת דילול תרופות לבדיקת FLICK זהה לכל בדיקת צלחת אחרת של 96 בארות. עם זאת, הכללת בקרות רבות המכסות חלקים שונים של נוף הצלחת תסייע ביצירת תובנות חזקות לגבי קינטיקה של צמיחה ויכולה לעזור לקבוע אם קיימת שונות צמיחה שיטתית. הימנעות מהבארות החיצוניות של צלחת תייצר נתוני צמיחה מדויקים יותר, מכיוון שבארות אלו רגישות יותר לתנודות טמפרטורה, חמצן או לחות, מה שמשפיע על צמיחתן. לבסוף, שימוש בדפוס סימום פסאודו-אקראי, כגון החלפת מיקום הסם על לוחות משוכפלים, יספק הערכה מדויקת יותר של התנהגות הסמים.
קיימות מגבלות מסוימות לשימוש המדויק במבחן FLICK. מגבלות אלו קשורות בעיקר להסקה קינטית, מכיוון שנתוני נקודת הקצה הם ערכים שנצפו בניסוי. גם קינטיקה של התפשטות תאים חיים וגם קינטיקה של מוות תלויים בהנחה כלשהי לגבי מסלול הצמיחה של תאים חיים לאורך זמן במצב המטופל בתרופות. בפרוטוקול זה, אנו מתארים את מסלול הצמיחה הזה כאקספוננציאלי ובעל קצב אחיד לאורך זמן. בעוד שסביר להניח שתכונות אלה ייצפו בהיעדר תרופה, ייתכן שלא ניתן להקפיד עליהן בנוכחות תרופה. שיטת FLICK מדויקת, לא בגלל שההנחות לגבי מסלולי הגדילה תמיד נכונות, אלא בגלל שההשפעה של הנחות שגויות כלשהן ממוזערת אם האוכלוסייה המטופלת בתרופות לא מתרבה הרבה (כלומר, פחות מ-2 או 3 הכפלות אוכלוסייה בתקופת הבדיקה). לא נתקלנו בתרופות שלא ניתן לאפיין במדויק ב-FLICK; עם זאת, התיאוריה מציעה כי בדיקת FLICK תפסיק להיות מדויקת עבור תרופה שבה המוות מתרחש לאט מאוד, לאורך תקופות זמן ארוכות שהן כמה כפולות מזמן הכפלת התא (הערה: לא זיהינו תרופות עם תכונות אלה3). עם זאת, ההשפעה של הנחות שגויות כלשהן על קינטיקה של הגידול של כלל האוכלוסייה לא תשפיע על זמן תחילת המוות המתקבל מהתאמת ה-LED או על ערכי ה-LF המקסימליים הסופיים, מכיוון שאלה מוגבלים על ידי ערכי התאים המתים שנמדדו אמפירית, וערכי התאים הכוללים הסופיים שנקבעו בניסוי בסוף הבדיקה.
למרות מגבלות אלה, בדיקת FLICK מאפשרת תובנות שמאתגרות להפיק באמצעות שיטות תגובה אחרות לתרופות. רוב שיטות התגובה לתרופות מייצרות אות פרופורציונלי למספר התאים החיים, וניתן להשתמש בשיטות אלה כדי לכמת כיצד תרופות משפיעות על קצב גידול האוכלוסייה נטו (כלומר, ערך GR או שווה ערך) אך אינן יכולות להבחין במדויק בין ההשפעות הציטוטוקסיות לעומת הציטוסטטיות של תרופה. לחלופין, מבחנים מבוססי מיקרוסקופיה המודדים תאים חיים ומתים יכולים בהחלט לייצר תמונה מקיפה. עם זאת, בדיקות מבוססות מיקרוסקופיה עשויות להתקשות לספור תאים מתים לאחר שהתאים מתפרקים לפסולת, כפי שיקרה במהירות בהקשר של צורות רבות שאינן אפופטוטיות של מוות תאי. מאפיין מרכזי של FLICK הוא שהמדידות נעשות בקורא צלחות, שצובר את סך הקרינה של התאים המתים במקום לספור ישירות תאים מתים. לפיכך, אות התא המת ב-FLICK אינו תלוי בתאים שלמים של תאים מתים, אשר, בהקשר של מוות תאים לא אפופטוטי, הוא חיוני באופן ייחודי. יתר על כן, מאפיין ייחודי של בדיקת FLICK הוא היכולת למדוד הן את אוכלוסיות התאים החיים והן את אוכלוסיות התאים המתים באמצעות אותו ריאגנט. לפיכך, בדיקת FLICK מייצרת מדידות של תאים חיים ומתים ברגישות שווה. תכונה זו מאפשרת ניתוח בשיטת GRADE ומשפרת את הדיוק של חישובי הגידול ושיעור התמותה בו-זמנית של GRADE. הטבלה המשלימה המצורפת 1 כוללת תבנית המשמשת להדמיה של תרופת GRADE, שבה המשתמש יכול להזין את אורך הבדיקה שלו, קצב גידול מחושב משלב 7.1, מספרי תאים התחלתיים וסופיים או קריאות פלואורסצנטיות מתנאי בקרה, ואת ה-LF וה-GR המחושבים משלבים 5.4 ו-6.3. הקובץ מכיל סימולציה של כל השילובים הזוגיים של 50 שיעורי גידול ותמותה שיתעדכנו אוטומטית על סמך הפרמטרים המוגדרים על ידי המשתמש. התבנית מייצרת הדמיה מבוססת GRADE של תגובת התרופה ושיעורי הצמיחה (p) והתמותה (d) המוסקים מ-GRADE.
עבודה עתידית צריכה לחקור את היישום של מבחן FLICK בתרחישי תרבית תאים תלת מימדיים או בהערכת תגובות תרופתיות של אורגנואידים שמקורם בגידול. הליניאריות והרגישות של נתוני FLICK לא נחקרו לעומק בהקשרים אלה, אך בתיאוריה, בדיקת FLICK אמורה להיות יעילה עם כמה שינויים כדי לשלב פלואורסצנטיות על פני דגימות תלת מימדיות. בנוסף, הוספת תוויות נוספות ספציפיות לתא תעזור להבחין בין שני סוגי תאים או יותר בתרבית משותפת. התקדמות זו תהיה בעלת ערך לחקר אינטראקציות של תאי חיסון עם תאים סרטניים. לבסוף, מבחנים כמו SPARKL מעוררים השראה לשימוש במדווחים ספציפיים למוות בפורמט בדיקת FLICK, שעשוי לשפר את התפוקה תוך שמירה על הדיוק של תובנות מוות תאים11,16.
Disclosures
למחברים אין ניגודי אינטרסים לחשוף.
Acknowledgements
אנו מודים לכל חברי מעבדת לי בעבר ובהווה על תרומתם לנקודת המבט של המעבדה שלנו על הערכת תגובות לתרופות. עבודה זו נתמכה על ידי מימון מהמכונים הלאומיים לבריאות ל-MJL (R21CA294000 ו-R35GM152194).
Materials
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
References
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved