Method Article
使用 FLICK 分析对药物反应进行全面分析
摘要
该方案描述了如何使用 FLICK 测定法评估药物反应,包括使用该测定法计算药物诱导的生长速率和死亡率以及评估药物诱导的细胞死亡机制的详细说明。
摘要
为了了解药物疗效,一个关键需求是表征药物诱导的细胞死亡的程度。量化药物诱导的细胞死亡水平的努力受到十几种分子不同形式的调节死亡的挑战,每一种都有自己的激活时间和生化标志特征。此外,对于某些坏死性死亡亚型,标志性特征仅短暂观察到,并且由于细胞破裂而迅速丢失。因此,即使使用死亡途径特异性检测的组合,准确量化细胞死亡总量或每种死亡亚型的相对贡献也具有挑战性。另一个问题是,许多死亡特异性检测忽略了药物如何影响细胞增殖,因此很难解释药物治疗的群体是扩大还是缩小。FLICK 测定允许以特定于死亡的方式量化刺激后细胞死亡的总水平,但在很大程度上与激活的死亡类型无关。此外,FLICK 检测保留了有关总群体大小和细胞增殖速率的信息。在本手稿中,我们描述了 FLICK 测定的基本用途,在使用不同类型的生物材料时如何排除该测定的故障,以及如何使用 FLICK 测定量化每种类型的细胞死亡对观察到的药物反应的贡献。
引言
对于抗癌药物,药物敏感性的临床前评估通常涉及测试药物如何影响培养物中细胞的活力1。药物暴露后的细胞活力是至少两种独立作用的产物:药物诱导的细胞增殖抑制和细胞死亡的激活2。不幸的是,尽管细胞死亡是持久药物反应所需的关键特征,但标准方法未能阐明药物激活细胞死亡的程度3。
常见的药物反应检测包括直接计数细胞的检测(例如,Coulter 计数器,流式细胞术或显微镜的某些用途),量化细胞增殖能力(例如,集落形成检测)或量化代谢活性(例如,CellTiter-Glo、基于四唑的 MTT 或 MTS 检测)。这些检测的一个共同特点是生成的数据与活细胞的数量成正比。由于药物在协调生长抑制和细胞死亡方面差异很大,因此药物暴露后活细胞的数量无法可靠地了解药物诱导的细胞死亡水平3。此外,由于癌细胞通常在细胞培养中迅速增殖,因此相对于未处理的细胞群,活细胞的数量可以显著减少,而不会诱导任何细胞死亡4。因此,一个核心缺陷是,如果不测量活细胞和死细胞的数量,就无法量化细胞死亡的程度。
由于多种原因,准确量化药物暴露后死细胞的数量具有挑战性2。首先,存在十几种受调节的细胞死亡途径 5,6,7,8。尽管通常存在用于识别每种类型的受控细胞死亡的生化标志物,但这些标志物的特异性各不相同,并且没有单一的测定法可用于同时量化所有死亡亚型。其次,每种形式的细胞死亡的激活时间可能会因环境而异,因此除非随着时间的推移量化死亡,否则无法形成完整的画面 9,10。许多用于定量细胞死亡的生化检测会产生终点测量,因此生成动力学数据可能具有挑战性且受成本限制。第三个复杂情况是,死细胞本身是活细胞状态和解离细胞碎片之间的瞬时中间状态。死细胞的稳定性因死亡亚型而异,某些类型(如细胞凋亡)会产生相对稳定的尸体,而其他类型的死亡会导致快速裂解。因此,需要收集和计数死细胞的死亡量化方法也会产生对细胞死亡的偏见理解。最后,第四个限制是量化细胞死亡程度的生化分析通常无法提供关于药物如何改变增殖的任何见解。因此,无法解释总体人口规模——更重要的是,人口是扩大还是缩小——是无法解释的。
一些基于显微镜的检测方法(如 STACK 和 SPARKL)可有效测量活细胞和死细胞随时间的变化,并且这些检测可以全面了解药物诱导的细胞死亡10,11。然而,这些检测需要专门的仪器,例如 Incucyte 显微镜,这限制了通量和使用这些方法的机会。此外,基于显微镜的技术要求死细胞在整个实验期间保持在显微镜的焦平面中,当死细胞失去粘附或随着时间的推移随着死细胞腐烂而失去粘附时,会影响对死细胞的定量能力。同样,当应用于悬浮培养时,基于显微镜的分析面临挑战,因为细胞会漂移进出给定的焦平面。
为了解决上述突出的问题,我们生成了一种称为 FLICK (基于荧光和裂解依赖性的细胞死亡动力学推断) 的测定法12,13。FLICK 检测的目标是确定药物诱导的细胞死亡水平,无论细胞是如何死亡的。FLICK 方法使用细胞不可渗透染料,其荧光取决于 DNA 结合。FLICK 的一个关键特征是使用这些荧光团通过其可接近的 DNA 来标记死细胞随时间推移的积累,然后在检测结束时进行基于机械去污剂的裂解以透化任何活细胞。这些数据与数学建模相结合,能够以连续的时间分辨率量化活细胞和死细胞群,而无需收集或处理死细胞。此外,使用酶标仪评估死细胞荧光可以评估死细胞,而无需死细胞保持完整,从而减轻对导致细胞破裂的坏死形式的死亡的偏见。最后,FLICK 分析需要最少的板处理,并且可以快速生成动力学测量,从而实现高通量药物筛选。在该方案中,我们重点介绍 FLICK 测定法的使用,包括如何使用 FLICK 推断药物诱导的生长速率、死亡率和/或细胞死亡机制。
研究方案
1. 优化每个目标细胞系的透化时间
注:描述的体积和数量用于优化一种细胞系。这些值应根据要测试的细胞系数量进行放大。
- 在 96 孔、光学底部、黑壁板的每个孔中接种所需数量的细胞。加入 100 μL 完全培养基,让细胞粘附在板上过夜。
注:应考虑细胞的生长速率、最佳密度和测定时间,优化铺板的细胞数量。在 72 小时内测量的贴壁癌细胞系的典型起始细胞数为每孔 1500 - 5000 个细胞。例如,可以将 U2OS 细胞以每孔 2000 个细胞的密度接种在含有 10% FBS、2 mM 谷氨酰胺和 1% Pen-Strep 的 DMEM 中,并在标准条件(5% CO 2,37 °C,湿度)下培养过夜。 - 在 15 mL 锥形离心管中,制备 1.5 mL 的 1.5% Triton-X 磷酸盐缓冲盐水 (PBS) 溶液。以最大速度涡旋 1.5% Triton-X 溶液 5 秒。置于 37 °C 水浴中至少 30 分钟。
- 目视检查溶液,确保 Triton-X 通过涡旋几秒钟完全溶解。确保解决方案是同质的。
- 向铺板细胞的每个孔中加入 10 μL 的 1.5% Triton-X 溶液。请勿混合。
注意:在此阶段,由于会形成气泡,因此不希望使用重复移液进行混合。 - 将细胞放回 5% CO2,37 °C 中,用湿度细胞培养箱至少 1 小时。
注:施用 Triton-X 溶液后,大多数细胞系在 2-3 小时内完全裂解。当使用 SYTOX Green 等染料时,等待长达 24 小时对荧光信号的影响最小。增加 Triton-X 的百分比可能会缩短难以裂解的细胞系的孵育时间。 - 使用 10 倍物镜在光学显微镜上观察细胞形态。每小时检查一次细胞,直到细胞体不再可见。记录细胞透化所需的时间。
注:如果有荧光显微镜,可以使用细胞不可渗透的 DNA 染料(如 SYTOX Green)观察裂解。染料可以以 10 倍终浓度添加到 Triton-X 溶液中,并且可以通过荧光显微镜确认透化。
2. DNA 染料的选择和校准
注:FLICK 检测的要求是使用细胞不可渗透性荧光团,该荧光团以 DNA 结合依赖性方式发出信号,不影响细胞活力,并产生随细胞数量线性缩放的信号。该协议使用 SYTOX Green。其他具有相似特性的染料也可能适用于 FLICK 测定,但应分别进行评估和校准。有关示例,请参阅 表 1 。
- 对于所选的 DNA 染料,根据制造商的建议确定要测试的浓度范围。
- 对于要测试的每种浓度,将 40,000 个细胞接种在 180 μL 细胞培养基中,沿 96 孔光学底部黑壁板最左侧的列一式三份。向剩余的孔中加入 90 μL 培养基。
注:这将用于在板的行中生成线性细胞滴定。第一批孔的最终浓度为 20,000 个细胞/孔。从孔顶部读取荧光的读板器不需要光学底板。细胞培养基应与用于培养被测细胞的培养基相同。例如,如果使用 U2OS 细胞,则应将 40,000 个细胞重悬于 180 μL 的 DMEM 中。 - 使用多通道移液器,将 90 μL 从最左侧的色谱柱转移到右侧的相邻色谱柱中,从而实现 1:2 的连续稀释。移液 15 次混合。
- 重复步骤 2.3,从第二列移动到第三列,然后从第三列移动到第四列,依此类推。在板的倒数第二列完成滴定。留下最后一列不含细胞,以便获得细胞培养基的背景信号。
- 对于倒数第二根色谱柱,去除 90 μL,以便所有孔都有 90 μL 含有不同数量细胞的培养基。让细胞在 37 °C 细胞培养箱中粘附 6 小时。
- 为每种待测染料浓度制备 10x DNA 染料溶液,将 DNA 染料稀释到用于培养目标细胞的完全生长培养基中。如步骤 1 中所述,在 PBS 中制备 1.5 mL 的 1.5% Triton-X 溶液。
- 向 96 孔板的每个孔中,加入 10 μL 的 10x DNA 染料。向每个孔中加入 10 μL 1.5% Triton-X 溶液以透化细胞。将细胞在该溶液中孵育最佳时间,该时间在步骤 1 中确定。
注:添加 Triton-X 后,DNA 染料的最终浓度略低于 1 倍,但这无关紧要。孵育后,应裂解整个板。手动细胞滴定将用于确定最佳 DNA 染料浓度和获取荧光的最佳设置。 - 测量整个细胞滴定板的荧光强度。在 DNA 染料制造商概述的一系列采集设置中定量荧光。根据所使用的读板器,修改激发和发射波长和/或数字增益。
- 通过减去不包含单元格的列的平均信号,从每次测量中删除背景信号。这些值可以在连续稀释板最右侧的孔列中找到。
- 确定每种采集设置下每种 DNA 染料浓度的线性。可以通过根据荧光信号绘制细胞数并执行线性回归来确定线性。决定系数 (r2) 报告荧光信号与细胞数量线性相关的程度。
- 选择具有线性和动态范围最佳组合的采集设置和 DNA 染料浓度。
注意:考虑从 0 到 20,000 个单元格的整个单元格编号范围。确保在低细胞数(介于 0 到 1000 个细胞之间)的增益和 DNA 染料浓度呈线性。几种仪器设置或 DNA 染料浓度可能在整个范围内提供很强的相关性。然而,低端灵敏度必须稳定,这样才能准确测量死细胞中的微小变化。
3. 药物处理板中的细胞铺板、孔内药物应用和死细胞荧光随时间的变化测量
注:药物稀释板可根据实验需要灵活设计。通常,药物稀释板将包括一种或几种药物的对数或半对数稀释系列。
- 考虑实验的最佳电镀布局。避免从 96 孔板的外孔进行测量,以减少与生长变化相关的噪音,因为与板的内孔相比,外孔经历不同的温度、氧合和蒸发。
注意:在整个板景观中包括对照将增加生长数据的稳健性。考虑在板的中心和边缘包括对照孔。 - 确定实验所需的板数。包括一个额外的板,在测定开始时进行裂解,以确定给药时每个孔的平均细胞数(即 T0 对照板)。
- 在完全生长培养基中制备细胞悬液。
- 根据实验所需的板数(在步骤 3.2 中计算),确定所需的细胞悬液体积。如果以每孔 90 μL 的浓度接种板,则每板大约需要 10 mL。将所需的板数乘以 10 mL。
注意:最好增加此体积以解决储液槽的死体积和移液过程中的体积损失。例如,计算出的交易量可以乘以 1.2。 - 对细胞进行计数,并根据每孔所需的细胞数量制备细胞稀释液。例如,如果目标是在 96 孔板的每孔 90 μL 培养基中接种 2000 个细胞,则总共 3 个板,计算实验所需的细胞总数如下:

同样,如果计数的细胞悬液为 400,000 个细胞/mL,则实验所需的细胞悬液体积为:

- 通过减去要添加的计数细胞悬液的体积来校正用于铺板的细胞培养基的体积。
36 mL 培养基 - 2 mL 细胞悬液 = 34 mL 校正培养基体积
- 根据实验所需的板数(在步骤 3.2 中计算),确定所需的细胞悬液体积。如果以每孔 90 μL 的浓度接种板,则每板大约需要 10 mL。将所需的板数乘以 10 mL。
- 使用血清移液管将计数的细胞悬液与正确的培养基体积混合。将此细胞悬液转移到 V 形底试剂储液槽中。
- 使用多通道移液器,向 96 孔板的每个孔中加入 90 μL 细胞悬液。用血清移液管定期重复移液,将试剂储液槽中的细胞悬液混合,以确保维持所需的单位体积细胞浓度。
- 让细胞在 37 °C 细胞培养箱中粘附过夜(通常为 12-24 小时)。
- 如步骤 1 中所述,在 PBS 中制备 1.5% Triton-X 溶液。该溶液的体积应足以裂解所有板。PBS 中约 1.5 mL 的 1.5% Triton-X 足以裂解一个 96 孔板。
注:PBS 中的 1.5% Triton-X 溶液可稳定保存一周以上,为方便起见,可以提前制备。 - 确定制备药物稀释板所需的细胞培养基量。在 U 形或 V 形底 96 孔透明板中制备药物稀释板。
- 为确保稀释药物的体积足以进行实验,请增加所需的最小体积,以解决移液过程中的体积损失。例如,如果要向 96 孔板的每个孔中加入 10 μL 药物,并且实验包括 3 块板,则每孔需要 30 μL 稀释的药物。每孔准备 40-50 μL 的药物培养基,以解决体积损失。向所需培养基体积中加入 1.5 mL,以考虑 T0 对照板所需的培养基。
- 在完全生长培养基中制备 10 倍浓度的选定 DNA 染料。该浓度基于步骤 2.11 中选择的浓度。该解决方案的总体积在步骤 3.8 中计算。
- 使用步骤 3.9 中的 DNA 染料 + 生长培养基,为每种待测药物制备 10 倍浓度。在药物稀释板中仅制备每种药物的最高剂量,并用多通道移液器连续稀释药物,每个孔之间混合 15 倍。
注:当在药物稀释板中为给定药物进行最高剂量时,应考虑药物的体积以确保浓度准确。 - 使用多通道移液器,将步骤 3.10 中的 10 μL 药物 + DNA 染料溶液添加到含有细胞的板中。
注:对于连续稀释的药物稀释板,如果从最低药物浓度开始向细胞中加入药物,并逐渐增加到最高浓度,则无需更换多通道移液器的吸头。但是,多通道移液器上的吸头应在每块板之间以及在含有不同药物或更高浓度药物的孔中使用时更换吸头。 - 读取添加药物的所有板的荧光。该荧光读数是 T0 读数(即,时间 = 0 小时的死细胞)。
- 读取 T0 荧光读数后,将板放回培养箱中。
- 将 10 μL 步骤 3.9 中制备的 10x DNA 染料溶液和 10 μL 步骤 3.7 中制备的 1.5% Triton-X 溶液添加到 T0 对照板中。将此板放回培养箱中,放置步骤 1.8 中选择的时间。
- 细胞完全裂解后读取 T0 对照板,如步骤 1.6 中所述。
4. 测量药物处理板随时间变化的死细胞荧光
注意:尽量减少板从培养箱中取出的时间。长时间的温度变化会影响细胞活力,而暴露在光线下会损害 DNA 荧光团,例如 SYTOX Green。
- 给药后每 3-4 小时获取所有药物处理板的荧光读数。除非需要非常精确的死亡动力学,否则不需要在一夜之间读取板。
注意:不需要以固定/标准间隔采集时间点。通常,时间点可以仅在正常工作时间内采集,而不会在死亡动力学的下游分析中导致错误。只要在死亡开始前、死亡上升阶段和饱和/平台期进行一些测量,就可以推断出动力学。 - 在所需的最终时间点,获取荧光读数。紧接着,加入 10 μL 在步骤 3.7 中制备的 1.5% Triton-X 溶液以裂解细胞。
- 将板放回培养箱中,让细胞在步骤 1 中确定的时间内透化。
- 透化后获取荧光读数。该荧光值与每个孔的细胞总数(即活细胞 + 死细胞)成正比。
5. 计算致死分数动力学
注:本协议中描述的计算可以用任何格式或软件进行分析。但是,使用 MATLAB、R 或 Python 等编程环境将允许更快、更灵活的分析。
- 使用 50% 修剪平均值计算 T0 对照板的平均荧光值。该值与给药前的细胞总数成正比。
- 使用曲线拟合和指数增长函数,计算实验中所有井的种群增长动力学。每个孔的初始种群大小是在步骤 5.1 中计算的平均 T 0 荧光值。每个孔的最终种群大小是在步骤 4.4 中计算的透化后值。测定的持续时间从 0 小时到步骤 4.4 中选择的测定终点。
- 使用步骤 5.2 中确定的生长参数,计算每个孔的测定中每个测量时间点的总细胞数。通过从步骤 5.2 中计算的总细胞中减去死细胞测量值,确定每个测量时间点的活细胞数。
注:由于检测中的噪音量较小,有时活细胞数可能是一个小的负数。这可以手动设置为 0,因为活细胞的数量不能为负数,并且很可能表示该孔中的所有细胞都已死亡。 - 通过将死细胞荧光信号除以每个时间点的总细胞信号来计算每个时间点的致死分数 (LF)。
- 将滞后指数死亡 (LED) 方程拟合到致命分数时间过程数据10。为避免不会引起任何显着致死性的药物剂量的任意动力学参数,请拟合斜率等于 0 的线性模型。根据分析中的噪音或在未用药条件下观察到的 LF 确定显著的致死率水平。
- 从 LED 方程中记录死亡开始时间 (DO)。LED 方程有四个参数:初始 LF (LFi)、平台期 LF (LFp)、初始死亡率 (DR) 和 DO。使用非线性回归(即曲线拟合)从数据中推断这些参数。
- 计算每种药物在每个剂量下的测定终点的分数活力 (FV)。从 1 中减去终点 LF 值,或将活细胞数除以总细胞数。
注意:FV 可以在任何时间点计算,而不仅仅是在测定终点。这些数据用于评估剂量药理学(例如,药物 IC50 或 EC50)。
6. 计算 GR 值
- 在测定开始时确定每个孔的平均活细胞数。使用步骤 3.15 中计算的透化后 T0 读数减去步骤 3.12 中收集的每个孔的 T0 读数来计算该值。在下面的 GR 公式中(步骤 6.3),该值称为 x0。
注:有时,药物的最高剂量会干扰 DNA 染料的荧光读数。可能需要使用对照孔计算时间 0 处活细胞的平均数量。 - 对于对照孔 (xctrl),确定测定终点处每个孔的平均活细胞数。确定每种药物处理条件的测定终点处的平均活细胞数 (x(c))。
注:根据整个板中细胞生长的变化,可以使用不同的 xctrl 值对每个孔进行归一化。例如,如果 xctrl 值在整个印版上系统地变化,则可能需要使用最接近的 xctrl 值,而不是印版的平均 xctrl 值。 - 对于每个药物处理的井,使用以下公式4 计算标准化生长速率抑制值 (GR):
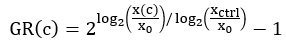
使用 4 参数 Logistic 回归执行曲线拟合。GR 的等级从 -1 到 1。
7. 使用 GRADE 方法计算药物诱导的生长率和死亡率
注:GR 代表净群体增长率,而不是真实细胞增殖率。药物诱导的种群增长和死亡率可以使用 GR 和致死分数 (LF) 的组合来计算。
- 使用 T0 对照读数 (x0) 中的平均活细胞数、测定终点处对照条件下的平均活细胞数 (xctrl) 以及以小时 (t) 为单位的测定长度,确定实验期间细胞系的倍增时间 (τ),以求解以下方程式:

注:细胞倍增时间 (τ) 的准确计算要求细胞在整个检测过程中连续倍增。对于经历接触抑制的细胞,应以起始密度接种细胞,该密度在所需的检测时间内不会汇合。例如,U2OS 的平均倍增时间为 24 小时。在 72 小时的测定中每孔接种 2000 个细胞是解释其生长速率的最佳选择。 - 确定增长率/死亡率与种群规模之间的关系。为此,请使用出生/死亡模型和模拟,该模拟使用生长率和死亡率的所有合理成对组合进行初始化。该模拟需要来自 T0 对照板的平均活细胞数 (x0)、测定长度 (t) 以及用户定义的合理增殖率范围 (p) 和死亡率 (d)。


- 为了确定生长速率的合理值,从细胞群倍增/小时 (1/T) 的未处理生长速率开始,作为最高生长速率,从 0 作为最低生长速率。将此范围划分为 500 个等距段。类似的作可以应用于死亡率,测试从 0 到 1 的范围。
注意:可以使用任何编程环境执行 500 个速率的所有成对组合,但如果使用某些软件,计算量可能会太大。将其减少到 50 个生长率和死亡率的所有成对组合可以缓解这个问题,同时降低推理的精度。此协议提供了电子表格模板。
- 为了确定生长速率的合理值,从细胞群倍增/小时 (1/T) 的未处理生长速率开始,作为最高生长速率,从 0 作为最低生长速率。将此范围划分为 500 个等距段。类似的作可以应用于死亡率,测试从 0 到 1 的范围。
- 确定每个模拟增殖 (p) 和死亡 (d) 速率对的 GR 和 LF 值(如上所述)。
注:步骤 7.2-7.3 将生成一个表格,其中包含增殖率和死亡率的每对理论值,以及该理论对的计算 GR 和 LF 值。这将用作查找表,将实验观察到的一对 GR 和 LF 值与一对药物诱导的增殖和死亡率相关联。测试 500 多个离散值的增殖率和死亡率将产生更精确的数值,但这种精度水平可能超出分析的精度。 - 计算查找表中每个实验计算的 GR/LF 对与每个理论 GR/LF 对之间的成对距离。推断与实验观察到的 GR/LF 值对的最小距离的理论对是真正的药物诱导的增殖和死亡率。
8. 使用通路选择性化学抑制剂确定死亡通路
注:单独的化学抑制剂不足以确定给定药物的死亡机制。应使用死亡途径的化学抑制剂来确定在后续实验中应探索哪些生化或表型反应,这可能包括形态学评估、途径特异性生化标志物和遗传依赖性评估。
- 通过在感兴趣的死亡途径的经典激活剂的背景下测试抑制剂浓度的剂量范围,优化目标细胞系中每种细胞死亡抑制剂的剂量(表 2)。理想情况下,所选剂量的抑制剂不应单独影响细胞活力(检查 GR 指标)。
注:并非所有细胞系都可以激活所有死亡途径。在测试不同死亡途径的激活剂/抑制剂时,可能需要进行生化或表型验证。 - 考虑抑制剂筛选的最佳接种布局。为了最大限度地减少批次效应,将每种药物与正在评估的抑制剂放在同一板上,并在单独的板上重复。
- 在 96 孔、光学底、黑壁板的每个孔中接种所需数量的细胞,类似于步骤 3.3。将铺板体积减少到 80 μL,以考虑抑制剂和药物的体积。
- 准备 10 倍协同的抑制剂,以便在完全培养基中进行测试,并向每个孔中加入 10 μL。用死亡途径抑制剂预处理细胞 2-4 小时。之后,添加感兴趣的药物并获得荧光读数,如步骤 3-5 所示。
- 评估死亡开始时间 (DO) 和/或最大致死分数的变化,以评估抑制剂的有效性。DO 是从滞后指数死亡 (LED) 模型中推断出的参数。请参阅步骤 5.6 - 5.7。
结果
使用该方案,我们探讨了 U2OS 细胞对 HDAC 抑制剂 Belinostat 的敏感性。使用 2 μM SYTOX Green 标记死细胞进行这些实验(图 1A)。使用荧光读板器以 130 增益设置进行动力学读数(图 1B)。测定结束时,将细胞在 PBS 中的 1.5% Triton-X 溶液中裂解 2 小时(图 1C-D)。
FLICK 方案可以深入了解种群增长率和药物诱导的细胞死亡率。这些见解可以单独查看,也可以使用药物 GRADE 可视化和分析框架一起查看(图 1E)。使用 GR 值评估 Belinostat 的灵敏度表明,1 μM Belinostat 导致 GR 值约为 0(图 2A)。在 GR 量表上,正值报告种群扩张率,负值表示种群收缩率。因此,GR 值为 0 表明种群仍处于停滞状态(即细胞抑制药物反应)。鉴于细胞抑制的常规解释,人们可以得出结论,1 μM Belinostat 会导致完全生长抑制,而没有任何细胞杀死。然而,评估药物诱导的致死分数表明,1 μM Belinostat 在该测定过程中导致大约 50% 的致死率(图 2B)。在大多数情况下,这两种见解来自不同的实验,无法以同类方式进行比较。重要的是,使用 FLICK 检测,两种见解都来自相同的实验数据,但分析方式不同,以捕捉 Belinostat 对群体生长与细胞死亡的影响。
这两个见解也可以使用 GRADE 分析方法进行整合(图 2C)。药物 GRADE 将与平均死亡率成正比的致死分数与 GR 值(净人口增长率)并列。以这种方式整合数据可以直观地了解药物如何协调生长抑制和死亡激活。在 GRADE 图内:抑制生长而不激活死亡的药物(即细胞抑制药物,使用通常的解释)将占据上边界,仅杀死而不改变细胞增殖速率的药物将占据右边界,首先引起生长抑制,然后生长停滞细胞死亡的药物将占据左边界(即 双相药物;图 2C)。可以推断,导致落在这些边界内的 GR/LF 反应的药物同时在一定程度上抑制增殖,同时也激活细胞死亡(即重合药物)3。重要的是,每种药物反应在 GR/LF 空间中的位置可用于计算每种药物浓度的真实增殖率 (p) 和平均死亡率 (d) (图 2D-E)。请注意,GR 值不是真实细胞增殖率,而是净种群增长率(即,结合真实增殖率和药物诱导死亡率的净效应)。此外,GRADE 分析中产生的死亡率(d,即平均死亡率)与 LED 动力学分析中的死亡率参数(DR,最大速度或最大死亡率)不同。
FLICK 检测可以生成死亡特异性数据,例如药物诱导的致死分数;然而,这些数据与细胞死亡的机制无关。为了深入了解使用 FLICK 的细胞死亡机制,最简单的方法是确定通过包含死亡途径特异性抑制剂如何改变致死部分动力学。抑制相关的死亡机制应导致药物诱导的致死分数降低和/或延迟死亡发病时间。在这里,我们表明 50 μM 泛半胱天冬酶抑制剂 z-VAD 挽救了 1 μM Belinostat 诱导的约 50% 的致死率(图 2F)。重要的是,与药物一样,抑制剂的特异性有限,可能会意外抑制或加剧其他死亡机制。因此,在得出明确结论之前,这些数据应辅以其他见解,例如细胞形态、活化生化标志物的测量和/或途径特异性遗传依赖性的评估14。
为了正式说明基于 FLICK 的评估和基于 GRADE 的分析如何改变对药物反应的解释,我们接下来探讨了三种具有不同类型生长/死亡协调的药物。Palbociclib 是一种 Cdk4/6 抑制剂,可导致细胞抑制反应 (生长抑制,无细胞死亡)。Camptothecin 是一种拓扑异构酶 I 抑制剂,可引起双相反应 (生长抑制,高剂量后细胞死亡)。Belinostat 是一种 HDAC 抑制剂,可引起重合反应 (每个剂量的部分生长抑制和死亡激活,但不同剂量的比例不同)。使用这些数据的常规分析,可以观察到所有三种药物都大大降低了 U2OS 细胞的相对活力(图 2G)。尽管 Palbociclib 的效力明显较低,但从这些数据中尚不清楚 Palbociclib 无法激活细胞死亡。使用基于 GR 的分析,该报告报告了药物如何影响净种群增长率,可以更准确地解释 Palbociclib 治疗的种群在所有剂量下继续扩大,而高剂量的喜树碱或 Belinostat 导致种群萎缩,因此,细胞死亡必须在高剂量这些药物后被激活(图 2H).然而,基于 GR 的分析未能解释生长和死亡之间的不同协调,这可能导致关于给定药物激活的细胞死亡水平的错误结论。例如,根据 GR 数据,可以得出结论,1 μM 喜树碱比 1 μM Belinostat 激活细胞死亡的程度更大,因为喜树碱的 GR 值为 -0.25,而 Belinostat 的 GR 值为 0;-0.25 GR 值意味着种群萎缩的速度与未经治疗的种群增长的速度一样快 25%。使用基于 GRADE 的可视化和分析,可以观察到喜树碱和 Belinostat 的生长/死亡协调不同。因此,在 1 μM 剂量下,Belinostat 以比喜树碱更高的速率激活死亡(Belinostat 为 0.8% 死亡/小时,而喜树碱为 0.7% 死亡/小时),但由于该药物诱导的生长抑制更明显,喜树碱处理的种群萎缩得更快(图 2I)。

图 1:FLICK 方案的流程图。 (A) 创建细胞稀释液以优化酶标仪上荧光测量的 DNA 染料浓度和线性。(B) 在 U2OS 细胞中,2 μM SYTOX Green 的细胞数增益滴定。增益 130 在所有单元编号中提供最线性的信号。(C) 使用 10 倍物镜,用 1.5% Triton-X 裂解的 U2OS 细胞随时间变化的相位图像。方框区域是 SYTOX Green 荧光的放大视图。比例尺代表所有图像。(D) 在指定时间裂解后的 SYTOX Green 信号。数据是 3 次生物学重复的平均 +/- SD。(E) 分析的概念概述,从设置到分析。关键步骤是创建药物稀释板、在多孔板中对细胞进行药物化、随时间获取荧光测量值以及计算生长和死亡指标。对于图 B 和 D,数据为 n = 3 个独立生物学重复的平均 ± SD。 请单击此处查看此图的较大版本。

图 2:使用 FLICK 测定和基于 GRADE 的分析表征药物反应。 (A) HDAC 抑制剂 Belinostat 在一剂剂量下的 GR 指标。显示的数据来自测定终点,即 66 小时。在 1 μM 时,Belinostat 的 GR 值约为 0,表明细胞抑制反应。(B) Belinostat 的致死分数动力学。在 1 μM Belinostat 时,可诱导近 50% 的致死率,而 GR 值未捕获该致死率。(C) 基于所有可能的生长和死亡率模拟的 GRADE 阶段图。模拟基于该测定中 U2OS 的倍增时间 (27.12 h)。(D) 检测终点处 Belinostat 跨剂量的 GRADE 图。(E) Belinostat 剂量的 GRADE 推断的增长(p,每小时种群倍增数)和死亡(d,每小时死亡的种群比例)的条形图。费率是根据 (C) 中的模拟费率和 (D) 中的数据确定的。(F) 1 μM Belinostat 处理(紫色)或未处理(黑色)50 μM 凋亡抑制剂 z-VAD 的致死分数图。ΔLF = 0.18 是通过从对照的平均最大 LF 中减去抑制剂处理条件的平均最大 LF 来计算的。(G) 暴露 72 小时后三种药物的相对活力(药物处理条件下的活细胞数除以未处理条件下的活细胞数)。所有药物都会降低细胞活力,但细胞死亡对反应的贡献尚不清楚。(H) (G) 中三种药物的 GR 指标。这些数据表明,三种药物中的两种导致种群萎缩,GR < 0。细胞死亡的贡献尚不清楚,尽管当 GR 值为负时,细胞死亡必须被激活。(I) 三种药物的 GRADE 推断率。Palbociclib 是非致命的,但会导致生长停滞。喜树碱是双相的;较低剂量导致生长停滞,而较高剂量导致生长停滞,然后致死。Belinostat 是一种巧合药物;剂量范围穿过 GRADE 图的中间,表明所有剂量都会导致生长扰动和细胞死亡激活。所有面板中的数据是三个独立生物重复实验的 SD ±平均值。 请单击此处查看此图的较大版本。
| 染 | 运动的 | 终点 |
| SYTOX 绿色 | Y | Y |
| CellTox 绿色 | Y | Y |
| NucSpot 500/515、594/615、750/780 系列 | Y | Y |
| YOYO-3 (悠悠球-3) | Y | Y |
| 碘化丙啶 | N | Y |
| 7-AAD | N | Y |
表 1:DNA 染料及其在 FLICK 中的应用。 许多类型的染料可用于仅终点的 FLICK 分析。满足整个检测过程中连续孵育条件的染料较少。其他染料,包括非绿色染料,尚未经过深入表征,应在使用前仔细评估。
| 死亡通路 | 抑制剂 | 典型剂量 |
| 内源性细胞凋亡 | ZVAD-FMK 系列 | 50 微米 |
| 外源性细胞凋亡 | Z-IETD 系列 | 30 微米 |
| 铁死亡 | Ferrostatin-1 (铁司他汀-1) | 10 微米 |
| 坏死性凋亡 | 坏死抑素-1 | 50 微米 |
| 焦亡 | VX-765 型 | 50 微米 |
| 帕塔纳托斯 | 鲁卡帕尼 | 1 微米 |
| 自噬依赖性细胞死亡 | 羟氯喹 (HCQ) | 10 微米 |
| 溶酶体依赖性细胞死亡 | E-64D 系列 | 10 微米 |
| 铜砾病 | 四硫钼酸盐 (TTM) | 5 微米 |
| Oxeiptosis(奥塞皮坤病) | N-乙酰基-l-半胱氨酸 (NAC) | 2 毫米 |
| MPT 驱动的坏死 | 环孢菌素 A (CsA) | 10 微米 |
表 2:细胞死亡途径及其抑制剂。 细胞死亡抑制剂应针对每种死亡途径的经典激活剂进行验证。理想情况下,选定的抑制剂剂量不应影响细胞活力。
补充表 1:GRADE 计算器。 用于从基于 FLICK 的 GR 和 FV 数据中计算药物诱导的生长率和死亡率的电子表格。用户应输入 GR 和 FV 数据,以及检测时长(以小时为单位)、细胞倍增时间(以小时为单位)和开始/结束群体大小。 请点击此处下载此文件。
讨论
FLICK 检测是一种可靠的方法,用于全面评估药物对细胞群生长和死亡的影响。由于该方法不直接计数细胞,因此该方案中的关键步骤是确保 Triton 透化步骤中的测定线性和完全裂解。正确的透化时间可以通过视觉识别,如本方案中强调的那样,或者通过读取板荧光随时间的变化并确定信号何时稳定来定量识别。然而,根据我们的经验,当有疑问时,等待更长时间似乎没有任何成本:对于我们在 FLICK 检测中使用的 DNA 染料,信号强度在裂解后几天内保持稳定。我们经常使用 SYTOX Green 核酸染色剂,虽然我们尚未验证表 1 中提到的每种荧光基团,但其他几种方案证明了它们的有效性 13,15,16。评估给定的细胞不可渗透性 DNA 染料在一系列细胞数量和仪器设置中的线性,将确保数据可靠且定量可靠。
一些细胞系比其他细胞系更难裂解,绝不应假设一种细胞系的裂解时间与另一种13 种细胞系的裂解时间相同。对于某些细胞系,在较高密度下,细胞裂解会导致细胞以片状从孔中抬起,这可能导致读板器错误。这可以通过降低起始接种密度或在裂解后和读取前对孔进行移液混合来避免。为了提高裂解时间或裂解效率,可以将 Triton-X 溶液调整为更高或更低的百分比,但我们还没有遇到需要这样做的情况。
当需要稳健的生长数据时,应仔细考虑实验设计和药物铺板布局。为 FLICK 测定创建药物稀释板与任何其他 96 孔板测定相同。然而,包括许多涵盖板图不同部分的对照将有助于对生长动力学产生强大的见解,并有助于确定是否存在系统性生长变异。避开板的外孔将产生更精确的生长数据,因为这些孔对温度、氧气或湿度波动更敏感,从而影响其生长。最后,使用伪随机药物模式,例如在复制板上切换药物的位置,将提供更准确的药物行为评估。
准确使用 FLICK 分析存在一些限制。这些限制主要与动力学推断有关,因为终点数据是实验观察到的值。活细胞增殖动力学和死亡动力学都依赖于关于在药物治疗条件下活细胞随时间推移的生长轨迹的一些假设。在该协议中,我们将这种增长轨迹描述为指数增长,并且随着时间的推移具有均匀的速率。虽然在没有药物的情况下可能会观察到这些特征,但在有药物的情况下可能无法遵守这些特征。FLICK 方法是准确的,不是因为关于生长轨迹的假设总是正确的,而是因为如果药物治疗的群体没有太多增殖(即,在分析期间少于 2 或 3 个群体倍增),则任何错误假设的影响都会最小化。我们还没有遇到在 FLICK 中无法准确分析的药物;然而,该理论表明,FLICK 测定对于死亡发生非常缓慢的药物将不再准确,持续时间是细胞倍增时间的几倍(注意:我们还没有发现任何具有这些特征的药物3)。然而,对总群体生长动力学的任何错误假设的影响不会影响从 LED 拟合获得的死亡开始时间或最终最大 LF 值,因为这些值受经验测量的死细胞值和最终总细胞值的限制在测定结束时通过实验确定。
尽管存在这些限制,但 FLICK 分析能够获得使用其他药物反应方法难以获得的见解。大多数药物反应方法产生的信号与活细胞的数量成正比,这些方法可用于量化药物如何影响净细胞群增长率(即 GR 值或等效值),但不能准确区分药物的细胞毒性和细胞抑制作用。或者,基于显微镜的检测方法同时测量活细胞和死细胞,当然可以产生全面的信息。然而,基于显微镜的检测可能难以在细胞分解成碎片后对死细胞进行计数,这在许多非凋亡形式的细胞死亡的情况下会迅速发生。FLICK 的一个关键特性是在读板器中进行测量,该读板器聚合总死细胞荧光,而不是直接计数死细胞。因此,FLICK 中的死细胞信号不依赖于死细胞的完整细胞性,这在非凋亡细胞死亡的情况下是独一无二的。此外,FLICK 分析的一个独特功能是能够使用相同的试剂测量活细胞群和死细胞群。因此,FLICK 测定法以相同的灵敏度产生活细胞和死细胞的测量值。此功能支持使用 GRADE 方法进行分析,并提高 GRADE 同时计算生长率和死亡率的准确性。包括 的补充表 1 包括一个用于药物 GRADE 可视化的模板,用户可以在其中输入他们的检测长度、步骤 7.1 中计算的生长速率、对照条件下的起始和结束细胞数或荧光读数,以及步骤 5.4 和 6.3 中计算的 LF 和 GR。该文件包含 50 种生长率和死亡率的所有成对组合的模拟,这些组合将根据用户定义的参数自动更新。该模板生成基于 GRADE 的药物反应可视化以及 GRADE 推断的生长 (p) 和死亡率 (d)。
未来的工作应探索 FLICK 检测在 3D 细胞培养场景或评估肿瘤衍生类器官的药物反应中的应用。在这些情况下,FLICK 数据的线性和灵敏度尚未得到深入询问,但从理论上讲,FLICK 分析应该在进行一些修改以整合 3 维样品中的荧光后是有效的。此外,添加额外的细胞特异性标记将有助于在共培养中区分两种或多种细胞类型。这些进展对于探索免疫细胞与癌细胞的相互作用很有价值。最后,像 SPARKL 这样的检测激发了 FLICK 检测形式的死亡特异性报告基因的使用,这可能会提高通量,同时保持细胞死亡洞察的准确性11,16。
披露声明
作者没有需要披露的利益冲突。
致谢
我们感谢 Lee 实验室的所有过去和现在的成员,感谢他们对我们实验室评估药物反应的观点做出的贡献。这项工作得到了美国国立卫生研究院对 MJL (R21CA294000 和 R35GM152194) 的资助。
材料
| Name | Company | Catalog Number | Comments |
| Belinostat | ApexBio | A4612 | |
| Camptothecin | ApexBio | A2877 | |
| Conical centrifuge tube, 15mL | Fisher Scientific | 12-565-269 | |
| DMEM | Corning | 10017CV | For seeding and drugging cells |
| DMSO | Fisher Scientific | MT-25950CQC | For seeding and drugging cells |
| Fisherbrand 96-Well, Cell Culture-Treated, U-Shaped-Bottom Microplate | Fisher Scientific | FB012932 | For seeding and drugging cells (pin plate) |
| Greiner Bio-One CELLSTAR μClear 96-well, Cell Culture-Treated, Flat-Bottom Microplate | Greiner | 655090 | For seeding and drugging cells |
| IncuCyte S3 | Essen Biosciences | Any phase microscope will work | |
| MATLAB | MathWorks | https://www.mathworks.com/products/matlab.html | MATLAB version R2023b, a license is required |
| Microplate fluorescence reader | Tecan | Spark | For measuring dead cell fluorescence |
| Palbociclib | ApexBio | A8316 | |
| PBS | Corning | 21-040-CM | Any PBS works |
| Spark Multimode Microplate Reader | Tecan | https://www.tecan.com/spark-overview | SparkControl software version 2.2 |
| Sterile reservoir, 25 mL | Fisher Scientific | 13-681-508 | For seeding and drugging cells |
| Sytox Green nucleic acid stain | Thermo Fisher Scientific | S7020 | DNA stain for measuring dead cell fluorescence |
| Triton-X 100 | Thermo Fisher Scientific | J66624-AP | For permeabilizing cells |
| U2OS | ATCC | HTB-96 | An example cell line used in this protocol |
| Z-VAD-FMK | ApexBio | A1902 |
参考文献
- Hafner, M., Niepel, M., Sorger, P. K. Alternative drug sensitivity metrics improve preclinical cancer pharmacogenomics. Nat Biotechnol. 35 (6), 500-502 (2017).
- Dixon, S. J., Lee, M. J. Quick tips for interpreting cell death experiments. Nat Cell Biol. 25 (12), 1720-1723 (2023).
- Schwartz, H. R., et al. Drug GRADE: An Integrated Analysis of Population Growth and Cell Death Reveals Drug-Specific and Cancer Subtype-Specific Response Profiles. Cell Rep. 31 (12), 107800(2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nat Methods. 13 (6), 1-11 (2016).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death Differ. 25 (3), 486-541 (2018).
- Tsvetkov, P., et al. Copper induces cell death by targeting lipoylated TCA cycle proteins. Science. 375 (6586), 1254-1261 (2022).
- Holze, C., et al. Oxeiptosis, a ROS-induced caspase-independent apoptosis-like cell-death pathway. Nat Immunol. 19 (2), 130-140 (2018).
- Maltese, W. A., Overmeyer, J. H. Methuosis Nonapoptotic Cell Death Associated with Vacuolization of Macropinosome and Endosome Compartments. Am J Pathol. 184 (6), 1630-1642 (2014).
- Inde, Z., Forcina, G. C., Denton, K., Dixon, S. J. Kinetic Heterogeneity of Cancer Cell Fractional Killing. Cell Rep. 32 (1), 107845(2020).
- Forcina, G. C., Conlon, M., Wells, A., Cao, J. Y., Dixon, S. J. Systematic Quantification of Population Cell Death Kinetics in Mammalian Cells. Cell Syst. 4 (6), 1-18 (2017).
- Gelles, J. D., et al. Single-Cell and Population-Level Analyses Using Real-Time Kinetic Labeling Couples Proliferation and Cell Death Mechanisms. Dev Cell. 51 (2), 277-291.e4 (2019).
- Richards, R., et al. Drug antagonism and single-agent dominance result from differences in death kinetics. Nat Chem Biol. 16 (7), 791-800 (2020).
- Richards, R., Honeywell, M. E., Lee, M. J. FLICK: an optimized plate reader-based assay to infer cell death kinetics. STAR Protoc. 2 (1), 100327(2021).
- Honeywell, M. E., et al. Functional genomic screens with death rate analyses reveal mechanisms of drug action. Nat Chem Biol. 20 (11), 1443-1452 (2024).
- Jannoo, R., Xia, Z., Row, P. E., Kanamarlapudi, V. Targeting of the Interleukin-13 Receptor (IL-13R)α2 Expressing Prostate Cancer by a Novel Hybrid Lytic Peptide. Biomolecules. 13 (2), 356(2023).
- Gelles, J. D., Chipuk, J. E. Why not add some SPARKL to your life (and death)! Mol Cell Oncol. 7 (1), 1685841(2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。