Method Article
Measuring Axonal Cargo Transport in Mouse Primary Cortical Cultured Neurons
요약
본 프로토콜은 축삭 수송에 대한 전체 분석 절차를 설명합니다. 특히, 일시정지를 포함하지 않는 수송 속도의 계산과 오픈 액세스 소프트웨어 "KYMOMAKER"를 사용한 시각화 방법을 소개합니다.
초록
뉴런 세포(neuronal cell)는 고정관념적으로 여러 개의 수상돌기와 축삭돌기를 품고 있는 고도로 분극화된 세포입니다. 축삭돌기의 길이는 운동 단백질에 의한 효율적인 양방향 수송을 필요로 합니다. 다양한 보고서에서 축삭 수송의 결함이 신경 퇴행성 질환과 관련이 있다고 제안했습니다. 또한 여러 운동 단백질의 조정 메커니즘은 매력적인 주제였습니다. 축삭돌기에는 단방향 미세소관이 있기 때문에 어떤 운동 단백질이 움직임에 관여하는지 더 쉽게 확인할 수 있습니다. 따라서 축삭 화물 운송의 기저에 있는 메커니즘을 이해하는 것은 신경 퇴행성 질환의 분자 메커니즘과 운동 단백질의 조절을 밝히는 데 중요합니다. 여기에서는 마우스 1차 피질 뉴런의 배양, 화물 단백질을 인코딩하는 플라스미드의 transfection, 일시 중지의 영향이 없는 방향 및 속도 분석을 포함한 축삭 수송 분석의 전체 프로세스를 소개합니다. 또한 오픈 액세스 소프트웨어 "KYMOMAKER"가 도입되어 카이모그래프를 생성하여 방향에 따라 전송 추적을 강조 표시하고 축삭 전송을 더 쉽게 시각화할 수 있습니다.
서문
Kinesin 패밀리 구성원과 세포질 dynein은 세포의 미세소관을 따라 이동하여 화물을 운반하는 운동 단백질입니다1. 대부분의 kinesin은 플러스 끝을 향해 이동하는 반면 dynein은 미세소관의 마이너스 끝을 향해 이동합니다. 뉴런 축삭돌기에서 화물 수송의 기능과 메커니즘은 광범위하게 조사되었습니다. 축삭돌기는 길이가 길기 때문에 뉴런의 건강을 유지하기 위해 안정적인 장거리 이동이 필요합니다. 아밀β로이드 단백질 전구체(APP)를 포함하는 미토콘드리아, 자가포식소체 및 소포의 수송 결함은 신경퇴행성 질환의 원인으로 보고되었습니다 2,3. 수많은 체외 연구를 통해 운동 단백질에 의한 조정 수송의 기저에 있는 메커니즘이 밝혀졌으며, 정제된 운동 단백질과 미세소관을 사용하는 다양한 연구에서 운동 분자가 미세소관을 따라 이동하는 방식을 밝혀냈습니다 4,5. 전형적으로, 다수의 모터가 단일 화물(6)에 관련되어 있다. 그러나 반대 모터가 화물 운송의 방향을 결정하는 방법에 대한 몇 가지 모델이 있습니다. 하나는 "연관/해리 모델"입니다. 여기서 단방향 모터만 단방향 운송 중에 화물과 연결됩니다. 두 번째, "조정 모델"에서는 두 모터가 동일한화물에 부착되고 모터의 한쪽 만 활성화됩니다. 세 번째 "줄다리기 모델"에서 kinesins와 dyneins 사이의 힘의 균형은 수송 방향 7,8,9를 결정합니다. 또한, 여러 보고서에서 활성화된 운동 단백질의 균형과 수가 체외 화물 운송 속도에 영향을 미친다고 제안했습니다 8,10.
대답되지 않은 질문은 kinesin 또는 dynein의 이러한 활동이 살아있는 세포에서 어떻게 조절되는지입니다. 이전 보고서는 축삭돌기에서 미세소관의 아세틸화를 보여주었으며, 신경영양 인자는 축삭 수송을 향상시킵니다11,12. 또한, 다양한 유형의 화물 및 화물 어댑터는 해당 방식으로 모터 활성제로서 기능한다(13,14). 많은 것들이 수송 소포의 막과 관련되어 있으며, 그 기능은 번역 후 변형(post-translational modifications)15과 같은 신호에 의해 조절됩니다. 따라서 살아있는 세포의 수송 방향과 속도를 관찰하면 체외 실험에서 화물 운송의 분자 조절에 대한 귀중한 통찰력을 얻을 수 있습니다. 축삭 수송의 관찰을 통해 kinesin-based 수송과 dynein 기반 수송을 구별할 수 있습니다. 축삭돌기는 플러스 말단 단방향 미세소관16을 품고 있기 때문에, 화물은 키네신(kinesin)에 의해 전방(즉, 소마에서 축삭 말단으로)으로 운송되고 다인(dyneins)에 의해 역행적으로(즉, 축삭 말단에서 소마로) 운송됩니다.
본 연구에서는 1차 배양 뉴런에서 축삭 수송을 관찰하고 분석하는 방법을 설명합니다. 일례로써, 막단백질-APP, 칼신테닌-1/알카데인 α(Alcα), 칼신테닌-3/알카데인 β(Alcβ)-함유 소포의 축삭 수송을 관찰하는 절차가 기재되어 있다. APP-함유 소포의 선행성 수송은 Alcα 함유 소포의 수송보다 상당히 빠른 것으로 알려져 있지만, 둘 다 kinesin-1에 의해 수송됩니다 17,18,19. 이전 보고서에서는 속도를 측정하기 위해 여러 가지 방법이 사용되었습니다. 가장 가변적인 단계는 운송 중 일시 중지를 처리하는 것입니다. 살아있는 세포에서 수송은 때때로 미세소관을 따라 있는 장애물에 의해 방해를 받습니다. 그러나, 모터는 일시정지(20)가 있거나 없는 영역을 우회할 수 있다. 더 긴 관측 시간에 대한 속도 계산은 일시 중지의 영향을 받을 수 있으며, 이로 인해 속도 추정이 느려질 수 있습니다. 여기서, 모터의 물리적 일시 중지의 영향을 배제하기 위해 세그먼트화된(200ms) 기간에 걸친 이동을 사용하는 방법이 설명됩니다. 마지막으로 "KYMOMAKER"21라는 오픈 액세스(Windows 전용) 소프트웨어 프로그램이 도입되었습니다. 카이모그래프는 소포 수송을 시각화하는 데 널리 사용되며 동영상 없이 각 화물의 운송 방향을 시각화하는 데 유용합니다. 이 소프트웨어는 이미지를 회전하는 동안 1차원 유역 알고리즘을 여러 번 적용하여 타임랩스 동영상에서 카이모그래프를 생성합니다. 이를 통해 결과 카이모그래프는 미세한 구조를 효율적이고 쉽게 표시할 수 있습니다. 또한 KYMOMAKER는 방향에 따라 트레일을 자동으로 감지하고 강조 표시하며 이해하기 쉬운 다이어그램을 만들 수 있습니다.
프로토콜
이 실험은 ARRIVE(Animal Research: Reporting of In Vivo Experiments) 지침에 따라 홋카이도 대학의 동물 연구 위원회의 승인을 받았습니다. 본 연구에는 암컷 C57BL/6J 마우스(임신, 15.5일)를 사용했습니다.
1. 마우스 1차 피질 배양 뉴런의 준비
- 0.1 M Tris (트리스 (하이드 록시 메틸) 아미노 메탄) -HCl (pH 8.5)에 0.1 mg / mL의 폴리 -L-Lysin을 8 웰 (No.1.0) 유리 바닥 챔버에 추가합니다 ( 재료 표 참조). 37°C 인큐베이터에서 최소 1시간 동안 챔버를 배양합니다.
- 챔버를 멸균된 물로 최소 3회 헹구고 조직 배양 후드에서 자연 건조합니다.
- 임신한 생쥐를 15.5일에 자궁경부 탈구로 희생시키거나 제도적으로 승인된 규정에 따라 희생시킵니다.
- 배아가 들어 있는 자궁 뿔을 꼬집고 가위를 사용하여 자궁 뿔 전체를 분리한 다음 얼음 위의 차가운 인산염 완충 식염수(PBS)에 담급니다.
- 차가운 PBS에서는 날카로운 핀셋을 사용하여 배아를 분리하고 배아에서 모든 양막을 제거합니다.
- 신선한 차가운 PBS로 배아를 다시 씻으십시오.
참고: 각 분리 단계에서 가능한 한 많은 혈액을 씻어내고 혈관이나 배아가 손상되지 않도록 하십시오. - 멸균된 거즈에 배아를 올려 차가운 페트리 접시에 담아 광학 현미경으로 검사합니다.
- 뇌를 분리하십시오. 구부러진 날카로운 핀셋 끝을 사용하여 배아의 머리, 피부, 두개골을 자릅니다. 피부와 두개골을 제거하여 뇌의 윗면과 옆구리를 노출시킵니다.
- 핀셋의 구부러진 부분을 사용하여 뇌 전체를 퍼내고 얼음처럼 차가운 1x Hanks의 균형 잡힌 소금 용액(HBSS; 10 mM HEPES [pH 7.6], 5.3 mM KCl, 0.44 mM KH2PO4, 140 mM NaCl, 4.2 mM NaHCO3, 0.34 mM Na2HPO4, 39 mM D-포도당, 및 0.5 μg/mL 겐타마이신; 재료 표 참조).
- 필요한 수의 뇌를 얻을 때까지 1.7-1.9단계를 반복합니다.
참고: 일반적으로 뇌당 5 x 106 에서 2 x 107 세포를 얻을 수 있습니다. - 차가운 1x HBSS에서 핀셋을 사용하여 뇌에서 피질22 를 분리하고 피질에서 수막을 떼어냅니다.
- 차가운 1x HBSS를 포함하는 새로운 페트리 접시로 피질을 옮깁니다. 수술용 칼날을 사용하여 피질을 반으로 자르거나 3등분한다.
- 팁 컷 1,000 μL 팁을 사용하여 피질을 15 mL 원심분리 튜브로 옮기고 여분의 1x HBSS를 제거합니다.
- 배아 당 파파인 100단위와 5mL의 파파인 희석 용액(5mg/mL D-포도당, 0.2mg/mL 소 혈청 알부민 및 0.2mg/mL L-시스테인, 0.2mg/mL L-시스테인)으로 희석한 200μg의 데옥시리보뉴클레아제 I(DNase I)을 추가하고 배아당 37°C 수조에서 15분 동안 배양합니다. 배양 중 5분마다 튜브를 뒤집습니다.
참고: 파파인 희석 용액에서 파파인을 사전 배양하는 것이 좋습니다. 37°C 수조에서 5분 동안 배양한 다음 DNase I을 추가합니다. - 용액을 버리십시오. 20% 열 비활성화 호스 세럼 2mL( 재료 표 참조)를 1x HBSS에 추가합니다. 1분 동안 배양합니다.
- 용액을 버리십시오. 4mL의 1x HBSS로 조직을 두 번 헹굽니다.
- 용액을 버리십시오. 도금 배지(2% B-27 보충제, 4mM 글루타맥스, 5% 열 불활성화 말 혈청 및 1x 페니실린-스트렙토마이신을 함유하는 neurobasal 배지)의 배아당 0.5mL를 추가합니다.
알림: 1.14-1.17 단계에서는 절단된 피질 조각이 원심분리 없이 튜브 바닥으로 가라앉기 때문에 원심분리가 필요하지 않습니다. 흡인기를 사용하지 말고 조각을 빨아먹지 마십시오. - 1,000 μL 팁 또는 10 mL 피펫으로 5-10회 절단하여 매우 부드럽게 재현탁합니다.
- 1,000μL 팁으로 매우 부드럽게 재현탁합니다.
- 실온(20-25° C)에서 250×g에서 3분 동안 원심분리기
- 상층액을 제거하고 1,000 μL 팁을 10회 사용하여 도금 배지 배아당 0.5 mL로 매우 부드럽게 재현탁합니다.
- 50mL 원심분리 튜브에 40μm 세포 여과기를 놓고 중력 흐름으로 세포 현탁액을 여과합니다. 여과된 세포 현탁액을 튜브에 수집합니다.
- 적절한 부피의 도금 매체와 함께 2 ×10 4 에서 5 × 104 cells/cm2의 챔버로 세포와 플레이트를 계산합니다. 밀도의 예가 보충 그림 1에 나와 있습니다.
- day-in-vitro (Div.) 2에서 배지의 절반을 배양 배지(2% B-27 보충제, 4mM 글루타맥스 및 1x 페니실린-스트렙토마이신을 함유하는 신경기저 배지)로 교체하고 5μM의 5-플루오로-2-데옥시우리딘을 추가합니다( 재료 표 참조).
- Div. 4 이후, 2일 또는 3일마다 배지의 절반을 배양 배지로 교체합니다.
2. 인산칼슘 방법을 사용하여 플라스미드를 1차 배양된 뉴런으로 형질주입
참고: 언급된 부피는 8웰 유리 바닥 챔버에서0.8cm 2 웰에 대한 것입니다( 재료 표 참조).
- 형광 표지 단백질, 6.2 μL의 2 M CaCl2 및 멸균된 물을 인코딩하는 200 ng - 2 μg의 플라스미드(pCAGGS의 APP-EGFP, pcDNA3.118의 Alcα-EGFP 또는 pcDNA3.1의 Alcβ-EGFP, 보충 표 1 참조)를 혼합하여 50 μL의 DNA/CaCl2 용액을 준비합니다.
- 50μL의 2x HEPES 완충 식염수(HBS)를 새로운 원심분리 튜브에 분취합니다.
- 6.2 μL의 DNA/CaCl2 용액을 2x HBS 용액에 추가합니다. 10회 피펫팅하여 부드럽게 섞습니다.
- 모든 DNA/CaCl2 용액이 전달될 때까지 2.3단계를 반복합니다.
- 실온에서 15분 동안 배양합니다.
- 유리 바닥 챔버에서 배양 배지를 수집하고 항생제가 없는 새로운 배양 배지로 교체합니다.
참고: 수집된 배지를 2.10단계까지 세포 배양 인큐베이터에 보관하십시오. - 웰당 DNA/CaCl2/HBS 혼합물 50μL를 뉴런에 적가합니다. 1시간 동안 문화.
- 배양 중 DMEM(Dulbecco's modified Eagle's medium)/Ham's F-12 배지가 포함된 Petri 접시를 37°C의 10% CO2 배양기에 넣어 산성화된 배지를 준비합니다.
- transfection 후 1시간 동안 배양한 후 DMEM/Ham's F-12(acidified medium)로 일차 뉴런을 3회 헹굽니다.
- 배지를 반쯤 수집된 배지와 반쯤 신선한 배지의 혼합물로 교체하십시오. 37 ° C 및 5 % CO2에서 현미경으로 관찰 할 때까지 배양.
3. 축삭 수송의 관찰
- TIRF(Total Internal Reflectance) 현미경( 재료 표 참조)이 장착된 챔버를 37°C에서 사전 배양합니다.
- CO2 컨트롤러가 장착된 경우 배양 배지는 5% CO2 미만에서 관찰 매체로 사용할 수 있습니다. 장착되지 않은 경우 배지를 4mM 글루타맥스 및 2% B-27 보충제와 함께 제공되는 Leibovitz L-15( 재료 표 참조)와 같은 pH 안정성 배지로 교체하십시오.
- 형질주입된 세포를 찾은 다음 축삭돌기를 식별합니다. 레이저가 완전히 반사될 때까지 레이저의 입사각을 높입니다. 그런 다음 축삭의 모든 소포가 시각화될 때까지(pseudo-TIRF) 각도를 줄입니다. 150프레임(30초) 동안 200ms 노출 시간으로 이미지를 획득합니다.
참고: 이 실험에서는 pseud-TIRF 조명을 사용하여 모든 축삭 소포를 캡처했습니다. 고속 캡처 시스템이 장착된 다른 현미경을 사용할 수 있습니다.
알림: 매번 축삭돌기의 방향을 기록하십시오 file 이름. 초기 축삭 분절 또는 말단 축삭 영역의 영역은 관찰에서 피했습니다.
4. 이미지 처리
- MetaMorph 소프트웨어를 사용하여 획득한 이미지를 엽니다( 재료 표 참조).
- 도구 모음을 열고 > 거리를 측정> Calibrate distance를 선택합니다. "열려 있는 모든 이미지에 적용"을 활성화하고 픽셀의 실제 거리를 입력합니다.
- 도구 모음> 열기를 > 회전을 표시합니다. "모든 비행기"에 대한 확인란을 활성화합니다. 축삭이 수평이 되고 축삭 말단 방향이 오른쪽이 되도록 회전합니다.
- 영역 > 도구 모음을 엽니다> 영역을 만듭니다. 클리핑할 영역의 크기를 설정하고 Create를 클릭합니다. 분석을 위해 이미지에 나타나는 프레임을 축삭 영역에 배치합니다. 생성된 이미지 시퀀스를 스택 파일로 저장합니다.
참고: 초점이 맞지 않는 축삭돌기나 파편이 없는 영역을 선택합니다. - 도구 모음을 열어 > 그래픽> 표시합니다. Calibration bar(보정 막대)를 선택하여 눈금 막대를 스탬프합니다. 데이터/시간을 선택하여 이미지에 타임스탬프를 추가합니다. "모든 비행기"를 활성화합니다. 개체를 적절한 공간으로 이동하고 스탬프를 클릭합니다.
- 도구 모음> Stack> Make movie를 엽니다. 적절한 프레임 속도를 입력하고 AVI를 선택합니다. 새 파일로 저장합니다.
5. KYMOMAKER를 사용하여 카이모그래프 그리기 및 추적 감지
- KYMOMAKER( 재료 표 참조)를 다운로드하고 KYMOMAKER 폴더에서 "Kymoanalysis.exe"를 엽니다.
- "kymoAnalysis.exe"를 열어 기본 창을 표시합니다(그림 1A). 파일 > 로드 를 선택하고 생성된 AVI 동영상을 엽니다. "Preview" 창이 나타납니다(그림 1B).
- Generate Kymograph 탭 > trimming 섹션에서 트리밍할 픽셀 번호를 입력합니다. 트리밍은 "Preview" 창에 즉시 반영되어 쉽게 조정할 수 있습니다(그림 1C). 전체 축척 막대와 타임스탬프가 제거되었는지 확인합니다.
- Generate 버튼을 클릭하여 카이모그래프를 생성합니다(그림 1C). KYMOMAKER는 "미리보기" 창에서 각 y축 내에서 가장 밝은 픽셀을 감지합니다. 파일을 통해 카이모그래프를 저장> 일반 카이모그래프> 저장합니다.
- 카이모그래프에서 저강도 트레이스를 감지하려면 Rotational Kymograph를 클릭합니다. "Rotational Kymograph" 섹션에서 숫자를 조정하고 회전 카이모그래프 를 클릭하여 결과를 확인합니다(그림 1D).
참고: "회전(도)"은 회전 유역 알고리즘21에 대한 차수 간격입니다. 예를 들어 10°는 유역 알고리즘이 10°마다 36회 수행된 다음 결과가 "회전 유역" 창에 결합됨을 의미합니다. - 카이모그래프에서 전행성 및 역행 트레이스를 감지하려면 Detection(탐지 ) 탭> Target(대상 ) 섹션에서 Rotational Kymograph가 생성되지 않은 경우 original( 원본 )을 선택합니다. 회전(Rotation)을 선택하면 생성된 회전 유역(Rotational Watershed)이 추적을 탐지하는 데 사용되는지 확인합니다. "Detecting Method"에서 유역 을 선택합니다. Detect 버튼을 클릭하면 트레이스를 자동으로 감지할 수 있습니다.
참고: 감지된 추적은 "작업" 창에 그려집니다. "마스킹"을 켜면 원래 카이모그래프에 트레이스가 그려집니다. - Detection > Filtering > Velocity 섹션에 숫자를 입력하여 이전 및 역행 추적을 감지합니다(그림 1E, F). 0.4 - 7.0 μm/s를 입력하여 선행성 트레이스를 감지합니다. -0.4 - -7.0 μm/s를 입력하여 역행 흔적을 감지합니다.
참고: 가장 낮은 속도에 대한 설정은 소포의 부동(imtilit) 상태의 정의에 따라 달라집니다. 6.7단계를 참조하십시오. - 파일을 저장합니다.
6. 속도 결정
- MetaMorph 소프트웨어를 엽니다.
참고: ImageJ의 "수동 추적" 플러그인도 같은 방식으로 속도를 계산할 수 있습니다. - 4.4단계에서 만든 파일을 엽니다. 보정이 이미지에 맞게 조정되었는지 확인합니다.
- 도구 모음> 앱을 열고 포인트를 추적합니다>. 트랙 추가를 클릭합니다. 선행성 또는 역행성으로 수송된 움직이는 소포를 찾습니다. 그런 다음 전송이 종료될 때까지 클릭하여 소포를 추적합니다. 완료를 클릭합니다.
- 모든 소포가 추적될 때까지 6.3단계를 반복합니다.
- 로그 열기를 클릭하여 CSV 파일을 엽니다. 그런 다음 로그 파일을 클릭하여 CSV 파일에 데이터를 표시합니다.
- CSV 파일을 저장합니다.
- 일시 중지의 영향을 받지 않는 분할된 속도를 계산하려면 "속도" 열에서 계산된 속도 0.37μm/s 이하를 제거합니다.
참고: 소포는 변칙적 확산에 의해 매우 짧게 이동할 수 있으며, 운동 단백질에 의해 처리되지 않는다23. 모터 구동 속도 계산에서 이러한 모션을 포함하지 않기 위해 이 프로토콜에서 가장 낮은 속도(1픽셀/200ms)가 계산에서 제거됩니다. - 5프레임마다(총 1초) 분할된 평균 속도를 계산합니다. 남은 프레임이 5개 미만인 경우 속도의 마지막 부분을 버립니다.
- 하나의 조건에서 모든 평균 속도를 결합합니다. 속도 분포를 시각화하는 데 도움이 되는 히스토그램을 생성합니다.
결과
E15.5 마우스 피질로부터 1차 배양된 뉴런을 설명된 바와 같이 유리 바닥 접시에서 배양했습니다. 예를 들어, APP-EGFP, Alcα-EGFP 또는 Alcβ-EGFP는 1차 피질 뉴런에서 발현되었습니다. APP와 Alcα는 kinesin-1 2,17에 의해 축삭돌기로 운반되는 것으로 알려져 있습니다. APP는 어댑터 단백질 JIP1(JNK-상호 작용 단백질 1)을 통해 kinesin-1과 결합하는 반면, Alcα는 이중 W-산성 모티프를 통해 직접 결합합니다. Alcβ는 Alcadein 계열의 일원이며 W-산성 모티프가 하나뿐이다24. 각 단백질을 Div. 5 뉴런으로 transfection하고, transfection 후 14-16시간 후에 TIRF 현미경을 사용하여 200ms마다 이미지를 획득했습니다. APP-EGFP, Alcα-EGFP 또는 Alcβ-EGFP로 형질주입된 뉴런에서 축삭돌기의 여러 움직이는 소포가 관찰되었습니다(각각 보충 영화 1-3).
KYMOMAKER를 사용하여 회전 유역 알고리즘을 사용하거나 사용하지 않고 Alcβ-EGFP에 대한 카이모그래프를 생성했습니다(각각 그림 1C, D). 또한 KYMOMAKER는 원래 및 유역 카이모그래프에서 전행성 및 역행 방향의 흔적을 추출할 수 있었습니다(그림 1E, F). 두 카이모그래프에서 주요 흔적이 감지되었습니다. 기존 방법과 비교하여 Watershed 알고리즘을 적용하면 저강도 트레이스를 더 잘 감지할 수 있습니다(그림 1F).
각 뉴런에 있는 소포의 속도를 추적하여 설명한 대로 계산했습니다. APP-EGFP의 속도는 3.05 ± 1.03 μm/s로 이전 결과18,19와 일치합니다. Alcα-EGFP는 1.89 ± 0.77 μm/s의 속도로 수송되었는데, 이는 APP보다 상당히 느렸으며, 이는 kinesin-1의 보고된 속도와 일치하였다. Alcβ-EGFP의 수송 속도는 2.68 ± 1.07 μm/s였으며, 이는 Alcα와 APP의 속도 사이에 있었습니다. 분포의 차이를 보여주기 위해 0.2 μm/s bin 너비를 사용하여 히스토그램을 생성했습니다. 또한 KYMOMAKER로 생성된 카이모그래프를 사용하여 운송 방향의 상대적 비율을 계산했습니다(그림 2A, B). 세 가지 단백질 모두 주로 전행성 방향으로 운반되었습니다(그림 2C).

그림 1: KYMOMAKER를 사용한 검출 방법의 워크플로우. (A) KYMOMAKER의 메인 창 설정. (B) 입수한 동영상을 KYMOMAKER에서 개봉하고, 축삭돌기가 포함되지 않은 부분을 "트리밍"으로 제거했습니다. (C) 생성을 클릭하면 원본 카이모그래프가 생성되었습니다. (D) 회전 카이모그래프 를 클릭하면 새로운 "회전 카이모그래프" 창이 열리며 (A)의 상단 대화 상자에서 감지 매개변수를 변경할 수 있습니다. (E) 각 전행성 또는 역행성 흔적은 (C)의 카이모그래프에서 검출되었습니다. 감지 매개변수는 (A)의 하단 대화 상자에서 변경할 수 있습니다. (F) (D)에서 감지된 흔적. 스케일 바 = 5μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
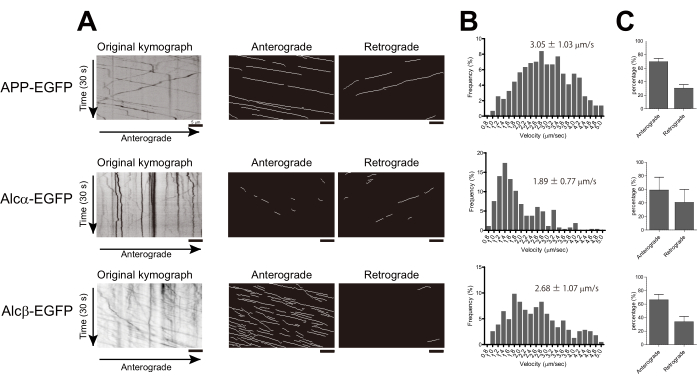
그림 2: kinesin-1 수송 소포의 속도 및 방향 계산. APP-EGFP, Alcα-EGFP 또는 Alcβ-EGFP를 마우스 1차 피질 배양 뉴런의 Div. 4-6에 형질주입하고, 형질주입 14-18시간 후에 축삭돌기의 이미지를 획득하였다. (A) 축삭 수송의 키모그래프는 KYMOMAKER를 사용하여 생성되었습니다. 왼쪽: 원본 카이모그래프; 중간: 선행성 흔적; 오른쪽: 역행 흔적. (B) 두 개의 독립적인 실험에서 전방 수송 속도의 히스토그램이 표시됩니다(두 개의 독립적인 실험에서 APP-EGFP의 경우 n = 583, Alcβ-EGFP의 경우 n = 388, Alcα-EGFP의 경우 n = 264). 평균 ± SD가 히스토그램에 표시됩니다. (C) 각 방향으로 운반된 트레이스의 백분율이 표시됩니다(각 막대는 각각 6-10개의 세포를 포함하는 독립적인 실험에서 평균 SD. n = 2± 나타냄). 스케일 바 = 5μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 그림 1: 유리 바닥 접시에 있는 1차 배양 뉴런의 배양 밀도. Div. 1 마우스 1차 피질 배양 뉴런의 미분 간섭 대비(DIC) 이미지, 1.23단계에서와 같이 도금됨. 스케일 바 = 50 μm. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 1: 이 프로토콜에 사용된 플라스미드의 구조. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 동영상 1: APP-EGFP의 축삭 수송. 영화는 30초 동안 200ms마다 촬영되었습니다. 눈금 막대 = 5μm. 이 동영상을 다운로드하려면 여기를 클릭하십시오.
보충 동영상 2: Alcα-EGFP의 축삭 수송. 영화는 30초 동안 200ms마다 촬영되었습니다. 눈금 막대 = 5μm. 이 동영상을 다운로드하려면 여기를 클릭하십시오.
보충 영화 3: Alcβ -EGFP의 축삭 수송. 영화는 30초 동안 200ms마다 촬영되었습니다. 눈금 막대 = 5μm. 이 동영상을 다운로드하려면 여기를 클릭하십시오.
토론
축삭 수송에 대한 분석 방법이 설명되며, 여기에는 분할 속도 계산 및 카이모그래프 생성이 포함됩니다. transfection 단계에서 중요한 단계는 배양된 뉴런의 건강을 유지하는 것입니다. Jiang과 Chen[29 ]에 의해 기술된 형질주입 방법은 약간의 수정을 가하였다. DNA/CaCl2 용액과 2x HBS를 부드럽게 혼합하면 침전물의 크기를 줄여 transfection의 효율성을 높일 수 있었습니다. 침전물이 세포를 손상시키는 것을 방지하기 위해 세포를 산성화된 매체로 헹궈냈습니다. 배양 없이 빠르게 헹구는 것만으로도(2회 또는 3회) 세포에서 침전물을 제거하기에 충분했습니다. 여기에서는 배양된 1차 뉴런을 사용했습니다. 쥐 좌골 신경의 수송은 수술을 통해 생체 내 컨포칼 현미경으로 관찰할 수 있으며(26), 최근 다광자 현미경의 개발로 생쥐 뇌의 축삭 수송을 관찰할 수 있게 되었다는 점을 언급할 가치가 있습니다27.
Kymographs는 축삭 수송의 속도와 방향을 시각화하는 데 널리 사용됩니다. 피지 플러그인 KymographBuilder를 포함하여 카이모그래프를 만드는 다양한 도구가 있습니다. KymoAnalyzer 또는 Kymolyzer와 같은 일부는 반수동28,29에 할당된 트랙의 매개변수를 계산할 수 있습니다. KYMOMAKER 소프트웨어는 트레이스 할당을 수정할 수 없지만 방향21에 따라 트레이스를 쉽게 시각화할 수 있는 장점이 있습니다. 트리밍 프로세스를 관리하고, 감지 매개변수를 조정하여 카이모그래프를 생성하고, 방향별로 트레이스를 자동으로 추출합니다. 회전 유역 방법을 사용하면 미세한 구조를 탐지할 수 있습니다. 대부분의 트레이스를 탐지할 수 있었지만, 두 트레이스가 독립적인지 아니면 일시적으로 연결이 끊어졌는지, 특히 트레이스가 교차하는 위치를 파악하기는 어려웠습니다. 또한 KYMOMAKER는 각 트레이스의 매개 변수 계산에 사용되지 않습니다. 선행성 및 역행 추적 횟수의 경우 감지된 추적을 빨간색 선으로 마스킹하는 "Kymograph" 창을 사용하는 것이 좋습니다.
이 계산 방법은 라이브 셀에 대한 예측할 수 없는 일시 중지의 영향을 제외하는 장점이 있습니다. 일시 중지는 다른 모터 및 화물과 같은 물리적 장애물과 tau20,30과 같은 미세소관 관련 단백질로 인해 발생할 수 있습니다. 수송 속도의 계산은 몇 가지 절차를 사용하여 수행되었습니다. 일반적으로 일시 중지를 포함하여 이동 거리를 결정하는 데 더 긴(몇 초 이상) 기간이 사용됩니다. 그러나 세그먼트화된(200ms) 기간을 사용하면 이 프로토콜은 계산에서 일시 중지를 보다 효율적으로 식별하고 제거할 수 있습니다. 어떤 경우에는 Alcβ의 경우와 같이 화물 속도가 2개 또는 3개의 피크 분포를 보였는데, 이는 관련된 운동 단백질의 다양한 유형 또는 상태를 반영할 수 있습니다. 따라서 속도의 분포를 시각화하기 위해 히스토그램을 생성하는 것이 좋으며, 이 보다 정확한 계산 방법은 최고 속도를 보다 명확하게 추정하는 데 기여할 수 있습니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
이 연구는 KAKENHI(22K15270, Grant-in-Aid for Young Scientists)와 아키야마 생명과학재단(Akiyama Life Science Foundation for YS)의 지원을 받았습니다. 홋카이도 대학 생명과학부 분자세포역학연구실의 긴조 마사타카 박사와 기타무라 아키라 박사가 연구에 큰 도움을 준 데 중요한 의견과 전문 지식을 제공해 주신 데 대해 감사의 말씀을 드립니다. TIRF 현미경을 사용한 관찰은 홋카이도 대학 첨단 생명 과학 학부 분자 세포 역학 실험실에 설치된 기기를 사용하여 수행되었습니다. 이 기기는 홋카이도 대학 크리에이티브 연구 기관(Creative Research Institution)의 글로벌 시설 센터(Global Facility Center)에서 관리하는 개방형 시설(Open Facility) 시스템(AP-100138)에 등록되어 있습니다. 일본 후쿠오카시 규슈 대학교 정보 과학 및 전기 공학부 첨단 정보 기술학과 휴먼 인터페이스 연구실의 Seiichi Uchida 박사에게 Kymomaker 응용 프로그램을 사용한 회의에 감사드립니다. 홋카이도 대학 약학연구과 치매 첨단 예방 및 연구소는 주식회사 Japan Medical Leaf의 지원을 받고 있습니다.
자료
| Name | Company | Catalog Number | Comments |
| 5-Fluoro-2-deoxyuridine | Sigma-Aldrich | F0503 | |
| Apo TIRF 100x/1.49 OIL | Nikon | ||
| B-27 Supplement (50x), serum free | Thermo fischer scientific | 17504044 | |
| Bovine serum albumin | Wako | 013-25773 | |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | |
| Cell strainer 40 µm Nylon | Falcon | 352340 | |
| CoolSNAP HQ | Photometrics | ||
| Deoxyribonuclease I | Sigma-Aldrich | DN-25 | |
| D-Glucose | Wako | 041-00595 | |
| DMEM/Ham’s F-12 | Wako | 042-30555 | |
| Dumont No. 7 forceps | Dumont | No.7 | |
| Feather surgical blade | Feather | No.11 | |
| Feather surgical blade handle | Feather | No. 3 | |
| Gentamicin | Wako | 079-02973 | |
| Gentamicin Sulfate | Wako | 075-04913 | |
| GlutaMAX Supplement | Thermo fischer scientific | 35050061 | |
| HEPES | DOJINDO | 342-01375 | |
| Horse Serum, heat inactivated | Thermo fischer scientific | 26050088 | |
| KCl | Wako | 163-03545 | |
| KH2PO4 | Wako | 169-04245 | |
| KYMOMAKER | http://www.pharm.hokudai.ac.jp/shinkei/Kymomaker.html | ||
| L-15 Medium (Leibovitz) | Sigma-Aldrich | L5520 | |
| MetaMorph version 6.2r1 | Metamorph | ||
| Na2HPO4 | Wako | 197-02865 | |
| NaCl | Wako | 197-01667 | |
| NaHCO3 | Wako | 191-01305 | |
| Neurobasal Medium | Thermo fischer scientific | 21103049 | |
| Nikon ECLIPSE TE 2000-E | Nikon | ||
| Nunc Lab-Tek 8 well Chambered Coverglass | Thermo fischer scientific | 155411 | |
| Papain | Worthington | LS003126 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140122 | |
| Poly-L-lysine hydrobromide | Sigma-Aldrich | P2636-500MG | |
| Trizma base | Merck | T1194-10PAK | solved with water to make 0.1 M Tris-HCl (pH.8.5) |
참고문헌
- Hirokawa, N., Noda, Y., Tanaka, Y., Niwa, S. Kinesin superfamily motor proteins and intracellular transport. Nature Reviews Molecular Cell Biology. 10 (10), 682-696 (2009).
- Stokin, G. B., et al. Axonopathy and transport deficits early in the pathogenesis of Alzheimer's diseases. Science. 307 (5713), 1282-1288 (2005).
- Guo, W., Stoklund Dittlau, K., vanden Bosch, L. Axonal transport defects and neurodegeneration: Molecular mechanisms and therapeutic implications. Seminars in Cell & Developmental Biology. 99, 133-150 (2020).
- Svoboda, K., Schmidt, C. F., Schnapp, B. J., Block, S. M. Direct observation of kinesin stepping by optical trapping interferometry. Nature. 365 (6448), 721-727 (1993).
- Carter, N. J., Cross, R. A. Mechanics of the kinesin step. Nature. 435 (7040), 308-312 (2005).
- Welte, M. A. Bidirectional transport along microtubules. Current Biology. 14 (13), R525-R537 (2004).
- Hendricks, A. G., et al. Motor coordination via a tug-of-war mechanism drives bidirectional vesicle transport. Current Biology. 20 (8), 697-702 (2010).
- Rezaul, K., et al. Engineered tug-of-war between kinesin and dynein controls direction of microtubule based transport in vivo. Traffic. 17 (5), 475-486 (2016).
- Encalada, S. E., Goldstein, L. S. B. Biophysical challenges to axonal transport: motor-cargo deficiencies and neurodegeneration. Annual Review of Biophysics. 43, 141-169 (2014).
- Monzon, G. A., et al. Stable tug-of-war between kinesin-1 and cytoplasmic dynein upon different ATP and roadblock concentrations. Journal of Cell Science. 133 (22), 226248 (2020).
- Guo, W., et al. HDAC6 inhibition reverses axonal transport defects in motor neurons derived from FUS-ALS patients. Nature Communications. 8 (1), 861 (2017).
- Tosolini, A. P., et al. BDNF-dependent modulation of axonal transport is selectively impaired in ALS. Acta Neuropathologica Communications. 10 (1), 121 (2022).
- Verhey, K. J., Hammond, J. W. Traffic control: regulation of kinesin motors. Nature Reviews Molecular Cell Biology. 10 (11), 765-777 (2009).
- Fu, M. M., Holzbaur, E. L. F. Integrated regulation of motor-driven organelle transport by scaffolding proteins. Trends in Cell Biology. 24 (10), 564-574 (2014).
- Kumari, D., Ray, K. Phosphoregulation of kinesins involved in long-range intracellular transport. Frontiers in Cell and Developmental Biology. 10, 873164 (2022).
- Wai Yau, K., et al. Cellular/molecular dendrites in vitro and in vivo contain microtubules of opposite polarity and axon formation correlates with uniform plus-end-out microtubule orientation. The Journal of Neuroscience. 36 (4), 1071-1085 (2016).
- Araki, Y., et al. The novel cargo Alcadein induces vesicle association of kinesin-1 motor components and activates axonal transport. The EMBO Journal. 26 (6), 1475-1486 (2007).
- Sobu, Y., et al. Phosphorylation of multiple sites within an acidic region of Alcadein α is required for kinesin-1 association and Golgi exit of Alcadein α cargo. Molecular Biology of the Cell. 28 (26), 3844-3856 (2017).
- Tsukamoto, M., et al. The cytoplasmic region of the amyloid β-protein precursor (APP) is necessary and sufficient for the enhanced fast velocity of APP transport by kinesin-1. FEBS Letters. 592 (16), 2716-2724 (2018).
- Ferro, L. S., Can, S., Turner, M. A., Elshenawy, M. M., Yildiz, A. Kinesin and dynein use distinct mechanisms to bypass obstacles. eLife. 8, e48629 (2019).
- Chiba, K., Shimada, Y., Kinjo, M., Suzuki, T., Uchida, S. Simple and direct assembly of kymographs from movies using KYMOMAKER. Traffic. 15 (1), 1-11 (2014).
- Xu, S. -. Y., Wu, Y. -. M., Ji, Z., Gao, X. -. Y., Pan, S. -. Y. A modified technique for culturing primary fetal rat cortical neurons. Journal of Biomedicine and Biotechnology. 2012, 803930 (2012).
- Han, D., et al. Deciphering anomalous heterogeneous intracellular transport with neural networks. eLife. 9, e52224 (2020).
- Araki, Y., et al. Novel cadherin-related membrane proteins, Alcadeins, enhance the X11-like protein-mediated stabilization of amyloid beta-protein precursor metabolism. The Journal of Biological Chemistry. 278 (49), 49448-49458 (2003).
- Jiang, M., Chen, G. High Ca2+-phosphate transfection efficiency in low-density neuronal cultures. Nature Protocols. 1 (2), 695-700 (2006).
- Tosolini, A. P., Villarroel-Campos, D., Schiavo, G., Sleigh, J. N. Expanding the toolkit for in vivo imaging of axonal transport. Journal of Visualized Experiments. (178), e63471 (2021).
- Takihara, Y., et al. In vivo imaging of axonal transport of mitochondria in the diseased and aged mammalian CNS. Proceedings of the National Academy of Sciences. 112 (33), 10515-10520 (2015).
- Neumann, S., Chassefeyre, R., Campbell, G. E., Encalada, S. E. KymoAnalyzer: a software tool for the quantitative analysis of intracellular transport in neurons. Traffic. 18 (1), 71-88 (2017).
- Basu, H., Ding, L., Pekkurnaz, G., Cronin, M., Kymolyzer Schwarz, T. L. a semi-autonomous kymography tool to analyze intracellular motility. Current Protocols in Cell Biology. 87 (1), 107 (2020).
- Dixit, R., Ross, J. L., Goldman, Y. E., Holzbaur, E. L. F. Differential regulation of dynein and kinesin motor proteins by tau. Science. 319 (5866), 1086-1089 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유