Method Article
Miglioramento della consegna e dell'espressione genica mediante iniezione intraossea di plasmidi editor di base adenina incapsulati con nanoparticelle di chitosano
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, presentiamo un approccio di terapia genica che somministra chitosano rivestito di ABE direttamente al midollo osseo mediante iniezione intraossea.
Abstract
La somministrazione di plasmidi esogeni negli animali da esperimento è fondamentale nella ricerca biomedica, compresa lo studio delle funzioni geniche, il chiarimento dei meccanismi della malattia e la valutazione dell'efficacia dei farmaci. Tuttavia, l'efficienza di trasfezione del metodo attuale è relativamente bassa e l'introduzione di plasmidi per l'espressione genica a lungo termine può essere influenzata dal sistema immunitario. Per affrontare queste limitazioni, abbiamo sviluppato e studiato un nuovo metodo che utilizza il chitosano per incapsulare i plasmidi dell'editor di basi adenine (ABE) e quindi somministrare direttamente il complesso al midollo osseo dei topi mediante iniezione intraossea. In questo studio, per colpire il gene CaMK II δ , che è strettamente correlato alla differenziazione degli osteoclasti, abbiamo utilizzato il chitosano per incapsulare i plasmidi ABE CaMK II δ . Abbiamo iniettato direttamente il carico plasmidico nella cavità del midollo osseo mediante iniezione intraossea. I risultati hanno mostrato un'elevata efficienza di editing in vivo del 14,27% al locus A1 e del 10,69% al locus A2 nei topi riceventi. Questa nuova strategia non solo è particolarmente adatta per le malattie causate da una funzione anomala degli osteoclasti, ma ha anche un potenziale significativo per far progredire il campo della terapia genica.
Introduzione
La terapia genica è emersa come un approccio promettente nel campo della ricerca biomedica 1,2. Offre il potenziale per trattare una varietà di disturbi introducendo plasmidi estranei in animali da esperimento per modulare l'espressione di geni specifici e studiarne gli effetti terapeutici. Tuttavia, i metodi di somministrazione convenzionali hanno riscontrato alcuni problemi che ne limitano l'efficacia e la sicurezza3. Le preoccupazioni includono la bassa efficienza di trasfezione, l'elevato danno biologico e la bassa efficienza dell'espressione genica. Pertanto, è necessario stabilire un nuovo metodo di somministrazione per la terapia genica in grado di superare queste limitazioni.
Il chitosano è un polisaccaride naturale con una buona biodegradabilità e biocompatibilità 4,5,6, che lo rende facile da degradare nei corpi animali senza causare grave tossicità biologica. È stato ampiamente utilizzato nella somministrazione di farmaci grazie al suo alto tasso di inclusione del farmaco 7,8, alla maggiore efficienza di somministrazione e alla riduzione dei danni agli animali.
La tecnologia di editing genetico ABE, che consente la conversione diretta di coppie di basi da A-T a G-C, è promettente nella ricerca genetica e medica. Rispetto all'attuale tecnologia di editing genetico tradizionale, la tecnologia ABE può ottenere un'accurata mutazione di base singola, riducendo così l'editing di sequenze di DNA non bersaglio, riducendo gli effetti off-target 9,10 e portando a zero rotture del doppio filamento del DNA11, il che riduce notevolmente il rischio di editing genetico12. La tecnologia ABE è anche altamente biocompatibile ed è più adatta per la ricerca sul trattamento delle malattie.
L'iniezione della vena caudale è un metodo comune utilizzato per la somministrazione in vivo di plasmidi, soprattutto nella terapia genica. Il gene bersaglio di questo studio è CaMK II δ, che è strettamente correlato al differenziamento degli osteoclasti13,14. L'uso dell'iniezione intraossea al posto della vena caudale consente al plasmide modificato di entrare negli osteoclasti direttamente nel midollo osseo. Questa trasmissione diretta al midollo osseo aumenta l'efficienza e la stabilità dell'espressione genica, il che è vantaggioso per il trattamento delle malattie legate alla disfunzione degli osteoclasti.
Qui, presentiamo un nuovo metodo che prevede l'incapsulamento del plasmide ABE con il chitosano e quindi l'introduzione diretta del complesso nei topi mediante iniezione intraossea. Attraverso questo metodo, speriamo di aprire la strada a trattamenti di terapia genica più efficaci per i plasmidi estranei che entrano negli organismi, in particolare le malattie legate alla disfunzione degli osteoclasti.
Protocollo
Tutti gli esperimenti sugli animali descritti sono stati approvati dal Comitato per la salute animale dell'Università di Anhui sull'uso e la cura degli animali. In questo studio, ABE è stato generosamente donato dal professor Tian Chi (Università di Scienza e Tecnologia di Shanghai, Shanghai, Cina; Figura 1D).
1. Costruzione del plasmide ABE
- Progettare il gRNA con la sequenza TCCATGATGCACAGACAGGA dove PAM è presentato da GAC.
- Secondo la guida all'elenco dei primer dell'azienda, aggiungere il primer al volume corrispondente di ddH2O per ottenere una concentrazione di 100 μM. Quindi, agitare e mescolare accuratamente per garantire una distribuzione uniforme. Per la reazione di ricottura, preparare una soluzione da 10 μM aggiungendo 10 μL della soluzione di primer stock alla nuova provetta per microcentrifuga, 90 μL di ddH2O e mescolare bene.
- Aggiungere 5 μL di primer a monte, 5 μL di primer a valle e 40 μL di ddH2O in una provetta da microcentrifuga. Per facilitare la reazione di ricottura, immergere il tubo in un contenitore riempito con acqua bollente per 5 minuti. Assicurarsi che il livello dell'acqua non raggiunga l'apertura del tubo per evitare che l'acqua penetri e contamini i primer. Raffreddare a temperatura ambiente per completare la reazione di ricottura.
- Aggiungere 2 μg di pLKO5.sgRNA.EFS.tRFP (Figura 1D), 1 μL di ESP3I, 4 μL di tampone e 33 μL di ddH2O in una provetta per microcentrifuga e lasciare reagire in un bagno d'acqua a temperatura costante a 37 °C per 45 minuti.
- Eseguire l'elettroforesi su gel di agarosio. Preparare un gel di agarosio all'1% pesando 0,5 g di agarosio e versarlo in una bottiglia di vetro. Aggiungere 50 ml di tampone 1x TAE nel flacone di vetro e agitare per sciogliere la polvere di agarosio. Scaldare nel forno a microonde per 2 minuti fino a completo scioglimento. Aggiungere 5 μl di YeaRed Nucleic Acid Gel Stain, mescolare accuratamente e versare nello stampo in gel; Dopo che il gel si è solidificato, inizia ad aggiungere i campioni.
NOTA: Per preparare 1xTAE, utilizzare i reagenti elencati nella Tabella 1. - Aggiungere 1x TAE alla vasca di elettroforesi e inserire il blocco di gel di agarosio configurato, che deve essere immerso nel TAE. Aggiungere 5 μl di tampone di carico 1x al prodotto di digestione dal passaggio 1.4 e mescolare. Aggiungere il campione al gel. Inoltre, aggiungi anche 6 μL di marcatore al gel.
- Eseguire il programma di elettroforesi a una tensione costante di 110 V per 35 minuti. Dopo l'elettroforesi, determinare la posizione della banda nel campione in base al marcatore e tagliare la striscia di gel con una lama pulita per il recupero del gel.
- Eseguire l'operazione di recupero del gel secondo le istruzioni del kit di maxi purificazione del gel. Impostare un bagnomaria a 50 °C. Aggiungere il volume corrispondente di soluzione di PN al tubo contenente il gel tagliato come da kit. Colpire il gel con la punta di una pipetta per facilitarne la dissoluzione e poi metterlo a bagnomaria. Agitare il tubo di tanto in tanto e osservare il processo di dissoluzione fino a quando il blocco di gel non è completamente sciolto.
- Prendi una colonna rotante, CA3, dal kit. Aggiungere 500 μl di soluzione BL dal kit nella colonna rotante e centrifugarla a 2.000 x g per 1 minuto per equilibrare la colonna. Dopo la centrifugazione, rimuovere il liquido di scarto sul fondo della provetta di raccolta e reinserire la colonna di adsorbimento nella provetta di raccolta.
- Continuare la centrifugazione a 2.000 x g per 1 minuto. Scartare il liquido di scarto sul fondo della provetta da centrifuga, aggiungere 600 μL di soluzione PN e centrifugare 2.000 x g per 30 s; Gettare nuovamente il liquido di scarto. Ripetere l'operazione e gettare nuovamente il liquido di scarto.
- Dopo la centrifugazione, sostituire la provetta di raccolta da 2 mL con una nuova provetta da 1,5 mL. Aprire il coperchio e lasciare asciugare in un luogo ventilato per 30 minuti per rimuovere completamente l'etanolo. Quindi, aggiungere 40 μl preriscaldati di ddH2O, centrifugare ed eluire il DNA richiesto.
NOTA: PN deve essere aggiunto con etanolo anidro secondo le istruzioni. L'uso di acqua preriscaldata può aumentare la resa. - Aggiungere 7 μL di prodotto di ricottura dalla fase 1.3, 3 μL di prodotto di recupero del gel dalla fase 1.11, 1 μL di DNA Ligasi T4, 2 μL di tampone Ligasi T4 10x e 7 μL di ddH2O in una provetta da microcentrifuga e conservare per una notte a 16 °C per ottenere il prodotto di legatura.
- Aggiungere 10 μL di prodotto di legatura e 70 μL di DH5α in una provetta da microcentrifuga su ghiaccio per 30 minuti. Impostare il bagnomaria a 42 °C. Dopo il bagno di ghiaccio, mettere il tubo nel bagnomaria per 90 s e poi di nuovo sul ghiaccio per 2 minuti.
- Aggiungere 600 μl di terreno liquido Luria-Bertani (LB) nella provetta e agitare su uno shaker per 30 minuti. Allo stesso tempo, irradiare una piastra media solida LB all'interno di una cappa a flusso laminare per 30 minuti. Dopo l'incubazione, distribuire uniformemente 100 μl di soluzione sulla piastra. Dopo il rivestimento, posizionare la piastra capovolta in un incubatore batterico a 37 °C per la coltura.
NOTA: Il DH5α viene solitamente conservato a -80 °C. Il mezzo solido LB viene preparato utilizzando i reagenti elencati nella Tabella 2. Il terreno liquido LB viene preparato utilizzando i reagenti elencati nella Tabella 3. - Selezionare una singola colonia ed espandere la colonia batterica per 14 ore su una piastra fresca. Prendere una provetta da centrifuga da 50 mL, aggiungere 50 mL di mezzo liquido LB, aggiungere 50 μL di ampicillina in rapporto 1:1000 e mescolare bene. Dividerlo in nuove provette a 15 ml per provetta.
- Controlla la piastra di coltura per colonie bianche rotonde con una buona crescita. Selezionarli con una pipetta da 10 μl e trasferirli nel terreno LB. Coltivarlo in un agitatore per 12 ore e poi inviare questo campione per il sequenziamento.
- Confrontare i risultati del sequenziamento con il gRNA target per identificare il ceppo con la sequenza corrispondente, Camk II δ-sgRNA, come ceppo target, e quindi estrarre il plasmide.
- Eseguire l'estrazione del plasmide secondo le istruzioni del kit mini plasmide. Aggiungere 500 μl di BL alla colonna di adsorbimento CP3, centrifugare per 1 minuto a 2.000 x g ed eliminare il liquido di scarto. Reinserire la colonna di adsorbimento nella provetta di raccolta. Aggiungere 500 μL di soluzione batterica e centrifugare a 4.000 x g per 10 min.
- Raccogli il flusso. Centrifugare nuovamente e, dopo aver rimosso il surnatante, aggiungere 250 μl di P1 alla provetta da centrifuga con precipitazione batterica e quindi trasferirla in una provetta da microcentrifuga e vortice. Aggiungere 250 μl di P2 e capovolgere delicatamente per 6x-8x. Aggiungere 350 μl di P3 e invertire delicatamente per 6x-8x. Appare un precipitato flocculante bianco.
- Centrifugare per 10 min a 2.000 x g. Trasferire il surnatante in una nuova colonna di adsorbimento CP3, centrifugare a 2.000 x g per 30 s e rimuovere il liquido di scarto. Aggiungere 600 μl di PW, centrifugare nuovamente a 2.000 x g per 30 s e rimuovere il liquido di scarto. Aggiungere nuovamente 600 μl di PW, centrifugare a 2.000 x g per 30 s e rimuovere il liquido di scarto.
- Posizionare la colonna di adsorbimento nella provetta di raccolta e centrifugare per 2 minuti a 2.000 x g per rimuovere il PW residuo. Quindi, posizionare la colonna di adsorbimento in una provetta da microcentrifuga pulita, lasciarla asciugare per 30 minuti e aggiungere 35 μl di ddH2O preriscaldato al centro del film di adsorbimento, quindi conservare il prodotto risultante a -20 °C per i test successivi.
NOTA: PW deve essere aggiunto con etanolo anidro secondo le istruzioni. L'uso di acqua preriscaldata può aumentare la resa.
2. Estrazione di cellule midollari
- Alcuni topi KM di età compresa tra 8 e 9 settimane, maschi e del peso di circa 30 g. Fissare i topi con la mano sinistra con la testa rivolta verso il basso e iniettare 150 μL di Pentobarbital sodico all'1% dal lato dell'addome con una siringa usando la mano destra. Confermare l'anestesia pizzicando le dita dei piedi.
- Eseguire la lussazione cervicale dei topi, rimuovere la tibia con forbici chirurgiche e pinzette e rimuovere il tessuto muscolare in eccesso circostante. Immergere la tibia rimossa in PBS e lavare, lasciando solo le ossa pulite.
- Assumere 1 mL di PBS in una siringa per eliminare le cellule del midollo osseo da un'estremità della tibia fino a quando l'osso non diventa bianco.
- Raccogliere la sospensione cellulare e centrifugare a 800 x g a 4 °C per 5 minuti. Dopo aver rimosso il surnatante, lisare i globuli rossi con 500 μL di lisato di globuli rossi. Dopo 1 minuto, aggiungere 1 mL di tampone di separazione per terminare il lisato dei globuli rossi. Dopo aver centrifugato a 800 x g a 4 °C per 5 minuti, rimuovere il surnatante e porre le cellule sul ghiaccio per un uso successivo.
NOTA: Se i globuli rossi sono completamente lisati, le cellule precipitano e non sono visibili globuli rossi. Se i globuli rossi possono ancora essere visti, possono essere lisati di nuovo, ma il tempo di incubazione non dovrebbe essere troppo lungo per evitare danni ad altre cellule.
3. Trasfezione del chitosano
- Preparazione del chitosano: Pesare 4 mg di chitosano e scioglierlo in 20 mL di soluzione di acido acetico 0,2 mg/mL. Regolare il pH a 5,5 con 10 M NaOH. Prelevare 500 μl di questa soluzione e aggiungerli alle singole provette per microcentrifuga.
- Aggiungere 2 μg, 3 μg, 4 μg e 5 μg di plasmidi ABE alle singole provette e scioglierli in 500 μl di 30 mM Na2SO4. Miscelare 500 μL di soluzione di chitosano e 500 μL di plasmide.
- Incubarli a bagnomaria a 50-55 °C per 15 min. Dopo aver mescolato bene le due soluzioni, agitare per 15-30 s e lasciare riposare per 30 min.
- Caratterizzazione del chitosano: Analizzare i diametri e i potenziali zeta del chitosano utilizzando la diffusione dinamica della luce (DLS), con la concentrazione di chitosano mantenuta a 0,1 mg/mL in ddH2O. Eseguire ogni campione 3 volte.
- Colate campioni da 0,1 mg/mL su un chip di silicio. Aggiungere 20 μl di campioni risospesi a griglie da 200 mesh e incubare a temperatura ambiente per 10 minuti. Colorare le griglie con acido fosfotungstico al 2% per 3 minuti e rimuovere il liquido rimanente con carta da filtro. Osservare con un microscopio elettronico a trasmissione.
- Efficienza di incapsulamento delle nanoparticelle: filtrare il surnatante con un filtro da 0,1 μm per rimuovere le particelle non precipitate. Caricare il plasmide e misurare la concentrazione con uno spettrofotometro a 260 nm. Le efficienze calcolate del plasmide ABE e del plasmide gRNA erano rispettivamente dell'84% ± 0,37% e dell'85% ± 0,53%.
- Iniettare 100 μL di chitosano incorporato con plasmide nei topi attraverso la cavità del midollo osseo (passaggio 5). Dopo 7 giorni dall'iniezione, isolare le cellule del midollo osseo e lisare i globuli rossi come descritto al punto 2.
- Aggiungere 2 mL di terreno DMEM contenente siero a piastre di coltura cellulare a 6 pozzetti e aggiungere le cellule del midollo osseo (circa 1 x 106) sospese in 500 μL di terreno DMEM contenente siero. Misurare l'intensità della fluorescenza con un microscopio a fluorescenza. Il plasmide CaMK II δ-sgRNA ha fluorescenza rossa TagRFP quando eccitato a 580 nm (Figura 2B).
NOTA: Il terreno DMEM contenente siero viene preparato utilizzando i reagenti elencati nella Tabella 4.
4. Citometria a flusso
- Preparare diversi dosaggi di plasmidi ABE: 2 μg, 3 μg, 4 μg e 5 μg e somministrare ai topi riceventi come descritto nel passaggio 3. Dopo 7 giorni, isolare le cellule del midollo osseo dai topi come descritto nel passaggio 3.
- Raccogliere le cellule del midollo osseo e centrifugare a 800 x g a 4 °C per 5 min. Eliminare il surnatante e lisare i globuli rossi con 500 μL di lisato di globuli rossi. Dopo 1 minuto, terminare la reazione con 1 mL di tampone di separazione e contare il numero di cellule con un contatore di cellule.
- Trasferire 1 x 106 cellule in una nuova provetta da microcentrifuga e centrifugare con 800 x g a 4 °C per 5 minuti. Dopo aver rimosso il surnatante, risospendere le cellule con 500 μL di PBS e trasferirle in una provetta di flusso. Misurare l'efficienza mediante citometria a flusso. Preparare un gruppo di controllo per citometria a flusso come descritto al punto 2.
NOTA: Sono stati esclusi i detriti cellulari e le celle morte con i segnali FSC più bassi e situati nell'angolo inferiore sinistro del grafico a dispersione FSC rispetto a SSC (meno di 400) (Figura 3). Poiché i plasmidi hanno fluorescenza rossa TagRFP, possono essere selezionati.
5. Iniezione nella cavità del midollo osseo
- Alcuni topi maschi KM di età compresa tra 8 e 9 settimane, del peso di circa 30 g. Anestetizzarli con un'iniezione intraperitoneale di 150 μL di pentobarbital sodico all'1% (passaggio 2.1). Confermare la profondità dell'anestesia pizzicando le dita dei piedi.
- Fissare il mouse anestetizzato in posizione supina sul tavolo operatorio e fissare l'arto anteriore del mouse con del nastro adesivo.
- Disinfettare la tibia posteriore dei topi con un tampone imbevuto di alcol. Riempire il plasmide da iniettare in una siringa da 1 ml con un ago da 26 G. Rimuovere le bolle d'aria e prepararsi per l'iniezione.
- Tocca lo stinco del mouse e tieni premuto lo stinco del mouse con le dita. Determinare la posizione dell'ago per iniezione in modo che l'asta tibiale sia penetrata dal piatto tibiale all'articolazione del ginocchio del topo. Ruotare l'ago per iniezione parallelamente alla tibia, infilzare nella cavità del midollo osseo e iniettare lentamente (circa 3 s) per ridurre al minimo il danno ai topi.
- Dopo l'iniezione, estrarre lentamente l'ago (circa 3 s) e utilizzare immediatamente un tampone imbevuto di alcol per fermare l'emorragia e disinfettare il sito di puntura.
- Posizionare delicatamente i topi in un ambiente caldo (20-26 °C), attendere che i topi si riprendano dall'anestesia e osservare il loro recupero.
6. Sequenziamento con metodo Sanger
- Dopo 7 giorni dall'iniezione, raccogliere le cellule del midollo osseo di topo come descritto nella fase 2 e isolare il DNA genomico utilizzando un kit di estrazione. Raccogliere le cellule centrifugando a 2.000 x g per 1 minuto. Scartare il surnatante, aggiungere 200 μL di soluzione GA e vibrare con un miscelatore a vortice. Aggiungere 20 μl di proteinasi K e 200 μl di GB, mescolare e quindi porre a bagnomaria a 70 °C per 10 minuti.
- Dopo l'incubazione, aggiungere 200 μl di etanolo anidro, agitare per 15 s, quindi mettere il liquido miscelato nella colonna di adsorbimento di supporto. Centrifugare a 2.500 x g per 1 minuto, eliminare il liquido di scarto sul fondo della provetta e aggiungere 500 μL di soluzione GD. Centrifugare e scartare il liquido di scarto, lavare il pellet 2 volte con PW e infine asciugarlo, eluendolo con 40 μL di acqua pura.
NOTA: PW deve essere aggiunto con etanolo anidro secondo le istruzioni. - Amplificare il gene bersaglio CaMKIIδ mediante PCR con la sequenza di primer diretta gctaaggtgataaatgtggcact e la sequenza di primer inversa di ctagtgtgcgggccagattc. Preparare la reazione PCR aggiungendo 25 μl di tampone 2x, 1 μl di dNTP Mix, 2 μl di primer diretto, 2 μl di primer inverso, 1 μl di DNA polimerasi, 2 μl di DNA stampo e 17 μl di ddH2O. Eseguire il seguente programma PCR: 95 °C per 3 min, 95 °C per 15 s, 53,7 °C per 30 s, 72 °C per 60 s, 12 °C per la conservazione del calore.
- Dopo la PCR, preparare un gel di agarosio all'1% ed eseguire l'elettroforesi su gel di agarosio a 110 V per 35 minuti (passaggio 1.5, passaggio 1.6). Tagliare l'area del gel target dopo l'elettroforesi e inviarla per il sequenziamento Sanger (Figura 4).
Risultati
I plasmidi in vitro sono stati iniettati nei topi mediante iniezione intraossea (Figura 1A). La dimensione media delle particelle delle nanoparticelle era di circa 202,9 nm, il potenziale era di 2,77 mV e il PDI era di 0,22 (Figura 1B). La Figura 1C mostra la forma superficiale delle nanoparticelle osservate come sferiche al microscopio elettronico. La Figura 1D mostra la mappa plasmidica del vettore ABE e la mappa plasmidica del vettore gRNA.
La trasduzione plasmidica è stata osservata al microscopio a fluorescenza. La Figura 2A mostra le cellule normali del midollo osseo. Dopo 7 giorni di trasferimento del plasmide nel topo, le cellule del midollo osseo del topo sono state osservate al microscopio a fluorescenza. Rispetto al gruppo di controllo, l'efficienza di trasfezione del gruppo di trasfezione del chitosano è stata significativamente migliorata (Figura 2B). Ciò indica che il chitosano come materiale di inclusione può migliorare l'efficienza del plasmide di trasduzione in vitro.
Dopo 7 giorni dalla somministrazione nei topi, il plasmide era completamente espresso. Sono state estratte cellule del midollo osseo dai topi e l'efficienza di trasfezione delle cellule del midollo osseo è stata rilevata mediante citometria a flusso. I risultati hanno mostrato che l'efficienza di trasfezione delle cellule trasfettate direttamente con il plasmide ABE era dello 0,92% ± dello 0,02%. L'efficienza di trasfezione delle cellule del midollo osseo trasfettate con 2 μg di ABE rivestite con chitosano è stata del 40,80% ± del 4,31%, quella delle cellule del midollo osseo trasfettate con 3 μg di ABE rivestite con chitosano è stata del 47,20% ± del 5,37%, quella delle cellule del midollo osseo trasfettate con 4 μg di ABE rivestite con chitosano è stata del 51,20% ± 2,02% e quella delle cellule del midollo osseo trasfettate con 5 μg di ABE rivestite con chitosano è stata del 48,2% ± del 7,39% (Figura 3B). L'efficienza del gruppo di trasfezione del chitosano era superiore a quella del gruppo di controllo (Figura 3A). I risultati della citometria a flusso hanno mostrato che il plasmide trasdotto potrebbe essere espresso in modo stabile ed efficiente nei topi utilizzando il metodo descritto in questo articolo. Dal punto di vista dell'efficienza di espressione completa e del risparmio di materiale, è stato possibile selezionare lo schema di trasduzione una tantum di 3 μg di plasmide.
Abbiamo estratto il DNA genomico dalle cellule del midollo osseo e amplificato il gene bersaglio mediante PCR. Dopo il sequenziamento con Sanger, l'efficienza di editing in vivo in A1 è stata del 14,27% ± 0,35% e quella in A2 è stata del 10,69% ± 0,30% (Figura 4).

Figura 1: Diagramma di flusso dell'iniezione ossea, caratterizzazione delle nanoparticelle e mappa vettoriale plasmidica. (A) Procedura per l'iniezione intraossea. (B) Dimensione delle particelle, potenziale Zeta e PDI delle nanoparticelle formate da plasmidi ABE incorporati con chitosano. (C) Immagine al microscopio elettronico di nanoparticelle. (D) Il lato sinistro mostra la mappa del plasmide vettore ABE e il lato destro mostra la mappa del plasmide vettore gRNA. Clicca qui per visualizzare una versione più grande di questa figura.
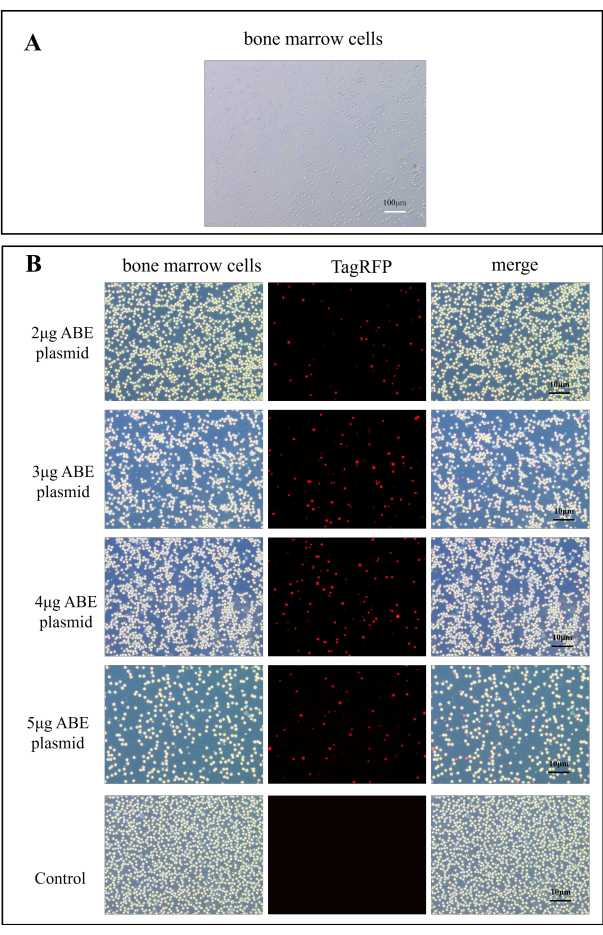
Figura 2: Immagini a fluorescenza per l'efficienza di trasfezione del plasmide ABE. (A) Immagine A100x di cellule del midollo osseo. (B) Le mappe di fluorescenza delle cellule del midollo osseo nel gruppo di trasfezione del chitosano sono state ottenute 7 giorni dopo la trasfezione di 2 μg, 3 μg, 4 μg e 5 μg di plasmide ABE, rispetto a quelle del gruppo di controllo dopo l'iniezione diretta di plasmide ABE Fare clic qui per visualizzare una versione più grande di questa figura.
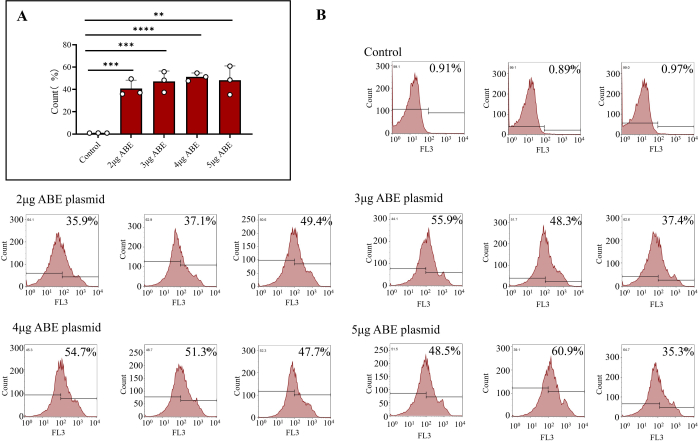
Figura 3: Risultati della citometria a flusso di cellule del midollo osseo trasfettate. (A) Cellule del midollo osseo trattate con citometria a flusso trasfettate con plasmidi ABE da 2 μg, 3 μg, 4 μg e 5 μg (gruppo di trasfezione del chitosano) e iniettate direttamente (gruppo di controllo). I risultati hanno mostrato che l'efficienza di trasfezione delle cellule del midollo osseo iniettate direttamente con plasmidi ABE era scarsa, mentre l'efficienza di trasfezione e l'effetto di trasfezione degli altri quattro gruppi erano più elevati. (B) I risultati della citometria a flusso includono l'efficienza di trasfezione del gruppo di controllo e diversi rapporti di plasmide ABE. Clicca qui per visualizzare una versione più grande di questa figura.
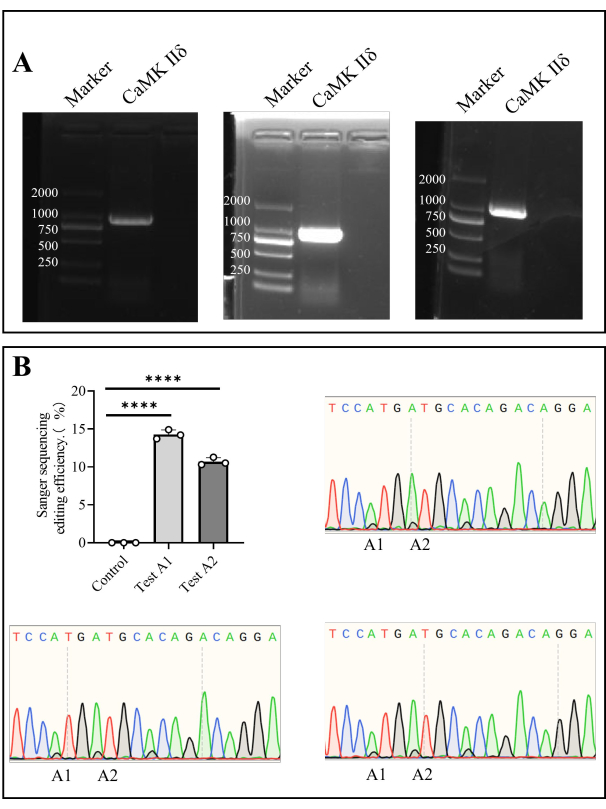
Figura 4: Sequenziamento Sanger di Camk II δ l'efficienza dell'editing nei topi. (A) Rilevamento dell'elettroforesi su gel di agarosio di Camk II δ. (B) Il grafico a barre mostra l'analisi dei risultati del sequenziamento Sanger dell'efficienza di editing di Camk II δ nei topi, mentre i diagrammi di sequenziamento Sanger mostrano i risultati del sequenziamento. Clicca qui per visualizzare una versione più grande di questa figura.
| Reagenti | Concentrazione utilizzata |
| Acido etilendiamminatetraacetico | 1,142 ml |
| Acido acetico glaciale | 2 ml |
| Tris | 4,84 grammi |
Tabella 1: Reagenti per la preparazione del tampone TAE 1X
| Reagente | Volume |
| Agar | 7,5 g |
| Amp | Aggiungere 500 μl quando sterilizzato e raffreddato a 70 °C |
| ddH2O | Fino a 500 μl |
| Cloruro di sodio | 5 g |
| triptone | 5 g |
| Estratto di lievito | 2,5 g |
Tabella 2: Reagenti per la preparazione del terreno solido LB.
| Reagente | Volume |
| ddH2O | fino a 1 l |
| peptone | 10 g |
| Cloruro di sodio | 10 g |
| Estratto di lievito | 5 g |
Tabella 3: Reagenti per la preparazione del terreno liquido LB.
| Reagente | Volume |
| DMEM | 450 ml |
| Siero fetale bovino | 5 ml |
| MEM | 5 ml |
| Penicillina/Streptomicina | 5 ml |
| β-mercaptoetanolo | 500 μl |
Tabella 4: Reagenti per la preparazione di un terreno DMEM contenente siero.
Discussione
Per la ricerca biomedica, la sfida nel fornire plasmidi esogeni agli animali consiste nel migliorare l'efficienza della somministrazione e dell'espressione genica, riducendo contemporaneamente al minimo i danni agli animali per ottenere l'effetto terapeutico desiderato15,16. Presentiamo un nuovo metodo di iniezione intraossea di rilascio di plasmidi ABE mediato da chitosano nelle cellule del midollo osseo di topi riceventi. Questa strategia migliora l'efficienza della somministrazione dei plasmidi e l'espressione genica.
In primo luogo, in questo studio, il chitosano, offerto come sistema vettoriale non virale, è stato utilizzato come metodo per la somministrazione di plasmidi. Poiché è stato dimostrato che l'efficienza di trasfezione del metodo convenzionale è relativamente bassa, l'introduzione di plasmidi per l'espressione genica a lungo termine può essere influenzata dal sistema immunitario17,18. Il chitosano, un polisaccaride naturale, è rinomato per la sua eccellente biocompatibilità, capacità di incapsulamento plasmidico e bassa tossicità. L'uso del chitosano come veicolo di somministrazione aiuta a prevenire potenziali effetti immuno-correlati durante l'espressione a lungo termine del plasmide esogeno, migliorando anche l'efficienza di trasferimento del plasmide 19,20,21. Per la ricerca futura, riconosciamo l'importanza di valutare la tossicità e i potenziali effetti sul midollo osseo delle nanoparticelle di chitosano quando somministrate per iniezione intraossea. Prevediamo di incorporare questi aspetti nel nostro futuro progetto sperimentale, compresa la conduzione di valutazioni complete della tossicità e analisi istopatologiche. Ciò comporterà la valutazione di vari parametri come la vitalità cellulare, le risposte infiammatorie e la morfologia del midollo osseo in diversi punti temporali e livelli di dose. Inoltre, prenderemo in considerazione l'utilizzo di tecniche di imaging avanzate e metodi di biologia molecolare per ottenere una comprensione più profonda del profilo di sicurezza e dei potenziali impatti sulla salute del midollo osseo dopo il trattamento con nanoparticelle di chitosano.
In secondo luogo, ABE è stato selezionato come editor genetico in grado di convertire i nucleotidi dell'adenina in nucleotidi della guanina senza introdurre rotture del DNA a doppio filamento. Rispetto a CRISPR/Cas9, ABE offre una maggiore precisione correggendo direttamente una singola base senza indurre rotture a doppio filamento, riducendo così gli effetti fuori bersaglio e i potenziali danni. Tuttavia, quando si progetta il gRNA target, è necessaria un'attenta considerazione per prevenire il legame non specifico e ridurre gli effetti off-target22. Abbiamo implementato misure per ridurre il rischio, ma evitarlo completamente rimane difficile e richiede ulteriori ricerche.
In terzo luogo, per la somministrazione in vivo, abbiamo sviluppato una strategia di iniezione intraossea. L'iniezione intraossea è un metodo sicuro ed efficace, come è stato riportato nella revisione del sistema da Betzler et al.23. Il gene bersaglio in questo studio è Camk II δ, che svolge un ruolo chiave nella regolazione del differenziamento degli osteoclasti13,14. Rispetto al metodo convenzionale di iniezione della vena caudale, l'iniezione intraossea può rilasciare il plasmide δ Camk II mirato direttamente ed efficientemente negli osteoclasti, raggiungendo così l'elevata efficienza di editing in vivo del 14,27% al locus A1, mentre del 10,69% al locus A2 nei topi riceventi.
Inoltre, va notato che l'iniezione intraossea è un'operazione che richiede un'attenzione particolare. Quindi, prima dell'esperimento, abbiamo usato il colorante blu come pratica per monitorare se l'iniezione viene iniettata correttamente nella cavità del midollo osseo dei topi. Inoltre, a causa della possibilità che i topi riceventi possano soffrire dei danni causati dall'iniezione intraossea, si consiglia di scegliere topi del peso di circa 30 g. Sottolineiamo l'importanza di diversi passaggi chiave nella procedura di iniezione intraossea: evitare di somministrare una quantità eccessiva di anestetico, garantire una disinfezione accurata sia prima che dopo l'iniezione per prevenire l'infezione e ruotare l'ago per evitare blocchi causati dal midollo osseo. Se bloccato, astenersi dal forzare il fluido nella cavità del midollo osseo del topo, che potrebbe potenzialmente portare alla morte del topo. Il plasmide deve essere iniettato lentamente ed è fondamentale monitorare le condizioni dei topi dopo l'iniezione per assicurarsi che non si verifichino decessi dovuti a una manipolazione impropria. Anche se abbiamo specificato che i topi pesano 30 g per un'iniezione intraossea di successo. In ambito clinico, è necessario considerare la variabilità specifica del paziente e i nostri studi futuri, come l'ottimizzazione delle tecniche di iniezione per diversi intervalli di peso e l'esplorazione di metodi di somministrazione alternativi, sono attualmente in corso.
Questo metodo può essere ampiamente utilizzato nell'editing di cellule derivate dal midollo osseo, inclusi osteoblasti, cellule eritroidi, linfociti, cellule ematopoietiche, ecc. L'elevata precisione ed efficienza del metodo consentono ai ricercatori di utilizzarlo non solo per le malattie causate da una funzione anomala degli osteoclasti, ma anche nel campo della terapia genica per altre malattie del sangue. Ad esempio, anche le malattie del sangue comuni possono essere ben allineate con i nostri metodi di trattamento. Le malattie del sangue causate da mutazioni di singole basi24 possono essere trattate con gli editor genici ABE e l'iniezione intraossea può anche fornire plasmidi direttamente alle cellule staminali ematopoietiche, ottenendo così migliori effetti terapeutici. Tuttavia, il metodo presenta limitazioni nella sua applicazione per i processi di modifica e consegna di tipi cellulari di origine non midollare, come le cellule muscolari e adipose.
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti.
Riconoscimenti
Questo lavoro è stato finanziato dalla Fondazione per le Scienze Naturali della Provincia di Anhui (2208085MC74, 2208085MC51) e dalla Fondazione per la Ricerca Scientifica del Dipartimento dell'Istruzione della Provincia di Anhui, Cina (KJ2021A0055).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 ml PCR Tubes, Flat cap | LABSELECT | PT-02-C | |
| 1 mL syringe | Anhui Jiangnan medical equipment Co., LTD | / | |
| 1% Pentobarbital sodium | / | / | |
| 1.5 ml Microcentrifuge Tubes | LABSELECT | MCT-001-150 | |
| 10 × DNA Loading Buffer | Vazyme | P022-01 | |
| 10X T4 DNA Ligase Buffer 1 ml | TaKaRa Biotechnology(Dalian)Co.,LTD | 2011A | |
| 1250 μl Pipette Tip 102.1mm | LABSELECT | T-001-1250 | |
| 200 μl Pipette Tips 50.55mm | LABSELECT | T-001-200 | |
| 2x Phanta Max Buffera | Vazyme | P505-d1 | |
| 4 ? centrifuge | Thermo Fisher | 75002425 | |
| 50 ml Centrifuge tube | LABSELECT | T-012-50 | |
| 6-well Cell Culture Plates | LABSELECT | 11110 | |
| Agar | Sangon Biotech | A505255-0250 | |
| Amp | Abiowell | / | |
| Chitosan | Sangon Biotech | A600614-0500 | |
| Constant temperature culture shaker | Shanghai Zhicheng Analytical Instrument Manufacturing Co., LTD | ZWY-200D | |
| Countess Automated Cell Counter | Thermo Fisher Scientific | Countess II/II FL | |
| Countess Cell Counting Chamber Slides and Holder, disposable | Thermo Fisher Scientific | C10228 | |
| CutSmart Buffer | New England Biolabs | B7204SVIAL | |
| DH5α | General Biosystems | CS01010 | |
| DMEM | gibco | C11995500BT | |
| dNTP Mix | Vazyme | P505-d1 | |
| Esp3I enzyme | NEBiolabs | R0734S | |
| Ethylenediaminetetraacetic acid | VETEC (sigma-aldrich) | V900106 | |
| Fetal bovine serum | OriCell | FBSAD-01011-500 | |
| Flow cytometer | BD FACSCalibur | 342975 | |
| Flow tube | Beyotime Biotechnology | FFC005-1bag | |
| Fluorescence microscope | Leica | 427019 | |
| gel maxi purification kit | TIANGEN | DP210 | |
| Genomic DNA extraction kit | TIANGEN | DP304 | |
| Glacial acetic acid | China National Pharmaceutical Group Corporation | 10000218 | |
| GoldBand DL2,000 DNA Marker | YESEN | 10501ES60 | |
| Ice machine | shanghaizhengqiao | BNS-30 | laboratory reserved |
| ImunoSep Buffer | Precision Biomedicals Co.,LTD | 604050 | |
| Megafuge 8 Small Benchtop Centrifuge Series | Thermo Fisher Scientific | 75004250 | |
| MEM | Life Technology | 11140050 | |
| NanoDrop 2000 | Thermo Fisher Scientific, USA | ||
| NaOH | SIGAM | S5881-500G | |
| PBS | XiGene | XG3650 | |
| PCMV-SPRY-ABE8E vector | / | / | |
| pcr amplification apparatus | Thermo Fisher | AKC96300441 | |
| Penicillin/Streptomycin | Solarbio | P1400 | |
| peptone | Sangon Biotech | A505247-0500 | |
| Phanta Max Super-Fidelity DNA Polymerase | Vazyme | P505-d1 | |
| Red cell lysate | Beyotime | C3702 | |
| Sodium chloride | China National Pharmaceutical Group Corporation | 10019318 | |
| Sodium sulfate | aladdin | S433911 | |
| T-001-10 10μl Pipette Tips 31.65mm | LABSELECT | T-001-10 | |
| T4 DNA Ligase | TaKaRa Biotechnology(Dalian)Co.,LTD | 2011A | |
| Tris | BioFroxx | 1115KG001 | |
| tryptone | Sangon Biotech | A505250-0500 | |
| vortex mixer | sigma | Z258423 | |
| Water bath | shanghaiyiheng | DK-80 | |
| YeaRed Nucleic Acid Gel Stain | YESEN | 10203ES76 | |
| Yeast extract | BBI | A610961-0500 | |
| Zetasizer Nano | Malvern Panalytical | Zetasizer Nano ZS | |
| β-mercaptoethanol | Sigma | 444203 |
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon