Method Article
Mejora de la entrega y expresión génica mediante inyección intraósea de nanopartículas de quitosano Encapsulado en plásmidos de base de adenina
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, presentamos un enfoque de terapia génica que administra quitosano recubierto de ABE directamente a la médula ósea mediante inyección intraósea.
Resumen
La administración de plásmidos exógenos en animales de experimentación es crucial en la investigación biomédica, incluida la investigación de las funciones de los genes, la elucidación de los mecanismos de la enfermedad y la evaluación de la eficacia de los fármacos. Sin embargo, la eficiencia de transfección del método actual es relativamente baja, y la introducción de plásmidos para la expresión génica a largo plazo puede verse afectada por el sistema inmunológico. Para abordar estas limitaciones, desarrollamos e investigamos un método novedoso que utiliza quitosano para encapsular plásmidos del editor de bases de adenina (ABE) y luego administrar directamente el complejo a la médula ósea de ratones mediante inyección intraósea. En este estudio, para dirigirse al gen CaMK II δ , que está estrechamente relacionado con la diferenciación de osteoclastos, utilizamos quitosano para encapsular los plásmidos de δ CaMK II de ABE. Inyectamos directamente la carga plasmídica en la cavidad de la médula ósea mediante inyección intraósea. Los resultados mostraron una alta eficiencia de edición in vivo del 14,27% en el locus A1 y del 10,69% en el locus A2 en ratones receptores. Esta novedosa estrategia no solo es especialmente adecuada para las enfermedades causadas por una función anormal de los osteoclastos, sino que también tiene un potencial significativo para avanzar en el campo de la terapia génica.
Introducción
La terapia génica se ha convertido en un enfoque prometedor en el campo de la investigación biomédica 1,2. Ofrece el potencial de tratar una variedad de trastornos mediante la introducción de plásmidos extraños en animales de experimentación para modular la expresión de genes específicos y estudiar sus efectos terapéuticos. Sin embargo, los métodos convencionales de administración han encontrado algunos problemas que limitan su efectividad y seguridad3. Las preocupaciones incluyen baja eficiencia de transfección, alto daño biológico y baja eficiencia de expresión génica. Por lo tanto, es necesario establecer un nuevo método de administración para la terapia génica que pueda superar estas limitaciones.
El quitosano es un polisacárido natural con buena biodegradabilidad y biocompatibilidad 4,5,6, lo que hace que sea fácil de degradar en el cuerpo de los animales sin causar una toxicidad biológica grave. Ha sido ampliamente utilizado en la administración de fármacos debido a su alta tasa de inclusión de fármacos 7,8, mejora de la eficiencia de la administración y reduce el daño a los animales.
La tecnología de edición genética ABE, que permite la conversión directa de pares de bases de A-T a G-C, es prometedora en la investigación genética y médica. En comparación con la tecnología de edición de genes convencional actual, la tecnología ABE puede lograr una mutación precisa de una sola base, reduciendo así la edición de secuencias de ADN no objetivo, reduciendo los efectos fuera del objetivo 9,10 y conduciendo a cero roturas de doble cadena de ADN11, lo que reduce en gran medida el riesgo de edición de genes12. La tecnología ABE también es altamente biocompatible y es más adecuada para la investigación del tratamiento de enfermedades.
La inyección de la vena de cola es un método común utilizado para la administración in vivo de plásmidos, especialmente en la terapia génica. El gen diana de este estudio es el CaMK II δ, que está estrechamente relacionado con la diferenciación de los osteoclastos13,14. El uso de la inyección intraósea en lugar de la vena de la cola permite que el plásmido editado ingrese a los osteoclastos directamente en la médula ósea. Esta transmisión directa a la médula ósea aumenta la eficiencia y la estabilidad de la expresión génica, lo que es ventajoso para el tratamiento de enfermedades relacionadas con la disfunción de los osteoclastos.
Aquí, presentamos un método novedoso que consiste en encapsular el plásmido ABE con quitosano y luego introducir directamente el complejo en ratones mediante inyección intraósea. A través de este método, esperamos allanar el camino para tratamientos de terapia génica más efectivos para plásmidos extraños que ingresan a los organismos, especialmente enfermedades relacionadas con la disfunción de los osteoclastos.
Protocolo
Todos los experimentos con animales descritos fueron aprobados por el Comité de Salud Animal de la Universidad de Anhui sobre el Uso y Cuidado de los Animales. En este estudio, el ABE fue generosamente donado por el profesor Tian Chi (Universidad de Ciencia y Tecnología de Shanghai, Shanghai, China; Figura 1D).
1. Construcción de plásmidos ABE
- Diseño de ARNg con la secuencia TCCATGATGCACAGACAGGA donde PAM es presentado por GAC.
- De acuerdo con la guía de la lista de cebadores de la compañía, agregue el cebador al volumen correspondiente de ddH2O para lograr una concentración de 100 μM. Luego, agite y mezcle bien para asegurar una distribución uniforme. Para la reacción de recocido, prepare una solución de 10 μM agregando 10 μL de la solución de cebador original al nuevo tubo de microcentrífuga, 90 μL de ddH2O, y mezcle bien.
- Agregue 5 μL de cebador aguas arriba, 5 μL de cebador aguas abajo y 40 μL de ddH2O en un tubo de microcentrífuga. Para facilitar la reacción de recocido, sumerja el tubo en un recipiente lleno de agua hirviendo durante 5 minutos. Asegúrese de que el nivel del agua no llegue a la abertura del tubo para evitar que el agua entre y contamine los cebadores. Enfríe a temperatura ambiente para completar la reacción de recocido.
- Añadir 2 μg de pLKO5.sgRNA.EFS.tRFP (Figura 1D), 1 μL de ESP3I, 4 μL de tampón y 33 μL de ddH2O en un tubo de microcentrífuga y dejar reaccionar en un baño de agua a temperatura constante a 37 °C durante 45 min.
- Realizar electroforesis en gel de agarosa. Prepare un gel de agarosa al 1% pesando 0,5 g de agarosa y viértalo en una botella de vidrio. Agregue 50 mL de 1x tampón TAE en el frasco de vidrio y agite para disolver el polvo de agarosa. Calentar en el horno microondas durante 2 min hasta que se derrita por completo. Agregue 5 μL de tinción de gel de ácido nucleico YeaRed, mezcle bien y vierta en el molde de gel; Después de que el gel se solidifique, comience a agregar las muestras.
NOTA: Para preparar 1xTAE, utilice los reactivos enumerados en la Tabla 1. - Agregue 1x TAE al tanque de electroforesis y colóquelo en el bloque de gel de agarosa configurado, que debe sumergirse en TAE. Añadir 5 μL de tampón de carga 1x al producto de digestión del paso 1.4 y mezclar. Añade la muestra al gel. Además, añade 6 μL de rotulador al gel.
- Ejecute el programa de electroforesis a un voltaje constante de 110 V durante 35 min. Después de la electroforesis, determine la posición de la banda en la muestra de acuerdo con el marcador y corte la tira de gel con una cuchilla limpia para la recuperación del gel.
- Realice la operación de recuperación de gel de acuerdo con las instrucciones del kit de purificación de gel maxi. Ponga un baño de agua a 50 °C. Agregue el volumen correspondiente de solución de PN al tubo que contiene el gel cortado según el kit. Pinche el gel con la punta de una pipeta para facilitar la disolución y luego póngalo en el baño de agua. Agite el tubo de vez en cuando y observe el proceso de disolución hasta que el bloque de gel se disuelva por completo.
- Tome una columna giratoria, CA3, del kit. Añada 500 μL de solución BL del kit a la columna de centrifugación y centrifugala a 2.000 x g durante 1 minuto para equilibrar la columna. Después de la centrifugación, retire el líquido residual en el fondo del tubo de recolección y vuelva a colocar la columna de adsorción en el tubo de recolección.
- Continúe la centrifugación a 2.000 x g durante 1 min. Deseche el líquido residual en el fondo del tubo de centrífuga, agregue 600 μL de solución de PN y centrifugue 2,000 x g durante 30 s; Deseche el líquido residual nuevamente. Repita la operación y vuelva a desechar el líquido residual.
- Después de la centrifugación, sustituya el tubo de recogida de 2 ml por un tubo nuevo de 1,5 mL. Abra la tapa y deje secar en un lugar ventilado durante 30 minutos para eliminar el etanol por completo. A continuación, añada 40 μL precalentados de ddH2O, centrífuga, y eluya el ADN requerido.
NOTA: El PN debe agregarse con etanol anhidro de acuerdo con las instrucciones. El uso de agua precalentada puede aumentar el rendimiento. - Añadir 7 μL de producto de recocido del paso 1.3, 3 μL de producto de recuperación en gel del paso 1.11, 1 μL de ADN ligasa T4, 2 μL de tampón de ligasa T4 10x y 7 μL de ddH2O en un tubo de microcentrífuga, y mantener toda la noche a 16 °C para obtener el producto de ligadura.
- Añadir 10 μL de producto de ligadura y 70 μL de DH5α en un tubo de microcentrífuga sobre hielo durante 30 min. Ajuste el baño de agua a 42 °C. Después del baño de hielo, coloque el tubo en el baño de agua durante 90 s y luego nuevamente en hielo durante 2 minutos.
- Añadir 600 μL de medio líquido Luria-Bertani (LB) al tubo y agitar en una coctelera durante 30 min. Al mismo tiempo, irradie una placa de medio sólido LB dentro de una cabina de flujo laminar durante 30 min. Después de la incubación, distribuya uniformemente 100 μL de la solución en la placa. Después del recubrimiento, coloque la placa boca abajo en una incubadora bacteriana a 37 °C para el cultivo.
NOTA: El DH5α se almacena generalmente a -80 °C. El medio sólido LB se prepara utilizando los reactivos enumerados en la Tabla 2. El medio líquido LB se prepara utilizando los reactivos enumerados en la Tabla 3. - Seleccione una sola colonia y amplíe la colonia bacteriana durante 14 h en un plato fresco. Tome un tubo de centrífuga de 50 mL, agregue 50 mL de medio líquido LB, agregue 50 μL de ampicilina en una proporción de 1:1000 y mezcle bien. Divida esto en tubos nuevos a 15 mL por tubo.
- Revise la placa de cultivo para ver si hay colonias blancas redondas con buen crecimiento. Selecciónelos con una pipeta de 10 μL y transfiéralos al medio LB. Cultive esto en un agitador durante 12 h y luego envíe esta muestra para su secuenciación.
- Compare los resultados de la secuenciación con el ARNg objetivo para identificar la cepa con la secuencia correspondiente, Camk II δ-sgRNA, como cepa objetivo y, a continuación, extraiga el plásmido.
- Realice la extracción de plásmidos de acuerdo con las instrucciones del mini kit de plásmidos. Añadir 500 μL de BL a la columna de adsorción CP3, centrifugar durante 1 min a 2.000 x g y desechar el líquido residual. Vuelva a colocar la columna de adsorción en el tubo de recolección. Añadir 500 μL de solución bacteriana y centrifugar a 4.000 x g durante 10 min.
- Recoja el flujo a través. Vuelva a centrifugar y, después de eliminar el sobrenadante, agregue 250 μL de P1 al tubo de centrífuga con precipitación bacteriana y luego transfiéralo a un tubo de microcentrífuga y vórtice. Agregue 250 μL de P2 e invierta suavemente para 6x-8x. Agregue 350 μL de P3 y nuevamente invierta suavemente para 6x-8x. Aparece el precipitado floculante blanco.
- Centrífuga durante 10 min a 2.000 x g. Transfiera el sobrenadante a una nueva columna de adsorción CP3, centrifugue a 2.000 x g durante 30 s y elimine el líquido residual. Añadir 600 μL de PW, centrifugar de nuevo a 2.000 x g durante 30 s y eliminar el líquido residual. Añadir de nuevo 600 μL de PW, centrifugar a 2.000 x g durante 30 s y eliminar el líquido residual.
- Coloque la columna de adsorción en el tubo de recolección y centrifugue durante 2 minutos a 2.000 x g para eliminar el PW residual. A continuación, coloque la columna de adsorción en un tubo de microcentrífuga limpio, déjela secar durante 30 minutos y añada 35 μL de ddH2O precalentado en el centro de la película de adsorción, almacene el producto resultante a -20 °C para pruebas posteriores.
NOTA: El PW debe agregarse con etanol anhidro de acuerdo con las instrucciones. El uso de agua precalentada puede aumentar el rendimiento.
2. Extracción de células de médula ósea
- Ratones KM seleccionados de 8-9 semanas de edad, machos, y con un peso aproximado de 30 g. Fije los ratones en la mano izquierda con la cabeza hacia abajo e inyecte 150 μL de pentobarbital sódico al 1% desde el costado del abdomen con una jeringa con la mano derecha. Confirme la anestesia con un pellizco en los dedos de los pies.
- Realice la luxación cervical de ratones, extirpe la tibia con tijeras quirúrgicas y pinzas, y elimine el exceso de tejido muscular circundante. Remoje la tibia extraída en PBS y lave, dejando solo los huesos limpios.
- Tome 1 ml de PBS en una jeringa para enjuagar las células de la médula ósea de un extremo de la tibia hasta que el hueso esté blanco.
- Recoja la suspensión de la célula y la centrífuga a 800 x g a 4 °C durante 5 min. Después de eliminar el sobrenadante, lisar los glóbulos rojos con 500 μL de lisado de glóbulos rojos. Después de 1 minuto, agregue 1 ml de tampón de separación para terminar el lisado de glóbulos rojos. Después de centrifugar a 800 x g a 4 °C durante 5 min, retire el sobrenadante y coloque las células en hielo para su uso posterior.
NOTA: Si los glóbulos rojos están completamente lisados, las células se precipitan y no hay glóbulos rojos visibles. Si los glóbulos rojos aún se pueden ver, se pueden lisar nuevamente, pero el tiempo de incubación no debe ser demasiado largo para evitar daños a otras células.
3. Transfección de quitosano
- Preparación de quitosano: Pesar 4 mg de quitosano y disolver en 20 ml de solución de ácido acético de 0,2 mg/mL. Ajuste el pH a 5,5 con 10 M de NaOH. Tome 500 μL de esta solución y añádala a los tubos individuales de la microcentrífuga.
- Agregue 2 μg, 3 μg, 4 μg y 5 μg de plásmidos ABE a los tubos individuales y disuélvalos en 500 μL de 30 mM Na2SO4. Mezclar 500 μL de solución de quitosano y 500 μL del plásmido.
- Incubarlos al baño maría a 50-55 °C durante 15 min. Después de mezclar bien las dos soluciones, agite el vórtice durante 15-30 s y deje reposar durante 30 min.
- Caracterización del quitosano: Analice los diámetros y potenciales zeta del quitosano utilizando dispersión dinámica de luz (DLS), con la concentración de quitosano mantenida en 0,1 mg/mL en ddH2O. Ejecute cada muestra 3 veces.
- Vierta muestras de 0,1 mg/mL en un chip de silicio. Agregue 20 μL de las muestras resuspendidas a rejillas de 200 mallas e incube a temperatura ambiente durante 10 minutos. Tiñe las rejillas con ácido fosfotúngstico al 2% durante 3 minutos y retira el líquido restante con papel de filtro. Observar con un microscopio electrónico de transmisión.
- Eficiencia de encapsulación de nanopartículas: Filtre el sobrenadante con un filtro de 0,1 μm para eliminar las partículas no precipitadas. Cargue el plásmido y mida la concentración con un espectrofotómetro a 260 nm. Las eficiencias calculadas del plásmido ABE y el plásmido de ARNg fueron del 84% ± 0,37% y del 85% ± 0,53%, respectivamente.
- Inyecte 100 μL de quitosano incrustado con plásmido en ratones a través de la cavidad de la médula ósea (paso 5). Después de 7 días de la inyección, aísle las células de la médula ósea y lise los glóbulos rojos como se describe en el paso 2.
- Agregue 2 mL de medio DMEM que contiene suero a placas de cultivo celular de 6 pocillos y agregue las células de médula ósea (aproximadamente 1 x 106) suspendidas en 500 μL de medio DMEM que contiene suero. Mida la intensidad de fluorescencia bajo un microscopio de fluorescencia. El plásmido CaMK II δ-sgRNA tiene fluorescencia roja TagRFP cuando se excita a 580 nm (Figura 2B).
NOTA: El medio DMEM que contiene suero se prepara utilizando los reactivos enumerados en la Tabla 4.
4. Citometría de flujo
- Prepare diferentes dosis de plásmidos ABE: 2 μg, 3 μg, 4 μg y 5 μg, y entréguelos a los ratones receptores como se describe en el paso 3. Después de 7 días, aísle las células de la médula ósea de los ratones como se describe en el paso 3.
- Recoja las células de la médula ósea y centrifugue a 800 x g a 4 °C durante 5 min. Deseche el sobrenadante y lise los glóbulos rojos con 500 μL de lisado de glóbulos rojos. Después de 1 minuto, finalice la reacción con 1 ml de tampón de separación y cuente el número de células con un contador de células.
- Transfiera 1 x 106 celdas a un nuevo tubo de microcentrífuga y centrifugue con 800 x g a 4 °C durante 5 min. Después de eliminar el sobrenadante, vuelva a suspender las células con 500 μL de PBS y transfiéralas a un tubo de flujo. Medir la eficiencia por citometría de flujo. Prepare un grupo de control de citometría de flujo como se describe en el paso 2.
NOTA: Se excluyeron los residuos de celdas y las celdas muertas con las señales FSC más bajas y ubicadas en la esquina inferior izquierda del diagrama de dispersión FSC versus SSC (menos de 400) (Figura 3). Debido a que los plásmidos tienen fluorescencia roja TagRFP, se pueden clasificar.
5. Inyección en la cavidad de la médula ósea
- Ratones machos KM seleccionados de 8 a 9 semanas de edad, con un peso aproximado de 30 g. Anestesiarlos con una inyección intraperitoneal de 150 μL de pentobarbital sódico al 1% (paso 2.1). Confirme la profundidad de la anestesia mediante un pellizco en los dedos de los pies.
- Fije el ratón anestesiado en posición supina en la mesa de operaciones y fije la extremidad delantera del ratón con cinta adhesiva.
- Desinfecte la tibia posterior de los ratones con un hisopo con alcohol. Llene el plásmido que se va a inyectar en una jeringa de 1 ml con una aguja de 26 G. Retire las burbujas de aire y prepárese para la inyección.
- Toque la espinilla del mouse y sostenga la espinilla del mouse con los dedos. Determine la posición de la aguja de inyección para que la diáfisis tibial penetre desde la meseta tibial en la articulación de la rodilla del ratón. Gire la aguja de inyección paralela a la tibia, apuñale la cavidad de la médula ósea e inyecte lentamente (aproximadamente 3 s) para minimizar el daño a los ratones.
- Después de la inyección, extraiga lentamente la aguja (aproximadamente 3 s) e inmediatamente use un hisopo con alcohol para detener el sangrado y desinfectar el sitio de la punción.
- Coloque suavemente a los ratones en un ambiente cálido (20-26 °C), espere a que los ratones se recuperen de la anestesia y observe su recuperación.
6. Secuenciación de Sanger
- Después de 7 días de la inyección, recoja células de médula ósea de ratón como se describe en el paso 2 y aísle el ADN genómico mediante un kit de extracción. Recoja las células centrifugando a 2.000 x g durante 1 min. Deseche el sobrenadante, agregue 200 μL de solución de GA y vibre con un mezclador de vórtice. Añadir 20 μL de proteinasa K y 200 μL de GB, mezclar y colocar en un baño de agua a 70 °C durante 10 min.
- Después de la incubación, agregue 200 μL de etanol anhidro, agite durante 15 s y luego coloque el líquido mezclado en la columna de adsorción de soporte. Centrifugar a 2.500 x g durante 1 min, desechar el líquido residual en el fondo del tubo y añadir 500 μL de solución GD. Centrifugar y desechar el líquido residual, lavar el pellet 2 veces con PW y finalmente secarlo, eluyéndolo con 40 μL de agua pura.
NOTA: El PW debe agregarse con etanol anhidro de acuerdo con las instrucciones. - Amplifique el gen diana CaMKIIδ por PCR con la secuencia de cebadores directa gctaaggtgataaatgtggcact y la secuencia de cebadores inversa de ctagtgtgcgggccagattc. Prepare la reacción de PCR añadiendo 25 μL de tampón 2x, 1 μL de dNTP Mix, 2 μL de cebador directo, 2 μL de cebador inverso, 1 μL de ADN polimerasa, 2 μL de ADN molde y 17 μL de ddH2O. Ejecute el siguiente programa de PCR: 95 °C durante 3 min, 95 °C durante 15 s, 53,7 °C durante 30 s, 72 °C durante 60 s, 12 °C para la conservación del calor.
- Después de la PCR, prepare un gel de agarosa al 1% y realice una electroforesis en gel de agarosa a 110 V durante 35 min (paso 1.5, paso 1.6). Corte el área del gel objetivo después de la electroforesis y envíe la secuenciación de Sanger (Figura 4).
Resultados
Los plásmidos in vitro se inyectaron en ratones mediante inyección intraósea (Figura 1A). El tamaño medio de partícula de las nanopartículas fue de unos 202,9 nm, el potencial fue de 2,77 mV y el PDI fue de 0,22 (Figura 1B). La Figura 1C muestra la forma de la superficie de las nanopartículas observadas como esféricas por microscopía electrónica. La Figura 1D muestra el mapa de plásmidos del vector ABE y el mapa de plásmidos del vector de ARNg.
La transducción de plásmidos se observó bajo un microscopio de fluorescencia. La figura 2A muestra las células normales de la médula ósea. Después de 7 días de transferencia de plásmido al ratón, las células de la médula ósea del ratón se observaron bajo un microscopio de fluorescencia. En comparación con el grupo control, la eficiencia de la transfección del grupo de transfección de quitosano mejoró significativamente (Figura 2B). Esto indica que el quitosano como material de inclusión puede mejorar la eficiencia del plásmido de transducción in vitro.
Después de 7 días de la entrega en los ratones, el plásmido se expresó completamente. Se extrajeron células de médula ósea de ratones y se detectó la eficiencia de transfección de las células de médula ósea mediante citometría de flujo. Los resultados mostraron que la eficiencia de transfección de las células transfectadas directamente con plásmido ABE fue del 0,92% ± del 0,02%. La eficiencia de transfección de las células de médula ósea transfectadas con 2 μg de ABE recubiertas con quitosano fue del 40,80% ± del 4,31%, la de las células de médula ósea transfectadas con 3 μg de ABE recubiertas con quitosano fue del 47,20% ± del 5,37%, la de las células de médula ósea transfectadas con 4 μg de ABE recubiertas con quitosano fue del 51,20% ± del 2,02%, y la de las células de médula ósea transfectadas con 5 μg de ABE recubiertas con quitosano fue del 48,2% ± del 7,39% (Figura 3B). La eficiencia del grupo de transfección de quitosano fue mayor que la del grupo control (Figura 3A). Los resultados de la citometría de flujo mostraron que el plásmido transducido podía expresarse de manera estable y eficiente en ratones utilizando el método descrito en este artículo. Desde la perspectiva de la eficiencia de expresión integral y el ahorro de material, se podría seleccionar el esquema de transducción única de plásmido de 3 μg.
Extrajeron ADN genómico de células de médula ósea y amplificamos el gen diana mediante PCR. Después de la secuenciación de Sanger, la eficiencia de edición in vivo en A1 fue del 14,27% ± 0,35%, y la de A2 fue del 10,69% ± 0,30% (Figura 4).

Figura 1: Diagrama de flujo de inyección ósea, caracterización de nanopartículas y mapa vectorial de plásmidos. (A) Procedimiento para inyección intraósea. (B) Tamaño de partícula, potencial Zeta y PDI de nanopartículas formadas por plásmidos ABE incrustados con quitosano. (C) Imagen microscópica electrónica de nanopartículas. (D) El lado izquierdo muestra el mapa del plásmido vectorial ABE, y el lado derecho muestra el mapa del plásmido vectorial de ARNg. Haga clic aquí para ver una versión más grande de esta figura.
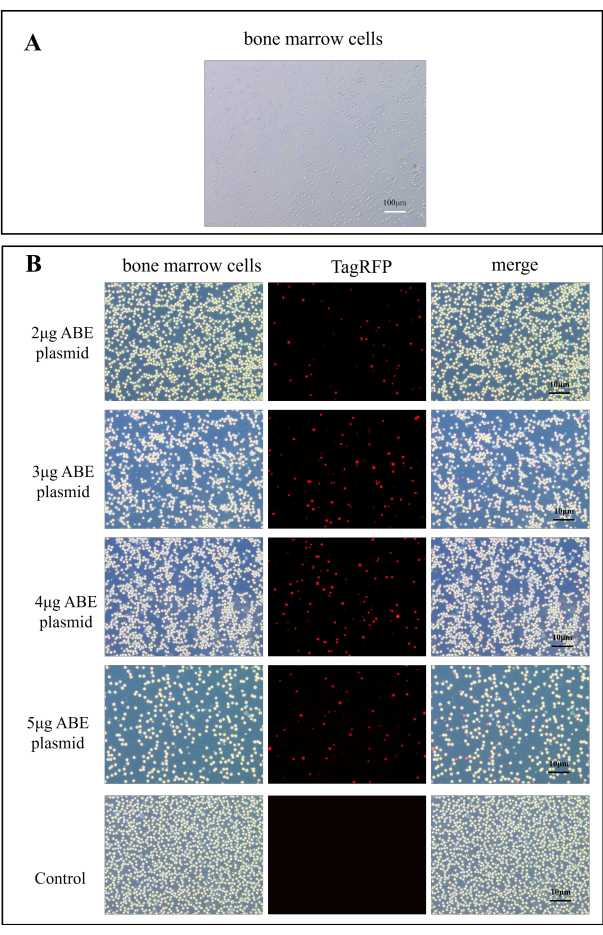
Figura 2: Imágenes de fluorescencia para la eficiencia de la transfección de plásmidos ABE. (A) Imagen A100x de células de médula ósea. (B) Los mapas de fluorescencia de las células de la médula ósea en el grupo de transfección de quitosano se obtuvieron 7 días después de la transfección de 2 μg, 3 μg, 4 μg y 5 μg de plásmido ABE, en comparación con los del grupo de control después de la inyección directa de plásmido ABE. Haga clic aquí para ver una versión más grande de esta figura.
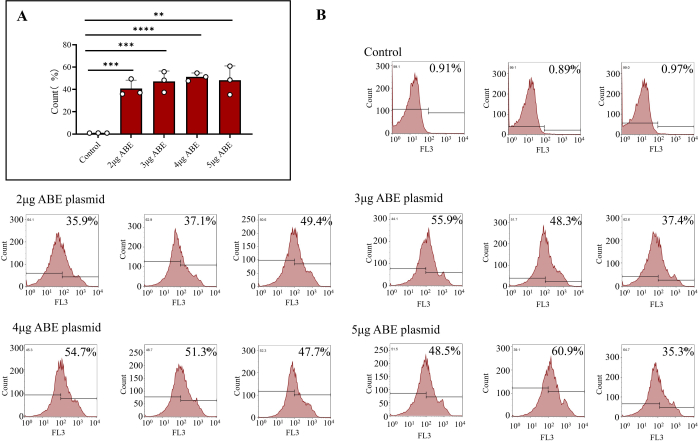
Figura 3: Resultados de la citometría de flujo de células de médula ósea transfectadas. (A) Células de médula ósea tratadas con citometría de flujo transfectadas con plásmidos ABE de 2 μg, 3 μg, 4 μg y 5 μg (grupo de transfección de quitosano) e inyectadas directamente (grupo control). Los resultados mostraron que la eficiencia de transfección de las células de médula ósea inyectadas directamente con plásmidos ABE fue pobre, mientras que la eficiencia de transfección y el efecto de transfección de los otros cuatro grupos fueron mayores. (B) Los resultados de la citometría de flujo incluyen la eficiencia de transfección del grupo de control y diferentes proporciones de plásmido ABE. Haga clic aquí para ver una versión más grande de esta figura.
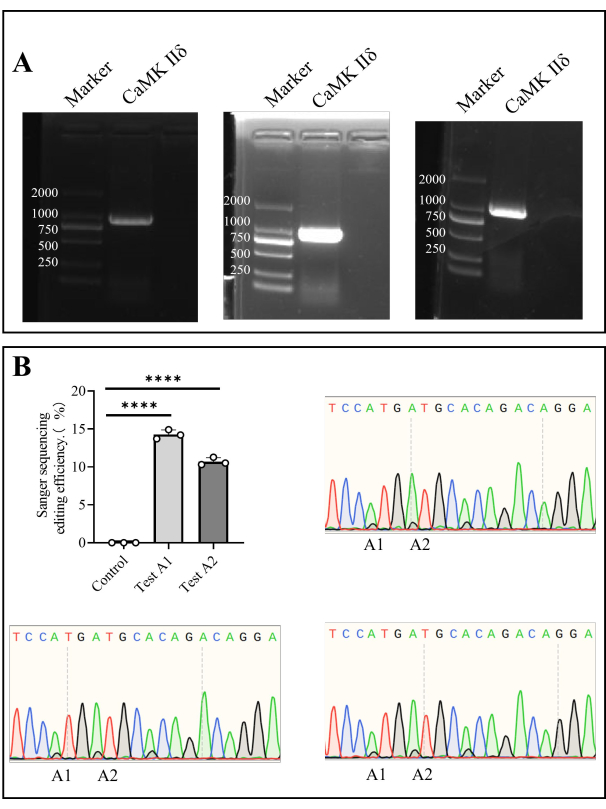
Figura 4: Secuenciación Sanger de Camk II δ eficiencia de edición en ratones. (A) Detección por electroforesis en gel de agarosa de Camk II δ. (B) El gráfico de barras muestra el análisis de resultados de secuenciación de Sanger de la eficiencia de edición de Camk II δ en ratones, y los diagramas de secuenciación de Sanger muestran los resultados de secuenciación. Haga clic aquí para ver una versión más grande de esta figura.
| Reactivos | Concentración utilizada |
| Ácido etilendiaminotetraacético | 1.142 mL |
| Ácido acético glacial | 2 mL |
| Tris | 4,84 g |
Tabla 1: Reactivos para la preparación de tampón 1X TAE
| Reactivo | Volumen |
| Agar-agar | 7,5 g |
| Amperio | Añadir 500 μL cuando esté esterilizado y enfriado a 70 °C |
| ddH2O | Hasta 500 μL |
| Cloruro de sodio | 5 g |
| Triptona | 5 g |
| Extracto de levadura | 2,5 g |
Tabla 2: Reactivos para la preparación de medio sólido LB.
| Reactivo | Volumen |
| ddH2O | Hasta 1 L |
| peptona | 10 g |
| Cloruro de sodio | 10 g |
| Extracto de levadura | 5 g |
Tabla 3: Reactivos para la preparación de medio líquido LB.
| Reactivo | Volumen |
| DMEM | 450 mL |
| Suero fetal bovino | 5 mL |
| MEM | 5 mL |
| Penicilina/Estreptomicina | 5 mL |
| β-mercaptoetanol | 500 μL |
Tabla 4: Reactivos para la preparación de medios DMEM que contienen suero.
Discusión
Para la investigación biomédica, el desafío de la administración de plásmidos exógenos a los animales implica mejorar la eficiencia de la administración y la expresión génica y, al mismo tiempo, minimizar el daño a los animales para lograr el efecto terapéutico deseado15,16. Presentamos un método novedoso de inyección intraósea de plásmidos ABE mediado por quitosano en las células de la médula ósea de ratones receptores. Esta estrategia mejora la eficiencia de la administración de plásmidos y la expresión génica.
En primer lugar, en este estudio, el quitosano, ofrecido como un sistema de vector no viral, se utilizó como método para la administración de plásmidos. Dado que se ha demostrado que la eficiencia de transfección del método convencional es relativamente baja, la introducción de plásmidos para la expresión génica a largo plazo puede verse afectada por el sistema inmune17,18. El quitosano, un polisacárido natural, es conocido por su excelente biocompatibilidad, capacidad de encapsulación de plásmidos y baja toxicidad. El uso del quitosano como vehículo de administración ayuda a prevenir posibles efectos relacionados con el sistema inmunitario durante la expresión a largo plazo de plásmidos exógenos, al tiempo que mejora la eficiencia de la transferencia de plásmidos 19,20,21. Para futuras investigaciones, reconocemos la importancia de evaluar la toxicidad y los posibles efectos en la médula ósea de las nanopartículas de quitosano cuando se administran por inyección intraósea. Planeamos incorporar estos aspectos en nuestro futuro diseño experimental, incluida la realización de evaluaciones exhaustivas de toxicidad y análisis histopatológicos. Esto implicará evaluar varios parámetros, como la viabilidad celular, las respuestas inflamatorias y la morfología de la médula ósea en diferentes puntos temporales y niveles de dosis. Además, consideraremos el uso de técnicas avanzadas de imagen y métodos de biología molecular para obtener una comprensión más profunda del perfil de seguridad y los posibles impactos en la salud de la médula ósea después del tratamiento con nanopartículas de quitosano.
En segundo lugar, se selecciona ABE como un editor de genes que puede convertir nucleótidos de adenina en nucleótidos de guanina sin introducir roturas de ADN de doble cadena. En comparación con CRISPR/Cas9, ABE ofrece una mayor precisión al corregir directamente una sola base sin inducir roturas de doble hebra, lo que reduce los efectos fuera del objetivo y el daño potencial. Sin embargo, a la hora de diseñar el ARNg diana, se requiere una cuidadosa consideración para evitar la unión inespecífica y reducir los efectos fuera de la diana22. Hemos implementado medidas para reducir el riesgo, pero la evitación completa sigue siendo un desafío y necesita más investigación.
En tercer lugar, para la administración in vivo, desarrollamos una estrategia de inyección intraósea. La inyección intraósea es un método seguro y eficaz, tal como ha sido reportado en la revisión del sistema por Betzler et al.23. El gen diana de este estudio es el Camk II δ, que desempeña un papel clave en la regulación de la diferenciación de los osteoclastos13,14. En comparación con el método convencional de inyección de la vena de cola, la inyección intraósea puede administrar el plásmido Camk II δ dirigido directamente y de manera eficiente a los osteoclastos, logrando así una alta eficiencia de edición in vivo del 14,27% en el locus A1, mientras que del 10,69% en el locus A2 en ratones receptores.
Además, hay que tener en cuenta que la inyección intraósea es una operación que requiere una atención especial. Entonces, antes del experimento, usamos tinte azul como práctica para monitorear si la inyección se inyecta correctamente en la cavidad de la médula ósea de los ratones. Además, debido a la posibilidad de que los ratones receptores sufran el daño causado por la inyección intraósea, se recomienda elegir ratones que pesen unos 30 g. Enfatizamos la importancia de varios pasos clave en el procedimiento de inyección intraósea: evitar administrar una cantidad excesiva de anestésico, asegurar una desinfección completa tanto antes como después de la inyección para prevenir infecciones y rotar la aguja para evitar obstrucciones causadas por la médula ósea. Si se bloquea, absténgase de forzar el líquido hacia la cavidad de la médula ósea del ratón, lo que puede provocar la muerte del ratón. El plásmido debe inyectarse lentamente, y es crucial monitorear la condición de los ratones después de la inyección para asegurarse de que no ocurran muertes debido a una manipulación inadecuada. Aunque especificamos que los ratones pesan 30 g para una inyección intraósea exitosa. En los entornos clínicos, se debe tener en cuenta la variabilidad específica del paciente, y nuestros estudios futuros, como la optimización de las técnicas de inyección para diferentes rangos de peso y la exploración de métodos de administración alternativos, están actualmente en curso.
Este método puede ser ampliamente utilizado en la edición de células derivadas de la médula ósea, incluidos osteoblastos, células eritroides, linfocitos, células hematopoyéticas, etc. La alta precisión y eficiencia del método permite a los investigadores utilizarlo no solo para enfermedades causadas por una función anormal de los osteoclastos, sino también en el campo de la terapia génica para otros trastornos sanguíneos. Por ejemplo, las enfermedades comunes de la sangre también pueden estar bien alineadas con nuestros métodos de tratamiento. Las enfermedades de la sangre causadas por mutaciones de una sola base24 se pueden tratar con editores de genes ABE, y la inyección intraósea también puede administrar plásmidos directamente a las células madre hematopoyéticas, logrando así mejores efectos terapéuticos. Sin embargo, el método presenta limitaciones en su aplicación para los procesos de edición y entrega de tipos celulares de origen no médula ósea, como las células musculares y adiposas.
Divulgaciones
Los autores declaran no tener intereses contrapuestos.
Agradecimientos
Este trabajo fue financiado por la Fundación de Ciencias Naturales de la Provincia de Anhui (2208085MC74, 2208085MC51) y la Fundación de Investigación Científica del Departamento de Educación de la Provincia de Anhui, China (KJ2021A0055).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 ml PCR Tubes, Flat cap | LABSELECT | PT-02-C | |
| 1 mL syringe | Anhui Jiangnan medical equipment Co., LTD | / | |
| 1% Pentobarbital sodium | / | / | |
| 1.5 ml Microcentrifuge Tubes | LABSELECT | MCT-001-150 | |
| 10 × DNA Loading Buffer | Vazyme | P022-01 | |
| 10X T4 DNA Ligase Buffer 1 ml | TaKaRa Biotechnology(Dalian)Co.,LTD | 2011A | |
| 1250 μl Pipette Tip 102.1mm | LABSELECT | T-001-1250 | |
| 200 μl Pipette Tips 50.55mm | LABSELECT | T-001-200 | |
| 2x Phanta Max Buffera | Vazyme | P505-d1 | |
| 4 ? centrifuge | Thermo Fisher | 75002425 | |
| 50 ml Centrifuge tube | LABSELECT | T-012-50 | |
| 6-well Cell Culture Plates | LABSELECT | 11110 | |
| Agar | Sangon Biotech | A505255-0250 | |
| Amp | Abiowell | / | |
| Chitosan | Sangon Biotech | A600614-0500 | |
| Constant temperature culture shaker | Shanghai Zhicheng Analytical Instrument Manufacturing Co., LTD | ZWY-200D | |
| Countess Automated Cell Counter | Thermo Fisher Scientific | Countess II/II FL | |
| Countess Cell Counting Chamber Slides and Holder, disposable | Thermo Fisher Scientific | C10228 | |
| CutSmart Buffer | New England Biolabs | B7204SVIAL | |
| DH5α | General Biosystems | CS01010 | |
| DMEM | gibco | C11995500BT | |
| dNTP Mix | Vazyme | P505-d1 | |
| Esp3I enzyme | NEBiolabs | R0734S | |
| Ethylenediaminetetraacetic acid | VETEC (sigma-aldrich) | V900106 | |
| Fetal bovine serum | OriCell | FBSAD-01011-500 | |
| Flow cytometer | BD FACSCalibur | 342975 | |
| Flow tube | Beyotime Biotechnology | FFC005-1bag | |
| Fluorescence microscope | Leica | 427019 | |
| gel maxi purification kit | TIANGEN | DP210 | |
| Genomic DNA extraction kit | TIANGEN | DP304 | |
| Glacial acetic acid | China National Pharmaceutical Group Corporation | 10000218 | |
| GoldBand DL2,000 DNA Marker | YESEN | 10501ES60 | |
| Ice machine | shanghaizhengqiao | BNS-30 | laboratory reserved |
| ImunoSep Buffer | Precision Biomedicals Co.,LTD | 604050 | |
| Megafuge 8 Small Benchtop Centrifuge Series | Thermo Fisher Scientific | 75004250 | |
| MEM | Life Technology | 11140050 | |
| NanoDrop 2000 | Thermo Fisher Scientific, USA | ||
| NaOH | SIGAM | S5881-500G | |
| PBS | XiGene | XG3650 | |
| PCMV-SPRY-ABE8E vector | / | / | |
| pcr amplification apparatus | Thermo Fisher | AKC96300441 | |
| Penicillin/Streptomycin | Solarbio | P1400 | |
| peptone | Sangon Biotech | A505247-0500 | |
| Phanta Max Super-Fidelity DNA Polymerase | Vazyme | P505-d1 | |
| Red cell lysate | Beyotime | C3702 | |
| Sodium chloride | China National Pharmaceutical Group Corporation | 10019318 | |
| Sodium sulfate | aladdin | S433911 | |
| T-001-10 10μl Pipette Tips 31.65mm | LABSELECT | T-001-10 | |
| T4 DNA Ligase | TaKaRa Biotechnology(Dalian)Co.,LTD | 2011A | |
| Tris | BioFroxx | 1115KG001 | |
| tryptone | Sangon Biotech | A505250-0500 | |
| vortex mixer | sigma | Z258423 | |
| Water bath | shanghaiyiheng | DK-80 | |
| YeaRed Nucleic Acid Gel Stain | YESEN | 10203ES76 | |
| Yeast extract | BBI | A610961-0500 | |
| Zetasizer Nano | Malvern Panalytical | Zetasizer Nano ZS | |
| β-mercaptoethanol | Sigma | 444203 |
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados