Method Article
Extracción de ADN basada en matrices para la secuenciación dirigida de próxima generación en muestras de esputo descontaminadas
* Estos autores han contribuido por igual
En este artículo
Resumen
Presentamos un método optimizado para la extracción de ADN a partir de esputo descontaminado utilizando un método de extracción basado en matriz combinado con una purificación magnética de perlas para la secuenciación de próxima generación dirigida posterior de Mycobacterium tuberculosis.
Resumen
En la actualidad, se reconoce que la secuenciación de nueva generación (NGS) es una herramienta poderosa para el diagnóstico oportuno y preciso de la tuberculosis resistente a los medicamentos (TB-DR). La NGS dirigida (tNGS) ofrece un enfoque simplificado al centrarse en genes específicos asociados con la resistencia a los medicamentos, evitando la necesidad de métodos tradicionales basados en cultivos con plazos de respuesta que van de semanas a meses. La Organización Mundial de la Salud (OMS) ha recomendado la tNGS como una estrategia valiosa para mejorar el diagnóstico de la tuberculosis (TB) con el fin de guiar el tratamiento y mejorar los resultados del tratamiento, especialmente en entornos con recursos limitados. Entre los ensayos de tNGS recomendados por la OMS, hemos seleccionado un método que proporciona pruebas rápidas y completas de susceptibilidad a los fármacos, determinación del linaje y tipificación de cepas. Si bien existen métodos estandarizados de extracción de ADN, pueden llevar mucho tiempo y mano de obra. Para abordar este desafío, optimizamos un protocolo simplificado de extracción de ADN basado en matrices en combinación con la purificación magnética de perlas. Este método ofrece un enfoque rápido y eficiente para extraer ADN directamente de sedimentos de esputo descontaminados, lo que permite un rápido análisis de tNGS aguas abajo. Al agilizar el proceso de extracción de ADN a partir de sedimentos de esputo, este protocolo podría facilitar una adopción más amplia de la tNGS en entornos clínicos rutinarios, contribuyendo en última instancia a mejorar los resultados de los pacientes y contribuyendo a los esfuerzos mundiales de control de la tuberculosis.
Introducción
Se estima que 3,7 millones de personas con tuberculosis no fueron diagnosticadas ni tratadas en todo el mundo en 2023, lo que pone de manifiesto la importante amenaza que representa la tuberculosis para la salud mundial1. La OMS estima que aproximadamente 400.000 personas desarrollaron tuberculosis resistente a la rifampicina (TB-RR) o multirresistente (TB-MDR) en 20231. El diagnóstico y el tratamiento rápido de la tuberculosis y la tuberculosis reproductiva es esencial para alcanzar los hitos y objetivos de reducción de la incidencia y la mortalidad por tuberculosis1.
Confiar en los métodos de cultivo convencionales y en las pruebas de susceptibilidad fenotípica a los fármacos (pDST) retrasa la determinación del perfil de resistencia de los aislados clínicos y del tratamiento, con plazos de respuesta de 6 a 8 semanas, y requiere una compleja infraestructura de biocontención. Las pruebas diagnósticas rutinarias, en combinación con la NGS de la TB-DR, pueden proporcionar un perfil completo de resistencia a los medicamentos y mejorar la personalización de los regímenes de TB-MDR, al tiempo que reducen el tiempo hasta el tratamiento efectivo de semanas o meses a días 2,3,4.
En 2023 y 2024, la OMS recomendó el uso de la tNGS como una nueva clase de diagnóstico para determinar rápidamente la susceptibilidad a los fármacos antituberculosos de primera y segunda línea5. Esto lo convierte en una herramienta valiosa para guiar las decisiones de tratamiento sin la necesidad de cultivo de Mycobacterium tuberculosis (MTB) en laboratorios de nivel de bioseguridad 3 (BSL-3). Un enfoque de tNGS es una forma condensada de secuenciación que utiliza la reacción en cadena de la polimerasa (PCR) para amplificar las dianas génicas que confieren resistencia a los fármacos antes de la secuenciación. Entre las pruebas de tNGS recomendadas por la OMS, hemos seleccionado el ensayo Deeplex Myc-TB, que se ha notificado que cumple los criterios basados en clases para detectar resistencia a la rifampicina, la isoniazida, el etambutol, la pirazinamida, las fluoroquinolonas, la amikacina, la estreptomicina, el linezolid, la bedaquilina y la clofazimina. Utilizamos este ensayo para evaluar la idoneidad del ADN extraído utilizando este protocolo para la tNGS posterior.
Además, la OMS publicó la2ª edición del catálogo de mutaciones asociadas a la resistencia a fármacos en MTB, proporcionando una hoja de ruta para el uso de la secuenciación del genoma completo (WGS) y la tNGS para predecir la susceptibilidad a los fármacos y guiar el tratamiento6. Una reciente revisión sistemática y metaanálisis mostraron que la tNGS tenía una sensibilidad y especificidad del 94,1% y del 98,1%, respectivamente, para la detección de resistencia a los fármacos, basada en 23 dianas en varias regiones del genoma del MTB, en comparación con la pDST7.
Sin embargo, la implementación de estos métodos sigue siendo un desafío debido a la complejidad y los costos asociados específicamente con los flujos de trabajo, la infraestructura y el equipo requeridos. Un desafío crítico es aislar suficiente ADN micobacteriano de alta calidad directamente del sedimento del esputo descontaminado, un paso crucial para las aplicaciones posteriores de tNGS. Para hacer frente a esto, presentamos un método de extracción de ADN rápido y sencillo adaptado a la tNGS.
El método estandarizado de extracción de ADN para el ensayo MTB tNGS seleccionado incluye un protocolo manual y automatizado interno8. Aquí describimos un protocolo simplificado de extracción de ADN basado en matrices (Figura 1). El método hace uso de la matriz InstaGene (IGM), que se une a metales y proteínas, lo que permite la extracción de ácidos nucleicos de calidad directamente de los sedimentos de esputo descontaminados. Esta alternativa proporciona un tiempo de respuesta más rápido y un rendimiento de ADN suficiente para tNGS aguas abajo. Este protocolo supera las complejidades de los métodos manuales y automatizados, al tiempo que garantiza un tNGS de calidad para el diagnóstico rápido de variantes que confieren resistencia en el MTB. Con el creciente interés en el uso de tNGS dentro del campo de la micobacteriología, este protocolo podría facilitar su adopción en los flujos de trabajo de diagnóstico de rutina.

Figura 1: Representación esquemática de los métodos para extraer ADN micobacteriano de muestras de sedimentos de esputo descontaminados utilizando una suspensión de matriz. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Esta investigación ha sido aprobada por el Comité de Ética en Investigación: Seguridad Biológica y Ambiental (REC: BES) de la Universidad de Stellenbosch: BES-2024-25384 y el Comité de Ética en Investigación en Humanos: N21/09/093 y N09/11/296.
1. Preparación de muestras de sedimentos de esputo
NOTA: Los siguientes pasos deben realizarse en un laboratorio de nivel de bioseguridad tres (BSL3) antes de la extracción de ADN.
- Prepare una alícuota de la muestra de sedimento de esputo previamente descontaminada con N-acetil-L-cisteína (NALC) e hidróxido de sodio (NaOH) (NALC/NaOH) transfiriendo 500 μL-2 mL a un tubo de baja unión de 1,5 mL o 2 mL.
- Coloque los tubos que contienen muestras de sedimentos de esputo en posición vertical en una rejilla de tubos y coloque las muestras en un horno a 80 °C e incube durante 1 h para inactivar las micobacterias mediante un tratamiento térmico.
- Después de 1 hora, retire la gradilla de tubos con muestras, limpie las superficies de los tubos y colóquelos en un recipiente adecuado para su transporte a un laboratorio de bioseguridad de nivel dos (BSL2).
NOTA: Deje que las muestras se enfríen a temperatura ambiente antes de manipularlas o almacenarlas.
2. Extracción de ADN mediante la suspensión de la matriz y un homogeneizador de alta velocidad (Figura 2)
- Antes de comenzar, agregue tres perlas de vidrio de 2 mm de diámetro a tubos con tapón de rosca de 1,5 o 2 mL, con tapa suelta, y autoclave (idealmente al menos un día antes de la extracción del ADN).
- Retire la suspensión de matriz del refrigerador y colóquela en un agitador magnético (se incluye un imán en la botella de reactivo) para llevar el reactivo a temperatura ambiente y comenzar a mezclar el reactivo.
- Encienda el bloque calefactor y precaliente a 56 °C (si dispone de dos bloques calefactores, precaliente el segundo a 100 °C.
NOTA: Los siguientes pasos se pueden realizar en una mesa de laboratorio en una instalación BSL2: - Centrifugar la muestra de sedimento de esputo inactivado por calor a 16.800 × g (o a toda velocidad) durante 15 minutos. Con una pipeta de 20-200 μL, aspire y deseche suavemente el sobrenadante, asegurándose de que el pellet permanezca inalterado.
- Mezcle la suspensión de la matriz agitándola suavemente (no haga vórtice) y agregue 200 μL de la matriz bien mezclada para volver a suspender el pellet.
- Pipetear todo el volumen en un tubo de rosca de 1,5 ml que contenga tres perlas de vidrio preesterilizadas de 2 mm de diámetro.
- Coloque las muestras en un bloque calefactor e incube a 56 °C durante 15 min.
NOTA: Inmediatamente después de retirar la muestra del bloque calefactor, ajuste el bloque calefactor a 100 °C después del paso 2.7, si solo hay un bloque calefactor disponible. - Homogeneizar las muestras utilizando un vórtice durante 10 s para dispersar las células.
- Coloque las muestras en un bloque calefactor e incube a 100 °C durante 10 min.
- Homogeneizar las muestras utilizando un homogeneizador de alta velocidad con los siguientes parámetros: un ciclo de 60 s a 4,0 m/s.
- Centrifugar a 12.000 × g durante 5 min. Transfiera 130 μL del sobrenadante que contiene ADN a un tubo fresco de baja unión de 1,5 mL sin alterar el pellet (matriz con restos celulares). Deseche el primer tubo que contiene la matriz y los restos de la célula.
NOTA: PUNTO DE PARADA SEGURO. Si se detiene, congele las muestras a -20 °C hasta que estén listas para continuar con la purificación del ADN. - Proceda con la purificación del ADN utilizando perlas magnéticas.
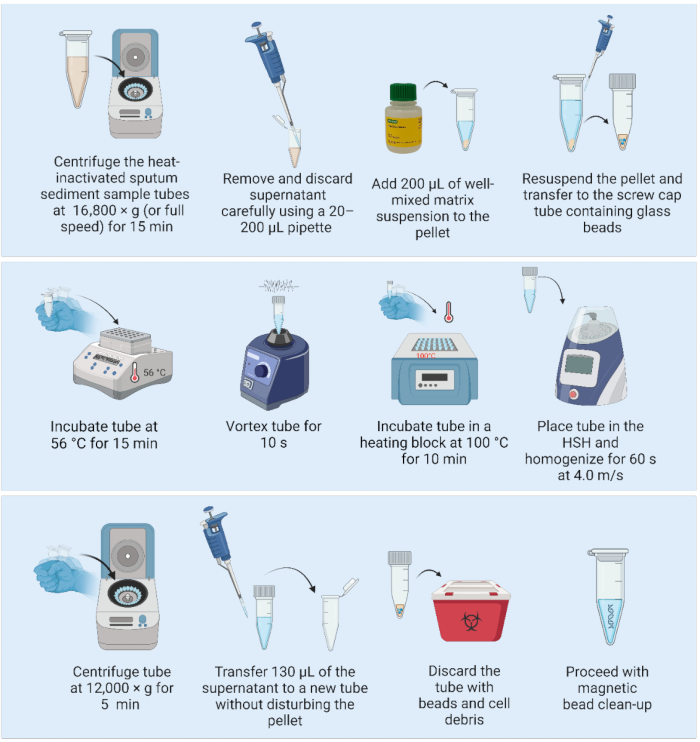
Figura 2: Método de extracción de ADN basado en matriz junto con un homogeneizador de alta velocidad. Abreviatura: HSH = homogeneizador de alta velocidad. Haga clic aquí para ver una versión más grande de esta figura.
3. Purificación del ADN mediante perlas magnéticas (Figura 3)
NOTA: Antes de continuar con la purificación del ADN, realice los pasos 3.1 y 3.2.
- Retire las cuentas magnéticas del refrigerador y coloque el recipiente a temperatura ambiente durante 30 minutos para que se equilibre. Alternativamente, vórtice el frasco de material de perlas magnéticas para resuspender todas las perlas y aspire una alícuota de 1.280 μL en un nuevo tubo (para la purificación de 21 muestras) y colóquelo a temperatura ambiente para equilibrarlo.
- Prepare un lote de 9 mL de etanol al 80% agregando 7,2 mL de alcohol etílico puro a 1,8 mL de agua filtrada y esterilizada.
NOTA: Los siguientes pasos se pueden realizar en una mesa de laboratorio en una instalación BSL2. - Agite minuciosamente el frasco de abalorios magnéticos o las alícuotas preparadas para asegurarse de que las perlas se resuspendan antes de su uso. Repita el vórtice después de cada 10 muestras para asegurar una suspensión homogénea.
- Agregue 1,2 veces el volumen (156 μL) de las perlas magnéticas al ADN extraído (130 μL).
NOTA: Para muestras con cantidades más grandes de ADN (cultivo), se puede procesar una alícuota más pequeña de la muestra (por ejemplo, 50 μL) y purificar con un volumen 1,2 veces mayor de perlas magnéticas. - Mezcle las muestras pipeteando 10 veces. Incubar las muestras durante 5 min a temperatura ambiente.
- Coloque las muestras en la gradilla magnética del tubo de 1,5 ml durante 3 minutos o hasta que el líquido se aclare. Aspire y deseche con cuidado el sobrenadante sin alterar las cuentas.
- Con los tubos en el imán, agregue 200 μL de etanol al 80%, asegurándose de que las perlas permanezcan inalteradas y permitan incubar durante 30 s.
- Aspire y deseche cuidadosamente el etanol sin alterar las perlas.
- Repita el paso 3.7 para un total de dos lavados.
- Después del segundo lavado, retire y deseche el etanol. Elimine cualquier residuo de etanol con una pipeta de 1-10 μL.
- Deja los tubos abiertos para que las cuentas se sequen al aire durante 10 minutos, o hasta que tengan una apariencia mate.
NOTA: No permita que las cuentas se sequen demasiado y se agrieten. - Una vez que las perlas tengan un aspecto mate, retire inmediatamente los tubos del imán y añada 50 μL de agua libre de nucleasas (NFW) directamente sobre las perlas de cada muestra. Después de agregar NFW a todos los tubos, mezcle cada muestra individual pipeteando 10 veces. Inspeccione el tubo en busca de cuentas que estén pegadas en el interior del tubo y repita el paso de mezcla si es necesario. Incubar las muestras durante 5 min a temperatura ambiente.
NOTA: La incubación se puede aumentar de 5 minutos a 10 minutos, pero no disminuya el tiempo de incubación a menos de 5 minutos. - Vuelva a colocar los tubos en la rejilla magnética durante 3 minutos o hasta que el líquido esté claro. Con los tubos en la rejilla magnética, transfiera el sobrenadante que contiene ADN a un tubo claramente marcado, estéril y de baja unión. Asegúrese de que las cuentas no se transfieran al sobrenadante.
NOTA: Si se ven perlas en la punta de la pipeta durante la transferencia, pipetee el sobrenadante de nuevo en el tubo de la rejilla magnética para separarlo durante 3 minutos más. No transfiera las cuentas al nuevo tubo.
PUNTO DE PARADA SEGURO. Si se detiene, congele las muestras a -20 °C hasta que estén listas para continuar con la cuantificación utilizando un método apropiado para NGS y la posterior amplificación por PCR utilizando un ensayo MTB tNGS.

Figura 3: Purificación y concentración de ADN mediante perlas magnéticas. Abreviaturas: NFW = agua libre de nucleasas; tNGS = secuenciación dirigida de próxima generación. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Descripción de la muestra
Un total de 165 muestras de sedimentos de esputo, positivas para bacilos acidorresistentes (AFB) en microscopía con una carga bacteriana de al menos 1+, fueron recolectadas y procesadas de forma rutinaria por los Servicios Nacionales de Laboratorio de Salud (NHLS) Green Point, Ciudad del Cabo, Sudáfrica. El ADN se extrajo de muestras de sedimentos de esputo utilizando dos volúmenes diferentes [aproximadamente 2 mL (n = 102) y 500 μL (n = 63)]. Esta comparación se llevó a cabo para evaluar si el menor volumen de muestra de sedimento podría producir suficiente ADN para el tNGS aguas abajo.
La Figura 4 muestra una comparación de diagrama de caja de la concentración total de ADN (ng/μL) en diferentes grados de frotis de esputo (1+, 2+ y 3+) y volúmenes de sedimentos de esputo (2 mL y 500 μL). El rendimiento de ADN generalmente disminuye en general cuando se utilizan 500 μL de sedimento para la extracción, con variabilidad entre los grados de frotis. Por lo tanto, las concentraciones medias de ADN extraídas de muestras de sedimentos con un volumen de al menos 2 mL fueron en promedio más altas que la concentración de ADN extraída de sedimentos de 500 μL, estratificada por AFB de la muestra.

Figura 4: Impacto del volumen de sedimento de esputo en el rendimiento de ADN a través de los grados de frotis. Haga clic aquí para ver una versión más grande de esta figura.
La Figura 5 muestra diagramas de caja que ilustran el número promedio de lecturas de secuenciación (profundidad de cobertura promedio) de todos los objetivos para muestras de sedimentos de esputo con diferentes grados de frotis (1+, 2+ y 3+) procesados a partir de dos volúmenes de entrada diferentes (2 mL y 500 μL). Los valores atípicos se representan mediante puntos individuales y el eje Y se muestra en una escala logarítmica para adaptarse a la variación en la cobertura. El alto rango intercuartílico para muestras 3+ extraídas de los sedimentos de 500 μL sugiere una mayor variabilidad con mayores cargas bacterianas.
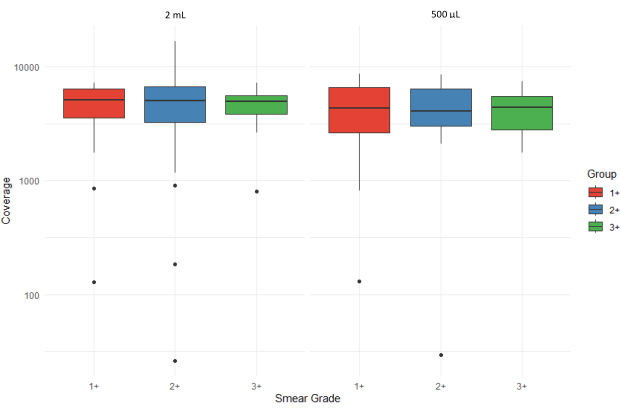
Figura 5: Comparación del número promedio de lecturas de secuenciación (profundidad de cobertura) para todos los objetivos en los grados de frotis y los volúmenes de entrada de la muestra. Haga clic aquí para ver una versión más grande de esta figura.
La Figura 6 muestra la evaluación de la aceptabilidad de los resultados de la secuenciación utilizando la canalización bioinformática de la aplicación web asociada. Las puntuaciones de calidad de la secuenciación se clasifican como +++ (muy aceptable), ++ (aceptable), + (marginalmente aceptable), - (inaceptable) y ND (no determinado). La ausencia de una barra en el gráfico indica que no se obtuvieron resultados para esa categoría específica. El frotis de grado 1+ muestra una mayor proporción de resultados inaceptables y ND en comparación con los grados de frotis 2+ y 3+ para el conjunto de muestras de volumen de entrada de 500 μL. Las muestras extraídas de 500 μL de sedimento de esputo tuvieron una profundidad de cobertura de secuenciación promedio de 4.316 en comparación con 4.810 para las muestras extraídas de 2 mL de sedimento de esputo. Esto sugiere que el ADN extraído del sedimento de esputo fue adecuado para realizar tNGS aguas abajo, independientemente del volumen de entrada.
Según la distribución de la aceptabilidad de los resultados de la secuenciación, las muestras con un grado de frotis de 3+ tuvieron la tasa más alta de éxito, y la mayoría de las secuencias de muestras se calificaron como ++ y +++ en comparación con las muestras de grado de frotis 1+ y 2+. Para las muestras de entrada de 500 μL, la proporción de muestras en las categorías - y + es relativamente mayor en todos los grados de frotis en comparación con las muestras de entrada de 2 mL. Esto sugiere que más muestras caen en las categorías de menor calidad, menos aceptables, para la entrada de 500 μL. Para las muestras de entrada de 2 mL, hay una mayor proporción de muestras en las categorías de mayor calidad, ++ y +++, particularmente para el frotis de grado 3+. Esto sugiere que las muestras con una entrada de 2 mL tienen más probabilidades de producir mejores puntuaciones de calidad de secuenciación en comparación con aquellas con una entrada de 500 μL.

Figura 6: Distribución de las puntuaciones de calidad por grado de frotis y volúmenes de entrada de muestra. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La combinación del método de extracción basado en matrices junto con un homogeneizador de alta velocidad fue publicada originalmente por Shea et al.9. Este método fue desarrollado y optimizado para WGS de ADN extraído de cultivos MTBC 9,10. Optimizamos este método para su uso con un ensayo MTB tNGS. La extracción y purificación del ADN de 21 muestras tuvo un tiempo de respuesta combinado de 4 h y 40 min, incluidas las etapas de incubación y centrifugación. La fiabilidad del protocolo descrito, junto con una limpieza de perlas para la extracción de ADN de muestras con grados de frotis que varían de 1+ a 3+, se demuestra en función del rendimiento medio (Figura 4), la profundidad general de la cobertura de todos los objetivos combinados (Figura 5) y la puntuación de aceptabilidad de los resultados de secuenciación determinada por la aplicación web asociada (Figura 6).
Un estudio reciente no encontró una relación consistente entre el grado de frotis y la concentración de ADN o la profundidad de lectura de secuenciación. Los autores sugirieron que la variabilidad en el procesamiento de muestras y los pasos de limpieza puede afectar el rendimiento en muestras de alto grado de frotis, particularmente cuando los contaminantes no se eliminan de manera eficiente. Además, la secuenciación a partir de sedimentos de esputo sigue siendo un desafío debido a la complejidad del grupo de ADN, donde el ADN no objetivo puede competir con el ADN MTB11. Si bien no observamos ninguna correlación clara entre la concentración de ADN de entrada y la profundidad de secuenciación, es importante tener en cuenta que la entrada de ADN antes de la PCR objetivo no debe exceder los 100 ng, según las instrucciones del fabricante, ya que esto podría conducir a la inhibición de la PCR. Además, las muestras de sedimentos de esputo 2+ y 3+ de mayor carga bacteriana podrían contener más contaminantes, como ADN humano, restos celulares y otros inhibidores, que pueden interferir con la amplificación de la PCR y la preparación de la biblioteca.
La etapa de purificación también funciona como una etapa de concentración, mejorando el rendimiento de ADN micobacteriano antes de la tNGS. Este método es eficaz para procesar muestras con una carga bacteriana baja y ha demostrado un rendimiento fiable con volúmenes tan pequeños como 500 μL, lo que lo hace adecuado para entornos de rutina en los que se dispone de un volumen de muestra limitado. Su aplicación puede reducir la necesidad de visitas de seguimiento a las clínicas, minimizando así el riesgo de pérdida de pacientes durante el seguimiento. Además, el método es sencillo y no requiere conocimientos avanzados de laboratorio, lo que respalda aún más su integración en entornos con recursos limitados.
Sin embargo, hay que tener en cuenta algunos puntos sobre el protocolo. Las muestras no deben congelarse ni refrigerarse inmediatamente después de la incubación a 80 °C. El enfriamiento rápido inmediatamente después del tratamiento térmico puede hacer que se forme condensación en las superficies internas del tubo, lo que lleva a la dilución de la muestra y puede afectar la extracción de ADN posterior. Además, el enfriamiento repentino puede aumentar el riesgo de degradación de la muestra al promover la fragmentación de ácidos nucleicos o la actividad enzimática que puede no haberse inactivado completamente durante el tratamiento térmico. Estas consideraciones son particularmente relevantes cuando se procesan muestras de esputo para la detección de MTB. De acuerdo con las recomendaciones de la OMS para la tinción de Ziehl-Neelsen, un grado de frotis de 1+ corresponde a 10-99 AFB por cada 100 campos de inmersión en aceite, 2+ indica 1-10 AFB por campo en al menos 50 campos, y 3+ representa más de 10 AFB por campo en al menos 20 campos12.
Una limitación de este método es su falta de automatización debido al requisito de homogeneización manual mediante un homogeneizador de alta velocidad o un dispositivo de batido de cordones. Para abordar esto, los esfuerzos en curso se centran en refinar el protocolo probando tiempos de incubación más cortos, temperaturas de incubación más bajas y enfoques alternativos a la homogeneización a alta velocidad.
Por lo tanto, este método de extracción de ADN basado en matriz, combinado con un homogeneizador de alta velocidad, es una técnica rápida de extracción de ADN que emplea calor y golpes de perlas para liberar ADN genómico y, al mismo tiempo, eliminar los inhibidores de PCR, como metales y proteínas.
Divulgaciones
El autor T. R. recibe apoyo financiero de FIND a través de un contrato de servicio con UC San Diego. El autor T. R. recibió una subvención de los NIH para desarrollar y evaluar una solución de tNGS para la tuberculosis resistente a los medicamentos (R01AI176401). El autor T. R. es cofundador, miembro de la junta directiva y accionista no remunerado de Verus Diagnostics Inc.
Agradecimientos
Los autores desean agradecer al equipo del laboratorio de tuberculosis del NHLS - Green Point, Sudáfrica, por brindar asistencia técnica. Este trabajo contó con el apoyo de los Institutos Nacionales de Salud (NIH) y la Fundación para Nuevos Diagnósticos Innovadores (FIND), proyecto TS ELiOT Grant ID: R01AI153213, Unitaid Grant ID: 2019-32-FIND MDR. Las figuras se crearon utilizando BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
Referencias
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados