Method Article
Матричная экстракция ДНК для целенаправленного секвенирования нового поколения на обеззараженных образцах мокроты
* Эти авторы внесли равный вклад
В этой статье
Резюме
Мы представляем оптимизированный метод экстракции ДНК из обеззараженной мокроты с использованием матричного метода экстракции в сочетании с очисткой магнитными шариками для последующего целевого секвенирования Mycobacterium tuberculosis следующего поколения.
Аннотация
Секвенирование нового поколения (NGS) в настоящее время признано мощным инструментом для своевременной и точной диагностики лекарственно-устойчивого туберкулеза (ЛУ-ТБ). Таргетная NGS (tNGS) предлагает оптимизированный подход, фокусируясь на конкретных генах, связанных с лекарственной устойчивостью, обходя необходимость в традиционных методах, основанных на культурах, со временем выполнения от нескольких недель до нескольких месяцев. Всемирная организация здравоохранения (ВОЗ) рекомендовала tNGS в качестве ценной стратегии для улучшения диагностики туберкулеза (ТБ) для руководства лечением и улучшения результатов лечения, особенно в условиях ограниченных ресурсов. Среди рекомендованных ВОЗ анализов tNGS мы выбрали метод, который обеспечивает быстрое и всестороннее тестирование на чувствительность к лекарственным препаратам, определение линии и типирование штамма. Несмотря на то, что стандартизированные методы экстракции ДНК доступны, они могут быть трудоемкими и трудоемкими. Чтобы решить эту проблему, мы оптимизировали упрощенный протокол экстракции ДНК на основе матрицы в сочетании с очисткой магнитными шариками. Этот метод обеспечивает быстрый и эффективный подход к извлечению ДНК непосредственно из обеззараженных отложений мокроты, что позволяет проводить быстрый анализ tNGS на последующих этапах. Оптимизируя процесс извлечения ДНК из осадка мокроты, этот протокол может способствовать более широкому внедрению tNGS в рутинных клинических условиях, что в конечном итоге способствует улучшению результатов лечения пациентов и способствует глобальным усилиям по борьбе с туберкулезом.
Введение
По оценкам, в 2023 г. во всем мире не диагностировались и не лечились 3,7 миллиона человек с туберкулезом, что подчеркивает значительную угрозу, которую туберкулез представляет для глобальногоздравоохранения1. По оценкам ВОЗ, в 2023 году примерно у 400 000 человек развился устойчивый к рифампицину (РР-ТБ) или туберкулез с множественной лекарственной устойчивостью (МЛУ-ТБ)1. Быстрая диагностика и лечение ТБ и ЛУ-ТБ имеют важнейшее значение для достижения контрольных показателей и целей по снижению заболеваемости ТБ и смертности от ТБ1.
Использование традиционных методов культивирования и фенотипического тестирования чувствительности к лекарственным препаратам (pDST) задерживает определение профиля резистентности клинических изолятов и лечение со временем обработки 6-8 недель и требует сложной инфраструктуры биосдерживания. Рутинные диагностические тесты в сочетании с NGS ЛУ-ТБ могут обеспечить комплексный профиль лекарственной устойчивости и улучшить персонализацию схем лечения ЛУ-ТБ, а также сократить время эффективного лечения с недель или месяцев до 2,3,4 дней.
В 2023 и 2024 годах ВОЗ рекомендовала использовать tNGS в качестве нового класса диагностики для быстрого определения чувствительности к противотуберкулезным препаратам первого и второгоряда5. Это делает его ценным инструментом для принятия решений о лечении без необходимости культивирования микобактерий туберкулеза (MTB) в лабораториях уровня биобезопасности 3 (BSL-3). Подход tNGS представляет собой конденсированную форму секвенирования, в которой используется полимеразная цепная реакция (ПЦР) для амплификации генов-мишеней, придающих устойчивость к лекарствам, перед секвенированием. Среди рекомендованных ВОЗ тестов tNGS мы выбрали тест Deeplex Myc-TB, который, как сообщается, соответствует критериям определения устойчивости к рифампицину, изониазиду, этамбутолу, пиразинамиду, фторхинолонам, амикацину, стрептомицину, линезолиду, бедаквилину и клофазимину. Мы используем этот анализ для оценки пригодности ДНК, экстрагированной с использованием этого протокола, для последующего tNGS.
Кроме того, ВОЗ опубликовала2-е издание каталога мутаций, связанных с лекарственной устойчивостью у МТБ, в котором представлена дорожная карта для использования полногеномного секвенирования (WGS) и tNGS для прогнозирования чувствительности к лекарствам и руководства лечением6. Недавний систематический обзор и метаанализ показали, что tNGS имеет чувствительность и специфичность 94,1% и 98,1% соответственно для выявления лекарственной устойчивости, основываясь на 23 мишенях в различных областях генома MTB, придающих устойчивость, по сравнению с pDST7.
Тем не менее, реализация этих методов остается сложной задачей из-за сложности и затрат, связанных с необходимыми рабочими процессами, инфраструктурой и оборудованием. Важнейшей задачей является выделение достаточного количества высококачественной ДНК микобактерий непосредственно из осадка обеззараженной мокроты, что является важнейшим этапом для последующих применений tNGS. Чтобы решить эту проблему, мы представляем быстрый и простой метод экстракции ДНК, адаптированный для tNGS.
Стандартизированный метод экстракции ДНК для выбранного анализа MTB tNGS включает в себя внутренний ручной и автоматизированный протокол8. Здесь мы опишем упрощенный, основанный на матрице протокол экстракции ДНК (рис. 1). В методе используется матрица InstaGene Matrix (IGM), которая связывает металлы и белки, позволяя извлекать качественные нуклеиновые кислоты непосредственно из обеззараженных отложений мокроты. Эта альтернатива обеспечивает более быстрое время выполнения заказа и достаточный выход ДНК для последующего tNGS. Этот протокол позволяет избежать сложностей ручных и автоматизированных методов, обеспечивая при этом качественную tNGS для быстрой диагностики вариантов, вызывающих устойчивость к MTB. В связи с растущим интересом к использованию tNGS в области микобактериологии, этот протокол может облегчить его внедрение в рутинные диагностические рабочие процессы.

Рисунок 1: Схематическое изображение методов извлечения ДНК микобактерий из обеззараженных образцов осадка мокроты с использованием матричной суспензии. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
протокол
Это исследование было одобрено Комитетом по этике научных исследований: Биологическая и экологическая безопасность (REC: BES) в Университете Стелленбоша: BES-2024-25384 и Комитетом по этике исследований человека: N21/09/093 и N09/11/296.
1. Подготовка проб осадка мокроты
ПРИМЕЧАНИЕ: Следующие этапы должны быть выполнены в лаборатории третьего уровня биобезопасности (BSL3) перед экстракцией ДНК.
- Приготовьте аликвоту образца осадка мокроты, предварительно обеззараженного с помощью N-ацетил-L-цистеина (NALC) и гидроксида натрия (NaOH) (NALC/NaOH), переложив 500 мкл-2 мл в пробирку объемом 1,5 мл или 2 мл с низким уровнем связывания.
- Поместите пробирки с образцами осадка мокроты вертикально в штатив для пробирок, поместите образцы в духовку при температуре 80 °C и инкубируйте в течение 1 часа, чтобы инактивировать микобактерии путем термической обработки.
- Через 1 ч снимите штатив с образцами, протрите поверхности пробирок и поместите их в соответствующий контейнер для транспортировки в лабораторию второго уровня биобезопасности (BSL2).
ПРИМЕЧАНИЕ: Дайте образцам остыть до комнатной температуры перед дальнейшей обработкой или хранением.
2. Экстракция ДНК с помощью матричной суспензии и высокоскоростного гомогенизатора (рис. 2)
- Перед началом добавьте три стеклянных шарика диаметром 2 мм в пробирки с завинчивающейся крышкой объемом 1,5 или 2 мл и проведите автоклав (в идеале не менее чем за сутки до экстракции ДНК).
- Достаньте матричную суспензию из холодильника и поместите ее на магнитную мешалку (магнит входит в комплект флакона с реагентом), чтобы довести реагент до комнатной температуры и начать перемешивание реагента.
- Включите нагревательный блок и разогрейте до 56 °C (если есть два нагревательных блока, разогрейте второй до 100 °C.
ПРИМЕЧАНИЕ: На лабораторном стенде в установке BSL2 можно выполнить следующие шаги: - Центрифугируйте инактивированный при нагревании образец осадка мокроты со скоростью 16 800 × g (или на полной скорости) в течение 15 минут. С помощью пипетки объемом 20-200 мкл аккуратно отсадите и удалите надосадочную жидкость, следя за тем, чтобы гранула оставалась нетронутой.
- Перемешайте матричную суспензию путем легкого встряхивания (не завихрите) и добавьте 200 мкл хорошо перемешанной матрицы для ресуспендирования гранул.
- Пипетируйте весь объем в пробирку с завинчивающейся крышкой объемом 1,5 мл, содержащую три предварительно стерилизованных стеклянных шарика диаметром 2 мм.
- Поместите образцы в нагревательный блок и инкубируйте при температуре 56 °C в течение 15 минут.
ПРИМЕЧАНИЕ: Сразу после извлечения образца из нагревательного блока установите температуру нагревательного блока на 100 °C после шага 2.7, если доступен только один нагревательный блок. - Гомогенизируйте образцы с помощью вихря в течение 10 с для диспергирования клеток.
- Поместите образцы в нагревательный блок и инкубируйте при 100 °C в течение 10 минут.
- Гомогенизировать образцы с помощью высокоскоростного гомогенизатора со следующими параметрами: один цикл 60 с со скоростью 4,0 м/с.
- Центрифуга при 12 000 × г в течение 5 мин. Перенесите 130 мкл ДНК-содержащей надосадочной жидкости в свежую пробирку с низким связыванием объемом 1,5 мл, не нарушая гранулу (матрицу с клеточным мусором). Выбросьте первую пробирку, содержащую матрикс и клеточный мусор.
ПРИМЕЧАНИЕ: БЕЗОПАСНАЯ ОСТАНОВКА. Если вы остановитесь, заморозьте образцы при температуре -20 °C до тех пор, пока не будете готовы продолжить очистку ДНК. - Приступайте к очищению ДНК с помощью магнитных шариков.
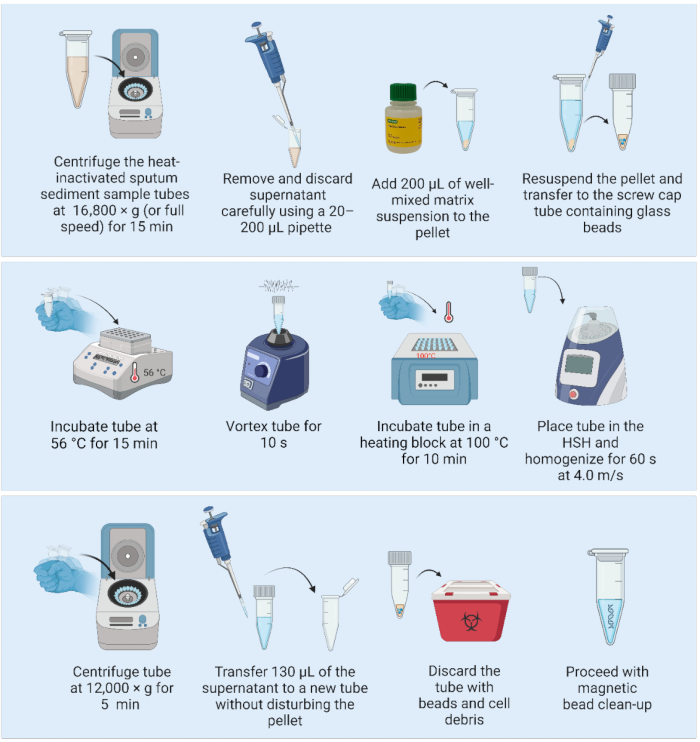
Рисунок 2: Метод экстракции ДНК на основе матрицы в сочетании с высокоскоростным гомогенизатором. Сокращение: HSH = высокоскоростной гомогенизатор. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
3. Очистка ДНК с помощью магнитных шариков (рис. 3)
ПРИМЕЧАНИЕ: Прежде чем приступить к очистке ДНК, выполните шаги 3.1 и 3.2.
- Достаньте магнитные шарики из холодильника и поставьте контейнер при комнатной температуре на 30 минут, чтобы уравновесить. В качестве альтернативы, сделайте бутылочку с магнитным шариком вихрем, чтобы снова суспендировать все шарики, и отасуньте аликвоту объемом 1280 мкл в новую пробирку (для очистки 21 образца) и поместите при комнатной температуре для балансировки.
- Приготовьте партию 80% этанола объемом 9 мл, добавив 7,2 мл чистого этилового спирта к 1,8 мл фильтрованной и стерилизованной воды.
ПРИМЕЧАНИЕ: Следующие шаги могут быть выполнены на лабораторном столе в установке BSL2. - Тщательно протрите магнитную бутылочку с бусинами или подготовленную аликвоту, чтобы убедиться, что бусины снова суспендированы перед использованием. Повторяйте вортексинг через каждые 10 образцов, чтобы обеспечить однородную суспензию.
- Добавьте в 1,2 раза больше объема (156 мкл) магнитных шариков к извлеченной ДНК (130 мкл).
ПРИМЕЧАНИЕ: Для образцов с большим количеством ДНК (культуры) можно обработать меньшую аликвоту образца (например, 50 мкл) и очистить с помощью магнитных гранул, в 1,2 раза превышающих объем. - Перемешайте образцы с помощью пипетирования 10 раз. Инкубируйте образцы в течение 5 минут при комнатной температуре.
- Поместите образцы на магнитный штатив для пробирок объемом 1,5 мл на 3 минуты или пока жидкость не станет прозрачной. Осторожно аспирируйте и выбросьте надосадочную жидкость, не потревожив бусины.
- С помощью трубок на магните добавьте 200 μл 80% этанола, следя за тем, чтобы шарики оставались нетронутыми, и дайте им инкубироваться в течение 30 секунд.
- Осторожно отсадите и выбросьте этанол, не потревожив шарики.
- Повторите шаг 3.7 в общей сложности два мытья.
- После второй промывки удалите и выбросьте этанол. Удалите остатки этанола с помощью пипетки объемом 1–10 мкл.
- Оставьте трубки открытыми, чтобы шарики высохли на воздухе в течение 10 минут или пока бусины не приобретут матовый вид.
ПРИМЕЧАНИЕ: Не допускайте пересыхания бусин и их растрескивания. - Как только шарики приобретут матовый вид, немедленно снимите трубки с магнита и добавьте 50 μL воды без нуклеаз (NFW) непосредственно на шарики в каждом образце. После добавления NFW во все пробирки перемешайте каждый отдельный образец с помощью пипетирования 10 раз. Осмотрите пробирку на наличие шариков, застрявших на внутренней стороне пробирки, и при необходимости повторите этап смешивания. Инкубируйте образцы в течение 5 минут при комнатной температуре.
ПРИМЕЧАНИЕ: Продолжительность инкубации можно увеличить с 5 до 10 минут, но не уменьшайте время инкубации менее чем до 5 минут. - Поместите пробирки обратно на магнитную решетку на 3 минуты или пока жидкость не станет прозрачной. С помощью пробирок на магнитном штативе перенесите содержащую ДНК надосадочную жидкость в четко маркированную, стерильную трубку с низким уровнем связывания. Следите за тем, чтобы бусины не переносились в надосадочную жидкость.
ПРИМЕЧАНИЕ: Если во время переноса на наконечнике пипетки видны шарики, введите надосадочную жидкость обратно в пробирку на магнитном штативе для разделения еще на 3 минуты. Не перекладывайте шарики в новую трубку.
БЕЗОПАСНАЯ ОСТАНОВКА. В случае остановки заморозьте образцы при температуре -20 °C до тех пор, пока они не будут готовы к количественному определению с использованием метода, соответствующего стандарту NGS, и последующей амплификации ПЦР с помощью анализа MTB tNGS.

Рисунок 3: Очистка и концентрация ДНК с помощью магнитных шариков. Сокращения: NFW = безнуклеазная вода; tNGS = целевое секвенирование нового поколения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Результаты
Описание примера
В общей сложности 165 образцов осадка мокроты, положительных на кислотоустойчивые бациллы (AFB) при микроскопии с бактериальной нагрузкой не менее 1+, были регулярно собраны и обработаны Национальной лабораторной службой здравоохранения (NHLS) в Грин-Пойнте, Кейптаун, Южная Африка. ДНК извлекали из образцов отложений мокроты с использованием двух разных объемов [приблизительно 2 мл (n = 102) и 500 μл (n = 63)]. Это сравнение было проведено для того, чтобы оценить, может ли меньший объем образца отложений дать достаточное количество ДНК для tNGS ниже по течению.
На рисунке 4 показано сравнение общей концентрации ДНК (нг/мкл) в мазке мокроты разных сортов (1+, 2+ и 3+) и объемах отложения мокроты (2 мл и 500 мкл). Выход ДНК обычно снижается при использовании 500 мкл осадка для экстракции, с вариабельностью в зависимости от качества мазка. Таким образом, средние концентрации ДНК, извлеченные из образцов отложений объемом не менее 2 мл, были в среднем выше, чем концентрация ДНК, извлеченная из 500 мкл отложений, стратифицированных по AFB образца.

Рисунок 4: Влияние объема осадка мокроты на выход ДНК в зависимости от сортов мазка. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
На рисунке 5 показаны ящичковые диаграммы, иллюстрирующие среднее количество прочтений секвенирования (средняя глубина покрытия) всех мишеней для образцов осадка мокроты с различным содержанием мазка (1+, 2+ и 3+), обработанных из двух разных входных объемов (2 мл и 500 мкл). Выбросы представлены отдельными точками, а ось Y отображается в логарифмической шкале, чтобы учесть изменения в покрытии. Высокий межквартильный диапазон для 3+ образцов, извлеченных из отложений объемом 500 μл, предполагает большую изменчивость при более высокой бактериальной нагрузке.
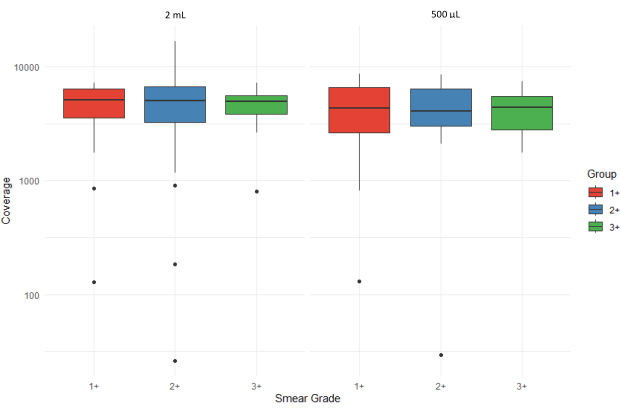
Рисунок 5: Сравнение среднего числа прочтений секвенирования (глубины покрытия) для всех мишеней по степеням мазка и объемам входных образцов. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
На рисунке 6 показана оценка приемлемости результатов секвенирования с использованием биоинформатического конвейера соответствующего веб-приложения. Оценки качества секвенирования подразделяются на следующие категории: +++ (высокоприемлемый), ++ (приемлемый), + (минимально приемлемый), - (неприемлемый) и ND (не определен). Отсутствие столбца на графике указывает на то, что по этой конкретной категории результаты не были получены. Мазок класса 1+ показывает более высокую долю неприемлемых результатов и ND по сравнению с мазком классов 2+ и 3+ для набора образцов входного объема 500 мкл. Пробы, извлеченные из 500 мкл осадка мокроты, имели среднюю глубину покрытия секвенирования 4 316 по сравнению с 4 810 для образцов, извлеченных из осадка мокроты объемом 2 мл. Это говорит о том, что ДНК, извлеченная из осадка мокроты, была достаточной для выполнения tNGS ниже по течению, независимо от входного объема.
Исходя из распределения приемлемости результатов секвенирования, образцы с оценкой мазка 3+ имели наибольший уровень успеха, при этом большинство последовательностей образцов были оценены как ++ и +++ по сравнению с образцами с классом мазка 1+ и 2+. Для входных образцов объемом 500 мкл доля образцов в категориях - и + относительно выше по всем сортам мазка по сравнению с входными образцами объемом 2 мкл. Это говорит о том, что большее количество образцов попадает в категории более низкого качества, менее приемлемых, для входного объема 500 μL. Для входных образцов объемом 2 мл наблюдается более высокая доля образцов в категориях более высокого качества, ++ и +++, особенно для мазка класса 3+. Это говорит о том, что образцы с входом 2 мл с большей вероятностью дадут лучшие показатели качества секвенирования по сравнению с образцами с входом 500 мкл.

Рисунок 6: Распределение оценок качества по степени мазка и объемам ввода образцов. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Обсуждение
Комбинация метода экстракции на основе матрицы в сочетании с высокоскоростным гомогенизатором была первоначально опубликована Shea et al.9. Этот метод был разработан и оптимизирован для WGS ДНК, выделенной из культур MTBC 9,10. Мы оптимизировали этот метод для использования с анализом MTB tNGS. Экстракция ДНК и очистка 21 образца имели общее время обработки 4 часа 40 минут, включая этапы инкубации и центрифугирования. Надежность описанного протокола в сочетании с очисткой гранул для извлечения ДНК из образцов с классом мазка от 1+ до 3+ демонстрируется на основе среднего выхода (рис. 4), общей глубины охвата всех мишеней в совокупности (рис. 5) и оценки приемлемости результата секвенирования, определенной соответствующим веб-приложением (рис. 6).
Недавнее исследование не обнаружило последовательной связи между сортностью мазка и концентрацией ДНК или глубиной чтения секвенирования. Авторы предположили, что вариативность в обработке образцов и этапах очистки может повлиять на производительность образцов с высоким содержанием мазка, особенно когда загрязнения не удаляются эффективно. Кроме того, секвенирование из осадка мокроты остается сложной задачей из-за сложности пула ДНК, где нецелевая ДНК может конкурировать с ДНК MTB11. Хотя мы не наблюдали какой-либо четкой корреляции между концентрацией входной ДНК и глубиной секвенирования, важно отметить, что ввод ДНК до целевой ПЦР не должен превышать 100 нг, в соответствии с инструкциями производителя, так как это может привести к ингибированию ПЦР. Кроме того, более высокая бактериальная нагрузка, образцы отложения мокроты 2+ и 3+ могут содержать больше загрязняющих веществ, таких как ДНК человека, клеточный мусор и другие ингибиторы, которые могут мешать амплификации ПЦР и подготовке библиотеки.
Стадия очистки также функционирует как стадия концентрации, увеличивая выход ДНК микобактерий до tNGS. Этот метод эффективен для обработки образцов с низкой бактериальной нагрузкой и продемонстрировал надежную работу при объемах до 500 мкл, что делает его пригодным для рутинных условий с ограниченным объемом образца. Его применение может снизить потребность в последующих визитах в клиники, тем самым сводя к минимуму риск потери пациента для последующего наблюдения. Кроме того, метод прост и не требует продвинутых лабораторных знаний, что еще больше способствует его интеграции в условиях ограниченных ресурсов.
Тем не менее, следует отметить некоторые моменты о протоколе. Образцы не следует замораживать или хранить в холодильнике сразу после инкубации при 80 °C. Быстрое охлаждение сразу после термической обработки может привести к образованию конденсата на внутренних поверхностях пробирки, что приведет к разбавлению образца и потенциально повлияет на последующую экстракцию ДНК. Кроме того, резкое охлаждение может увеличить риск деградации образца, способствуя фрагментации нуклеиновых кислот или ферментативной активности, которая, возможно, не была полностью инактивирована во время термической обработки. Эти соображения особенно актуальны при обработке образцов мокроты для обнаружения MTB. Согласно рекомендациям ВОЗ по окрашиванию по Цилю-Нильсену, класс мазка 1+ соответствует 10-99 AFB на 100 нефтяных месторождений, 2+ указывает на 1-10 AFB на месторождение не менее чем на 50 месторождениях, а 3+ представляет более 10 AFB на поле не менее чем на 20месторождениях12.
Ограничением данного метода является его недостаточная автоматизация из-за необходимости ручной гомогенизации с помощью высокоскоростного гомогенизатора или устройства для взбивания валика. Для решения этой проблемы в настоящее время предпринимаются усилия по совершенствованию протокола путем тестирования более короткого времени инкубации, более низких температур инкубации и альтернативных подходов к высокоскоростной гомогенизации.
Таким образом, этот метод экстракции ДНК на основе матрицы в сочетании с высокоскоростным гомогенизатором представляет собой метод быстрой экстракции ДНК, который использует тепло и биение гранул для высвобождения геномной ДНК при одновременном удалении ингибиторов ПЦР, таких как металлы и белки.
Раскрытие информации
Автор Т. Р. получает финансовую поддержку от FIND по контракту на обслуживание с Калифорнийским университетом в Сан-Диего. Автор Т. Р. получил грант от NIH на разработку и оценку решения tNGS для лечения лекарственно-устойчивого туберкулеза (R01AI176401). Автор Т. Р. является соучредителем, членом совета директоров и неоплачиваемым акционером Verus Diagnostics Inc.
Благодарности
Авторы хотели бы поблагодарить команду лаборатории по борьбе с туберкулезом в NHLS - Green Point, Южная Африка, за предоставленную техническую помощь. Эта работа была поддержана Национальными институтами здравоохранения (NIH) и Фондом инновационной новой диагностики (FIND), проектом TS ELiOT Grant ID: R01AI153213, Unitaid Grant ID: 2019-32-FIND MDR. Фигуры создавались с использованием BioRender.com.
Материалы
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
Ссылки
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены