Method Article
Extraction d’ADN basée sur la matrice pour un séquençage ciblé de nouvelle génération sur des échantillons d’expectorations décontaminés
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons une méthode optimisée pour l’extraction de l’ADN à partir d’expectorations décontaminées à l’aide d’une méthode d’extraction matricielle combinée à une purification par billes magnétiques pour le séquençage ciblé de nouvelle génération de Mycobacterium tuberculosis en aval.
Résumé
Le séquençage de nouvelle génération (NGS) est désormais reconnu comme un outil puissant pour le diagnostic rapide et précis de la tuberculose pharmacorésistante. Le NGS ciblé (tNGS) offre une approche rationalisée en se concentrant sur des gènes spécifiques associés à la résistance aux médicaments, en contournant le besoin de méthodes traditionnelles basées sur la culture avec des délais d’exécution allant de quelques semaines à quelques mois. L’Organisation mondiale de la Santé (OMS) a recommandé le tNGS comme une stratégie précieuse pour améliorer le diagnostic de la tuberculose (TB) afin d’orienter le traitement et d’améliorer les résultats du traitement, en particulier dans les pays aux ressources limitées. Parmi les tests tNGS recommandés par l’OMS, nous avons sélectionné une méthode qui fournit des tests rapides et complets de sensibilité aux médicaments, la détermination de la lignée et le typage des souches. Bien qu’il existe des méthodes d’extraction d’ADN standardisées, elles peuvent prendre du temps et demander beaucoup de main-d’œuvre. Pour relever ce défi, nous avons optimisé un protocole d’extraction d’ADN simplifié basé sur la matrice en combinaison avec la purification par billes magnétiques. Cette méthode offre une approche rapide et efficace pour extraire l’ADN directement des sédiments d’expectorations décontaminés, permettant une analyse rapide du tNGS en aval. En rationalisant le processus d’extraction de l’ADN à partir des sédiments d’expectorations, ce protocole pourrait faciliter une adoption plus large du tNGS dans les contextes cliniques de routine, contribuant ainsi à améliorer les résultats pour les patients et à contribuer aux efforts mondiaux de lutte contre la tuberculose.
Introduction
On estime que 3,7 millions de personnes atteintes de tuberculose n’ont pas été diagnostiquées et traitées dans le monde en 2023, ce qui souligne la menace importante que représente la tuberculose pour la santé mondiale1. L’OMS estime qu’environ 400 000 personnes ont développé une tuberculose résistante à la rifampicine (TB-RR) ou une tuberculose multirésistante (TB-MR) en 20231. Il est essentiel de diagnostiquer et de traiter rapidement la tuberculose et la tuberculose pharmacorésistante pour atteindre les étapes et les objectifs de réduction de l’incidence et de la mortalité liés à la tuberculose1.
Le recours à des méthodes de culture conventionnelles et à des tests de sensibilité phénotypique aux médicaments (pDST) retarde la détermination du profil de résistance des isolats cliniques et du traitement, avec des délais d’exécution de 6 à 8 semaines, et nécessite une infrastructure de bioconfinement complexe. Les tests de diagnostic de routine, associés au NGS de la TB-DR, peuvent fournir un profil complet de résistance aux médicaments et améliorer la personnalisation des schémas thérapeutiques de la TB-MR, tout en réduisant le délai de traitement efficace de plusieurs semaines ou mois à quelques jours 2,3,4.
En 2023 et 2024, l’OMS a recommandé l’utilisation du tNGS comme nouvelle classe de diagnostic afin de déterminer rapidement la sensibilité aux antituberculeux de première et de deuxième intention5. Cela en fait un outil précieux pour guider les décisions de traitement sans avoir besoin de culture de Mycobacterium tuberculosis (MTB) dans des laboratoires de niveau de biosécurité 3 (BSL-3). Une approche tNGS est une forme condensée de séquençage qui utilise la réaction en chaîne par polymérase (PCR) pour amplifier les cibles génétiques conférant une résistance aux médicaments avant le séquençage. Parmi les tests tNGS recommandés par l’OMS, nous avons sélectionné le test Deeplex Myc-TB, dont il a été rapporté qu’il répondait aux critères de détection de la résistance à la rifampicine, à l’isoniazide, à l’éthambutol, au pyrazinamide, aux fluoroquinolones, à l’amikacine, à la streptomycine, au linézolide, à la bédaquiline et à la clofazimine. Nous utilisons ce test pour évaluer la pertinence de l’ADN extrait à l’aide de ce protocole pour le tNGS en aval.
De plus, l’OMS a publié la 2eédition du catalogue des mutations associées à la résistance aux médicaments dans le MTB, fournissant une feuille de route pour l’utilisation du séquençage du génome entier (WGS) et du tNGS pour prédire la sensibilité aux médicaments et guider le traitement6. Une revue systématique et une méta-analyse récentes ont montré que le tNGS avait une sensibilité et une spécificité de 94,1 % et 98,1 %, respectivement, pour la détection de la résistance aux médicaments, sur la base de 23 cibles dans diverses régions conférant une résistance dans le génome du MTB, par rapport à la pDST7.
Cependant, la mise en œuvre de ces méthodes reste difficile en raison de la complexité et des coûts spécifiquement associés aux flux de travail, à l’infrastructure et à l’équipement requis. L’un des principaux défis consiste à isoler suffisamment d’ADN mycobactérien de haute qualité directement à partir des sédiments des expectorations décontaminées, une étape cruciale pour les applications tNGS en aval. Pour y remédier, nous présentons une méthode d’extraction d’ADN simple et rapide adaptée au tNGS.
La méthode d’extraction d’ADN standardisée pour le test MTB tNGS sélectionné comprend un manuel interne et un protocole automatisé8. Nous décrivons ici un protocole d’extraction d’ADN simplifié, basé sur une matrice (Figure 1). La méthode utilise la matrice InstaGene (IGM), qui lie les métaux et les protéines, permettant une extraction d’acides nucléiques de qualité directement à partir de sédiments d’expectorations décontaminés. Cette alternative offre un délai d’exécution plus rapide et un rendement d’ADN suffisant pour le tNGS en aval. Ce protocole s’affranchit des complexités des méthodes manuelles et automatisées tout en assurant un tNGS de qualité pour le diagnostic rapide des variants qui confèrent une résistance au MTB. Avec l’intérêt croissant pour l’utilisation du tNGS dans le domaine de la mycobactériologie, ce protocole pourrait faciliter son adoption dans les flux de travail de diagnostic de routine.

Figure 1 : Représentation schématique des méthodes d’extraction de l’ADN mycobactérien à partir d’échantillons de sédiments d’expectorations décontaminés à l’aide d’une suspension matricielle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Cette recherche a été approuvée par le Comité d’éthique de la recherche : Sécurité biologique et environnementale (REC : BES) de l’Université de Stellenbosch : BES-2024-25384 et le Comité d’éthique de la recherche sur l’homme : N21/09/093 et N09/11/296.
1. Préparation d’échantillons de sédiments d’expectorations
REMARQUE : Les étapes suivantes doivent être effectuées dans un laboratoire de niveau de biosécurité trois (BSL3) avant l’extraction de l’ADN.
- Préparez une aliquote de l’échantillon de sédiment d’expectoration préalablement décontaminé à l’aide de N-acétyl-L-cystéine (NALC) et d’hydroxyde de sodium (NaOH) (NALC/NaOH) en transférant 500 μL-2 mL dans un tube à faible liaison de 1,5 mL ou 2 mL.
- Placez les tubes contenant des échantillons de sédiments d’expectorations à la verticale dans une grille à tubes et placez les échantillons dans un four à 80 °C et incubez pendant 1 h pour inactiver les mycobactéries par traitement thermique.
- Après 1 h, retirez le support de tubes contenant les échantillons, essuyez les surfaces des tubes et placez-les dans un récipient approprié pour le transport vers un laboratoire de niveau de biosécurité deux (BSL2).
REMARQUE : Laissez les échantillons refroidir à température ambiante avant de les manipuler ou de les stocker.
2. Extraction de l’ADN à l’aide de la suspension matricielle et d’un homogénéisateur à grande vitesse (Figure 2)
- Avant de commencer, ajoutez trois billes de verre de 2 mm de diamètre dans des tubes à bouchon à vis de 1,5 ou 2 ml, légèrement bouchés, et en autoclave (idéalement au moins un jour avant l’extraction de l’ADN).
- Retirez la suspension matricielle du réfrigérateur et placez-la sur un agitateur magnétique (un aimant est inclus dans le flacon de réactif) pour amener le réactif à température ambiante et commencer à mélanger le réactif.
- Allumez le bloc chauffant et préchauffez à 56 °C (si deux blocs chauffants sont disponibles, préchauffez le second à 100 °C.
REMARQUE : Les étapes suivantes peuvent être effectuées sur une paillasse de laboratoire dans une installation BSL2 : - Centrifuger l’échantillon de sédiments d’expectorations inactivé par la chaleur à 16 800 × g (ou à pleine vitesse) pendant 15 min. À l’aide d’une pipette de 20 à 200 μL, aspirez et jetez doucement le surnageant, en veillant à ce que la pastille ne soit pas dérangée.
- Mélangez la suspension matricielle en agitant doucement (ne pas vortex) et ajoutez 200 μL de la matrice bien mélangée pour remettre la pastille en suspension.
- Pipeter tout le volume dans un tube à bouchon à vis de 1,5 mL contenant trois billes de verre préstérilisées de 2 mm de diamètre.
- Placez les échantillons dans un bloc chauffant et incubez à 56 °C pendant 15 min.
REMARQUE : Immédiatement après avoir retiré l’échantillon du bloc chauffant, réglez le bloc chauffant à 100 °C après l’étape 2.7, si un seul bloc chauffant est disponible. - Homogénéiser les échantillons à l’aide d’un vortex pendant 10 s pour disperser les cellules.
- Placez les échantillons dans un bloc chauffant et incubez à 100 °C pendant 10 min.
- Homogénéiser les échantillons à l’aide d’un homogénéisateur à grande vitesse avec les paramètres suivants : un cycle de 60 s à 4,0 m/s.
- Centrifugeuse à 12 000 × g pendant 5 min. Transférez 130 μL du surnageant contenant de l’ADN dans un tube frais de 1,5 mL à faible liaison sans perturber la pastille (matrice avec débris cellulaires). Jetez le premier tube contenant la matrice et les débris de la cellule.
REMARQUE : POINT D’ARRÊT SÛR. En cas d’arrêt, congelez les échantillons à -20 °C jusqu’à ce qu’ils soient prêts à poursuivre la purification de l’ADN. - Procédez à la purification de l’ADN à l’aide de billes magnétiques.
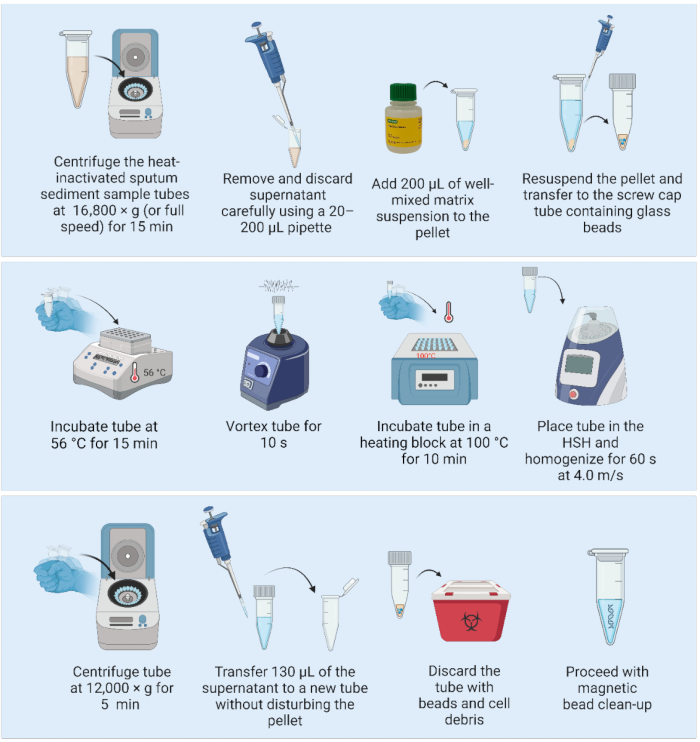
Figure 2 : Méthode d’extraction d’ADN basée sur la matrice couplée à un homogénéisateur à grande vitesse. Abréviation : HSH = homogénéisateur à grande vitesse. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Purification de l’ADN à l’aide de billes magnétiques (Figure 3)
REMARQUE : Avant de procéder à la purification de l’ADN, effectuez les étapes 3.1 et 3.2.
- Sortez les billes magnétiques du réfrigérateur et placez le récipient à température ambiante pendant 30 min pour équilibrer. Alternativement, vortex la bouteille de stock de billes magnétiques pour remettre en suspension toutes les billes et aspirer une aliquote de 1 280 μL dans un nouveau tube (pour la purification de 21 échantillons) et placer à température ambiante pour équilibrer.
- Préparez un lot de 9 ml d’éthanol à 80 % en ajoutant 7,2 ml d’alcool éthylique pur à 1,8 ml d’eau filtrée et stérilisée.
REMARQUE : Les étapes suivantes peuvent être effectuées sur une paillasse de laboratoire dans une installation BSL2. - Manipulez soigneusement la bouteille de stock de billes magnétiques ou l’aliquote préparée pour vous assurer que les billes sont remises en suspension avant utilisation. Répétez le vortex tous les 10 échantillons pour assurer une suspension homogène.
- Ajouter 1,2 fois le volume (156 μL) des billes magnétiques à l’ADN extrait (130 μL).
REMARQUE : Pour les échantillons contenant de plus grandes quantités d’ADN (culture), une plus petite partie de l’échantillon peut être traitée (p. ex., 50 μL) et purifiée avec 1,2 fois le volume de billes magnétiques. - Mélangez les échantillons par pipetage 10x. Incuber les échantillons pendant 5 min à température ambiante.
- Placez les échantillons sur la grille magnétique du tube de 1,5 mL pendant 3 minutes ou jusqu’à ce que le liquide devienne clair. Aspirez et jetez soigneusement le surnageant sans déranger les billes.
- Avec les tubes sur l’aimant, ajoutez 200 μL d’éthanol à 80 %, en veillant à ce que les billes ne soient pas dérangées et laissez incuber pendant 30 s.
- Aspirez et jetez soigneusement l’éthanol sans déranger les billes.
- Répétez l’étape 3.7 pour un total de deux lavages.
- Après le deuxième lavage, retirez et jetez l’éthanol. Éliminez tout résidu d’éthanol à l’aide d’une pipette de 1 à 10 μL.
- Laissez les tubes ouverts pour sécher les billes à l’air libre pendant 10 min, ou jusqu’à ce que les billes aient un aspect mat.
REMARQUE : Ne laissez pas les billes trop sécher et se fissurer. - Une fois que les billes ont un aspect mat, retirez immédiatement les tubes de l’aimant et ajoutez 50 μL d’eau sans nucléase (NFW) directement sur les billes de chaque échantillon. Après avoir ajouté NFW à tous les tubes, mélangez chaque échantillon individuel en pipetant 10x. Inspectez le tube pour voir s’il n’y a pas de billes collées à l’intérieur du tube et répétez l’étape de mélange si nécessaire. Incuber les échantillons pendant 5 min à température ambiante.
REMARQUE : L’incubation peut être augmentée de 5 min à 10 min, mais ne diminuez pas le temps d’incubation à moins de 5 min. - Replacez les tubes sur la grille magnétique pendant 3 minutes ou jusqu’à ce que le liquide soit clair. Avec les tubes sur le support magnétique, transférez le surnageant contenant de l’ADN dans un tube clairement marqué, stérile et à faible liaison. Assurez-vous que les billes ne sont pas transférées au surnageant.
REMARQUE : Si des billes sont visibles dans la pointe de la pipette pendant le transfert, pipetez le surnageant dans le tube sur le support magnétique pour le séparer pendant 3 minutes supplémentaires. Ne transférez pas les billes dans le nouveau tube.
POINT D’ARRÊT SÛR. En cas d’arrêt, congeler les échantillons à -20 °C jusqu’à ce qu’ils soient prêts à poursuivre la quantification à l’aide d’une méthode appropriée au NGS et l’amplification PCR ultérieure à l’aide d’un test MTB tNGS.

Figure 3 : Purification et concentration de l’ADN à l’aide de billes magnétiques. Abréviations : NFW = eau exempte de nucléases ; tNGS = séquençage ciblé de nouvelle génération. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Description de l’échantillon
Au total, 165 échantillons de sédiments d’expectorations, positifs pour les bacilles acido-résistants (BAAR) au microscope avec une charge bactérienne d’au moins 1+, ont été systématiquement collectés et traités par le National Health Laboratory Services (NHLS) de Green Point, au Cap, en Afrique du Sud. L’ADN a été extrait d’échantillons de sédiments d’expectorations à l’aide de deux volumes différents [environ 2 mL (n = 102) et 500 μL (n = 63)]. Cette comparaison a été effectuée pour évaluer si le plus petit volume d’échantillon de sédiments pouvait produire suffisamment d’ADN pour le tNGS en aval.
La figure 4 montre une comparaison en boîte à moustaches de la concentration totale d’ADN (ng/μL) entre différents grades de frottis d’expectoration (1+, 2+ et 3+) et volumes de sédiments d’expectoration (2 mL et 500 μL). Le rendement en ADN diminue généralement dans l’ensemble lorsque l’on utilise 500 μL de sédiments pour l’extraction, avec une variabilité entre les niveaux de frottis. Ainsi, les concentrations moyennes d’ADN extraites d’échantillons de sédiments d’un volume d’au moins 2 mL étaient en moyenne plus élevées que la concentration d’ADN extraite de sédiments de 500 μL, stratifiés par AFB de l’échantillon.

Figure 4 : Impact du volume des sédiments d’expectoration sur le rendement en ADN à travers les grades de frottis. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La figure 5 montre des boîtes à moustaches qui illustrent le nombre moyen de lectures de séquençage (profondeur de couverture moyenne) de toutes les cibles pour des échantillons de sédiments d’expectorations avec différents degrés de frottis (1+, 2+ et 3+) traités à partir de deux volumes d’entrée différents (2 mL et 500 μL). Les valeurs aberrantes sont représentées par des points individuels, et l’axe des y est affiché sur une échelle logarithmique pour tenir compte des variations de couverture. L’écart interquartile élevé pour les échantillons 3+ extraits des sédiments de 500 μL suggère une plus grande variabilité avec des charges bactériennes plus élevées.
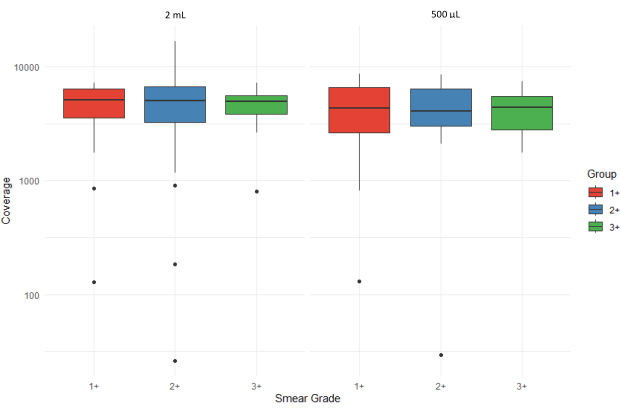
Figure 5 : Comparaison du nombre moyen de lectures de séquençage (profondeur de couverture) pour toutes les cibles entre les niveaux de frottis et les volumes d’entrée d’échantillon. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La figure 6 illustre l’évaluation de l’acceptabilité des résultats de séquençage à l’aide du pipeline bioinformatique de l’application Web associée. Les scores de qualité du séquençage sont classés comme suit : +++ (très acceptable), ++ (acceptable), + (marginalement acceptable), - (inacceptable) et ND (non déterminé). L’absence de barre dans le graphique indique qu’aucun résultat n’a été obtenu pour cette catégorie spécifique. Le grade de frottis 1+ montre une proportion plus élevée de résultats inacceptables et ND par rapport aux grades de frottis 2+ et 3+ pour l’ensemble d’échantillons de volume d’entrée de 500 μL. Les échantillons extraits de 500 μL de sédiments d’expectorations avaient une profondeur de séquençage moyenne de 4 316, comparativement à 4 810 pour les échantillons extraits de sédiments d’expectorations de 2 mL. Cela suggère que l’ADN extrait des sédiments d’expectoration était adéquat pour effectuer le tNGS en aval, quel que soit le volume d’entrée.
D’après la distribution de l’acceptabilité des résultats de séquençage, les échantillons avec un frottis de 3+ ont eu le taux de réussite le plus élevé, la plupart des séquences d’échantillons étant notées ++ et +++ par rapport aux échantillons de 1+ et 2+ de grade frottis. Pour les échantillons d’entrée de 500 μL, la proportion d’échantillons dans les catégories - et + est relativement plus élevée pour tous les niveaux de frottis par rapport aux échantillons d’entrée de 2 mL. Cela suggère qu’un plus grand nombre d’échantillons entrent dans les catégories de qualité inférieure et moins acceptables pour l’entrée de 500 μL. Pour les échantillons d’entrée de 2 ml, il y a une proportion plus élevée d’échantillons dans les catégories de qualité supérieure, ++ et +++, en particulier pour le frottis de grade 3+. Cela suggère que les échantillons avec une entrée de 2 ml sont plus susceptibles de produire de meilleurs scores de qualité de séquençage que ceux avec une entrée de 500 μL.

Figure 6 : Répartition des scores de qualité par degré de frottis et volumes d’échantillons d’entrée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
La combinaison de la méthode d’extraction matricielle couplée à un homogénéisateur à grande vitesse a été initialement publiée par Shea et al.9. Cette méthode a été développée et optimisée pour le séquençage par intervalles de l’ADN extrait de cultures de CMTB 9,10. Nous avons optimisé cette méthode pour l’utiliser avec un test MTB tNGS. L’extraction et la purification de l’ADN de 21 échantillons ont eu un temps d’exécution combiné de 4 h et 40 min, y compris les étapes d’incubation et de centrifugation. La fiabilité du protocole décrit, associée à un nettoyage des billes pour l’extraction de l’ADN à partir d’échantillons dont les grades variant de 1+ à 3+, est démontrée sur la base du rendement moyen (figure 4), de la profondeur globale de couverture de toutes les cibles combinées (figure 5) et du score d’acceptabilité des résultats de séquençage déterminé par l’application Web associée (figure 6).
Une étude récente n’a trouvé aucune relation cohérente entre le grade de frottis et la concentration d’ADN ou la profondeur de lecture du séquençage. Les auteurs ont suggéré que la variabilité des étapes de traitement et de nettoyage des échantillons peut affecter le rendement des échantillons à frottis élevé, en particulier lorsque les contaminants ne sont pas éliminés efficacement. De plus, le séquençage à partir des sédiments d’expectorations reste difficile en raison de la complexité du pool d’ADN, où l’ADN non cible peut entrer en compétition avec l’ADN MTB11. Bien que nous n’ayons pas observé de corrélation claire entre la concentration d’ADN d’entrée et la profondeur de séquençage, il est important de noter que l’entrée d’ADN avant la PCR cible ne doit pas dépasser 100 ng, selon les instructions du fabricant, car cela pourrait entraîner une inhibition de la PCR. De plus, une charge bactérienne plus élevée, les échantillons de sédiments d’expectorations 2+ et 3+ pourraient contenir plus de contaminants, tels que de l’ADN humain, des débris cellulaires et d’autres inhibiteurs, qui peuvent interférer avec l’amplification de la PCR et la préparation de la banque.
L’étape de purification fonctionne également comme une étape de concentration, améliorant le rendement de l’ADN mycobactérien avant le tNGS. Cette méthode est efficace pour le traitement d’échantillons à faible charge bactérienne et a démontré des performances fiables avec des volumes aussi petits que 500 μL, ce qui la rend adaptée aux environnements de routine où le volume d’échantillon disponible est limité. Son application peut réduire le besoin de visites de suivi dans les cliniques, minimisant ainsi le risque de perte de patients lors du suivi. De plus, la méthode est simple et ne nécessite pas d’expertise de laboratoire avancée, ce qui renforce son intégration dans des environnements aux ressources limitées.
Cependant, certains points concernant le protocole doivent être notés. Les échantillons ne doivent pas être congelés ou réfrigérés immédiatement après l’incubation à 80 °C. Un refroidissement rapide immédiatement après le traitement thermique peut provoquer la formation de condensation sur les surfaces internes du tube, entraînant une dilution de l’échantillon et affectant potentiellement l’extraction de l’ADN en aval. De plus, un refroidissement soudain peut augmenter le risque de dégradation de l’échantillon en favorisant la fragmentation des acides nucléiques ou l’activité enzymatique qui n’a peut-être pas été complètement inactivée pendant le traitement thermique. Ces considérations sont particulièrement pertinentes lors du traitement d’échantillons d’expectorations pour la détection des MTB. Selon les recommandations de l’OMS pour la coloration Ziehl-Neelsen, un grade de frottis de 1+ correspond à 10-99 AFB pour 100 champs d’immersion dans l’huile, 2+ indique 1-10 AFB par champ dans au moins 50 champs, et 3+ représente plus de 10 AFB par champ dans au moins 20 champs12.
L’une des limites de cette méthode est son manque d’automatisation en raison de la nécessité d’une homogénéisation manuelle à l’aide d’un homogénéisateur à grande vitesse ou d’un dispositif de battage de billes. Pour remédier à cette situation, les efforts en cours sont axés sur l’amélioration du protocole en testant des temps d’incubation plus courts, des températures d’incubation plus basses et d’autres approches d’homogénéisation à grande vitesse.
Ainsi, cette méthode d’extraction d’ADN basée sur la matrice, combinée à un homogénéisateur à grande vitesse, est une technique d’extraction rapide de l’ADN qui utilise la chaleur et le battement des billes pour libérer de l’ADN génomique tout en éliminant simultanément les inhibiteurs de PCR, tels que les métaux et les protéines.
Déclarations de divulgation
L’auteur T. R. reçoit un soutien financier de FIND par le biais d’un contrat de service avec UC San Diego. L’auteur T. R. a reçu une subvention des NIH pour développer et évaluer une solution tNGS pour la tuberculose résistante aux médicaments (R01AI176401). L’auteur T. R. est cofondateur, membre du conseil d’administration et actionnaire non rémunéré de Verus Diagnostics Inc.
Remerciements
Les auteurs tiennent à remercier l’équipe du laboratoire de tuberculose du NHLS - Green Point, Afrique du Sud pour son assistance technique. Ce travail a été soutenu par les National Institutes of Health (NIH) et la Foundation for Innovative New Diagnostics (FIND), projet TS ELiOT Grant ID : R01AI153213, Unitaid Grant ID : 2019-32-FIND MDR. Les figurines ont été créées à l’aide de BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
Références
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon