Method Article
Matrixbasierte DNA-Extraktion für gezieltes Next-Generation-Sequencing an dekontaminierten Sputumproben
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir stellen eine optimierte Methode zur DNA-Extraktion aus dekontaminiertem Sputum vor, bei der eine matrixbasierte Extraktionsmethode in Kombination mit einer magnetischen Bead-Aufreinigung für die nachgelagerte gezielte Next-Generation-Sequenzierung von Mycobacterium tuberculosis verwendet wird.
Zusammenfassung
Next-Generation-Sequencing (NGS) ist heute als leistungsstarkes Instrument für die rechtzeitige und genaue Diagnose von arzneimittelresistenter Tuberkulose (DR-TB) anerkannt. Targeted NGS (tNGS) bietet einen optimierten Ansatz, indem es sich auf bestimmte Gene konzentriert, die mit Arzneimittelresistenz verbunden sind, und die Notwendigkeit traditioneller kulturbasierter Methoden mit Durchlaufzeiten von Wochen bis Monaten umgeht. Die Weltgesundheitsorganisation (WHO) hat tNGS als wertvolle Strategie zur Verbesserung der Diagnose von Tuberkulose (TB) empfohlen, um die Behandlung zu steuern und die Behandlungsergebnisse zu verbessern, insbesondere in Umgebungen mit begrenzten Ressourcen. Unter den von der WHO empfohlenen tNGS-Assays haben wir eine Methode ausgewählt, die schnelle und umfassende Empfindlichkeitstests für Medikamente, die Bestimmung der Abstammung und die Typisierung von Stämmen ermöglicht. Es stehen zwar standardisierte DNA-Extraktionsmethoden zur Verfügung, diese können jedoch zeit- und arbeitsintensiv sein. Um dieser Herausforderung zu begegnen, haben wir ein vereinfachtes, matrixbasiertes DNA-Extraktionsprotokoll in Kombination mit der Aufreinigung von magnetischen Beads optimiert. Diese Methode bietet einen schnellen und effizienten Ansatz für die Extraktion von DNA direkt aus dekontaminierten Sputumsedimenten und ermöglicht so eine schnelle nachgelagerte tNGS-Analyse. Durch die Rationalisierung des DNA-Extraktionsprozesses aus Sputumsediment könnte dieses Protokoll eine breitere Einführung von tNGS in klinischen Routineumgebungen erleichtern, was letztendlich zu verbesserten Patientenergebnissen beiträgt und zu den weltweiten Bemühungen zur TB-Kontrolle beiträgt.
Einleitung
Schätzungsweise 3,7 Millionen Menschen mit TB blieben im Jahr 2023 weltweit undiagnostiziert und unbehandelt, was die erhebliche Bedrohung durch TB für die globale Gesundheit unterstreicht1. Die WHO schätzt, dass im Jahr 2023 etwa 400.000 Menschen an Rifampicin-resistenter (RR-TB) oder multiresistenter Tuberkulose (MDR-TB) erkrankt sind1. Die schnelle Diagnose und Behandlung von TB und DR-TB ist unerlässlich, um die Meilensteine und Ziele zur Reduzierung der TB-Inzidenz und -Mortalität zu erreichen1.
Der Rückgriff auf konventionelle Kulturmethoden und phänotypische Arzneimittelempfindlichkeitstests (pDST) verzögert die Bestimmung des Resistenzprofils klinischer Isolate und der Behandlung mit Durchlaufzeiten von 6-8 Wochen und erfordert eine komplexe Biocontainment-Infrastruktur. Routinemäßige diagnostische Tests in Kombination mit NGS von DR-TB können ein umfassendes Arzneimittelresistenzprofil liefern und die Personalisierung von MDR-TB-Therapien verbessern, während gleichzeitig die Zeit bis zur wirksamen Behandlung von Wochen oder Monaten auf Tage verkürztwird 2,3,4.
In den Jahren 2023 und 2024 empfahl die WHO den Einsatz von tNGS als neue diagnostische Klasse, um die Anfälligkeit für Erst- und Zweitlinienmedikamente gegen TB schnell zu bestimmen5. Dies macht es zu einem wertvollen Instrument für Behandlungsentscheidungen, ohne dass in Labors der Biosicherheitsstufe 3 (BSL-3) eine Mycobacterium tuberculosis (MTB)-Kultur erforderlich ist. Ein tNGS-Ansatz ist eine kondensierte Form der Sequenzierung, bei der die Polymerase-Kettenreaktion (PCR) verwendet wird, um Wirkstoffresistenz-verleihende Genziele vor der Sequenzierung zu amplifizieren. Unter den von der WHO empfohlenen tNGS-Tests haben wir den Deeplex Myc-TB-Assay ausgewählt, von dem berichtet wurde, dass er die klassenbasierten Kriterien für den Nachweis von Resistenzen gegen Rifampicin, Isoniazid, Ethambutol, Pyrazinamid, Fluorchinolone, Amikacin, Streptomycin, Linezolid, Bedaquilin und Clofazimin erfüllt. Wir verwenden diesen Assay, um die Eignung der mit diesem Protokoll extrahierten DNA für nachgeschaltete tNGS zu bewerten.
Darüber hinaus veröffentlichte die WHO die 2. Auflage des Katalogs der Mutationen, die mit Arzneimittelresistenz bei MTB assoziiert sind, und lieferte einen Fahrplan für den Einsatz von Ganzgenomsequenzierung (WGS) und tNGS zur Vorhersage der Arzneimittelempfindlichkeit und zur Steuerung der Behandlung6. Eine kürzlich durchgeführte systematische Übersichtsarbeit und Metaanalyse zeigte, dass tNGS eine Sensitivität und Spezifität von 94,1 % bzw. 98,1 % für den Nachweis von Arzneimittelresistenz aufwies, basierend auf 23 Zielen in verschiedenen resistenzverleihenden Regionen im MTB-Genom, verglichen mit pDST7.
Die Implementierung dieser Methoden bleibt jedoch aufgrund der Komplexität und der Kosten, die speziell mit den erforderlichen Arbeitsabläufen, der Infrastruktur und der Ausrüstung verbunden sind, eine Herausforderung. Eine entscheidende Herausforderung besteht darin, ausreichend hochwertige mykobakterielle DNA direkt aus dem Sediment des dekontaminierten Sputums zu isolieren, ein entscheidender Schritt für nachgelagerte tNGS-Anwendungen. Um dies zu beheben, stellen wir eine schnelle und einfache DNA-Extraktionsmethode vor, die auf tNGS zugeschnitten ist.
Die standardisierte DNA-Extraktionsmethode für den ausgewählten MTB-tNGS-Assay umfasst ein hauseigenes manuelles und automatisiertes Protokoll8. Hier beschreiben wir ein vereinfachtes, matrixbasiertes DNA-Extraktionsprotokoll (Abbildung 1). Die Methode nutzt die InstaGene-Matrix (IGM), die Metalle und Proteine bindet und so eine qualitativ hochwertige Nukleinsäureextraktion direkt aus dekontaminierten Sputumsedimenten ermöglicht. Diese Alternative bietet eine schnellere Durchlaufzeit und eine ausreichende DNA-Ausbeute für nachgeschaltete tNGS. Dieses Protokoll überwindet die Komplexität der manuellen und automatisierten Methoden und gewährleistet gleichzeitig eine qualitativ hochwertige tNGS für die schnelle Diagnose von Varianten, die Resistenz bei MTB verleihen. Mit dem wachsenden Interesse an der Verwendung von tNGS im Bereich der Mykobakteriologie könnte dieses Protokoll seine Einführung in routinemäßige diagnostische Arbeitsabläufe erleichtern.

Abbildung 1: Schematische Darstellung der Verfahren zur Extraktion mykobakterieller DNA aus dekontaminierten Sputumsedimentproben unter Verwendung einer Matrixsuspension. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
Diese Forschung wurde von der Forschungsethikkommission: Biologische und Umweltsicherheit (REC: BES) an der Universität Stellenbosch: BES-2024-25384 und der Ethikkommission für die Humanforschung: N21/09/093 und N09/11/296 genehmigt.
1. Vorbereitung der Sputumsedimentprobe
HINWEIS: Die folgenden Schritte sind vor der DNA-Extraktion in einem Labor der Biosicherheitsstufe drei (BSL3) durchzuführen.
- Bereiten Sie ein Aliquot der Sputumsedimentprobe vor, die zuvor mit N-Acetyl-L-Cystein (NALC) und Natriumhydroxid (NaOH) (NALC/NaOH) dekontaminiert wurde, indem Sie 500 μl-2 mL in ein 1,5 mL oder 2 mL Low-Bind-Röhrchen überführen.
- Die Röhrchen mit den Sputumsedimentproben werden aufrecht in ein Röhrchengestell gelegt und in einen Ofen bei 80 °C gestellt und 1 h inkubiert, um die Mykobakterien durch Wärmebehandlung zu inaktivieren.
- Entfernen Sie nach 1 Stunde das Röhrchengestell mit den Proben, wischen Sie die Röhrchenoberflächen ab und legen Sie sie in einen geeigneten Behälter für den Transport in ein Labor der Biosicherheitsstufe zwei (BSL2).
HINWEIS: Lassen Sie die Proben auf Raumtemperatur abkühlen, bevor Sie sie weiter handhaben oder lagern.
2. DNA-Extraktion mit der Matrixsuspension und einem Hochgeschwindigkeits-Homogenisator (Abbildung 2)
- Bevor Sie beginnen, geben Sie drei Glasperlen mit einem Durchmesser von 2 mm in 1,5- oder 2-ml-Schraubverschlussröhrchen, die locker verschlossen sind, und autoklavieren Sie sie (idealerweise mindestens einen Tag vor der DNA-Extraktion).
- Nehmen Sie die Matrixsuspension aus dem Kühlschrank und legen Sie sie auf einen Magnetrührer (ein Magnet ist in der Reagenzflasche enthalten), um das Reagenz auf Raumtemperatur zu bringen und mit dem Mischen des Reagenzes zu beginnen.
- Heizblock einschalten und auf 56 °C vorheizen (wenn zwei Heizblöcke zur Verfügung stehen, den zweiten auf 100 °C vorheizen.
HINWEIS: Die folgenden Schritte können auf einem Labortisch in einer BSL2-Einrichtung durchgeführt werden: - Zentrifugieren Sie die hitzeinaktivierte Sputumsedimentprobe bei 16.800 × g (oder voller Geschwindigkeit) für 15 min. Mit einer 20-200 μL-Pipette den Überstand vorsichtig ansaugen und entsorgen, um sicherzustellen, dass das Pellet ungestört bleibt.
- Mischen Sie die Matrixsuspension durch leichtes Schütteln (nicht vortexen) und fügen Sie 200 μl der gut gemischten Matrix hinzu, um das Pellet wieder zu suspendieren.
- Pipettieren Sie das gesamte Volumen in ein 1,5-ml-Schraubverschlussröhrchen, das drei vorsterilisierte Glaskügelchen mit einem Durchmesser von 2 mm enthält.
- Legen Sie die Proben in einen Heizblock und inkubieren Sie sie 15 Minuten lang bei 56 °C.
HINWEIS: Unmittelbar nach der Entnahme der Probe aus dem Heizblock den Heizblock nach Schritt 2.7 auf 100 °C einstellen, wenn nur ein Heizblock verfügbar ist. - Homogenisieren Sie die Proben mit einem Wirbel für 10 s, um die Zellen zu dispergieren.
- Legen Sie die Proben in einen Heizblock und inkubieren Sie sie 10 Minuten lang bei 100 °C.
- Homogenisieren Sie die Proben mit einem Hochgeschwindigkeits-Homogenisator mit folgenden Parametern: ein Zyklus von 60 s bei 4,0 m/s.
- Bei 12.000 × g 5 min zentrifugieren. 130 μL des DNA-haltigen Überstands werden in ein frisches 1,5 mL Röhrchen mit geringer Bindung überführt, ohne das Pellet (Matrix mit Zelltrümmern) zu stören. Entsorgen Sie das erste Röhrchen mit der Matrix und den Zelltrümmern.
HINWEIS: SICHERER HALTEPUNKT. Wenn Sie aufhören, frieren Sie die Proben bei -20 °C ein, bis sie mit der DNA-Aufreinigung fortfahren können. - Fahren Sie mit der DNA-Aufreinigung mit magnetischen Kügelchen fort.
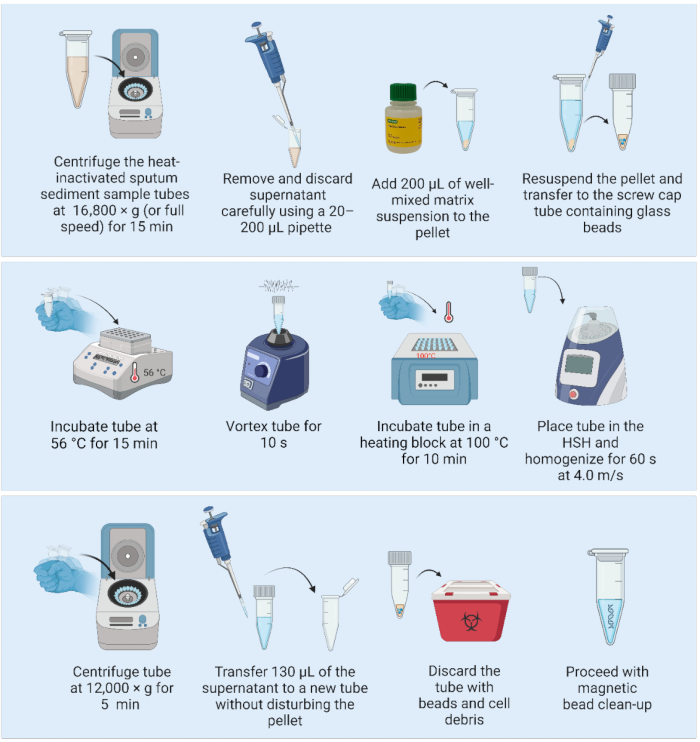
Abbildung 2: Matrixbasierte DNA-Extraktionsmethode gekoppelt mit einem Hochgeschwindigkeits-Homogenisator. Abkürzung: HSH = High-Speed-Homogenisator. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. DNA-Aufreinigung mit magnetischen Kügelchen (Abbildung 3)
HINWEIS: Bevor Sie mit der DNA-Aufreinigung fortfahren, führen Sie die Schritte 3.1 und 3.2 aus.
- Nehmen Sie die magnetischen Kügelchen aus dem Kühlschrank und stellen Sie den Behälter 30 Minuten lang bei Raumtemperatur auf, um ihn auszugleichen. Alternativ können Sie die magnetische Bead-Vorratsflasche vortexen, um alle Kügelchen zu resuspendieren, und ein Aliquot von 1.280 μl in ein neues Röhrchen aspirieren (für die Aufreinigung von 21 Proben) und zum Äquilibrieren bei Raumtemperatur platzieren.
- Bereiten Sie eine 9-ml-Charge mit 80 % Ethanol vor, indem Sie 7,2 mL reinen Ethylalkohol zu 1,8 mL gefiltertem und sterilisiertem Wasser hinzufügen.
HINWEIS: Die folgenden Schritte können auf einem Labortisch in einer BSL2-Einrichtung durchgeführt werden. - Wirbeln Sie die magnetische Perlenschaftflasche oder das vorbereitete Aliquot gründlich ein, um sicherzustellen, dass die Kügelchen vor der Verwendung wieder aufgewirbelt werden. Wiederholen Sie das Vortexen nach jeweils 10 Proben, um eine homogene Suspension zu gewährleisten.
- Fügen Sie der extrahierten DNA (130 μl) das 1,2-fache Volumen (156 μL) der magnetischen Kügelchen hinzu.
HINWEIS: Bei Proben mit größeren DNA-Mengen (Kultur) kann ein kleineres Aliquot der Probe verarbeitet (z. B. 50 μl) und mit dem 1,2-fachen Volumen der magnetischen Beads gereinigt werden. - Mischen Sie die Proben, indem Sie 10x pipettieren. Inkubieren Sie die Proben 5 Minuten lang bei Raumtemperatur.
- Legen Sie die Proben für 3 Minuten oder bis die Flüssigkeit klar ist, auf das 1,5-ml-Röhrchen-Magnetgestell. Saugen Sie den Überstand vorsichtig ab und entsorgen Sie ihn, ohne die Kügelchen zu stören.
- Wenn die Röhrchen auf den Magneten gesetzt sind, fügen Sie 200 μl 80%iges Ethanol hinzu, um sicherzustellen, dass die Kügelchen ungestört bleiben und 30 s lang inkubieren können.
- Aspirieren und entsorgen Sie das Ethanol vorsichtig, ohne die Kügelchen zu stören.
- Wiederholen Sie Schritt 3.7 für insgesamt zwei Wäschen.
- Nach dem zweiten Waschen das Ethanol entfernen und entsorgen. Entfernen Sie alle Ethanolreste mit einer 1-10 μl Pipette.
- Lassen Sie die Tuben offen, um die Perlen 10 Minuten lang an der Luft trocknen zu lassen oder bis die Perlen ein mattes Aussehen haben.
HINWEIS: Lassen Sie die Kügelchen nicht zu stark trocknen und rissig werden. - Sobald die Kügelchen ein mattes Aussehen haben, entfernen Sie sofort die Röhrchen vom Magneten und fügen Sie 50 μl nukleasefreies Wasser (NFW) direkt auf die Kügelchen in jeder Probe hinzu. Nachdem Sie NFW zu allen Röhrchen hinzugefügt haben, mischen Sie jede einzelne Probe durch 10-faches Pipettieren. Untersuchen Sie das Rohr auf Perlen, die an der Innenseite des Rohrs haften geblieben sind, und wiederholen Sie den Mischschritt gegebenenfalls. Inkubieren Sie die Proben 5 Minuten lang bei Raumtemperatur.
HINWEIS: Die Inkubationszeit kann von 5 Minuten auf 10 Minuten erhöht werden, aber verkürzen Sie die Inkubationszeit nicht auf weniger als 5 Minuten. - Legen Sie die Röhrchen für 3 Minuten oder bis die Flüssigkeit klar ist, wieder auf das Magnetgestell. Übertragen Sie mit den Röhrchen auf dem Magnetgestell den DNA-haltigen Überstand in ein eindeutig gekennzeichnetes, steriles, niedrig bindendes Röhrchen. Es ist darauf zu achten, dass keine Kügelchen auf den Überstand übertragen werden.
HINWEIS: Wenn während des Transfers Kügelchen in der Pipettenspitze sichtbar sind, pipettieren Sie den Überstand zurück in das Röhrchen auf dem Magnetgestell, um ihn für weitere 3 Minuten zu trennen. Übertragen Sie keine Perlen in die neue Tube.
SICHERER HALTEPUNKT. Wenn die Probe abbricht, frieren Sie sie bei -20 °C ein, bis sie mit der Quantifizierung mit einer NGS-geeigneten Methode und der anschließenden PCR-Amplifikation mit einem MTB-tNGS-Assay fortgesetzt werden kann.

Abbildung 3: DNA-Aufreinigung und -Konzentration mit magnetischen Kügelchen. Abkürzungen: NFW = nukleasefreies Wasser; tNGS = gezieltes Next-Generation-Sequencing. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Beispiel-Beschreibung
Insgesamt 165 Sputumsedimentproben, die in der Mikroskopie positiv auf säurefeste Bazillen (AFB) mit einer Bakterienlast von mindestens 1+ waren, wurden routinemäßig vom National Health Laboratory Services (NHLS) Green Point, Kapstadt, Südafrika, gesammelt und verarbeitet. Die DNA wurde aus Sputumsedimentproben mit zwei verschiedenen Volumina extrahiert [ca. 2 mL (n = 102) und 500 μL (n = 63)]. Dieser Vergleich wurde durchgeführt, um zu beurteilen, ob das kleinere Sedimentprobenvolumen genügend DNA für nachgeschaltetes tNGS liefern kann.
Abbildung 4 zeigt einen Boxplot-Vergleich der Gesamt-DNA-Konzentration (ng/μl) über verschiedene Sputumabstriche (1+, 2+ und 3+) und Sputumsedimentvolumen (2 mL und 500 μL). Die DNA-Ausbeute nimmt im Allgemeinen insgesamt ab, wenn 500 μl Sediment für die Extraktion verwendet werden, wobei die Abstrichqualitäten variieren. Somit waren die mittleren DNA-Konzentrationen, die aus Sedimentproben mit einem Volumen von mindestens 2 ml extrahiert wurden, im Durchschnitt höher als die DNA-Konzentration, die aus 500 μl-Sedimenten extrahiert wurde, die nach AFB der Probe geschichtet wurden.

Abbildung 4: Einfluss des Sputumsedimentvolumens auf die DNA-Ausbeute über die Schmiergehalte hinweg. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung 5 zeigt Boxplots, die die durchschnittliche Anzahl der Sequenzierungs-Reads (durchschnittliche Abdeckungstiefe) aller Ziele für Sputumsedimentproben mit unterschiedlichen Abstrichgraden (1+, 2+ und 3+) veranschaulichen, die von zwei verschiedenen Eingangsvolumina (2 mL und 500 μL) verarbeitet wurden. Ausreißer werden durch einzelne Punkte dargestellt, und die Y-Achse wird auf einer logarithmischen Skala angezeigt, um Schwankungen in der Abdeckung Rechnung zu tragen. Der hohe Interquartilsabstand für 3+ Proben, die aus den 500-μL-Sedimenten extrahiert wurden, deutet auf eine größere Variabilität bei höherer Bakterienbelastung hin.
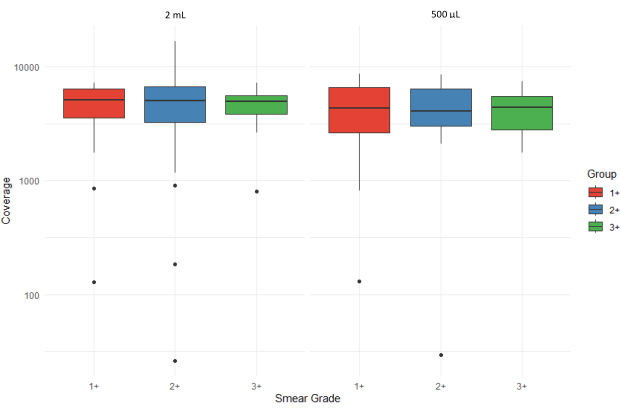
Abbildung 5: Vergleich der durchschnittlichen Anzahl von Sequenzierungs-Reads (Abdeckungstiefe) für alle Ziele über Abstrichgrade und Probeneingangsmengen hinweg. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung 6 zeigt die Bewertung der Akzeptabilität von Sequenzierungsergebnissen unter Verwendung der Bioinformatik-Pipeline der zugehörigen Webanwendung. Die Qualitätswerte für die Sequenzierung werden als +++ (sehr akzeptabel), ++ (akzeptabel), + (geringfügig akzeptabel), - (inakzeptabel) und ND (nicht bestimmt) kategorisiert. Das Fehlen eines Balkens in der Grafik deutet darauf hin, dass für diese spezielle Kategorie keine Ergebnisse erzielt wurden. Der Abstrich Grad 1+ zeigt einen höheren Anteil an inakzeptablen und ND-Ergebnissen im Vergleich zu den Abstrichen der Grade 2+ und 3+ für den Probensatz mit einem Eingangsvolumen von 500 μl. Proben, die aus 500 μl Sputumsediment extrahiert wurden, hatten eine durchschnittliche Sequenzierungsabdeckungstiefe von 4.316 im Vergleich zu 4.810 bei Proben, die aus 2 ml Sputumsediment extrahiert wurden. Dies deutet darauf hin, dass die aus dem Sputumsediment extrahierte DNA unabhängig vom Eingangsvolumen für die Durchführung von nachgeschalteten tNGS geeignet war.
Basierend auf der Verteilung der Akzeptabilität der Sequenzierungsergebnisse hatten Proben mit einem Abstrichgrad von 3+ die höchste Erfolgsquote, wobei die meisten Probensequenzen mit ++ und +++ bewertet wurden, verglichen mit Proben mit 1+ und 2+ Abstrich. Bei den 500-μl-Eingangsproben ist der Anteil der Proben in den Kategorien - und + über alle Abstrichqualitäten hinweg relativ höher als bei den 2-ml-Eingangsproben. Dies deutet darauf hin, dass mehr Proben in die Kategorien mit geringerer Qualität, weniger akzeptabel, für die 500-μl-Eingabe fallen. Bei den 2-ml-Eingangsproben gibt es einen höheren Anteil an Proben in den höherwertigen Kategorien ++ und +++, insbesondere bei Abstrichgrad 3+. Dies deutet darauf hin, dass Proben mit einem Input von 2 ml mit größerer Wahrscheinlichkeit bessere Sequenzierungsqualitätswerte liefern als Proben mit einem Input von 500 μl.

Abbildung 6: Verteilung der Qualitätswerte nach Abstrichgrad und Probeneingangsvolumen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Kombination aus der matrixbasierten Extraktionsmethode in Verbindung mit einem Hochgeschwindigkeits-Homogenisator wurde ursprünglich von Shea et al.9 veröffentlicht. Diese Methode wurde für WGS von DNA entwickelt und optimiert, die aus MTBC-Kulturen extrahiert wurde 9,10. Wir haben diese Methode für die Verwendung mit einem MTB-tNGS-Assay optimiert. Die DNA-Extraktion und -Reinigung von 21 Proben hatte eine kombinierte Durchlaufzeit von 4 h und 40 min, einschließlich Inkubations- und Zentrifugationsschritten. Die Zuverlässigkeit des beschriebenen Protokolls in Verbindung mit einer Bead-Reinigung für die DNA-Extraktion aus Proben mit Abstrichen von 1+ bis 3+ wird auf der Grundlage der durchschnittlichen Ausbeute (Abbildung 4), der Gesamtabdeckungstiefe aller Ziele zusammen (Abbildung 5) und der Akzeptanzbewertung des Sequenzierungsergebnisses, wie sie von der zugehörigen Webanwendung bestimmt wird (Abbildung 6), nachgewiesen.
Eine kürzlich durchgeführte Studie fand keinen konsistenten Zusammenhang zwischen dem Abstrichgrad und der DNA-Konzentration oder der Lesetiefe der Sequenzierung. Die Autoren schlugen vor, dass die Variabilität bei der Probenverarbeitung und den Reinigungsschritten die Leistung bei Proben mit hohem Abstrichgehalt beeinträchtigen kann, insbesondere wenn Verunreinigungen nicht effizient entfernt werden. Darüber hinaus bleibt die Sequenzierung aus Sputumsedimenten aufgrund der Komplexität des DNA-Pools, in dem Nicht-Ziel-DNA mit MTB-DNA konkurrieren kann, eine Herausforderung11. Obwohl wir keine klare Korrelation zwischen der DNA-Konzentration der Eingabe und der Sequenzierungstiefe feststellen konnten, ist es wichtig zu beachten, dass die DNA-Eingabe vor der Ziel-PCR gemäß den Anweisungen des Herstellers 100 ng nicht überschreiten sollte, da dies zu einer PCR-Hemmung führen könnte. Darüber hinaus können Sputumsedimentproben mit höherer Bakterienlast 2+ und 3+ mehr Verunreinigungen wie menschliche DNA, Zelltrümmer und andere Inhibitoren enthalten, die die PCR-Amplifikation und die Vorbereitung der Bibliothek beeinträchtigen können.
Der Reinigungsschritt fungiert auch als Konzentrationsschritt, der die Ausbeute an mykobakterieller DNA vor der tNGS erhöht. Diese Methode ist effektiv für die Verarbeitung von Proben mit geringer Bakterienlast und hat eine zuverlässige Leistung bei Volumina von nur 500 μl gezeigt, wodurch sie sich für Routineumgebungen eignet, bei denen nur ein begrenztes Probenvolumen zur Verfügung steht. Seine Anwendung kann den Bedarf an Nachsorgebesuchen in Kliniken reduzieren und dadurch das Risiko eines Patientenverlusts bei der Nachsorge minimieren. Darüber hinaus ist die Methode einfach und erfordert keine fortgeschrittenen Laborkenntnisse, was die Integration in ressourcenbegrenzte Umgebungen weiter unterstützt.
Allerdings sollten einige Punkte zum Protokoll beachtet werden. Die Proben sollten nicht unmittelbar nach der Inkubation bei 80 °C eingefroren oder gekühlt werden. Eine schnelle Abkühlung unmittelbar nach der Wärmebehandlung kann dazu führen, dass sich Kondenswasser an den Innenflächen des Röhrchens bildet, was zu einer Verdünnung der Probe führt und möglicherweise die nachgelagerte DNA-Extraktion beeinträchtigt. Darüber hinaus kann eine plötzliche Abkühlung das Risiko einer Probendegradation erhöhen, indem sie die Fragmentierung von Nukleinsäuren oder die enzymatische Aktivität fördert, die während der Wärmebehandlung möglicherweise nicht vollständig inaktiviert wurde. Diese Überlegungen sind besonders relevant bei der Verarbeitung von Sputumproben für den MTB-Nachweis. Gemäß den WHO-Empfehlungen für die Ziehl-Neelsen-Färbung entspricht ein Abstrich von 1+ 10-99 AFB pro 100 Ölimmersionsfelder, 2+ bedeutet 1-10 AFB pro Feld in mindestens 50 Feldern und 3+ steht für mehr als 10 AFB pro Feld in mindestens 20 Feldern12.
Eine Einschränkung dieses Verfahrens ist die mangelnde Automatisierung, da eine manuelle Homogenisierung mit einem Hochgeschwindigkeits-Homogenisator oder einer Bead-Beating-Vorrichtung erforderlich ist. Um dies zu beheben, konzentrieren sich die laufenden Bemühungen auf die Verfeinerung des Protokolls, indem kürzere Inkubationszeiten, niedrigere Inkubationstemperaturen und alternative Ansätze für die Hochgeschwindigkeits-Homogenisierung getestet werden.
Daher handelt es sich bei dieser matrixbasierten DNA-Extraktionsmethode, kombiniert mit einem Hochgeschwindigkeits-Homogenisator, um eine schnelle DNA-Extraktionstechnik, bei der Hitze und Bead-Beating verwendet werden, um genomische DNA freizusetzen und gleichzeitig PCR-Inhibitoren wie Metalle und Proteine zu entfernen.
Offenlegungen
Autor T. R. erhält finanzielle Unterstützung von FIND durch einen Servicevertrag mit der UC San Diego. Autor T. R. erhielt Zuschüsse vom NIH für die Entwicklung und Evaluierung einer tNGS-Lösung für arzneimittelresistente TB (R01AI176401). Autor T. R. ist Mitbegründer, Vorstandsmitglied und unbezahlter Aktionär von Verus Diagnostics Inc.
Danksagungen
Die Autoren danken dem TB-Laborteam der NHLS - Green Point, Südafrika für die technische Unterstützung. Diese Arbeit wurde unterstützt von den National Institutes of Health (NIH) und der Foundation for Innovative New Diagnostics (FIND), TS ELiOT-Projekt Förderkennzeichen: R01AI153213, Unitaid Förderkennzeichen: 2019-32-FIND MDR. Die Figuren wurden mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
Referenzen
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten