Method Article
Extração de DNA baseada em matriz para sequenciamento de próxima geração direcionado em amostras de escarro descontaminadas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Apresentamos um método otimizado para extração de DNA de escarro descontaminado usando um método de extração baseado em matriz combinado com uma purificação de esferas magnéticas para sequenciamento de próxima geração direcionado a jusante de Mycobacterium tuberculosis.
Resumo
O sequenciamento de próxima geração (NGS) é agora reconhecido como uma ferramenta poderosa para o diagnóstico oportuno e preciso da tuberculose resistente a medicamentos (DR-TB). O NGS direcionado (tNGS) oferece uma abordagem simplificada, concentrando-se em genes específicos associados à resistência a medicamentos, ignorando a necessidade de métodos tradicionais baseados em cultura com tempos de resposta que variam de semanas a meses. A Organização Mundial da Saúde (OMS) recomendou o tNGS como uma estratégia valiosa para melhorar o diagnóstico de tuberculose (TB) para orientar o tratamento e melhorar os resultados do tratamento, particularmente em ambientes com recursos limitados. Entre os ensaios tNGS recomendados pela OMS, selecionamos um método que fornece testes rápidos e abrangentes de suscetibilidade a medicamentos, determinação de linhagem e tipagem de cepas. Embora os métodos padronizados de extração de DNA estejam disponíveis, eles podem ser demorados e trabalhosos. Para enfrentar esse desafio, otimizamos um protocolo simplificado de extração de DNA baseado em matriz em combinação com a purificação de esferas magnéticas. Este método oferece uma abordagem rápida e eficiente para extrair DNA diretamente de sedimentos de escarro descontaminados, permitindo uma rápida análise de tNGS a jusante. Ao simplificar o processo de extração de DNA do sedimento do escarro, este protocolo pode facilitar a adoção mais ampla do tNGS em ambientes clínicos de rotina, contribuindo para melhorar os resultados dos pacientes e contribuindo para os esforços globais de controle da TB.
Introdução
Estima-se que 3,7 milhões de pessoas com TB não foram diagnosticadas e tratadas globalmente em 2023, destacando a ameaça significativa representada pela TB para a saúde global1. A OMS estima que aproximadamente 400.000 pessoas desenvolveram TB resistente à rifampicina (RR-TB) ou multirresistente (MDR-TB) em 20231. Diagnosticar e tratar TB e DR-TB rapidamente é essencial para atingir os marcos e metas para reduzir a incidência e mortalidade por TB1.
Confiar em métodos convencionais de cultura e testes de suscetibilidade a medicamentos fenotípicos (pDST) atrasa a determinação do perfil de resistência de isolados clínicos e tratamento, com tempos de resposta de 6 a 8 semanas, e requer infraestrutura complexa de biocontenção. Os testes diagnósticos de rotina, em combinação com NGS de TB-DR, podem fornecer um perfil abrangente de resistência a medicamentos e melhorar a personalização dos esquemas de TB-MDR, além de reduzir o tempo para o tratamento eficaz de semanas ou meses para dias 2,3,4.
Em 2023 e 2024, a OMS recomendou o uso de tNGS como uma nova classe de diagnóstico para determinar rapidamente a suscetibilidade aos medicamentos anti-TB de primeira e segunda linha5. Isso o torna uma ferramenta valiosa para orientar as decisões de tratamento sem a necessidade de cultura de Mycobacterium tuberculosis (MTB) em laboratórios de nível de biossegurança 3 (BSL-3). Uma abordagem tNGS é uma forma condensada de sequenciamento que usa a reação em cadeia da polimerase (PCR) para amplificar os alvos genéticos que conferem resistência a medicamentos antes do sequenciamento. Entre os testes de tNGS recomendados pela OMS, selecionamos o ensaio Deeplex Myc-TB, que foi relatado como atendendo aos critérios baseados em classe para detectar resistência à rifampicina, isoniazida, etambutol, pirazinamida, fluoroquinolonas, amicacina, estreptomicina, linezolida, bedaquilina e clofazimina. Usamos este ensaio para avaliar a adequação do DNA extraído usando este protocolo para tNGS a jusante.
Além disso, a OMS publicou a2ª edição do catálogo de mutações associadas à resistência a medicamentos em MTB, fornecendo um roteiro para o uso de sequenciamento do genoma completo (WGS) e tNGS para prever a suscetibilidade a medicamentos e orientar o tratamento6. Uma recente revisão sistemática e meta-análise mostrou que o tNGS teve uma sensibilidade e especificidade de 94,1% e 98,1%, respectivamente, para a detecção de resistência a medicamentos, com base em 23 alvos em várias regiões que conferem resistência no genoma do MTB, em comparação com o pDST7.
No entanto, a implementação desses métodos continua sendo um desafio devido à complexidade e aos custos especificamente associados aos fluxos de trabalho, infraestrutura e equipamentos necessários. Um desafio crítico é isolar DNA micobacteriano de alta qualidade suficiente diretamente do sedimento do escarro descontaminado, uma etapa crucial para aplicações de tNGS a jusante. Para resolver isso, apresentamos um método rápido e simples de extração de DNA adaptado para tNGS.
O método padronizado de extração de DNA para o ensaio MTB tNGS selecionado inclui um protocolo interno manual e automatizado8. Aqui descrevemos um protocolo simplificado de extração de DNA baseado em matriz (Figura 1). O método faz uso da Matriz InstaGene (IGM), que se liga a metais e proteínas, permitindo a extração de ácidos nucléicos de qualidade diretamente de sedimentos de escarro descontaminados. Esta alternativa fornece um tempo de resposta mais rápido e um rendimento de DNA suficiente para tNGS a jusante. Este protocolo supera as complexidades dos métodos manuais e automatizados, garantindo tNGS de qualidade para o diagnóstico rápido de variantes que conferem resistência no MTB. Com o crescente interesse em usar tNGS no campo da micobacteriologia, este protocolo pode facilitar sua adoção em fluxos de trabalho de diagnóstico de rotina.

Figura 1: Representação esquemática dos métodos de extração de DNA micobacteriano de amostras de sedimentos de escarro descontaminados usando uma suspensão de matriz. Clique aqui para ver uma versão maior desta figura.
Protocolo
Esta pesquisa foi aprovada pelo Comitê de Ética em Pesquisa: Segurança Biológica e Ambiental (REC: BES) da Universidade de Stellenbosch: BES-2024-25384 e Comitê de Ética em Pesquisa em Humanos: N21/09/093 e N09/11/296.
1. Preparação da amostra de sedimento de escarro
NOTA: As etapas a seguir devem ser realizadas em um laboratório de nível de biossegurança três (BSL3) antes da extração de DNA.
- Preparar uma alíquota da amostra de sedimento de expetoração previamente descontaminada com N-acetil-L-cisteína (NALC) e hidróxido de sódio (NaOH) (NALC/NaOH) transferindo 500 μL-2 ml para um tubo de ligação baixa de 1,5 ml ou 2 ml.
- Coloque os tubos contendo amostras de sedimentos de escarro na vertical em um rack de tubos e coloque as amostras em um forno a 80 ° C e incube por 1 h para inativar as micobactérias por meio de tratamento térmico.
- Após 1 h, remova o suporte para tubos com amostras, limpe as superfícies dos tubos e coloque-os em um recipiente apropriado para transporte para um laboratório de nível de biossegurança dois (BSL2).
NOTA: Deixe as amostras esfriarem até a temperatura ambiente antes de manuseá-las ou armazená-las.
2. Extração de DNA usando a suspensão da matriz e um homogeneizador de alta velocidade (Figura 2)
- Antes de começar, adicione três esferas de vidro de 2 mm de diâmetro a tubos com tampa de rosca de 1,5 ou 2 mL, com tampa frouxa, e autoclave (idealmente pelo menos um dia antes da extração de DNA).
- Remova a suspensão da matriz da geladeira e coloque-a em um agitador magnético (um ímã está incluído no frasco do reagente) para levar o reagente à temperatura ambiente e comece a misturar o reagente.
- Ligue o bloco de aquecimento e pré-aqueça a 56 °C (se dois blocos de aquecimento estiverem disponíveis, pré-aqueça o segundo a 100 °C.
NOTA: As seguintes etapas podem ser executadas em uma bancada de laboratório em uma instalação BSL2: - Centrifugue a amostra de sedimento de escarro inativada pelo calor a 16.800 × g (ou velocidade máxima) por 15 min. Com uma pipeta de 20-200 μL, aspire e descarte suavemente o sobrenadante, garantindo que o pellet permaneça intacto.
- Misture a suspensão da matriz agitando suavemente (não vórtice) e adicione 200 μL da matriz bem misturada para ressuspender o pellet.
- Pipete todo o volume em um tubo com tampa de rosca de 1,5 mL contendo três esferas de vidro pré-esterilizadas de 2 mm de diâmetro.
- Colocar as amostras num bloco de aquecimento e incubar a 56 °C durante 15 min.
NOTA: Imediatamente após remover o sample do bloco de aquecimento, ajuste o bloco de aquecimento para 100 °C após a etapa 2.7, se apenas um bloco de aquecimento estiver disponível. - Homogeneizar as amostras usando um vórtice por 10 s para dispersar as células.
- Colocar as amostras num bloco de aquecimento e incubar a 100 °C durante 10 min.
- Homogeneizar as amostras usando um homogeneizador de alta velocidade com os seguintes parâmetros: um ciclo de 60 s a 4,0 m/s.
- Centrifugue a 12.000 × g por 5 min. Transfira 130 μL do sobrenadante contendo DNA para um novo tubo de baixa ligação de 1,5 mL sem perturbar o pellet (matriz com detritos celulares). Descarte o primeiro tubo contendo a matriz e os detritos celulares.
NOTA: PONTO DE PARAGEM SEGURO. Se parar, congele as amostras a -20 °C até que estejam prontas para continuar com a purificação do DNA. - Prossiga com a purificação do DNA usando esferas magnéticas.
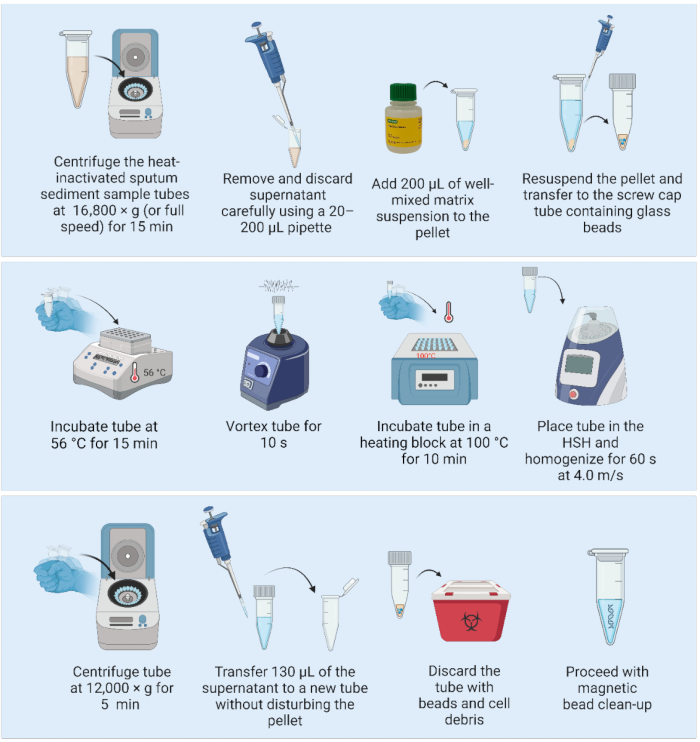
Figura 2: Método de extração de DNA baseado em matriz acoplado a um homogeneizador de alta velocidade. Abreviatura: HSH = homogeneizador de alta velocidade. Clique aqui para ver uma versão maior desta figura.
3. Purificação do DNA usando esferas magnéticas (Figura 3)
NOTA: Antes de prosseguir com a purificação do DNA, execute as etapas 3.1 e 3.2.
- Retire as contas magnéticas da geladeira e coloque o recipiente em temperatura ambiente por 30 min para equilibrar. Alternativamente, vortice o frasco de estoque de esferas magnéticas para ressuspender todas as esferas e aspirar uma alíquota de 1.280 μL em um novo tubo (para purificação de 21 amostras) e coloque em temperatura ambiente para equilibrar.
- Prepare um lote de 9 mL de etanol a 80% adicionando 7,2 mL de álcool etílico puro a 1,8 mL de água filtrada e esterilizada.
NOTA: As etapas a seguir podem ser executadas em uma bancada de laboratório em uma instalação BSL2. - Vortex o frasco de estoque de contas magnéticas ou a alíquota preparada completamente para garantir que as contas sejam ressuspensas antes do uso. Repita o vórtice a cada 10 amostras para garantir uma suspensão homogênea.
- Adicione 1,2x o volume (156 μL) das esferas magnéticas ao DNA extraído (130 μL).
NOTA: Para amostras com maiores quantidades de DNA (cultura), uma alíquota menor da amostra pode ser processada (por exemplo, 50 μL) e purificada com 1,2x o volume de esferas magnéticas. - Misture as amostras pipetando 10x. Incube as amostras por 5 min em temperatura ambiente.
- Coloque as amostras no rack magnético do tubo de 1,5 mL por 3 min ou até que o líquido fique claro. Aspire e descarte cuidadosamente o sobrenadante sem perturbar as contas.
- Com os tubos no ímã, adicione 200 μL de etanol a 80%, garantindo que os grânulos permaneçam intactos e deixe incubar por 30 s.
- Aspire e descarte cuidadosamente o etanol sem perturbar as contas.
- Repita a etapa 3.7 para um total de duas lavagens.
- Após a segunda lavagem, retire e descarte o etanol. Remova qualquer etanol residual com uma pipeta de 1-10 μL.
- Deixe os tubos abertos para secar ao ar por 10 min ou até que tenham uma aparência fosca.
NOTA: Não permita que as contas sequem demais e fiquem rachadas. - Assim que os grânulos tiverem uma aparência fosca, remova imediatamente os tubos do ímã e adicione 50 μL de água livre de nuclease (NFW) diretamente nos grânulos em cada amostra. Depois de adicionar NFW a todos os tubos, misture cada amostra individual pipetando 10x. Inspecione o tubo quanto a grânulos presos no interior do tubo e repita a etapa de mistura, se necessário. Incube as amostras por 5 min em temperatura ambiente.
NOTA: A incubação pode ser aumentada de 5 min para 10 min, mas não diminua o tempo de incubação para menos de 5 min. - Coloque os tubos de volta no rack magnético por 3 min ou até que o líquido esteja límpido. Com os tubos no rack magnético, transfira o sobrenadante contendo DNA para um tubo claramente marcado, estéril e de baixa ligação. Certifique-se de que as contas não sejam transferidas para o sobrenadante.
NOTA: Se os grânulos forem visíveis na ponta da pipeta durante a transferência, pipete o sobrenadante de volta para o tubo no suporte magnético para separar por mais 3 min. Não transfira contas para o novo tubo.
PONTO DE PARAGEM SEGURO. Se parar, congelar as amostras a -20 °C até que estejam prontas para continuar com a quantificação usando um método apropriado para NGS e subsequente amplificação por PCR usando um ensaio MTB tNGS.

Figura 3: Purificação e concentração de DNA usando esferas magnéticas. Abreviaturas: NFW = água livre de nuclease; tNGS = sequenciamento de próxima geração direcionado. Clique aqui para ver uma versão maior desta figura.
Resultados
Descrição da amostra
Um total de 165 amostras de sedimento de escarro, positivas para bacilos álcool-ácido resistentes (BAAR) em microscopia com carga bacteriana de pelo menos 1+, foram rotineiramente coletadas e processadas pelo National Health Laboratory Services (NHLS) Green Point, Cidade do Cabo, África do Sul. O DNA foi extraído de amostras de sedimento de escarro usando dois volumes diferentes [aproximadamente 2 mL (n = 102) e 500 μL (n = 63)]. Essa comparação foi conduzida para avaliar se o menor volume de amostra de sedimento poderia produzir DNA suficiente para tNGS a jusante.
A Figura 4 mostra uma comparação boxplot da concentração total de DNA (ng/μL) em diferentes graus de baciloscopia de escarro (1+, 2+ e 3+) e volumes de sedimento de escarro (2 mL e 500 μL). O rendimento de DNA geralmente diminui em geral ao usar 500 μL de sedimento para extração, com variabilidade entre os graus de esfregaço. Assim, as concentrações médias de DNA extraídas de amostras de sedimentos com volume de pelo menos 2 mL foram, em média, maiores do que a concentração de DNA extraída de sedimentos de 500 μL, estratificada por BAAR da amostra.

Figura 4: Impacto do volume de sedimentos de escarro no rendimento do DNA em todos os graus de esfregaço. Clique aqui para ver uma versão maior desta figura.
A Figura 5 mostra boxplots que ilustram o número médio de leituras de sequenciamento (profundidade média de cobertura) de todos os alvos para amostras de sedimentos de escarro com graus variados de esfregaço (1+, 2+ e 3+) processados a partir de dois volumes de entrada diferentes (2 mL e 500 μL). Os valores discrepantes são representados por pontos individuais e o eixo y é exibido em uma escala logarítmica para acomodar a variação na cobertura. A alta faixa interquartil para 3+ amostras extraídas dos sedimentos de 500 μL sugere maior variabilidade com cargas bacterianas mais altas.
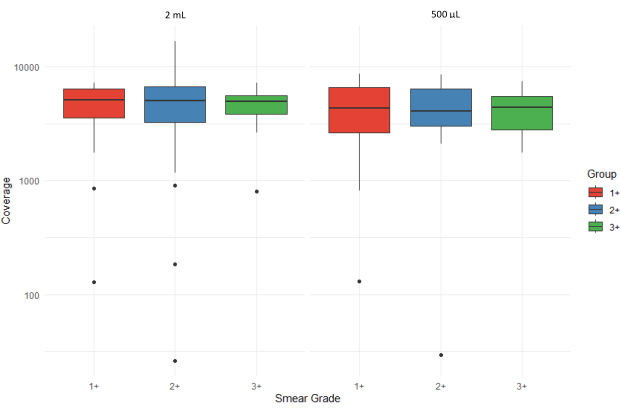
Figura 5: Comparação do número médio de leituras de sequenciamento (profundidade de cobertura) para todos os alvos em graus de esfregaço e volumes de entrada de amostra. Clique aqui para ver uma versão maior desta figura.
A Figura 6 mostra a avaliação da aceitabilidade dos resultados do sequenciamento usando o pipeline de bioinformática do aplicativo da web associado. Os índices de qualidade do sequenciamento são categorizados como +++ (altamente aceitável), ++ (aceitável), + (marginalmente aceitável), - (inaceitável) e ND (não determinado). A ausência de uma barra no gráfico indica que nenhum resultado foi obtido para essa categoria específica. O esfregaço grau 1+ mostra uma proporção maior de resultados inaceitáveis e ND em comparação com os esfregaços graus 2+ e 3+ para o conjunto de amostras de volume de entrada de 500 μL. As amostras extraídas de 500 μL de sedimento de escarro tiveram uma profundidade média de cobertura de sequenciamento de 4.316 em comparação com 4.810 para amostras extraídas de sedimentos de escarro de 2 mL. Isso sugere que o DNA extraído do sedimento do escarro foi adequado para realizar tNGS a jusante, independentemente do volume de entrada.
Com base na distribuição da aceitabilidade dos resultados do sequenciamento, as amostras com um grau de esfregaço de 3+ tiveram a maior taxa de sucesso, com a maioria das sequências de amostra sendo pontuadas como ++ e +++ em comparação com amostras de grau de esfregaço 1+ e 2+. Para as amostras de entrada de 500 μL, a proporção de amostras nas categorias - e + é relativamente maior em todos os graus de esfregaço em comparação com as amostras de entrada de 2 mL. Isso sugere que mais amostras se enquadram nas categorias de qualidade inferior e menos aceitáveis para a entrada de 500 μL. Para as amostras de entrada de 2 mL, há uma proporção maior de amostras nas categorias de maior qualidade, ++ e +++, particularmente para esfregaço grau 3+. Isso sugere que as amostras com entrada de 2 mL são mais propensas a produzir melhores pontuações de qualidade de sequenciamento em comparação com aquelas com entrada de 500 μL.

Figura 6: Distribuição dos índices de qualidade por grau de esfregaço e volumes de entrada de amostra. Clique aqui para ver uma versão maior desta figura.
Discussão
A combinação do método de extração baseado em matriz acoplado a um homogeneizador de alta velocidade foi originalmente publicada por Shea et al.9. Este método foi desenvolvido e otimizado para WGS de DNA extraído de culturas de MTBC 9,10. Otimizamos esse método para uso com um ensaio MTB tNGS. A extração e purificação do DNA de 21 amostras tiveram um tempo de resposta combinado de 4 h e 40 min, incluindo as etapas de incubação e centrifugação. A confiabilidade do protocolo descrito, juntamente com uma limpeza de esferas para extração de DNA de amostras com graus de esfregaço variando de 1+ a 3+, é demonstrada com base no rendimento médio (Figura 4), na profundidade geral de cobertura de todos os alvos combinados (Figura 5) e na pontuação de aceitabilidade do resultado do sequenciamento, conforme determinado pelo aplicativo da web associado (Figura 6).
Um estudo recente não encontrou nenhuma relação consistente entre o grau do esfregaço e a concentração de DNA ou a profundidade de leitura do sequenciamento. Os autores sugeriram que a variabilidade nas etapas de processamento e limpeza da amostra pode afetar o desempenho em amostras de alto grau de esfregaço, principalmente quando os contaminantes não são removidos com eficiência. Além disso, o sequenciamento do sedimento do escarro continua sendo um desafio devido à complexidade do pool de DNA, onde o DNA não-alvo pode competir com o DNA MTB11. Embora não tenhamos observado nenhuma correlação clara entre a concentração de DNA de entrada e a profundidade de sequenciamento, é importante observar que a entrada de DNA antes da PCR alvo não deve exceder 100 ng, de acordo com as instruções do fabricante, pois isso pode levar à inibição da PCR. Além disso, amostras de sedimento de escarro 2+ e 3+ com carga bacteriana mais alta podem conter mais contaminantes, como DNA humano, detritos celulares e outros inibidores, que podem interferir na amplificação da PCR e na preparação da biblioteca.
A etapa de purificação também funciona como uma etapa de concentração, aumentando o rendimento do DNA micobacteriano antes do tNGS. Este método é eficaz para processar amostras com baixa carga bacteriana e demonstrou desempenho confiável com volumes tão pequenos quanto 500 μL, tornando-o adequado para configurações de rotina onde o volume de amostra limitado está disponível. Sua aplicação pode reduzir a necessidade de visitas de acompanhamento às clínicas, minimizando assim o risco de perda de seguimento do paciente. Além disso, o método é simples e não requer conhecimento avançado em laboratório, apoiando ainda mais sua integração em ambientes com recursos limitados.
No entanto, alguns pontos sobre o protocolo devem ser observados. As amostras não devem ser congeladas ou refrigeradas imediatamente após a incubação a 80 °C. O resfriamento rápido imediatamente após o tratamento térmico pode causar a formação de condensação nas superfícies internas do tubo, levando à diluição da amostra e afetando potencialmente a extração de DNA a jusante. Além disso, o resfriamento repentino pode aumentar o risco de degradação da amostra, promovendo a fragmentação do ácido nucleico ou a atividade enzimática que pode não ter sido completamente inativada durante o tratamento térmico. Essas considerações são particularmente relevantes ao processar amostras de escarro para detecção de MTB. De acordo com as recomendações da OMS para a coloração de Ziehl-Neelsen, um grau de esfregaço de 1+ corresponde a 10-99 BAAR por 100 campos de imersão em óleo, 2+ indica 1-10 BAAR por campo em pelo menos 50 campos e 3+ representa mais de 10 BAAR por campo em pelo menos 20 campos12.
Uma limitação deste método é a falta de automação devido à necessidade de homogeneização manual usando um homogeneizador de alta velocidade ou dispositivo de batimento de esferas. Para resolver isso, os esforços contínuos estão focados em refinar o protocolo, testando tempos de incubação mais curtos, temperaturas de incubação mais baixas e abordagens alternativas para homogeneização de alta velocidade.
Assim, este método de extração de DNA baseado em matriz, combinado com um homogeneizador de alta velocidade, é uma técnica de extração rápida de DNA que emprega calor e batimento de esferas para liberar DNA genômico enquanto remove simultaneamente inibidores de PCR, como metais e proteínas.
Divulgações
O autor T. R. recebe apoio financeiro da FIND por meio de um contrato de serviço com a UC San Diego. O autor T. R. recebeu financiamento do NIH para desenvolver e avaliar uma solução tNGS para TB resistente a medicamentos (R01AI176401). O autor T. R. é cofundador, membro do conselho e acionista não remunerado da Verus Diagnostics Inc.
Agradecimentos
Os autores gostariam de agradecer à equipe do laboratório de TB do NHLS - Green Point, África do Sul, pela assistência técnica. Este trabalho foi apoiado pelos Institutos Nacionais de Saúde (NIH) e pela Fundação para Novos Diagnósticos Inovadores (FIND), projeto TS ELiOT Grant ID: R01AI153213, Unitaid Grant ID: 2019-32-FIND MDR. As figuras foram criadas usando BioRender.com.
Materiais
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
Referências
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados