Method Article
Kostengünstige Mikrofluidik-Designs für die Laminierung von Polyethylenterephthalat für die Multiplex-Bildgebung von Zebrafischen
In diesem Artikel
Zusammenfassung
Ein innovatives Verfahren zur Herstellung von mikrofluidischen Geräten mit Polyethylenterephthalat (PET)-Laminierung reduziert die Kosten und die Komplexität des Einfangens und der Bildgebung mehrerer lebender Zebrafischembryonen erheblich.
Zusammenfassung
Zebrafischembryonen sind transparent und eignen sich daher hervorragend für die nicht-invasive intravitale Bildgebung grundlegender Prozesse, wie z.B. der Wundheilung und der Immunzellmigration. Mikrofluidische Geräte werden für das Einfangen verwendet, um die Langzeitbildgebung von mehrzelligen Organismen, einschließlich Zebrafischen, zu unterstützen. Die Herstellung dieser Geräte mittels weicher Lithografie erfordert jedoch spezielle Einrichtungen und Kompetenzen im 3D-Druck, die möglicherweise nicht jedem Labor zugänglich sind. Unsere Adaption eines zuvor entwickelten, kostengünstigen Polyethylenterephthalat-Laminierungsverfahrens für die Konstruktion von mikrofluidischen Geräten erhöht die Zugänglichkeit, indem es Design, Herstellung und Iteration für einen Bruchteil der technischen Investitionen herkömmlicher Techniken ermöglicht. Wir verwenden ein Gerät, das mit dieser Methode hergestellt wurde, den Rotational Assistant for Danio Imaging of Subsequent Healing (RADISH), um die medikamentöse Behandlung, manuelle Verwundung und Langzeitbildgebung von bis zu vier Embryonen im selben Sichtfeld zu ermöglichen. Mit diesem neuen Design gelingt es uns, grobe morphologische Merkmale des Kalziumsignals um Laserablations- und manuelle Durchtrennungswunden für mehrere Embryonen in den 2 Stunden unmittelbar nach der Verletzung sowie die Rekrutierung von Neutrophilen am Wundrand für 24 Stunden zu erfassen.
Einleitung
Die Fähigkeit, angemessen auf Verletzungen zu reagieren, ist entscheidend für das Überleben jedes Organismus auf jeder Ebene, von einer einzelnen Zelle bis hin zu mehreren Geweben. Die Verwundung und die damit verbundenen Reaktionen, wie z.B. die Rekrutierung von Phagozyten in geschädigte Areale1, sind daher wichtige Themen in der Zell- und Gewebebiologie. Wunden werden unmittelbar nach dem Durchbrechen der Gewebebarriere ertastet, was zu einer Gewebegradientenreaktion mit Wundkontraktion 2,3 führt, die die Wundheilung und das anschließende Nachwachsen koordiniert4. Aufgrund der mechanischen Natur dieser Kontraktion dürfen Werkzeuge, die während des Experiments verwendet werden, die Bewegung der Zellen in der Nähe der Verletzungsstelle nicht physisch behindern.
Zebrafischembryonen sind aufgrund ihrer Pflegeleichtigkeit, genetischen Rückverfolgbarkeit und optischen Transparenz ein hervorragendes Modell für die Untersuchung von Entwicklung und Krankheit, einschließlich der Wundreaktion von Wirbeltieren. 5,6. Es ist jedoch erforderlich, den gesamten Organismus über einen längeren Zeitraum zu immobilisieren, um das längerfristige Verhalten von Zellen in der Nähe einer Verletzungsstelle zu untersuchen. Die Einbettung von Embryonen in niedrigschmelzende Agarose ist ausreichend für die kurzfristige Bildgebung von unverwundetem Gewebe. Die umgebende Gelmatrix schränkt jedoch die Kontraktion und Entspannung der Wunden ein und behindert das Wachstum der sich entwickelnden Embryonen im Laufe der Zeit 7,8,9.
Mikrofluidik-Geräte können Zebrafischembryonen in verschiedenen Entwicklungsstadienimmobilisieren 10,11,12,13,14,15,16, und einige, wie z.B. das zWEDGI7, ermöglichen manuelle Wunden. Es gibt jedoch einige Nachteile bei den verfügbaren Gerätedesigns. So sind beispielsweise viele Geräte, die direkt auf Kunststoff gedruckt werden, aufgrund der schlechten optischen Transparenz nicht mit der Bildgebung auf inversen Mikroskopsystemen kompatibel. Darüber hinaus ermöglichen parallele Kanäle eine Mehrpositionsbildgebung10,11, aber die physikalische Bewegung des Mikroskoptisches führt zu einer Verzögerung bei der Bildaufnahme. Wiederholte Iterationen und Optimierungen zur Lösung dieser Probleme sind schwierig, wenn man die finanziellen und technischen Investitionen berücksichtigt, die für jedes neue Design erforderlich sind, insbesondere für die aktuellen Goldstandardmethoden zur Herstellung von Geräten mit weicher Lithographie aus Polydimethylsiloxan (PDMS). Der allererste Schritt, die Erstellung einer Urform, erfordert oft Kenntnisse in 3D-Modellierungssoftware, Zugang zu speziellen Fertigungsgeräten und (je nach verwendetem Material) eine zusätzliche Antiadhäsionsbehandlung wie Silanisierung, bevor die Form überhaupt verwendet wird. Das Aushärten des PDMS dauert nach dem Gießen mindestens eine Stunde bei hohen Temperaturen und muss in der Regel unter einem Vakuum oder mit einer Klemme erfolgen, um beste Ergebnisse zu erzielen, oft in einem Reinraum 7,17,18. Wie in der Entwicklungsbiologie können sich diese Kosten bei Experimenten, die viele einzigartige mikrofluidische Umgebungen über mehrere Modelle hinweg erfordern, schnell als unerschwinglich erweisen.
Von den verfügbaren alternativen Baumaterialien ist Polyethylenterephthalat (PET) langlebig, ungiftig und leicht zu handhaben. PET-Platten sind weit verbreitete Kunststoff-Laminierbeutel, die in den meisten Bürobedarfsgeschäften erhältlich sind und mit voraufgetragenem, thermisch aktiviertem Klebstoff (in der Regel Ethylenvinylacetat) versehen sind. Durch das Formen dieser PET-Platten mit handelsüblichen Bastelschneidern (d. h. computergesteuerten Schneideplottern, die für Heimwerker entwickelt wurden) und das Aneinanderkleben der Schichten mit Standard-thermischen Laminiergeräten kann schnell eine breite Palette potenzieller Designs generiert und iteriert werden. Wir haben daher eine zuvor beschriebene Bemessungs- und Konstruktionsmethode mit gestapeltem PET18 angepasst, um das Rotational Asistant für dieD anio matierung von Ssupra keent Healing (RADISH) zu schaffen (Abbildung 1A,B). Eine rotierende Anordnung mehrerer annähernd keilförmiger Rückhaltekanäle optimiert die Nähe und bietet gleichzeitig Raum für eine manuelle Schwanzflossendurchtrennung, die eine Bildgebung unmittelbar nach der Verwundung und eine gleichzeitige Langzeitbildgebung mehrerer verwundeter Zebrafischembryonen im selben Sichtfeld ermöglicht. Darüber hinaus reduziert diese Konstruktionsmethode die Investitionskosten und den Zeitaufwand für den Gerätebau drastisch, während die Wiederverwendbarkeit erhalten bleibt.
Protokoll
In dieser Studie wurden Embryonen 3 Tage nach der Befruchtung (dpf) verwendet, es können aber auch Embryonen mit 2-14 dpf verwendet werden. Das Zebrafisch-Experiment wurde nach international anerkannten Standards durchgeführt. Das Animal Care and Use Protocol wurde vom Purdue Animal Care and Use Committee (PACUC) genehmigt und hält sich an die Richtlinien für die Verwendung von Zebrafisch im NIH Intramural Research Program (Protokollnummer: 1401001018).
1. Montage des PET-Mikrofluidik-Geräts
HINWEIS: Jeder Teil dieses Protokolls kann jederzeit pausiert werden, mit Ausnahme der Schritte 1.8 und 1.9, die abhängig von der Trocknungszeit von Cyanacrylat sind.
- Entwerfen Sie das Muster für das Gerät in der Software Ihrer Wahl (z. B. Adobe Illustrator, Abbildung 1A). Je nach Alter und Größe des Embryos passen Sie die Breite des Kanals und die Größe der zentralen Bildgebungskammer für die beste Passform an.
- Speichern Sie jede Ebene als einzelne Bilddatei mit identischen Abmessungen. Benennen Sie jede Datei so, dass die Dicke des zu verwendenden Blechs und die Reihenfolge der Montage ersichtlich sind.
- Initialisieren Sie die Schneidemaschine gemäß den Anweisungen des Herstellers.
- Laden Sie jedes entworfene Muster zum Schneiden in die Designsoftware hoch.
- Wählen Sie im Design-Arbeitsbereich die neu hochgeladenen Designs aus. Gruppieren Sie Modelle, die für die gleiche Dicke von PET-Platten vorgesehen sind, im gleichen Schnitt (Abbildung 2A). Passen Sie bei Bedarf die Größe der Muster in der Designsoftware auf die richtigen Abmessungen an.
- Bestätigen Sie die Anzahl und Art der Muster im Schnitt und wählen Sie dann das Schnittmaterial aus.
HINWEIS: PET-Platten sind in der Designsoftware möglicherweise keine Materialauswahl, auch nicht unter Namen wie "Kunststofflaminierbeutel". Stattdessen kann jedes Material mit ähnlicher Dicke und Textur (z. B. transparente Folien oder nicht klebendes Vinyl) ausgewählt werden. - Befestigen Sie die PET-Platten an der Schneidematte, drücken Sie fest, um Luftblasen zu entfernen, und legen Sie die Matte in die Schneidemaschine ein. Befolgen Sie die Anweisungen auf dem Bildschirm, um die Schneidemaschine aufzufordern, mit dem Schnitt zu beginnen.
- Nehmen Sie fertige Schnitte von der Schneidematte. Passen Sie bei Bedarf die Schnittmuster mit einem Bastelmesser oder einer Skalpellklinge an.
HINWEIS: Die Bilddateien, die zur Erstellung des RADISH-Designs verwendet wurden (Abbildung 1A), stehen als Vektor- und PNG-Dateiformate zum Download zur Verfügung (Ergänzende Datei 1). Wenn die Schneidemaschine die PET-Folie anhebt, reißt oder nicht sauber durchschneidet, sollten Sie die Schneidematte austauschen.
- Richten Sie die Schichten auf dem Deckglas aus, ohne fremde Hilfe oder mit vorgestanzten Passermarken. Wenn mehrere Schichten vor dem Laminieren aneinander befestigt werden können, richten Sie sie gleichzeitig aus.
- Sichern Sie die Schichten mit Büroklebeband, um ein Verrutschen während der Laminierung zu vermeiden (Abbildung 2B). Montieren Sie je nach Gerätegröße und Laminierkapazität kleine Designs mit mehr Klebeband auf einer größeren Unterlage, um ein Verklemmen oder Verstopfen der Laminiergerätezuführung zu vermeiden (Abbildung 2C).
HINWEIS: Die bei der Laminierung verwendete Unterlage ist NICHT die gleiche wie die Schneidematte und dient in erster Linie dazu, die Traktion des Laminiergeräts zu gewährleisten. Jedes hitzebeständige Material mit vernachlässigbarer Dicke eignet sich als Unterlage (einschließlich, aber nicht beschränkt auf Karton, gefaltetes Büropapier oder ein größeres PET-Blatt). - Schalten Sie das Laminiergerät ein und laminieren Sie gemäß den Anweisungen des Herstellers. Entfernen Sie das Büroklebeband, bevor Sie die nächste Schicht ausrichten (Abbildung 2D).
- Wiederholen Sie die Schritte 1.4 bis 1.6, bis alle Layer eingehalten wurden.
- Befestigen Sie das Gerät an einer 35-mm-Schüssel, die mit Cyanacrylatkleber mit einem 3/4-Zoll-Loch gebohrt wurde (Abbildung 2E,F). Passen Sie die Form und den Typ der Schale entsprechend den experimentellen Anforderungen an (z. B. einen Behälter, der groß genug ist, um das Gerät aufzunehmen - eine Vertiefung in einer 24-Well-Platte oder eine größere Petrischale). Verwenden Sie genügend Kleber, um das Gerät vollständig mit der Schüssel abzudichten, ohne sie zu überfluten.
HINWEIS: Das Befestigen des Geräts an einer Halteschale ist für die Funktion des Geräts nicht erforderlich. ACHTUNG: Cyanacrylat verbindet Haut und Augen in Sekundenschnelle und reizt die Atemwege, Augen und Haut. Tragen Sie einen Hand- und Augenschutz und arbeiten Sie in einem gut belüfteten Bereich. - Imprägnieren Sie das PET-Laminat, indem Sie die äußeren Kanten des Geräts gründlich mit Cyanacrylatkleber beschichten (Abbildung 2F). Verwende so viel Kleber, dass die Außenkanten vollständig bedeckt sind. Lassen Sie den Kleber vor der Verwendung mindestens 2 h aushärten.
HINWEIS: Stellen Sie sicher, dass ausreichend Platz für die Entlüftung der Cyanacrylatdämpfe vorhanden ist. Die Ansammlung von Dämpfen kann dazu führen, dass sich Cyanacrylat auf dem Gerät ablagert und optisch transparentes Material verdeckt.
2. Positionierung der Embryonen in der Radieschen und Vorbereitung für die Bildgebung
- Die Zebrafischembryonen werden im Standard-Embryoaufzuchtmedium E319 aufgezogen. Falls erforderlich, dechorionieren Sie die Embryonen mindestens 1 h vor der Bildgebung, damit sie sich abflachen können. In diesem Experiment wurden Embryonen mit 3 dpf verwendet, aber Embryonen können bis zu 14 dpf haben (Abbildung 1E).
- Färben oder behandeln Sie die Embryonen je nach experimentellem Bedarf.
HINWEIS: In diesen Experimenten wurden die Zellmembranen der äußeren Epithelschicht gegebenenfalls durch Inkubation mit 100 nM MemGlow 560 gefärbt. - Anästhesieren Sie Embryonen mit 164 mg/l MS-22220.
HINWEIS: Die genaue Konzentration und das verwendete Anästhetikum können je nach Vorliebe des Experimentators angepasst werden. Die Anästhesiekonzentration kann reduziert werden, um eine erfolgreiche Entwicklung während der Langzeitbilder zu ermöglichen (siehe Schritt 4.1). - Füllen Sie die Warmhalteschale mit dem montierten Gerät mit 5 mL E3 mit 164 mg/l MS-222.
- Legen Sie mit einer Transferpipette einen betäubten Embryo in jede Haltekammer des Geräts. Stellen Sie sicher, dass die Embryonen für die Dauer der Bildgebung in Flüssigkeit getaucht bleiben.
HINWEIS: Das RADISH-Design kann 1-4 Embryonen immobilisieren. - Richten Sie die Embryonen mit einer Haarschlaufe so aus, dass der Schwanz des Embryos in die zentrale Bildgebungskammer hineinragt und das Eigelb in dem keilförmigen Kanal sicher immobilisiert ist (Abbildung 1C).
HINWEIS: Die Orientierung der Embryonen kann zur Erleichterung der Manipulation unter einem Stereomikroskop durchgeführt werden. - Falls gewünscht, sichern Sie den Kopf des Embryos, indem Sie mit einer Mikropipette 1,5 % niedrigschmelzende Agarose in die Haltekammer geben.
- Bringen Sie das Gerät mit den Embryonen zum Bildgebungssystem Ihrer Wahl, um mit der Bildgebung zu beginnen.
3. Bildgebung von Calciumtransienten nach einer Verwundung
HINWEIS: Dieser Abschnitt und Abschnitt 4 sind optional und werden als mögliche Beispielversuche zur Verfügung gestellt.
- Wählen Sie das geeignete System und Objektiv für die Bildgebung aus.
HINWEIS: Hier wurde ein inverses Laserscanning konfokal mit einem 20x Objektiv ausgestattet. - Passen Sie den Probenfokus und die Bildgebungsparameter an (z. B. Laserleistung: 5, Verstärkung: 90, Lochblendengröße: 2 AE, Belichtungszeit [Scangeschwindigkeit: 1 Bild/s]) für ein optimales Signal-Rausch-Verhältnis und eine optimale Aufnahmegeschwindigkeit.
HINWEIS: Es wird empfohlen, die Proben vor dem Verwunden vorscharfzustellen und die Parameter für jeden Kanal des Bildgebungssystems der Wahl anzupassen, um die Verzögerung zwischen Wundbildung und Bildaufnahme zu verringern. - Wunde einen Tg(krt4:Gal4, UAS:GCaMP6f) x (cdh1-dTomate)xt1821,22 Embryo.
- Um den Embryo durch manuelle Durchtrennung zu wickeln, üben Sie mit einer Skalpellklinge unter einem Stereomikroskop Druck auf das Schwanzflossengewebe an der Spitze der Chorda aus (Video 1) und stellen Sie das Gerät dann wieder auf den Tisch.
ACHTUNG: Übermäßiger Druck mit der Skalpellklinge kann das darunter liegende Glas zerbrechen. Wenn das Glas zerbricht, entsorgen Sie das Gerät und starten Sie es mit Schritt 1.2 neu. - Um den Embryo durch Laserstimulation zu wickeln, definieren Sie eine Region of Interest (ROI) in der Nähe des Randes der Schwanzflossenfalte und stimulieren Sie entsprechend der verfügbaren Laserleistung.
HINWEIS: Hier reichte eine Laserleistung von 2,4 W für 30 s bei 800 nm aus, um eine Wunde in voller Dicke zu induzieren.
- Um den Embryo durch manuelle Durchtrennung zu wickeln, üben Sie mit einer Skalpellklinge unter einem Stereomikroskop Druck auf das Schwanzflossengewebe an der Spitze der Chorda aus (Video 1) und stellen Sie das Gerät dann wieder auf den Tisch.
- Bild bei Raumtemperatur mit GFP (Anregung 488 nm, Emission 510 nm) und RFP (Anregung 561 nm, Emission 610 nm) unter Verwendung eines Bildintervalls von 1 min für insgesamt 1 h.
- (Optional): Geben Sie die Embryonen bei Bedarf nach der Bildgebung durch sanftes Absaugen in der Nähe des Kopfes mit einer Transferpipette oder Schwenken der Schale zur Entnahme frei.
4. Bildgebung der Rekrutierung von Neutrophilen nach Verwundung
- Mindestens 1 Stunde vor der Bildgebung die Embryonen auf E3 übertragen, das die Hälfte der Standardkonzentration des Anästhetikums enthält, in diesem Fall 82 mg/L MS-222.
- Wählen Sie das geeignete Objektiv für die Bildgebung aus (hier ein inverses konfokales Laserscanning, das mit einem 10-fach-Objektiv ausgestattet ist).
- Passen Sie den Probenfokus und die Bildgebungsparameter an (z. B. Laserleistung: 5, Verstärkung: 90, Lochblende: 2 AE, Belichtungszeit [Scangeschwindigkeit: 0,5 Bilder/s])) für ein optimales Signal-Rausch-Verhältnis und eine optimale Aufnahmegeschwindigkeit.
HINWEIS: Es wird empfohlen, die Proben vor dem Verwunden vorscharfzustellen und die Parameter für jeden Kanal des Bildgebungssystems der Wahl anzupassen, um die Verzögerung zwischen Wundbildung und Bildaufnahme zu verringern. - Wickeln Sie vier Tol2(mpx:Dendra2)23-Embryonen durch manuelle Durchtrennung auf, indem Sie mit einer Skalpellklinge unter einem Stereomikroskop Druck auf das Schwanzflossengewebe ausüben, und bringen Sie das Gerät dann wieder auf den Tisch.
- Nehmen Sie das Bild auf dem GFP-Kanal (Anregung 488 nm, Emission 510 nm) bei Raumtemperatur mit einem Bildintervall von 5 Minuten für 24 h auf.
- Geben Sie die Embryonen bei Bedarf nach der Bildgebung durch sanftes Absaugen in der Nähe des Kopfes mit einer Transferpipette oder Schwenken der Schale zur Entnahme frei.
Ergebnisse
Zum Vergleich der Bildqualität, die durch ein konfokales Laserscanning mit und ohne Hilfe des RADISH erzeugt wurde, wurden Embryonen, die den intensitätometrischen Calcium-Biosensor GCaMP6f22,24 in der äußeren Epithelschicht exprimieren, mit MemGlow 560 gefärbt und mit einem inversen konfokalen Laserscanning, das die gesamte Dicke der Schwanzflossenfalte abdeckte, in einem Zeitintervall von 1 min pro Bild abgebildet (Video 2). Ohne das Gerät (Abbildung 3A) drifteten die Proben erheblich und wiesen Streifenartefakte auf, die sich aus der Begrenzung des Z-Stapels auf 25 Schichten ergaben, um im Vergleich zur Bildgebung mit dem Gerät innerhalb der angegebenen zeitlichen Auflösung zu passen (Abbildung 3B). Das RADISH wurde weiterhin verwendet, um Calciumtransienten unmittelbar nach der Verwundung in Embryonen zu untersuchen, die Tg(krt4:Gal4, UAS:GCaMP6f) x (cdh1-dTomato)xt1821 exprimieren, wobei endogenes E-Cadherin in rot und zytoplasmatisches Calcium im apikalen Epithel grün hervorgehoben wurde. Die Embryonen wurden parallel zu den Seiten der zentralen Kammer mit einer Skalpellklinge (Abbildung 4A und Video 3) oder durch direkte Laserablation mit einem IR-Laser (Abbildung 4B) verwunden und dann durch die gesamte Dicke der Schwanzflossenfalte abgebildet.
Um die Wirksamkeit des RADISH für die längerfristige Bildgebung zu demonstrieren, untersuchten wir die Rekrutierung von Immunzellen an den Wundrand für 24 Stunden nach der Wunde. Vier Zebrafischembryonen mit 3 dph, die Tol2(mpx:Dendra2) exprimierten, um Neutrophile in grün23 zu markieren, wurden durch Schwanzdurchtrennung verwundet und 24 Stunden lang abgebildet (Abbildung 5). Bildgebende Artefakte, die typischerweise mit späten Embryonen mit 3 bis 4 dpf in Verbindung gebracht werden, wie z. B. Kippen und Schweben, waren bemerkenswerterweise nicht vorhanden (Video 4), und das Nachwachsen des Gewebes wurde während des Experiments ungehindert fortgesetzt. Die Rekrutierung von Neutrophilen bis zum Wundrand setzt sich im gesamten Bild fort (Abbildung 5B), wobei die meisten migrierenden Zellen 24 Stunden nach der Wunde wieder verschwinden (Abbildung 5C), was mit der erwarteten Entzündungsauflösung übereinstimmt25,26.

Abbildung 1: Design und Verwendung eines PET-Laminat-Mikrofluidik-Geräts (RADISH). (A) Schematische Veranschaulichung der Anordnung und Dicke der einzelnen PET-Schichten, der Montage und Verwendung des Geräts. (B) Endprodukt, montiert in einem 3/4"-Ausschnitt in einer 35-mm-Schale. (C) 3 dpf-Embryonen, (D) 5 dpf-Embryonen, (E) 14 dpf-Embryonen, die im Gerät untergebracht sind. Maßstabsleisten = 1.000 μm (C,D,E). Abkürzungen: PET = Polyethylenterephthalat; RADISH = Rotationsassistent für die Danio-Bildgebung der nachfolgenden Heilung; dpf = Tage nach der Befruchtung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Fotografische Schritt-für-Schritt-Illustration der Montage von PET-Geräten. (A) Einzelne PET-Schichten vor dem Laminieren. (B) PET-Schicht, die mit Büroklebeband auf einem 25-mm-Deckglas befestigt ist. (C) Das Gerät in Arbeit ist zur Laminierung auf einer Kunststoffunterlage befestigt. (D) Fertiges PET-Laminatgerät. (E) Modifizierte 35-mm-Schüssel vor der Gerätemontage. (F) Fertiges Gerät. Abkürzung: PET = Polyethylenterephthalat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Verbesserte Qualität der konfokalen Laserscanning-Bilder von 3-dph-Embryonen im RADISH-Design. (A) Repräsentatives Bild eines 3 dpf Tg(krt4:Gal4, UAS:GCaMP6f) Embryos, der mit MemGlow 560 gefärbt wurde, ohne den RADISH zu verwenden. (B) Repräsentatives Bild eines Tg(krt4:Gal4, UAS:GCaMP6f) Embryos, der mit MemGlow 560 gefärbt wurde, montiert in der RADISH. Maßstabsleiste = 100 μm. Abkürzungen: RADISH = Rotationsassistent für die Bildgebung der anschließenden Heilung; dpf = Tage nach der Befruchtung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
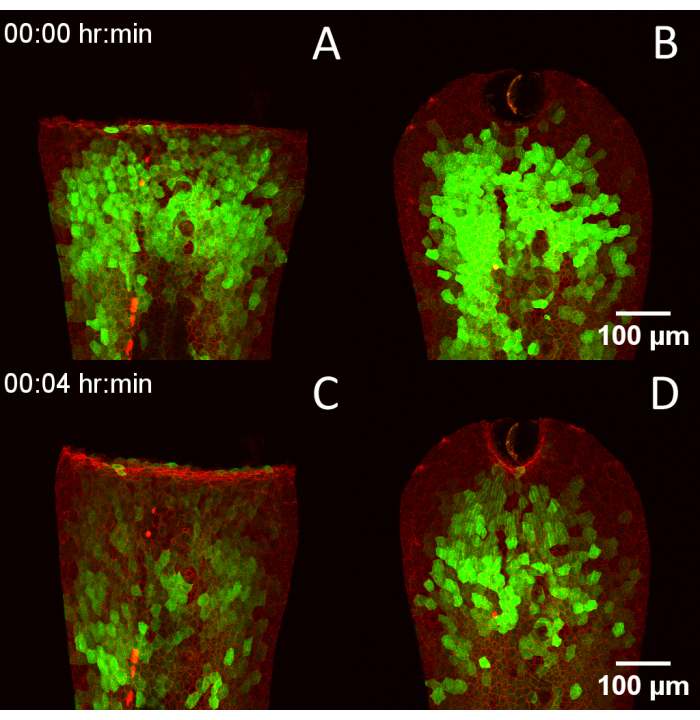
Abbildung 4: Kompatibilität des RADISH-Designs mit mehreren Verwundungstechniken. (A) 3 dpf Tg(krt4:Gal4, UAS:GCaMP6f) x (cdh1-dTomato)xt18 Embryo, der durch manuelle Schwanzflossendurchtrennung verwundet wurde. (B) Embryo, der durch Laserablation verletzt wurde. (C,D) Die gleichen Embryonen 4 Minuten später, die das Fortschreiten der Kalziumtransienten nach der Verwundung zeigen. Maßstabsleisten = 100 μm. Abkürzungen: RADISH = Rotationsassistent für die Bildgebung der anschließenden Heilung; dpf = Tage nach der Befruchtung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Langzeiteinschluss von Embryonen für die Bildgebung der Phagozytenmigration. (A) Tol2(mpx:Dendra2)- Embryonen unmittelbar nach der Verwundung und dieselben Embryonen (B) 25 min, (C) 12 h und (D) 24 h später. Maßstabsbalken = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Vektor- und PNG-Dateien für das Design von RADISH-Geräten, wie in Abbildung 1A gezeigt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Video 1: Hellfeld-Video der manuellen Heckflossendurchtrennung mit dem RADISH-Gerätedesign. Abkürzung: RADISH = Rotational Assistant für Danio Bildgebung der nachfolgenden Heilung. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Video 2: Vergleich von ungefassten und eingerahmten 3 dpf Tg(krt4:Gal4, UAS:GCaMP6f) Embryonen aus Abbildung 3, aufgenommen mit 1 frame/min. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Video 3: Calciumtransienten in Tg(krt4:Gal4, UAS:GCaMP6f) x (cdh1-dTomato)xt18 Embryonen, die durch Skalpelldurchtrennung oder Laserablation verwundet wurden, aus Abbildung 4, aufgenommen bei 1 frame/min. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Video 4: Langzeitbildgebung der Leukozytenrekrutierung und der Regeneration der Schwanzflosse in Tol2(mpx:Dendra2)- Embryonen. Abkürzungen: RADISH = Rotationsassistent für die Bildgebung der anschließenden Heilung; HPW = Stunden nach der Verwundung. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Diskussion
Die Kernprämisse des PET-Laminierungsverfahrens ist die Kostensenkung und Minimierung technischer Eintrittsbarrieren im Vergleich zu herkömmlichen Mitteln zur Herstellung mikrofluidischer Geräte, wie z. B. Softlithographie oder PDMS-Spritzguss. Daher sind die einzigen kritischen Schritte in diesem Protokoll die genaue Ausrichtung der PET-Schichten während der Laminierung und die Abdichtung der Gerätekanten nach der Konstruktion. Alle anderen Teile des Verfahrens können je nach experimentellem Bedarf modifiziert werden (z. B. wenn eine Immobilisierung jüngerer oder älterer Embryonen erforderlich ist, kann die Dicke der PET-Schichten angepasst werden, um den Kanal entsprechend zu verengen oder zu erweitern). Designs können in jeder Form erstellt und auf jedes Grundmaterial laminiert werden, solange das Material hitzebeständig und dünn genug ist, um durch das Laminiergerät geschickt zu werden18,27. Da PET-Platten mit handelsüblichen Bastelschneidern geschnitten werden, die zweidimensionale statt dreidimensionale Bilddateien lesen, erfordern das Design und die Iteration eines bestimmten Geräts nur die Fähigkeit, Schwarz-Weiß-Formen in einem Bildbearbeitungsprogramm zu bearbeiten, was die Schwierigkeit drastisch verringert. Diese Flexibilität bei der Auswahl der Software und die kommerzielle Verfügbarkeit der verwendeten Materialien stellen eine dramatische Verbesserung der Zugänglichkeit von Mikrofluidik-Geräten im Allgemeinen dar und ermöglichen ihren Einsatz in jedem Labor oder Klassenzimmer zu Vorabkosten von weniger als 500 US-Dollar zum Zeitpunkt der Erstellung dieses Papiers, verglichen mit über 2000 US-Dollar an Startausrüstungskosten für die Herstellung von PDMS-Formen17.
Darüber hinaus wird der Zeitaufwand, der für die Erstellung eines einzigen nutzbaren Geräts mit dieser Methode benötigt wird, im Vergleich zur typischen Softlithographie drastisch reduziert. Unter der Voraussetzung, dass Sie mit der erforderlichen Software vertraut sind, kann ein einzigartiges mikrofluidisches Gerät für PET-Laminat in ~3 h konzipiert, hergestellt und sofort verwendet werden, einschließlich der Aushärtungszeit von Cyanacrylat. Die Iteration dauert sogar noch weniger, abhängig vom Umfang der erforderlichen Änderungen an den ursprünglichen Designdateien, und mehrere Varianten können parallel mit minimalem Zeitaufwand und ohne zusätzliche Ausrüstung hergestellt werden. Obwohl Geräte, die mit dieser Methode hergestellt wurden, aus verschiedenen Gründen weniger haltbar sind als solche, die mit PDMS hergestellt wurden (einschließlich Klebeversagen und innerer Spannungen bei Formen mit vielen Schichten und langen Seitenverhältnissen), bedeuten ihre kostengünstige Natur und skalierbare Herstellung, dass sie viel einfacher ausgetauscht werden können. Da kommerzielle Schneideplotter mit der Möglichkeit vermarktet werden, denselben präzisen Schnitt immer wieder zu wiederholen, und PET kein Verfallsdatum hat, können die einzelnen Schichten eines fertigen Designs in großen Mengen für die Montage und Verwendung zu einem beliebigen Zeitpunkt in der Zukunft vorbereitet werden, wodurch der Gesamtzeitaufwand weiter reduziert wird.
Geschwindigkeit und Variabilität gehen jedoch auf Kosten der Präzision. Die Haupteinschränkung der PET-Laminierung im Vergleich zum herkömmlichen PDMS-Spritzguss ist die räumliche Auflösung. Kommerzielle Bastelschneider haben oft Probleme mit scharfen Winkeln bei Formen, die kleiner als ca. 500 μm sind, und PET-Kaschierungsfolien sind bei Dicken unter 33 μm nicht erhältlich (ohne die Dicke des Klebstoffs selbst, die nicht in der Messung auf der Verpackung enthalten ist), was den Maßstab, bei dem diese Technik angewendet werden kann, erheblich einschränkt. Die maximale Anzahl der Schichten ist auch durch die Größe des Kaschiergeräts begrenzt, die von Hersteller zu Hersteller variieren kann.
Die endgültige Version des Protokolls, einschließlich Variationen in der Registrierungs- und Ausrichtungstechnik, den Materiallieferanten und der Software, hängt weitgehend von den Benutzerpräferenzen ab. Die Zugänglichkeit und die allgemeine Flexibilität der Methode reduzieren jedoch die Kosten für die Anpassung. Einzelne Benutzer werden ermutigt, so viel wie nötig zu iterieren, um den Ansatz an ihre individuellen Bedürfnisse anzupassen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Arbeit wurde durch Forschungsgelder der National Institutes of Health (R35GM119787 bis QD) unterstützt. Diese Arbeit basiert auf Bemühungen, die vom EMBRIO Institute, NSF-Vertrag #2120200, einem Biology Integration Institute der National Science Foundation (NSF), unterstützt werden. Die konfokale Bildgebung wurde in der Purdue Imaging Facility durchgeführt. Wir danken Dr. Guangjun Zhang (Purdue University) für die Bereitstellung der Tg(UAS:GCaMP6f)-Linie. Wir danken Dr. David Tobin (Duke University) für die Bereitstellung der (cdh1-tdtomato)xt18-Linie.
Materialien
| Name | Company | Catalog Number | Comments |
| 25 mm diameter round coverglass #1 thickness | Chemglass Life Sciences | CLS-1760-025 | Base mounting material. Thickness was chosen based on the specifications of the microscope lens. |
| Adobe Illustrator v28.5 | Adobe | N/A | Vector editor for designing the device pattern. |
| Calcium Chloride Dihydrate | Fisher | C79 | For making E3 medium. |
| Design PNG Files (Online Mirror) | N/A | N/A | https://i.ibb.co/QPYj7BL/multipositionalv7-1.png https://i.ibb.co/FDpQYXc/multipositionalv7-2.png https://i.ibb.co/5TG4SB6/multipositionalv7-3.png |
| Design Space for Desktop v8.39 | Cricut | N/A | Proprietary software to drive the craft cutter. |
| Fusion Plus 7000L | GBC | 1703098 | Thermal laminator. This specific model was selected for quality of life features, such as maximum sheet stack size. |
| German Carbide Premium Blade | Cricut | 10396595 | Replacement blade for craft cutter. |
| Gray Basic Tool Set | Cricut | 10307854 | Tool set for weeding cuts, cleaning mats, and mounting PET sheets. Optional. |
| Magnesium Chloride Hexahydrate | Acros Organic | 223211000 | For making E3 medium. |
| Maker 3 | Cricut | 10669040 | Craft cutter. The Cricut Maker 3 was selected over the Silhouette Cameo 4 for software user friendliness, but any craft cutter capable of reading black and white images will work. |
| MemGlow 560 | Cytoskeleton | MG02-10 | Red live cell dye for cell membranes. No washing needed. Used in Figure 3. |
| No. 22 Carbon Scalpel Blade | Surgical Design | 22-079-697 | Scalpel blade for manual cut adjustments. Any similar craft knife (e.g., X-acto #2 knife) will also work. |
| Potassium Chloride | Fisher | BP366 | For making E3 medium. |
| Sodium Chloride | Fisher | BP358 | For making E3 medium. |
| StandardGrip Adhesive Cutting Mat | Cricut | 10138842 | Mounting mat for craft cutter. The LightGrip cutting mat will also work, but avoid the StrongGrip and FabricGrip cutting mats as the strength of the adhesive may warp the final cut during weeding and removal. Adhesive on mats will eventually wear out with continued use; replace mats as needed. |
| Stationery Tape 12 mm | Deli | 30014 | Office tape. Deli brand was chosen via testing for ease of removal after lamination. |
| Super glue, liquid | Loctite | 1364076 | Cyanoacrylate glue. Glue used must be liquid. Gel formulations will not sufficiently seal or waterproof device edges. |
| PET Thermal Laminating Pouches, 3 mil | Scotch | TP3854 | PET sheets with thermal adhesive. Sheets arrive as sandwiched pairs and should be separated before use. Cut with adhesive side facing down. |
| PET Thermal Laminating Pouches, 5 mil | Scotch | TP5854 | PET sheets with thermal adhesive. Sheets arrive as sandwiched pairs and should be separated before use. Cut with adhesive side facing down. |
| Tricaine (MS-222) | Syndel | IC10310680 | Fish anaesthetic. |
Referenzen
- De Oliveira, S., Rosowski, E. E., Huttenlocher, A. Neutrophil migration in infection and wound repair: going forward in reverse. Nat Rev Immunol. 16 (6), 378-391 (2016).
- Xu, S., Hsiao, T. I., Chisholm, A. D. The wounded worm. Worm. 1 (2), 134-138 (2012).
- Belacortu, Y., Paricio, N. Drosophila as a model of wound healing and tissue regeneration in vertebrates. Dev Dyn. 240 (11), 2379-2404 (2011).
- Yoo, S. K., Freisinger, C. M., LeBert, D. C., Huttenlocher, A. Early redox, Src family kinase, and calcium signaling integrate wound responses and tissue regeneration in zebrafish. J Cell Biol. 199 (2), 225-234 (2012).
- Kawakami, A., Fukazawa, T., Takeda, H. Early fin primordia of zebrafish larvae regenerate by a similar growth control mechanism with adult regeneration. Dev Dyn. 231 (4), 693-699 (2004).
- Gault, W. J., Enyedi, B., Niethammer, P. Osmotic surveillance mediates rapid wound closure through nucleotide release. J Cell Biol. 207 (6), 767-782 (2014).
- Huemer, K., et al. zWEDGI: Wounding and entrapment device for imaging live zebrafish larvae. Zebrafish. 14 (1), 42-50 (2017).
- Kaufmann, A., Mickoleit, M., Weber, M., Huisken, J. Multilayer mounting enables long-term imaging of zebrafish development in a light sheet microscope. Development. 139 (17), 3242-3247 (2012).
- Hirsinger, E., Steventon, B. A versatile mounting method for long term imaging of zebrafish development. J Vis Exp. (119), e55210 (2017).
- Wielhouwer, E. M., et al. Zebrafish embryo development in a microfluidic flow-through system. Lab Chip. 11 (10), 1815-1824 (2011).
- Khalili, A., Rezai, P. Microfluidic devices for embryonic and larval zebrafish studies. Brief Funct Genomics. 18 (6), 419-432 (2019).
- Choudhury, D., et al. Fish and Chips: a microfluidic perfusion platform for monitoring zebrafish development. Lab Chip. 12 (5), 892-900 (2012).
- Zhang, G., et al. An integrated microfluidic system for zebrafish larva organs injection. , 8563-8566 (2017).
- Erickstad, M., Hale, L. A., Chalasani, S. H., Groisman, A. A microfluidic system for studying the behavior of zebrafish larvae under acute hypoxia. Lab Chip. 15 (3), 857-866 (2015).
- Mani, K., Hsieh, Y. C., Panigrahi, B., Chen, C. Y. A noninvasive light driven technique integrated microfluidics for zebrafish larvae transportation. Biomicrofluidics. 12 (2), 021101 (2018).
- Lee, Y., Seo, H. W., Lee, K. J., Jang, J. W., Kim, S. A microfluidic system for stable and continuous EEG monitoring from multiple larval zebrafish. Sensors (Basel). 20 (20), 5903 (2020).
- Sonnen, K. F., Merten, C. A. Microfluidics as an emerging precision tool in developmental biology. Dev Cell. 48 (3), 293-311 (2019).
- Levis, M., et al. Microfluidics on the fly: Inexpensive rapid fabrication of thermally laminated microfluidic devices for live imaging and multimodal perturbations of multicellular systems. Biomicrofluidics. 13 (2), 024111 (2019).
- . E3 medium (for zebrafish embryos). Cold Spring Harbor Protocols. 2011 (10), (2011).
- Matthews, M., Varga, Z. M. Anesthesia and euthanasia in zebrafish. ILAR J. 53 (2), 192-204 (2012).
- Cronan, M. R., Tobin, D. M. Endogenous tagging at the cdh1 locus for live visualization of E-cadherin dynamics. Zebrafish. 16 (3), 324 (2019).
- Thiele, T. R., Donovan, J. C., Baier, H. Descending control of swim posture by a midbrain nucleus in zebrafish. Neuron. 83 (3), 679-691 (2014).
- Yoo, S. K., Huttenlocher, A. Spatiotemporal photolabeling of neutrophil trafficking during inflammation in live zebrafish. J Leukoc Biol. 89 (5), 661-667 (2011).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Li, L., Yan, B., Shi, Y. -. Q., Zhang, W. -. Q., Wen, Z. -. L. Live imaging reveals differing roles of macrophages and neutrophils during zebrafish tail fin regeneration. J Biol Chem. 287 (30), 25353-25360 (2012).
- Mathias, J. R., et al. Resolution of inflammation by retrograde chemotaxis of neutrophils in transgenic zebrafish. J Leukoc Biol. 80, 1281-1288 (2006).
- Levis, M., Ontiveros, F., Juan, J., Kavanagh, A., Zartman, J. J. Rapid fabrication of custom microfluidic devices for research and educational applications. J Vis Exp. 2019, (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten