Method Article
Genetik Olarak Yeniden Programlanmış Murin Melanomundan FFPE Örneklerini Analiz Etmek için DNA Barkod Tabanlı Multipleks İmmünofloresan Görüntüleme
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu protokol, murin FFPE melanom dokularının DNA barkod tabanlı görüntülenmesi için bir multipleks immünofloresan antikor paneli tasarlamak için adım adım bir kılavuz sağlar. Ayrıca, murin melanom tümörü bağışıklık mikro ortamına ilişkin uzamsal proteomik içgörüler oluşturmak için açık kaynaklı araçlar kullanan bir görüntü analizi boru hattını da açıklıyoruz.
Özet
Burada, doku mikro ortamlarının uzamsal proteomiklerini analiz eden Co-Detection-by-indEXing'e dayalı, gelişmekte olan bir DNA barkod tabanlı multipleks görüntüleme tekniği sunulmaktadır. Başarılı görüntüleme, iyi tasarlanmış ve uygun şekilde doğrulanmış antikor panellerinden oluşan bir repertuar gerektirir, ancak formalinle sabitlenmiş parafine gömülü (FFPE) örnekler için şu anda çok azı mevcuttur. FFPE, taze dondurulmuş numunelere göre yaygın bulunabilirlik, kullanım ve saklama kolaylığı ve doku mikrodizileri (TMA'lar) yapma yeteneği gibi çeşitli avantajlar sunar. Burada, tümör mikroçevresinin yeniden programlanması için plazmit DNA kodlayan immünolojik sinyalleri ileten nanopartiküllerle tedavi edilmiş bir murin melanom modelinden FFPE dokularını görselleştirmek ve analiz etmek için bir antikor paneli geliştirmek için bir protokol sunuyoruz. Ayrıca, dokulara açıklama eklemek, hücreleri segmentlere ayırmak, proteomik verileri işlemek, hücre popülasyonlarını fenotiplemek ve uzamsal ölçümleri ölçmek için açık kaynaklı hesaplama araçlarını kullanan bir görüntü analizi boru hattını da açıklıyoruz. Protokol, murin FFPE'de antikor panelleri tasarlamak ve karmaşık doku mikro ortamlarının mekansal proteomikleri hakkında yeni bilgiler üretmek için uygulamalar sunar.
Giriş
Kutanöz melanom en sık görülen cilt kanseridir ve tanı zamanına ve birinci basamak sağlık hizmetine bağlı olarak dünya genelinde değişen hastalık ve mortalite oranları ile1. Son on yılda, melanomun biyolojik olarak anlaşılmasının artması, katı tümörleri tedavi etmek için yeni kanser modellerinin geliştirilmesine yardımcı olmuştur2. İmmünoterapinin son zamanlardaki yükselişi, endojen bağışıklık sistemini aktive etmeye dayanan devrim niteliğinde bir kanser tedavisi konseptine yol açmıştır 3,4.
Tümör mikroçevresi (TME), çeşitli bağışıklık hücreleri, kanserle ilişkili fibroblastlar, perisitler, endotel hücreleri ve çeşitli dokuda yerleşik hücrelerden oluşan oldukça karmaşıktır5. Geçmişte, TME'yi incelemek için akış sitometrisi ve tek hücre dizilimi gibi, tümör dokusunu yok etmek için gerekli oldukları için uzamsal bağlamı tehlikeye atan çeşitli teknikler uygulanmıştır. İmmünofloresan (IF) ve immünohistokimya (IHC) gibi geleneksel mikroskop görüntüleme, numune dokularını tahrip etmeden protein biyobelirteçlerinin görselleştirilmesine olanak tanır. Bununla birlikte, bu yaklaşımlar iki veya üç biyobelirteç ile sınırlıdır ve karmaşık TME 6 içindeki mekansal ve yapısal ilişkilerin tam olarak anlaşılmasını sağlayamaz.
Bu sorunu ele almak için, kompleks TME'yi mekansal olarak görselleştirmek için çeşitli multipleks görüntüleme teknikleri geliştirilmiştir 7,8,9,10. Bunlardan biri, DNA oligonükleotid konjuge antikorlarına11 dayanan PhenoCycler sistemi olarak yeniden adlandırılan CoDetection-by-inDEXing'dir. Sistem, insan örnekleri için 100'den fazla biyobelirteçin tek hücreli görüntülemesini ve analizini sağlayabilir. Bununla birlikte, murin örneklerini, özellikle Formalin-Sabit Parafin-Gömülü (FFPE) numuneleri görselleştirmek ve analiz etmek için çok az sayıda envantere alınmış antikormevcuttur.12. FFPE, Taze Dondurulmuş (FF) korumaya göre, kullanım ve saklama kolaylığı, zaman içinde iyi korunmuş morfoloji ve en önemlisi, tek bir slaytta birkaç örneğin görselleştirilmesine izin veren doku/tümör mikrodizileri (TMA) hazırlama yeteneği gibi çeşitli avantajlar sunar. Yakın zamanda bir murin FFPE CODEX / PhenoCycler antikor paneli tasarladık ve geliştirdik ve genetik olarak yeniden programlanmış murin melanom örneklerinin uzamsal proteomiklerini görselleştirmek ve analiz etmek için başarıyla uyguladık13.
Bu protokolün genel amacı, bir murin FFPE antikor paneli tasarlamak için adım adım bir kılavuz sağlamak ve antikor-barkod konjugasyonu, doku boyama ve görüntüleme sürecini tanımlamaktır. Ek olarak, QuPath ve R paketleri gibi açık kaynaklı araçları kullanan ayrıntılı bir görüntü analizi hattı sunuyoruz. Bu protokolü izledikten sonra, araştırmacılar özel konjuge bir antikor panelinin nasıl tasarlanacağını, bir Phenocycler-Fusion cihazı kullanarak multipleks görüntülemenin nasıl gerçekleştirileceğini ve melanom TME'nin uzamsal proteomiği hakkında yeni bilgiler kazanmayı öğrenecekler. Ayrıca, bu protokol çeşitli tümör immün mikroçevrelerini incelemek için uyarlanabilir ve mevcut mekansal transkriptomik tekniklerle birleştirilebilir.
Protokol
Tüm hayvan çalışmaları, Johns Hopkins Hayvan Bakım ve Kullanım Komitesi tarafından belirlenen yönergelere uygun olarak, onaylanmış protokol numaraları MO18M388 ve MO21M384 kullanılarak gerçekleştirildi.
1. Antikor seçimi
- İlgilenilen dokuya ve belirli biyobelirteçin bolluğuna dayalı bir antikor paneli tasarlayın. Literatürdeki başarılı immünofloresan (IF) ve immünohistokimya (IHC) boyama uygulamalarına dayanarak antikor klonlarını seçin. Bu antikorların taşıyıcısız versiyonlarını güvenli hale getirin.
NOT: Taşıyıcıdan arındırılmış saflaştırılmış bir antikor, barkod konjugasyonu için önemlidir. BSA, glüten, gliserol vb. gibi protein bazlı koruyuculardan kaçının. Taşıyıcısız antikor mevcut değilse, tedarikçiden özel bir antikor isteyin veya taşıyıcısız antikoru yapmak için bir BSA çıkarma kiti kullanın. Sodyum azid, antikor konjugasyonuna müdahale etmez. IgM izotipleri yerine IgG izotipleri önerilir. - Konvansiyonel immünofloresan (IF) görüntüleme11 kullanarak bu seçilen klonları ilgilenilen doku üzerinde doğrulayın. Belirli klonların özgüllüğünü doğrulamak için çift IF görüntüleme kullanın (örneğin, düzenleyici T hücreleri için boyamayı doğrulamak için FOXP3 ve CD4 ile çift IF görüntüleme yapın).
NOT: Mümkün olduğunda, boyama için kullanılacak olan IF doğrulaması için aynı dokunun kullanılması önemlidir. Uygun antijen alma solüsyonunu seçin. Bu protokolde, tüm murin melanom FFPE doku epitoplarını almak için Akoya Biosciences tarafından sağlanan bir AR9 tamponu kullandık. AR6 veya Universal gibi diğer antijen alma solüsyonları, epitop gereksinimlerine bağlı olarak kullanılabilir.
2. Antikor konjugasyonu ve doğrulanması
- ATTO550 (Cy3), AF647 (Cy5) veya AF750 (Cy7) floroforlarına bağlı ücretsiz raporlayıcıları olan barkodlara doğrulanmış antikorlar atayın. Düşük miktarda antijenler genellikle daha düşük sinyaller üretir; bu tür antikorları AF647 (Cy5) gibi düşük otofloresan kanallarına konjuge edin. Otofloresan olasılığı nedeniyle ATTO550 (Cy3) ve AF750'ye (Cy7) yüksek oranda eksprese edilen antijenleri konjuge edin.
NOT: Bu kritik adım, kanal duyarlılığını ve antijen bolluğunu dikkate alır. Daha yüksek otofloresansı nedeniyle FFPE dokusu için AF488 kanalının kullanılması önerilmez. - Antikor-barkod konjugasyonu (4,5 saat) için antikor konjugasyon kitini edinin. -20 °C'de tek kullanımlık küçük şişeler olarak saklanması gereken indirgeme çözeltisi 1 hariç tüm reaktifleri 4 °C'de saklayın.
- Bir spektrofotometre kullanarak antikor konsantrasyonunu ölçün.
- Antikorların filtreye spesifik olmayan bağlanmasını bloke etmek için 50 kDa MWCO'luk bir filtre kolonuna 500 μL filtre bloke edici solüsyon uygulayarak antikor konjugasyonunu başlatın. Oda sıcaklığında (RT) 2 dakika boyunca 12.000 x g'da döndürün. 200 μL'lik bir mikropipet kullanarak kolondaki fazla sıvıyı çıkarın.
NOT: İndirgeme çözeltisi 1, bir seferde üç antikor konjugasyonu için yeterli olan tek kullanımlık bir çözelti şişesidir. Çözüldükten sonra çözeltiyi tekrar kullanmayın; Kalan reaktifleri atın. 50 kDa MWCO filtre sütunu üzerinde filtre kullanmayın, çünkü bu zayıf saflaştırma ve konjugasyona neden olabilir. - 50 μg eşdeğer hacimde antikor çözeltisi kullanın ve 50 kDa MWCO filtresine ekleyin. Gerekirse PBS kullanarak toplam hacmi 100 μL'ye ayarlayın. 4 ° C'de 8 dakika boyunca 12.000 x g'da döndürün. Bu süre zarfında, üç antikor konjugasyonu için 19.8 μL indirgeme çözeltisi 1 + 825 μL indirgeme çözeltisi 2 kullanarak indirgeme ana karışımını hazırlayın.
- Akışı atın ve her bir filtre ünitesinin üstüne 260 μL indirgeme ana karışımı ekleyin. Filtre ünitesinde 2-3 saniye vorteks çözeltisi ve RT'de 30 dakika inkübe edin.
NOT: Antikorların aşırı azalmasını önlemek için 30 dakikalık inkübasyonu aşmamak çok önemlidir, çünkü bu onlara zarar verir ve başarısız konjugasyona neden olabilir. - 30 dakikalık inkübasyondan sonra, 4 ° C'de 8 dakika boyunca 12.000 x g'da döndürün ve alttan akışı atın. Kolonun üstüne 450 μL konjugasyon çözeltisi ekleyin ve 4 ° C'de 8 dakika boyunca 12.000 x g'da döndürün. Santrifüjleme sırasında CODEX barkod çözeltisini hazırlayın. Barkodu -20 °C'den aldıktan sonra hızlı hareket edin, çünkü barkod bozulmaya başlar.
NOT: Barkodlar genellikle -20 °C'de saklanan liyofilize ürünler (pul veya toz) içeren küçük cam şişelerde sağlanır. Bu şişeler bir kerede 50 μg antikoru konjuge etmek için kullanılabilir. Bir seferde üçten fazla antikorun konjuge edilmemesi önerilir. - Cam şişenin altındaki liyofilize barkodu dikkatlice bulun (duvarlara da yapışmış olabilir). Katıları dibe getirmek için cam şişeyi bir masanın üzerine hafifçe vurun. Liyofilize ürünün yerinin belirlenmesi, 10 μL nükleaz içermeyen moleküler biyoloji dereceli suda çözülürken çok önemlidir. 10 μL nükleaz içermeyen su ekledikten sonra, her barkoda 210 μL konjugasyon solüsyonu ekleyin. Tüm liyofilize malzemeleri çözün ve yukarı ve aşağı pipetleyerek nazikçe karıştırın. Kenara.
- Adım 2.2.5'teki dönüşü tamamladıktan sonra, akışı atın ve adım 2.2.6'daki ilgili barkod çözümünü her bir filtre ünitesinin üstüne ekleyin. İlk konjuge olmayan antikorun 1 μg'ını kaydedin; bu, jel elektroforez validasyonu sırasında konjuge 5 μL antikor numunesi ile birlikte çalıştırılacaktır.
- Her tüpü ilgili antikor ve barkod adıyla etiketleyin. Kapağı kapatın ve çözeltiyi 2-3 saniye girdaplayarak karıştırın. Antikor-barkod konjugasyon reaksiyonunu RT'de 2 saat inkübe edin.
- 2 saatlik inkübasyondan sonra, 5 μL konjuge antikoru çıkarın ve jel elektroforezi ile konjugasyonu doğrulamak için 0.2 mL'lik bir PCR tüpünde saklayın.
- Kalan konjuge antikor çözeltisini 12.000 x g'da 4 ° C'de 8 dakika boyunca döndürün. Kolonun üstüne 450 μL arıtma solüsyonu ekleyin, 4 ° C'de 8 dakika boyunca 12.000 x g'da döndürün ve akışı atın. Bu saflaştırma adımını 2 kez tekrarlayın, her seferinde 450 μL saflaştırma solüsyonu ekleyin ve aşağı döndürün.
- 3. santrifüjlemeden sonra, akışı atın. Üst filtre, barkodla konjuge antikorları içerecektir.
- Konjuge antikoru toplamak için, filtre sütununu tutan yeni dış tüpü karşılık gelen antikor ve barkodla etiketleyin. Santrifüjlemeden önce dış tüpün kapağını kesin. Her filtre ünitesine 100 μL antikor depolama solüsyonu ekleyin ve yeni etiketlenmiş dış tüpü filtre kolonunun üzerine baş aşağı yerleştirin.
- Konjuge antikoru dış tüpe toplamak için filtre kolonunu ters çevirin ve RT'de 2 dakika boyunca 3.000 x g'da döndürün. Bu, yaklaşık 120 μL konjuge antikor çözeltisi toplamalıdır. Steril bir mikrosantrifüj tüpüne aktarın ve 4 ° C'de 18-24 ay saklayın.
NOT: Konjugasyondan sonra yüksek arka plan nükleer lekelenmesini önlemek için, bu antikorların en az 2 gün kullanılmaması önerilir.
- Daha önce yayınlanmış protokol13'te açıklandığı gibi jel elektroforezi ile konjugasyon onayını gerçekleştirin.
NOT: Bu işlem yalnızca barkodları antikorlarla konjuge etmek için kullanılan kimyasal konjugasyon reaksiyonunun başarısını doğrular. Antikor validasyonu ancak dokunun başarılı bir şekilde boyanması ve görüntülenmesinden sonra mümkün olduğu için isteğe bağlıdır.
3. Murin FFPE numune hazırlama
- B16F10 yan tümörleri implante edilmiş C57BL / 6J dişi farelerden FFPE doku örnekleri alın. Fareleri, implantasyondan 9, 11, 16 ve14 gün sonra intraperitoneal anti-PD1 uygulaması ile birlikte 4-1BBL ve IL-12 plazmitleri içeren poli (β-amino ester) bazlı nanopartiküllerin intratümöral enjeksiyonları ile tedavi edin. 20. günde fareleri kurban edin ve tümörleri parafine gömülü% 10 formalin içinde düzeltin, ardından Johns Hopkins Onkoloji Doku Hizmetleri merkezinde bir bölüm.
- Kullanım kılavuzunda verilen görüntüleme alanı özelliklerini dikkatlice değerlendirin ve 5 μm'lik numuneleri görüntüleme alanının içindeki bir slayta monte edin. Dört farklı tümör örneğini tek bir slayta monte edebildik.
NOT: Leica Apex Yapışkan Slaytlar veya Fisherbrand Superfrost Plus Slaytlar gibi görüntüleme için mevcut olan önerilen slaytların kullanılması önemlidir. Bu, akış hücresinin kızağa sıkıca yapışmasına yardımcı olacaktır.
4. FFPE doku boyama ve görüntüleme
- Antikor konsantrasyonunun ve maruz kalma sürelerinin doğrulanması ve optimizasyonu: Antikor-barkod konjugasyonunun başarısını doğrulamak için, ilgilenilen dokuyu 6-9 konjuge antikorla boyayın. Multipleks görüntüleme cihazını çalıştırın ve her bir antikor konsantrasyonunu ve maruz kalma süresini not edin. Seyreltmeleri ve maruz kalma sürelerini gerektiği gibi ayarlayın, ardından daha önce doğrulanmış olanlarla 6-9 yeni antikor çalıştırın ve herhangi bir ayarlamayı not edin. Boyama ve görüntüleme için aşağıdaki adımları izleyin.
- Doku boyama ve görüntüleme
- Boyamadan bir gün önce, FFPE doku slaytlarını gece boyunca 60 °C fırında pişirin. Ertesi gün, deparafinizasyon ve doku rehidrasyon adımlarına başlamadan önce slaytları RT'de 10 dakika soğutun.
- Her biri 5 dakika boyunca 2 kez bir ksilen çözeltisi içinde inkübe ederek dokunun deparafinizasyonuna başlayın.
- Dokuyu yeniden sulandırmak için, her biri 5 dakika boyunca iki tur %100 etanol çözeltisinden geçirin. Ardından, her biri 5 dakika boyunca %90'dan %70'e, %50 ve %30'a kadar bir dizi alkol solüsyonundan geçirin. Son olarak, dokuyu her biri 5 dakika boyunca iki kez damıtılmış suyla yıkayın.
NOT: Deparafinizasyon adımları için Neoclear çözeltisi gibi ksilene daha az toksik alternatifler kullanılabilir. Çeker ocak altında deparafinizasyon ve rehidrasyon gerçekleştirin. - Damıtılmış su kullanarak 10x AR9 tamponunu 1x'e seyreltin. Bir Coplin kavanozunu 50 mL 1x AR9 tamponu ile doldurun ve slaytların tamamen antijen alma tamponuna daldırıldığından emin olun.
- Bir düdüklü tencereyi, seviye Coplin kavanozunun yarısına gelecek şekilde suyla doldurun, ardından yüksek basınç ayarında 20 dakika inkübe edin.
NOT: 10 dakika boyunca 120 °C'ye ayarlayarak daha iyi antijen alımı için özel bir düdüklü tencere veya Boşaltma Odası kullanmak mümkündür. - Düdüklü tencere adımını tamamladıktan sonra slaytları 45-60 dk kadar soğutun. Slaytı 2 kez damıtılmış suyla 2 dakika durulayın. Ardından, slaytı 1x PBS içeren kavanoza aktarın.
- Otofloresanı en aza indirmek için, antikorlarla boyamadan önce doku üzerinde bir ağartma adımı gerçekleştirin. Önerilen reaktifleri ve konsantrasyonları kullanarak taze bir ağartma çözeltisi (25 mL 1x PBS + 0.8 mL 1M NaOH + 4.5 mLH2O2) hazırlayın. Slaytları, ağartma solüsyonunu içeren plastik bir kapta, iki LED lamba arasında oda sıcaklığında 45 dakika inkübe edin. Taze ağartma solüsyonu ile 45 dakika daha tekrarlayın.
- Bu ağartma adımı sırasında, özellikle panelde çeşitli seyreltmelere sahip birkaç antikor varsa, antikor kokteyl solüsyonunu hazırlamaya başlayın. Dört engelleyiciyi de (G, S, J ve N) alın ve bazılarının -20 °C'den çözülmesi zaman alacağından buzun üzerine yerleştirin.
- Foto ağartma işleminden sonra, dokuyu 1x PBS ile 2x, her biri 2 dakika boyunca yıkayın. Dokuyu hidrasyon tamponu içeren kavanozlara taşıyın ve her birini 2 dakika boyunca 2 kez yıkayın.
- Doku slaytlarını boyama tamponuna aktarın ve 30 dakika boyunca dengelenmelerine izin verin. Ağartma aşaması sırasında hazırlanmadıysa, bu süre zarfında antikor kokteyl solüsyonunu hazırlayın.
- İki slaytı boyamak için 300 μL antikor kokteyli çözeltisi hazırlayın (genellikle, bir hibridizasyon plastik odası kullanılıyorsa, her slaytı lekelemek için 100-120 μL antikor kokteyli yeterlidir.)
- Gece boyunca 4 °C'de nem odasının içine bir antikor kokteyl solüsyonu uygulayarak slaytları lekeleyin.
NOT: Buradaki tüm antikorlar, gece boyunca 4 °C'de boyandığında pozitif bir sinyal gösterir. Bununla birlikte, her antikorun boyama süresi ve sıcaklığı değişebilir ve optimize edilmelidir. En sık kullanılan boyama koşulları RT'de 3 saat veya gece boyunca 4 °C'dir. Gerekirse, başlangıçta farklı sıcaklıklarda birkaç antikoru ve daha sonra kalan antikorları gece boyunca 4 °C'de boyamak da mümkündür. Sıralı boyama gerekiyorsa dokuyu daha sonra PBS ile yıkayın. - Ertesi gün, boyama sonrası fiksasyon yapın. Plastik hazneyi çıkarın ve toplanan antikor kokteylini 4 °C'de saklayın. Slaytları 2 kez boyama tamponunda her biri 2 dakika yıkayın. Ardından, slaytları 40 mL'lik bir boyama sonrası sabitleme solüsyonuna (4 mL %16 Paraformaldehit + 36 mL depolama tamponu) taşıyın ve 10 dakika inkübe edin. PBS 3x ile her biri 2 dakika durulayın.
- Slaytları 5 dakika buz gibi soğuk metanole koyun, ardından PBS 3x ile her biri 2 dakika durulayın. Bir nem odası altındaki slaytlara son bir fiksatif solüsyon (20 μL bir tüp + 1 mL PBS) uygulayın. Genellikle, slayt başına 200 μL fiksatif çözelti yeterlidir. Sabitleyicinin RT'de 20 dakika oturmasına izin verin. PBS'de her biri 2 dakika boyunca üç son yıkama gerçekleştirin.
NOT: Fiksatif tüpü önceden -20 °C'den çıkarmayın; Sadece uygulamadan önce çözdürün. - Hemen görüntülüyorsanız, tüy bırakmayan bir mendil kullanarak dokunun etrafındaki slaytı silin ve akış hücresini, akış hücresi montaj ekipmanı kullanarak 30 saniye boyunca bastırarak uygulayın. Daha sonra görüntülüyorsanız, slaytı (akış hücresini uygulamadan) 4 °C'de depolama tamponunda saklayın. Görüntüye hazır olduğunuzda, akış hücresini uygulamadan önce slaytları depolama arabelleğinden 10 dakika boyunca PBS'ye aktarın.
- Deneyde istenen döngülere göre katkı maddesi ile 1x çalışma tamponunu hazırlamaya başlayın. İstenen döngüler için gerekli olan 4 parçalı çalışma arabelleği ile 1 parçalı DMSO ve 4 parçalı çalışma arabelleği ile 9 parçalı DMSO olarak karıştırarak düşük (1:4) ve yüksek DMSO tamponları (9:1) hazırlayın. Bir muhabir plakası yapmak için katkı maddesi ile yaklaşık 20 mL 1x tampon tasarruf edin. Katkı maddesi ve yüksek/düşük DMSO tamponları ile gerekli 1x tamponu hesaplamak için cihaz yöneticisine danışın.
- Muhabir plakasını hazırlamaya başlayın. İlk olarak, toplam döngü sayısı için gerekli olan raportör stok çözümünü yapın. Siyah veya kehribar renkli 1 mL mikrosantrifüj tüplerini döngü adlarıyla etiketleyin. İlgili tüpe 250 μL raportör stok çözeltisi ve her raportörden 5 μL ekleyin. Ardından, çözeltiyi 96 oyuklu siyah bir plakaya aktarın ve yapışkan folyo ile kapatın.
- Cihazı başlatın, parametreleri ve pozlama sürelerini (Tablo 1'e göre) ayarlamak için cihaz yöneticisini kullanın ve görüntüleri alın. Şekil 1 , boyama öncesi ve sonrası işlemleri özetlemektedir.
5. Doku açıklaması ve hücre segmentasyonu
- Bir proje ve çalışma alanı ayarlayın.
- Dijital patoloji analiz yazılımının en son sürümünü yükleyin (ör. QuPath sürüm 0.5.1 veya üstü). Proje alanı için bir hedef klasör seçmek için Proje oluştur'a tıklayın.
- Görüntü ekle'ye > Dosyaları seç'e tıklayın, ardından QPTIFF dosyasına gidin (multipleks immünofloresan görüntülemeden üretilir). Görüntü türünü Floresan olarak ayarlayın, diğer tüm varsayılan ayarları koruyun ve İçe Aktar'ı tıklatın.
- QuPath kullanıyorsanız, bir çalışma alanı açmak için yeni görüntüye çift tıklayın. Projede yapılan değişiklikleri takip etmek için Dosya > Düzenli aralıklarla kaydet'e tıklayın. Araç çubuğunun ortasına yakın bir yerde bulunan Parlaklık ve kontrastı (yarım ay simgesi) kullanarak işaretçi görünürlüğünü ve görüntüleme ayarlarını değiştirin.
NOT: Görüntü listesindeki bir görüntüyü yeniden adlandırmak için sağ tıklatın. Bu, daha sonra fenotip sınıflandırmalarını görselleştirirken önemli olacaktır.
- Tam doku bölümüne, intratümöral bölmeye ve stromal bölmeye açıklama ekleyin.
- Tüm doku bölümünün etrafına bir açıklama çizmek için fırça ve/veya değnek aracını kullanın. Bu ek açıklamayı Full_Tissue olarak tanımlayın. Üstteki cildi ve / veya analiz edilmemesi gereken bölgeleri hariç tutun. Negatif bir ek açıklama oluşturmak ve/veya ek açıklama sınırını küçültmek için, ek açıklama araçlarından birini kullanırken Alt tuşunu basılı tutun.
- Tam doku ek açıklamasını çoğaltın. Ek Açıklamalar sekmesi altında Ek Açıklama'yı seçin, ardından Nesneler > Ek Açıklamalar'a gidin... > Seçili ek açıklamaları çoğaltın.
- SOX10 kanalını açın. Çoğaltılan ek açıklamada, Alt + fırça/değnek aracını kullanarak intratümöral bölmeyi yakalamak için ek açıklama sınırını küçültün. Bu ek açıklamayı bir Tümör olarak tanımlayın (Şekil 2).
- Tam doku ek açıklamasını seçerek Nesneler > Ek Açıklamalar'a gidin... > Ek açıklamaları genişletin. Genişleme yarıçapını 1 μm olarak tanımlayın ve Çalıştır'a tıklayın.
- Yeni ek açıklamayı Full_Tissue_Expansion olarak yeniden adlandırın. Tümör Ek Açıklaması'nı seçin, sağ tıklayın ve Hiyerarşiye ekle'yi seçin.
- Tümör Ek Açıklamasını yeniden seçin, Nesneler > Ek Açıklamalar'a gidin... > Tersini yapın. Bu yeni ek açıklamayı Stroma olarak tanımlayın. Tüm doku bölümleri için tekrarlayın.
- Hücre segmentasyonunu çalıştırın ve sonuçları dışa aktarın.
- Görüntünün piksel genişliğini ve yüksekliğini bulmak için Görüntü sekmesine gidin (Şekil 3A).
- StarDist uzantısını https://github.com/qupath/qupath-extension-stardist/releases adresinden indirin. qupath-extension-stardist-[version].jar dosyasını QuPath penceresine sürükleyin, ardından QuPath penceresinin sağ üst köşesindeki dişli simgesine tıklayın. Uzantılar altında, QuPath kullanıcı dizinini StarDist klasörünün konumu olarak tanımlayın.
- StarDist harika komut dosyalarını ve StarDist model dosyasını https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis'dan indirin (StarDist komut dosyaları orijinal olarak Akoya Biosciences'tan düzenlenmiştir).
- Her doku bölümü için hem Tümör hem de Stroma ek açıklamalarını seçin (birden fazla ek açıklama seçmek için Ctrl tuşunu kullanın). Üst ayarlar çubuğunda, komut dosyaları arayüzünü açmak için Komut Dosyası düzenleyicisini otomatikleştir > tıklayın. Görüntü piksel boyutuna karşılık gelen uygun StarDist hücre segmentasyon komut dosyasını (harika dosya) açın. Hücre segmentasyon dosyasını seçmeniz istendiğinde, stardist_cell_seg_model.pb dosyasını açın.
- Hücre segmentasyonundan sonra görüntüyü kaydedin. Ölçü > Ölçümleri dışa aktar'a gidin, ardından İlgili görüntüleri seçin. Dışa Aktarma türünü Hücreler ve Ayırıcı olarak .csv olarak atayın, ardından Çıktı dosyası konumunu seçin. İlgilendiğiniz metrikleri dahil etmek için Sütunların yanındaki Doldur'a tıklayın. Dışa aktarılacak önemli sütunlar Görüntü, Nesne Kimliği, Üst, Centroid X, Centroid Y ve Nucleus Area'dır.
NOT: Nesne Kimliği, sınıflandırmaları dijital patoloji analiz yazılımındaki multipleks immünofloresan görüntüsüne geri eşlemek için sonraki adımlarda kullanılır. Üst sütun, hücrenin ait olduğu ek açıklama adını gösterir. Mekansal analiz için X ve Y verileri kullanılır. - Kümelemede/fenotiplemede kullanılacak her köken işaretçisi için, dışa aktarılacak bir ortalama değer seçin. Nükleer belirteçler için (örneğin, SOX10 ve FOXP3), Nucleus: Mean seçeneğini dışa aktarın. Sitoplazmik/membranöz belirteçler için (örn., CD45, CD3, CD4), Sitoplazma: Ortalama seçeneğini dışa aktarın.
6. Proteomik veri ön işleme ve normalleştirme
- Dışa aktarılan CSV dosyasını açın. Kümelemede/fenotiplemede kullanılacak her işaretçi için, sütun başlığının adını yalnızca işaretçiyi içerecek şekilde kesin. Örneğin, SOX10: Nucleus: Mean'ı SOX10 ve CD3: Cytoplasm: Mean'ı CD3 olarak yeniden adlandırın.
- R ve RStudio'nun en son sürümünü yükleyin. https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis'dan Marker Normalization.R dosyasını indirin ve betiği çalıştırın. Bu ilk önce hücrelerin nükleer boyuta göre filtrelenmesini içerir.
- Her işaretçi için, ifade aralığındaki en düşük MFI değeri 0 olarak ve 99,7. yüzdebirlik dilimdeki MFI değeri 1 olarak ayarlanacak şekilde bir min-maks normalleştirme gerçekleştirilir. 99.7. yüzdelik dilimin üzerindeki tüm yoğunluklar 1'de kırpılır. İşlem tamamlandıktan sonra, normalleştirilmiş verilerle oluşturulan yeni CSV dosyasını kaydedin.
7. Kümeleme ve fenotipleme
- İşaretleyici ifade profillerine göre hücreleri kümelemek için bir algoritma seçin. https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis'de, Seurat v4.415 ve FlowSOM v2.13.9 (kümeleme ve görselleştirme aracı)16 kullanılarak kümeleme için R komut dosyaları sağlanır.
- Anahtar soy belirteçlerinin işaretleyici ifade profillerine göre fenotip hücreleri. Burada sunulan protokol, kümeleme ve fenotipleme için 14 köken belirteci kullandı: SOX10, CD45, CD3, CD4, CD8, FOXP3, CD11C, F4/80, CD68, CD86, CD163, CD206, NK1.1 ve CD31.
- Tümör hücrelerini SOX10hi olarak tanımlayın. CD4 T hücrelerini CD45mod-hi/ CD4hi/CD8düşük/FOXP3düşük olarak tanımlayın. CD8 T hücrelerini CD45mod-hi/ CD8hi/CD4düşük olarak tanımlayın. Treg'leri CD45mod-hi/CD4hi/CD8low/FOXP3hi olarak tanımlayın. Diğer T hücrelerini CD45mod-hi/CD3hi/CD4düşük/CD8düşük olarak tanımlayın.
- M1 makrofajlarını CD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163düşük /CD206düşük olarak tanımlayın. M2 makrofajlarını CD45mod-hi/F4/80hi/CD68mod-hi/CD86düşük/CD163hi/CD206düşük, CD45mod-hi/F4/80hi/CD68mod-hi/CD86düşük/CD163düşük/CD206hi veya CD45mod-hi/F4/80hi/CD68mod-hi/CD86düşük/CD163hi/CD206hi olarak tanımlayın. Diğer makrofajları CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/ CD163low/CD206low veya CD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hi olarak tanımlayınız.
- Dendritik hücreleri CD45mod-hi/CD11chi/CD86mod-hi olarak tanımlayın. NK hücrelerini CD45mod-hi/NK1.1hi olarak tanımlayın. Yukarıda ayrıntıları verilen koşulları karşılamayan diğer bağışıklık hücrelerini CD45mod-hi olarak tanımlayın. Endotel hücrelerini CD31mod-hi olarak tanımlayın. Diğer tüm ifade profillerini kaldırın.
8. Fenotip sınıflandırmalarının yeniden haritalandırılması ve kalite kontrolünün yapılması
- https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis'dan Remapping Classifications.groovy betiğini indirin (bu betik petebankhead tarafından https://forum.image.sc/ tarihinde yayınlanan bir gönderiden uyarlanmıştır).
- Fenotiplenmiş CSV dosyasını açın ve Görüntü sekmesi altındaki tüm etiketlerin, yeniden eşleme için kullanılacak dijital patoloji analiz yazılımındaki görüntü dosyasının adıyla tam olarak eşleştiğinden emin olun. CSV dosyasındaki büyük miktarlardaki etiketleri hızlı bir şekilde değiştirmek için Tümünü Değiştir işlevini kullanın.
- QuPath kullanıyorsanız, Komut Dosyası düzenleyicisini otomatikleştir'e tıklayarak sınıflandırmaları yeniden eşleme harika > çalıştırın.
- Ek Açıklamalar sekmesinde, Otomatik ayar düğmesinin yanındaki Üç Nokta'ya tıklayın, ardından Mevcut nesnelerden doldur'a tıklayın > Tüm sınıflar (alt sınıflar dahil) > Evet'e tıklayın.
- Farklı fenotip sınıflandırmalarını görüntülemek ve renk şemasını gerektiği gibi ayarlamak için işaretçi listesinde aşağı kaydırın. Fenotipini (Sınıflandırma etiketinin yanında) ve kümesini (Ad etiketinin yanında) görüntülemek için herhangi bir hücreye çift tıklayın. Fenotip atamalarının doğruluğunu belirlemek için temsili görüş alanlarını kontrol edin ve fenotip stratejisini gerektiği gibi revize edin (Şekil 3B).
9. Yoğunluk ölçümü ve mekansal analiz
- Fenotipleme tamamlandıktan sonra, Dijital Patoloji Analiz Yazılımında Ek Açıklamalar sekmesi altındaki açıklama alanlarını edinin. Alan değerlerini mm2'ye dönüştürün, ardından yoğunlukları hesaplamak için sayıları açıklama alanlarına bölün.
- https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis'dan SPIAT Spatial Analysis.R betiğini indirin ve çalıştırın. Ortalama minimum mesafeler (AMD), komşuluk bileşimleri ve normalleştirilmiş karıştırma puanları (NMS) hakkında veri toplayın.
- Bulguları sunmak için istatistiksel analiz yazılımında rakamlar oluşturun. Tedavi koşullarının ortalama minimum mesafeleri için tek bir ısı haritası oluştururken, farklı doku bölümlerinden elde edilen sonuçların ortalamasını alın.
Sonuçlar
Burada, murin FFPE dokusu için bir antikor paneli tasarlamak, multipleks immünofloresan görüntüleme yapmak ve proteomik miktar tayini ve uzamsal ilişkiler için görüntüleri analiz etmek için bir protokol sunuyoruz. Doğrulanmış panel, melanom hücrelerini (SOX10), lökositleri (CD45), T hücrelerini (CD3, CD4, CD8, FOXP3), B hücrelerini (CD20), makrofajları ve alt tiplerini (F4/80, CD68, CD86, CD163, CD206), dendritik hücreleri (CD11c), NK hücrelerini (NK1.1) ve endotel hücrelerini (CD31) görselleştirmek için belirteçler sağlayan 27 antikor içerir. Tam panel ayrıca diğer immün popülasyonlar (CD11b, CD38), proliferasyon aktivitesi (Ki67), T hücresi işlevselliği (T-bet, Eomesodermin, granzyme B), antijen sunumu (LMP2, beta-2 mikroglobulin, MHC II) ve kontrol noktası ekspresyonu (TIM3, LAG3, PD-L1) için belirteçler içerir12. Temsili bir jel elektroforez görüntüsü, Şekil 4'te gösterildiği gibi, DNA oligonükleotid barkodlarının taşıyıcısız antikorlara başarılı bir şekilde konjugasyonunu doğrular. Bu adım sadece kimyasal reaksiyonu doğrular ve görüntü doğrulaması ancak bu antikorlar ilgilenilen doku üzerinde multipleks görüntüleme cihazı ile kontrol edildikten sonra yapılabilir. Panel27'teki 13 işaretçinin tümünün doğrulanmış görüntülerini görüntülemek için aşağıdaki referansa bakın. İntratümöral 4-1BBL/IL-12 nanopartikül enjeksiyonları ve sistemik anti-PD1 ile tedavi edilen üç murin melanom doku kesitini boyayan anahtar soy belirteçlerinin füzyon görüntüsü Şekil 5'te sunulmuştur. Bu bölümler, bu protokolde daha sonraki görüntü analizi için kullanılmıştır.
Doku anotasyonu, hücre segmentasyonu ve proteomik veri ön işlemesinden sonra, daha önce tek hücreli transkriptomik ve/veya proteomik analiz için uygulanmış olan iki kümeleme algoritması arasında bir karşılaştırma sunuyoruz17,18. Fenotipli popülasyonlar için ekspresyon profilleri, her iki yaklaşım için de Şekil 6'da sunulmuştur. FlowSOM'un, makrofaj alt tipleri gibi benzer hücre popülasyonları arasındaki farkları ayırt etmek için daha geniş bir yoğunluk değerleri aralığı (~ 0.7'ye karşı ~ 0.5) sunduğunu gösteriyoruz. Seurat, kümeleme sırasında Louvain algoritmasını uyguladı ve 29 küme oluşturdu (Ek Şekil 1). Buna karşılık, FlowSOM kendi kendini organize eden haritalar uyguladı ve 100 küme oluşturabilir (Ek Şekil 2). Daha fazla sayıda küme, fenotipleme için daha fazla zaman gerektiği anlamına gelir, ancak bu ikinci FlowSOM yaklaşımı, benzer hücre popülasyonlarını sınıflandırırken daha fazla nüans sunar. Kalitatif olarak, FlowSOM'un aynı görüş alanındaki Seurat fenotiplemesi ile karşılaştırıldığında, daha fazla intratümöral makrofajı M1 veya M2 alt tipi olarak sınıflandırabildiğini görüyoruz (Şekil 7). Aynı sonuç, makrofaj yoğunluklarını ölçtüğümüzde de görülür, FlowSOM, Seurat'a kıyasla hem M1 hem de M2 makrofajlarının daha yüksek bir yoğunluğunu yakalar (Şekil 8A-B) ve ardından diğer/sınıflandırılmamış makrofajların önemli ölçüde daha düşük bir yoğunluğunu yakalar (p = 0.0028; Şekil 8C). Bununla birlikte, iki analiz yaklaşımı, CD4 T hücreleri, CD8 T hücreleri ve endotel hücreleri gibi diğer hücre popülasyon yoğunluklarını tanımlarken de benzer sonuçlar vermiştir (Şekil 8D-F).
Ayrıca, kümeleme ve fenotiplemeden sonra aşağı akış mekansal analiz bulgularını da sunuyoruz. Şekil 9'da , bu protokol kullanılarak oluşturulabilecek bazı uzamsal ölçümler gösterilmektedir. Tüm fenotipli popülasyonlara göre, M2 makrofajları ve NK hücreleri, 4-1BBL/IL-12 nanopartikülleri ve anti-PD1 ile tedaviyi takiben en yüksek AMD'lere sahipti (Şekil 9A). Benzer şekilde, intratümöral CD8 T hücreleri ile M1 makrofajları arasındaki NMS, CD8 T hücreleri ile M2 makrofajları arasındakinden çok daha yüksekti (Şekil 9B). Ayrıca, M2 makrofajları, intratümöral CD8 T hücrelerini çevreleyen 100 μm'lik komşulukta ~% 1 katkıda bulunurken, M1 makrofajları bu komşulukların% 9-13'ünü oluşturmuştur (Şekil 9C). Birlikte ele alındığında, bu sonuçlar 4-1BBL/IL-12 tedavi rejiminin, tümörle ilişkili makrofajları bir M1 alt tipine doğru polarize ettiğini ve M2 makrofajlarını tümör immün mikroçevresinden dışladığını göstermektedir.
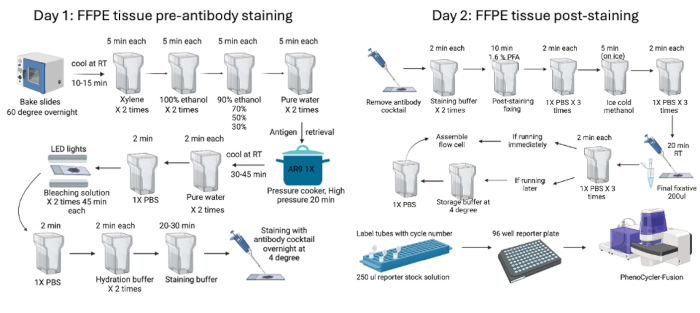
Şekil 1: FFPE doku boyama ve görüntüleme iş akışının özeti. Murin FFPE dokuları, iki günlük boyama öncesi (1. gün) ve boyama sonrası (2. gün) işlemlere başlamadan önce dokunun gece boyunca pişirilmesiyle başlayan ön boyama prosedürleri kullanılarak işlendi. Son olarak cihaz ile görüntüleme yapılmadan önce raportör plakası hazırlandı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
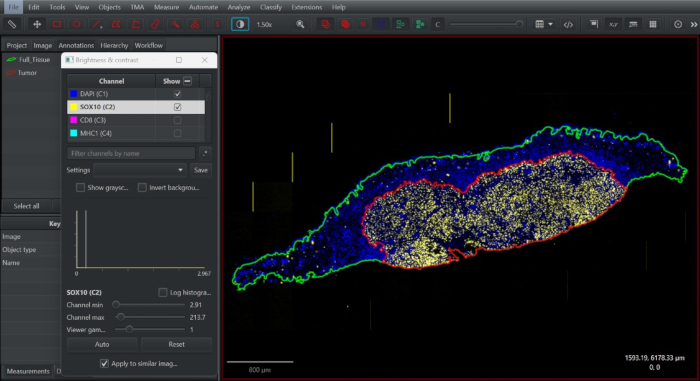
Şekil 2: Açık erişimli dijital patoloji yazılımı QuPath'te doku açıklamalarının ekran görüntüsü. Tam doku anotasyonu çizildi (yeşil), çoğaltıldı ve tümör sınırındaki SOX10+ melanositlerin dağılımına (kırmızı) göre intratümöral kompartmanı tanımlamak için küçültüldü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
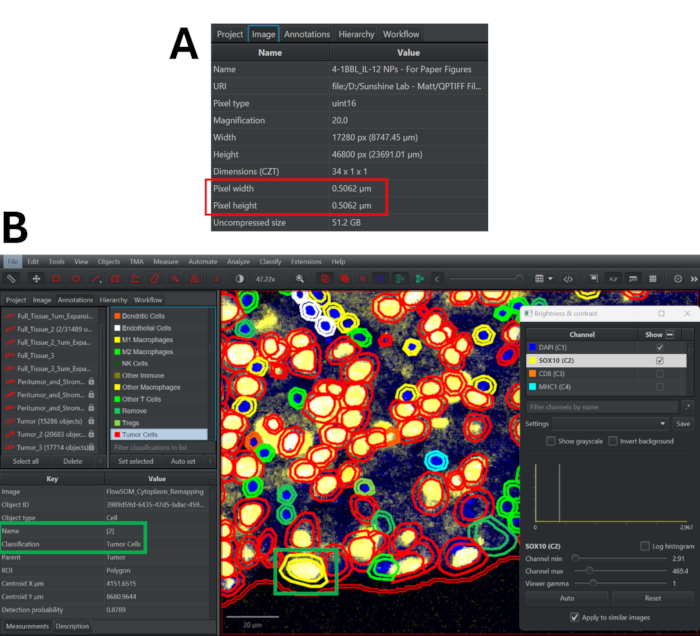
Şekil 3: QuPath'te StarDist algoritması kullanılarak hücre segmentasyonu ve fenotip sınıflandırmalarını gözden geçirmek için kalite kontrol süreci. (A) Multipleks immünofloresan görüntüsünün piksel genişliğini ve yüksekliğini belirlemek için Görüntü sekmesine gidin. (B) Sınıflandırmaları yeniden eşledikten sonra, küme atamasını ve fenotipini görmek için herhangi bir hücreye (sarı renkle vurgulanır) çift tıklayın. Bu sınıflandırma yaklaşımının doğru olup olmadığını belirlemek için panel işaretçilerini açın/kapatın. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 4: Antikor-DNA barkod konjugasyon onayının temsili jel görüntüsü. Protein jel elektroforezi, ağır zincir bölgesinde ek bantlar tarafından gözlemlenen DNA oligonükleotid barkodları ile antikor konjugasyonunu doğrular. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
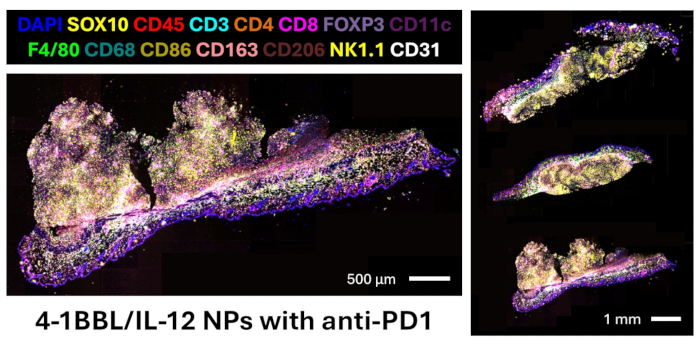
Şekil 5: Sistemik anti-PD1 ile 4-1BBL/IL-12 nanopartiküllerin intratümöral enjeksiyonları ile tedavi edilen B16F10 yan tümörlerinin DNA barkod tabanlı multipleks görüntülemesi. Panelimizdeki belirteçler gösterilmemiştir: CD11b, CD20, CD38, TIM3, LAG3, T-bet, Eomesodermin, granzim B, Ki67, LMP2, beta-2 mikroglobulin, MHC II, PD-L1. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 6: Fenotiplenmiş hücre popülasyonlarının proteomik ekspresyon ısı haritaları. (A) Seurat kümeleme ve fenotipleme yaklaşımı, (B) FlowSOM kümeleme ve fenotipleme ile karşılaştırıldığında biraz daha küçük bir işaretleyici ifade değerleri aralığı oluşturur. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 7: FlowSOM fenotiplemesi, daha geniş bir yelpazede farklı makrofaj alt tiplerini tanımlar. Üst panel, aynı görüş alanı için Seurat (solda) ve FlowSOM (sağda) makrofaj fenotiplemesini gösterir. Alt panel, aynı görüş alanındaki temel makrofaj soy belirteçlerinin multipleks immünofloresan görüntülerini gösterir. Tüm ölçek çubukları 20 μm'dir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 8: Kümeleme/fenotipleme sonrası farklı hücre popülasyonlarının intratümöral yoğunluklarının karşılaştırılması. İntratümöral (A) M1 makrofajları, (B) M2 makrofajları, (C) diğer makrofajlar, (D) CD4 T hücreleri, (E) CD8 T hücreleri ve (F) endotel hücreleri için yoğunluk karşılaştırmaları gösterilmiştir. Hata çubukları, ortalamanın (SEM) standart hatalarıdır ve anlamlılık, eşleşmemiş t-testleri kullanılarak test edilmiştir (**p ≤ 0.01). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 9: 4-1BBL/IL-12 intratümöral nanopartikül enjeksiyonları ve sistemik anti-PD1 ile tedavi edilen üç yan tümör için intratümöral mekansal ölçümlerin profilinin çıkarılması. (A) İntratümöral fenotipli popülasyonlar arasındaki ortalama minimum mesafelerin ısı haritası. Ölçümler μm cinsindendir. (B) Anahtar intratümöral fenotip çiftleri arasında normalleştirilmiş karışım skorları. (C) İntratümöral CD8 T hücre popülasyonları etrafında 100 μm'lik bir yarıçapta komşuluk bileşimlerinin parçalanması. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Antikor | Seyreltme | Maruz kalma süresi (ms) |
| SOX-10 | 50 | 450 |
| CD8 Serisi | 100 | 450 |
| CD3 (İngilizce) | 100 | 450 |
| TilkiP3 | 50 | 350 |
| CD4 (İngilizce) | 50 | 450 |
| MHC-II | 100 | 450 |
| PDL1 (İngilizce) | 50 | 450 |
| CD45 Serisi | 100 | 450 |
| Ki-67 Uçağı | 100 | 300 |
| F4/80 Serisi | 100 | 150 |
| CD20 Serisi | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| CD206 Serisi | 100 | 450 |
| CD68 Serisi | 100 | 450 |
| Granzime-B | 50 | 450 |
| CD86 Serisi | 100 | 150 |
| CD31 Serisi | 100 | 450 |
| CD11C Sertifikası | 50 | 450 |
| CD11b | 50 | 450 |
| EOMES (Türkçe) | 50 | 450 |
| TİM-3 | 50 | 300 |
| CD38 Serisi | 50 | 200 |
| LAG3 | 50 | 450 |
| CD163 Serisi | 50 | 200 |
| T-bahis | 50 | 300 |
| LMP2 | 100 | 100 |
| Beta2 MG | 200 | 50 |
Tablo 1: Antikor seyreltme ve maruz kalma süresi ayarları.
Ek Şekil 1: Seurat kümelemesinden sonra oluşturulan ilk proteomik ekspresyon ısı haritası. Bu rakamı indirmek için lütfen buraya tıklayın.
Ek Şekil 2: FlowSOM kümelemesinden sonra oluşturulan ilk proteomik ekspresyon ısı haritası. Bu rakamı indirmek için lütfen buraya tıklayın.
Tartışmalar
Görüntülemenin başarısı, iyi tasarlanmış ve doğrulanmış bir antikor paneline bağlıdır. FFPE örneklerinin multipleks immünofloresan görüntülemesi, yüksek otofloresan ve parafin gömülmesi ile maskelenen epitopların alınmasının zorluğu nedeniyle zorluklar sunar. Bununla birlikte, FFPE'nin FF örneklerine kıyasla çeşitli avantajlar sunduğu göz önüne alındığında, FFPE antikor panellerinin tasarlanması ve doğrulanması esastır. İmmünofloresan (IF) görüntüleme sırasında pozitif sinyaller gösteren antikor klonlarının sonlandırılması ilk adımdır; daha sonra, bunları DNA barkodları ile dikkatli bir şekilde birleştirmek önemlidir. Antikor konjugasyonu, bir barkodla maleimid grup reaksiyonu sırasında kullanılan SH bağları oluşturmak için antikorun kısmen indirgenmesini gerektirir. Her antikor klonu bu adıma dayanamaz ve bazı reaksiyonlar antikorda geri dönüşü olmayan hasara neden olabilir, bu da başarılı konjugasyona rağmen görüntüleme başarısızlığına neden olabilir. Bu nedenle, bazı antikorlar konvansiyonel IF validasyonu sırasında pozitif sinyaller gösterebilse de, antikor konjugasyonunun nihai başarısını değerlendirmek için, her bir antikorun ilgilenilen gerçek doku üzerinde doğrulanması ve o doku için istenen maruz kalma sürelerinin kaydedilmesi önemlidir. Gelecekteki uygulamalarda, bu teknik, ek içgörüler oluşturmak için ardışık slaytlarda/aynı slaytta mevcut uzamsal transkriptomik analizle birleştirilebilir. Bu yöntemin sınırlamalarından biri, paneldeki her bir antikorun hedefe ve doku tipine göre dikkatli bir şekilde seçilmesini ve doğrulanmasını gerektirmesidir.
Görüntü analizi ile ilgili olarak QuPath, proteomik belirteçlerin yüksek kaliteli görselleştirilmesi, yoğunluk ölçümlerini dışa aktarmak ve fenotip sınıflandırmaları için kalite kontrol gerçekleştirmek için geniş işlevsellik ve kullanıcı tarafından oluşturulan komut dosyaları için iyi esneklik ile değerli bir açık erişim aracı sunar. https://forum.image.sc/ gibi çevrimiçi forumlar, belirli analiz görevlerinin nasıl gerçekleştirileceğini tartışmak ve komut dosyalarını diğer kullanıcılarla paylaşmak için ek bir kaynaktır. Bu protokolde, Seurat ve FlowSOM kullanarak iki kümeleme ve fenotipleme yaklaşımını karşılaştırıyoruz. FlowSOM, TME'nin bağışıklık hücresi alt popülasyonları hakkında daha ayrıntılı bilgiler üretme yeteneği nedeniyle tercih edilebilirken, proteomik analiz için gereken süre de dikkate alınmalıdır. Bir kullanıcının yalnızca bir veya iki doku örneği içindeki hücreleri fenotiplemesi gerekiyorsa, 100 küme oluşturmak gereksiz olabilir. Bu durumlarda Seurat, görüntü analizi için daha hızlı ve daha verimli bir boru hattı sunabilir. Buna karşılık, 40 veya 50'den fazla doku kesitine sahip bir TMA'nın analiz edilmesinin, her iki analiz yaklaşımında da daha fazla sayıda hücre kümesi üretme olasılığı daha yüksektir ve FlowSOM, daha incelikli fenotip sınıflandırmaları oluşturmak için tercih edilen metodoloji olabilir.
Hücre kümeleme/fenotipleme ve sonraki tüm görüntü analizi adımları büyük ölçüde hücre segmentasyonuna bağlıdır. Mevcut çalışmamız, HALO (Indica Labs) ve StarDist algoritmalarında hücre segmentasyonunu araştırdı ve her iki yaklaşımın da nükleer DAPI sinyallerine dayalı olarak hücreleri aşırı segmentlere ayırma eğiliminde olduğunu bulduk. Mesmer19 ve InstanSeg20 gibi birçok alternatif segmentasyon algoritması da mevcuttur. Bu, daha fazla keşif ve optimizasyon gerektiren, büyüyen bir hesaplama araştırması alanıdır.
Açıklamalar
J.C.S., Emerson Collective, LLC ve Ulusal Sağlık Enstitüleri'nden mali destek aldığını kabul eder. JJG ayrıca Ulusal Sağlık Enstitüleri'nden fon aldığını da kabul eder. J.C.S.'nin Palleon Pharmaceuticals Inc. ile hibeleri finanse etmeyi içeren bir ilişkisi vardır. SYT ve JJG'nin OncoSwitch Therapeutics ile hisse senedi veya hisse senedi içeren bir ilişkisi vardır. S.Y.T., J.J.G., J.C.S. ve K.M.L.'nin bekleyen bir patenti var. Diğer tüm yazarlar, bu yazıda sunulan araştırmayı etkilediği şeklinde algılanabilecek bilinen bir rekabet eden finansal çıkarları veya kişisel ilişkileri olmadığını belirtmektedir.
Teşekkürler
J.C.S., Yazarın kariyerini ilerlettiği için Dermatoloji Vakfı'na ve Dermatopatoloji Kariyer Geliştirme Ödülü'ne teşekkür eder. Yazarlar, hesaplamalı analiz tekniklerine yardımcı olduğu için Ulusal Kanser Enstitüsü'nün Kanser Veri Bilimi Laboratuvarı'ndan Hsin-Pei Lee'ye teşekkür ediyor. Bu araştırma Emerson Collective ve Ulusal Sağlık Enstitüleri'nden (R37CA246699, P41EB028239 ve R01CA228133) fon aldı. Ek olarak, Johns Hopkins Onkoloji Doku Hizmetleri (OTS) çekirdeği, Ulusal Sağlık Enstitüleri (P30CA006973) tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
Referanslar
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır