Method Article
הדמיית אימונופלואורסצנציה מולטיפלקס מבוססת ברקוד DNA לניתוח דגימות FFPE ממלנומה עכברית מתוכנתת מחדש גנטית
* These authors contributed equally
In This Article
Summary
פרוטוקול זה מספק מדריך שלב אחר שלב לתכנון פאנל נוגדנים אימונופלואורסצנטי מרובה להדמיה מבוססת ברקוד DNA של רקמות מלנומה FFPE של עכברים. אנו מתארים גם צינור ניתוח תמונה באמצעות כלי קוד פתוח ליצירת תובנות פרוטאומיקה מרחביות לגבי המיקרו-סביבה החיסונית של גידול מלנומה בעכברים.
Abstract
מוצגת כאן טכניקת הדמיה מולטיפלקס מבוססת ברקוד DNA המבוססת על זיהוי משותף על ידי indEXing המנתחת את הפרוטאומיקה המרחבית של מיקרו-סביבות רקמות. הדמיה מוצלחת דורשת רפרטואר של לוחות נוגדנים מתוכננים היטב ומאומתים כהלכה, אך מעט מאוד קיימים כיום עבור דגימות משובצות פרפין קבועות בפורמלין (FFPE). FFPE מציע מספר יתרונות על פני דגימות קפואות טריות, כגון זמינות נרחבת, קלות טיפול ואחסון, והיכולת לייצר מיקרו-מערכי רקמות (TMAs). כאן, אנו מציגים פרוטוקול לפיתוח פאנל נוגדנים להדמיה וניתוח רקמות FFPE ממודל מלנומה של עכברים שטופלו בננו-חלקיקים, המספקים DNA פלסמיד המקודד אותות אימונולוגיים לתכנות מחדש של מיקרו-סביבת גידול. אנו מתארים גם צינור ניתוח תמונה באמצעות כלים חישוביים בקוד פתוח לביאור רקמות, פילוח תאים, עיבוד נתוני פרוטאומיקה, פנוטיפ אוכלוסיות תאים וכימות מדדים מרחביים. הפרוטוקול מציע יישומים לתכנון לוחות נוגדנים ב-FFPE של עכברים ויצירת תובנות חדשות לגבי הפרוטאומיקה המרחבית של מיקרו-סביבות רקמה מורכבות.
Introduction
מלנומה עורית היא סרטן העור הנפוץ ביותר, עם שיעורי מחלות ותמותה משתנים ברחבי העולם בהתאם לזמן האבחון והטיפול הראשוני1. במהלך העשור האחרון, הבנה ביולוגית מוגברת של מלנומה סייעה להניע את הפיתוח של מודלים חדשים של סרטן לטיפול בגידולים מוצקים2. עלייתה של האימונותרפיה לאחרונה הובילה לתפיסה מהפכנית של טיפול בסרטן המבוססת על הפעלת מערכת החיסון האנדוגנית 3,4.
המיקרו-סביבה של הגידול (TME) מורכבת מאוד, ומורכבת מתאי חיסון מגוונים, פיברובלסטים הקשורים לסרטן, פריציטים, תאי אנדותלותאים שונים השוכנים ברקמות. מספר טכניקות יושמו בעבר לחקר ה-TME, כגון ציטומטריית זרימה וריצוף תא בודד, הפוגעות בהקשר המרחבי מכיוון שהן נדרשות להרוס את רקמת הגידול. הדמיית מיקרוסקופ מסורתית, כגון אימונופלואורסצנציה (IF) ואימונוהיסטוכימיה (IHC), מאפשרת הדמיה של סמנים ביולוגיים של חלבונים מבלי להרוס רקמות דגימה. עם זאת, גישות אלה מוגבלות לשניים או שלושה סמנים ביולוגיים ואינן מסוגלות לספק הבנה מלאה של יחסים מרחביים ומבניים בתוך TME 6 המורכב.
כדי לטפל בבעיה זו, פותחו מספר טכניקות הדמיה מרובות כדי להמחיש את ה-TME המורכב מרחבית 7,8,9,10. אחד מהם הוא CoDetection-by-inDEXing, ששמו שונה למערכת PhenoCycler, המבוססת על נוגדנים מצומדים ל-DNA אוליגונוקלאוטידים11. המערכת יכולה לספק הדמיה של תא בודד וניתוח של למעלה מ-100 סמנים ביולוגיים לדגימות אנושיות. עם זאת, מעט מאוד נוגדנים זמינים להמחשה וניתוח דגימות עכברים, במיוחד דגימות מוטבעות פרפין קבועות פורמלין (FFPE)12. FFPE מציע מספר יתרונות על פני שימור קפוא טרי (FF), כגון קלות טיפול ואחסון, מורפולוגיה שמורה היטב לאורך זמן, והכי חשוב, היכולת להכין מיקרו-מערכי רקמות/גידולים (TMA) המאפשרים הדמיה של מספר דגימות בשקופית אחת. לאחרונה עיצבנו ופיתחנו פאנל נוגדנים FFPE CODEX/PhenoCycler לעכברים ויישמנו אותו בהצלחה כדי להמחיש ולנתח את הפרוטאומיקה המרחבית של דגימות מלנומה של עכברים שתוכנתו מחדש גנטית13.
המטרה הכוללת של פרוטוקול זה היא לספק מדריך שלב אחר שלב לתכנון פאנל נוגדנים FFPE לעכברים ולתאר את תהליך צימוד נוגדנים-ברקוד, צביעת רקמות והדמיה. בנוסף, אנו מציגים צינור ניתוח תמונה מפורט תוך שימוש בכלי קוד פתוח כגון חבילות QuPath ו-R. לאחר ביצוע פרוטוקול זה, החוקרים ילמדו כיצד לתכנן פאנל נוגדנים מצומד בהתאמה אישית, לבצע הדמיה מרובה באמצעות מכשיר Phenocycler-Fusion, ולקבל תובנות חדשות לגבי הפרוטאומיקה המרחבית של המלנומה TME. יתר על כן, ניתן להתאים פרוטוקול זה לחקר מיקרו-סביבות חיסוניות שונות של גידול ולשלב אותו עם טכניקות טרנסקריפטומיקה מרחבית קיימות.
Protocol
כל העבודה בבעלי חיים בוצעה בהתאם להנחיות שנקבעו על ידי הוועדה לטיפול ושימוש בבעלי חיים של ג'ונס הופקינס, תוך שימוש במספרי הפרוטוקול המאושרים MO18M388 ו-MO21M384.
1. בחירת נוגדנים
- תכנן פאנל נוגדנים המבוסס על הרקמה המעניינת והשפע של הסמן הביולוגי המסוים. בחר שיבוטי נוגדנים על סמך יישומים קודמים מוצלחים של צביעת אימונופלואורסצנציה (IF) ואימונוהיסטוכימיה (IHC) בספרות. גרסאות מאובטחות ללא נשא של נוגדנים אלה.
הערה: נוגדן מטוהר ללא נשא חשוב לצימוד הברקוד. הימנעו מחומרים משמרים על בסיס חלבון כגון BSA, גלוטן, גליצרול וכו'. אם הנוגדן נטול הנשא אינו זמין, בקש נוגדן מותאם אישית מהספק או השתמש בערכת הסרת BSA כדי ליצור את הנוגדן ללא נשא. נתרן אזיד אינו מפריע להצמדת נוגדנים. איזוטיפים של IgG מומלצים על פני איזוטיפים של IgM. - אמת את השיבוטים הנבחרים הללו על הרקמה המעניינת באמצעות הדמיה אימונופלואורסצנטית קונבנציונלית (IF)11. השתמש בהדמיית IF כפולה כדי לאשר את הספציפיות של שיבוטים מסוימים (למשל, בצע הדמיית IF כפולה עם FOXP3 ו-CD4 כדי לאשר צביעה עבור תאי T רגולטוריים).
הערה: במידת האפשר, חשוב להשתמש באותה רקמה לצורך אימות IF שתשמש לצביעה. בחר את פתרון אחזור האנטיגן המתאים. בפרוטוקול זה, השתמשנו במאגר AR9 שסופק על ידי Akoya Biosciences כדי לאחזר את כל האפיטופים של רקמת המלנומה של העכברים FFPE. ניתן להשתמש בתמיסות אחרות לאחזור אנטיגן, כגון AR6 או Universal, על סמך דרישות האפיטופ.
2. צימוד ואישור נוגדנים
- הקצה נוגדנים מאומתים לברקודים שיש להם מדווחים משלימים המצורפים לפלואורופורים ATTO550 (Cy3), AF647 (Cy5) או AF750 (Cy7). אנטיגנים בשפע נמוך מייצרים בדרך כלל אותות נמוכים יותר; להצמיד נוגדנים כאלה לתעלות אוטופלואורסצנטיות נמוכות כגון AF647 (Cy5). מצומדים אנטיגנים בעלי ביטוי גבוה ל-ATTO550 (Cy3) ו-AF750 (Cy7) בשל האפשרות של אוטופלואורסצנטיות.
הערה: שלב קריטי זה לוקח בחשבון את רגישות הערוצים ושפע האנטיגן. השימוש בערוץ AF488 עבור רקמת FFPE אינו מומלץ בשל האוטופלואורסצנטיות הגבוהה יותר שלו. - לצימוד נוגדנים-ברקוד (4.5 שעות), השג את ערכת צימוד הנוגדנים. אחסן את כל הריאגנטים ב-4 מעלות צלזיוס למעט תמיסת הפחתה 1, אותה יש לאחסן ב-20 מעלות צלזיוס כבקבוקונים קטנים לשימוש חד פעמי.
- למדוד את ריכוז הנוגדנים באמצעות ספקטרופוטומטר.
- התחל צימוד נוגדנים על ידי החלת 500 מיקרוליטר של תמיסת חסימת המסנן על עמודת מסנן MWCO של 50 kDa כדי לחסום את הקישור הלא ספציפי של נוגדנים למסנן. יש להסתובב ב-12,000 x גרם למשך 2 דקות בטמפרטורת החדר (RT). הסר נוזל נוסף מהעמודה באמצעות מיקרופיפטה של 200 מיקרוליטר.
הערה: תמיסת הפחתה 1 היא בקבוקון תמיסה חד פעמי, המספיק לשלושה צימודי נוגדנים בכל פעם. אין לעשות שימוש חוזר בתמיסה לאחר ההפשרה; השלך את כל המגיב שנותר. אל תשתמש במסנן מעל עמודת המסנן של 50 kDa MWCO, מכיוון שהדבר עלול לגרום לטיהור וצימוד לקויים. - השתמש בנפח שווה ערך של 50 מיקרוגרם של תמיסת הנוגדן והוסף אותו למסנן MWCO של 50 kDa. התאם את הנפח הכולל ל-100 מיקרוליטר באמצעות PBS במידת הצורך. יש להסתובב בטמפרטורה של 12,000 x גרם למשך 8 דקות בטמפרטורה של 4 מעלות צלזיוס. במהלך תקופה זו, הכינו את תערובת האב להפחתה באמצעות 19.8 מיקרוליטר של תמיסת הפחתה 1 + 825 מיקרוליטר של תמיסת הפחתה 2 לשלושה צימודי נוגדנים.
- השליכו את הזרימה והוסיפו 260 מיקרוליטר של תערובת אב הפחתה לחלק העליון של כל יחידת מסנן. תמיסת מערבולת ביחידת המסנן למשך 2-3 שניות ודגירה למשך 30 דקות ב-RT.
הערה: חשוב לא לחרוג מ-30 דקות דגירה כדי למנוע הפחתת יתר של נוגדנים, מכיוון שהדבר פוגע בהם ועלול לגרום לצימוד כושל. - לאחר 30 דקות דגירה, סובב כלפי מטה ב-12,000 x גרם למשך 8 דקות ב-4 מעלות צלזיוס, והשליך את הזרימה בתחתית. הוסף 450 מיקרוליטר של תמיסת צימוד לחלק העליון של העמודה וסובב מטה ב-12,000 x g למשך 8 דקות ב-4 מעלות צלזיוס. במהלך הצנטריפוגה, הכן את פתרון הברקוד CODEX. זוז במהירות לאחר אחזור הברקוד מ-20 מעלות צלזיוס, כאשר הברקוד מתחיל להתקלקל.
הערה: ברקודים מסופקים בדרך כלל בבקבוקוני זכוכית קטנים המכילים מוצרים ליופיליים (פתיתים או אבקה) המאוחסנים בטמפרטורה של -20 מעלות צלזיוס. ניתן להשתמש בבקבוקונים אלה כדי להצמיד 50 מיקרוגרם של נוגדן בבת אחת. מומלץ לא להצמיד יותר משלושה נוגדנים בכל פעם. - אתר בזהירות את הברקוד הליופילי בתחתית בקבוקון הזכוכית (הוא עשוי להיות דבוק גם לקירות). הקש על בקבוקון הזכוכית על שולחן כדי להביא את המוצקים לתחתית. איתור המוצר הליופילי חיוני בעת המסתו ב-10 מיקרוליטר של מים בדרגת ביולוגיה מולקולרית נטולת נוקלאז. לאחר הוספת 10 מיקרוליטר של מים נטולי נוקלאז, הוסף 210 מיקרוליטר של תמיסת צימוד לכל ברקוד. ממיסים את כל החומר הליופילי ומערבבים בעדינות על ידי פיפטינג למעלה ולמטה. מניחים בצד.
- לאחר השלמת הסחיטה בשלב 2.2.5, השלך את הזרימה והוסף את פתרון הברקוד המתאים משלב 2.2.6 לחלק העליון של כל יחידת סינון. שמור 1 מיקרוגרם מהנוגדן הראשוני הלא מצומד; זה יופעל יחד עם דגימת הנוגדנים המצומדת של 5 מיקרוליטר במהלך אימות אלקטרופורזה של ג'ל.
- סמן כל צינור עם הנוגדן ושם הברקוד המתאימים. סוגרים את המכסה ומערבבים את התמיסה על ידי מערבולת למשך 2-3 שניות. דגור על תגובת צימוד הנוגדנים-ברקוד למשך שעתיים ב-RT.
- לאחר הדגירה של שעתיים, הסר 5 מיקרוליטר של נוגדן מצומד ואחסן בצינור PCR של 0.2 מ"ל כדי לאשר את הצימוד על ידי אלקטרופורזה בג'ל.
- סובב את תמיסת הנוגדנים המצומדת הנותרת ב-12,000 x גרם למשך 8 דקות ב-4 מעלות צלזיוס. הוסף 450 מיקרוליטר של תמיסת טיהור לראש העמודה, סובב ב-12,000 x גרם למשך 8 דקות ב-4 מעלות צלזיוס, והשליך את הזרימה דרך. חזור על שלב טיהור זה פעמיים, הוסף תמיסת טיהור של 450 מיקרוליטר בכל פעם וסובב אותו כלפי מטה.
- לאחר הצנטריפוגההשלישית , השליכו את הזרימה דרכם. המסנן העליון יכיל את הנוגדנים המצומדים בברקוד.
- כדי לאסוף את הנוגדן המצומד, סמן את הצינור החיצוני החדש המחזיק את עמודת המסנן עם הנוגדן והברקוד המתאימים. חותכים את המכסה מהצינור החיצוני לפני הצנטריפוגה. הוסף 100 מיקרוליטר של תמיסת אחסון נוגדנים לכל יחידת סינון והנח את הצינור החיצוני החדש שסומן הפוך על גבי עמודת המסנן.
- הפוך את עמודת המסנן כדי לאסוף את הנוגדן המצומד לתוך הצינור החיצוני וסובב מטה ב-3,000 x g למשך 2 דקות ב-RT. זה אמור לאסוף כ-120 מיקרוליטר של תמיסת נוגדנים מצומדת. מעבירים לצינור מיקרוצנטריפוגה סטרילי ומאחסנים בטמפרטורה של 4 מעלות צלזיוס למשך 18-24 חודשים.
הערה: כדי למנוע כתמים גרעיניים ברקע גבוה לאחר הצימוד, מומלץ לא להשתמש בנוגדנים אלה במשך יומיים לפחות.
- בצע אישור צימוד על ידי אלקטרופורזה של ג'ל כמתואר בפרוטוקול13 שכבר פורסם.
הערה: תהליך זה רק מאשר את הצלחת תגובת הצימוד הכימית המשמשת לצימוד הברקודים עם נוגדנים. זה אופציונלי, שכן אימות נוגדנים אפשרי רק לאחר צביעה והדמיה מוצלחת של הרקמה.
3. הכנת דגימת FFPE של עכברים
- השג דגימות רקמת FFPE מעכברים נקבות C57BL/6J שהושתלו בגידולי צד B16F10. טפל בעכברים עם זריקות תוך-גידוליות של ננו-חלקיקים מבוססי פולי (β-אמינו אסטר) המכילים פלסמידים 4-1BBL ו-IL-12, יחד עם אספקה תוך-צפקית של אנטי-PD1 ב-9, 11, 16 ו-18 ימים לאחר ההשתלה14. הקריבו עכברים ביום ה-20 וקבעו גידולים בפורמלין 10%, משובץ בפרפין, ואז חתכו בליבת שירותי הרקמות האונקולוגיות של ג'ונס הופקינס.
- שקול היטב את מפרטי אזור ההדמיה המופיעים במדריך למשתמש והתקן דגימות של 5 מיקרומטר על שקופית בתוך אזור ההדמיה. הצלחנו להרכיב ארבע דגימות גידול שונות על שקופית אחת.
הערה: חיוני להשתמש בשקופיות המומלצות הזמינות להדמיה, כגון Leica Apex Adhesive Slides או Fisherbrand Superfrost Plus Slides. זה יעזור לתא הזרימה להיצמד היטב לשקופית.
4. צביעה והדמיה של רקמות FFPE
- אימות ואופטימיזציה של ריכוז הנוגדנים וזמני החשיפה: כדי לאמת את הצלחת צימוד נוגדנים-ברקוד, צבעו את הרקמה המעניינת ב-6-9 נוגדנים מצומדים. הפעל את מכשיר ההדמיה המולטיפלקס ושים לב לכל ריכוז נוגדנים וזמן חשיפה. התאם את הדילולים וזמני החשיפה לפי הצורך, ולאחר מכן הפעל 6-9 נוגדנים חדשים עם אלה שאומתו בעבר ושים לב להתאמות. בצע את השלבים הבאים לצביעה והדמיה.
- צביעת רקמות והדמיה
- יום לפני הצביעה, אופים את שקופיות הטישו FFPE בתנור של 60 מעלות למשך הלילה. למחרת, קררו את המגלשות למשך 10 דקות ב-RT לפני שתתחילו בשלבי הדפראפיניזציה והחזרת הרקמות.
- התחל דה-פרפיניזציה של רקמות על ידי דגירה בתמיסת קסילן פעמיים למשך 5 דקות כל אחת.
- כדי להחזיר לחות לרקמה, העבירו אותה דרך שני סבבים של תמיסת אתנול 100% למשך 5 דקות כל אחד. לאחר מכן, העבירו אותו דרך סדרה של תמיסות אלכוהול מ-90% ל-70%, 50% ו-30% למשך 5 דקות כל אחד. לבסוף, שטפו את הטישו במים מזוקקים פעמיים למשך 5 דקות כל אחת.
הערה: ניתן להשתמש בחלופות פחות רעילות לקסילן, כגון תמיסת Neoclear, לשלבי דה-פרפיניזציה. בצע דה-פרפיניזציה והחזרת נוזלים מתחת למכסה אדים. - יש לדלל 10x AR9 buffer ל-1x באמצעות מים מזוקקים. מלאו צנצנת קופלין ב-50 מ"ל של מאגר 1x AR9, וודאו שהשקופיות שקועות לחלוטין במאגר אחזור האנטיגן.
- מלאו סיר לחץ במים כך שהמפלס יהיה באמצע צנצנת קופלין, ואז דגרו בהגדרת לחץ גבוה למשך 20 דקות.
הערה: אפשר להשתמש בסיר לחץ מיוחד או בתא השרייה לאחזור אנטיגן טוב יותר על ידי הגדרתו ל-120 מעלות צלזיוס למשך 10 דקות. - לאחר השלמת שלב סיר הלחץ, יש לקרר את המגלשות למשך 45-60 דקות. שוטפים את השקופית במים מזוקקים פעמיים למשך 2 דקות כל אחד. לאחר מכן, העבירו את השקופית לצנצנת המכילה 1x PBS.
- כדי למזער אוטופלואורסצנטיות, בצע שלב הלבנה על הרקמה לפני הצביעה בנוגדנים. הכינו טרי תמיסת הלבנה (25 מ"ל של 1x PBS + 0.8 מ"ל של 1M NaOH + 4.5 מ"ל של H2O2) באמצעות הריאגנטים והריכוזים המומלצים. דגרו את השקופיות במיכל פלסטיק המכיל את תמיסת ההלבנה בין שתי מנורות לד למשך 45 דקות בטמפרטורת החדר. חזור על הפעולה עם תמיסת הלבנה טרייה למשך 45 דקות נוספות.
- התחל להכין את תמיסת קוקטייל הנוגדנים בשלב הלבנה זה, במיוחד אם ישנם מספר נוגדנים בפאנל עם דילולים שונים. אחזר את כל ארבעת החוסמים (G, S, J ו-N) והנח אותם על קרח, מכיוון שלחלקם לוקח זמן להפשיר מ-20 מעלות צלזיוס.
- לאחר ההלבנה יש לשטוף את הטישו עם 1x PBS, 2x למשך 2 דקות כל אחד. העבירו את הטישו לצנצנות המכילות חוצץ לחות ושטפו פעמיים למשך 2 דקות כל אחת.
- העבירו את שקופיות הרקמה למאגר הצביעה ואפשרו להן להתייצב למשך 30 דקות. אם לא מוכנים בשלב ההלבנה, הכינו את תמיסת קוקטייל הנוגדנים במהלך תקופה זו.
- הכן 300 מיקרוליטר של תמיסת קוקטייל נוגדנים כדי להכתים שתי שקופיות (בדרך כלל, אם משתמשים בתא פלסטיק היברידי, 100-120 מיקרוליטר של קוקטייל נוגדנים מספיקים כדי להכתים כל שקופית.)
- הכתים את השקופיות על ידי מריחת תמיסת קוקטייל נוגדנים בתוך תא הלחות למשך הלילה בטמפרטורה של 4 מעלות צלזיוס.
הערה: כל הנוגדנים כאן מראים אות חיובי כאשר הם מוכתמים במהלך הלילה ב-4 מעלות צלזיוס. עם זאת, זמן הצביעה והטמפרטורה של כל נוגדן עשויים להשתנות ויש לייעל אותם. תנאי הצביעה הנפוצים ביותר הם 3 שעות ב-RT או לילה ב-4 מעלות צלזיוס. במידת הצורך, ניתן גם לצבוע כמה נוגדנים בתחילה בטמפרטורות שונות ולאחר מכן את הנוגדנים הנותרים למשך הלילה ב-4 מעלות צלזיוס. שטפו טישו לאחר מכן עם PBS אם יש צורך בצביעה רציפה. - למחרת, בצע קיבוע לאחר צביעה. הסר את תא הפלסטיק ואחסן את קוקטייל הנוגדנים שנאסף בטמפרטורה של 4 מעלות צלזיוס. שטפו את השקופיות פעמיים במאגר הצביעה למשך 2 דקות כל אחת. לאחר מכן, העבירו את השקופיות לתמיסת קיבוע לאחר צביעה של 40 מ"ל (4 מ"ל של 16% פרפורמלדהיד + 36 מ"ל של מאגר אחסון) ודגרו למשך 10 דקות. יש לשטוף עם PBS 3 פעמים למשך 2 דקות כל אחד.
- העבירו את המגלשות למתנול קר כקרח למשך 5 דקות, ולאחר מכן שטפו עם PBS 3x למשך 2 דקות כל אחת. מרחו תמיסת קיבוע סופית (20 מיקרוליטר צינור אחד + 1 מ"ל PBS) על המגלשות מתחת לתא לחות. בדרך כלל, 200 מיקרוליטר של תמיסת הקיבוע מספיקים לכל שקופית. אפשר לקיבוע לשבת במשך 20 דקות ב-RT. בצע שלוש שטיפות אחרונות ב-PBS למשך 2 דקות כל אחת.
הערה: אל תסיר את צינור הקיבוע מ-20 מעלות צלזיוס לפני כן; יש להפשיר רק לפני היישום. - אם הדמיה מיד, נגב את השקופית סביב הרקמה באמצעות רקמה נטולת מוך והחל את תא הזרימה על ידי לחיצה עליו למשך 30 שניות באמצעות ציוד להרכבת תאי זרימה. אם הדמיה מאוחר יותר, אחסן את השקופית (מבלי להחיל את תא הזרימה) במאגר האחסון ב-4 מעלות צלזיוס. כאשר אתה מוכן לצילום, העבר את השקופיות ממאגר האחסון ל-PBS למשך 10 דקות לפני החלת תא הזרימה.
- התחל להכין מאגר ריצה 1x עם תוסף על סמך המחזורים הרצויים בניסוי. הכן מאגרי DMSO נמוכים (1:4) וגבוהים (9:1) על ידי ערבוב כ-DMSO של חלק אחד עם מאגר רץ של 4 חלקים ו-DMSO של 9 חלקים עם מאגר ריצה של חלק אחד הנדרש למחזורים הרצויים. חסוך כ-20 מ"ל של מאגר 1x עם תוסף להכנת לוחית עיתונאי. כדי לחשב את המאגר הנדרש 1x עם מאגרי תוסף ומאגרי DMSO גבוהים/נמוכים, התייעץ עם מנהל המכשירים.
- התחל להכין את לוחית הכתב. ראשית, הפוך את פתרון מלאי הכתב הנדרש למספר המחזורים הכולל. סמן צינורות מיקרו-צנטריפוגה שחורים או בצבע ענבר של 1 מ"ל עם שמות מחזורים. הוסף 250 מיקרוליטר של תמיסת מלאי מדווח ו-5 מיקרוליטר מכל מדווח לצינור המתאים. לאחר מכן, העבירו את התמיסה לצלחת שחורה של 96 בארות ואטמו אותה בנייר כסף.
- הפעל את המכשיר, השתמש במנהל המכשירים כדי להגדיר את הפרמטרים וזמני החשיפה (לפי טבלה 1), ורכוש את התמונות. איור 1 מסכם את התהליכים לפני ואחרי הצביעה.
5. הערת רקמות ופילוח תאים
- הגדרת פרוייקט וסביבת עבודה.
- התקן את הגרסה העדכנית ביותר של תוכנת ניתוח פתולוגיה דיגיטלית (למשל, QuPath גרסה 0.5.1 ומעלה). לחץ על צור פרויקט כדי לבחור תיקיית יעד עבור שטח הפרויקט.
- לחץ על הוסף תמונות > בחר קבצים, ולאחר מכן נווט לקובץ QPTIFF (המופק מהדמיה אימונופלואורסצנטית מרובה). קבעו את סוג התמונה ל'פלואורסצנטי', שמרו על כל קביעות ברירת המחדל האחרות ולחצו על 'ייבוא'.
- אם אתה משתמש ב- QuPath, לחץ פעמיים על התמונה החדשה כדי לפתוח סביבת עבודה. לחץ על קובץ > שמור מעת לעת כדי לעקוב אחר שינויים שבוצעו בפרוייקט. החלף את הגדרות הנראות והתצוגה של הסמן באמצעות Brightness & contrast (סמל חצי ירח) ליד אמצע סרגל הכלים.
הערה: לחץ לחיצה ימנית על תמונה ברשימת התמונות כדי לשנות את שמה. זה יהיה חשוב מאוחר יותר בעת הדמיית סיווגי פנוטיפ.
- ביאורים לקטע רקמה מלא, תא תוך-גידולי ותא סטרומל.
- השתמש בכלי המברשת ו/או השרביט כדי לצייר הערה סביב כל קטע הרקמה. הגדר ביאור זה כ-Full_Tissue. אל תכלול מעטפת עליונה ו/או אזורים שאין לנתח. כדי ליצור ביאור שלילי ו/או לכווץ את גבול הביאור, החזק את מקש Alt תוך שימוש באחד מכלי הביאור.
- שכפל את ביאור הרקמה המלא. בחר את ההערה תחת הכרטיסייה ביאורים, ולאחר מכן עבור אל אובייקטים > ביאורים... > שכפול ביאורים שנבחרו.
- הפעל את ערוץ SOX10. בביאור המשוכפל, כווץ את גבול ההערות כדי ללכוד את התא התוך-גידולי באמצעות כלי המברשת/שרביט Alt +. הגדר את ההערה הזו כגידול (איור 2).
- בחירת ביאור הרקמה המלא, עבור אל אובייקטים > ביאורים... > הרחב ביאורים. הגדר את רדיוס ההרחבה כ-1 מיקרומטר ולחץ על הפעל.
- שנה את שם הביאור החדש ל-Full_Tissue_Expansion. בחר את ביאור הגידול, לחץ באמצעות לחצן העכבר הימני ובחר הוסף בהירארכיה.
- בחר מחדש את ביאור הגידול, עבור אל אובייקטים > ביאורים... > עשה הפוך. הגדר את ההערה החדשה הזו כ-Stroma. חזור על הפעולה עבור כל חלקי הרקמות.
- הפעל פילוח תאים וייצא תוצאות.
- עבור אל הכרטיסייה תמונה כדי למצוא את רוחב הפיקסלים והגובה של התמונה (איור 3A).
- הורד את התוסף StarDist בשעה https://github.com/qupath/qupath-extension-stardist/releases. גרור את הקובץ qupath-extension-stardist-[version].jar לחלון QuPath, ולאחר מכן לחץ על סמל גלגל השיניים בפינה השמאלית העליונה של חלון QuPath. תחת הרחבות, הגדר את ספריית המשתמש של QuPath כמיקום של התיקיה StarDist.
- הורד את קבצי הסקריפט הגרוביים של StarDist ואת קובץ המודל StarDist מ-https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (סקריפטים של StarDist אורגנו במקור מ-Akoya Biosciences).
- עבור כל מקטע רקמה, בחר גם את הביאורים גידול וגם סטרומה (השתמש ב-Ctrl כדי לבחור ביאורים מרובים). בסרגל ההגדרות העליון, לוחצים על אוטומציה של עורך הסקריפטים > כדי לפתוח את ממשק הסקריפטים. פתח את סקריפט פילוח התאים המתאים של StarDist (קובץ גרובי) המתאים לגודל הפיקסלים של התמונה. כאשר תתבקש לבחור את קובץ פילוח התאים, פתח את הקובץ stardist_cell_seg_model.pb.
- לאחר פילוח התאים, שמור את התמונה. עבור אל מדידה > ייצוא מדידות ולאחר מכן בחר את התמונות המתאימות. הקצה את סוג הייצוא כתאים ואת המפריד כ-.csv ולאחר מכן בחר את מיקום קובץ הפלט . לחץ על אכלס לצד עמודות כדי לכלול מדדים מעניינים. עמודות חשובות לייצוא הן תמונה, מזהה אובייקט, אב, מרכז X, מרכז Y ואזור גרעין.
הערה: מזהה האובייקט משמש בשלבים הבאים כדי למפות מחדש סיווגים בחזרה לתמונת האימונופלואורסצנציה המולטיפלקס בתוכנת ניתוח הפתולוגיה הדיגיטלית. העמודה אב מציינת את שם הביאור שאליו שייך התא. נתוני X ו-Y משמשים לניתוח מרחבי. - עבור כל סמן שושלת שישמש באשכולות/פנוטיפ, בחר ערך ממוצע אחד לייצוא. עבור סמנים גרעיניים (למשל, SOX10 ו-FOXP3), ייצא את האפשרות Nucleus: Mean. עבור סמנים ציטופלזמיים/קרומיים (למשל, CD45, CD3, CD4), ייצא את הציטופלזמה: אפשרות ממוצעת.
6. עיבוד מקדים ונורמליזציה של נתוני פרוטאומיקה
- פתח את קובץ ה- CSV המיוצא. כדי שכל סמן ישמש באשכולות/פנוטיפ, חתוך את שם כותרת העמודה כך שיכלול רק את הסמן. לדוגמה, שנה את שם SOX10: גרעין: ממוצע ל- SOX10 ו- CD3: ציטופלזמה: ממוצע ל- CD3.
- התקן את הגרסה העדכנית ביותר של R ו-RStudio. הורד את הקובץ Marker Normalization.R מ-https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis והפעל את קובץ ה- Script. זה כרוך תחילה בסינון תאים לפי גודל גרעיני.
- עבור כל סמן, לאחר מכן מתבצעת נורמליזציה מינימלית-מקסימלית כך שערך ה-MFI הנמוך ביותר בטווח הביטוי מוגדר ל-0 וערך ה-MFI באחוזוןה-99.7 מוגדר ל-1. כל העוצמות מעל האחוזוןה-99.7 נחתכות ב-1. לאחר השלמתו, שמור את קובץ ה- CSV החדש שנוצר עם הנתונים המנורמלים.
7. אשכולות ופנוטיפ
- בחר אלגוריתם לאשכול תאים לפי פרופילי ביטוי סמן. ב-https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis, סקריפטים R מסופקים לאשכולות באמצעות Seurat v4.415 ו-FlowSOM v2.13.9 (כלי אשכולות והדמיה)16.
- תאי פנוטיפ על פי פרופילי ביטוי הסמנים של סמני שושלת מפתח. הפרוטוקול המוצג כאן השתמש ב-14 סמני שושלת עבור אשכולות ופנוטיפים: SOX10, CD45, CD3, CD4, CD8, FOXP3, CD11C, F4/80, CD68, CD86, CD163, CD206, NK1.1 ו-CD31.
- הגדר תאי גידול כ-SOX10היי. הגדר תאי CD4 T כ-CD45mod-hi/ CD4hi/CD8נמוך/FOXP3נמוך. הגדר תאי CD8 T כ-CD45mod-hi/ CD8hi/CD4נמוך. הגדר Tregs כ-CD45mod-hi/CD4hi/CD8low/FOXP3hi. הגדר תאי T אחרים כ-CD45mod-hi/CD3hi/CD4נמוך/CD8נמוך.
- הגדר מקרופאגים M1 כ-CD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163low /CD206נמוך. הגדר מקרופאגים M2 כ-CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206נמוך, CD45mod-hi/F4/80hi/CD68mod-hi/CD86נמוך/CD163נמוך/CD206hi, או CD45mod-hi/F4/80hi/CD68mod-hi/CD86נמוך/CD163hi/CD206hi. הגדירו מקרופאגים אחרים כ-CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/ CD163low/CD206low או CD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hi.
- הגדר תאים דנדריטיים כ-CD45mod-hi/CD11chi/CD86mod-hi. הגדר תאי NK כ- CD45mod-hi/NK1.1hi. הגדירו תאים חיסוניים אחרים כ-CD45mod-hi שלא עמדו בתנאים המפורטים לעיל. הגדר תאי אנדותל כ-CD31mod-hi. הסר את כל פרופילי הביטוי האחרים.
8. מיפוי מחדש של סיווגי פנוטיפ וביצוע בקרת איכות
- הורד את הסקריפט Remapping Classifications.groovy מ-https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (סקריפט זה הותאם מפוסט ב-https://forum.image.sc/ מאת petebankhead).
- פתח את קובץ ה- CSV הפנוטיפי וודא שכל התוויות תחת הכרטיסייה תמונה תואמות בדיוק לשם קובץ התמונה בתוכנת ניתוח הפתולוגיה הדיגיטלית שתשמש למיפוי מחדש. כדי לשנות כמויות גדולות של תוויות במהירות בקובץ ה- CSV, השתמש בפונקציה החלף הכל.
- אם אתה משתמש ב-QuPath, הפעל את הסיווג למיפוי מחדש של קובץ גרובי על-ידי לחיצה על אוטומציה של עורך > Script.
- בכרטיסייה ביאורים, לחץ על שלוש הנקודות לצד כפתור ההגדרה האוטומטית, ולאחר מכן לחץ על אכלוס מאובייקטים קיימים > כל המחלקות (כולל תת-מחלקות) > כן.
- גלול מטה ברשימת הסמנים כדי להציג את סיווגי הפנוטיפים השונים ולהתאים את ערכת הצבעים לפי הצורך. לחץ פעמיים על תא כלשהו כדי להציג את הפנוטיפ שלו (לצד תג הסיווג) ואת האשכול (לצד תג השם). בדוק שדות ראייה מייצגים כדי לקבוע את הדיוק של הקצאות פנוטיפ ולשנות את אסטרטגיית הפנוטיפ לפי הצורך (איור 3B).
9. כימות צפיפות וניתוח מרחבי
- לאחר סיום הפנוטיפ, השג אזורי הערות בתוכנת ניתוח הפתולוגיה הדיגיטלית תחת הכרטיסייה ביאורים. המר ערכי שטח למ"מ2, ולאחר מכן חלק את הספירות באזורי הערות כדי לחשב צפיפות.
- הורד את סקריפט SPIAT Spatial Analysis.R מ-https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis והפעל. אסוף נתונים על מרחקי מינימום ממוצעים (AMD), הרכבי שכונה וציוני ערבוב מנורמל (NMS).
- הפקת נתונים בתוכנת ניתוח סטטיסטי להצגת ממצאים. בעת יצירת מפת חום בודדת למרחקים מינימליים ממוצעים של תנאי טיפול, ממוצע את התוצאות המתקבלות מקטעי רקמה שונים.
תוצאות
כאן, אנו מציגים פרוטוקול לתכנון פאנל נוגדנים לרקמת FFPE של עכברים, ביצוע הדמיה אימונופלואורסצנטית מרובה וניתוח תמונות לכימות פרוטאומיקה ויחסים מרחביים. הפאנל המאומת מכיל 27 נוגדנים המספקים סמנים להמחשת תאי מלנומה (SOX10), לויקוציטים (CD45), תאי T (CD3, CD4, CD8, FOXP3), תאי B (CD20), מקרופאגים ותתי סוגים (F4/80, CD68, CD86, CD163, CD206), תאים דנדריטיים (CD11c), תאי NK (NK1.1) ותאי אנדותל (CD31). הפאנל המלא מכיל גם סמנים לאוכלוסיות חיסוניות אחרות (CD11b, CD38), פעילות התפשטות (Ki67), פונקציונליות של תאי T (T-bet, Eomesodermin, granzyme B), הצגת אנטיגן (LMP2, בטא-2 מיקרוגלובולין, MHC II) וביטוי נקודות ביקורת (TIM3, LAG3, PD-L1)12. תמונת אלקטרופורזה מייצגת של ג'ל מאשרת את הצימוד המוצלח של ברקודי אוליגונוקלאוטידים של DNA לנוגדנים נטולי נשאים, כפי שמוצג באיור 4. שלב זה רק מאשר את התגובה הכימית, ואישור תמונה יכול להיעשות רק לאחר בדיקת נוגדנים אלה עם מכשיר ההדמיה המולטיפלקס על הרקמה המעניינת. ראו את ההפניה הבאה להצגת תמונות מאומתות של כל 27 הסמנים בחלונית13. תמונת היתוך של סמני שושלת מרכזיים המכתימים שלושה קטעי רקמת מלנומה של עכברים שטופלו בהזרקות ננו-חלקיקים תוך-גידוליות 4-1BBL/IL-12 ואנטי-PD1 מערכתי מוצגת באיור 5. סעיפים אלה שימשו לניתוח תמונה לאחר מכן בפרוטוקול זה.
לאחר הערת רקמות, פילוח תאים ועיבוד מקדים של נתוני פרוטאומיקה, אנו מציגים השוואה בין שני אלגוריתמי אשכולות שיושמו בעבר עבור ניתוח טרנסקריפטומי ו/או פרוטאומי של תא בודד17,18. פרופילי ביטוי עבור אוכלוסיות פנוטיפיות מוצגים באיור 6 עבור שתי הגישות. אנו מראים ש-FlowSOM מציע טווח רחב יותר של ערכי עוצמה (~0.7 לעומת ~0.5) להבחנה בהבדלים בין אוכלוסיות תאים דומות, כגון תת-סוגים של מקרופאגים. Seurat יישם את אלגוריתם Louvain במהלך אשכולות ויצר 29 אשכולות (איור משלים 1). לשם השוואה, FlowSOM יישם מפות בארגון עצמי ויכול ליצור 100 אשכולות (איור משלים 2). מספר גדול יותר של אשכולות מתורגם לזמן רב יותר הנדרש לפנוטיפ, אך גישת FlowSOM האחרונה מציעה גם ניואנסים רבים יותר בעת סיווג אוכלוסיות תאים דומות. מבחינה איכותית, אנו רואים ש-FlowSOM הצליח לסווג יותר מקרופאגים תוך-גידוליים כתת-סוג M1 או M2 בהשוואה לפנוטיפ Seurat באותו שדה ראייה (איור 7). אותה תוצאה נראית כאשר אנו מכמתים את צפיפות המקרופאגים, כאשר FlowSOM לוכד צפיפות גבוהה יותר של מקרופאגים M1 ו-M2 בהשוואה ל-Seurat (איור 8A-B) ובהמשך צפיפות נמוכה משמעותית של מקרופאגים אחרים/לא מסווגים (p = 0.0028; איור 8C). עם זאת, שתי גישות הניתוח הניבו גם תוצאות דומות כאשר תיארו צפיפויות אוכלוסיית תאים אחרות, כגון תאי CD4 T, תאי CD8 T ותאי אנדותל (איור 8D-F).
אנו מציגים גם את ממצאי הניתוח המרחבי במורד הזרם לאחר אשכולות ופנוטיפים. איור 9 מדגים כמה מהמדדים המרחביים שניתן ליצור באמצעות פרוטוקול זה. יחסית לכל האוכלוסיות הפנוטיפיות, למקרופאגים M2 ולתאי NK היו ה-AMD הגבוהים ביותר לאחר טיפול בננו-חלקיקים 4-1BBL/IL-12 ואנטי-PD1 (איור 9A). באופן דומה, NMS בין תאי CD8 T תוך-גידוליים למקרופאגים M1 היה גבוה בהרבה מאשר בין תאי CD8 T למקרופאגים M2 (איור 9B). יתר על כן, מקרופאגים M2 תרמו ~1% בשכונת 100 מיקרומטר המקיפה תאי CD8 T תוך-גידוליים, בעוד שמקרופאגים M1 היוו 9%-13% מהשכונות הללו (איור 9C). יחד, תוצאות אלה מצביעות על כך שמשטר הטיפול 4-1BBL/IL-12 קיטב מקרופאגים הקשורים לגידול לכיוון תת-סוג M1 והוציא מקרופאגים M2 מהמיקרו-סביבה החיסונית של הגידול.
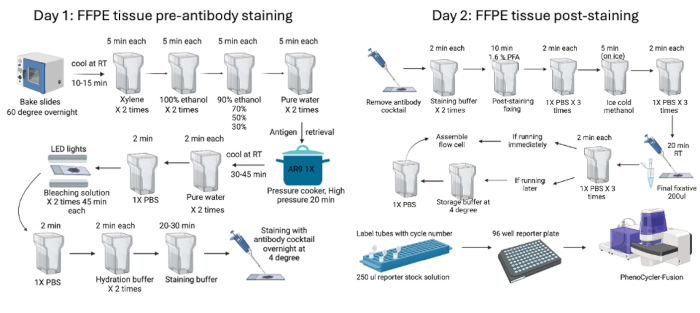
איור 1: סיכום זרימת העבודה של צביעת רקמות FFPE והדמיה. רקמות FFPE של עכברים עובדו באמצעות הליכי צביעה שהחלו באפיית הרקמה למשך הלילה לפני תחילת התהליכים של יומיים לפני הצביעה (יום 1) ואחרי הצביעה (יום 2). לבסוף, לוחית הכתב הוכנה לפני ההדמיה עם המכשיר. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
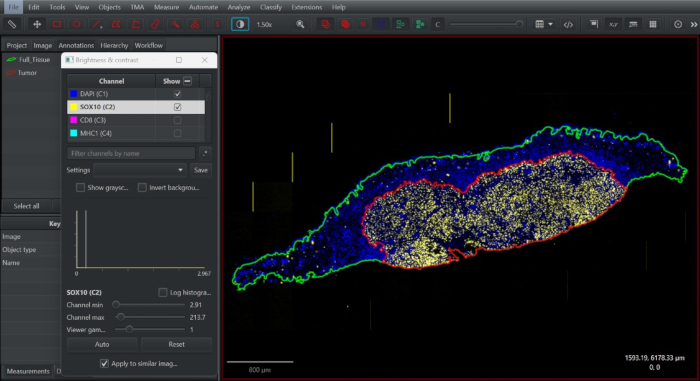
איור 2: צילום מסך של הערת רקמות בתוכנת הפתולוגיה הדיגיטלית בגישה פתוחה QuPath. הערת רקמה מלאה צוירה (ירוק), שוכפלה ומוזערה למטה כדי להגדיר את התא התוך-גידולי בהתאם להתפלגות המלנוציטים SOX10+ בגבול הגידול (אדום). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
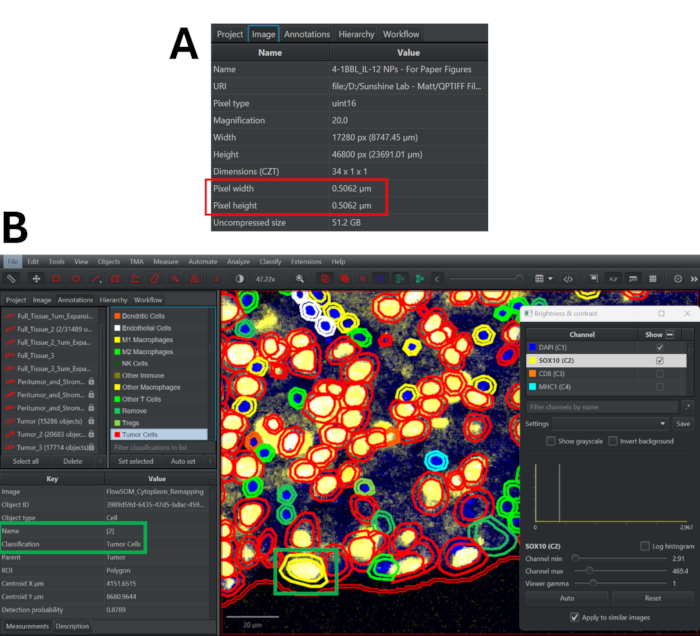
איור 3: פילוח תאים באמצעות אלגוריתם StarDist ב-QuPath ותהליך בקרת איכות לסקירת סיווגי פנוטיפ. (A) נווט אל הכרטיסייה תמונה כדי לקבוע את רוחב וגובה הפיקסלים של תמונת האימונופלואורסצנציה המולטיפלקס. (B) לאחר מיפוי מחדש של סיווגים, לחצו פעמיים על תא כלשהו (מודגש בצהוב) כדי לראות את הקצאת האשכול והפנוטיפ שלו. הפעל/כבה את סמני החלונית כדי לקבוע אם גישת סיווג זו מדויקת. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 4: תמונת ג'ל מייצגת של אישור צימוד ברקוד נוגדן-DNA. אלקטרופורזה של ג'ל חלבון מאשרת צימוד נוגדנים עם ברקודים של אוליגונוקלאוטידים של DNA, שנצפו על ידי להקות נוספות באתר השרשרת הכבדה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
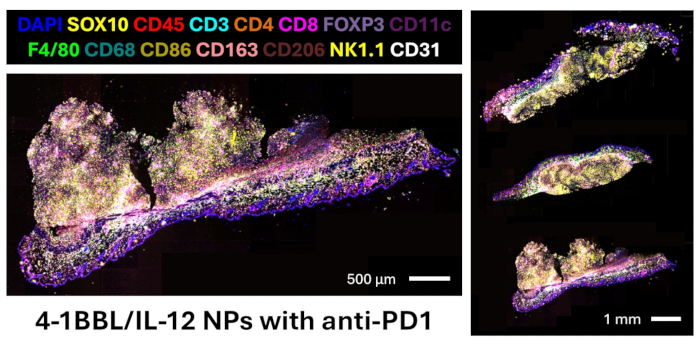
איור 5: הדמיית מולטיפלקס מבוססת ברקוד DNA של גידולי צד B16F10 שטופלו בהזרקות תוך-גידוליות של ננו-חלקיקים 4-1BBL/IL-12 עם אנטי-PD1 מערכתי. סמנים בפאנל שלנו לא מוצגים: CD11b, CD20, CD38, TIM3, LAG3, T-bet, Eomesodermin, granzyme B, Ki67, LMP2, בטא-2 מיקרוגלובולין, MHC II, PD-L1. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 6: מפות חום של ביטוי פרוטאומיקה של אוכלוסיות תאים פנוטיפיות. (A) גישת אשכולות ופנוטיפ Seurat מייצרת טווח מעט קטן יותר של ערכי ביטוי סמנים בהשוואה ל- (B) אשכולות ופנוטיפ של FlowSOM. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 7: פנוטיפ FlowSOM מזהה מגוון גדול יותר של תת-סוגים שונים של מקרופאגים. החלונית העליונה מציגה פנוטיפ מקרופאגים של Seurat (משמאל) ו-FlowSOM (מימין) עבור אותו שדה ראייה. הפאנל התחתון מציג תמונות אימונופלואורסצנטיות מרובות של סמני שושלת מקרופאגים מרכזיים באותו שדה ראייה. כל מוטות הסולם הם 20 מיקרומטר. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 8: השוואת צפיפויות תוך-גידוליות של אוכלוסיות תאים שונות לאחר אשכולות/פנוטיפ. השוואות צפיפות מוצגות עבור מקרופאגים תוך-גידוליים (A) M1, (B) מקרופאגים M2, (C) מקרופאגים אחרים, (D) תאי CD4 T, (E) תאי CD8 T ו-(F) תאי אנדותל. קווי שגיאה הם שגיאות תקן של הממוצע (SEM), והמובהקות נבדקה באמצעות מבחני t לא מזווגים (**p ≤ 0.01). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 9: פרופיל מדדים מרחביים תוך-גידוליים עבור גידולים בשלושה צדדים שטופלו בהזרקות ננו-חלקיקים תוך-גידוליים 4-1BBL/IL-12 ואנטי-PD1 מערכתי. (A) מפת חום של מרחקים מינימליים ממוצעים בין אוכלוסיות פנוטיפיות תוך-גידוליות. המדידות הן במיקרומטר. (B) ציוני ערבוב מנורמל בין זוגות פנוטיפ תוך-גידוליים מרכזיים. (C) פירוט הרכבים שכונתיים ברדיוס של 100 מיקרומטר סביב אוכלוסיות תאי CD8 T תוך-גידוליים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| נוגדן | דילול | זמן חשיפה (אלפיות השנייה) |
| SOX-10 | 50 | 450 |
| תקליטור 8 | 100 | 450 |
| תקליטור 3 | 100 | 450 |
| פוקס P3 | 50 | 350 |
| תקליטור 4 | 50 | 450 |
| MHC-II | 100 | 450 |
| PDL1 | 50 | 450 |
| CD45 | 100 | 450 |
| קי-67 | 100 | 300 |
| F4/80 | 100 | 150 |
| תקליטור 20 | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| תקליטור 206 | 100 | 450 |
| CD68 | 100 | 450 |
| גרנזים-בי | 50 | 450 |
| CD86 | 100 | 150 |
| תקליטור 31 | 100 | 450 |
| CD11C | 50 | 450 |
| CD11b | 50 | 450 |
| EOMES | 50 | 450 |
| טים-3 | 50 | 300 |
| תקליטור 38 | 50 | 200 |
| LAG3 | 50 | 450 |
| תקליטור 163 | 50 | 200 |
| טי-הימור | 50 | 300 |
| LMP2 | 100 | 100 |
| בטא 2 מ"ג | 200 | 50 |
טבלה 1: דילול נוגדנים והגדרות זמן חשיפה.
איור משלים 1: מפת חום ראשונית של ביטוי פרוטאומיקה שנוצרה לאחר אשכולות Seurat. אנא לחץ כאן להורדת איור זה.
איור משלים 2: מפת חום ראשונית של ביטוי פרוטאומיקה שנוצרה לאחר אשכול FlowSOM. אנא לחץ כאן להורדת איור זה.
Discussion
הצלחת ההדמיה תלויה בפאנל נוגדנים מתוכנן ומאומת היטב. הדמיה אימונופלואורסצנטית מרובה של דגימות FFPE מציגה אתגרים עקב אוטופלואורסצנציה גבוהה והקושי לאחזר אפיטופים מוסווים על ידי הטמעת פרפין. עם זאת, בהתחשב בכך ש-FFPE מציע מספר יתרונות בהשוואה לדגימות FF, חיוני לתכנן ולאמת לוחות נוגדנים FFPE. סיום שיבוטי הנוגדנים המראים אותות חיוביים במהלך הדמיית אימונופלואורסצנציה (IF) הוא הצעד הראשון; לאחר מכן, חשוב להצמיד אותם בזהירות עם ברקודי DNA. צימוד נוגדנים דורש הפחתה חלקית של הנוגדן ליצירת קשרי SH, המשמשים במהלך תגובת קבוצת המלימיד עם ברקוד. לא כל שיבוט נוגדן יכול לעמוד בשלב זה, ותגובות מסוימות עלולות לגרום לנזק בלתי הפיך לנוגדן, וכתוצאה מכך לכשל בהדמיה למרות צימוד מוצלח. מסיבה זו, למרות שחלק מהנוגדנים עשויים להראות אותות חיוביים במהלך אימות IF קונבנציונלי, כדי להעריך את ההצלחה הסופית של צימוד הנוגדנים, חשוב לתקף כל נוגדן על הרקמה המעניינת בפועל ולתעד את זמני החשיפה הרצויים לאותה רקמה. ביישומים עתידיים, ניתן לשלב טכניקה זו עם ניתוח טרנסקריפטומיקה מרחבית קיים בשקופיות עוקבות/אותה שקופית כדי ליצור תובנות נוספות. אחת המגבלות של שיטה זו היא שהיא דורשת בחירה ותיקוף קפדני של כל נוגדן בפאנל על סמך המטרה וסוג הרקמה.
בכל הנוגע לניתוח תמונה, QuPath מציעה כלי רב ערך לגישה פתוחה עם הדמיה איכותית של סמני פרוטאומיקה, פונקציונליות רחבה לייצוא מדידות עוצמה וביצוע בקרת איכות לסיווגי פנוטיפ, וגמישות טובה עבור סקריפטים שנוצרו על ידי משתמשים. פורומים מקוונים כגון https://forum.image.sc/ הם משאב נוסף לדיון כיצד לבצע משימות ניתוח ספציפיות ולשיתוף סקריפטים עם משתמשים אחרים. בפרוטוקול זה, אנו משווים שתי גישות אשכולות ופנוטיפים באמצעות Seurat ו-FlowSOM. בעוד ש-FlowSOM עשוי להיות מועדף בשל יכולתו לייצר תובנות מפורטות יותר לגבי תת-אוכלוסיות תאי חיסון של ה-TME, יש לקחת בחשבון גם את הזמן הנדרש לניתוח פרוטאומיקה. יצירת 100 אשכולות עשויה להיות מיותרת אם משתמש צריך רק תאים פנוטיפיים בתוך דגימת רקמה אחת או שתיים. במצבים אלה, Seurat עשוי להציע צינור מהיר ויעיל יותר לניתוח תמונה. לעומת זאת, ניתוח TMA עם למעלה מ-40 או 50 קטעי רקמה נוטה יותר לייצר מספר גדול יותר של אשכולות תאים בשתי גישות הניתוח, ו-FlowSOM עשויה להיות המתודולוגיה המועדפת ליצירת סיווגי פנוטיפ ניואנסים יותר.
אשכול תאים/פנוטיפ וכל שלבי ניתוח התמונה הבאים תלויים במידה רבה בפילוח התאים. העבודה הנוכחית שלנו חקרה פילוח תאים באלגוריתמים HALO (מעבדות אינדיקה) ו-StarDist, ומצאנו ששתי הגישות נוטות לפלח יתר של תאים על סמך אותות DAPI גרעיניים. קיימים גם אלגוריתמי פילוח חלופיים רבים, כגון Mesmer19 ו-InstanSeg20. זהו תחום הולך וגדל של מחקר חישובי הדורש חקירה ואופטימיזציה נוספת.
Disclosures
J.C.S. מודה על תמיכה כספית מאמרסון קולקטיב LLC והמכונים הלאומיים לבריאות. J.J.G. גם מכיר במימון מהמכונים הלאומיים לבריאות. ל-JCS יש מערכת יחסים עם Palleon Pharmaceuticals Inc., הכוללת מענקי מימון. ל-S.Y.T. ו-J.J.G. יש מערכת יחסים עם OncoSwitch Therapeutics הכוללת הון עצמי או מניות. ל-S.Y.T., J.J.G., J.C.S. ו-K.M.L. יש פטנט תלוי ועומד. כל שאר המחברים מצהירים כי לא ידוע על אינטרסים פיננסיים מתחרים או קשרים אישיים שיכולים להיתפס כמשפיעים על המחקר המוצג במאמר זה.
Acknowledgements
J.C.S. מכיר בקרן הדרמטולוגיה ובפרס פיתוח הקריירה של דרמטופתולוגיה על קידום הקריירה של המחבר. המחברים מודים ל-Hsin-Pei Lee במעבדה למדעי נתוני הסרטן של המכון הלאומי לסרטן על הסיוע בטכניקות ניתוח חישוביות. מחקר זה קיבל מימון מקולקטיב אמרסון ומהמכונים הלאומיים לבריאות (R37CA246699, P41EB028239 ו-R01CA228133). בנוסף, ליבת שירותי הרקמות האונקולוגיות של ג'ונס הופקינס (OTS) נתמכת על ידי המכונים הלאומיים לבריאות (P30CA006973).
Materials
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
References
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved