Method Article
Imágenes de inmunofluorescencia múltiple basadas en códigos de barras de ADN para analizar muestras de FFPE de melanoma murino reprogramado genéticamente
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo proporciona una guía paso a paso para diseñar un panel de anticuerpos de inmunofluorescencia multiplex para la obtención de imágenes basadas en códigos de barras de ADN de tejidos murinos de melanoma FFPE. También describimos una línea de análisis de imágenes que utiliza herramientas de código abierto para generar información proteómica espacial sobre el microambiente inmunológico del tumor de melanoma murino.
Resumen
Aquí se presenta una técnica emergente de imagen múltiple basada en códigos de barras de ADN basada en la co-detección por indEX que analiza la proteómica espacial de los microentornos tisulares. El éxito de la obtención de imágenes requiere un repertorio de paneles de anticuerpos bien diseñados y debidamente validados, pero actualmente existen muy pocos para muestras fijadas en formol e incluidas en parafina (FFPE). El FFPE ofrece varias ventajas sobre las muestras frescas congeladas, como la disponibilidad generalizada, la facilidad de manejo y almacenamiento, y la capacidad de hacer micromatrices de tejidos (TMA). Aquí, presentamos un protocolo para desarrollar un panel de anticuerpos para visualizar y analizar tejidos FFPE a partir de un modelo de melanoma murino tratado con nanopartículas, que entregan ADN plasmídico que codifica señales inmunológicas para la reprogramación del microambiente tumoral. También describimos una línea de análisis de imágenes que utiliza herramientas computacionales de código abierto para anotar tejidos, segmentar células, procesar datos proteómicos, fenotipar poblaciones de células y cuantificar métricas espaciales. El protocolo ofrece aplicaciones para el diseño de paneles de anticuerpos en FFPE murinos y la generación de nuevos conocimientos sobre la proteómica espacial de microentornos de tejidos complejos.
Introducción
El melanoma cutáneo es el cáncer de piel más común, con tasas de enfermedad y mortalidad variables en todo el mundo según el momento del diagnóstico y la atención primaria1. Durante la última década, una mayor comprensión biológica del melanoma ha ayudado a impulsar el desarrollo de nuevos modelos de cáncer para tratartumores sólidos. El reciente auge de la inmunoterapia ha dado lugar a un concepto revolucionario de tratamiento del cáncer basado en la activación del sistema inmunitario endógeno 3,4.
El microambiente tumoral (TME) es muy complejo y está formado por diversas células inmunitarias, fibroblastos asociados al cáncer, pericitos, células endoteliales y diversas células residentes en los tejidos5. En el pasado se han aplicado varias técnicas para estudiar la TME, como la citometría de flujo y la secuenciación de una sola célula, que comprometen el contexto espacial, ya que son necesarias para destruir el tejido tumoral. Las imágenes de microscopio tradicionales, como la inmunofluorescencia (IF) y la inmunohistoquímica (IHC), permiten la visualización de biomarcadores de proteínas sin destruir los tejidos de la muestra. Sin embargo, estos enfoques se limitan a dos o tres biomarcadores y no pueden proporcionar una comprensión completa de las relaciones espaciales y estructurales dentro del complejo TME 6.
Para abordar este problema, se han desarrollado varias técnicas de imagen múltiplex para visualizar espacialmente el complejo TME 7,8,9,10. Uno de ellos es la CoDetection-by-inDEXing, rebautizado como sistema PhenoCycler, basado en anticuerpos conjugados con oligonucleótidos de ADN11. El sistema puede proporcionar imágenes de una sola célula y análisis de más de 100 biomarcadores para muestras humanas. Sin embargo, se dispone de muy pocos anticuerpos inventariados para visualizar y analizar muestras murinas, en particular muestras de formol fijadas en parafina (FFPE)12. El FFPE ofrece varias ventajas sobre la conservación fresca congelada (FF), como la facilidad de manejo y almacenamiento, la morfología bien conservada a lo largo del tiempo y, lo que es más importante, la capacidad de preparar micromatrices de tejidos/tumores (TMA) que permiten la visualización de varias muestras en un solo portaobjetos. Recientemente diseñamos y desarrollamos un panel de anticuerpos murino FFPE CODEX/PhenoCycler y lo aplicamos con éxito para visualizar y analizar la proteómica espacial de especímenes de melanoma murino reprogramados genéticamente13.
El objetivo general de este protocolo es proporcionar una guía paso a paso para diseñar un panel de anticuerpos FFPE murino y describir el proceso de conjugación de anticuerpos-códigos de barras, tinción de tejidos e imágenes. Además, presentamos una canalización detallada de análisis de imágenes utilizando herramientas de código abierto como los paquetes QuPath y R. Después de seguir este protocolo, los investigadores aprenderán a diseñar un panel de anticuerpos conjugados a medida, realizar imágenes multiplexadas utilizando un dispositivo Phenocycler-Fusion y obtener nuevos conocimientos sobre la proteómica espacial del melanoma TME. Además, este protocolo puede adaptarse para estudiar diversos microambientes inmunes tumorales y combinarse con las técnicas de transcriptómica espacial existentes.
Protocolo
Todo el trabajo con animales se realizó de acuerdo con las pautas establecidas por el Comité de Cuidado y Uso de Animales de Johns Hopkins, utilizando los números de protocolo aprobados MO18M388 y MO21M384.
1. Selección de anticuerpos
- Diseñar un panel de anticuerpos basado en el tejido de interés y la abundancia del biomarcador en particular. Elija clones de anticuerpos en función de las aplicaciones anteriores exitosas de tinción de inmunofluorescencia (IF) e inmunohistoquímica (IHQ) en la literatura. Versiones seguras sin portadores de estos anticuerpos.
NOTA: Un anticuerpo purificado libre de portador es importante para la conjugación del código de barras. Evite los conservantes a base de proteínas como BSA, gluten, glicerol, etc. Si el anticuerpo libre de portadores no está disponible, solicite un anticuerpo personalizado al proveedor o use un kit de extracción de BSA para producir el anticuerpo libre de portadores. La azida sódica no interfiere con la conjugación de anticuerpos. Los isotipos de IgG se recomiendan sobre los isotipos de IgM. - Verificar estos clones seleccionados en el tejido de interés utilizando imágenes de inmunofluorescencia (IF) convencional11. Utilice imágenes de FI dobles para confirmar la especificidad de ciertos clones (por ejemplo, realice imágenes de FI dobles con FOXP3 y CD4 para confirmar la tinción de células T reguladoras).
NOTA: Siempre que sea posible, es importante utilizar el mismo tejido para la validación de FI que se utilizará para la tinción. Seleccione la solución de recuperación de antígeno adecuada. En este protocolo, utilizamos un tampón AR9 suministrado por Akoya Biosciences para recuperar todos los epítopos tisulares FFPE del melanoma murino. Se pueden utilizar otras soluciones de recuperación de antígenos, como AR6 o Universal, en función de los requisitos del epítopo.
2. Conjugación y confirmación de anticuerpos
- Asigne anticuerpos verificados a códigos de barras que tengan reporteros complementarios adjuntos a los fluoróforos ATTO550 (Cy3), AF647 (Cy5) o AF750 (Cy7). Los antígenos poco abundantes suelen producir señales más bajas; conjugan dichos anticuerpos con canales de autofluorescencia bajos como AF647 (Cy5). Conjugamos antígenos altamente expresados a ATTO550 (Cy3) y AF750 (Cy7) debido a la posibilidad de autofluorescencia.
NOTA: Este paso crítico tiene en cuenta la sensibilidad del canal y la abundancia de antígenos. No se recomienda el uso del canal AF488 para tejido FFPE debido a su mayor autofluorescencia. - Para la conjugación anticuerpo-código de barras (4,5 h), obtenga el kit de conjugación de anticuerpos. Almacene todos los reactivos a 4 °C, excepto la solución de reducción 1, que debe almacenarse a -20 °C en forma de pequeños viales de un solo uso.
- Mida la concentración de anticuerpos mediante el uso de un espectrofotómetro.
- Inicie la conjugación de anticuerpos aplicando 500 μL de la solución de bloqueo del filtro a una columna de filtro MWCO de 50 kDa para bloquear la unión inespecífica de los anticuerpos al filtro. Girar a 12.000 x g durante 2 min a temperatura ambiente (RT). Elimine el exceso de líquido de la columna con una micropipeta de 200 μL.
NOTA: La solución reductora 1 es un vial de solución de un solo uso, que es suficiente para tres conjugaciones de anticuerpos a la vez. No reutilice la solución después de descongelarla; Deseche cualquier reactivo restante. No utilice un filtro sobre la columna de filtro MWCO de 50 kDa, ya que esto puede dar lugar a una purificación y conjugación deficientes. - Utilice un volumen equivalente de 50 μg de la solución de anticuerpos y añádalo a un filtro MWCO de 50 kDa. Ajuste el volumen total a 100 μL usando PBS si es necesario. Centrifugar a 12.000 x g durante 8 min a 4 °C. Durante este tiempo, prepare la mezcla maestra de reducción utilizando 19,8 μL de solución de reducción 1 + 825 μL de solución de reducción 2 para tres conjugaciones de anticuerpos.
- Deseche el flujo continuo y agregue 260 μL de mezcla maestra de reducción en la parte superior de cada unidad de filtro. Solución de vórtice en la unidad de filtro durante 2-3 segundos e incubar durante 30 min en RT.
NOTA: Es fundamental no exceder los 30 minutos de incubación para evitar la sobrerreducción de anticuerpos, ya que esto los daña y puede resultar en una conjugación fallida. - Después de 30 minutos de incubación, centrifugar a 12.000 x g durante 8 minutos a 4 °C y desechar el flujo por el fondo. Añadir 450 μL de solución de conjugación en la parte superior de la columna y centrifugar a 12.000 x g durante 8 min a 4 °C. Durante la centrifugación, prepare la solución de código de barras del CODEX. Muévase rápidamente después de recuperar el código de barras de -20 °C, ya que el código de barras comienza a degradarse.
NOTA: Los códigos de barras generalmente se suministran en pequeños viales de vidrio que contienen productos liofilizados (escamas o polvo) almacenados a -20 °C. Estos viales se pueden utilizar para conjugar 50 μg de anticuerpo a la vez. Se recomienda no conjugar más de tres anticuerpos a la vez. - Ubique cuidadosamente el código de barras liofilizado en el fondo del frasco de vidrio (también puede estar pegado a las paredes). Golpee el frasco de vidrio sobre una mesa para llevar los sólidos al fondo. La localización del producto liofilizado es esencial cuando se disuelve en 10 μL de agua de grado de biología molecular libre de nucleasas. Después de agregar 10 μL de agua sin nucleasas, agregue 210 μL de solución de conjugación a cada código de barras. Disuelva todo el material liofilizado y mezcle suavemente pipeteando hacia arriba y hacia abajo. Reservar.
- Después de completar el centrifugado en el paso 2.2.5, deseche el flujo y agregue la solución de código de barras respectiva del paso 2.2.6 a la parte superior de cada unidad de filtro. Guarde 1 μg del anticuerpo inicial no conjugado; esto se ejecutará junto con la muestra de anticuerpos conjugados de 5 μL durante la validación de la electroforesis en gel.
- Etiquete cada tubo con el anticuerpo respectivo y el nombre del código de barras. Cierre la tapa y mezcle la solución en vórtice durante 2-3 s. Incubar la reacción de conjugación anticuerpo-código de barras durante 2 h en RT.
- Después de 2 h de incubación, extraer 5 μL de anticuerpo conjugado y almacenar en un tubo de PCR de 0,2 mL para confirmar la conjugación por electroforesis en gel.
- Centrifugar la solución de anticuerpos conjugados restante a 12.000 x g durante 8 minutos a 4 °C. Añadir 450 μL de solución de purificación en la parte superior de la columna, centrifugar a 12.000 x g durante 8 min a 4 °C y desechar el flujo. Repita este paso de purificación 2 veces, agregando 450 μL de solución de purificación cada vez y girándola.
- Después de la3ª centrifugación, deseche el flujo a través. El filtro superior contendrá los anticuerpos conjugados con código de barras.
- Para recoger el anticuerpo conjugado, etiquete el nuevo tubo exterior que contiene la columna del filtro con el anticuerpo y el código de barras correspondientes. Corte la tapa del tubo exterior antes de la centrifugación. Agregue 100 μL de solución de almacenamiento de anticuerpos a cada unidad de filtro y coloque el tubo exterior recién etiquetado boca abajo en la parte superior de la columna de filtro.
- Invierta la columna del filtro para recoger el anticuerpo conjugado en el tubo exterior y gire hacia abajo a 3.000 x g durante 2 minutos en RT. Esto debería recoger unos 120 μL de solución de anticuerpos conjugados. Transferir a un tubo de microcentrífuga estéril y almacenar a 4 °C durante 18-24 meses.
NOTA: Para evitar una tinción nuclear de fondo alta después de la conjugación, se recomienda que estos anticuerpos no se utilicen durante al menos 2 días.
- Realizar la confirmación de la conjugación mediante electroforesis en gel como se describe en el protocolo13 ya publicado.
NOTA: Este proceso solo confirma el éxito de la reacción de conjugación química utilizada para conjugar los códigos de barras con anticuerpos. Es opcional, ya que la validación de anticuerpos solo es posible después de una tinción e imagen exitosas del tejido.
3. Preparación de muestras murinas de FFPE
- Obtención de muestras de tejido FFPE de ratones hembra C57BL/6J implantados con tumores de flanco B16F10. Tratar ratones con inyecciones intratumorales de nanopartículas basadas en poli(β-amino-éster) que contienen plásmidos 4-1BBL e IL-12, junto con la administración intraperitoneal de anti-PD1 a los 9, 11, 16 y 18 días después de la implantación14. Sacrifique ratones el día 20 y repare los tumores en formalina al 10%, incrustada en parafina, y luego corte en el núcleo de los Servicios de Tejidos Oncológicos de Johns Hopkins.
- Considere cuidadosamente las especificaciones del área de imagen proporcionadas en el manual del usuario y monte muestras de 5 μm en un portaobjetos dentro del área de imagen. Pudimos montar cuatro muestras tumorales diferentes en un solo portaobjetos.
NOTA: Es esencial utilizar los portaobjetos recomendados disponibles para la obtención de imágenes, como los portaobjetos adhesivos Leica Apex o los portaobjetos Superfrost Plus de Fisherbrand. Esto ayudará a que la celda de flujo se adhiera firmemente a la corredera.
4. Tinción e imagen de tejidos FFPE
- Verificación y optimización de la concentración de anticuerpos y los tiempos de exposición: Para verificar el éxito de la conjugación anticuerpo-código de barras, tiñe el tejido de interés con 6-9 anticuerpos conjugados. Ejecute el dispositivo de imágenes multiplex y anote la concentración de cada anticuerpo y el tiempo de exposición. Ajuste las diluciones y los tiempos de exposición según sea necesario, luego ejecute de 6 a 9 anticuerpos nuevos con los verificados previamente y anote los ajustes. Siga los pasos a continuación para la tinción y la obtención de imágenes.
- Tinción de tejidos e imágenes
- El día antes de teñir, hornee los portaobjetos de papel FFPE en un horno a 60 °C durante la noche. Al día siguiente, enfríe los portaobjetos durante 10 minutos en RT antes de comenzar los pasos de desparafinación y rehidratación de tejidos.
- Inicie la desparafinación del tejido incubando en una solución de xileno 2 veces durante 5 minutos cada una.
- Para rehidratar el tejido, muévalo a través de dos rondas de solución de etanol al 100% durante 5 minutos cada una. A continuación, muévalo a través de una serie de soluciones de alcohol de 90% a 70%, 50% y 30% durante 5 minutos cada una. Finalmente, lave el pañuelo con agua destilada dos veces durante 5 minutos cada una.
NOTA: Se pueden utilizar alternativas menos tóxicas al xileno, como una solución Neoclear, para los pasos de desparafinación. Realizar la desparafinación y rehidratación bajo una campana extractora. - Diluya 10x tampón AR9 a 1x usando agua destilada. Llene un frasco de Coplin con 50 ml de 1x tampón AR9, asegurándose de que los portaobjetos estén completamente sumergidos en el tampón de recuperación de antígenos.
- Llene una olla a presión con agua hasta que el nivel esté a la mitad del frasco de Coplin, luego incube a alta presión durante 20 minutos.
NOTA: Es posible utilizar una olla a presión especializada o una cámara de desobstrucción para una mejor recuperación del antígeno ajustándola a 120 °C durante 10 min. - Después de completar el paso de la olla a presión, enfríe los portaobjetos durante 45-60 minutos. Enjuague el portaobjetos con agua destilada 2 veces durante 2 minutos cada una. Luego, transfiera la diapositiva al frasco que contiene 1x PBS.
- Para minimizar la autofluorescencia, realice un paso de blanqueo en el tejido antes de teñir con anticuerpos. Prepare una solución blanqueadora (25 mL de 1x PBS + 0,8 mL de 1M de NaOH + 4,5 mL deH2O2) utilizando los reactivos y concentraciones recomendados. Incubar los portaobjetos en un recipiente de plástico que contenga la solución blanqueadora entre dos lámparas LED durante 45 minutos a temperatura ambiente. Repita con una solución blanqueadora fresca durante otros 45 minutos.
- Comience a preparar la solución de cóctel de anticuerpos durante este paso de blanqueo, especialmente si hay varios anticuerpos en el panel con varias diluciones. Recupere los cuatro bloqueadores (G, S, J y N) y colóquelos en hielo, ya que algunos tardan en descongelarse a partir de -20 °C.
- Después del fotoblanqueo, lave el pañuelo con 1x PBS, 2x durante 2 minutos cada uno. Mueva el pañuelo a frascos que contengan tampón de hidratación y lave 2 veces durante 2 minutos cada uno.
- Transfiera los portaobjetos de tejido al tampón de tinción y deje que se equilibren durante 30 minutos. Si no se prepara durante el paso de blanqueo, prepare la solución de cóctel de anticuerpos durante este tiempo.
- Prepare 300 μL de solución de cóctel de anticuerpos para teñir dos portaobjetos (por lo general, si se usa una cámara de plástico de hibridación, 100-120 μL de cóctel de anticuerpos es suficiente para teñir cada portaobjetos).
- Tiñir los portaobjetos aplicando una solución de cóctel de anticuerpos dentro de la cámara de humedad durante la noche a 4 °C.
NOTA: Todos los anticuerpos aquí muestran una señal positiva cuando se tiñen durante la noche a 4 °C. Sin embargo, el tiempo y la temperatura de tinción de cada anticuerpo pueden variar y deben optimizarse. Las condiciones de tinción más utilizadas son 3 h a RT o toda la noche a 4 °C. Si es necesario, también es posible teñir algunos anticuerpos inicialmente a diferentes temperaturas y luego los anticuerpos restantes durante la noche a 4 °C. Lave el pañuelo después con PBS si es necesario tinción secuencial. - Al día siguiente, realice la fijación posterior a la tinción. Retire la cámara de plástico y guarde el cóctel de anticuerpos recogido a 4 °C. Lave los portaobjetos 2 veces en el tampón de tinción durante 2 minutos cada uno. A continuación, pase los portaobjetos a una solución de fijación posterior a la tinción de 40 mL (4 mL de paraformaldehído al 16% + 36 mL de tampón de almacenamiento) e incube durante 10 min. Enjuague con PBS 3 veces durante 2 minutos cada uno.
- Mueva los portaobjetos a metanol helado durante 5 minutos, luego enjuague con PBS 3x durante 2 minutos cada uno. Aplique una solución fijadora final (20 μL de un tubo + 1 mL de PBS) a los portaobjetos bajo una cámara de humedad. Por lo general, 200 μL de la solución fijadora son suficientes por portaobjetos. Deje que el fijador se asiente durante 20 minutos en RT. Realice tres lavados finales en PBS durante 2 minutos cada uno.
NOTA: No retire previamente el tubo fijador a -20 °C; Solo descongele antes de aplicar. - Si toma imágenes de inmediato, limpie el portaobjetos alrededor del tejido con un pañuelo sin pelusa y aplique la celda de flujo presionándola durante 30 segundos con el equipo de ensamblaje de la celda de flujo. Si toma imágenes más tarde, guarde el portaobjetos (sin aplicar la celda de flujo) en el tampón de almacenamiento a 4 °C. Cuando esté listo para tomar imágenes, transfiera las diapositivas del búfer de almacenamiento a PBS durante 10 minutos antes de aplicar la celda de flujo.
- Comience a preparar el búfer de ejecución 1x con aditivo en función de los ciclos deseados en el experimento. Prepare tampones DMSO bajos (1:4) y altos (9:1) mezclándolos como DMSO de 1 parte con tampón de ejecución de 4 partes y DMSO de 9 partes con tampón de ejecución de 1 parte requerido para los ciclos deseados. Ahorre alrededor de 20 ml de tampón 1x con aditivo para hacer una placa reportera. Para calcular el tampón 1x requerido con tampones aditivos y DMSO alto/bajo, consulte al administrador de instrumentos.
- Comience a preparar la placa reportera. En primer lugar, haga que la solución de stock informante sea necesaria para el número total de ciclos. Etiquete los tubos de microcentrífuga de 1 ml de color negro o ámbar con los nombres de los ciclos. Añadir 250 μL de solución madre reportera y 5 μL de cada tubo reportero al tubo correspondiente. Luego, transfiera la solución a una placa negra de 96 pocillos y séllela con papel de aluminio.
- Encienda el dispositivo, use el administrador de instrumentos para configurar los parámetros y los tiempos de exposición (según la Tabla 1) y adquiera las imágenes. En la figura 1 se resumen los procesos previos y posteriores a la tinción.
5. Anotación de tejidos y segmentación celular
- Configurar un proyecto y un espacio de trabajo.
- Instale la última versión del software de análisis de patología digital (por ejemplo, QuPath versión 0.5.1 o superior). Haga clic en Crear proyecto para elegir una carpeta de destino para el espacio del proyecto.
- Haga clic en Agregar imágenes > Elegir archivos, luego navegue hasta el archivo QPTIFF (producido a partir de imágenes de inmunofluorescencia multiplex). Establezca el tipo de imagen en Fluorescencia, mantenga todos los demás ajustes predeterminados y haga clic en Importar.
- Si utiliza QuPath, haga doble clic en la nueva imagen para abrir un espacio de trabajo. Haga clic en Archivo > Guardar periódicamente para realizar un seguimiento de los cambios realizados en el proyecto. Alterna la visibilidad del marcador y la configuración de visualización con el icono de brillo y contraste (media luna) cerca del centro de la barra de herramientas.
NOTA: Haga clic con el botón derecho en una imagen de la lista de imágenes para cambiarle el nombre. Esto será importante más adelante cuando se visualicen las clasificaciones de fenotipos.
- Anote la sección completa del tejido, el compartimento intratumoral y el compartimento estromal.
- Utilice la herramienta de pincel y/o varita para dibujar una anotación alrededor de toda la sección de tejido. Defina esta anotación como Full_Tissue. Excluya la piel suprayacente y/o cualquier región que no deba analizarse. Para crear una anotación negativa y/o reducir el límite de la anotación, mantenga presionada la tecla Alt mientras usa una de las herramientas de anotación.
- Duplique la anotación de tejido completo. Seleccione la Anotación en la pestaña Anotaciones, luego vaya a Objetos > Anotaciones... > Duplicar las anotaciones seleccionadas.
- Encienda el canal SOX10. En la anotación duplicada, reduzca el límite de la anotación para capturar el compartimento intratumoral con la herramienta Alt + pincel/varita. Defina esta anotación como un tumor (Figura 2).
- Al seleccionar la anotación de tejido completo, vaya a Objetos > anotaciones... > Expandir anotaciones. Defina el radio de expansión como 1 μm y haga clic en Ejecutar.
- Cambie el nombre de la nueva anotación a Full_Tissue_Expansion. Seleccione la anotación del tumor, haga clic con el botón derecho y seleccione Insertar en jerarquía.
- Vuelva a seleccionar la anotación del tumor, vaya a Objetos > anotaciones... > Haz lo inverso. Defina esta nueva anotación como Estroma. Repita para todas las secciones de tejido.
- Ejecute la segmentación de celdas y exporte los resultados.
- Vaya a la pestaña Imagen para encontrar el ancho y el alto de píxeles de la imagen (Figura 3A).
- Descargue la extensión StarDist en https://github.com/qupath/qupath-extension-stardist/releases. Arrastre el archivo qupath-extension-stardist-[version].jar a la ventana de QuPath, luego haga clic en el icono de ajustes en la esquina superior derecha de la ventana de QuPath. En Extensiones, defina el directorio de usuario de QuPath como la ubicación de la carpeta StarDist.
- Descargue los archivos de script de StarDist groovy y el archivo de modelo de StarDist de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (los scripts de StarDist se organizaron originalmente desde Akoya Biosciences).
- Para cada sección de tejido, seleccione las anotaciones Tumor y Estroma (use Ctrl para seleccionar varias anotaciones). En la barra de configuración superior, haga clic en Automatizar > editor de scripts para abrir la interfaz de scripts. Abra el script de segmentación de celdas StarDist apropiado (archivo groovy) que corresponda con el tamaño de píxel de la imagen. Cuando se le pida que seleccione el archivo de segmentación de celda, abra el archivo stardist_cell_seg_model.pb.
- Después de la segmentación de celdas, guarde la imagen. Ve a Medir > Exportar medidas y, a continuación, selecciona la (s) imagen(es) correspondiente(s). Asigne el Tipo de exportación como Celdas y el Separador como .csv y, a continuación, elija la ubicación del archivo de salida . Haga clic en Rellenar junto a Columnas para incluir métricas de interés. Las columnas importantes que se deben exportar son Imagen, Id. de objeto, Padre, Centroide X, Centroide Y y Área del núcleo.
NOTA: El ID de objeto se utiliza en los pasos posteriores para reasignar las clasificaciones a la imagen de inmunofluorescencia multiplexada en el software de análisis de patología digital. La columna Principal indica el nombre de la anotación a la que pertenece la celda. Los datos X e Y se utilizan para el análisis espacial. - Para cada marcador de linaje que se utilizará en la agrupación en clústeres/fenotipado, seleccione un valor medio para exportar. En el caso de los marcadores nucleares (por ejemplo, SOX10 y FOXP3), exporte la opción Nucleus: Mean. En el caso de los marcadores citoplasmáticos/membranosos (p. ej., CD45, CD3, CD4), exporte la opción Citoplasma: Media.
6. Preprocesamiento y normalización de datos proteómicos
- Abra el archivo CSV exportado. Para cada marcador que se vaya a utilizar en la agrupación en clústeres o el fenotipado, trunque el nombre del encabezado de la columna para incluir solo el marcador. Por ejemplo, cambie el nombre de SOX10: Núcleo: Media a SOX10 y CD3: Citoplasma: Media a CD3.
- Instale la versión más reciente de R y RStudio. Descargue el archivo Marker Normalization.R de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis y ejecute el script. En primer lugar, se trata de filtrar las células por tamaño nuclear.
- Para cada marcador, se realiza una normalización mínima-máxima de modo que el valor de MFI más bajo en el rango de expresión se establezca en 0 y el valor de MFI enel percentil 99,7 se establezca en 1. Todas las intensidades por encimadel percentil 99,7 se recortan en 1. Una vez completado, guarde el nuevo archivo CSV generado con los datos normalizados.
7. Agrupamiento y fenotipado
- Elija un algoritmo para agrupar células de acuerdo con los perfiles de expresión de marcadores. En https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis, se proporcionan scripts de R para la agrupación en clústeres utilizando Seurat v4.415 y FlowSOM v2.13.9 (herramienta de agrupación y visualización)16.
- Fenotipo de las células según los perfiles de expresión de los marcadores de linaje clave. El protocolo presentado aquí utilizó 14 marcadores de linaje para la agrupación y el fenotipado: SOX10, CD45, CD3, CD4, CD8, FOXP3, CD11C, F4/80, CD68, CD86, CD163, CD206, NK1.1 y CD31.
- Defina las células tumorales como SOX10hi. Defina las células T CD4 como CD45mod-hi/CD4hi/CD8low/FOXP3low. Defina las células T CD8 como CD45mod-hi/CD8hi/CD4low. Defina las Tregs como CD45mod-hi/CD4hi/CD8low/FOXP3hi. Defina otros linfocitos T como CD45mod-hi/CD3hi/CD4low/CD8low.
- Defina los macrófagos M1 como CD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163low /CD206low. Defina los macrófagos M2 como CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206low, CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206hi, o CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206hi. Defina otros macrófagos como CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206low o CD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hi.
- Defina las células dendríticas como CD45mod-hi/CD11chi/CD86mod-hi. Defina las celdas NK como CD45mod-hi/NK1.1hi. Defina otras células inmunitarias como CD45mod-hi que no cumplieron las condiciones detalladas anteriormente. Defina las células endoteliales como CD31mod-hi. Elimine todos los demás perfiles de expresión.
8. Reasignación de clasificaciones de fenotipos y realización de controles de calidad
- Descargue el script Remapping Classifications.groovy de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (este script fue adaptado de una publicación en https://forum.image.sc/ por petebankhead).
- Abra el archivo CSV fenotipado y asegúrese de que todas las etiquetas de la pestaña Imagen coincidan exactamente con el nombre del archivo de imagen en el software de análisis de patología digital que se utilizará para la reasignación. Para cambiar rápidamente grandes cantidades de etiquetas en el archivo CSV, utilice la función Reemplazar todo.
- Si utiliza QuPath, ejecute el archivo groovy de reasignación de clasificaciones haciendo clic en Automatizar > editor de scripts.
- En la pestaña Anotaciones, haga clic en los tres puntos junto al botón Establecer automáticamente y, a continuación, haga clic en Rellenar a partir de objetos existentes > Todas las clases (incluidas las subclases) > Sí.
- Desplácese hacia abajo en la lista de marcadores para ver las diferentes clasificaciones de fenotipos y ajustar el esquema de color según sea necesario. Haga doble clic en cualquier celda para ver su fenotipo (junto a la etiqueta Clasificación) y su clúster (junto a la etiqueta Nombre). Verifique los campos de visión representativos para determinar la precisión de las asignaciones de fenotipos y revise la estrategia de fenotipado según sea necesario (Figura 3B).
9. Cuantificación de la densidad y análisis espacial
- Una vez finalizado el fenotipado, obtenga las áreas de anotación en el software de análisis de patología digital en la pestaña Anotaciones. Convierta los valores de área amm 2 y, a continuación, divida los recuentos por áreas de anotación para calcular las densidades.
- Descargue el script SPIAT Spatial Analysis.R de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis y ejecute. Recopile datos sobre distancias mínimas medias (AMD), composiciones de vecindad y puntuaciones de mezcla normalizadas (NMS).
- Generar cifras en software de análisis estadístico para presentar hallazgos. Al generar un único mapa de calor para distancias mínimas medias de las condiciones de tratamiento, promedie los resultados obtenidos de diferentes secciones de tejido.
Resultados
Aquí, presentamos un protocolo para diseñar un panel de anticuerpos para tejido FFPE murino, realizar imágenes de inmunofluorescencia multiplex y analizar imágenes para la cuantificación proteómica y las relaciones espaciales. El panel validado contiene 27 anticuerpos que proporcionan marcadores para visualizar células de melanoma (SOX10), leucocitos (CD45), células T (CD3, CD4, CD8, FOXP3), células B (CD20), macrófagos y subtipos (F4/80, CD68, CD86, CD163, CD206), células dendríticas (CD11c), células NK (NK1.1) y células endoteliales (CD31). El panel completo también contiene marcadores para otras poblaciones inmunes (CD11b, CD38), actividad de proliferación (Ki67), funcionalidad de las células T (T-bet, Eomesodermin, granzima B), presentación de antígenos (LMP2, microglobulina beta-2, MHC II) y expresión de puntos de control (TIM3, LAG3, PD-L1)12. Una imagen representativa de electroforesis en gel confirma la conjugación exitosa de los códigos de barras de oligonucleótidos de ADN con los anticuerpos libres de portadores, como se muestra en la Figura 4. Este paso solo confirma la reacción química, y la confirmación de la imagen solo se puede realizar después de verificar estos anticuerpos con el dispositivo de imagen multiplex en el tejido de interés. Consulte la siguiente referencia para ver imágenes validadas de los 27 marcadores en el panel13. En la Figura 5 se presenta una imagen de fusión de marcadores de linaje clave que tiñen tres secciones de tejido de melanoma murino tratadas con inyecciones intratumorales de nanopartículas 4-1BBL/IL-12 y anti-PD1 sistémico. Estas secciones se utilizaron para el análisis posterior de imágenes en este protocolo.
Después de la anotación de tejidos, la segmentación celular y el preprocesamiento de datos proteómicos, presentamos una comparación entre dos algoritmos de agrupamiento que se han aplicado previamente para el análisis transcriptómico y/o proteómico de una sola célula17,18. Los perfiles de expresión para las poblaciones fenotipadas se presentan en la Figura 6 para ambos enfoques. Demostramos que FlowSOM ofrece una gama más amplia de valores de intensidad (~0,7 frente a ~0,5) para discernir las diferencias entre poblaciones celulares similares, como los subtipos de macrófagos. Seurat aplicó el algoritmo de Lovaina durante la agrupación y generó 29 agrupaciones (Figura complementaria 1). En comparación, FlowSOM aplicó mapas autoorganizados y puede generar 100 clústeres (Figura complementaria 2). Un mayor número de clústeres se traduce en más tiempo necesario para el fenotipado, pero este último enfoque de FlowSOM también ofrece más matices a la hora de clasificar poblaciones de células similares. Cualitativamente, vemos que FlowSOM fue capaz de clasificar más macrófagos intratumorales como un subtipo M1 o M2 en comparación con el fenotipado de Seurat en el mismo campo de visión (Figura 7). El mismo resultado se observa cuando cuantificamos las densidades de macrófagos, con FlowSOM capturando una mayor densidad de macrófagos M1 y M2 en comparación con Seurat (Figura 8A-B) y, posteriormente, una densidad significativamente menor de otros macrófagos/no clasificados (p = 0,0028; Figura 8C). Sin embargo, los dos enfoques de análisis también generaron resultados similares al describir otras densidades de población celular, como las células T CD4, las células T CD8 y las células endoteliales (Figura 8D-F).
También presentamos los resultados del análisis espacial posterior después de la agrupación y el fenotipado. En la figura 9 se muestran algunas de las métricas espaciales que se pueden generar con este protocolo. En relación con todas las poblaciones fenotipadas, los macrófagos M2 y las células NK presentaron los niveles más altos de DMAE tras el tratamiento con nanopartículas 4-1BBL/IL-12 y anti-PD1 (Figura 9A). De manera similar, el SNM entre los linfocitos T CD8 intratumorales y los macrófagos M1 fue mucho mayor que entre los linfocitos T CD8 y los macrófagos M2 (Figura 9B). Además, los macrófagos M2 contribuyeron ~1% en el vecindario de 100 μm que rodea a las células T CD8 intratumorales, mientras que los macrófagos M1 constituyeron el 9%-13% de estos vecindarios (Figura 9C). En conjunto, estos resultados sugieren que el régimen de tratamiento 4-1BBL/IL-12 polarizó los macrófagos asociados al tumor hacia un subtipo M1 y excluyó a los macrófagos M2 del microambiente inmunitario tumoral.
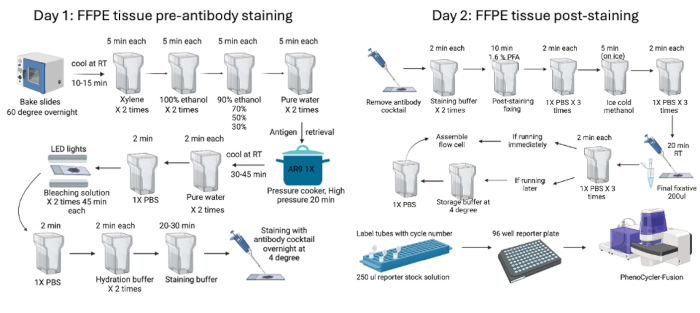
Figura 1: Resumen del flujo de trabajo de tinción e imagen de tejidos FFPE. Los tejidos murinos de FFPE se procesaron utilizando procedimientos previos a la tinción que comenzaron con la cocción del tejido durante la noche antes de comenzar los procesos de pretinción (día 1) y posttinción (día 2) de dos días. Finalmente, se preparó la placa reportera antes de la obtención de imágenes con el dispositivo. Haga clic aquí para ver una versión más grande de esta figura.
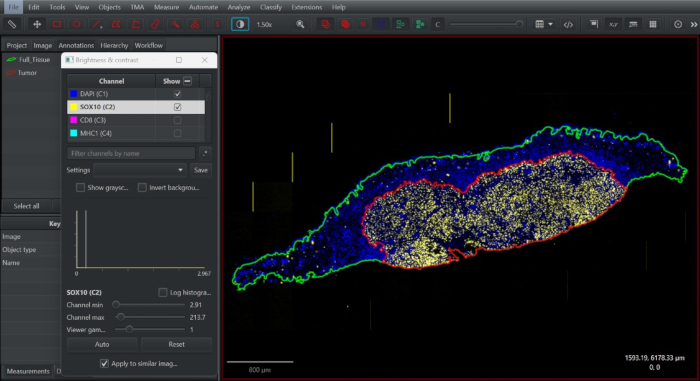
Figura 2: Captura de pantalla de la anotación de tejido en el software de patología digital de acceso abierto QuPath. Se dibujó una anotación de tejido completo (verde), se duplicó y se minimizó para definir el compartimento intratumoral de acuerdo con la distribución de los melanocitos SOX10+ en el límite tumoral (rojo). Haga clic aquí para ver una versión más grande de esta figura.
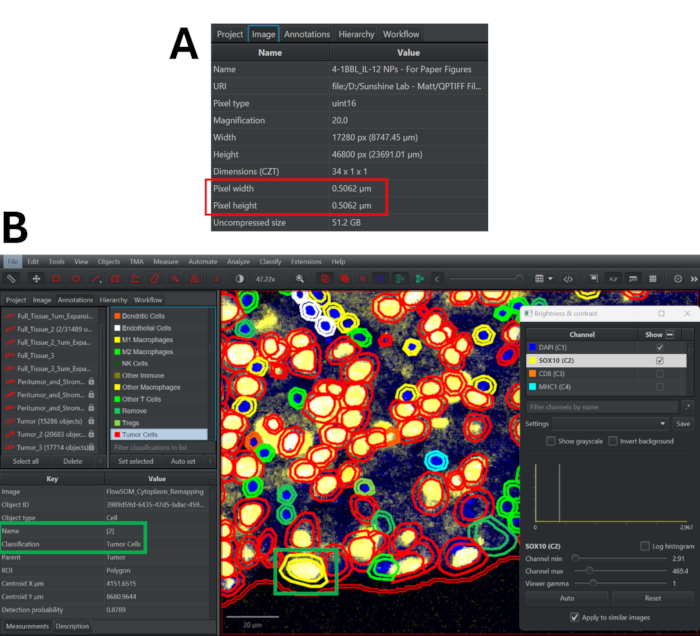
Figura 3: Segmentación celular utilizando el algoritmo StarDist en QuPath y proceso de control de calidad para revisar las clasificaciones de fenotipos. (A) Navegue hasta la pestaña Imagen para determinar el ancho y la altura de píxeles de la imagen de inmunofluorescencia multiplexada. (B) Después de reasignar las clasificaciones, haga doble clic en cualquier celda (se resalta en amarillo) para ver su asignación de clúster y fenotipo. Activa o desactiva los marcadores del panel para determinar si este enfoque de clasificación es preciso. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Imagen representativa en gel de la confirmación de la conjugación del código de barras anticuerpo-ADN. La electroforesis en gel de proteínas confirma la conjugación de anticuerpos con códigos de barras de oligonucleótidos de ADN, observados por bandas adicionales en el sitio de la cadena pesada. Haga clic aquí para ver una versión más grande de esta figura.
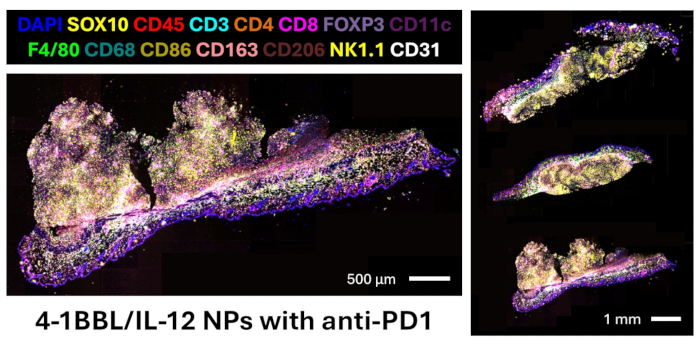
Figura 5: Imágenes multiplex basadas en códigos de barras de ADN de tumores de flanco B16F10 tratados con inyecciones intratumorales de nanopartículas 4-1BBL/IL-12 con anti-PD1 sistémico. No se muestran los marcadores en nuestro panel: CD11b, CD20, CD38, TIM3, LAG3, T-bet, Eomesodermin, granzima B, Ki67, LMP2, microglobulina beta-2, MHC II, PD-L1. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Mapas de calor de expresión proteómica de poblaciones celulares fenotipadas. (A) El enfoque de agrupamiento y fenotipado de Seurat genera un rango ligeramente menor de valores de expresión de marcadores en comparación con (B) el agrupamiento y fenotipado de FlowSOM. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: El fenotipado de FlowSOM identifica una mayor gama de diferentes subtipos de macrófagos. El panel superior muestra el fenotipado de macrófagos Seurat (izquierda) y FlowSOM (derecha) para el mismo campo de visión. El panel inferior muestra imágenes de inmunofluorescencia multiplex de marcadores clave del linaje de macrófagos en el mismo campo de visión. Todas las barras de escala son de 20 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 8: Comparación de las densidades intratumorales de diferentes poblaciones celulares después de la agrupación/fenotipado. Se muestran comparaciones de densidad para macrófagos intratumorales (A) M1, (B) macrófagos M2, (C) otros macrófagos, (D) linfocitos T CD4, (E) linfocitos T CD8 y (F) linfocitos endoteliales. Las barras de error son errores estándar de la media (SEM), y la significación se probó mediante pruebas t no apareadas (**p ≤ 0,01). Haga clic aquí para ver una versión más grande de esta figura.

Figura 9: Perfil de las métricas espaciales intratumorales para tres tumores de flanco tratados con inyecciones de nanopartículas intratumorales 4-1BBL/IL-12 y anti-PD1 sistémico. (A) Mapa de calor de las distancias mínimas medias entre poblaciones fenotipadas intratumorales. Las medidas se expresan en μm. (B) Puntuaciones de mezcla normalizadas entre pares de fenotipos intratumorales clave. (C) Desglose de las composiciones de vecindad en un radio de 100 μm alrededor de poblaciones de células T CD8 intratumorales. Haga clic aquí para ver una versión más grande de esta figura.
| Anticuerpo | Dilución | Tiempo de exposición (ms) |
| SOX-10 | 50 | 450 |
| CD8 | 100 | 450 |
| CD3 | 100 | 450 |
| FoxP3 | 50 | 350 |
| CD4 | 50 | 450 |
| MHC-II | 100 | 450 |
| PDL1 | 50 | 450 |
| CD45 | 100 | 450 |
| Ki-67 | 100 | 300 |
| F4/80 | 100 | 150 |
| CD20 | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| CD206 | 100 | 450 |
| CD68 | 100 | 450 |
| Granzima-B | 50 | 450 |
| CD86 | 100 | 150 |
| CD31 | 100 | 450 |
| CD11C | 50 | 450 |
| CD11b | 50 | 450 |
| EOMES | 50 | 450 |
| TIM-3 | 50 | 300 |
| CD38 | 50 | 200 |
| LAG3 | 50 | 450 |
| CD163 | 50 | 200 |
| Apuesta T | 50 | 300 |
| LMP2 | 100 | 100 |
| Beta2 MG | 200 | 50 |
Tabla 1: Dilución de anticuerpos y ajustes de tiempo de exposición.
Figura complementaria 1: Mapa de calor de expresión proteómica inicial generado después de la agrupación de Seurat. Haga clic aquí para descargar esta figura.
Figura complementaria 2: Mapa de calor de expresión proteómica inicial generado después de la agrupación en clústeres de FlowSOM. Haga clic aquí para descargar esta figura.
Discusión
El éxito de las imágenes depende de un panel de anticuerpos bien diseñado y validado. Las imágenes de inmunofluorescencia múltiple de muestras de FFPE presentan desafíos debido a la alta autofluorescencia y la dificultad de recuperar epítopos enmascarados por la inclusión en parafina. Sin embargo, dado que la FFPE ofrece varias ventajas en comparación con las muestras de FF, es esencial diseñar y validar los paneles de anticuerpos FFPE. El primer paso es finalizar los clones de anticuerpos que muestran señales positivas durante las imágenes de inmunofluorescencia (IF); posteriormente, es importante conjugarlos cuidadosamente con códigos de barras de ADN. La conjugación de anticuerpos requiere una reducción parcial del anticuerpo para crear enlaces SH, que se utilizan durante la reacción del grupo de maleimida con un código de barras. No todos los clones de anticuerpos pueden resistir este paso, y algunas reacciones pueden causar daños irreversibles al anticuerpo, lo que resulta en un fracaso de las imágenes a pesar de la conjugación exitosa. Por esta razón, a pesar de que algunos anticuerpos pueden mostrar señales positivas durante la validación convencional del FI, para evaluar el éxito final de la conjugación de anticuerpos, es importante validar cada anticuerpo en el tejido real de interés y registrar los tiempos de exposición deseados para ese tejido. En aplicaciones futuras, esta técnica se puede combinar con el análisis de transcriptómica espacial existente en diapositivas consecutivas o en la misma diapositiva para generar información adicional. Una de las limitaciones de este método es que requiere una cuidadosa selección y validación de cada anticuerpo en el panel en función del objetivo y el tipo de tejido.
Con respecto al análisis de imágenes, QuPath ofrece una valiosa herramienta de acceso abierto con visualización de alta calidad de marcadores proteómicos, una amplia funcionalidad para exportar mediciones de intensidad y realizar controles de calidad para clasificaciones de fenotipos, y una buena flexibilidad para scripts generados por el usuario. Los foros en línea, como https://forum.image.sc/, son un recurso adicional para analizar cómo realizar tareas de análisis específicas y para compartir scripts con otros usuarios. En este protocolo, comparamos dos enfoques de agrupamiento y fenotipado utilizando Seurat y FlowSOM. Si bien se puede preferir FlowSOM por su capacidad para generar información más granular sobre las subpoblaciones de células inmunitarias del TME, también se debe tener en cuenta el tiempo requerido para el análisis proteómico. La generación de 100 clústeres puede ser innecesaria si un usuario solo necesita fenotipar células dentro de una o dos muestras de tejido. En estas situaciones, Seurat puede ofrecer un canal más rápido y eficiente para el análisis de imágenes. Por el contrario, es más probable que el análisis de una TMA con más de 40 o 50 secciones de tejido produzca un mayor número de grupos de células en ambos enfoques de análisis, y FlowSOM puede ser la metodología preferida para generar clasificaciones de fenotipos más matizadas.
El agrupamiento/fenotipado de células y todos los pasos posteriores de análisis de imágenes dependen en gran medida de la segmentación de células. Nuestro trabajo actual ha explorado la segmentación celular en los algoritmos HALO (Indica Labs) y StarDist, y hemos encontrado que ambos enfoques tienden a sobresegmentar las células en función de las señales nucleares DAPI. También hay disponibles muchos algoritmos de segmentación alternativos, como Mesmer19 e InstanSeg20. Este es un campo creciente de investigación computacional que requiere más exploración y optimización.
Divulgaciones
J.C.S. agradece el apoyo financiero de Emerson Collective LLC y los Institutos Nacionales de Salud. J.J.G. también agradece la financiación de los Institutos Nacionales de Salud. J.C.S. tiene una relación con Palleon Pharmaceuticals Inc., que implica subvenciones de financiación. S.Y.T. y J.J.G. tienen una relación con OncoSwitch Therapeutics que involucra acciones o acciones. S.Y.T., J.J.G., J.C.S. y K.M.L. tienen una patente pendiente. Todos los demás autores afirman que no tienen intereses financieros o relaciones personales contrapuestos conocidos que puedan percibirse como influyentes en la investigación presentada en este artículo.
Agradecimientos
J.C.S. reconoce a la Fundación de Dermatología y al Premio al Desarrollo Profesional en Dermatopatología por avanzar en la carrera del autor. Los autores agradecen a Hsin-Pei Lee, del Laboratorio de Ciencia de Datos del Cáncer del Instituto Nacional del Cáncer, por su ayuda con las técnicas de análisis computacional. Esta investigación recibió fondos del Colectivo Emerson y de los Institutos Nacionales de Salud (R37CA246699, P41EB028239 y R01CA228133). Además, el núcleo de Servicios de Tejidos Oncológicos (OTS) de Johns Hopkins cuenta con el apoyo de los Institutos Nacionales de Salud (P30CA006973).
Materiales
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
Referencias
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados