Method Article
基于 DNA 条形码的多重免疫荧光成像分析来自基因重编程小鼠黑色素瘤的 FFPE 标本
* 这些作者具有相同的贡献
摘要
该方案为设计用于小鼠 FFPE 黑色素瘤组织基于 DNA 条形码的成像的多重免疫荧光抗体面板提供了分步指南。我们还描述了一个使用开源工具的图像分析管道,用于生成对小鼠黑色素瘤肿瘤免疫微环境的空间蛋白质组学见解。
摘要
这里介绍的是一种新兴的基于 DNA 条形码的多重成像技术,基于 IndEXing 共检测,可分析组织微环境的空间蛋白质组学。成功的成像需要一系列精心设计和适当验证的抗体组合,但目前很少有用于福尔马林固定石蜡包埋 (FFPE) 样品的抗体组合。与新鲜冷冻标本相比,FFPE 具有多项优势,例如广泛可用、易于处理和储存以及能够制备组织微阵列 (TMA)。在这里,我们提出了一种开发抗体面板的方案,用于可视化和分析来自用纳米颗粒处理的小鼠黑色素瘤模型的 FFPE 组织,这些纳米颗粒为肿瘤微环境重编程提供编码免疫信号的质粒 DNA。我们还描述了一个使用开源计算工具的图像分析管道,用于注释组织、分割细胞、处理蛋白质组学数据、表型细胞群和量化空间指标。该方案为在小鼠 FFPE 中设计抗体面板和为复杂组织微环境的空间蛋白质组学提供新的见解。
引言
皮肤黑色素瘤是最常见的皮肤癌,根据诊断时间和初级保健,全球的疾病和死亡率各不相同1。在过去十年中,对黑色素瘤的生物学认识的提高有助于推动治疗实体瘤的新癌症模型的开发2。最近免疫疗法的兴起导致了基于激活内源性免疫系统的革命性癌症治疗概念 3,4。
肿瘤微环境 (TME) 非常复杂,由多种免疫细胞、癌症相关成纤维细胞、周细胞、内皮细胞和各种组织驻留细胞组成5。过去已经应用了几种技术来研究 TME,例如流式细胞术和单细胞测序,这些技术会损害空间环境,因为它们需要破坏肿瘤组织。传统的显微镜成像,如免疫荧光 (IF) 和免疫组织化学 (IHC),可以在不破坏样品组织的情况下实现蛋白质生物标志物的可视化。然而,这些方法仅限于两个或三个生物标志物,无法全面了解复杂的 TME 6 中的空间和结构关系。
为了解决这个问题,已经开发了几种多重成像技术,可以在空间上可视化复杂的 TME 7,8,9,10。其中一种是 CoDetection-by-inDEXing,更名为 PhenoCycler 系统,基于 DNA 寡核苷酸偶联抗体11。该系统可提供人类标本 100 多种生物标志物的单细胞成像和分析。然而,很少有库存抗体可用于观察和分析小鼠标本,尤其是福尔马林固定石蜡包埋 (FFPE) 样品12。与新鲜冷冻 (FF) 保存相比,FFPE 具有多项优势,例如易于处理和储存、随着时间的推移保存完好的形态,最重要的是,能够制备组织/肿瘤微阵列 (TMA),从而允许在单个载玻片上可视化多个标本。我们最近设计并开发了小鼠 FFPE CODEX/PhenoCycler 抗体面板,并成功将其应用于可视化和分析遗传重编程小鼠黑色素瘤标本的空间蛋白质组学13。
该方案的总体目标是为设计小鼠 FFPE 抗体组合提供分步指南,并描述抗体-条形码偶联、组织染色和成像的过程。此外,我们还提供了一个利用开源工具(如 QuPath 和 R 包)的详细图像分析管道。遵循此协议后,研究人员将学习如何设计定制的偶联抗体组,使用 Phenocycler-Fusion 设备进行多重成像,并获得对黑色素瘤 TME 的空间蛋白质组学的新见解。此外,该方案可以适应研究各种肿瘤免疫微环境,并与现有的空间转录组学技术相结合。
研究方案
所有动物工作均按照约翰霍普金斯大学动物护理和使用委员会制定的指南进行,使用经批准的方案编号 MO18M388 和 MO21M384。
1. 抗体选择
- 根据感兴趣的组织和特定生物标志物的丰度设计抗体组合。根据文献中免疫荧光 (IF) 和免疫组织化学 (IHC) 染色的先前成功应用来选择抗体克隆。这些抗体的安全无载体蛋白版本。
注:不含载体的纯化抗体对于条形码偶联很重要。避免使用任何基于蛋白质的防腐剂,如 BSA、麸质、甘油等。如果无法获得无载体蛋白抗体,请向供应商索取定制抗体或使用 BSA 去除试剂盒制备无载体蛋白抗体。叠氮化钠不会干扰抗体偶联。建议使用 IgG 同种型而不是 IgM 同种型。 - 使用常规免疫荧光 (IF) 成像在目标组织上验证这些选定的克隆11。使用双 IF 成像来确认某些克隆的特异性(例如,使用 FOXP3 和 CD4 进行双 IF 成像以确认调节性 T 细胞的染色)。
注意:只要有可能,使用与染色相同的组织进行 IF 验证非常重要。选择合适的抗原修复溶液。在该协议中,我们使用 Akoya Biosciences 提供的 AR9 缓冲液来检索所有小鼠黑色素瘤 FFPE 组织表位。根据表位要求,可以使用其他抗原修复解决方案,例如 AR6 或 Universal。
2. 抗体偶联和确认
- 将经过验证的抗体分配给 ATTO550 (Cy3)、AF647 (Cy5) 或 AF750 (Cy7) 荧光基团上附有互补报告基因的条形码。低丰度抗原通常产生较低的信号;将此类抗体与低自发荧光通道(如 AF647 (Cy5))偶联。由于存在自发荧光,因此将高表达抗原与 ATTO550 (Cy3) 和 AF750 (Cy7) 偶联。
注意:这个关键步骤考虑了通道敏感性和抗原丰度。不建议将 AF488 通道用于 FFPE 组织,因为它的自发荧光较高。 - 对于抗体 - 条形码偶联(4.5 小时),获得抗体偶联试剂盒。将所有试剂储存在4°C,但还原溶液1除外,还原溶液1需要作为一次性小瓶储存在-20°C下。
- 使用分光光度计测量抗体浓度。
- 通过将 500 μL 过滤器封闭溶液涂抹到 50 kDa MWCO 过滤柱上来开始抗体偶联,以阻止抗体与过滤器的非特异性结合。在室温 (RT) 下以 12,000 x g 离心 2 分钟。使用 200 μL 微量移液器从色谱柱中去除多余的液体。
注:还原液 1 是一次性溶液小瓶,一次足以进行三种抗体偶联。解冻后不要重复使用溶液;丢弃所有剩余的试剂。请勿在 50 kDa MWCO 过滤柱上使用过滤器,因为这会导致纯化和偶联效果不佳。 - 使用 50 μg 等效体积的抗体溶液,将其添加到 50 kDa MWCO 过滤器中。如果需要,使用 PBS 将总体积调节至 100 μL。在 4 °C 下以 12,000 x g 旋转 8 分钟。 在此期间,使用 19.8 μL 还原液 1 + 825 μL 还原液 2 制备还原预混液,用于三种抗体偶联。
- 弃去流通液,在每个过滤器单元的顶部添加 260 μL 还原预混液。在过滤装置中涡旋溶液 2-3 秒,并在室温下孵育 30 分钟。
注:为避免抗体过度还原,孵育时间不要超过 30 分钟,因为这会损坏抗体并可能导致偶联失败。 - 孵育 30 分钟后,在 4 °C 下以 12,000 x g 离心 8 分钟,并丢弃底部流出的液体。将 450 μL 偶联溶液添加到色谱柱顶部,并在 4 °C 下以 12,000 x g 的速度离心 8 分钟。 离心过程中,制备 CODEX 条形码溶液。从 -20 °C 检索条形码后,当条形码开始降解时,请快速移动。
注:条形码通常装在小玻璃瓶中,其中包含储存在 -20 °C 的冻干产品(薄片或粉末)。 这些样品瓶可用于一次偶联 50 μg 抗体。建议一次不要偶联超过 3 种抗体。 - 小心地找到玻璃瓶底部的冻干条形码(它也可能粘在壁上)。轻敲桌子上的玻璃瓶,使固体到达底部。将冻干产品溶解在 10 μL 无核酸酶分子生物学级水中时,必须找到冻干产品。加入 10 μL 无核酸酶水后,向每个条形码中加入 210 μL 偶联溶液。溶解所有冻干材料,上下吹打轻轻混匀。留。
- 完成步骤 2.2.5 中的离心后,丢弃流出液,并将步骤 2.2.6 中的相应条形码解决方案添加到每个过滤器单元的顶部。保存 1 μg 初始未偶联抗体;在凝胶电泳验证期间,该样品将与偶联的 5 μL 抗体样品一起运行。
- 用相应的抗体和条形码名称标记每个试管。盖上盖子,涡旋 2-3 秒混合溶液。在 RT 下将抗体 - 条形码偶联反应孵育 2 小时。
- 孵育 2 小时后,取出 5 μL 偶联抗体并储存在 0.2 mL PCR 管中,以通过凝胶电泳确认偶联。
- 在 4°C 下以 12,000 x g 的速度离心剩余的偶联抗体溶液 8 分钟。 向柱顶部添加 450 μL 纯化溶液,在 4 °C 下以 12,000 x g 离心 8 分钟,然后弃去流出液。重复此纯化步骤 2 次,每次加入 450 μL 纯化溶液并离心。
- 第 3 次离心后,丢弃流出物。顶部过滤器将包含条形码偶联的抗体。
- 要收集偶联抗体,请用相应的抗体和条形码标记容纳过滤柱的新外管。离心前切掉外管的盖子。向每个过滤单元中加入 100 μL 抗体储存溶液,并将新标记的外管倒置在过滤柱顶部。
- 倒置过滤柱以将偶联抗体收集到外管中,并在 RT 下以 3,000 x g 的速度旋转 2 分钟。这应该收集大约 120 μL 的偶联抗体溶液。转移到无菌微量离心管中,并在 4 °C 下储存 18-24 个月。
注:为避免偶联后出现高背景核染色,建议这些抗体至少 2 天不要使用。
- 如已发布的方案13 中所述,通过凝胶电泳进行偶联确认。
注:此过程仅确认用于将条形码与抗体偶联的化学偶联反应成功。这是可选的,因为只有在成功对组织进行染色和成像后才能进行抗体验证。
3. 小鼠 FFPE 标本制备
- 从植入 B16F10 侧腹肿瘤的 C57BL/6J 雌性小鼠中获取 FFPE 组织样本。在植入后 9、11、16 和 18 天,瘤内注射含有 4-1BBL 和 IL-12 质粒的基于 β-氨基酯的基于聚(-氨基酯)的纳米颗粒,以及腹膜内递送抗 PD1治疗小鼠 14。在第 20 天处死小鼠,将肿瘤固定在 10% 福尔马林中,包埋在石蜡中,然后在约翰霍普金斯大学肿瘤学组织服务核心处切片。
- 仔细考虑用户手册中提供的成像区域规格,并将 5 μm 标本安装在成像区域内的载玻片上。我们能够在一张载玻片上安装四个不同的肿瘤标本。
注意:必须使用可用于成像的推荐载玻片,例如 Leica Apex 粘合载玻片或 Fisherbrand Superfrost Plus 载玻片。这将有助于流通池牢固地粘附在玻片上。
4. FFPE 组织染色和成像
- 抗体浓度和曝光时间的验证和优化:为了验证抗体-条形码偶联是否成功,用 6-9 种偶联抗体对目标组织进行染色。运行多重成像设备并记录每种抗体浓度和曝光时间。根据需要调整稀释度和曝光时间,然后使用先前验证的抗体运行 6-9 种新抗体,并记录任何调整。请按照以下步骤进行染色和成像。
- 组织染色和成像
- 染色前一天,将 FFPE 组织载玻片在 60 °C 烘箱中烘烤过夜。第二天,在开始脱蜡和组织再水化步骤之前,在 RT 下将载玻片冷却 10 分钟。
- 通过在二甲苯溶液中孵育 2 次,每次 5 分钟来开始组织脱蜡。
- 要使组织再水化,请将其移入两轮 100% 乙醇溶液中,每轮 5 分钟。接下来,将其从90%移至70%、50%和30%的一系列酒精溶液,每次5分钟。最后,用蒸馏水洗涤组织两次,每次 5 分钟。
注:二甲苯的毒性较小的替代品,例如 Neoclear 溶液,可用于脱蜡步骤。在通风橱下进行脱蜡和再水化。 - 使用蒸馏水将 10x AR9 缓冲液稀释至 1x。用 50 mL 的 1x AR9 缓冲液填充 Coplin 罐,确保载玻片完全浸入抗原修复缓冲液中。
- 在高压锅中装满水,使液位达到 Coplin 罐的一半,然后在高压设置下孵育 20 分钟。
注意:可以使用专门的压力锅或脱衣室,将其设置为 120 °C 10 分钟,以获得更好的抗原修复。 - 完成压力锅步骤后,将载玻片冷却 45-60 分钟。用蒸馏水冲洗载玻片 2 次,每次 2 分钟。然后,将载玻片转移到含有 1x PBS 的罐子中。
- 为了尽量减少自发荧光,请在用抗体染色之前对组织进行漂白步骤。使用推荐的试剂和浓度新鲜制备漂白溶液(25 mL 1x PBS + 0.8 mL 1M NaOH + 4.5 mL H2O2)。在室温下,将载玻片在含有漂白溶液的塑料容器中在两个 LED 灯之间孵育 45 分钟。用新鲜的漂白溶液重复再 45 分钟。
- 在此漂白步骤中开始制备抗体混合物溶液,特别是当检测组合中有多种不同稀释度的抗体时。取回所有四种阻滞剂(G、S、J 和 N)并将它们放在冰上,因为有些阻滞剂需要时间才能从 -20 °C 解冻。
- 光漂白后,用 1x PBS 洗涤组织,每次 2 次 2 分钟。将组织移至装有水合缓冲液的罐子中,洗涤 2 次,每次 2 分钟。
- 将组织载玻片转移到染色缓冲液中,让它们平衡 30 分钟。如果在漂白步骤中未制备,请在此时制备抗体混合物溶液。
- 准备 300 μL 抗体混合物溶液对两张玻片进行染色(通常,如果使用杂交塑料室,100-120 μL 抗体混合物足以对每张玻片进行染色。
- 通过在湿度室内在 4 °C 下将抗体混合物溶液涂抹过夜来对载玻片进行染色。
注:此处的所有抗体在 4 °C 下染色过夜时均显示阳性信号。 但是,每种抗体的染色时间和温度可能会有所不同,必须进行优化。最常用的染色条件是在室温下 3 小时或在 4 °C 下过夜。 如果需要,也可以先在不同温度下对一些抗体进行染色,然后在 4 °C 下对剩余抗体进行过夜染色。 如果需要连续染色,然后用 PBS 洗涤组织。 - 第二天,进行染色后固定。取下塑料室并将收集的抗体混合物储存在 4 °C 下。 在染色缓冲液中洗涤载玻片 2 次,每次 2 分钟。接下来,将载玻片移至 40 mL 染色后固定溶液(4 mL 16% 多聚甲醛 + 36 mL 储存缓冲液)中并孵育 10 分钟。用 PBS 冲洗 3 次,每次 2 分钟。
- 将玻片移入冰冷的甲醇中 5 分钟,然后用 PBS 冲洗 3 次,每次 2 分钟。将最终固定液(20 μL 单管 + 1 mL PBS)涂抹在湿度室下的载玻片上。通常,每张玻片 200 μL 固定液就足够了。让固定剂在 RT 下静置 20 分钟。在 PBS 中进行 3 次最后一次洗涤,每次 2 分钟。
注意:请勿事先将固定管从 -20 °C 中取出;仅在应用前解冻。 - 如果立即成像,请使用无绒组织擦拭组织周围的载玻片,并使用流通池组装设备按压流动池 30 秒以应用流动池。如果稍后成像,请将载玻片(不使用流通池)储存在 4 °C 的储存缓冲液中。 准备好成像后,在应用流通池之前,将载玻片从储存缓冲液转移到 PBS 中 10 分钟。
- 根据实验中所需的循环,开始制备含有添加剂的 1x 运行缓冲液。通过混合制备低 (1:4) 和高浓度 DMSO 缓冲液 (9:1),制备所需循环所需的 1 份 DMSO 与 4 份电泳缓冲液和 9 份 DMSO 与 1 份电泳缓冲液。节省约 20 mL 的 1x 缓冲液与添加剂,用于制作报告板。要计算所需的 1x 缓冲液(含添加剂和高/低 DMSO 缓冲液),请咨询仪器管理器。
- 开始准备报告板。首先,使报告基因原液达到总循环数所需的结果。用循环名称标记黑色或琥珀色的 1 mL 微量离心管。向相应的试管中加入 250 μL 报告基因储备液和 5 μL 每个报告基因。然后,将溶液转移到黑色 96 孔板中,并用胶箔密封。
- 启动设备,使用仪器管理器设置参数和曝光时间(如表 1),并获取图像。 图 1 总结了染色前和染色后的过程。
5. 组织注释和细胞分割
- 设置项目和工作区。
- 安装最新版本的数字病理学分析软件(例如,QuPath 版本 0.5.1 或更高版本)。单击 Create project 以选择项目空间的目标文件夹。
- 单击 Add images > Choose files,然后导航到 QPTIFF 文件(由多重免疫荧光成像生成)。将图像类型设置为 Fluorescence(荧光),保留所有其他默认设置,然后单击 Import(导入)。
- 如果使用 QuPath,请双击新图像以打开工作区。定期单击 File > Save 以跟踪对项目所做的更改。使用工具栏中间附近的 亮度和对比度 (半月图标)切换标记可见性和查看设置。
注意:右键单击图像列表中的图像可对其进行重命名。这在以后可视化表型分类时将很重要。
- 注释整个组织切片、瘤内隔室和基质隔室。
- 使用画笔和/或魔棒工具在整个组织切片周围绘制注释。将此注释定义为 Full_Tissue。排除覆盖的皮肤和/或任何不应分析的区域。要创建负性注释和/或收缩注释边界,请在使用其中一个注释工具时按住 Alt 键。
- 复制完整的组织注释。选择 注释 选项卡下的 注释 ,然后转到 对象 > 注释...> 复制选定的注释。
- 打开 SOX10 通道。在复制的注释上,缩小注释边界以使用 Alt + 画笔/魔杖工具捕获肿瘤内隔室。将此注释定义为肿瘤(图 2)。
- 选择完整的组织注释,转到 Objects > Annotations...> 展开注释。将 Expansion radius (扩展半径) 定义为 1 μm,然后单击 Run(运行)。
- 将新注释重命名为 Full_Tissue_Expansion。选择 Tumor Annotation,右键单击,然后选择 Insert in hierarchy。
- 重新选择 Tumor Annotation,转到 Objects > Annotations...> Make inverse(反函数)。将此新注释定义为 Stroma。对所有组织切片重复此作。
- 运行细胞分割并导出结果。
- 转到 图像 选项卡以查找图像的像素宽度和高度(图 3A)。
- 在 https://github.com/qupath/qupath-extension-stardist/releases 下载 StarDist 扩展。将 qupath-extension-stardist-[version].jar 文件拖到 QuPath 窗口中,然后单击 QuPath 窗口右上角的齿轮图标。在 Extensions (扩展) 下,将 QuPath 用户目录 定义为 StarDist 文件夹的位置。
- 从 https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 下载 StarDist groovy 脚本文件和 StarDist 模型文件(StarDist 脚本最初由 Akoya Biosciences 组织)。
- 对于每个组织切片,同时选择 Tumor 和 Stroma 注释 (使用 Ctrl 选择多个注释)。在顶部设置栏中,单击 自动化 > 脚本编辑器 打开脚本界面。打开与图像像素大小对应的相应 StarDist 单元格分割脚本(groovy 文件)。当系统提示选择单元格分割文件时,打开 stardist_cell_seg_model.pb 文件。
- 单元格分割后,保存图像。转到 Measure > Export measurements(导出测量值),然后选择 相应的图像。分配 导出类型 作为 单元格 和 分隔符 作为 .csv,然后选择 输出文件 位置。单击 Columns 旁边的 Populate 以包含感兴趣的量度。要导出的重要列包括 Image、Object ID、Parent、Centroid X、Centroid Y 和 Nucleus Area。
注意:在后续步骤中使用对象 ID 将分类重新映射回数字病理学分析软件中的多重免疫荧光图像。Parent 列指示单元格所属的注释名称。X 和 Y 数据用于空间分析。 - 对于要用于聚类/表型分析的每个谱系标记,请选择一个平均值进行导出。对于细胞核标记物(例如 SOX10 和 FOXP3),请导出 Nucleus: Mean 选项。对于细胞质/膜标志物(例如 CD45、CD3、CD4),请导出 Cytoplasm: Mean 选项。
6. 蛋白质组学数据预处理和归一化
- 打开导出的 CSV 文件。对于要在聚类/表型分析中使用的每个标记,截断列标题的名称以仅包含标记。例如,将 SOX10: Nucleus: Mean 重命名为 SOX10,将 CD3: Cytoplasm: Mean 重命名为 CD3。
- 安装最新版本的 R 和 RStudio。从 https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 下载 Marker Normalization.R 文件并运行脚本。首先涉及通过细胞核大小过滤掉细胞。
- 然后对于每个标记,执行最小-最大标准化,以便将表达式范围内的最低 MFI 值设置为 0,并将第 99.7个 百分位数的 MFI 值设置为 1。所有高于第 99.7 个百分位数的强度都被裁剪为 1。完成后,保存使用规范化数据生成的新 CSV 文件。
7. 聚类和表型
- 根据标记物表达谱选择一种算法来聚类细胞。https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 上提供了 R 脚本,用于使用 Seurat v4.415 和 FlowSOM v2.13.9(聚类和可视化工具)16 进行聚类。
- 根据关键谱系标记物的标记表达谱对细胞进行表型分析。这里介绍的方案使用了 14 个谱系标记物进行聚类和表型分析:SOX10、CD45、CD3、CD4、CD8、FOXP3、CD11C、F4/80、CD68、CD86、CD163、CD206、NK1.1 和 CD31。
- 将肿瘤细胞定义为 SOX10hi。将 CD4 T 细胞定义为 CD45mod-hi/ CD4hi/CD8低/FOXP3低。将 CD8 T 细胞定义为 CD45mod-hi/ CD8hi/CD4low。将 Treg 定义为 CD45mod-hi/CD4hi/CD8low/FOXP3hi。将其他 T 细胞定义为 CD45mod-hi/CD3hi/CD4low/CD8low。
- 将 M1 巨噬细胞定义为 CD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163low /CD206low。将 M2 巨噬细胞定义为 CD45mod-hi/F4/80hi/CD68mod-hi/CD86低/CD163hi/CD206低、CD45mod-hi/F4/80hi/CD68mod-hi/CD86低/CD163低/CD206hi 或 CD45mod-hi/F4/80hi/CD68mod-hi/CD86低/CD163hi/CD206hi。将其他巨噬细胞定义为 CD45mod-hi/F4/80hi/CD68mod-hi/CD86低/CD163低/CD206低 或 CD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hi。
- 将树突状细胞定义为 CD45mod-hi/CD11chi/CD86mod-hi。将 NK 细胞定义为 CD45mod-hi/NK1.1hi。将其他免疫细胞定义为不满足上述详细条件的 CD45mod-hi 。将内皮细胞定义为 CD31mod-hi。删除所有其他表达谱。
8. 重新映射表型分类并进行质量控制
- 从 https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 下载 Remapping Classifications.groovy 脚本(此脚本改编自 petebankhead 在 https://forum.image.sc/ 上的一篇博文)。
- 打开表型 CSV 文件,并确保 Image 选项卡下的所有标签与将用于重新映射的数字病理学分析软件中的图像文件名称完全匹配。要在 CSV 文件中快速更改大量标签,请使用 Replace All 函数。
- 如果使用 QuPath,请单击 Automate > Script editor(自动脚本编辑器)来运行分类重新映射 groovy 文件。
- 在 Annotations 选项卡上,单击 Auto set 按钮旁边的 Three Dots ,然后单击 Populate from existing objects > All classes (including subclasses) > Yes。
- 在标记列表中向下滚动以查看不同的表型分类,并根据需要调整配色方案。双击任何单元格可查看其表型 (在 Classification 标签旁边) 和 cluster (在 Name 标签旁边)。检查代表性视野以确定表型分配的准确性,并根据需要修改表型策略(图 3B)。
9. 密度量化和空间分析
- 一旦表型分析完成,在数字病理学分析软件的 Annotations 选项卡下获取注释区域。将面积值转换为 mm2,然后将计数除以注释区域以计算密度。
- 从 https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 下载 SPIAT Spatial Analysis.R 脚本并运行。收集有关平均最小距离 (AMD)、邻域组成和归一化混合分数 (NMS) 的数据。
- 在统计分析软件中生成数字以呈现结果。当为治疗条件的平均最小距离生成单个热图时,对从不同组织切片获得的结果进行平均。
结果
在这里,我们提出了一种方案,用于设计小鼠 FFPE 组织的抗体面板,进行多重免疫荧光成像,以及分析图像以进行蛋白质组学定量和空间关系。经过验证的检测组合包含 27 种抗体,这些抗体为可视化黑色素瘤细胞 (SOX10)、白细胞 (CD45)、T 细胞(CD3、CD4、CD8、FOXP3)、B 细胞 (CD20)、巨噬细胞和亚型(F4/80、CD68、CD86、CD163、CD206)、树突状细胞 (CD11c)、NK 细胞 (NK1.1) 和内皮细胞 (CD31)。该检测组合还包含其他免疫群体(CD11b、CD38)、增殖活性 (Ki67)、T 细胞功能(T-bet、Eomesodermin、颗粒酶 B)、抗原呈递(LMP2、β-2 微球蛋白、MHC II)和检查点表达(TIM3、LAG3、PD-L1)的标志物12。代表性的凝胶电泳图像证实了 DNA 寡核苷酸条形码与无载体蛋白抗体的成功偶联,如图 4 所示。此步骤仅确认化学反应,只有在使用多重成像设备检查这些抗体对目标组织后才能进行图像确认。 请参阅以下参考资料以查看面板13 中所有 27 个标记的验证图像。 图 5 显示了用瘤内 4-1BBL/IL-12 纳米颗粒注射和全身抗 PD1 处理的三个小鼠黑色素瘤组织切片染色的关键谱系标志物的融合图像。这些切片用于本协议中的后续图像分析。
在组织注释、细胞分割和蛋白质组学数据预处理之后,我们比较了以前应用于单细胞转录组学和/或蛋白质组学分析的两种聚类算法17,18。两种方法的表型群体的表达谱如图 6 所示。我们表明,FlowSOM 提供了更广泛的强度值范围(~0.7 vs ~0.5),用于识别相似细胞群(例如巨噬细胞亚型)之间的差异。Seurat 在聚类过程中应用了鲁汶算法,并生成了 29 个聚类(补充图 1)。相比之下,FlowSOM 应用了自组织映射,可以生成 100 个集群(补充图 2)。更多的簇数意味着表型分析所需的时间更多,但后一种 FlowSOM 方法在对相似细胞群进行分类时也提供了更多的细微差别。从定性上讲,我们看到,与相同视野中的 Seurat 表型相比,FlowSOM 能够将更多的瘤内巨噬细胞分类为 M1 或 M2 亚型(图 7)。当我们量化巨噬细胞密度时可以看到相同的结果,与 Seurat 相比,FlowSOM 捕获的 M1 和 M2 巨噬细胞密度更高(图 8A-B),随后其他/未分类巨噬细胞的密度显着降低 (p = 0.0028;图 8C)。然而,在描述其他细胞群密度(例如 CD4 T 细胞、CD8 T 细胞和内皮细胞)时,这两种分析方法也产生了相似的结果(图 8D-F)。
我们还介绍了聚类和表型分析后的下游空间分析结果。 图 9 演示了可以使用此协议生成的一些空间指标。相对于所有表型群体,M2 巨噬细胞和 NK 细胞在用 4-1BBL/IL-12 纳米颗粒和抗 PD1 处理后具有最高的 AMDs(图 9A)。同样,瘤内 CD8 T 细胞和 M1 巨噬细胞之间的 NMS 远高于 CD8 T 细胞和 M2 巨噬细胞之间的 NMS(图 9B)。此外,M2 巨噬细胞在瘤内 CD8 T 细胞周围的 100 μm 邻域中贡献了 ~1%,而 M1 巨噬细胞占这些邻域的 9%-13%(图 9C)。综上所述,这些结果表明,4-1BBL/IL-12 治疗方案将肿瘤相关巨噬细胞极化为 M1 亚型,并将 M2 巨噬细胞排除在肿瘤免疫微环境中。
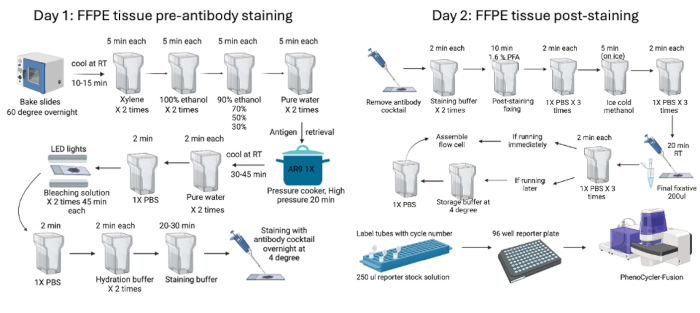
图 1:FFPE 组织染色和成像工作流程总结。 小鼠 FFPE 组织使用预染色程序进行处理,该程序从烘烤组织过夜开始,然后开始为期两天的预染色(第 1 天)和后染色(第 2 天)过程。最后,在使用设备成像之前准备报告板。 请单击此处查看此图的较大版本。
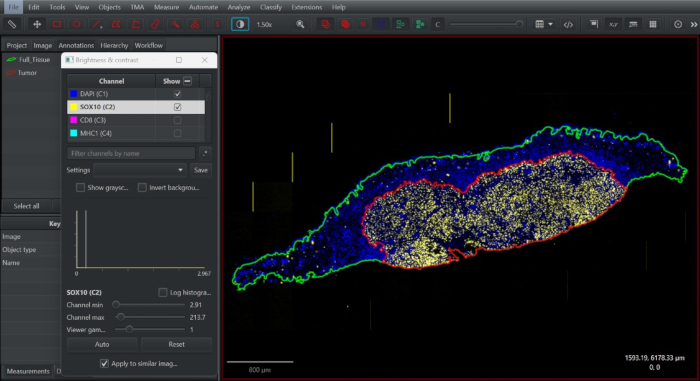
图 2:开放访问数字病理学软件 QuPath 中的组织注释屏幕截图。 绘制完整的组织注释(绿色)、复制并最小化,以根据肿瘤边界 (红色) 的 SOX10 + 黑色素细胞的分布定义瘤内隔室。 请单击此处查看此图的较大版本。
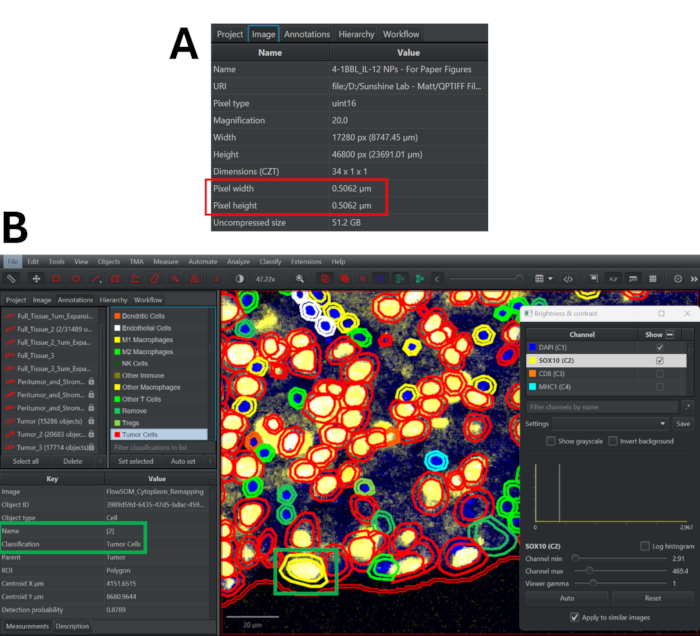
图 3:在 QuPath 中使用 StarDist 算法进行细胞分割,以及用于查看表型分类的质量控制过程。 (A) 导航到“图像”选项卡,以确定多重免疫荧光图像的像素宽度和高度。(B) 重新映射分类后,双击任何单元格(以黄色突出显示)以查看其簇分配和表型。打开/关闭面板标记以确定此分类方法是否准确。 请单击此处查看此图的较大版本。

图 4:抗体-DNA 条形码偶联确认的代表性凝胶图像。 蛋白质凝胶电泳证实抗体与 DNA 寡核苷酸条形码的偶联,通过重链位点的其他条带观察到。 请单击此处查看此图的较大版本。
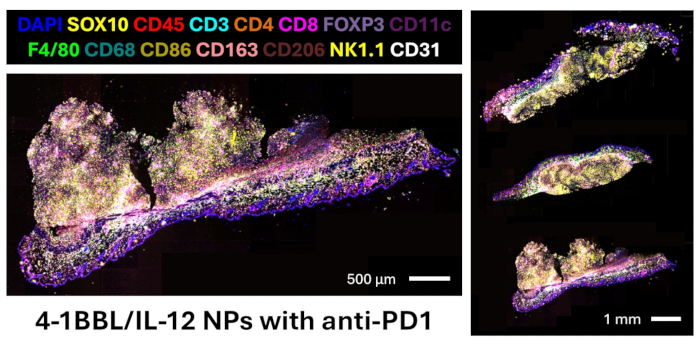
图 5:用全身抗 PD1 瘤内注射 4-1BBL/IL-12 纳米颗粒治疗的 B16F10 侧腹肿瘤的基于 DNA 条形码的多重成像。 我们面板中的标志物未显示:CD11b、CD20、CD38、TIM3、LAG3、T-bet、Eomesodermin、颗粒酶 B、Ki67、LMP2、β-2 微球蛋白、MHC II、PD-L1。 请单击此处查看此图的较大版本。

图 6:表型细胞群的蛋白质组学表达热图。 (A) 与 (B) FlowSOM 聚类和表型分析相比,Seurat 聚类和表型方法产生的标记物表达值范围略小。 请单击此处查看此图的较大版本。

图 7:FlowSOM 表型可识别更大范围的不同巨噬细胞亚型。 上图显示了相同视野下的 Seurat(左)和 FlowSOM(右)巨噬细胞表型。下图显示了同一视野中关键巨噬细胞谱系标记物的多重免疫荧光图像。所有比例尺均为 20 μm。 请单击此处查看此图的较大版本。

图 8:聚类/表型后比较不同细胞群的瘤内密度。 显示了瘤内 (A) M1 巨噬细胞、 (B) M2 巨噬细胞、 (C) 其他巨噬细胞、 (D) CD4 T 细胞、 (E) CD8 T 细胞和 (F) 内皮细胞的密度比较。误差线是均值 (SEM) 的标准误差,使用未配对的 t 检验 (**p ≤ 0.01) 检验显著性。 请单击此处查看此图的较大版本。

图 9:分析用 4-1BBL/IL-12 瘤内纳米颗粒注射和全身抗 PD1 治疗的三个侧腹肿瘤的瘤内空间指标。 (A) 瘤内表型群体之间平均最小距离的热图。测量值以 μm 为单位。(B) 关键瘤内表型对之间的标准化混合评分。(C) 瘤内 CD8 T 细胞群周围 100 μm 半径内的邻域组成分解。 请单击此处查看此图的较大版本。
| 抗体 | 稀释 | 曝光时间 (ms) |
| SOX-10 系列 | 50 | 450 |
| CD8型 | 100 | 450 |
| CD3 系列 | 100 | 450 |
| 福克斯P3 | 50 | 350 |
| CD4 | 50 | 450 |
| MHC-II 型 | 100 | 450 |
| PDL1 | 50 | 450 |
| CD45的 | 100 | 450 |
| Ki-67 系列 | 100 | 300 |
| F4/80 | 100 | 150 |
| CD20的 | 100 | 450 |
| NK1.1/CD161 蛋白 | 50 | 450 |
| CD206的 | 100 | 450 |
| CD68的 | 100 | 450 |
| 颗粒酶-B | 50 | 450 |
| CD86的 | 100 | 150 |
| CD31的 | 100 | 450 |
| CD11C型 | 50 | 450 |
| CD11乙 | 50 | 450 |
| EOMES | 50 | 450 |
| TIM-3 (蒂姆-3) | 50 | 300 |
| CD38的 | 50 | 200 |
| LAG3 | 50 | 450 |
| CD163的 | 50 | 200 |
| T-bet | 50 | 300 |
| LMP2 | 100 | 100 |
| β2 毫克 | 200 | 50 |
表 1:抗体稀释度和曝光时间设置。
补充 图 1:Seurat 聚类后生成的初始蛋白质组学表达热图。 请点击此处下载此图表。
补充 图 2:FlowSOM 聚类后生成的初始蛋白质组学表达热图。 请点击此处下载此图表。
讨论
成像的成功取决于精心设计和验证的抗体组合。由于高自发荧光和难以检索被石蜡包埋掩盖的表位,FFPE 样品的多重免疫荧光成像带来了挑战。然而,鉴于 FFPE 与 FF 样本相比具有多项优势,因此设计和验证 FFPE 抗体组合至关重要。第一步是最终确定在免疫荧光 (IF) 成像过程中显示阳性信号的抗体克隆;随后,将它们与 DNA 条形码小心地偶联非常重要。抗体偶联需要部分还原抗体以产生 SH 键,在马来酰亚胺基团反应过程中与条形码利用 SH 键。并非每个抗体克隆都能承受这一步,有些反应会对抗体造成不可逆的损伤,导致尽管偶联成功,但成像失败。因此,即使一些抗体在常规 IF 验证过程中可能显示阳性信号,但要评估抗体偶联的最终成功,必须在实际目标组织上验证每种抗体并记录该组织所需的暴露时间。在未来的应用中,该技术可以与连续载玻片/同一载玻片的现有空间转录组学分析相结合,以产生额外的见解。这种方法的局限性之一是,它需要根据靶标和组织类型仔细选择和验证检测组合中的每种抗体。
在图像分析方面,QuPath 提供了一个有价值的开放访问工具,具有蛋白质组学标志物的高质量可视化、用于导出强度测量值和执行表型分类质量控制的广泛功能,以及用户生成脚本的良好灵活性。在线论坛(如 https://forum.image.sc/)是讨论如何完成特定分析任务以及与其他用户共享脚本的附加资源。在该协议中,我们比较了使用 Seurat 和 FlowSOM 的两种聚类和表型方法。虽然 FlowSOM 可能是首选,因为它能够对 TME 的免疫细胞亚群产生更精细的见解,但也必须考虑蛋白质组学分析所需的时间。如果用户只需要对一个或两个组织样本中的细胞进行表型分析,则可能不需要生成 100 个簇。在这些情况下,Seurat 可能会提供更快、更高效的图像分析管道。相比之下,在两种分析方法中,分析具有 40 或 50 个组织切片的 TMA 更有可能产生更多的细胞簇,而 FlowSOM 可能是生成更细微的表型分类的首选方法。
细胞聚类/表型和所有后续图像分析步骤在很大程度上取决于细胞分割。我们目前的工作探索了 HALO (Indica Labs) 和 StarDist 算法中的细胞分割,我们发现这两种方法都倾向于基于核 DAPI 信号对细胞进行过度分割。此外,还提供了许多替代分割算法,例如 Mesmer19 和 InstanSeg20。这是一个不断发展的计算研究领域,需要进一步探索和优化。
披露声明
J.C.S. 感谢 Emerson Collect, LLC 和美国国立卫生研究院的财政支持。J.J.G. 还感谢美国国立卫生研究院 (National Institutes of Health) 的资助。JCS 与 Palleon Pharmaceuticals Inc. 建立了关系,这涉及资助赠款。S.Y.T. 和 J.J.G. 与 OncoSwitch Therapeutics 存在涉及股权或股票的关系。S.Y.T.、J.J.G.、J.C.S. 和 K.M.L. 有一项正在申请的专利。所有其他作者都表示,他们没有已知的竞争性经济利益或个人关系,这些利益或个人关系可能会被认为会影响本文所介绍的研究。
致谢
JCS 感谢皮肤病学基金会和皮肤病理学职业发展奖,以推动作者的职业生涯。作者感谢美国国家癌症研究所癌症数据科学实验室的 Hsin-Pei Lee 在计算分析技术方面的帮助。这项研究得到了 Emerson Collective 和美国国立卫生研究院 (R37CA246699、P41EB028239 和 R01CA228133) 的资助。此外,约翰霍普金斯大学肿瘤学组织服务 (OTS) 核心得到了美国国立卫生研究院 (P30CA006973) 的支持。
材料
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
参考文献
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。