Method Article
التصوير المناعي متعدد الإرسال القائم على الباركود الشريطي للحمض النووي لتحليل عينات FFPE من الورم الميلانيني الفئراني المعاد برمجته وراثيا
* These authors contributed equally
In This Article
Summary
يوفر هذا البروتوكول دليلا تفصيليا لتصميم لوحة الأجسام المضادة للتألق المناعي متعددة الإرسال للتصوير المستند إلى الباركود الحمض النووي لأنسجة الورم الميلانيني FFPE في الفئران. نصف أيضا خط أنابيب تحليل الصور باستخدام أدوات مفتوحة المصدر لتوليد رؤى البروتينات المكانية في البيئة المكروية المناعية لورم الورم الميلانيني الفئراني.
Abstract
يتم تقديم تقنية التصوير المتعدد الإرسال الناشئة القائمة على الباركود الشريطي للحمض النووي تعتمد على الاكتشاف المشترك عن طريق indEXing الذي يحلل البروتينات المكانية للبيئات الدقيقة للأنسجة. يتطلب التصوير الناجح ذخيرة من لوحات الأجسام المضادة المصممة جيدا والتي تم التحقق من صحتها بشكل صحيح ، ولكن يوجد عدد قليل جدا حاليا لعينات البارافين المثبتة بالفورمالين (FFPE). يوفر FFPE العديد من المزايا مقارنة بالعينات المجمدة الطازجة ، مثل التوافر على نطاق واسع ، وسهولة المناولة والتخزين ، والقدرة على صنع المصفوفات الدقيقة للأنسجة (TMAs). هنا ، نقدم بروتوكولا لتطوير لوحة أجسام مضادة لتصور وتحليل أنسجة FFPE من نموذج الورم الميلانيني في الفئران المعالج بجسيمات نانوية ، والتي توفر إشارات مناعية تشفير الحمض النووي البلازميد لإعادة برمجة البيئة المكروية للورم. نصف أيضا خط أنابيب تحليل الصور باستخدام أدوات حسابية مفتوحة المصدر للتعليق على الأنسجة ، وتجزئة الخلايا ، ومعالجة بيانات البروتينات ، والتنميط الظاهري لمجموعات الخلايا ، وتحديد المقاييس المكانية. يقدم البروتوكول تطبيقات لتصميم ألواح الأجسام المضادة في FFPE للفئران وتوليد رؤى جديدة حول البروتينات المكانية للبيئات الدقيقة للأنسجة المعقدة.
Introduction
الورم الميلانيني الجلدي هو أكثر أنواع سرطان الجلد شيوعا ، مع معدلات متفاوتة للأمراض والوفيات في جميع أنحاء العالم اعتمادا على وقت التشخيص والرعاية الأولية1. على مدى العقد الماضي ، ساعد الفهم البيولوجي المتزايد للورم الميلانيني في دفع تطوير نماذج جديدة للسرطان لعلاج الأورام الصلبة2. أدى الارتفاع الأخير للعلاج المناعي إلى مفهوم ثوري لعلاج السرطان يعتمد على تنشيط الجهاز المناعي الداخلي3،4.
البيئة المكروية للورم (TME) معقدة للغاية ، وتتكون من خلايا مناعية متنوعة ، والخلايا الليفية المرتبطة بالسرطان ، والخلايا المحيطة ، والخلايا البطانية ، والعديد من الخلايا المقيمة في الأنسجة5. تم تطبيق العديد من التقنيات في الماضي لدراسة TME ، مثل قياس التدفق الخلوي وتسلسل الخلية المفردة ، مما يضر بالسياق المكاني لأنه مطلوب لتدمير أنسجة الورم. يسمح التصوير المجهري التقليدي ، مثل التألق المناعي (IF) والكيمياء المناعية (IHC) ، بتصور المؤشرات الحيوية للبروتين دون تدمير أنسجة العينة. ومع ذلك ، فإن هذه الأساليب تقتصر على اثنين أو ثلاثة مؤشرات حيوية وغير قادرة على توفير فهم كامل للعلاقات المكانية والهيكلية داخل TME 6 المعقد.
لمعالجة هذه المشكلة ، تم تطوير العديد من تقنيات التصوير متعدد الإرسال لتصور TME المعقد مكانيا7،8،9،10. أحد هذه الأجسام هو CoDetection-by-inDEXing ، الذي أعيدت تسميته باسم نظام PhenoCycler ، استنادا إلى الأجسام المضادة المترافقة بقليل النوكليوتيدات DNA11. يمكن للنظام توفير تصوير وتحليل أحادي الخلية لأكثر من 100 مؤشر حيوي للعينات البشرية. ومع ذلك ، يتوفر عدد قليل جدا من الأجسام المضادة المخزنة لتصور وتحليل عينات الفئران ، وخاصة عينات البارافين المدمجة بالفورمالين (FFPE)12. يوفر FFPE العديد من المزايا مقارنة بحفظ المجمدة الطازجة (FF) ، مثل سهولة المناولة والتخزين ، والتشكل المحفوظ جيدا بمرور الوقت ، والأهم من ذلك ، القدرة على إعداد المصفوفات الدقيقة للأنسجة / الورم (TMA) التي تسمح بتصور العديد من العينات على شريحة واحدة. لقد قمنا مؤخرا بتصميم وتطوير لوحة الأجسام المضادة للفئران FFPE CODEX / PhenoCycler وقمنا بتطبيقها بنجاح لتصور وتحليل البروتينات المكانية لعينات الورم الميلانيني الفئراني المعاد برمجتها وراثيا13.
الهدف العام من هذا البروتوكول هو توفير دليل خطوة بخطوة لتصميم لوحة الأجسام المضادة FFPE للفئران ووصف عملية اقتران الباركود بين الأجسام المضادة وتلطيخ الأنسجة والتصوير. بالإضافة إلى ذلك ، نقدم خط أنابيب مفصل لتحليل الصور باستخدام أدوات مفتوحة المصدر مثل حزم QuPath و R. بعد اتباع هذا البروتوكول ، سيتعلم الباحثون كيفية تصميم لوحة الأجسام المضادة المترافقة حسب الطلب ، وإجراء التصوير المتعدد باستخدام جهاز Phenocycler-Fusion ، واكتساب رؤى جديدة حول البروتينات المكانية للورم الميلانيني TME. علاوة على ذلك ، يمكن تكييف هذا البروتوكول لدراسة البيئات الدقيقة المناعية المختلفة للورم ودمجها مع تقنيات النسخ المكانية الحالية.
Protocol
تم تنفيذ جميع أعمال وفقا للإرشادات التي وضعتها لجنة رعاية واستخدام بجامعة جونز هوبكنز ، باستخدام أرقام البروتوكول المعتمدة MO18M388 و MO21M384.
1. اختيار الأجسام المضادة
- صمم لوحة أجسام مضادة بناء على الأنسجة ذات الأهمية ووفرة العلامة الحيوية المعينة. اختر استنساخ الأجسام المضادة بناء على التطبيقات السابقة الناجحة للتألق المناعي (IF) وتلوين الكيمياء النسيجية المناعية (IHC) في الأدبيات. إصدارات آمنة خالية من الناقلات من هذه الأجسام المضادة.
ملاحظة: الجسم المضاد المنقى الخالي من الناقل مهم لاقتران الباركود. تجنب أي مواد حافظة تحتوي على البروتين مثل BSA والغلوتين والجلسرين وما إلى ذلك. إذا كان الجسم المضاد الخالي من الناقلات غير متوفر ، فاطلب جسما مضادا مخصصا من المورد أو استخدم مجموعة إزالة BSA لجعل الجسم المضاد خاليا من الناقل. أزيد الصوديوم لا يتداخل مع اقتران الأجسام المضادة. يوصى باستخدام الأنماط الإسوية IgG على الأنماط المتماثلة IgM. - تحقق من هذه المستنسخة المختارة على الأنسجة ذات الأهمية باستخدام التصوير المناعي التقليدي (IF)11. استخدم تصوير IF المزدوج لتأكيد خصوصية بعض المستنسخة (على سبيل المثال ، قم بإجراء تصوير IF مزدوج باستخدام FOXP3 و CD4 لتأكيد تلطيخ الخلايا التائية التنظيمية).
ملاحظة: كلما أمكن ، من المهم استخدام نفس الأنسجة للتحقق من صحة IF التي سيتم استخدامها للتلطيخ. حدد محلول استرجاع المستضد المناسب. في هذا البروتوكول ، استخدمنا المخزن المؤقت AR9 الذي توفره Akoya Biosciences لاسترداد جميع حواتم أنسجة الورم الميلانيني الفئراني FFPE. يمكن استخدام حلول استرجاع المستضد الأخرى ، مثل AR6 أو Universal ، بناء على متطلبات الحاتمة.
2. اقتران الأجسام المضادة وتأكيدها
- قم بتعيين الأجسام المضادة التي تم التحقق منها للرموز الشريطية التي تحتوي على مراسلين مجانيين متصلين بالفلوروفورات ATTO550 (Cy3) أو AF647 (Cy5) أو AF750 (Cy7). عادة ما تنتج المستضدات منخفضة الوفرة إشارات أقل. اقتران هذه الأجسام المضادة بقنوات منخفضة التألق الذاتي مثل AF647 (Cy5). اقتران المستضدات عالية التعبير مع ATTO550 (Cy3) و AF750 (Cy7) بسبب إمكانية التألق الذاتي.
ملاحظة: تأخذ هذه الخطوة الحاسمة في الاعتبار حساسية القناة ووفرة المستضد. لا ينصح باستخدام قناة AF488 لأنسجة FFPE بسبب التألق الذاتي الأعلى. - لاقتران الباركود بين الأجسام المضادة (4.5 ساعة) ، احصل على مجموعة اقتران الأجسام المضادة. قم بتخزين جميع الكواشف عند 4 درجات مئوية باستثناء محلول الاختزال 1 ، والذي يجب تخزينه عند -20 درجة مئوية كقوارير صغيرة تستخدم لمرة واحدة.
- قياس تركيز الأجسام المضادة باستخدام مقياس الطيف الضوئي.
- ابدأ اقتران الأجسام المضادة عن طريق تطبيق 500 ميكرولتر من محلول حجب المرشح على عمود مرشح MWCO 50 كيلو دالتون لمنع الارتباط غير المحدد للأجسام المضادة بالمرشح. قم بالدوران عند 12,000 × جم لمدة دقيقتين في درجة حرارة الغرفة (RT). قم بإزالة السائل الزائد من العمود باستخدام ماصة دقيقة سعة 200 ميكرولتر.
ملاحظة: محلول الاختزال 1 عبارة عن قارورة محلول تستخدم مرة واحدة ، وهي كافية لثلاثة اقترانات للأجسام المضادة في وقت واحد. لا تعيد استخدام المحلول بعد الذوبان ؛ تجاهل أي كاشف متبقي. لا تستخدم مرشحا فوق عمود مرشح MWCO 50 كيلو دالتون ، حيث يمكن أن يؤدي ذلك إلى ضعف التنقية والاقتران. - استخدم حجما مكافئا يبلغ 50 ميكروغرام من محلول الجسم المضاد وأضفه إلى مرشح MWCO 50 كيلو دالتون. اضبط مستوى الصوت الإجمالي إلى 100 ميكرولتر باستخدام PBS إذا لزم الأمر. قم بالدوران عند 12,000 × جم لمدة 8 دقائق عند 4 درجات مئوية. خلال هذا الوقت ، قم بإعداد مزيج الاختزال الرئيسي باستخدام 19.8 ميكرولتر من محلول الاختزال 1 + 825 ميكرولتر من محلول الاختزال 2 لثلاثة اقترانات للأجسام المضادة.
- تخلص من التدفق وأضف 260 ميكرولتر من مزيج الاختزال الرئيسي إلى الجزء العلوي من كل وحدة مرشح. محلول الدوامة في وحدة المرشح لمدة 2-3 ثوان واحتضانه لمدة 30 دقيقة في RT.
ملاحظة: من الأهمية بمكان ألا تتجاوز 30 دقيقة من الحضانة لتجنب الإفراط في تقليل الأجسام المضادة ، لأن هذا يضر بها ويمكن أن يؤدي إلى فشل الاقتران. - بعد 30 دقيقة من الحضانة ، قم بالدوران عند 12,000 × جم لمدة 8 دقائق عند 4 درجات مئوية ، وتخلص من التدفق في الأسفل. أضف 450 ميكرولتر من محلول الاقتران إلى أعلى العمود وقم بالدوران لأسفل عند 12,000 × جم لمدة 8 دقائق عند 4 درجات مئوية. أثناء الطرد المركزي ، قم بإعداد محلول الباركود CODEX. تحرك بسرعة بعد استرداد الباركود من -20 درجة مئوية ، حيث يبدأ الباركود في التدهور.
ملاحظة: عادة ما يتم توفير الرموز الشريطية في قوارير زجاجية صغيرة تحتوي على منتجات مجففة بالتجميد (رقائق أو مسحوق) مخزنة عند -20 درجة مئوية. يمكن استخدام هذه القوارير لاقتران 50 ميكروغرام من الأجسام المضادة في وقت واحد. يوصى بعدم اقتران أكثر من ثلاثة أجسام مضادة في المرة الواحدة. - حدد موقع الرمز الشريطي المجفف بالتجميد بعناية في الجزء السفلي من القارورة الزجاجية (قد يكون أيضا عالقا على الجدران). اضغط على القارورة الزجاجية على طاولة لإحضار المواد الصلبة إلى الأسفل. يعد تحديد موقع المنتج المجفف بالتجميد أمرا ضروريا عند إذابته في 10 ميكرولتر من الماء الخالي من البيولوجيا الجزيئية الخالية من النوكلياز. بعد إضافة 10 ميكرولتر من الماء الخالي من النوكلياز ، أضف 210 ميكرولتر من محلول الاقتران إلى كل رمز شريطي. قم بإذابة جميع المواد المجففة بالتجميد واخلطها برفق عن طريق سحب العينات لأعلى ولأسفل. جانبا.
- بعد الانتهاء من الدوران في الخطوة 2.2.5 ، تجاهل التدفق وأضف حل الباركود المعني من الخطوة 2.2.6 إلى أعلى كل وحدة مرشح. حفظ 1 ميكروغرام من الجسم المضاد الأولي غير المترافق ؛ سيتم تشغيل هذا مع عينة الأجسام المضادة المترافقة 5 ميكرولتر أثناء التحقق من صحة الرحلان الكهربائي للهلام.
- قم بتسمية كل أنبوب بالجسم المضاد واسم الباركود المعنيين. أغلق الغطاء واخلط المحلول عن طريق الدوامة لمدة 2-3 ثوان. احتضان تفاعل اقتران الباركود بين الأجسام المضادة لمدة 2 ساعة في RT.
- بعد حضانة 2 ساعة ، قم بإزالة 5 ميكرولتر من الجسم المضاد المترافق وتخزينها في أنبوب PCR سعة 0.2 مل لتأكيد الاقتران بواسطة الرحلان الكهربائي للهلام.
- قم بتدوير محلول الأجسام المضادة المترافق المتبقي عند 12,000 × جم لمدة 8 دقائق عند 4 درجات مئوية. أضف 450 ميكرولتر من محلول التنقية إلى الجزء العلوي من العمود ، وقم بالدوران عند 12,000 × جم لمدة 8 دقائق عند 4 درجات مئوية ، وتخلص من التدفق من خلاله. كرر خطوة التنقية هذه 2x ، مع إضافة محلول تنقية 450 ميكرولتر في كل مرة وقم بتدويره.
- بعد الطردالمركزي 3 ، تخلص من التدفق من خلال. سيحتوي المرشح العلوي على الأجسام المضادة المترافقة بالباركود.
- لجمع الجسم المضاد المترافق ، قم بتسمية الأنبوب الخارجي الجديد الذي يحمل عمود المرشح بالجسم المضاد والرمز الشريطي المقابلين. اقطع غطاء الأنبوب الخارجي قبل الطرد المركزي. أضف 100 ميكرولتر من محلول تخزين الأجسام المضادة إلى كل وحدة مرشح وضع الأنبوب الخارجي المسمى حديثا رأسا على عقب أعلى عمود المرشح.
- اقلب عمود المرشح لتجميع الجسم المضاد المترافق في الأنبوب الخارجي وقم بالدوران عند 3,000 × جم لمدة دقيقتين في RT. يجب أن يجمع هذا حوالي 120 ميكرولتر من محلول الأجسام المضادة المترافقة. انقله إلى أنبوب طرد مركزي دقيق معقم وخزنه في درجة حرارة 4 درجات مئوية لمدة 18-24 شهرا.
ملاحظة: لتجنب تلطيخ نووي عالي الخلفية بعد الاقتران ، يوصى بعدم استخدام هذه الأجسام المضادة لمدة يومين على الأقل.
- قم بإجراء تأكيد الاقتران بواسطة الرحلان الكهربائي للهلام كما هو موضح في البروتوكول13 المنشور بالفعل.
ملاحظة: تؤكد هذه العملية فقط نجاح تفاعل الاقتران الكيميائي المستخدم لاقتران الرموز الشريطية بالأجسام المضادة. إنه اختياري ، حيث لا يمكن التحقق من صحة الأجسام المضادة إلا بعد تلطيخ الأنسجة وتصويرها بنجاح.
3. إعداد عينة FFPE الفئران
- الحصول على عينات من أنسجة FFPE من إناث الفئران C57BL / 6J المزروعة بأورام الخاصرة B16F10. عالج الفئران بالحقن داخل الورم من الجسيمات النانوية القائمة على poly (β-amino ester) التي تحتوي على 4-1BBL و IL-12 بلازميدات ، جنبا إلى جنب مع توصيل داخل الصفاق ل anti-PD1 في 9 و 11 و 16 و 18 يوما بعد الزرع14. قم بالتضحية بالفئران في اليوم 20 وقم بإصلاح الأورام في 10٪ فورمالين ، مدمج في البارافين ، ثم قسم في مركز خدمات أنسجة الأورام بجامعة جونز هوبكنز.
- ضع في اعتبارك بعناية مواصفات منطقة التصوير الواردة في دليل المستخدم وقم بتركيب عينات 5 ميكرومتر على شريحة داخل منطقة التصوير. تمكنا من تركيب أربع عينات مختلفة من الورم على شريحة واحدة.
ملاحظة: من الضروري استخدام الشرائح الموصى بها المتاحة للتصوير ، مثل Leica Apex Adhesive Slides أو Fisherbrand Superfrost Plus Slides. سيساعد ذلك خلية التدفق على الالتصاق بقوة بالشريحة.
4. تلطيخ أنسجة FFPE والتصوير
- التحقق من تركيز الأجسام المضادة وأوقات التعرض: للتحقق من نجاح اقتران الباركود بين الأجسام المضادة ، قم بتلطيخ الأنسجة ذات الأهمية ب 6-9 أجسام مضادة مترافقة. قم بتشغيل جهاز التصوير المتعدد ولاحظ تركيز كل جسم مضاد ووقت التعرض. اضبط التخفيفات وأوقات التعرض حسب الحاجة ، ثم قم بتشغيل 6-9 أجسام مضادة جديدة مع تلك التي تم التحقق منها مسبقا ولاحظ أي تعديلات. اتبع الخطوات أدناه للتلطيخ والتصوير.
- تلطيخ الأنسجة والتصوير
- في اليوم السابق للتلطيخ ، اخبز شرائح مناديل FFPE في فرن 60 درجة مئوية طوال الليل. في اليوم التالي ، قم بتبريد الشرائح لمدة 10 دقائق في RT قبل البدء في خطوات إزالة البارافينات وتجفيف الأنسجة.
- ابدأ بإزالة الأنسجة عن طريق احتضانها في محلول زيلين 2x لمدة 5 دقائق لكل منهما.
- لإعادة ترطيب الأنسجة ، حركها من خلال جولتين من محلول الإيثانول بنسبة 100٪ لمدة 5 دقائق لكل منهما. بعد ذلك ، انقله عبر سلسلة من محاليل الكحول من 90٪ إلى 70٪ و 50٪ و 30٪ لمدة 5 دقائق لكل منها. أخيرا ، اغسل المنديل بالماء المقطر مرتين لمدة 5 دقائق لكل منهما.
ملاحظة: يمكن استخدام بدائل أقل سمية للزيلين ، مثل محلول Neoclear ، لخطوات إزالة البارافين. قم بإجراء إزالة البارافين وإعادة الجفاف تحت غطاء الدخان. - قم بتخفيف 10x AR9 المؤقت إلى 1x باستخدام الماء المقطر. املأ جرة Coplin ب 50 مل من المخزن المؤقت 1x AR9 ، وتأكد من أن الشرائح مغمورة بالكامل في المخزن المؤقت لاسترجاع المستضد.
- املأ قدر الضغط بالماء بحيث يكون المستوى في منتصف الطريق أعلى جرة كوبلين ، ثم احتضنه تحت إعداد الضغط العالي لمدة 20 دقيقة.
ملاحظة: من الممكن استخدام قدر ضغط متخصص أو غرفة إزالة التشويه لاسترجاع المستضد بشكل أفضل عن طريق ضبطه على 120 درجة مئوية لمدة 10 دقائق. - بعد الانتهاء من خطوة قدر الضغط ، قم بتبريد الشرائح لمدة 45-60 دقيقة. اشطف الشريحة بالماء المقطر 2x لمدة دقيقتين لكل منهما. ثم انقل الشريحة إلى البرطمان الذي يحتوي على 1x PBS.
- لتقليل التألق الذاتي ، قم بإجراء خطوة تبييض على الأنسجة قبل تلطيخها بالأجسام المضادة. قم بإعداد محلول تبييض طازج (25 مل من 1x PBS + 0.8 مل من 1M هيدروكسيد الصوديوم + 4.5 مل من H2O2) باستخدام الكواشف والتركيزات الموصى بها. احتضان الشرائح في وعاء بلاستيكي يحتوي على محلول التبييض بين مصباحي LED لمدة 45 دقيقة في درجة حرارة الغرفة. كرر مع محلول التبييض الطازج لمدة 45 دقيقة أخرى.
- ابدأ في تحضير محلول كوكتيل الأجسام المضادة خلال خطوة التبييض هذه ، خاصة إذا كان هناك العديد من الأجسام المضادة في اللوحة مع تخفيفات مختلفة. استرجع جميع الحاصرات الأربعة (G و S و J و N) وضعها على الجليد ، حيث يستغرق بعضها وقتا لإذابة الجليد من -20 درجة مئوية.
- بعد التبييض الضوئي ، اغسل المنديل ب 1x PBS ، 2x لمدة دقيقتين لكل منهما. انقل الأنسجة إلى برطمانات تحتوي على محلول ترطيب واغسلها 2x لمدة دقيقتين لكل منهما.
- انقل شرائح الأنسجة إلى مخزن التلوين واتركها تتوازن لمدة 30 دقيقة. إذا لم يتم تحضيره أثناء خطوة التبييض ، فقم بإعداد محلول كوكتيل الأجسام المضادة خلال هذا الوقت.
- قم بإعداد 300 ميكرولتر من محلول كوكتيل الأجسام المضادة لتلطيخ شريحتين (عادة ، إذا كنت تستخدم حجرة بلاستيكية مهجنة ، فإن 100-120 ميكرولتر من كوكتيل الأجسام المضادة يكفي لتلطيخ كل شريحة.)
- قم بتلطيخ الشرائح عن طريق وضع محلول كوكتيل الأجسام المضادة داخل حجرة الرطوبة طوال الليل عند 4 درجات مئوية.
ملاحظة: تظهر جميع الأجسام المضادة هنا إشارة إيجابية عند تلطيخها طوال الليل عند 4 درجات مئوية. ومع ذلك ، قد يختلف وقت تلطيخ كل جسم مضاد ودرجة حرارته ويجب تحسينه. ظروف التلوين الأكثر استخداما هي 3 ساعات في RT أو بين عشية وضحاها عند 4 درجات مئوية. إذا لزم الأمر ، من الممكن أيضا تلطيخ بعض الأجسام المضادة في البداية في درجات حرارة مختلفة ثم الأجسام المضادة المتبقية طوال الليل عند 4 درجات مئوية. اغسل المناديل بعد ذلك باستخدام PBS إذا كانت هناك حاجة إلى تلطيخ متسلسل. - في اليوم التالي ، قم بإجراء تثبيت ما بعد التلوين. قم بإزالة الحجرة البلاستيكية وتخزين كوكتيل الأجسام المضادة المجمعة عند 4 درجات مئوية. اغسل الشرائح 2x في مخزن التلوين لمدة دقيقتين لكل منهما. بعد ذلك ، انقل الشرائح إلى محلول تثبيت بعد التلوين سعة 40 مل (4 مل من 16٪ بارافورمالدهيد + 36 مل من المخزن المؤقت للتخزين) واحتضنه لمدة 10 دقائق. اشطفها باستخدام PBS 3x لمدة دقيقتين لكل منهما.
- انقل الشرائح إلى الميثانول المثلج لمدة 5 دقائق ، ثم اشطفها باستخدام PBS 3x لمدة دقيقتين لكل منهما. ضع محلول مثبت نهائي (20 ميكرولتر أنبوب واحد + 1 مل من PBS) على الشرائح الموجودة أسفل غرفة الرطوبة. عادة ما يكون 200 ميكرولتر من محلول التثبيت كافيا لكل شريحة. اترك المثبت لمدة 20 دقيقة في RT. قم بإجراء ثلاث غسلات أخيرة في PBS لمدة 2 دقيقة لكل منهما.
ملاحظة: لا تقم بإزالة الأنبوب المثبت من -20 درجة مئوية مسبقا ؛ قم بإذابة الثلج فقط قبل التقديم. - في حالة التصوير على الفور ، امسح الشريحة حول الأنسجة باستخدام منديل خال من النسالة وقم بتطبيق خلية التدفق بالضغط عليها لمدة 30 ثانية باستخدام معدات تجميع خلايا التدفق. في حالة التصوير لاحقا، قم بتخزين الشريحة (دون تطبيق خلية التدفق) في المخزن المؤقت للتخزين عند 4 درجات مئوية. عندما تكون جاهزا للتصوير، انقل الشرائح من المخزن المؤقت للتخزين إلى PBS لمدة 10 دقائق قبل تطبيق خلية التدفق.
- ابدأ في تحضير المخزن المؤقت الجاري 1x مع مادة مضافة بناء على الدورات المطلوبة في التجربة. قم بإعداد مخازن DMSO منخفضة (1:4) وعالية (9:1) عن طريق الخلط كذاكرة DMSO من جزء واحد مع مخزن مؤقت للتشغيل مكون من 4 أجزاء و DMSO مكون من 9 أجزاء مع مخزن مؤقت للتشغيل مكون من جزء واحد مطلوب للدورات المطلوبة. وفر حوالي 20 مل من المخزن المؤقت 1x مع مادة مضافة لصنع لوحة مراسل. لحساب المخزن المؤقت 1x المطلوب مع المخازن المؤقتة المضافة والمرتفعة/المنخفضة DMSO، استشر مدير الأدوات.
- ابدأ في إعداد لوحة المراسل. أولا ، قم بعمل حل مخزون المراسل المطلوب لإجمالي عدد الدورات. قم بتسمية أنابيب الطرد المركزي الدقيقة باللون الأسود أو الكهرماني 1 مل بأسماء الدورات. أضف 250 ميكرولتر من محلول مخزون المراسل و 5 ميكرولتر من كل مراسل إلى الأنبوب المقابل. بعد ذلك ، انقل المحلول إلى صفيحة سوداء سعة 96 بئرا وأغلقها بورق لاصق.
- ابدأ تشغيل الجهاز ، واستخدم مدير الأدوات لتعيين المعلمات وأوقات التعرض (كما هو موضح في الجدول 1) ، واحصل على الصور. يلخص الشكل 1 عمليات ما قبل وبعد التلوين.
5. شرح الأنسجة وتجزئة الخلايا
- قم بإعداد مشروع ومساحة عمل.
- قم بتثبيت أحدث إصدار من برنامج تحليل علم الأمراض الرقمي (على سبيل المثال ، QuPath الإصدار 0.5.1 أو أعلى). انقر فوق إنشاء مشروع لاختيار مجلد وجهة لمساحة المشروع.
- انقر فوق إضافة صور > اختر الملفات ، ثم انتقل إلى ملف QPTIFF (تم إنتاجه من التصوير المناعي متعدد الإرسال). قم بتعيين نوع الصورة إلى Fluorescence، واحتفظ بجميع الإعدادات الافتراضية الأخرى، ثم انقر فوق Import.
- إذا كنت تستخدم QuPath ، فانقر نقرا مزدوجا فوق الصورة الجديدة لفتح مساحة عمل. انقر فوق ملف > حفظ بشكل دوري لتعقب التغييرات التي تم إجراؤها على المشروع. قم بتبديل إعدادات رؤية العلامة والعرض باستخدام السطوع والتباين (أيقونة نصف القمر) بالقرب من منتصف شريط الأدوات.
ملاحظة: انقر بزر الماوس الأيمن فوق صورة في قائمة الصور لإعادة تسميتها. سيكون هذا مهما لاحقا عند تصور تصنيفات النمط الظاهري.
- على قسم الأنسجة الكامل ، والحيز داخل الورم ، والحيز اللحمي.
- استخدم أداة الفرشاة و / أو العصا لرسم تعليق توضيحي حول قسم الأنسجة بالكامل. حدد هذا التعليق التوضيحي على أنه Full_Tissue. استبعاد الجلد المغطى و / أو أي مناطق لا ينبغي تحليلها. لإنشاء تعليق توضيحي سلبي و / أو تقليص حدود التعليقات التوضيحية ، اضغط مع الاستمرار على المفتاح Alt أثناء استخدام إحدى أدوات التعليقات التوضيحية.
- تكرار التعليق التوضيحي الكامل للنسجة. حدد التعليق التوضيحي ضمن علامة التبويب التعليقات التوضيحية ، ثم انتقل إلى الكائنات > التعليقات التوضيحية ... > تكرار التعليقات التوضيحية المحددة.
- قم بتشغيل قناة SOX10. في التعليق التوضيحي المكرر ، قم بتقليص حدود التعليقات التوضيحية لالتقاط الحيز داخل الورم باستخدام أداة Alt + brush / wand. حدد هذا التعليق التوضيحي على أنه ورم (الشكل 2).
- عند تحديد التعليق التوضيحي الكامل للأنسجة ، انتقل إلى الكائنات > التعليقات التوضيحية ... > توسيع التعليقات التوضيحية. حدد نصف قطر التوسع على أنه 1 ميكرومتر وانقر فوق تشغيل.
- أعد تسمية التعليق التوضيحي الجديد إلى Full_Tissue_Expansion. حدد التعليق التوضيحي للورم، وانقر بزر الماوس الأيمن، وحدد إدراج في التدرج الهرمي.
- أعد تحديد التعليق التوضيحي للورم، وانتقل إلى الكائنات > التعليقات التوضيحية... > اصنع معكوسا. حدد هذا التعليق التوضيحي الجديد على أنه Stroma. كرر ذلك لجميع أقسام الأنسجة.
- تشغيل تجزئة الخلايا وتصدير النتائج.
- انتقل إلى علامة التبويب "صورة" للعثور على عرض البكسل وارتفاع الصورة (الشكل 3 أ).
- قم بتنزيل امتداد StarDist في https://github.com/qupath/qupath-extension-stardist/releases. اسحب ملف qupath-extension-stardist-[version].jar إلى نافذة QuPath ، ثم انقر فوق رمز الترس في الزاوية اليمنى العليا من نافذة QuPath. ضمن الامتدادات، حدد دليل مستخدم QuPath كموقع مجلد StarDist.
- قم بتنزيل ملفات البرنامج النصي الرائع StarDist وملف نموذج StarDist من https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (تم تنظيم نصوص StarDist في الأصل من Akoya Biosciences).
- لكل قسم من أقسام الأنسجة، حدد كلا من التعليقات التوضيحية للورم والستروما (استخدم Ctrl لتحديد تعليقات توضيحية متعددة). في شريط الإعدادات العلوي ، انقر فوق أتمتة محرر البرنامج النصي > لفتح واجهة البرامج النصية. افتح البرنامج النصي المناسب لتجزئة خلايا StarDist (ملف رائع) الذي يتوافق مع حجم بكسل الصورة. عند مطالبتك بتحديد ملف تجزئة الخلية، افتح الملف stardist_cell_seg_model.pb.
- بعد تجزئة الخلية، احفظ الصورة. انتقل إلى قياس > تصدير القياسات، ثم حدد الصورة (الصور) المقابلة. قم بتعيين نوع التصدير كخلايا والفاصل على أنه .csv، ثم اختر موقع ملف الإخراج . انقر فوق تعبئة بجانب الأعمدة لتضمين مقاييس الاهتمام. الأعمدة المهمة المراد تصديرها هي الصورة ومعرف الكائن والأصل والنقطة الوسطى X والنقطة الوسطى Y ومنطقة النواة.
ملاحظة: يتم استخدام معرف الكائن في الخطوات اللاحقة لإعادة تعيين التصنيفات مرة أخرى إلى صورة التألق المناعي متعدد الإرسال في برنامج تحليل علم الأمراض الرقمي. يشير العمود الأصل إلى اسم التعليق التوضيحي الذي تنتمي إليه الخلية. تستخدم بيانات X و Y للتحليل المكاني. - لكل علامة نسب لاستخدامها في التجميع / التنميط الظاهري ، حدد قيمة متوسطة واحدة للتصدير. بالنسبة للعلامات النووية (على سبيل المثال ، SOX10 و FOXP3) ، قم بتصدير خيار Nucleus: Mean. بالنسبة للعلامات السيتوبلازمية / الغشائية (على سبيل المثال ، CD45 ، CD3 ، CD4) ، قم بتصدير خيار السيتوبلازم: متوسط.
6. المعالجة المسبقة لبيانات البروتينات والتطبيع
- افتح ملف CSV الذي تم تصديره. لكل علامة سيتم استخدامها في التجميع / التنميط الظاهري ، اقتطاع اسم عنوان العمود لتضمين العلامة فقط. على سبيل المثال ، أعد تسمية SOX10: النواة: يعني إلى SOX10 و CD3: السيتوبلازم: يعني إلى CD3.
- قم بتثبيت أحدث إصدار من R و RStudio. قم بتنزيل ملف Marker Normalization.R من https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis وقم بتشغيل البرنامج النصي. يتضمن هذا أولا تصفية الخلايا حسب الحجم النووي.
- لكل علامة، يتم بعد ذلك إجراء تطبيع الحد الأدنى للحد الأقصى بحيث يتم تعيين أقل قيمة MFI في نطاق التعبير إلى 0 ويتم تعيين قيمة MFI عندالنسبة المئوية 99.7 إلى 1. يتم قص جميع الشدة فوقالنسبة المئوية 99.7 عند 1. بمجرد الانتهاء، احفظ ملف CSV الجديد الذي تم إنشاؤه باستخدام البيانات الطبيعية.
7. التجميع والتنميط الظاهري
- اختر خوارزمية لتجميع الخلايا وفقا لملفات تعريف تعبير العلامة. في https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis، يتم توفير البرامج النصية R للتجميع باستخدام Seurat v4.415 و FlowSOM v2.13.9 (أداة التجميع والتصور)16.
- خلايا النمط الظاهري وفقا لملفات تعريف تعبير العلامة لعلامات النسب الرئيسية. استخدم البروتوكول المقدم هنا 14 علامة نسب للتجميع والتنميط الظاهري: SOX10 و CD45 و CD3 و CD4 و CD8 و FOXP3 و CD11C و F4 / 80 و CD68 و CD86 و CD163 و CD206 و NK1.1 و CD31.
- تعريف الخلايا السرطانية على أنها SOX10مرحبا. حدد خلايا CD4 T على أنها CD45mod-hi / CD4hi / CD8منخفض / FOXP3منخفض. حدد خلايا CD8 T على أنها CD45mod-hi / CD8hi / CD4منخفضة. حدد Tregs على أنها CD45mod-hi / CD4hi / CD8منخفضة / FOXP3مرحبا. حدد الخلايا التائية الأخرى على أنها CD45mod-hi / CD3hi / CD4منخفضة / CD8منخفضة.
- حدد البلاعم M1 على أنها CD45mod-hi / F4 / 80hi / CD68hi / CD86hi / CD163منخفضة / CD206منخفضة. حدد البلاعم M2 على أنها CD45mod-hi / F4 / 80hi / CD68mod-hi / CD86منخفضة / CD163hi / CD206منخفضة ، CD45mod-hi / F4 / 80hi / CD68mod-hi / CD86منخفضة / CD163منخفضة / CD206مرحبا ، أو CD45mod-hi / F4 / 80hi / CD68mod-hi / CD86منخفضة / CD163hi / CD206مرحبا. حدد البلاعم الأخرى على أنها CD45mod-hi / F4 / 80hi / CD68mod-hi / CD86منخفض / CD163منخفض / CD206منخفض أو CD45mod-hi / F4 / 80hi / CD68mod-hi / CD86hi / CD163hi / CD206مرحبا.
- تعريف الخلايا المتغصنة على أنها CD45mod-hi / CD11chi / CD86mod-hi. حدد خلايا NK على أنها CD45mod-hi/NK1.1hi. حدد الخلايا المناعية الأخرى على أنها CD45mod-hi التي لا تفي بالشروط المفصلة أعلاه. تعريف الخلايا البطانية على أنها CD31mod-hi. قم بإزالة جميع ملفات تعريف التعبير الأخرى.
8. إعادة تعيين تصنيفات النمط الظاهري وإجراء مراقبة الجودة
- قم بتنزيل البرنامج النصي Remapping Classifications.groovy من https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (تم اقتباس هذا البرنامج النصي من منشور على https://forum.image.sc/ بواسطة petebankhead).
- افتح ملف CSV الظاهري وتأكد من أن جميع الملصقات الموجودة أسفل علامة التبويب صورة تتطابق تماما مع اسم ملف الصورة في برنامج تحليل علم الأمراض الرقمي الذي سيتم استخدامه لإعادة التعيين. لتغيير كميات كبيرة من التسميات بسرعة في ملف CSV، استخدم الدالة استبدال الكل.
- إذا كنت تستخدم QuPath ، فقم بتشغيل ملف رائع لإعادة تعيين التصنيفات بالنقر فوق أتمتة محرر البرنامج النصي >.
- في علامة التبويب التعليقات التوضيحية ، انقر فوق النقاط الثلاث بجوار الزر التعيين التلقائي ، ثم انقر فوق تعبئة من الكائنات الموجودة > جميع الفئات (بما في ذلك الفئات الفرعية) > نعم.
- قم بالتمرير لأسفل في قائمة العلامات لعرض تصنيفات النمط الظاهري المختلفة وضبط نظام الألوان حسب الحاجة. انقر نقرا مزدوجا فوق أي خلية لعرض النمط الظاهري (بجانب علامة التصنيف) والمجموعة (بجانب علامة الاسم). تحقق من مجالات الرؤية التمثيلية لتحديد دقة تعيينات النمط الظاهري ومراجعة استراتيجية التنميط الظاهري حسب الحاجة (الشكل 3 ب).
9. القياس الكمي للكثافة والتحليل المكاني
- بمجرد الانتهاء من التنميط الظاهري ، احصل على مناطق التعليقات التوضيحية في برنامج تحليل علم الأمراض الرقمي ضمن علامة التبويب التعليقات التوضيحية. قم بتحويل قيم المنطقة إلى مم2 ، ثم اقسم الأعداد على مناطق التعليقات التوضيحية لحساب الكثافات.
- قم بتنزيل البرنامج النصي SPIAT Spatial Analysis.R من https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis وقم بتشغيله. جمع البيانات حول متوسط المسافات الدنيا (AMD) ، وتركيبات الجوار ، ودرجات الخلط الطبيعية (NMS).
- إنشاء أرقام في برنامج التحليل الإحصائي لتقديم النتائج. عند إنشاء خريطة حرارية واحدة لمتوسط المسافات الدنيا لظروف العلاج ، قم بحساب متوسط النتائج التي تم الحصول عليها من أقسام الأنسجة المختلفة.
النتائج
هنا ، نقدم بروتوكولا لتصميم لوحة الأجسام المضادة لأنسجة FFPE للفئران ، وإجراء تصوير التألق المناعي متعدد الإرسال ، وتحليل الصور لقياس البروتينات والعلاقات المكانية. تحتوي اللوحة التي تم التحقق من صحتها على 27 جسما مضادا توفر علامات لتصور خلايا الورم الميلانيني (SOX10) ، والخلايا البيضاء (CD45) ، والخلايا التائية (CD3 ، CD4 ، CD8 ، FOXP3) ، والخلايا البائية (CD20) ، والضامة والأنواع الفرعية (F4 / 80 ، CD68 ، CD86 ، CD163 ، CD206) ، الخلايا المتغصنة (CD11c) ، الخلايا الطبيعية (NK1.1) ، والخلايا البطانية (CD31). تحتوي اللوحة الكاملة أيضا على علامات لمجموعات مناعية أخرى (CD11b ، CD38) ، ونشاط الانتشار (Ki67) ، ووظيفة الخلايا التائية (T-bet ، و Eomesodermin ، و granzyme B) ، وعرض المستضد (LMP2 ، microglobulin beta-2 ، معقد التوافق النسيجي الكبير II) ، وتعبير نقطة التفتيش (TIM3 ، LAG3 ، PD-L1) 12. تؤكد صورة الرحلان الكهربائي الهلامي التمثيلي الاقتران الناجح للباركود قليل النوكليوتيد للحمض النووي بالأجسام المضادة الخالية من الناقل ، كما هو موضح في الشكل 4. تؤكد هذه الخطوة التفاعل الكيميائي فقط ، ولا يمكن تأكيد الصورة إلا بعد فحص هذه الأجسام المضادة باستخدام جهاز التصوير المتعدد على الأنسجة ذات الاهتمام. راجع المرجع التالي لعرض الصور التي تم التحقق من صحتها لجميع العلامات ال 27 في اللوحة13. يتم عرض صورة اندماج لعلامات النسب الرئيسية التي تلطخ ثلاثة أقسام لأنسجة الورم الميلانيني في الفئران المعالجة بحقن الجسيمات النانوية 4-1BBL / IL-12 داخل الورم ومضاد PD1 الجهازي في الشكل 5. تم استخدام هذه الأقسام لتحليل الصور اللاحق في هذا البروتوكول.
بعد التعليق التوضيحي للأنسجة ، وتجزئة الخلايا ، والمعالجة المسبقة لبيانات البروتينات ، نقدم مقارنة بين خوارزميتين للتجميع تم تطبيقهما مسبقا على تحليل النسخ و / أو البروتينات أحادي الخلية17،18. يتم عرض ملفات تعريف التعبير للمجموعات السكانية ذات النمط الظاهري في الشكل 6 لكلا النهجين. نوضح أن FlowSOM يقدم نطاقا أوسع من قيم الشدة (~ 0.7 مقابل ~ 0.5) لتمييز الاختلافات بين مجموعات الخلايا المتشابهة ، مثل الأنواع الفرعية للبلاعم. طبق Seurat خوارزمية Louvain أثناء التجميع وأنتج 29 مجموعة (الشكل التكميلي 1). وبالمقارنة ، طبقت FlowSOM خرائط ذاتية التنظيم ويمكنها إنشاء 100 مجموعة (الشكل التكميلي 2). يترجم عدد أكبر من المجموعات إلى مزيد من الوقت المطلوب للتنميط الظاهري ، ولكن نهج FlowSOM الأخير هذا يوفر أيضا مزيدا من الفروق الدقيقة عند تصنيف مجموعات الخلايا المتشابهة. من الناحية النوعية ، نرى أن FlowSOM كان قادرا على تصنيف المزيد من البلاعم داخل الورم إما على أنها نوع فرعي M1 أو M2 عند مقارنتها بالتنميط الظاهري Seurat في نفس مجال الرؤية (الشكل 7). تظهر نفس النتيجة عندما نحدد كثافات البلاعم ، حيث يلتقط FlowSOM كثافة أعلى لكل من الضامة M1 و M2 مقارنة ب Seurat (الشكل 8A-B) وبالتالي كثافة أقل بكثير من الضامة الأخرى / غير المصنفة (p = 0.0028 ؛ ص = 0.0028 ؛ الشكل 8 ج). ومع ذلك ، فقد أنتج نهجان التحليل أيضا نتائج مماثلة عند وصف كثافات الخلايا الأخرى ، مثل خلايا CD4 T وخلايا CD8 T والخلايا البطانية (الشكل 8D-F).
نقدم أيضا نتائج التحليل المكاني المصب بعد التجميع والتنميط الظاهري. يوضح الشكل 9 بعض المقاييس المكانية التي يمكن إنشاؤها باستخدام هذا البروتوكول. بالنسبة لجميع المجموعات السكانية ذات النمط الظاهري ، كان لدى M2 الضامة والخلايا القاتلة الطبيعية أعلى AMDs بعد العلاج بالجسيمات النانوية 4-1BBL / IL-12 ومضاد PD1 (الشكل 9 أ). وبالمثل ، كان NMS بين خلايا CD8 T داخل الورم والضامة M1 أعلى بكثير من الخلايا التائية CD8 والضامة M2 (الشكل 9 ب). علاوة على ذلك ، ساهمت البلاعم M2 ~ 1٪ في الحي 100 ميكرومتر المحيط بخلايا CD8 T داخل الورم ، بينما شكلت البلاعم M1 9٪ -13٪ من هذه الأحياء (الشكل 9 ج). مجتمعة ، تشير هذه النتائج إلى أن نظام العلاج 4-1BBL / IL-12 استقطب البلاعم المرتبطة بالورم نحو نوع فرعي M1 واستبعد البلاعم M2 من البيئة المكروية المناعية للورم.
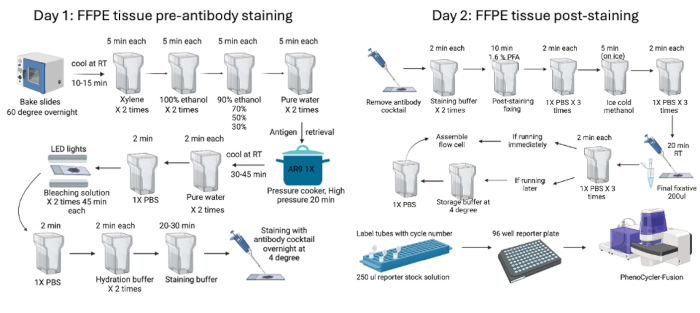
الشكل 1: ملخص لتلطيخ أنسجة FFPE وسير عمل التصوير. تمت معالجة أنسجة FFPE الفئران باستخدام إجراءات ما قبل التلوين التي بدأت بخبز الأنسجة طوال الليل قبل البدء في عمليات ما قبل التلوين لمدة يومين (اليوم 1) وما بعد التلوين (اليوم 2). أخيرا ، تم إعداد لوحة المراسل قبل التصوير بالجهاز. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
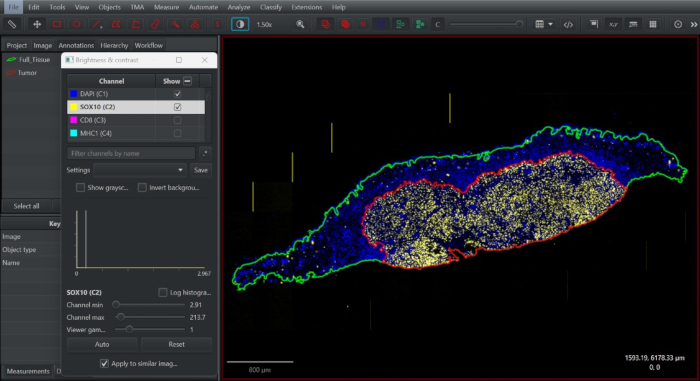
الشكل 2: لقطة شاشة للتعليق التوضيحي للأنسجة في برنامج علم الأمراض الرقمي مفتوح الوصول QuPath. تم رسم تعليق توضيحي كامل للأنسجة (أخضر) ، وتكراره ، وتصغيره لتحديد المقصورة داخل الورم وفقا لتوزيع الخلايا الصباغية SOX10 + عند حدود الورم (الأحمر). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
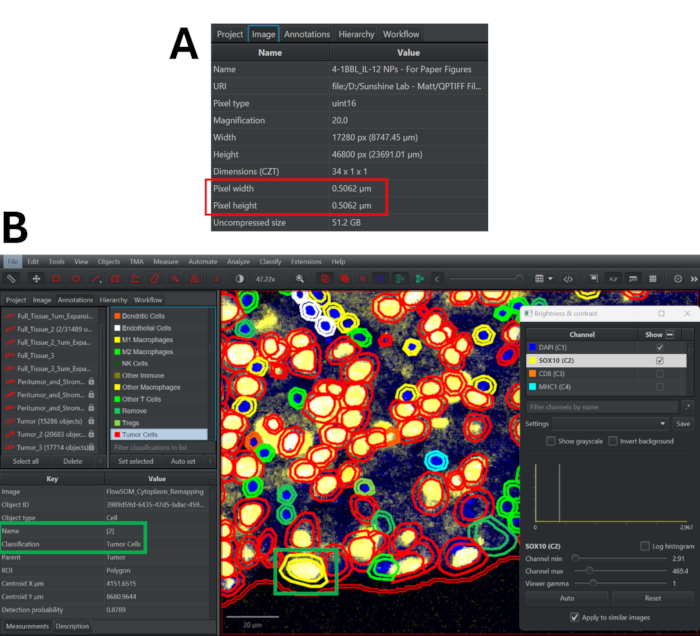
الشكل 3: تجزئة الخلايا باستخدام خوارزمية StarDist في QuPath وعملية مراقبة الجودة لمراجعة تصنيفات النمط الظاهري. (أ) انتقل إلى علامة التبويب صورة لتحديد عرض البكسل وارتفاعه لصورة التألق المناعي متعدد الإرسال. (ب) بعد إعادة تعيين التصنيفات ، انقر نقرا مزدوجا فوق أي خلية (تصبح مظللة باللون الأصفر) لرؤية تعيين نظام المجموعة والنمط الظاهري. قم بتبديل علامات لوحة التشغيل / الإيقاف لتحديد ما إذا كان نهج التصنيف هذا دقيقا أم لا. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 4: صورة هلامية تمثيلية لتأكيد اقتران الباركود للجسم المضاد والحمض النووي. يؤكد الرحلان الكهربائي لهلام البروتين اقتران الأجسام المضادة مع الرموز الشريطية قليلة النوكليوتيد للحمض النووي ، والتي لوحظتها نطاقات إضافية في موقع السلسلة الثقيلة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
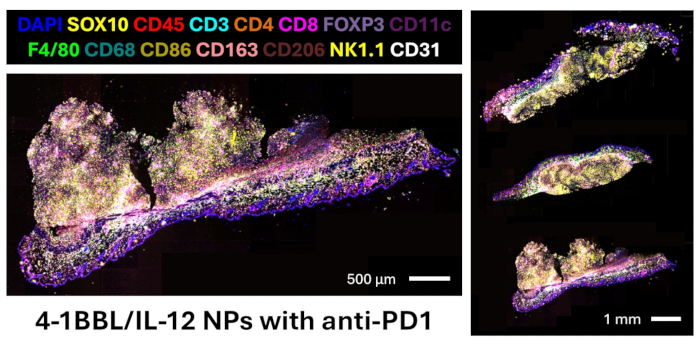
الشكل 5: التصوير المتعدد المستند إلى الباركود للحمض النووي لأورام الخاصرة B16F10 التي تم علاجها بالحقن داخل الأورام لجسيمات نانوية 4-1BBL / IL-12 مع مضاد PD1 الجهازي. العلامات الموجودة في لوحتنا غير معروضة: CD11b ، CD20 ، CD38 ، TIM3 ، LAG3 ، T-bet ، Eomesodermin ، granzyme B ، Ki67 ، LMP2 ، beta-2 microglobulin ، MHC II ، PD-L1. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 6: خرائط حرارية للتعبير عن البروتينات لمجموعات الخلايا ذات النمط الظاهري. (أ) يولد نهج تجميع Seurat والتنميط الظاهري نطاقا أصغر قليلا من قيم تعبير العلامات مقارنة ب (ب) تجميع FlowSOM والتنميط الظاهري. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 7: يحدد التنميط الظاهري ل FlowSOM نطاقا أكبر من الأنواع الفرعية المختلفة للبلاعم. تعرض اللوحة العلوية التنميط الظاهري للبلاعم Seurat (يسار) و FlowSOM (يمين) لنفس مجال الرؤية. تعرض اللوحة السفلية صورا متعددة الإرسال للتألق المناعي لعلامات سلالة البلاعم الرئيسية في نفس مجال الرؤية. جميع قضبان المقياس 20 ميكرومتر. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 8: مقارنة الكثافات داخل الورم لمجموعات الخلايا المختلفة بعد التجميع / التنميط الظاهري. تظهر مقارنات الكثافة للضامة داخل الورم (A) M1 ، (B) M2 الضامة ، (C) الضامة الأخرى ، (D) خلايا CD4 T ، (E) خلايا CD8 T ، و (F) الخلايا البطانية. أشرطة الخطأ هي أخطاء قياسية للمتوسط (SEM) ، وتم اختبار الأهمية باستخدام اختبارات t غير المزدوجة (** p ≤ 0.01). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 9: تحديد المقاييس المكانية داخل الورم لثلاثة أورام الخاصرة المعالجة بحقن الجسيمات النانوية داخل الورم 4-1BBL / IL-12 ومضاد PD1 الجهازي. (أ) خريطة حرارية لمتوسط المسافات الدنيا بين المجموعات السكانية ذات النمط الظاهري داخل الورم. القياسات بالميكرومتر. (ب) درجات الخلط الطبيعية بين أزواج النمط الظاهري الرئيسية داخل الورم. (ج) انهيار تركيبات الجوار في دائرة نصف قطرها 100 ميكرومتر حول مجموعات الخلايا التائية CD8 داخل الورم. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
| جسم | التخفيف | وقت التعرض (مللي ثانية) |
| سوكس -10 | 50 | 450 |
| CD8 | 100 | 450 |
| CD3 | 100 | 450 |
| فوكس بي 3 | 50 | 350 |
| CD4 | 50 | 450 |
| معقد التوافق النسيجي الثاني | 100 | 450 |
| PDL1 | 50 | 450 |
| سي دي 45 | 100 | 450 |
| كي -67 | 100 | 300 |
| F4 / 80 | 100 | 150 |
| سي دي 20 | 100 | 450 |
| NK1.1 / CD161 | 50 | 450 |
| CD206 | 100 | 450 |
| CD68 | 100 | 450 |
| جرانزيم ب | 50 | 450 |
| سي دي 86 | 100 | 150 |
| CD31 | 100 | 450 |
| سي دي 11 ج | 50 | 450 |
| CD11b | 50 | 450 |
| EOMES | 50 | 450 |
| تيم-3 | 50 | 300 |
| سي دي 38 | 50 | 200 |
| لاغ3 | 50 | 450 |
| سي دي 163 | 50 | 200 |
| تي رهان | 50 | 300 |
| LMP2 | 100 | 100 |
| بيتا 2 ملغ | 200 | 50 |
الجدول 1: إعدادات تخفيف الأجسام المضادة ووقت التعرض.
الشكل التكميلي 1: خريطة حرارية لتعبير البروتينات الأولية التي تم إنشاؤها بعد تجميع Seurat. الرجاء النقر هنا لتنزيل هذا الرقم.
الشكل التكميلي 2: خريطة حرارية تعبير البروتينات الأولية التي تم إنشاؤها بعد تجميع FlowSOM. الرجاء النقر هنا لتنزيل هذا الرقم.
Discussion
يعتمد نجاح التصوير على لوحة أجسام مضادة جيدة التصميم والتحقق من صحتها. يمثل التصوير المناعي المتعدد الإرسال لعينات FFPE تحديات بسبب التألق الذاتي العالي وصعوبة استرداد الحلقات المقنعة بتضمين البارافين. ومع ذلك ، نظرا لأن FFPE يوفر العديد من المزايا مقارنة بعينات FF ، فمن الضروري تصميم لوحات الأجسام المضادة FFPE والتحقق من صحتها. الخطوة الأولى هي الانتهاء من استنساخ الأجسام المضادة التي تظهر إشارات إيجابية أثناء التصوير المناعي (IF) ؛ بعد ذلك ، من المهم اقترانها بعناية مع الرموز الشريطية للحمض النووي. يتطلب اقتران الأجسام المضادة اخزالا جزئيا للجسم المضاد لإنشاء روابط SH ، والتي يتم استخدامها أثناء تفاعل مجموعة maleimide مع الباركود. لا يمكن لكل استنساخ للأجسام المضادة أن يتحمل هذه الخطوة ، ويمكن أن تسبب بعض التفاعلات ضررا لا رجعة فيه للجسم المضاد ، مما يؤدي إلى فشل التصوير على الرغم من الاقتران الناجح. لهذا السبب ، على الرغم من أن بعض الأجسام المضادة قد تظهر إشارات إيجابية أثناء التحقق من صحة IF التقليدي ، لتقييم النجاح النهائي لاقتران الأجسام المضادة ، فمن المهم التحقق من صحة كل جسم مضاد على الأنسجة الفعلية ذات الاهتمام وتسجيل أوقات التعرض المطلوبة لهذا الأنسجة. في التطبيقات المستقبلية ، يمكن دمج هذه التقنية مع تحليل النسخ المكاني الحالي على شرائح متتالية / نفس الشريحة لتوليد رؤى إضافية. أحد قيود هذه الطريقة هو أنها تتطلب اختيارا دقيقا والتحقق من صحة كل جسم مضاد في اللوحة بناء على الهدف ونوع الأنسجة.
فيما يتعلق بتحليل الصور ، تقدم QuPath أداة وصول مفتوح قيمة مع تصور عالي الجودة لعلامات البروتينات ، ووظائف واسعة لتصدير قياسات الكثافة وإجراء مراقبة الجودة لتصنيفات الأنماط الظاهرية ، ومرونة جيدة للنصوص التي ينشئها المستخدم. تعد المنتديات عبر الإنترنت مثل https://forum.image.sc/ مصدرا إضافيا لمناقشة كيفية إنجاز مهام تحليل محددة ومشاركة البرامج النصية مع مستخدمين آخرين. في هذا البروتوكول ، نقارن نهجين للتجميع والتنميط الظاهري باستخدام Seurat و FlowSOM. في حين أن FlowSOM قد يكون مفضلا لقدرته على توليد المزيد من الأفكار الدقيقة حول مجموعات الخلايا المناعية الفرعية في TME ، يجب أيضا مراعاة الوقت اللازم لتحليل البروتينات. قد يكون إنشاء 100 مجموعة غير ضروري إذا كان المستخدم يحتاج فقط إلى خلايا النمط الظاهري داخل عينة أو عينتين من الأنسجة. في هذه الحالات ، قد تقدم Seurat خط أنابيب أسرع وأكثر كفاءة لتحليل الصور. في المقابل ، من المرجح أن يؤدي تحليل TMA الذي يحتوي على ما يزيد عن 40 أو 50 قسما من الأنسجة إلى إنتاج عدد أكبر من مجموعات الخلايا في كلا نهجي التحليل ، وقد يكون FlowSOM هو المنهجية المفضلة لتوليد تصنيفات أكثر دقة للنمط الظاهري.
يعتمد تجميع الخلايا / التنميط الظاهري وجميع خطوات تحليل الصور اللاحقة إلى حد كبير على تجزئة الخلايا. استكشف عملنا الحالي تجزئة الخلايا في خوارزميات HALO (Indica Labs) و StarDist ، ووجدنا أن كلا النهجين يميلان إلى الإفراط في تقسيم الخلايا بناء على إشارات DAPI النووية. تتوفر أيضا العديد من خوارزميات التجزئة البديلة ، مثل Mesmer19 و InstanSeg20. هذا مجال متنامي للبحث الحسابي يتطلب مزيدا من الاستكشاف والتحسين.
Disclosures
تعترف JCS بالدعم المالي من Emerson Collective LLC والمعاهد الوطنية للصحة. كما يقر JG بالتمويل من المعاهد الوطنية للصحة. JCS لديها علاقة مع شركة Palleon Pharmaceuticals Inc. ، والتي تنطوي على منح التمويل. S.Y.T. و JJG لديهما علاقة مع OncoSwitch Therapeutics التي تنطوي على الأسهم أو الأسهم. S.Y.T. ، J.J.G. ، J.C.S. و K.M.L. لديها براءة اختراع معلقة. يذكر جميع المؤلفين الآخرين أنه ليس لديهم مصالح مالية متنافسة معروفة أو علاقات شخصية يمكن اعتبارها تؤثر على البحث المقدم في هذه الورقة.
Acknowledgements
تعترف JCS بمؤسسة الأمراض الجلدية وجائزة التطوير الوظيفي لأمراض الأمراض الجلدية للنهوض بمهنة المؤلف. يشكر المؤلفون هسين باي لي في مختبر علوم بيانات السرطان التابع للمعهد الوطني للسرطان للمساعدة في تقنيات التحليل الحسابي. تلقى هذا البحث تمويلا من Emerson Collective والمعاهد الوطنية للصحة (R37CA246699 و P41EB028239 و R01CA228133). بالإضافة إلى ذلك ، يتم دعم أساسي خدمات أنسجة الأورام في جونز هوبكنز (OTS) من قبل المعاهد الوطنية للصحة (P30CA006973).
Materials
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
References
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved