Method Article
Imagem de tempo de vida de fluorescência NAD (P) H para a análise metabólica do intestino murino e parasitas durante a infecção por nematóides
Neste Artigo
Resumo
O presente protocolo descreve a imagem de fluorescência NAD(P)H de um intestino murino explantado infectado com o parasita natural Heligmosomoides polygyrus, o que permite investigar processos metabólicos tanto no tecido do hospedeiro quanto no parasita de maneira espacialmente resolvida.
Resumo
Os parasitas geralmente têm um efeito negativo na saúde de seu hospedeiro. Eles representam um enorme fardo para a saúde, pois afetam globalmente a saúde do ser humano ou animal infestado a longo prazo e, portanto, afetam os resultados agrícolas e socioeconômicos. No entanto, efeitos imunorreguladores impulsionados por parasitas foram descritos, com potencial relevância terapêutica para doenças autoimunes. Embora o metabolismo do hospedeiro e dos parasitas contribua para sua defesa e seja a base para a sobrevivência dos nematóides no intestino, ele permaneceu amplamente pouco estudado devido à falta de tecnologias adequadas. Desenvolvemos e aplicamos imagens de fluorescência NAD (P) H ao tecido intestinal murino explantado durante a infecção com o nematóide natural Heligmosomoides polygyrus para estudar os processos metabólicos no hospedeiro e nos parasitas de maneira espacialmente resolvida. A exploração do tempo de vida de fluorescência das coenzimas nicotinamida adenina dinucleotídeo (NADH) e nicotinamida adenina dinucleotídeo fosfato (NADPH), doravante NAD (P) H, que são preservadas entre as espécies, depende de seu status de ligação e do local de ligação nas enzimas que catalisam os processos metabólicos. Concentrando-se nas enzimas dependentes de NAD(P)H mais abundantemente expressas, as vias metabólicas associadas à glicólise anaeróbica, fosforilação oxidativa/glicólise aeróbica e explosão oxidativa baseada em NOX, como um importante mecanismo de defesa, foram distinguidas, e a interferência metabólica entre o hospedeiro e o parasita durante a infecção foi caracterizada.
Introdução
As infecções parasitárias impõem um enorme fardo à saúde humana 1,2. Uma correlação entre o aumento das doenças autoimunes e o declínio das infecções parasitárias foi observada nos países industrializados. Sabe-se que os parasitas podem ter efeitos benéficos ao amortecer as respostas imunes excessivas do hospedeiro. O H. polygyrus é um parasita natural encontrado no intestino de roedores, e sabe-se que esse parasita induz mecanismos imunorreguladores que reduzem a resposta imune antiparasitária do hospedeiro por meio de, entre outros mecanismos, da indução de células T reguladoras (Treg) no hospedeiro infectado 3,4,5,6,7,8,9,10,11 . Esses mecanismos regulatórios são especialmente interessantes em doenças autoimunes degenerativas.
A análise da interferência metabólica entre o hospedeiro e os nematóides intestinais permanece amplamente negligenciada, embora o metabolismo desempenhe um papel importante tanto no hospedeiro quanto nos parasitas para defesa, sobrevivência e função. Propomos adaptar e aplicar imagens de tempo de vida de fluorescência de NADH e NADPH após excitação de dois fótons, uma tecnologia já amplamente utilizada em diferentes situações fisiológicas e fisiopatológicas em células e tecidos de mamíferos12, para investigar o metabolismo do hospedeiro e do nematóide em tecidos vivos de forma correlativa.
NADH e NADPH, conhecidos como NAD (P) H, são moléculas onipresentes que são preservadas em todas as formas de vida baseadas em células e desempenham o papel de coenzimas em várias vias metabólicas. Por exemplo, eles estão envolvidos na produção de energia, biossíntese redutiva e produção de espécies reativas de oxigênio (ROS) mediadas por NADPH oxidase, que estão principalmente ligadas à defesa celular e comunicação celular 13,14,15,16,17,20. Ambas as coenzimas emitem fluorescência a ~ 450 nm após excitação de dois fótons a 750 nm, permitindo assim imagens metabólicas sem marcadores em células e tecidos19,21. Excitar NADH e NADPH com apenas um comprimento de onda é possível devido aos seus espectros de excitação de dois fótons semelhantes e bastante amplos21.
O tempo de vida de fluorescência da coenzima NAD (P) H depende diretamente da enzima à qual ela se liga 18,21,22,23. Devido à sua estrutura química que permite a transferência de energia intramolecular, a molécula excitada de NADH ou NADPH perde energia por meio de processos de conversão interna, a uma taxa que depende de suas propriedades de ligação, às enzimas (catalisador) antes de relaxar e emitir um fóton de fluorescência. Este tempo de vida fornece informações sobre o local de ligação do NAD (P) H na enzima e, portanto, a reação bioquímica preferencial que ocorre19 , 21 , 22 , 23 , 24 , 25 . O tempo de vida de fluorescência das moléculas livres de NADH e NADPH é de ~ 450 ps, enquanto seu tempo de vida de fluorescência quando ligado a uma enzima é muito mais longo (~ 2.000 ps) e depende de seu local de ligação na respectiva enzima21.
Existem mais de 370 enzimas envolvidas em processos ligados a NAD(P)H; no entanto, apenas o mais abundante será capaz de contribuir para o tempo de vida de fluorescência NAD (P) H resultante dentro da faixa de excitação do microscópio. Usando dados de RNASeq de células de mamíferos, identificamos as enzimas dependentes de NAD(P)H mais abundantes e geramos uma referência de tempo de vida de fluorescência para interpretar os dados gerados em amostras de tecidos e células18. Assim, este trabalho distinguiu, por exemplo, entre a atividade preferencial da lactato desidrogenase (LDH), que está associada às vias metabólicas glicolíticas anaeróbias, e a atividade da isocitrato desidrogenase (IDH) e da piruvato desidrogenase (PDH), que estão envolvidas principalmente nas vias metabólicas da glicólise aeróbica/fosforilação oxidativa16,20. Além disso, a ligação do NADPH às NADPH oxidases, que são as principais enzimas responsáveis pela explosão oxidativa, pode ser facilmente resolvida devido à localização característica dessas enzimas na célula (ligada à membrana) e devido ao tempo de vida de fluorescência particularmente longo do NADPH (3.650 ps) 18 , 24 , 29 , 30 , 32 . Os dados de RNASeq de H. polygyrus mostram que a referência gerada para células de mamíferos também se aplica de forma adaptada a esse nematóide27.
Assim, neste trabalho, ao realizar imagens de fluorescência de NAD(P)H (FLIM) em amostras de duodeno recém-explantadas de camundongos infectados com H. polygyrus, foram adquiridas informações sobre a relação entre NAD(P)H livre e ligado à enzima, que retratou a atividade metabólica geral em todos os tecidos, bem como a enzima predominantemente ativa em cada pixel da imagem (ou seja, a enzima à qual o NAD(P)H se liga preferencialmente naquele local específico). O sucesso desses experimentos depende da preparação precisa da amostra do intestino explantado, da imagem ao vivo confiável da vida útil da fluorescência NAD(P)H na resolução subcelular e da avaliação padronizada de dados, conforme discutido neste protocolo.
Protocolo
Todos os experimentos foram realizados de acordo com as Diretrizes Nacionais de Proteção Animal e aprovados pelo Comitê Alemão de Ética Animal para a proteção de animais (G0176/16 e G0207/19). O protocolo descreve a aquisição de dados de imagem de fluorescência NAD (P) H e a avaliação de dados, que permitem avaliar a atividade metabólica geral e as vias metabólicas específicas no intestino do hospedeiro e nos parasitas após a infecção com o nematóide intestinal murino natural, H. polygyrus. Para isso, camundongos C57BL/6 fêmeas com idade entre 10 e 12 semanas foram infectados com 200 larvas de estágio 3 (L3). Em diferentes momentos da infecção, os camundongos infectados foram sacrificados e os duodenos foram excisados e preparados para imagem conforme descrito anteriormente33. Os duodenos de camundongos não infectados, pareados por idade e sexo, foram preparados e fotografados de forma semelhante para fins de controle. Para manter as propriedades teciduais necessárias para imagens e análises adicionais, as amostras devem ser processadas imediatamente após o explante, e as próximas etapas (etapas 1.1-1.7) devem ser executadas rapidamente (Figura 1B).
1. Preparação da amostra
- Corte pedaços de ~ 1 cm de comprimento do duodeno inicialmente excisado.
- Cole o tubo de tecido esticado no meio de uma pequena placa de Petri com cola de tecido de cura rápida (47 mm de diâmetro, 10 mm de altura da borda, consulte a Tabela de Materiais) (Figura 1A2).
- Aplique uma fina camada de cola adicional no fundo da placa de Petri sobre uma grande área ao redor do tecido com um micropincel (Figura 1A2).
- Use uma tesoura romba para fazer uma incisão ao longo de todo o comprimento do intestino fisicamente fixado nas proximidades do fundo da placa de Petri (Figura 1A3).
- Desdobre o intestino com uma pinça romba para que o lado abluminal entre em contato completo com a cola. Assim, o intestino fica fixado pela cola com o lado luminal voltado para cima (Figura 1A4).
- No caso de um camundongo infectado, conte os vermes em um microscópio estereoscópico (ampliação de 10x) para garantir que a infecção foi bem-sucedida (Figura 1A5).
- Sele o intestino com agarose para protegê-lo de secar. Aqui, a agarose a 0,5% a 0,9% foi usada para proteger o tecido intestinal sensível da queima devido ao seu ponto de fusão de baixa temperatura de aproximadamente 38 °C. Retire 1 mL de agarose em uma pipeta e regue cuidadosamente no intestino com contato suave entre a ponta da pipeta e o tecido, de modo que uma fina camada de cerca de 0,5 mm de espessura envolva completamente o tecido ( Figura 1A6 ).
- Encha as placas de Petri com as amostras seladas em agarose com PBS (10%) à temperatura ambiente e feche-as com a tampa. Fotografe as amostras diretamente ou coloque-as no gelo em uma caixa com isolamento térmico para fazer fila para medição. Colocar a primeira amostra preparada sob a objetiva do microscópio numa placa de aquecimento regulada para 37 °C (figura 1A8).
2. Exames de imagem
NOTA: O sistema de microscópio usado para realizar NAD (P) H-FLIM em amostras de tecido duodenal infectado e saudável consiste nos dispositivos listados e descritos na Figura 2 e na Tabela de Materiais. Use o ImSpector 208 como software de controle para todos os módulos usados.
- Para encontrar inicialmente a região de interesse (ROI), coloque a amostra na placa de Petri sob a objetiva e mova-se ao longo do plano x e y manualmente para encontrar uma ROI adequada por inspeção visual usando o modo de microscopia de fluorescência de campo amplo.
- Mude o sistema de imagem para excitação de dois fótons usando detecção de PMT ou detecção de contagem de fótons únicos correlacionada ao tempo (TCSPC) alternando os modos no software. Tome cuidado especial para garantir que o ambiente seja escuro e livre de vibrações, para envolver o microscópio em cortinas à prova de luz e usar uma mesa óptica suspensa pneumática, se possível.
- Mergulhe a lente objetiva na placa de Petri que contém a amostra com o software de controle clicando no ícone do sistema de lentes e girando a roda do mouse para alterar a posição z.
- Ajuste o laser para 765 nm e defina a potência máxima do laser para 10%, o que corresponde a 30 mW sob a objetiva, digitando o comprimento de onda desejado no painel de comprimento de onda do software. Ajuste a potência do laser, se necessário. Meça abaixo de um ganho de 40% (~ 30 mW a 100 mW) para evitar fotodanos teciduais e alterações metabólicas indesejadas.
- No software, defina o tamanho do passo do estágio z para 2 μm, que corresponde à resolução axial do microscópio a 765 nm no tecido.
- Defina a frequência de varredura de linha para 400 Hz no painel de espelho galvo, resultando em um tempo de permanência de pixel de 4.95 μs.
- Defina a média da imagem em duas a quatro imagens escolhendo o número médio no menu suspenso do software (ou seja, digitalize o ROI duas a quatro vezes para adquirir imagens mais suaves). Desta forma, o tempo total de permanência do pixel é aumentado para 9,9-19,8 μs para o benefício de uma relação sinal-ruído (SNR) mais alta no processo de análise.
- Defina o campo de visão da imagem (FOV) para 505 pixels x 505 pixels (500 x 500 μm²) escolhendo os parâmetros no painel FOV no software.
- Para cada medição, calibre o módulo com antecedência para garantir o funcionamento ideal da eletrônica TCSPC clicando em inicializar na janela de calibração no menu de hardware do TCSPC.
- Ajuste o fotodiodo rápido externamente para detectar o trem de pulso de laser Ti:Sa usando um osciloscópio rápido.
NOTA: O sinal do fotodiodo é usado para acionar a detecção de hPMT e a contagem de fótons pelo TCSPC em relação ao pulso de excitação. A detecção é repetida a cada 12,5 ns (ou seja, o tempo entre dois pulsos de laser consecutivos), correspondendo à taxa de repetição do laser Ti:Sa (80 MHz). - Defina a faixa da profundidade de medição definindo primeiro a média para 1 e o ganho da potência do laser para ~10%, depois iniciando o modo de vídeo (clique no botão de vídeo ), procurando a profundidade da qual o sinal ainda pode ser adquirido clicando no ícone do sistema de lentes novamente e usando a roda do mouse para progredir para a amostra enquanto aumenta o ganho progressivamente.
- Para as imagens NAD(P)H-FLIM das amostras de tecido medidas no modo FLIM, defina o sistema para 765 nm a uma potência nominal máxima de 100 mW. Detecte o sinal de fluorescência de NAD(P)H (466/60 nm) com o PMT híbrido com um ganho de 97%, que é definido no software. O módulo TCSPC conta fótons em 227 janelas de tempo (bins), cada uma com 55 ps.
- Clique com o botão direito do mouse na imagem exibida durante a medição no software e escolha exibir > T-PROFILE para visualizar a curva de decaimento adquirida pelo sistema.
- Observe o número de caixas antes do pulso de laser na curva de decaimento para análise posterior.
- Adquira os dados clicando em iniciar medição.
NOTA: Os dados devem corresponder a um formato específico. Cada fatia, registrada em qualquer profundidade de tecido, é separada por uma distância de 2 μm e tem uma área de 500 μm x 500 μm, e as fatias são adquiridas como uma pilha de fatias de 500 μm x 500 μm em pontos de tempo de 227 x 55 ps (Figura 3A). Cada voxel contém um histograma de tempo de chegada de fótons espacialmente resolvido (no plano x e no plano y). Isso representa o decaimento da fluorescência (Figura 3B). Um volume normalmente medido deve estar na forma de uma hiperpilha com dimensões de ~500 μm x 500 μm x 100-300 μm e 227 compartimentos, onde cada fatia no plano z contém os dados de intensidade dependentes do tempo, conforme descrito acima. Para um conjunto exemplar de dados de medição, isso resulta em (505 x 505 x 227) x 100 pixels (16 bits) e corresponde a cerca de 4 GB.
3. Análise dos dados
NOTA: Para a análise fasorial das imagens NAD(P)H-FLIM, o programa para calcular os tempos de vida é um código escrito sob medida em Python33.
- Use o Anaconda com uma distribuição Python 3.7 no IDE Spyder (consulte Tabela de Materiais). O código usa as bibliotecas padrão do Pythons.
- Carregue o código no IDE e execute. Uma caixa de diálogo de caminho de arquivo é aberta.
- Escolha a pasta com os dados brutos a serem analisados. O código é programado para escolher três parâmetros antes da análise por meio de uma caixa de diálogo de entrada do usuário do Tkinter com caixas de seleção, um menu suspenso e campos de texto.
- Escolha um deslocamento na caixa de diálogo de entrada. Determine o deslocamento como o número das primeiras fatias na pilha de tempo que não são analisadas.
NOTA: O parâmetro corta os pontos de tempo antes do pulso de excitação. Com a calibração do sistema e do diodo de disparo, esse valor deve ser de aproximadamente cinco fatias ou o número de compartimentos mortos antes da excitação do laser, conforme descrito na etapa 2.14. - Escolha a representação dos gráficos fasoriais no menu suspenso da caixa de diálogo de entrada. Aqui, escolha a aparência dos pontos de dados (semelhantes a nuvens ou topográficos), bem como os tiques do eixo de tempo do semicírculo (enzimas ou tempo [ps]), como opções no menu suspenso.
- Clique em OK.
- Verifique se o código calcula dois tipos de informações das pilhas z NAD(P)H-FLIM.
- Primeiro, meça as informações espaciais de cada fatia de volume pelo colapso das pilhas TCSPC, onde os 227 histogramas de contagem de fótons resolvidos no tempo são projetados em uma única fatia chamada imagem de projeção de intensidade (Figura 3G), e da análise baseada em Fourier das curvas de decaimento exponencial (conforme descrito por Leben et al.18), e da parte real e imaginária normalizada para cada pixel em cada fatia de volume (Figura 3C).
- A partir das imagens reais e imaginárias, obtenha os gráficos fasoriais (Figura 3D) e a imagem de tempo médio de fluorescência (t) (constantes médias de decaimento codificadas por cores e espacialmente resolvidas para cada voxel) (Figura 3E).
NOTA: Conforme descrito na introdução, o tempo de vida de fluorescência de NAD (P) H, quando ligado a enzimas, é determinado pelo local de ligação da coenzima à respectiva enzima. - Determine a contribuição da respectiva enzima para a atividade metabólica gerando o vetor entre o vetor de fase de cada pixel e o vetor de fase de NAD(P)H não ligado e projetando-o no semicírculo no gráfico fasorial. O semicírculo representa todos os possíveis decaimentos mono-exponenciais do tempo de vida da fluorescência em compostos puros.
- Usando a referência gerada anteriormente dos tempos de vida de fluorescência de NAD (P) H ligados às enzimas dependentes de NAD (P) H mais abundantes18,33 (Figura Suplementar 1), e calcule a probabilidade de ativação dessas enzimas.
NOTA: Um código de cores da enzima mais dominante (ou seja, da enzima para a qual a maior probabilidade de ativação é calculada) é atribuído a cada pixel, produzindo assim um mapa enzimático (Figura 3F).
- A partir da razão entre NAD(P)H livre (não ligado) e ligado a enzimas, especificamente a partir da razão vetorial no gráfico fasorial entre NAD(P)H livre a 450 ps e o estado ligado a enzimas, calcule a atividade metabólica geral como uma porcentagem entre 0% *apenas NAD(P)H não ligado) e 100% (apenas NAD(P)H ligado a enzimas). Ao atribuir um valor de atividade metabólica (0% -100%) a cada pixel, um mapa de atividade (codificado por cores) é gerado (Figura 3F).
- Sobreponha os mapas resultantes com as imagens de intensidade para obter informações morfológicas adicionais. Aqui, use uma macro ImageJ (consulte Tabela de materiais) para sobrepor o matiz e a saturação dos mapas de enzimas ou atividades com o brilho das imagens de intensidade.
- Certifique-se de que a macro itere em toda a pilha. Para cada fatia (profundidade) na pilha, ela deve dividir a imagem de intensidade e o mapa de escolha, respectivamente, em HUE, SATURATION e BRIGHTNESS (tipo de > de imagem > pilha HSB).
- Separe os canais principais para ambas as pilhas HSB (pilhas de > de imagem > pilha para imagens).
- Feche o HUE e a SATURAÇÃO da pilha de intensidade, bem como o BRILHO da pilha de mapas (close(slicename_HUE), close(slicename_BRIGHTNESS), ...).
- Recombine os canais restantes para formar uma nova pilha HSB que consiste na TONALIDADE e SATURAÇÃO da fatia do mapa de interesse e no BRILHO da imagem de intensidade (a imagem > se acumula > as imagens a serem empilhadas).
- Altere o tipo de imagem para RGB para uma melhor aparência visual (Tipo de > imagem > cor RGB).
4. Segmentação de tecidos
NOTA: Use uma rede baseada em U-Net pré-treinada (ILASTIK, consulte a Tabela de Materiais) para segmentar o hospedeiro intestinal e o tecido do nematóide, respectivamente, e, além disso, o epitélio e a lâmina própria no hospedeiro e as áreas de sinal de fluorescência de alto NAD (P) H e áreas de sinal de fluorescência de baixo NAD (P) H no nematóide.
- Abra o ILASTIK e escolha um novo projeto e classificação de pixels.
- Carregue a projeção de intensidade calculada anteriormente na etapa 3.7.1 clicando em Adicionar novo > Adicionar imagens separadas. Carregue fatias aleatórias de várias medições no recipiente.
- Clique em Seleção de recursos > selecionar recursos. Adicione um valor sigma de 50 (peso sem unidade) e marque todos os recursos a serem ativos.
NOTA: Embora o valor sigma determine o peso dos recursos, os recursos determinam as classes e a capacidade da rede de reconhecer bordas, formas, textura ou cor. - Clique em Treinamento e nomeie os rótulos de acordo com o tecido a ser segmentado (por exemplo, Epitélio, Fundo, etc.) clicando neles. Selecione um rótulo e pinte todos os pixels da imagem correspondente a esse rótulo.
- Repita para os outros rótulos.
- Repita para aproximadamente metade das imagens no conjunto de dados carregado.
- Clique em Live Update e deixe o modelo prever os rótulos para as imagens não rotuladas restantes.
- Corrija aproximadamente metade das imagens apenas previstas e não rotuladas rotulando novamente as segmentações falsamente estimadas e repita a atualização ao vivo.
- Repita esta etapa até que a rede tenha aprendido a estimar corretamente o tecido de interesse desejado; Certifique-se de que a confiança da rede mostrada à esquerda perto dos nomes dos rótulos varie entre 95% e 98%.
- Carregue um novo conjunto de dados conforme descrito na etapa 3.11, desta vez com todas as fatias de uma medição, e clique em Exportação de Previsão.
- No menu suspenso "Fonte", escolha Segmentação Simples; em "Formato", escolha a saída a ser no formato tiff; e em "Escolher configurações de exportação de imagem", selecione o caminho de salvamento para a saída. Clique em exportar.
- Isso cria máscaras binárias para o tipo de tecido de interesse. Como consistem em fatias em que o tecido segmentado de interesse tem um valor de pixel de 1 e o restante tem um valor de pixel de 0 (Figura 3H), basta multiplicar as máscaras binárias pelos dados gerados, conforme listado na etapa 1.1 e nas etapas 1.4-1.7. Isso leva a dados mascarados (Figura 3 I, J, M). Use ImageJ para esta etapa (Processar > Calculadora de imagem > multiplicar).
- Para cada fatia de volume, calcule uma relação sinal-ruído (SNR) a partir das imagens de intensidade mascaradas. Use uma macro ImageJ para essa finalidade. Crie seu programa de forma que ele calcule a relação sinal-ruído (SNR) a partir das imagens segmentadas e mascaradas.
- Use o plano de fundo segmentado da etapa 3.21 como plano de fundo (BG) e o tecido segmentado de interesse como sinal (SIG) iterando sobre cada pixel de cada fatia com um valor maior que 0 com dois loops for aninhados. Calcule o SNR com a seguinte fórmula33:
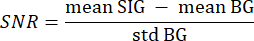
onde SIG médio refere-se ao valor médio do histograma de sinal, BG médio refere-se ao valor médio do histograma de fundo e BG padrão refere-se ao desvio padrão do histograma de fundo. - Descarte as fatias de volume com um valor de SNR inferior a 5 da análise posterior.
- Use o plano de fundo segmentado da etapa 3.21 como plano de fundo (BG) e o tecido segmentado de interesse como sinal (SIG) iterando sobre cada pixel de cada fatia com um valor maior que 0 com dois loops for aninhados. Calcule o SNR com a seguinte fórmula33:
- Com o script Python 18,33, crie um gráfico de frequência enzimática a partir dos mapas enzimáticos para cada tipo de tecido, somando e calculando a média das enzimas em fatias e, em seguida, normalizando-as para um volume de tecido de 100 μm³ (Figura 3K, N).
- Codifique o script de forma que ele carregue o mapa enzimático mascarado.
- Itere sobre cada pixel, calcule a soma dos pixels da mesma enzima e divida pelo volume analisado (o total de todos os pixels).
- Agrupe as abundâncias calculadas em seus estados metabólicos e calcule a média dividindo pelo número de enzimas em cada grupo. Escreva um código que atribua estados metabólicos agrupados em torno de LDH a vias anaeróbicas semelhantes à glicólise, atividade enzimática agrupada em torno de PDH/IDH/GAPDH a vias aeróbicas semelhantes à glicólise/oxFos e atividade NADPH oxidase (atividade NOX) a explosão oxidativa/estresse oxidativo usado para defesa (Figura 3L, O).
- Use a biblioteca matplotlib do Python para visualizar os dados com boxplots.
- Calcule a atividade do tecido a partir dos mapas de atividade mascarados (Figura 3F) como o valor médio em cada fatia de volume para todos os pixels com um valor maior que 0 usando uma macro ImageJ executando Analyze > Measure.
- Visualize todas as imagens geradas lado a lado com o ImageJ/FIJI.
Resultados
Usando o procedimento NAD (P) H-FLIMatual 28 , 29 , 33 combinado com o método de análise fasorial descrito, a atividade metabólica e as vias metabólicas em duodenos saudáveis e infectados foram medidas no dia 6, dia 10, dia 12 e dia 14 pós-infecção com o nematóide intestinal murino H. polygyrus.
Viabilidade do tecido intestinal preservado no duodeno excisado revelada por NAD(P)H-FLIM
A fim de investigar a atividade tecidual ex vivo e determinar por quanto tempo as amostras de tecido excisadas permanecem metabolicamente ativas em comparação com o tecido em organismos vivos, o NAD (P) H-FLIM foi realizado em tecido duodenal excisado de camundongos saudáveis que foi (i) recém-excisado ou (ii) mantido em gelo por 3 h, e os resultados foram comparados com dados NAD (P) H-FLIM já publicados adquiridos no intestino de camundongos anestesiados32. O preparo do duodeno excisado e o exame de imagem foram realizados conforme descrito no protocolo de preparo da amostra (etapa 1).
O tempo de vida de fluorescência do NAD (P) H foi medido durante um período de 3 h e em quatro profundidades de tecido diferentes (ponta das vilosidades: ~ 0 μm, vilosidades superiores: ~ − 50 μm, vilosidades inferiores: ~ − 100 μm, base das vilosidades: ~ − 200 μm). A partir dos dados NAD(P)H-FLIM adquiridos dessa forma, foram gerados mapas de atividade mostrando a atividade metabólica geral nas diferentes regiões das vilosidades ao longo do tempo. A atividade metabólica geral (NAD (P) H livre a ligada à enzima) é considerada um indicador da degeneração do tecido ( Figura 4A , B ).
Uma segunda série de medições com amostras de tecido semelhantes foi realizada com outro pedaço de intestino. Aqui, as medições não foram feitas diretamente após o sacrifício, mas o tecido foi colocado no gelo por 3 h após a preparação para imitar o tempo máximo de espera para amostras colocadas no gelo no experimento real. Após um tempo de aquecimento de ~ 15 min usando uma placa de aquecimento, foram realizadas imagens em diferentes profundidades de tecido ( Figura 4C, D ). A atividade metabólica medida nas duas configurações descritas anteriormente estava em boa concordância com a atividade metabólica medida por NAD (P) H-FLIM sob condições intravitais no intestino murino32.
Detecção de diferentes atividades metabólicas e enzimáticas no tecido intestinal do hospedeiro e H. polygyrus ao longo da infecção aguda usando NAD(P)H-FLIM
Ao longo da doença, desde o dia 6 após a infecção, quando as larvas do verme se desenvolvem na submucosa, irrompem no lúmen no dia 10 e se infiltram no nicho intestinal, foi observado um aumento na atividade metabólica geral dos parasitas. De um estado mais dormente no dia 6 58,0% ± 2,2%, a atividade metabólica aumentou constantemente para 73,1% ± 5,9%.
O estado inicial do parasita durante a infecção inicial é refletido em sua impressão digital metabólica; Especificamente, no dia 6, a produção de energia foi equilibrada entre as vias anaeróbica e aeróbica, indicando um avanço iminente no lúmen. Houve então uma mudança na produção de energia para um comportamento fortemente anaeróbico semelhante à glicólise, presumivelmente devido ao aumento da necessidade de energia para migrar para o lúmen (dia 10), e os parasitas mostraram uma via semelhante à fosforilação oxidativa / glicólise aeróbica para a produção de energia mais tarde (medido até o dia 14). A atividade metabólica geral do hospedeiro permaneceu comparável ao estado saudável em 74,0% ± 7,6% (dia 6 a 14), enquanto as vias metabólicas no tecido do hospedeiro foram predominantemente fosforilação aeróbica / oxidativa durante o curso da infecção aguda.
A reação de defesa dos parasitas é baseada na ativação do DUOX2, como o único membro da família NADPH oxidase nesta espécie. Essa reação é insignificante no estágio inicial da infecção, no dia 6, mas aumenta em momentos posteriores durante a infecção aguda Portanto, detectamos um aumento drástico na ativação do DUOX2 a partir do dia 10, após a transmigração luminal e durante toda a fase aguda da infecção. (Figura 5E, F)
O comportamento inflamatório do hospedeiro, por outro lado, aumentou drasticamente em comparação com um estado saudável no dia 10 após a infecção, com o aparecimento do parasita no lúmen, e cessou mais tarde no dia 14 após a infecção. Esse comportamento é paralelo ao fato de que os parasitas induzem um fenótipo regulatório no sistema imunológico do hospedeiro nos estágios posteriores (crônicos) da infecção 4,6,7 (Figura 5A-D).

Figura 1: Preparação do tecido intestinal murino para NAD (P) H-FLIM com o microscópio de varredura a laser multifotônico. (A) A preparação requer a remoção do pedaço de intestino e o corte de cerca de 1 cm de comprimento (1), espalhando cola de tecido em uma placa de Petri e colocando o tubo sobre ela (2), cortando o tubo longitudinalmente com uma tesoura romba perto do fundo (3), desdobrar o tubo para abrir o lado luminal com uma pinça romba (4), contar os vermes em um estereomicroscópio (5), selar o tecido com agarose de baixa concentração em uma camada fina de cerca de 0,5 mm de espessura (6), encher o prato com 10% de PBS (7) e obter imagens da amostra de tecido ou colocá-la em gelo para aguardar o processamento (8). (B) Eixo de tempo para processar uma única amostra de tecido em minutos, desde a remoção do duodeno até a colocação ao microscópio. O tempo de medição de uma amostra envolvia encontrar um ROI, aquecer as amostras resfriadas em um elemento de aquecimento a 37 °C nesse meio tempo e, em seguida, adquirir as imagens. (C) O tempo médio de medição de 45 min (determinado posteriormente a partir dos dados) é plotado em (C) em relação aos quatro estágios de infecção investigados, bem como ao tecido saudável. Clique aqui para ver uma versão maior desta figura.

Figura 2: Esquema da configuração experimental. O sistema consiste em um laser ajustável Ti:Sa titânio:safira LASER Chameleon ULTRA II (690-1.080 nm, 80 MHz, largura de pulso de 140 fs) com um cabeçote de varredura comercial Trimscope II. Uma lente objetiva de imersão em água (20x, NA 1.05) focaliza a luz de excitação. Um sistema de espelhos dicróicos, filtros de interferência (525/50 nm, 593/40 nm, 655/40 nm) e PMTs detecta o sinal de fluorescência. Os dados de vida útil da fluorescência são detectados usando um PMT híbrido (GaAsP) (hPMT) no canal 466/60 nm, que é resfriado por um módulo de resfriamento (CM) e é conectado a um módulo de contagem de fótons único correlacionado ao tempo; esse processo envolve a detecção de fótons dentro de um compartimento de 55 ps e a aquisição de dados em 9 ns com uma função de resposta de instrumento em forma de gaussiana de 250 ps FWHM (TCSPC). Para acionar o TCSPC, 5% -8% da intensidade do pulso inicial é separada do feixe de excitação e é redirecionada em um fotodiodo ultrarrápido (PD). Para obter a excitação de dois fótons em modo de tempo real sem o TCSPC, são usados PMTs com filtros de cores (525 nm, 593 nm, 655 nm). Uma lâmpada de vapor de mercúrio (MV) de banda larga e uma via ocular são usadas para a observação da sonda (OP) para microscopia de fluorescência visual e posicionamento da amostra, e a ROI é encontrada com um estágio intravital (IS) com um tamanho de passo de 1 μm (IS). Clique aqui para ver uma versão maior desta figura.
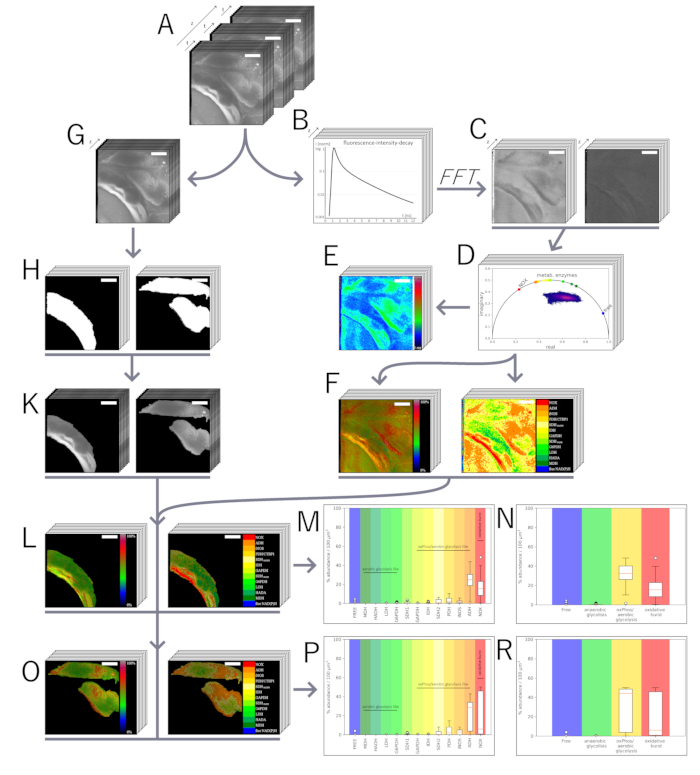
Figura 3: Análise fasorial dos dados NAD (P) H-FLIM no domínio do tempo. (A) Os dados brutos são adquiridos em quatro dimensões: a informação espacial (x, y, z; 505 pixels = 500 μm para xy) e os dados TCSPC (227 pontos de tempo de 55 ps cada para t). (B) Cada pixel nos dados brutos do TCSPC ao longo de t, conforme mostrado em (A), contém o decaimento da intensidade de fluorescência após um pulso de excitação em 227 caixas de tempo de 55 ps cada. Os dados TCSPC no domínio do tempo são pré-complicados gaussianos e transferidos para um domínio de fase virtual calculando a transformada de Fourier normalizada e discreta numérica (abordagem fasorial). Isso resulta em um número complexo de cada pixel na imagem, contendo uma parte real e imaginária, e isso pode ser dividido e representado como duas imagens codificadas por intensidade por fatia medida: (C, esquerda) a parte real e (C, direita) a parte imaginária. (D) As partes real e imaginária fornecem as coordenadas no gráfico fasorial, onde cada ponto no gráfico tem uma contraparte espacial e suas informações de decaimento de (B). O eixo de tempo semicircular indica tempos de vida mono-exponenciais. (E) Os tempos de vida de fluorescência são calculados e codificados por cores a partir de (C) usando a transformação contínua de Fourier (de volta). Isso é equivalente a uma análise mono-exponencial da curva de decaimento de intensidade em (B). Seguindo uma estrutura de análise desenvolvida anteriormente, (F, à direita) um mapa de enzimas dependentes de NAD(P)H e (F, à esquerda) um mapa da atividade enzimática são gerados. (G) Paralelamente, os dados brutos do TCSPC são recolhidos na projeção de intensidade, perdendo as informações temporais e preservando apenas as informações espaciais. (H) A partir disso, as máscaras são geradas usando ILASTIK para o tecido de interesse desejado por algoritmos de segmentação treinados, que são sobrepostos com a projeção de intensidade; um exemplo é mostrado aqui para (I, esquerda) parasita e (I, direita) hospedeiro no dia 14 pós-infecção. (J, à esquerda) O mapa de atividade mascarado e o mapa enzimático mascarado de (J, à direita) do parasita e de (M) do tecido hospedeiro. A partir dos mapas mascarados, gráficos de frequência enzimática para o parasita são gerados contando as enzimas e normalizando a porcentagem de abundância para 100 μm3 (K). Após a análise de Liublin et al.33, as abundâncias enzimáticas foram agrupadas de acordo com as vias metabólicas de glicólise aeróbia e fosforilação oxidativa/glicólise anaeróbia, bem como explosão oxidativa e um estado metabolicamente inativo (NAD(P)H livre e não ligado) para (L) o parasita e para (N,O) o tecido hospedeiro. Todas as barras de escala são de 100 μm. Clique aqui para ver uma versão maior desta figura.

Figura 4: Pré-avaliação da vitalidade do tecido explantado. (A) Mapa da atividade enzimática do duodeno recém-explantado de um camundongo C57 / bl6 saudável, conforme descrito na seção 3. O tecido foi fotografado diretamente após a preparação em intervalos de 20 minutos por um tempo de 106 minutos no total em uma placa de aquecimento a 37 ° C. As medições foram realizadas em quatro profundidades de tecido diferentes: as pontas das vilosidades (0 μm), o meio superior (−50 μm), o meio inferior (−100 μm) e a base das vilosidades (−200 μm). Os exemplos mostrados em (A) são a região média superior das vilosidades 5 min e 68 min após o preparo. Para cada ponto de medição, a atividade enzimática média em todas as profundidades foi calculada a partir dos mapas de atividade segmentada da lâmina própria e do tecido epitelial e plotada ao longo do tempo de medição. (B) O gráfico mostra o comportamento de vitalidade e degeneração do tecido explantado após sacrifício e preparo ao longo de 106 min. Para cobrir o comportamento de degeneração tecidual das amostras preparadas que foram colocadas em gelo para enfileirar para medição, conforme descrito na seção 1, o duodeno recém-explantado de um camundongo C57 / bl6 saudável foi preparado e armazenado em gelo por 3 h para imitar o tempo máximo de espera que ocorreu durante os experimentos. (C) O tecido foi então aquecido até 37 ° C e fotografado com NAD (P) H-FLIM, e um mapa de atividade foi gerado. As atividades médias correspondentes do tecido foram calculadas a partir dos dados segmentados descritos para (B) e (D), o que resultou em uma atividade de 57,8% ± 4,6% para a lâmina própria e 68,5% ± 5,7% para o epitélio). Todas as barras de escala são de 250 μm. Clique aqui para ver uma versão maior desta figura.

Figura 5: NAD (P) H-FLIM das vilosidades do hospedeiro e tecido nematóide indicando o estado metabólico e as vias metabólicas preferenciais no hospedeiro e no parasita. (A) A atividade metabólica geral média determinada a partir dos mapas de atividade mascarada para o epitélio (EP) (78,4% ± 5,5%) e lâmina própria (LP) (76,3% ± 5,2%) no duodeno murino saudável. (B) Seguindo nossa análise, as abundâncias enzimáticas foram agrupadas de acordo com as vias metabólicas, incluindo glicólise aeróbica, fosforilação oxidativa/glicólise anaeróbia e explosão oxidativa para o epitélio (EP) e lâmina própria (LP) no duodeno murino saudável. (C, D) Semelhante a (A) e (B), foi realizada uma análise para o tecido hospedeiro adquirido ao longo da infecção aguda. A atividade metabólica média do tecido hospedeiro no dia 6 foi determinada em 74,0% ± 7,6%, no dia 10 foi de 72,4% ± 10,5% e no dia 14 foi de 73,5% ± 11,4%. (E, F) Como em (C) e (D), o tecido do parasita foi analisado ao longo da fase aguda da doença. A atividade metabólica média do parasito no dia 6 foi de (58,0% ± 2%, no dia 10 foi de 61,1% ± 9,9%, no dia 12 foi de 64,5% ± 5,8% e no dia 14 foi de 73,1% ± 5,9%. A análise estatística foi realizada por meio de um teste ANOVA (ns p > 0,05, *p < 0,05, **p < 0,01, *** p < 0,001). Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Vida útil de fluorescência dependente do estado de ligação NAD(P)H. Representação esquemática de (A I) NAD (livre) não ligado (livre) H após excitação por absorção de fótons no ponto de tempo 0 na linha do tempo e seu tempo de vida de fluorescência específico do estado de ligação (τ) a 450 ps. O mesmo processo é mostrado para (A II) NADH ligado a LDH e (A III) PDH, com tempos de vida de fluorescência a 1.600 ps e 2.470 ps, respectivamente, e NADPH ligado a NADPH oxidases (NOX) (A IV) com seu tempo de vida de fluorescência em 3.650 ps. (B) Tabela dos tempos de vida de fluorescência caracterizados de NAD (P) H ligados às enzimas mais abundantes. Clique aqui para baixar este arquivo.
Discussão
As etapas críticas dentro do protocolo ocorrem durante a preparação e ao encontrar o ROI. As fibras de alimentos parcialmente digeridos representam um desafio para a obtenção de imagens, principalmente devido à luminescência endógena das fibras que se sobrepõem à fluorescência do NAD(P)H, mas também devido ao seu sinal de geração harmônica. É de grande importância encontrar ROIs livres de fezes. Nosso objetivo foi evitar a medição de áreas contendo fezes. A lavagem foi evitada porque isso afeta a integridade das vilosidades frágeis e influencia a viscosidade do muco. Além disso, todo o procedimento de preparação deve durar preferencialmente cerca de 5 a 10 minutos por amostra, para evitar a degeneração e o ressecamento do tecido. Além disso, a agarose de baixa concentração usada imita a viscosidade do muco intestinal34. Assim, pode-se garantir que as vilosidades não colapsem enquanto os vermes se movem livremente durante a medição. A medição não deve durar mais de 45 minutos (Figura 1A,B e Figura 1A,B,C) para preservar as melhores condições para a obtenção de imagens do metabolismo celular. Portanto, a pesquisa da região de interesse (ROI) não deve demorar mais de 15 minutos. No entanto, para garantir que o tecido mantido no gelo aqueça até os 37 °C desejados na placa de aquecimento, a imagem deve ser realizada após ~10 min. A imagem normalmente dura 45 minutos (consulte a análise de dados TCSPC para controlar a viabilidade do tecido antes e durante a medição; Figura 1C e Figura 4). Ao procurar um ROI adequado, certifique-se de que pelo menos 20% do lado luminal do tecido duodenal (dias 10-14 após a infecção) sob a lente objetiva esteja coberto por tecido de verme e que o movimento do parasita seja visível. Para indivíduos saudáveis e do dia 6, um ROI adequado é definido como aquele em que a integridade do tecido é mantida após a preparação (normalmente, longe das bordas da amostra). Isso garante as melhores condições de treinamento para o modelo de segmentação e fornece pontos de dados suficientes para análise posterior. Para os estágios de infecção a partir do dia 10, quando os vermes invadem o lúmen e se fixam nas vilosidades, os dados são segmentados apenas de acordo com o background e o tecido parasitário e intestinal com alta atividade enzimática, uma vez que na presença dos vermes, as vilosidades são em sua maioria desestruturadas devido à nidificação, e a integridade tecidual das vilosidades não está mais presente. Para o tecido explantado no dia 6 e de camundongos saudáveis, a integridade do tecido permite a segmentação da lâmina própria, bem como do epitélio em relação ao fundo. Assim, o uso de modelos treinados separadamente para as condições é altamente recomendado. O sinal sobrecarrega os detectores em busca de material altamente autofluorescente, ocasionalmente acumulando-se dentro do volume medido. Neste trabalho, um controle dinâmico de potência do laser foi usado em toda a profundidade de imagem no software.
As limitações desta técnica estão relacionadas às limitações físicas do volume de excitação. A resolução axial da lente objetiva foi previamente determinada em 1,3 μm a 850 nm28, mas como normalmente se degrada no tecido, descobrimos que um tamanho de passo de 2 μm é apropriado. Com um FOV de 0,5 mm x 0,5 mm, cada fatia tem 505 pixels x 505 pixels. A resolução lateral da objetiva é de ~ 350 nm; No entanto, devido à degradação da resolução24,28, um tamanho de pixel de ~ 1 μm foi considerado suficiente. Isso significa que a medição segue uma abordagem estocástica. A curva de decaimento adquirida é composta pela soma de todas as curvas de decaimento, ou seja, todos os tempos de vida de fluorescência dentro do volume de saída de ~ 1 x 1 x 2 μm3. A contagem de fótons registrada ao longo do tempo para cada voxel (o tempo de vida de fluorescência medido ou pilha TCSPC) segue a forma de uma curva de decaimento multi-exponencial ou a combinação linear de curvas de decaimento multi-exponencial, o que é difícil de prever. Uma análise normalizada baseada em Fourier sem modelo foi usada para analisar a complexa curva de decaimento de fluorescência em cada voxel, denominada análise fasorial. As pilhas foram convolvidas antecipadamente com um filtro gaussiano (σ2) para reduzir o ruído nas imagens de fluorescência resolvidas no tempo. A perda de resolução espacial é aceitável e é uma compensação para uma melhor resolução de tempo de vida de fluorescência.
O tempo de aquisição de quadros está na faixa de 2,5 s a 7 s (média), e o tempo de aquisição para uma pilha z típica de 500 x 500 x 200 μm³ (505 x 505 x 101 voxel) leva ~ 450 s (7,5 min). Esses tempos geralmente resultam em artefatos móveis causados pelos parasitas durante uma medição. Os dados permanecem utilizáveis neste caso. Ocasionalmente, os parasitas se movem para fora do volume medido durante a medição.
Usando imagens de tempo de vida de fluorescência de NADH e NADPH de tecido duodenal murino excisado de camundongos infectados com o nematóide H. polygyrus e nossa estrutura de análise publicada anteriormente usando os tempos de vida de fluorescência de referência de NAD (P) H ligado a enzimas dependentes de NAD (P) H frequentes, a atividade metabólica geral e as vias metabólicas distintas no tecido intestinal do hospedeiro e no tecido do parasita foram visualizadas e analisadas; Esta análise revelou a interferência metabólica entre os dois. Alternativamente, se as informações sobre a atividade metabólica geral ou sobre o equilíbrio entre NADH e NADPH forem de interesse, algoritmos robustos e rotinas de software geralmente aplicáveis estão disponíveis e podem ser usados22,23.
O protocolo aqui apresentado e o procedimento de análise estabelecido representam um método generalizado para medir o metabolismo e as vias metabólicas que é aplicável a outros órgãos e outros tipos de patologias 13,15,16,17,19,20.
Comparado, por exemplo, com a análise de célula única com citometria, a elegância do método reside na possibilidade de obter informações espaciais subcelulares sobre vias metabólicas com intervenção mínima e de forma livre de marcação em sistemas biológicos, com a possibilidade do uso adicional de marcadores ou corantes para uma maior densidade de informação. A interferência com o tecido biológico e os processos de infecção a ele ligados são mínimos devido à escolha de um parasita que ocorre naturalmente em roedores, bem como ao exame preliminar do tecido intestinal e sua comparabilidade com o tecido vivo em termos de vitalidade. Com segmentação e normalização apropriadas, medições comparáveis podem ser geradas na quarta dimensão (tempo).
Divulgações
Os autores declaram não haver interesses financeiros concorrentes.
Agradecimentos
Agradecemos a Robert Günther por seu excelente suporte técnico. Apoio financeiro do Conselho Alemão de Pesquisa (DFG) sob a concessão SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.) e RA2544/1-1 (S.R.), sob a concessão SFB1444, P14 (R.A.N., A.E.H.), sob a concessão HA5354/8-2 (A.E.H.), e sob a concessão GRK2046 B4 e B5 (S.H., S.R.) e HA2542 / 8-1 (SH) são muito reconhecidos. W.L. recebeu uma bolsa de doutorado da Berliner Hochschule für Technik, Escola de Ciências Aplicadas de Berlim em Física Médica/Engenharia Física.
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
Referências
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados