Method Article
NAD(P)H Fluoreszenz-Lebensdauer-Bildgebung für die metabolische Analyse des murinen Darms und von Parasiten während einer Nematodeninfektion
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt die NAD(P)H-Fluoreszenzlebensdauer-Bildgebung eines explantierten Mausdarms, der mit dem natürlichen Parasiten Heligmosomoides polygyrus infiziert ist, was es ermöglicht, Stoffwechselprozesse sowohl im Wirts- als auch im Parasitengewebe ortsaufgelöst zu untersuchen.
Zusammenfassung
Parasiten wirken sich in der Regel negativ auf die Gesundheit ihres Wirts aus. Sie stellen eine enorme gesundheitliche Belastung dar, da sie global langfristig die Gesundheit des befallenen Menschen oder Tieres beeinträchtigen und somit die landwirtschaftlichen und sozioökonomischen Ergebnisse beeinflussen. Es wurden jedoch auch parasitengetriebene immunregulatorische Effekte beschrieben, die bei Autoimmunerkrankungen therapeutisch relevant sein könnten. Während der Stoffwechsel sowohl im Wirt als auch in den Parasiten zu deren Abwehr beiträgt und die Grundlage für das Überleben von Nematoden im Darm ist, ist er aufgrund eines Mangels an geeigneten Technologien weitgehend unerforscht geblieben. Wir haben die NAD(P)H-Fluoreszenz-Lebensdauer-Bildgebung entwickelt und auf explantiertes murines Darmgewebe während einer Infektion mit dem natürlichen Fadenwurm Heligmosomoides polygyrus angewendet, um die Stoffwechselprozesse sowohl im Wirt als auch in den Parasiten räumlich aufgelöst zu untersuchen. Die Ausnutzung der Fluoreszenzlebensdauer der Co-Enzyme Nicotinamidadenindinukleotid (NADH) und Nicotinamidadenindinukleotidphosphat (NADPH), im Folgenden NAD(P)H, die artenübergreifend erhalten sind, hängt von ihrem Bindungsstatus und der Bindungsstelle an den Enzymen ab, die Stoffwechselprozesse katalysieren. Unter Berücksichtigung der am häufigsten exprimierten NAD(P)H-abhängigen Enzyme wurden die Stoffwechselwege unterschieden, die mit der anaeroben Glykolyse, der oxidativen Phosphorylierung/aeroben Glykolyse und dem NOX-basierten oxidativen Burst als wichtiger Abwehrmechanismus verbunden sind, und die metabolische Wechselwirkung zwischen dem Wirt und dem Parasiten während der Infektion wurde charakterisiert.
Einleitung
Parasitäre Infektionen stellen eine enorme Belastung für die menschliche Gesundheit dar 1,2. In den Industrieländern wurde ein Zusammenhang zwischen dem Anstieg von Autoimmunerkrankungen und dem Rückgang parasitärer Infektionen beobachtet. Es ist bekannt, dass Parasiten positive Auswirkungen haben können, indem sie übermäßige Immunreaktionen des Wirts dämpfen. H. polygyrus ist ein natürlicher Parasit, der im Darm von Nagetieren vorkommt, und dieser Parasit ist dafür bekannt, immunregulatorische Mechanismen zu induzieren, die die antiparasitäre Immunantwort des Wirts reduzieren, unter anderem durch die Induktion von regulatorischen T-Zellen (Treg) im infizierten Wirt 3,4,5,6,7,8,9,10,11. Diese Regulationsmechanismen sind vor allem bei degenerativen Autoimmunerkrankungen von Interesse.
Die Analyse der metabolischen Wechselwirkung zwischen dem Wirt und den Darmnematoden wird nach wie vor weitgehend vernachlässigt, obwohl der Stoffwechsel sowohl im Wirt als auch im Parasiten eine wichtige Rolle für die Abwehr, das Überleben und die Funktion spielt. Wir schlagen vor, die NADH- und NADPH-Fluoreszenzlebensdauer-Bildgebung nach Zwei-Photonen-Anregung anzupassen und anzuwenden, eine Technologie, die bereits in verschiedenen physiologischen und pathophysiologischen Situationen in Säugetierzellen und -geweben weit verbreitet ist12, um den Wirts- und Nematodenstoffwechsel in lebenden Geweben korrelativ zu untersuchen.
NADH und NADPH, auch als NAD(P)H bezeichnet, sind ubiquitäre Moleküle, die in allen zellbasierten Lebensformen erhalten sind und die Rolle von Co-Enzymen in verschiedenen Stoffwechselwegen spielen. Zum Beispiel sind sie an der Energieproduktion, der reduktiven Biosynthese und der NADPH-Oxidase-vermittelten Produktion von reaktiven Sauerstoffspezies (ROS) beteiligt, die hauptsächlich mit der Zellabwehr und Zellkommunikation verbunden sind 13,14,15,16,17,20. Beide Co-Enzyme emittieren Fluoreszenz bei ~450 nm bei Zwei-Photonen-Anregung bei 750 nm und ermöglichen so eine markerfreie metabolische Bildgebung in Zellen und Geweben19,21. Die Anregung sowohl von NADH als auch von NADPH mit nur einer Wellenlänge ist aufgrund ihrer ähnlichen und recht breiten Zwei-Photonen-Anregungsspektrenmöglich 21.
Die Fluoreszenzlebensdauer des Co-Enzyms NAD(P)H hängt direkt von dem Enzym ab, an das es bindet 18,21,22,23. Aufgrund seiner chemischen Struktur, die einen intramolekularen Energietransfer ermöglicht, verliert das angeregte NADH- oder NADPH-Molekül durch interne Umwandlungsprozesse Energie an die Enzyme (Katalysator), bevor es sich entspannt und ein Fluoreszenzphoton emittiert. Diese Lebensdauer gibt Einblick in die NAD(P)H-Bindungsstelle am Enzym und damit in die bevorzugte biochemische Reaktion, die stattfindet 19,21,22,23,24,25. Die Fluoreszenzlebensdauer von freien NADH- und NADPH-Molekülen beträgt ~450 ps, während ihre Fluoreszenzlebensdauer bei Bindung an ein Enzym viel länger ist (~2.000 ps) und von ihrer Bindungsstelle auf dem jeweiligen Enzymabhängt 21.
Es gibt mehr als 370 Enzyme, die an NAD(P)H-verknüpften Prozessen beteiligt sind; Allerdings können nur die am häufigsten vorkommenden innerhalb des Anregungsbereichs des Mikroskops zur resultierenden NAD(P)H-Fluoreszenzlebensdauer beitragen. Unter Verwendung von RNASeq-Daten aus Säugetierzellen identifizierten wir die am häufigsten vorkommenden NAD(P)H-abhängigen Enzyme und generierten eine Referenz für die Fluoreszenzlebensdauer, um die in Gewebe- und Zellproben generierten Daten zu interpretieren18. Dabei wurde z.B. unterschieden zwischen der präferenziellen Aktivität der Laktatdehydrogenase (LDH), die mit anaeroben glykolytischen Stoffwechselwegen assoziiert ist, und der Aktivität der Isocitratdehydrogenase (IDH) und der Pyruvatdehydrogenase (PDH), die hauptsächlich an den Stoffwechselwegen der aeroben Glykolyse/oxidativen Phosphorylierung beteiligt sind16,20. Darüber hinaus kann die Bindung von NADPH an NADPH-Oxidasen, die Enzyme, die hauptsächlich für den oxidativen Burst verantwortlich sind, aufgrund der charakteristischen Position dieser Enzyme in der Zelle (membrangebunden) und aufgrund der besonders langen NADPH-Fluoreszenzlebensdauer (3.650 ps) leicht aufgelöst werden18,24,29,30,32. RNASeq-Daten von H. polygyrus zeigen, dass die für Säugetierzellen generierte Referenz in angepasster Form auch auf diesen Fadenwurm zutrifft27.
Daher wurde in dieser Arbeit durch die Durchführung von NAD(P)H-Fluoreszenz-Lebensdauer-Bildgebung (FLIM) in frisch explantierten Zwölffingerdarmproben von Mäusen, die mit H. polygyrus infiziert waren, Informationen über das Verhältnis zwischen freiem und enzymgebundenem NAD(P)H gewonnen, die die allgemeine metabolische Aktivität in allen Geweben sowie das überwiegend aktive Enzym in jedem Pixel des Bildes abbildeten (d.h. das Enzym, an das NAD(P)H bevorzugt an dieser spezifischen Stelle bindet). Der Erfolg dieser Experimente hängt von der genauen Probenvorbereitung des explantierten Darms, der zuverlässigen Live-Bildgebung der NAD(P)H-Fluoreszenzlebensdauer bei subzellulärer Auflösung und der standardisierten Datenauswertung ab, wie in diesem Protokoll beschrieben.
Protokoll
Alle Versuche wurden in Übereinstimmung mit den Nationalen Tierschutzrichtlinien durchgeführt und von der Deutschen Tierethikkommission zum Schutz von Tieren (G0176/16 und G0207/19) genehmigt. Das Protokoll beschreibt die Erfassung und Datenauswertung der NAD(P)H-Fluoreszenzlebensdauer-Bildgebung, die es ermöglicht, die allgemeine Stoffwechselaktivität und die spezifischen Stoffwechselwege sowohl im Wirtsdarm als auch bei den Parasiten nach einer Infektion mit dem natürlichen murinen Darmnematoden, H. polygyrus, zu beurteilen. Zu diesem Zweck wurden weibliche C57BL/6-Mäuse im Alter von 10-12 Wochen mit 200 Larven des Stadiums 3 (L3) infiziert. Zu verschiedenen Zeitpunkten der Infektion wurden die infizierten Mäuse getötet, und die Zwölffingerdarme wurden herausgeschnitten und für die Bildgebung vorbereitet, wie zuvor beschrieben33. Die Zwölffingerdarme von nicht infizierten, alters- und geschlechtsgleichen Mäusen wurden zu Kontrollzwecken auf ähnliche Weise präpariert und abgebildet. Um die für die weitere Bildgebung und Analyse erforderlichen Gewebeeigenschaften zu erhalten, müssen die Proben unmittelbar nach der Explantation verarbeitet und die nächsten Schritte (Schritte 1.1-1.7) zügig durchgeführt werden (Abbildung 1B).
1. Vorbereitung der Probe
- Schneiden Sie Stücke von ~1 cm Länge aus dem ursprünglich herausgeschnittenen Zwölffingerdarm ab.
- Kleben Sie das begradigte Geweberöhrchen in der Mitte einer kleinen Petrischale mit schnell aushärtendem Gewebekleber (47 mm Durchmesser, 10 mm Randhöhe, siehe Materialtabelle) (Abbildung 1A2).
- Tragen Sie mit einem Mikropinsel eine dünne Schicht zusätzlichen Kleber auf den Boden der Petrischale über eine große Fläche um das Gewebe herum auf (Abbildung 1A2).
- Verwenden Sie eine stumpfe Schere, um einen Schnitt über die gesamte Länge des physisch fixierten Darms in der Nähe des Bodens der Petrischale zu machen (Abbildung 1A3).
- Entfalten Sie den Darm mit einer stumpfen Pinzette, so dass die Bauchseite vollständig mit dem Kleber in Berührung kommt. Somit liegt der Darm mit der luminalen Seite nach oben fixiert durch den Kleber (Abbildung 1A4).
- Im Falle einer infizierten Maus zählen Sie die Würmer unter einem Stereomikroskop (10-fache Vergrößerung), um sicherzustellen, dass die Infektion erfolgreich war (Abbildung 1A5).
- Versiegeln Sie den Darm mit Agarose, um ihn vor dem Austrocknen zu schützen. Hier wurden 0,5%-0,9% Agarose verwendet, um das empfindliche Darmgewebe aufgrund seines niedrigen Schmelzpunktes von ca. 38 °C vor Verbrennungen zu schützen. Ziehen Sie 1 ml Agarose in eine Pipette und träufeln Sie sie vorsichtig auf den Darm, wobei Sie einen sanften Kontakt zwischen der Pipettenspitze und dem Gewebe haben, so dass eine dünne Schicht von etwa 0,5 mm Dicke das Gewebe vollständig umschließt (Abbildung 1A6).
- Füllen Sie die Petrischalen mit den in Agarose mit PBS (10%) bei Raumtemperatur versiegelten Proben und verschließen Sie sie dann mit dem Deckel. Bilden Sie die Proben entweder direkt ab oder legen Sie sie auf Eis in eine wärmeisolierende Box, um sie für die Messung in die Warteschlange zu stellen. Legen Sie die erste vorbereitete Probe unter das Mikroskopobjektiv auf eine auf 37 °C eingestellte Heizplatte (Abbildung 1A8).
2. Bildgebung
HINWEIS: Das Mikroskopsystem zur Durchführung von NAD(P)H-FLIM in infizierten und gesunden Zwölffingerdarmgewebeproben besteht aus den in Abbildung 2 und der Materialtabelle aufgeführten und beschriebenen Geräten. Verwenden Sie ImSpector 208 als Steuerungssoftware für alle verwendeten Module.
- Um zunächst den Region of Interest (ROI) zu finden, platzieren Sie die Probe in der Petrischale unter dem Objektiv und bewegen Sie sich von Hand entlang der x- und y-Ebene, um durch visuelle Inspektion mit dem Weitfeld-Fluoreszenzmikroskopiemodus einen geeigneten ROI zu finden.
- Schalten Sie das Bildgebungssystem auf Zwei-Photonen-Anregung um, indem Sie entweder die PMT-Detektion oder die zeitkorrelierte Einzelphotonenzählung (TCSPC) verwenden, indem Sie die Modi in der Software umschalten. Achten Sie besonders darauf, dass die Umgebung dunkel und vibrationsfrei ist, das Mikroskop mit lichtdichten Vorhängen einzuschließen und wenn möglich einen pneumatisch hängenden optischen Tisch zu verwenden.
- Tauchen Sie die Objektivlinse mit der Steuerungssoftware in die Petrischale ein, in der sich die Probe befindet, indem Sie auf das Symbol des Linsensystems klicken und das Mausrad drehen, um die Z-Position zu ändern.
- Stellen Sie den Laser auf 765 nm ein und stellen Sie die maximale Laserleistung auf 10 % ein, was 30 mW unter dem Objektiv entspricht, indem Sie die gewünschte Wellenlänge in das Wellenlängenfeld der Software eingeben. Passen Sie die Laserleistung bei Bedarf an. Messen Sie unter einer Verstärkung von 40 % (~30 mW bis 100 mW), um Lichtschäden am Gewebe und unerwünschte Stoffwechselverschiebungen zu vermeiden.
- Stellen Sie in der Software die Schrittweite des z-Tisches auf 2 μm ein, was der axialen Auflösung des Mikroskops bei 765 nm im Gewebe entspricht.
- Stellen Sie die Zeilenabtastfrequenz in der Galvo-Spiegelblende auf 400 Hz ein, was zu einer Pixelverweilzeit von 4,95 μs führt.
- Stellen Sie den Bildmittelwert auf zwei bis vier Bilder ein, indem Sie die Mittelungszahl aus dem Dropdown-Menü in der Software auswählen (d. h. scannen Sie den ROI zwei- bis viermal, um glattere Bilder zu erhalten). Auf diese Weise wird die Gesamtverweilzeit des Pixels auf 9,9-19,8 μs erhöht, was einem höheren Signal-Rausch-Verhältnis (SNR) im Analyseprozess zugute kommt.
- Stellen Sie das Bildsichtfeld (FOV) auf 505 Pixel x 505 Pixel (500 x 500 μm²) ein, indem Sie die Parameter im FOV-Panel in der Software auswählen.
- Für jede Messung kalibrieren Sie das Modul rechtzeitig im Voraus, um die optimale Funktion der TCSPC-Elektronik zu gewährleisten, indem Sie im Kalibrierfenster im Hardware-Menü des TCSPC auf Initialisieren klicken.
- Stellen Sie die schnelle Fotodiode extern ein, um die Ti:Sa-Laserpulsfolge mit einem schnellen Oszilloskop zu erfassen.
HINWEIS: Das Signal der Fotodiode wird verwendet, um die hPMT-Detektion und die Photonenzählung durch den TCSPC in Bezug auf den Anregungsimpuls auszulösen. Die Detektion wird alle 12,5 ns (d.h. die Zeit zwischen zwei aufeinanderfolgenden Laserpulsen) wiederholt, was der Wiederholrate des Ti:Sa-Lasers (80 MHz) entspricht. - Definieren Sie den Bereich der Messtiefe, indem Sie zuerst die Mittelung auf 1 und die Verstärkung der Laserleistung auf ~10% einstellen, dann den Videomodus starten (klicken Sie auf die Video-Schaltfläche), nach der Tiefe suchen, aus der das Signal noch erfasst werden kann, indem Sie erneut auf das Objektivsystemsymbol klicken, und mit dem Mausrad in die Probe vordringen, während Sie die Verstärkung schrittweise erhöhen.
- Für die NAD(P)H-FLIM-Bilder der im FLIM-Modus gemessenen Gewebeproben stellen Sie das System auf 765 nm bei einer maximalen Nennleistung von 100 mW ein. Detektieren Sie das Fluoreszenzsignal von NAD(P)H (466/60 nm) mit dem Hybrid-PMT bei einer Verstärkung von 97%, die in der Software eingestellt ist. Das TCSPC-Modul zählt Photonen über 227 Zeitfenster (Bins) mit jeweils 55 ps.
- Klicken Sie mit der rechten Maustaste auf das angezeigte Bild während der Messung in der Software und wählen Sie Anzeige > T-PROFIL , um die vom System erfasste Abklingkurve zu visualisieren.
- Notieren Sie sich die Anzahl der Bins vor dem Laserpuls in der Abklingkurve für eine spätere Analyse.
- Erfassen Sie die Daten, indem Sie auf Messung starten klicken.
HINWEIS: Die Daten müssen einem bestimmten Format entsprechen. Jede Schicht, die in beliebiger Gewebetiefe aufgezeichnet wird, ist durch einen Abstand von 2 μm voneinander getrennt und hat eine Fläche von 500 μm x 500 μm, und die Schichten werden als Stapel von 500 μm x 500 μm Schichten zu Zeitpunkten von 227 x 55 ps (Abbildung 3A) erfasst. Jedes Voxel enthält ein räumlich aufgelöstes (in der x-Ebene und in der y-Ebene) Histogramm der Photonenankunftszeit. Dies stellt den Fluoreszenzzerfall dar (Abbildung 3B). Ein typischerweise gemessenes Volumen muss in Form eines Hyper-Stacks mit Abmessungen von ~500 μm x 500 μm x 100-300 μm und 227 Bins vorliegen, wobei jede Schicht in der Z-Ebene die zeitabhängigen Intensitätsdaten enthält, wie oben beschrieben. Bei einem beispielhaften Satz von Messdaten ergibt sich daraus (505 x 505 x 227) x 100 Pixel (16 Bit) und entspricht etwa 4 GB.
3. Datenanalyse
HINWEIS: Für die Phasoranalyse der NAD(P)H-FLIM-Bilder ist das Programm zur Berechnung der Lebensdauern ein eigens geschriebener Code in Python33.
- Verwenden Sie Anaconda mit einer Python 3.7-Distribution auf der Spyder-IDE (siehe Materialtabelle). Der Code verwendet die Standardbibliotheken von Python.
- Laden Sie den Code in die IDE, und führen Sie ihn aus. Ein Dialogfeld für den Dateipfad wird geöffnet.
- Wählen Sie den Ordner mit den zu analysierenden Rohdaten aus. Der Code ist so programmiert, dass er vor der Analyse drei Parameter über einen Tkinter-Benutzereingabedialog mit Kontrollkästchen, einem Dropdown-Menü und Textfeldern auswählt.
- Wählen Sie im Eingabedialog einen Versatz aus. Bestimmen Sie den Offset als die Anzahl der ersten Slices im Zeitstapel, die nicht analysiert werden.
HINWEIS: Der Parameter schneidet die Zeitpunkte vor dem Anregungsimpuls ab. Bei der Kalibrierung des Systems und der Triggerdiode muss dieser Wert ungefähr fünf Schichten oder die Anzahl der toten Bins vor der Laseranregung betragen, wie in Schritt 2.14 beschrieben. - Wählen Sie die Darstellung der Phasordiagramme im Dropdown-Menü des Eingabedialogfelds aus. Wählen Sie hier das Aussehen der Datenpunkte (wolkenartig oder topografisch) sowie die Achsenticks der halbkreisförmigen Zeitachse (Enzyme oder Zeit [ps]) als Optionen aus dem Dropdown-Menü aus.
- Klicken Sie auf OK.
- Stellen Sie sicher, dass der Code zwei Arten von Informationen aus den NAD(P)H-FLIM-Z-Stacks berechnet.
- Messen Sie zunächst die räumlichen Informationen jeder Volumenschicht durch den Kollaps der TCSPC-Stapel, wobei die 227 zeitaufgelösten Photonenzählhistogramme auf eine einzelne Schicht projiziert werden, die als Intensitätsprojektionsbild bezeichnet wird (Abbildung 3G), und durch die Fourier-basierte Analyse der exponentiellen Zerfallskurven (wie von Leben et al.18 beschrieben) und durch den normalisierten Real- und Imaginärteil für jedes Pixel in jeder Volumenschicht (Abbildung 3C).
- Aus den realen und imaginären Bildern erhalten Sie die Phasordiagramme (Abbildung 3D) und das Bild der durchschnittlichen Fluoreszenzlebensdauer (t) (farbcodierte, räumlich aufgelöste durchschnittliche Zerfallskonstanten für jedes Voxel) (Abbildung 3E).
HINWEIS: Wie in der Einleitung beschrieben, wird die Fluoreszenzlebensdauer von NAD(P)H, wenn es an Enzyme gebunden ist, durch die Bindungsstelle des Co-Enzyms an das jeweilige Enzym bestimmt. - Bestimmen Sie den Beitrag des jeweiligen Enzyms zur Stoffwechselaktivität, indem Sie den Vektor zwischen dem Phasenvektor jedes Pixels und dem Phasenvektor von ungebundenem NAD(P)H erzeugen und auf den Halbkreis im Phasordiagramm projizieren. Der Halbkreis stellt alle möglichen monoexponentiellen Zerfälle der Fluoreszenzlebensdauer in reinen Verbindungen dar.
- Unter Verwendung der zuvor generierten Referenz der Fluoreszenzlebensdauer von NAD(P)H, die an die am häufigsten vorkommenden NAD(P)H-abhängigen Enzymegebunden ist 18,33 (Ergänzende Abbildung 1), und berechnen Sie die Wahrscheinlichkeit der Aktivierung dieser Enzyme.
HINWEIS: Jedem Pixel wird ein Farbcode des dominantesten Enzyms (d. h. des Enzyms, für das die höchste Aktivierungswahrscheinlichkeit berechnet wird) zugeordnet, wodurch eine Enzymkarte erstellt wird (Abbildung 3F).
- Aus dem Verhältnis von freiem (ungebundenem) zu enzymgebundenem NAD(P)H, konkret aus dem vektoriellen Verhältnis im Phasordiagramm zwischen freiem NAD(P)H bei 450 ps und dem enzymgebundenen Zustand, berechnen Sie die allgemeine metabolische Aktivität in Prozent zwischen 0% *nur ungebundenem NAD(P)H) und 100% (nur enzymgebundenes NAD(P)H). Indem jedem Pixel ein Wert für die metabolische Aktivität (0%-100%) zugewiesen wird, wird eine (farbcodierte) Aktivitätskarte erstellt (Abbildung 3F).
- Überlagern Sie die resultierenden Karten mit den Intensitätsbildern, um zusätzliche morphologische Informationen zu erhalten. Verwenden Sie hier ein ImageJ-Makro (siehe Materialtabelle), um den Farbton und die Sättigung der Enzym- oder Aktivitätskarten mit der Helligkeit der Intensitätsbilder zu überlagern.
- Stellen Sie sicher, dass das Makro über den gesamten Stack iteriert. Für jedes Slice (Tiefe) im Stapel muss das Intensitätsbild bzw. das Map der Wahl in Farbton, SÄTTIGUNG und HELLIGKEIT aufgeteilt werden (Bild > Typ > HSB-Stapel).
- Trennen Sie die Hauptkanäle für beide HSB-Stacks (Image > Stacks > Stack zu Bildern).
- Schließen Sie den Farbton und die Sättigung des Intensitätsstapels sowie die HELLIGKEIT des Map-Stapels (close(slicename_HUE), close(slicename_BRIGHTNESS), ...).
- Kombinieren Sie die verbleibenden Kanäle neu, um einen neuen HSB-Stapel zu bilden, der aus dem Farbton und der SÄTTIGUNG aus dem Ausschnitt der Karte von Interesse und der HELLIGKEIT aus dem Intensitätsbild besteht (Bild > stapelt > Bilder zum Stapel).
- Ändern Sie den Bildtyp in RGB, um ein besseres Erscheinungsbild zu erzielen (Bild > Typ > RGB-Farbe).
4. Segmentierung des Gewebes
HINWEIS: Verwenden Sie ein vortrainiertes U-Net-basiertes Netzwerk (ILASTIK, siehe Materialtabelle) für die Segmentierung des Darmwirts- bzw. Nematodengewebes und darüber hinaus des Epithels und der Lamina propria im Wirt sowie der hohen NAD(P)H-Fluoreszenzsignalbereiche und der niedrigen NAD(P)H-Fluoreszenzsignalbereiche im Fadenwurm.
- Öffnen Sie ILASTIK und wählen Sie ein neues Projekt und eine neue Pixelklassifizierung aus.
- Laden Sie die zuvor berechnete Intensitätsprojektion aus Schritt 3.7.1, indem Sie auf Neu hinzufügen > Separate Bilder hinzufügen klicken. Laden Sie zufällige Scheiben aus mehreren Messungen in den Behälter.
- Klicken Sie auf Feature-Auswahl > wählen Sie Features aus. Fügen Sie einen Sigma-Wert von 50 (einheitenlose Gewichtung) hinzu und markieren Sie alle Funktionen, die aktiv sein sollen.
HINWEIS: Während der Sigma-Wert die Gewichtung der Features bestimmt, bestimmen die Features die Klassen und die Fähigkeit des Netzwerks, Kanten, Formen, Texturen oder Farben zu erkennen. - Klicken Sie auf Training und benennen Sie die Beschriftungen entsprechend dem zu segmentierenden Gewebe (z. B. Epithel, Hintergrund usw.), indem Sie darauf klicken. Wählen Sie eine Beschriftung aus und färben Sie alle Pixel innerhalb des Bildes ein, die dieser Beschriftung entsprechen.
- Wiederholen Sie den Vorgang für die anderen Beschriftungen.
- Wiederholen Sie diesen Vorgang für etwa die Hälfte der Bilder im geladenen Datensatz.
- Klicken Sie auf Live-Update, und lassen Sie das Modell die Beschriftungen für die verbleibenden unbeschrifteten Bilder vorhersagen.
- Korrigieren Sie etwa die Hälfte der ausschließlich vorhergesagten und unbeschrifteten Bilder, indem Sie die falsch geschätzten Segmentierungen neu beschriften, und wiederholen Sie die Live-Aktualisierung.
- Wiederholen Sie diesen Schritt, bis das Netzwerk gelernt hat, das gewünschte Gewebe von Interesse richtig einzuschätzen. Stellen Sie sicher, dass die Zuverlässigkeit des Netzwerks, das links in der Nähe der Bezeichnungsnamen angezeigt wird, zwischen 95 % und 98 % liegt.
- Laden Sie einen neuen Datensatz, wie in Schritt 3.11 beschrieben, diesmal mit allen Slices aus einer Messung, und klicken Sie auf Prediction Export.
- Wählen Sie im Dropdown-Menü "Quelle" die Option Einfache Segmentierung; Wählen Sie unter "Format" die Ausgabe im tiff-Format aus. und wählen Sie unter "Einstellungen für den Export von Bildern auswählen" die Option Speicherpfad für die Ausgabe aus. Klicken Sie auf Exportieren.
- Dadurch werden binäre Masken für den interessierenden Gewebetyp erstellt. Da sie aus Schichten bestehen, bei denen das segmentierte Gewebe von Interesse einen Pixelwert von 1 und der Rest einen Pixelwert von 0 hat (Abbildung 3H), multiplizieren Sie einfach die binären Masken mit den generierten Daten, wie in Schritt 1.1 und Schritt 1.4-1.7 aufgeführt. Dies führt zu maskierten Daten (Abbildung 3 I, J, M). Verwenden Sie ImageJ für diesen Schritt (> Bildrechner verarbeiten > multiplizieren).
- Berechnen Sie für jedes Lautstärke-Slice ein Signal-Rausch-Verhältnis (SNR) aus den maskierten Intensitätsbildern. Verwenden Sie zu diesem Zweck ein ImageJ-Makro. Erstellen Sie Ihr Programm so, dass es das Signal-Rausch-Verhältnis (SNR) aus den segmentierten und maskierten Bildern berechnet.
- Verwenden Sie den segmentierten Hintergrund aus Schritt 3.21 als Hintergrund (BG) und das segmentierte Tissue of Interest als Signal (SIG), indem Sie mit zwei verschachtelten for-Schleifen über jedes Pixel jedes Slice mit einem Wert größer als 0 iterieren. Berechnen Sie das SNR mit der folgenden Formel33:
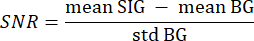
Dabei bezieht sich der mittlere SIG auf den Mittelwert des Signalhistogramms, der mittlere BG auf den Mittelwert des Hintergrundhistogramms und std BG auf die Standardabweichung des Hintergrundhistogramms. - Verwerfen Sie Volume-Slices mit einem SNR-Wert unter 5 aus der weiteren Analyse.
- Verwenden Sie den segmentierten Hintergrund aus Schritt 3.21 als Hintergrund (BG) und das segmentierte Tissue of Interest als Signal (SIG), indem Sie mit zwei verschachtelten for-Schleifen über jedes Pixel jedes Slice mit einem Wert größer als 0 iterieren. Berechnen Sie das SNR mit der folgenden Formel33:
- Erstellen Sie mit dem Python-Skript18,33 ein Enzymfrequenzdiagramm aus den Enzymkarten für jeden Gewebetyp, indem Sie die Enzyme schichtweise summieren und mittelnieren und dann auf ein Gewebevolumen von 100 μm³ normalisieren (Abbildung 3K, N).
- Codieren Sie das Skript so, dass es die maskierte Enzymkarte lädt.
- Iterieren Sie über jedes Pixel, berechnen Sie die Summe der Pixel desselben Enzyms und dividieren Sie dann durch das analysierte Volumen (die Summe aller Pixel).
- Gruppieren Sie die berechneten Häufigkeiten in ihren Stoffwechselzuständen und mitteln, indem Sie durch die Anzahl der Enzyme in jeder Gruppe dividieren. Schreiben Sie Code, der Stoffwechselzustände, die um LDH gruppiert sind, anaeroben Glykolyse-ähnlichen Signalwegen, Enzymaktivität, die um PDH/IDH/GAPDH gruppiert ist, aerober Glykolyse/oxPhos-ähnlichen Signalwegen und NADPH-Oxidase-Aktivität (NOX-Aktivität) oxidativem Stress, der zur Abwehr verwendet wird, zuordnet (Abbildung 3L, O).
- Verwenden Sie die matplotlib-Bibliothek von Python, um die Daten mit Boxplots zu visualisieren.
- Berechnen Sie die Aktivität des Gewebes aus den maskierten Aktivitätskarten (Abbildung 3F) als Mittelwert über jede Volumenschicht für alle Pixel mit einem Wert größer 0 mithilfe eines ImageJ-Makros, indem Sie Analyze > Measure ausführen.
- Visualisieren Sie alle generierten Bilder nebeneinander mit ImageJ/FIJI.
Ergebnisse
Unter Verwendung des aktuellen NAD(P)H-FLIM-Verfahrens 28,29,33 in Kombination mit der beschriebenen Phasoranalysemethode wurden die metabolische Aktivität und die Stoffwechselwege in gesunden und infizierten Zwölffingerdarmen an Tag 6, Tag 10, Tag 12 und Tag 14 nach der Infektion mit dem murinen intestinalen Nematoden H. polygyrus gemessen.
Erhaltene Lebensfähigkeit des Darmgewebes im exzidierten Zwölffingerdarm durch NAD(P)H-FLIM nachgewiesen
Um die Gewebeaktivität ex vivo zu untersuchen und zu bestimmen, wie lange exzidierte Gewebeproben im Vergleich zu Gewebe in lebenden Organismen metabolisch aktiv bleiben, wurde NAD(P)H-FLIM an exzidiertem Zwölffingerdarmgewebe von gesunden Mäusen durchgeführt, das (i) frisch exzidiert oder (ii) 3 h auf Eis gehalten wurde, und die Ergebnisse wurden mit bereits veröffentlichten NAD(P)H-FLIM-Daten verglichen, die im Darm von anästhesierten Mäusen gewonnen wurden32. Die Präparation des exzidierten Zwölffingerdarms und das bildgebende Experiment wurden wie im Probenvorbereitungsprotokoll beschrieben (Schritt 1) durchgeführt.
Die Fluoreszenzlebensdauer von NAD(P)H wurde über einen Zeitraum von 3 h und in vier verschiedenen Gewebetiefen gemessen (Zottenspitze: ~0 μm, obere Zotten: ~−50 μm, untere Zotten: ~−100 μm, Zottenbasis: ~−200 μm). Aus den so gewonnenen NAD(P)H-FLIM-Daten wurden Aktivitätskarten erstellt, die die allgemeine Stoffwechselaktivität in den verschiedenen Regionen der Zotten im Zeitverlauf zeigen. Die allgemeine metabolische Aktivität (frei zu enzymgebundenem NAD(P)H) gilt als Indikator für die Degeneration des Gewebes (Abbildung 4A,B).
Eine zweite Messreihe mit ähnlichen Gewebeproben wurde mit einem weiteren Stück Darm durchgeführt. Hier wurden die Messungen nicht direkt nach der Tötung durchgeführt, sondern das Gewebe nach der Präparation für 3 h auf Eis gelegt, um die maximale Wartezeit für Proben, die im realen Experiment auf Eis gelegt wurden, nachzuahmen. Nach einer Aufwärmzeit von ~15 min mit einer Heizplatte wurde eine Bildgebung in verschiedenen Gewebetiefen durchgeführt (Abbildung 4C,D). Die in den beiden zuvor beschriebenen Aufbauten gemessene Stoffwechselaktivität stimmte gut mit der mit NAD(P)H-FLIM gemessenen Stoffwechselaktivität unter intravitalen Bedingungen im murinen Darmüberein 32.
Nachweis unterschiedlicher Stoffwechsel- und Enzymaktivität im Darmgewebe des Wirts und von H. polygyrus im Verlauf einer akuten Infektion mittels NAD(P)H-FLIM
Im Verlauf der Erkrankung ab dem 6. Tag nach der Infektion, wenn sich die Wurmlarven in der Submukosa entwickeln, am 10. Tag in das Lumen eindringen und die Darmnische infiltrieren, wurde eine Zunahme der allgemeinen Stoffwechselaktivität der Parasiten beobachtet. Ausgehend von einem Ruhezustand am 6. Tag stieg die Stoffwechselaktivität stetig auf 73,1 % ±± 5,9 % 5,9 % an.
Der Ausgangszustand des Parasiten während der frühen Infektion spiegelt sich in seinem metabolischen Fingerabdruck wider; Konkret war an Tag 6 die Energieproduktion zwischen anaeroben und aeroben Wegen ausgeglichen, was auf einen bevorstehenden Durchbruch in das Lumen hindeutet. Es kam dann zu einer Verschiebung der Energieproduktion hin zu einem stark anaeroben Glykolyse-ähnlichen Verhalten, vermutlich aufgrund des erhöhten Energiebedarfs für die Migration in das Lumen (Tag 10), und die Parasiten zeigten später (gemessen bis Tag 14) einen oxidativen Phosphorylierungs-/aeroben Glykolyse-ähnlichen Weg zur Energieproduktion. Die allgemeine metabolische Aktivität des Wirts blieb mit 74,0 % ± 7,6 % (Tag 6 bis 14) mit dem gesunden Zustand vergleichbar, während die Stoffwechselwege im Wirtsgewebe im Verlauf der akuten Infektion überwiegend eine aerobe/oxidative Phosphorylierung waren.
Die Abwehrreaktion der Parasiten beruht auf der Aktivierung von DUOX2, da es bei dieser Spezies das einzige Mitglied der NADPH-Oxidase-Familie ist. Diese Reaktion ist im frühen Stadium der Infektion, am Tag 6, vernachlässigbar, nimmt aber zu einem späteren Zeitpunkt während der akuten Infektion zu. Daher konnten wir einen drastischen Anstieg der DUOX2-Aktivierung ab dem 10. Tag, nach luminaler Transmigration und während der gesamten akuten Phase der Infektion feststellen. (Abbildung 5E,F)
Das Entzündungsverhalten des Wirts hingegen stieg im Vergleich zu einem gesunden Zustand am Tag 10 nach der Infektion, mit dem Auftreten des Parasiten im Lumen, dramatisch an und hörte später am Tag 14 nach der Infektion auf. Dieses Verhalten geht einher mit der Tatsache, dass Parasiten in den späteren Stadien (chronischen) Stadien der Infektion einen regulatorischen Phänotyp im Immunsystem des Wirts induzieren 4,6,7 (Abbildung 5A-D).

Abbildung 1: Präparation des murinen Darmgewebes für NAD(P)H-FLIM mit dem Multiphotonen-Laser-Scanning-Mikroskop. (A) Die Präparation erfordert das Entfernen des Darmstücks und das Trimmen auf eine Länge von ca. 1 cm (1), das Auftragen von Gewebekleber auf eine Petrischale und das Auflegen des Röhrchens darauf (2), das Röhrchen in Längsrichtung mit einer stumpfen Schere in der Nähe des Bodens (3), Aufklappen des Röhrchens, um die luminale Seite mit einer stumpfen Pinzette zu öffnen (4), Zählen der Würmer unter einem Stereomikroskop (5), Versiegeln des Gewebes mit niedrig konzentrierter Agarose in einer dünnen, etwa 0,5 mm dicken Schicht (6), Befüllen der Schale mit 10 % PBS (7) und Bildgebung der Gewebeprobe oder Ablegen auf Eis zur Vorbereitung auf die Verarbeitung (8). (B) Zeitachse für die Verarbeitung einer einzelnen Gewebeprobe in Minuten von der Entnahme des Zwölffingerdarms bis zur Platzierung unter dem Mikroskop. Die Messzeit einer Probe bestand darin, einen ROI zu finden, die abgekühlten Proben auf einem Heizelement in der Zwischenzeit auf 37 °C zu erwärmen und dann die Bilder aufzunehmen. (C) Die durchschnittliche Messzeit von 45 min (nachträglich aus den Daten bestimmt) wird in (C) gegen die vier untersuchten Infektionsstadien sowie gegen gesundes Gewebe aufgetragen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Schematische Darstellung des Versuchsaufbaus. Das System besteht aus einem durchstimmbaren Ti:Sa-Laser Titan:Saphir LASER Chameleon ULTRA II (690-1.080 nm, 80 MHz, Pulsbreite von 140 fs) mit einem kommerziellen Scankopf Trimscope II. Eine Wasserimmersionsobjektiv (20x, NA 1,05) fokussiert das Anregungslicht. Ein System aus dichroitischen Spiegeln, Interferenzfiltern (525/50 nm, 593/40 nm, 655/40 nm) und PMTs detektiert das Fluoreszenzsignal. Die Fluoreszenzlebensdauerdaten werden mit Hilfe eines hybriden PMT (GaAsP) (hPMT) am Kanal 466/60 nm detektiert, das durch ein Kühlmodul (CM) gekühlt und mit einem zeitkorrelierten Einzelphotonen-Zählmodul verbunden ist; Bei diesem Prozess werden Photonen in einem Bin-Bereich von 55 ps detektiert und Daten über 9 ns mit einer Gauß-förmigen Instrumentenantwortfunktion von 250 ps FWHM (TCSPC) erfasst. Um den TCSPC auszulösen, werden 5%-8% der anfänglichen Pulsintensität vom Anregungsstrahl gespalten und in einer ultraschnellen Photodiode (PD) umgeleitet. Um die Zwei-Photonen-Anregung im Echtzeitmodus ohne TCSPC zu erhalten, werden PMTs mit Farbfiltern (525 nm, 593 nm, 655 nm) verwendet. Eine breitbandige Quecksilberdampflampe (MV) und ein Augenweg werden für die Beobachtung der Sonde (OP) für die visuelle Fluoreszenzmikroskopie und die Positionierung der Probe verwendet, und der ROI wird bei einem Intravitalstadium (IS) mit einer Schrittweite von 1 μm (IS) gefunden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
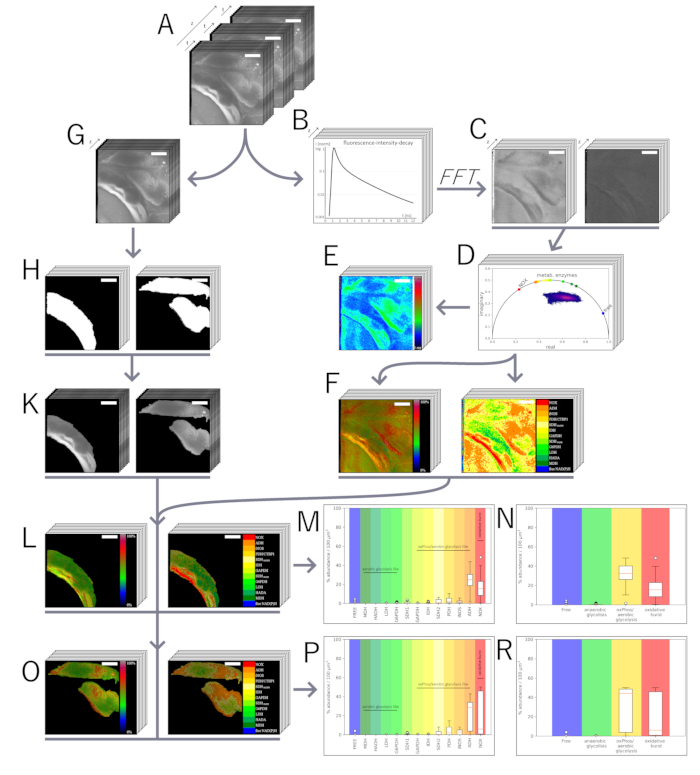
Abbildung 3: Phasoranalyse der NAD(P)H-FLIM-Daten im Zeitbereich. (A) Die Rohdaten werden in vier Dimensionen erfasst: den räumlichen Informationen (x, y, z; 505 Pixel = 500 μm für xy) und den TCSPC-Daten (227 Zeitpunkte à 55 ps für t). (B) Jedes Pixel in den TCSPC-Rohdaten entlang t, wie in (A) gezeigt, enthält den Abfall der Fluoreszenzintensität nach einem Anregungsimpuls in 227 Zeiträumen von jeweils 55 ps. Die Zeitbereichs-TCSPC-Daten werden durch Berechnung der normalisierten, diskreten numerischen Fourier-Transformation (Phasor-Ansatz) Gaußscher vorgefaltet und in einen virtuellen Phasenbereich übertragen. Daraus ergibt sich eine komplexe Anzahl jedes Pixels im Bild, die einen realen und einen imaginären Teil enthält, und dies kann aufgeteilt und als zwei intensitätskodierte Bilder pro gemessener Schicht dargestellt werden: (C, links) der reale Teil und (C, rechts) der imaginäre Teil. (D) Der reale und der imaginäre Teil liefern die Koordinaten im Phasorplot, wobei jeder Punkt im Diagramm ein räumliches Gegenstück und seine Zerfallsinformation von (B) hat. Die halbkreisförmige Zeitachse zeigt monoexponentielle Lebensdauern an. (E) Die Fluoreszenzlebensdauer wird aus (C) mit Hilfe der kontinuierlichen Fourier-Transformation (Rück-)Transformation berechnet und farbcodiert. Dies entspricht einer mono-exponentiellen Analyse der Intensitätsabfallkurve in (B). In Anlehnung an ein zuvor entwickeltes Analyseframework wird (F, rechts) eine Karte der NAD(P)H-abhängigen Enzyme und (F, links) eine Karte der enzymatischen Aktivität erstellt. (G) Parallel dazu werden die TCSPC-Rohdaten in die Intensitätsprojektion eingeklappt, wobei die zeitlichen Informationen verloren gehen und nur die räumlichen Informationen erhalten bleiben. (H) Daraus werden mittels ILASTIK Masken für das gewünschte Gewebe von Interesse durch trainierte Segmentierungsalgorithmen generiert, die mit der Intensitätsprojektion überlagert werden; ein Beispiel ist hier für (I, links) Parasit und (I, rechts) Wirt am Tag 14 nach der Infektion zu sehen. (J, links) Die maskierte Aktivitätskarte und die maskierte Enzymkarte von (J, rechts) des Parasiten und von (M) dem Wirtsgewebe. Aus den maskierten Karten werden Enzymhäufigkeitsdiagramme für den Parasiten erstellt, indem die Enzyme gezählt und der Abundanzprozentsatz auf 100 μm3 (K) normalisiert wird. Nach der Analyse von Liublin et al.33 wurden die Enzymhäufigkeiten nach den Stoffwechselwegen der aeroben Glykolyse und der oxidativen Phosphorylierung/anaeroben Glykolyse sowie dem oxidativen Burst und einem metabolisch inaktiven Zustand (ungebundenes, freies NAD(P)H) für (L) den Parasiten und für (N,O) das Wirtsgewebe gruppiert. Alle Maßstabsstäbe sind 100 μm groß. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Vorabbewertung der Vitalität des explantierten Gewebes. (A) Karte der enzymatischen Aktivität aus dem frisch explantierten Zwölffingerdarm einer gesunden C57/bl6-Maus, wie in Abschnitt 3 beschrieben. Das Gewebe wurde direkt nach der Präparation im Abstand von 20 min für eine Zeit von insgesamt 106 min auf einer Heizplatte bei 37 °C abgebildet. Die Messungen wurden in vier verschiedenen Gewebetiefen durchgeführt: an den Zottenspitzen (0 μm), in der oberen Mitte (-50 μm), in der unteren Mitte (-100 μm) und an der Basis der Zotten (-200 μm). Die in (A) gezeigten Beispiele zeigen den oberen mittleren Bereich der Zotten 5 min und 68 min nach der Präparation. Für jeden Messzeitpunkt wurde die über alle Tiefen gemittelte enzymatische Aktivität aus den segmentierten Aktivitätskarten der Lamina propria und des Epithelgewebes berechnet und über den Messzeitpunkt aufgetragen. (B) Die Grafik zeigt die Vitalität und das Degenerationsverhalten des explantierten Gewebes nach der Tötung und Präparation über 106 min. Um das Gewebedegenerationsverhalten der präparierten Proben abzudecken, die auf Eis gelegt wurden, um sich für die Messung in die Warteschlange zu stellen, wie in Abschnitt 1 beschrieben, wurde frisch explantiertes Zwölffingerdarm einer gesunden C57/bl6-Maus präpariert und 3 h lang auf Eis gelagert, um die maximale Wartezeit nachzuahmen, die während der Experimente auftrat. (C) Das Gewebe wurde dann auf bis zu 37 °C erhitzt und mit NAD(P)H-FLIM abgebildet, und es wurde eine Aktivitätskarte erstellt. Die entsprechenden durchschnittlichen Aktivitäten des Gewebes wurden aus den segmentierten Daten berechnet, wie bei (B) und (D) beschrieben, was zu einer Aktivität von 57,8 % ± 4,6 % für die Lamina propria und 68,5 % ± 5,7 % für das Epithel führte). Alle Maßstabsstäbe haben eine Größe von 250 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: NAD(P)H-FLIM von Wirtszotten und Nematodengewebe, die den Stoffwechselzustand und die bevorzugten Stoffwechselwege im Wirt und im Parasiten anzeigen. (A) Die durchschnittliche allgemeine metabolische Aktivität, bestimmt aus den maskierten Aktivitätskarten für das Epithel (EP) (78,4 % ± 5,5 %) und die Lamina propria (LP) (76,3 % ± 5,2 %) im gesunden murinen Zwölffingerdarm. (B) Nach unserer Analyse wurden die Enzymhäufigkeiten nach den Stoffwechselwegen gruppiert, einschließlich aerober Glykolyse, oxidativer Phosphorylierung/anaerober Glykolyse und oxidativer Burst für das Epithel (EP) und die Lamina propria (LP) im gesunden murinen Zwölffingerdarm. (C,D) Ähnlich wie bei (A) und (B) wurde eine Analyse für das im Verlauf der akuten Infektion erworbene Wirtsgewebe durchgeführt. Die durchschnittliche metabolische Aktivität des Wirtsgewebes betrug an Tag 6 74,0 % ± 7,6 %, an Tag 10 72,4 % ± 10,5 % und an Tag 14 73,5 % ± 11,4 %. (E,F) Wie bei (C) und (D) wurde das Parasitengewebe im Verlauf der akuten Phase der Erkrankung analysiert. Die durchschnittliche metabolische Aktivität des Parasiten betrug an Tag 6 (58,0 % ± 2 %, an Tag 10 61,1 % ± 9,9 %, an Tag 12 64,5 % ± 5,8 % und an Tag 14 73,1 % ± 5,9 %. Die statistische Auswertung erfolgte mittels eines ANOVA-Tests (ns p > 0,05, *p < 0,05, **p < 0,01, ***p < 0,001). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: NAD(P)H-Bindungszustandsabhängige Fluoreszenzlebensdauer. Schematische Darstellung von (A I) ungebundenem (freiem) NAD(P)H bei Anregung durch Photonenabsorption zum Zeitpunkt 0 auf der Zeitachse und seiner bindungszustandsspezifischen Fluoreszenzlebensdauer (τ) bei 450 ps. Der gleiche Prozess wird für (A II) NADH gezeigt, das an LDH gebunden ist, und (A III) PDH, mit einer Fluoreszenzlebensdauer bei 1.600 ps bzw. 2.470 ps, und für NADPH, gebunden an NADPH-Oxidasen (NOX) (A IV) mit einer Fluoreszenzlebensdauer von 3.650 ps. (B) Tabelle der charakterisierten Fluoreszenzlebensdauer von NAD(P)H, gebunden an die am häufigsten vorkommenden Enzyme. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die kritischen Schritte innerhalb des Protokolls erfolgen während der Vorbereitung und bei der Ermittlung des ROI. Fasern von teilweise verdauten Lebensmitteln stellen eine Herausforderung für die Bildgebung dar, vor allem aufgrund der endogenen Lumineszenz der Fasern, die sich mit der NAD(P)H-Fluoreszenz überlappen, aber auch aufgrund ihres harmonischen Generationssignals. Es ist von großer Bedeutung, ROIs zu finden, die frei von Fäkalien sind. Unser Ziel war es, Messbereiche mit Fäkalien zu vermeiden. Auf Waschen wurde verzichtet, da dies die Integrität der empfindlichen Zotten beeinträchtigt und die Schleimviskosität beeinflusst. Darüber hinaus sollte der gesamte Vorbereitungsvorgang vorzugsweise etwa 5-10 Minuten pro Probe dauern, um eine Degeneration und Austrocknung des Gewebes zu vermeiden. Des Weiteren ahmt die verwendete niedrig konzentrierte Agarose die Viskosität des Darmschleims34 nach. So kann sichergestellt werden, dass die Zotten nicht kollabieren, während sich die Würmer während der Messung frei bewegen. Die Messung darf nicht länger als 45 Minuten dauern (Abbildung 1A,B und Abbildung 1A,B,C), um die besten Bedingungen für die Abbildung des Zellstoffwechsels zu erhalten. Daher sollte die Suche nach dem Bereich of Interest (ROI) nicht länger als 15 Minuten dauern. Um jedoch sicherzustellen, dass sich das auf Eis gehaltene Gewebe auf der Heizplatte auf die gewünschten 37 °C erwärmt, sollte die Bildgebung nach ~10 min erfolgen. Die Bildgebung dauert in der Regel 45 Minuten (siehe TCSPC-Datenanalyse zur Kontrolle der Gewebeviabilität vor und während der Messung; Abbildung 1C und Abbildung 4). Bei der Suche nach einem geeigneten ROI ist darauf zu achten, dass mindestens 20 % der luminalen Seite des Zwölffingerdarmgewebes (Tage 10-14 nach der Infektion) unter der Objektivlinse von Wurmgewebe bedeckt sind und dass die Bewegung des Parasiten sichtbar ist. Für Personen am Tag 6 und gesunde Personen ist ein geeigneter ROI definiert als einer, bei dem die Gewebeintegrität nach der Präparation erhalten bleibt (in der Regel weit von den Grenzen der Probe entfernt). Dies gewährleistet die besten Trainingsbedingungen für das Segmentierungsmodell und liefert ausreichend Datenpunkte für die weitere Analyse. Für die Infektionsstadien ab dem 10. Tag, wenn die Würmer in das Lumen eindringen und sich an die Zotten anheften, werden die Daten nur nach dem Hintergrund und dem Parasiten- und Darmgewebe mit hoher Enzymaktivität segmentiert, da in Gegenwart der Würmer die Zotten durch Nestbildung meist unstrukturiert sind und die Gewebeintegrität der Zotten nicht mehr vorhanden ist. Für das Gewebe, das am Tag 6 explantiert wurde, und von gesunden Mäusen ermöglicht die Gewebeintegrität die Segmentierung der Lamina propria sowie des Epithels gegenüber dem Hintergrund. Daher wird die Verwendung von separat trainierten Modellen für die Bedingungen dringend empfohlen. Das Signal überlastet die Detektoren bei stark autofluoreszierendem Material, das gelegentlich innerhalb des gemessenen Volumens anfällt. In dieser Arbeit wurde eine dynamische Laserleistungsregelung über die gesamte Bildgebungstiefe in der Software verwendet.
Die Grenzen dieser Technik beziehen sich auf die physikalischen Grenzen des Anregungsvolumens. Die axiale Auflösung der Objektivlinse wurde zuvor mit 1,3 μm bei 850 nmbestimmt 28, aber da sie typischerweise im Gewebe abgebaut wird, hielten wir eine Schrittweite von 2 μm für angemessen. Bei einem Sichtfeld von 0,5 mm x 0,5 mm ist jedes Slice 505 Pixel x 505 Pixel groß. Die laterale Auflösung des Objektivs beträgt ~ 350 nm; Aufgrund der Auflösungsverschlechterung24,28 wurde jedoch eine Pixelgröße von ~ 1 μm als ausreichend befunden. Das bedeutet, dass die Messung einem stochastischen Ansatz folgt. Die erfasste Zerfallskurve setzt sich aus der Summe aller Zerfallskurven zusammen, d.h. aus allen Fluoreszenzlebensdauern innerhalb des Ausgangsvolumens von ~1 x 1 x 2μm 3. Die über die Zeit für jedes Voxel aufgezeichnete Photonenzahl (die gemessene Fluoreszenzlebensdauer oder TCSPC-Stapel) folgt der Form einer multiexponentiellen Zerfallskurve oder der linearen Kombination von multiexponentiellen Zerfallskurven, die schwer vorherzusagen ist. Eine modellfreie normierte Fourier-basierte Analyse wurde verwendet, um die komplexe Fluoreszenzzerfallskurve in jedem Voxel zu analysieren, die sogenannte Phasoranalyse. Die Stacks wurden im Vorfeld mit einem Gaußschen Filter (σ2) gefaltet, um das Rauschen in den zeitaufgelösten Fluoreszenzbildern zu reduzieren. Der Verlust der räumlichen Auflösung ist akzeptabel und stellt einen Kompromiss für eine bessere Auflösung der Fluoreszenzlebensdauer dar.
Die Frame-Erfassungszeit liegt im Bereich von 2,5 s bis 7 s (Mittelwertbildung), und die Erfassungszeit für einen typischen Z-Stapel von 500 x 500 x 200 μm³ (505 x 505 x 101 Voxel) beträgt ~ 450 s (7,5 min). Diese Zeiten führen oft dazu, dass sich während einer Messung die Parasiten bewegende Artefakte verursachen. Die Daten bleiben in diesem Fall verwendbar. Gelegentlich bewegen sich die Parasiten während der Messung außerhalb des gemessenen Volumens.
Unter Verwendung der NADH- und NADPH-Fluoreszenzlebensdauer-Bildgebung von exzidiertem murinem Zwölffingerdarmgewebe von Mäusen, die mit dem Fadenwurm H. polygyrus infiziert waren, und unserem zuvor veröffentlichten Analyserahmen unter Verwendung der Referenzfluoreszenzlebensdauer von NAD(P)H, die an häufige NAD(P)H-abhängige Enzyme gebunden sind, wurden die allgemeine Stoffwechselaktivität und die unterschiedlichen Stoffwechselwege im Darmgewebe des Wirts und im Parasitengewebe sichtbar gemacht und analysiert; Diese Analyse enthüllte die metabolische Wechselwirkung zwischen den beiden. Alternativ, wenn die Informationen über die gesamte metabolische Aktivität oder über das Gleichgewicht zwischen NADH und NADPH von Interesse sind, stehen allgemein anwendbare robuste Algorithmen und Softwareroutinen zur Verfügung, die verwendet werden können22,23.
Das hier vorgestellte Protokoll und das etablierte Analyseverfahren stellen eine verallgemeinerte Methode zur Messung des Stoffwechsels und der Stoffwechselwege dar, die auf andere Organe und andere Arten von Pathologien anwendbar ist 13,15,16,17,19,20.
Im Vergleich zu z.B. der Einzelzellanalyse mit Zytometrie liegt die Eleganz der Methode in der Möglichkeit, mit minimalen Eingriffen und markierungsfrei subzelluläre räumliche Informationen über Stoffwechselwege in biologischen Systemen zu gewinnen, mit der Möglichkeit des zusätzlichen Einsatzes von Markern oder Farbstoffen für eine höhere Informationsdichte. Der Eingriff in das biologische Gewebe und die damit verbundenen Infektionsprozesse sind aufgrund der Wahl eines natürlich bei Nagetieren vorkommenden Parasiten sowie der Voruntersuchung des Darmgewebes und seiner Vergleichbarkeit mit lebendem Gewebe in Bezug auf die Vitalität minimal. Mit entsprechender Segmentierung und Normalisierung können vergleichbare Messungen über die vierte Dimension (Zeit) generiert werden.
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden finanziellen Interessen bestehen.
Danksagungen
Wir danken Robert Günther für den hervorragenden technischen Support. Finanzielle Unterstützung durch die Deutsche Forschungsgemeinschaft (DFG) im Rahmen von Grant SPP2332 HA2542/12-1 (S.H.), NI1167/7-1 (R.A.N.), HA5354/11-1 (A.E.H.) und RA2544/1-1 (S.R.), im Rahmen von Grant SFB1444, P14 (R.A.N., A.E.H.), unter Grant HA5354/8-2 (A.E.H.) und unter Grant GRK2046 B4 und B5 (S.H., S.R.) und HA2542/8-1 (S.H.) werden sehr anerkannt. W.L. erhielt ein Promotionsstipendium der Berliner Hochschule für Technik, School of Applied Sciences, Berlin in Medizinischer Physik/Physikalischer Technik.
Materialien
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
Referenzen
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten