Method Article
NAD(P)H 荧光寿命成像用于线虫感染期间小鼠肠道和寄生虫的代谢分析
摘要
本方案描述了感染天然寄生虫 Heligmosomoides polygyrus 的外植小鼠肠道的 NAD(P)H 荧光寿命成像,这允许人们以空间分辨的方式研究宿主和寄生虫组织中的代谢过程。
摘要
寄生虫通常会对宿主的健康产生负面影响。它们代表着巨大的健康负担,因为它们在全球范围内长期影响受感染的人或动物的健康,从而影响农业和社会经济成果。然而,已经描述了寄生虫驱动的免疫调节作用,与自身免疫性疾病具有潜在的治疗相关性。虽然宿主和寄生虫的新陈代谢都有助于它们的防御,并且是线虫在肠道中存活的基础,但由于缺乏足够的技术,它在很大程度上仍然没有得到充分的研究。我们开发并应用 NAD(P)H 荧光寿命成像在天然线虫 Heligmosomoides polygyrus 感染期间移植的小鼠肠道组织,以空间分辨方式研究宿主和寄生虫的代谢过程。辅助酶烟酰胺腺嘌呤二核苷酸 (NADH) 和烟酰胺腺嘌呤二核苷酸磷酸 (NADPH),以下简称 NAD(P)H,在物种中保留,其荧光寿命的利用取决于它们的结合状态和催化代谢过程的酶上的结合位点。聚焦表达最丰富的 NAD(P)H 依赖性酶,区分了与厌氧糖酵解、氧化磷酸化/需氧糖酵解和基于 NOX 的氧化爆发相关的代谢途径,作为主要防御机制,并表征了感染过程中宿主和寄生虫之间的代谢串扰。
引言
寄生虫感染给人类健康带来了巨大的负担 1,2。在工业化国家观察到自身免疫性疾病的增加与寄生虫感染的减少之间存在相关性。众所周知,寄生虫可以通过抑制过度的宿主免疫反应来产生有益效果。H. polygyrus 是啮齿动物肠道中发现的一种天然寄生虫,已知这种寄生虫可诱导免疫调节机制,通过诱导受感染宿主中的调节性 T 细胞 (Treg) 来降低宿主的抗寄生虫免疫反应 3,4,5,6,7,8,9,10,11 .这些调节机制在退行性自身免疫性疾病中尤其令人感兴趣。
尽管新陈代谢在宿主和寄生虫的防御、生存和功能方面都起着重要作用,但对宿主和肠道线虫之间代谢串扰的分析仍然被广泛忽视。我们建议在双光子激发上适应和应用 NADH 和 NADPH 荧光寿命成像,该技术已广泛用于哺乳动物细胞和组织的不同生理和病理生理情况12,以研究活组织中的宿主和线虫代谢。
NADH 和 NADPH,简称 NAD(P)H,是存在于所有基于细胞的生命形式中普遍存在的分子,并在各种代谢途径中发挥辅酶的作用。例如,它们参与能量产生、还原性生物合成和 NADPH 氧化酶介导的活性氧 (ROS) 产生,这主要与细胞防御和细胞通讯有关 13,14,15,16,17,20。两种辅酶在 750 nm 的双光子激发下在 ~450 nm 处发射荧光,因此可以在细胞和组织中进行无标记物代谢成像19,21。由于双光子激发光谱相似且相当宽,因此可以仅用一个波长激发 NADH 和 NADPH21。
辅酶 NAD(P)H 的荧光寿命直接取决于它所结合的酶 18,21,22,23。由于其化学结构允许分子内能量转移,激发的 NADH 或 NADPH 分子在弛豫并发射荧光光子之前,会以取决于其结合特性的速率通过内部转化过程失去能量到酶(催化剂)中。这个寿命可以深入了解酶上的 NAD(P)H 结合位点,因此,优先发生生化反应 19,21,22,23,24,25。游离 NADH 和 NADPH 分子的荧光寿命为 ~450 ps,而它们与酶结合时的荧光寿命要长得多 (~2,000 ps),并且取决于它们在相应酶上的结合位点21。
有超过 370 种酶参与 NAD(P)H 连接过程;然而,只有最丰富的才能在显微镜的激发范围内为产生的 NAD(P)H 荧光寿命做出贡献。使用来自哺乳动物细胞的 RNASeq 数据,我们确定了最丰富的 NAD(P)H 依赖性酶,并生成了荧光寿命参考,以解释组织和细胞样品中生成的数据18。因此,这项工作区分了乳酸脱氢酶 (LDH) 的优先活性,它与厌氧糖酵解代谢途径相关,以及异柠檬酸脱氢酶 (IDH) 和丙酮酸脱氢酶 (PDH) 活性,它们主要参与有氧糖酵解/氧化磷酸化代谢途径16,20.此外,由于这些酶在细胞中的特征位置(膜结合)以及特别长的 NADPH 荧光寿命 (3,650 ps)18,24,29,30,32,NADPH 与 NADPH 氧化酶的结合可以很容易地解决。来自 H. polygyrus 的 RNASeq 数据表明,为哺乳动物细胞生成的参考也以适应的形式适用于该线虫27。
因此,在这项工作中,通过在感染 H. polygyrus 的小鼠新鲜移植的十二指肠样本中进行 NAD(P)H 荧光寿命成像 (FLIM),获得了有关游离和酶结合 NAD(P)H 之间比率的信息,它描述了所有组织中的一般代谢活动,以及图像每个像素中的主要活性酶(即 NAD(P)H 优先在该特定位置结合的酶)。这些实验的成功取决于外植肠道的准确样品制备、亚细胞分辨率下 NAD(P)H 荧光寿命的可靠实时成像以及标准化数据评估,如本协议所述。
研究方案
所有实验均按照国家动物保护指南进行,并经德国动物伦理委员会(G0176/16 和 G0207/19)批准。该方案描述了 NAD(P)H 荧光寿命成像数据采集和数据评估,这允许人们评估感染天然小鼠肠道线虫 H. polygyrus 后宿主肠道和寄生虫的一般代谢活动和特定代谢途径。为此,10-12 周龄的雌性 C57BL/6 小鼠感染了 200 只 3 期幼虫 (L3)。在感染的不同时间点,处死受感染的小鼠,切除十二指肠并准备成像,如前所述33。未感染、年龄和性别匹配的小鼠的十二指肠也以类似方式制备和成像以用于控制目的。为了保持进一步成像和分析所需的组织特性,必须在植入后立即处理样品,并且必须迅速执行下一步(步骤 1.1-1.7)(图 1B)。
1. 样品制备
- 从最初切除的十二指肠切下 ~1 cm 长的块。
- 用快速固化的组织胶(直径 47 毫米,边缘高度 10 毫米,参见 材料表)将拉直的组织管粘合在小培养皿的中间(图 1A2)。
- 用微刷在培养皿底部涂上一层薄薄的附加胶水,覆盖组织周围的大面积(图 1A2)。
- 使用钝剪刀在培养皿底部附近沿着物理固定肠的整个长度切开(图 1A3)。
- 用钝镊子展开肠道,使腔侧与胶水完全接触。因此,肠道由胶水固定,管腔侧朝上(图 1A4)。
- 在感染小鼠的情况下,在立体显微镜下(放大 10 倍)对蠕虫进行计数,以确保感染成功(图 1A5)。
- 用琼脂糖密封肠道以防止其变干。在这里,由于 0.5%-0.9% 琼脂糖的低温熔点约为 38 °C,因此用于保护敏感的肠道组织免受灼伤。 将 1 mL 琼脂糖吸入移液管中,小心地淋在肠道上,移液器吸头和组织之间轻轻接触,使约 0.5 mm 厚的薄层完全包围组织(图 1A6)。
- 在室温下用PBS(10%)密封在琼脂糖中的样品填充培养皿,然后用盖子关闭它们。可以直接对样品进行成像,也可以将它们放在保温箱中的冰上排队进行测量。将第一个制备的样品放在显微镜物镜下设置为 37 °C 的加热板上(图 1A8)。
2. 成像
注:用于在感染和健康的十二指肠组织样本中执行 NAD(P)H-FLIM 的显微镜系统由 图 2 和 材料表中列出和描述的设备组成。使用 ImSpector 208 作为所有使用模块的控制软件。
- 要初步找到感兴趣区域 (ROI),请将样品放在物镜下方的培养皿中,然后沿 x 和 y 平面手动移动,通过使用宽场荧光显微镜模式进行目视检查来找到合适的 ROI。
- 通过在软件中切换模式,使用 PMT 检测或时间相关单光子计数 (TCSPC) 检测将成像系统切换到双光子激发。请特别注意确保环境黑暗且无振动,将显微镜封闭在避光窗帘中,并尽可能使用气动悬挂式光学平台。
- 通过单击 透镜系统 图标并转动鼠标滚轮以改变 z 位置,使用控制软件将物镜浸入包含样品的培养皿中。
- 通过在软件的波长面板中输入所需的波长,将激光调谐到 765 nm,并将最大激光功率设置为 10%,相当于物镜下的 30 mW。如果需要,调整激光功率。在增益低于 40%(~30 mW 至 100 mW)的情况下进行测量,以避免组织光损伤和不必要的代谢变化。
- 在软件中,将 z 载物台的步长设置为 2 μm,这对应于显微镜在组织中 765 nm 处的轴向分辨率。
- 在振镜面板中将线扫描频率设置为 400 Hz,导致像素停留时间为 4.95 μs。
- 通过从软件的下拉菜单中选择平均数字,将图像平均设置为 2 到 4 张图像(即,扫描 ROI 两到四次以获得更平滑的图像)。通过这种方式,总像素停留时间增加到 9.9-19.8 μs,从而在分析过程中获得更高的信噪比 (SNR)。
- 通过在软件的 FOV 面板中选择参数,将图像视野 (FOV) 设置为 505 像素 x 505 像素 (500 x 500 μm²)。
- 对于每次测量,请提前校准模块时间,以确保 TCSPC 电子设备的最佳功能,方法是单击 TCSPC 硬件菜单中校准窗口中的 初始化 。
- 使用快速示波器从外部调整快速光电二极管以检测 Ti:Sa 激光脉冲序列。
注意:光电二极管的信号用于触发 TCSPC 相对于激发脉冲的 hPMT 检测和光子计数。每 12.5 ns(即两个连续激光脉冲之间的时间)重复检测,对应于 Ti:Sa 激光的重复率 (80 MHz)。 - 定义测量深度的范围,首先将平均设置为 1,将激光功率增益设置为 ~10%,然后启动视频模式(单击 视频 按钮),通过再次单击 镜头系统 图标来搜索仍然可以采集信号的深度,并使用鼠标滚轮进入样品,同时逐渐增加增益。
- 对于在 FLIM 模式下测量的组织样品的 NAD(P)H-FLIM 图像,将系统设置为 765 nm,最大标称功率为 100 mW。用杂交 PMT 以 97% 的增益检测 NAD(P)H (466/60 nm) 的荧光信号,该增益在软件中设置。TCSPC 模块对 227 个时间窗口(bin)上的光子进行计数,每个时间窗口 55 ps。
- 在软件中测量过程中右键单击 显示的图像 ,然后选择 Display > T-PROFILE ) 以可视化系统采集的衰减曲线。
- 记下衰减曲线中激光脉冲之前的 bin 数,以便以后分析。
- 通过单击 Start measurement 来获取数据。
注意:数据必须与特定格式匹配。以任何组织深度记录的每个切片相距 2 μm,面积为 500 μm x 500 μm,切片以 227 x 55 ps(图 3A)时间点作为 500 μm x 500 μm 切片堆栈获取。每个体素都包含一个空间分辨(在 x 平面和 y 平面中)光子到达时间直方图。这表示荧光衰减(图 3B)。通常测量的体积必须采用尺寸为 ~500 μm x 500 μm x 100-300 μm 和 227 个区间的超堆栈形式,其中 z 平面中的每个切片都包含如上所述的时间相关强度数据。对于一组示例性测量数据,这将产生 (505 x 505 x 227) x 100 像素(16 位),对应于大约 4 GB。
3. 数据分析
注意:对于 NAD(P)H-FLIM 图像的相量分析,用于计算寿命的程序是 Python33 中自定义编写的代码。
- 在 Spyder IDE 上将 Anaconda 与 Python 3.7 发行版一起使用(请参阅 材料表)。该代码使用 Python 的标准库。
- 将代码加载到 IDE 中,然后执行。此时将打开一个文件路径对话框。
- 选择包含要分析的原始数据的文件夹。该代码被编程为在分析之前通过 Tkinter 用户输入对话框选择三个参数,该对话框带有复选框、下拉菜单和文本字段。
- 在输入对话框中选择偏移量。将偏移量确定为时间堆栈中未分析的第一个切片的数量。
注:该参数在激发脉冲之前剪切时间点。校准系统和触发二极管后,该值必须大约为 5 个切片或激光激发前的死区间数,如步骤 2.14 中所述。 - 在输入对话框的下拉菜单中选择相量图的表示形式。在这里,从下拉菜单中选择数据点的外观(云状或地形),以及半圆时间轴的轴刻度(酶或时间 [ps])作为选项。
- 点击 OK。
- 检查代码是否从 NAD(P)H-FLIM z 堆栈计算了两种类型的信息。
- 首先,通过 TCSPC 堆栈的折叠来测量每个体积切片的空间信息,其中 227 个时间分辨光子计数直方图被投影到称为强度投影图像的单个切片上(图 3G),以及来自指数衰减曲线的基于傅里叶的分析(如 Leben 等人描述的 18),以及来自每个体积切片中每个像素的归一化实部和虚部(图 3C)。
- 从真实和虚构图像中,获得相量图(图 3D)和平均荧光寿命 (t) 图像(每个体素的颜色编码、空间分辨平均衰减常数)(图 3E)。
注:如引言中所述,NAD(P)H 与酶结合时的荧光寿命由辅酶与相应酶的结合位点决定。 - 通过生成每个像素的相位向量与未结合的 NAD(P)H 的相位向量之间的向量并将其投影到相量图中的半圆上,确定相应酶对代谢活性的贡献。半圆表示纯化合物中荧光寿命的所有可能的单指数衰减。
- 使用先前生成的 NAD(P)H 与最丰富的 NAD(P)H 依赖性酶结合的荧光寿命参考18,33(补充图 1),并计算这些酶激活的概率。
注意:最主要的酶(即,计算出最高激活概率的酶)的颜色代码归因于每个像素,从而产生酶图(图 3F)。
- 根据游离(未结合)与酶结合的 NAD(P)H 的比率,特别是从 450 ps 时游离 NAD(P)H 与酶结合状态之间的相量图中的矢量比率,计算一般代谢活性作为 0% *仅未结合的 NAD(P)H) 和 100%(仅酶结合的 NAD(P)H)之间的百分比。通过将代谢活动 (0%-100%) 值归因于每个像素,生成(颜色编码的)活动图(图 3F)。
- 将生成的地图与强度图像叠加以获得额外的形态学信息。在这里,使用 ImageJ 宏(参见 材料表)将酶或活性图的色调和饱和度与强度图像的亮度叠加。
- 确保宏遍历整个堆栈。对于堆栈中的每个切片(深度),它必须将强度图像和所选映射分别拆分为 HUE、饱和度和 BRIGHTNESS(HSB 堆栈>图像 > 类型)。
- 将两个 HSB 堆栈的主通道(图像>堆栈>堆栈到图像)分开。
- 关闭强度堆栈的 HUE 和 SATURATION,以及映射堆栈的 BRIGHTNESS (close(slicename_HUE)、close(slicename_BRIGHTNESS)、...)。
- 重新组合剩余的通道以形成一个新的 HSB 堆栈,该堆栈由来自感兴趣图切片的 HUE 和 SATURATION 以及来自强度图像的 BRIGHTNESS(图像 > 堆栈>要堆叠的图像)。
- 将图像类型更改为 RGB 以获得更好的视觉外观(图像>类型> RGB 颜色)。
4. 组织分割
注意:使用预先训练的基于 U-Net 的网络(ILASTIK,参见 材料表)分别分割肠道宿主和线虫组织,此外,还分割宿主中的上皮和固有层以及线虫中的高 NAD(P)H 荧光信号区域和低 NAD(P)H 荧光信号区域。
- 打开 ILASTIK,然后选择新项目和像素分类。
- 通过单击添加新 >添加单独的图像,从步骤 3.7.1 加载先前计算的强度投影。将多个测量值中的随机切片加载到容器中。
- 单击 Feature selection >选择要素。添加 sigma 值 50(无单位权重),然后勾选所有功能以激活。
注意:虽然 sigma 值决定了特征的权重,但特征决定了网络识别边缘、形状、纹理或颜色的类和能力。 - 单击 Training,然后单击标签以根据要分割的组织(例如,上皮、背景等)命名标签。选择一个标签,然后为图像中与该标签对应的所有像素着色。
- 对其他标签重复上述步骤。
- 对加载的数据集中大约一半的图像重复此作。
- 单击 Live Update,然后让模型预测剩余未标记图像的标签。
- 通过重新标记错误估计的分段,更正大约一半的单独预测和未标记的图像,然后重复 实时更新。
- 重复此步骤,直到网络学会正确估计所需的感兴趣组织;确保标签名称附近左侧显示的网络的置信度介于 95% 和 98% 之间。
- 按照步骤 3.11 中的说明加载新数据集,这次包含来自一次测量的所有切片,然后单击 Prediction Export。
- 在 “Source dropdown” 菜单中,选择 Simple Segmentation (简单分段);在“格式”中,选择要 tiff 格式的输出;,然后在“Choose Export Image Settings(选择导出图像设置)”中,选择输出的 保存路径 。单击 export。
- 这将为感兴趣的组织类型创建二进制掩码。由于它们由切片组成,其中感兴趣的分割组织的像素值为 1,其余组织的像素值为 0(图 3H),因此只需将二进制掩码与步骤 1.1 和步骤 1.4-1.7 中列出的生成数据相乘即可。这导致掩码数据(图 3 I、J、M)。此步骤使用 ImageJ(处理 > 图像计算器>乘法)。
- 对于每个体积切片,从遮罩强度图像计算信噪比 (SNR)。为此,请使用 ImageJ 宏。创建您的程序,以便它从分割和蒙版图像中计算信噪比 (SNR)。
- 使用步骤 3.21 中的分割背景作为背景 (BG),使用感兴趣的分割组织作为信号 (SIG),方法是使用两个嵌套的 for 循环迭代每个切片的每个像素,其值大于 0。使用以下公式33 计算 SNR:
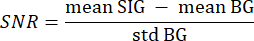
其中,均值 SIG 是指信号直方图的平均值,均值 BG 是指背景直方图的平均值,标准差 BG 是指背景直方图的标准差。 - 丢弃 SNR 值小于 5 的体积切片,从进一步分析中丢弃。
- 使用步骤 3.21 中的分割背景作为背景 (BG),使用感兴趣的分割组织作为信号 (SIG),方法是使用两个嵌套的 for 循环迭代每个切片的每个像素,其值大于 0。使用以下公式33 计算 SNR:
- 使用 Python 脚本18,33,通过对酶进行切片求和平均,然后将它们归一化为 100 μm³ 组织体积,从每种组织类型的酶图中创建一个酶频率图(图 3K,N)。
- 对脚本进行编码,使其加载掩码酶图。
- 迭代每个像素,计算相同酶的像素总和,然后除以分析的体积(所有像素的总和)。
- 将计算出的代谢状态丰度分组,并通过除以每组中的酶数量来取平均值。编写代码,将围绕 LDH 分组的代谢状态归因于厌氧糖酵解样途径,将围绕 PDH/IDH/GAPDH 分组的酶活性归因于需氧糖酵解/oxPhos 样途径,将 NADPH 氧化酶活性(NOX 活性)归因于用于防御的氧化爆发/氧化应激(图 3L,O)。
- 使用 Python 中的 matplotlib 库通过箱线图可视化数据。
- 通过运行分析>测量,使用 ImageJ 宏计算掩蔽活动图(图 3F)中的组织活动,作为值大于 0 的所有像素的每个体积切片的平均值。
- 使用 ImageJ/FIJI 并排可视化所有生成的图像。
结果
使用当前的 NAD(P)H-FLIM 程序 28,29,33 结合所描述的相量分析方法,在感染小鼠肠道线虫后第 6 天、第 10 天、第 12 天和第 14 天测量健康和感染十二指肠细胞的代谢活性和代谢途径多回线虫。
NAD(P)H-FLIM 揭示的切除十二指肠中保留的肠道组织活力
为了研究 离体 组织活性并确定与活生物体中的组织相比,切除的组织样品保持代谢活性的时间,对 (i) 新鲜切除或 (ii) 在冰上保存 3 小时的健康小鼠切除的十二指肠组织进行 NAD(P)H-FLIM,并将结果与已发表的麻醉小鼠肠道中获得的 NAD(P)H-FLIM 数据进行比较32.切除的十二指肠的制备和成像实验按照样品制备方案(步骤 1)中的描述进行。
在 3 小时内和四种不同的组织深度(绒毛尖端:~0 μm,上绒毛:~-50 μm,下绒毛:~-100 μm,绒毛基部:~-200 μm)测量 NAD(P)H 的荧光寿命。从以这种方式获得的 NAD(P)H-FLIM 数据中,生成了活性图,显示了绒毛不同区域随时间推移的一般代谢活动。一般代谢活性(游离到酶结合的 NAD(P)H))被认为是组织退化的指标(图 4A,B)。
使用另一段肠道对相似组织样本进行第二系列测量。在这里,不是在牺牲后立即进行测量,而是在制备后将组织置于冰上 3 小时,以模拟实际实验中置于冰上样品的最大等待时间。使用加热板预热 ~15 分钟后,在不同组织深度进行成像(图 4C,D)。在先前描述的两种设置中测量的代谢活性与在小鼠肠道活体条件下通过 NAD(P)H-FLIM 测量的代谢活性非常一致32。
使用 NAD(P)H-FLIM 检测急性感染过程中宿主肠道组织和 多回嗜 血杆菌中的不同代谢和酶活性
从感染后第 6 天开始的病程中,当蠕虫幼虫在粘膜下层发育,在第 10 天爆入管腔并浸润肠道生态位时,观察到寄生虫的整体代谢活性增加。从第 6 天的更休眠状态 58.0% ± 2.2%,代谢活性稳步增加到 73.1% ± 5.9%。
寄生虫在感染早期的初始状态反映在其代谢指纹上;具体来说,在第 6 天,能量产生在厌氧和好氧途径之间平衡,表明即将突破管腔。然后能量产生转变为强厌氧糖酵解样行为,可能是由于迁移到管腔的能量需求增加(第 10 天),并且寄生虫在稍后(测量到第 14 天)显示出氧化磷酸化/需氧糖酵解样能量产生途径。宿主的一般代谢活性与 74.0% ± 7.6% (第 6 至 14 天) 的健康状态相当,而宿主组织中的代谢途径在急性感染过程中主要是有氧/氧化磷酸化。
寄生虫的防御反应基于 DUOX2 激活,作为该物种中 NADPH 氧化酶家族的唯一成员。这种反应在感染的早期阶段,即第 6 天可以忽略不计,但在急性感染的后期时间点会增加因此,我们检测到 DUOX2 激活从第 10 天开始,在管腔迁移后,以及整个感染的急性期急剧增加。(图 5E、F)
另一方面,与感染后第 10 天的健康状态相比,宿主的炎症行为急剧增加,寄生虫出现在管腔中,并在感染后第 14 天停止。这种行为与寄生虫在感染的后期(慢性)阶段在宿主免疫系统中诱导调节表型的事实相似 4,6,7(图 5A-D)。

图 1:使用多光子激光扫描显微镜制备用于 NAD(P)H-FLIM 的小鼠肠道组织。 (A) 准备工作需要取出肠子并将其修剪成约 1 厘米长 (1),在培养皿上涂抹组织胶并将管子放在上面 (2),用钝剪刀纵向剪断管子靠近底部 (3), 展开管子,用钝镊子打开管腔侧 (4),在立体显微镜下计数蠕虫 (5),用低浓度琼脂糖将组织密封在约 0.5 mm 厚的薄层中 (6),用 10% PBS 填充培养皿 (7),并对组织样品进行成像或将其置于冰上等待处理 (8)。(B) 从切除十二指肠到放置在显微镜下,在几分钟内处理单个组织样本的时间轴。样品的测量时间包括找到 ROI,同时将加热元件上的冷却样品加热至 37 °C,然后获取图像。(C) 45 分钟的平均测量时间(随后根据数据确定)绘制在 (C) 中,对照所研究的四个感染阶段以及健康组织。 请单击此处查看此图的较大版本。

图 2:实验装置示意图。 该系统由可调谐 Ti:Sa 激光钛蓝宝石 LASER Chameleon ULTRA II(690-1,080 nm,80 MHz,脉冲宽度 140 fs)和商用扫描头 Trimscope II 组成。水浸物镜(20 倍,NA 1.05)聚焦激发光。由二向色镜、干涉滤光片(525/50 nm、593/40 nm、655/40 nm)和 PMT 组成的系统检测荧光信号。使用通道 466/60 nm 的混合 PMT (GaAsP) (hPMT) 检测荧光寿命数据,该通道由冷却模块 (CM) 冷却并连接到时间相关单光子计数模块;此过程包括检测 55 ps 分箱内的光子,并使用 250 ps FWHM (TCSPC) 的高斯形仪器响应函数采集 9 ns 的数据。为了触发 TCSPC,5%-8% 的初始脉冲强度从激发光束中分离出来,并在超快光电二极管 (PD) 中重定向。为了在没有 TCSPC 的情况下以实时模式获得双光子激发,使用带有彩色滤光片(525 nm、593 nm、655 nm)的 PMT。宽带汞蒸气灯 (MV) 和眼通路用于观察用于视觉荧光显微镜的探针 (OP) 和定位样品,并且 ROI 是通过步长为 1 μm (IS) 的活体阶段 (IS) 找到的。 请单击此处查看此图的较大版本。
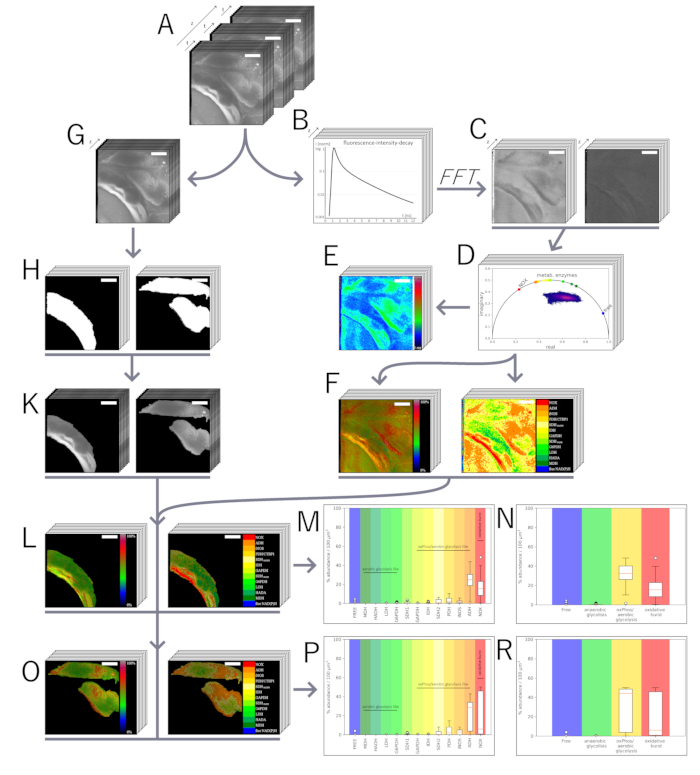
图 3:时域 NAD(P)H-FLIM 数据的相量分析。(A) 原始数据在四个维度上获取:空间信息(x、y、z;xy 为 505 像素 = 500 μm)和 TCSPC 数据(t 有 227 个时间点,每个时间点 55 ps)。(B) 如 (A) 所示,沿 t 的 TCSPC 原始数据中的每个像素都包含 227 个时间区间(每个 55 ps)中激发脉冲后的荧光强度衰减。时域 TCSPC 数据是高斯预卷积的,并通过计算归一化的离散傅里叶变换数值(相量方法)转移到虚拟相域。这导致图像中每个像素的复数,包含实部和虚部,这可以被分割并表示为每个切片测量的两个强度编码图像:(C,左)实部和(C,右)虚部。(D) 实部和虚部提供相量图中的坐标, 其中,图中的每个点都有一个空间对应物及其来自 (B) 的衰减信息。半圆时间轴表示单指数生命周期。(E) 使用连续傅里叶(反向)变换计算荧光寿命并从 (C) 进行颜色编码。这相当于 (B) 中强度衰减曲线的单指数分析。按照先前开发的分析框架,生成 (F, 右) NAD(P)H 依赖性酶的图谱和 (F, 左) 酶活性图谱。(G) 同时,TCSPC 原始数据被折叠到强度投影中,丢失了时间信息,只保留了空间信息。(H) 由此,通过经过训练的分割算法使用 ILASTIK 为所需的感兴趣组织生成掩模,这些算法与强度投影叠加;此处显示了感染后第 14 天的 (I, 左) 寄生虫和 (I, 右) 宿主的示例。(J,左)(J,右)寄生虫和 (M) 宿主组织的掩蔽活性图和掩蔽酶图。从掩蔽图中,通过计数酶并将丰度百分比归一化至 100 μm3 (K) 来生成寄生虫的酶频率图。在 Liublin 等人 33 的分析之后,根据 (L) 寄生虫和 (N,O) 宿主组织的有氧糖酵解和氧化磷酸化/厌氧糖酵解的代谢途径,以及氧化爆发和代谢失活状态(未结合、游离 NAD(P)H)对酶丰度进行分组。所有比例尺均为 100 μm。请单击此处查看此图的较大版本。

图 4:外植组织活力的预评估。 (A) 健康 C57/bl6 小鼠新鲜外植十二指肠的酶活性图谱,如第 3 节所述。制备后,在 37 °C 的加热板上以 20 分钟的间隔直接对组织进行成像,总共 106 分钟。 在四种不同的组织深度进行测量:绒毛尖端 (0 μm)、中上部 (-50 μm)、中下部 (-100 μm) 和绒毛基部 (-200 μm)。(A) 中显示的示例是制备后 5 分钟和 68 分钟绒毛的中上部区域。对于每个测量时间点,根据固有层和上皮组织的分段活性图计算所有深度平均的酶活性,并在测量时间内绘制。(B) 该图显示了 106 分钟内处死和制备后外植组织的活力和变性行为。如第 1 节所述,为了涵盖将制备的样品放在冰上排队进行测量的组织变性行为,制备了来自健康 C57/bl6 小鼠的新鲜移植的十二指肠并在冰上储存 3 小时,以模拟实验期间发生的最大等待时间。(C) 然后将组织加热至 37 °C,并用 NAD(P)H-FLIM 成像,并生成活性图。根据 (B) 中描述的分段数据计算组织的相应平均活性,并且 (D) 这导致固有层活性为 57.8% ± 4.6%,上皮活性为 68.5% ± 5.7%)。所有比例尺均为 250 μm。 请点击此处查看此图的较大版本。

图 5:宿主绒毛和线虫组织的 NAD(P)H-FLIM,表明宿主和寄生虫的代谢状态和优先代谢途径。(A) 根据健康小鼠十二指肠中上皮 (EP) (78.4% ± 5.5%) 和固有层 (LP) (76.3% ± 5.2%) 的掩蔽活动图确定的平均一般代谢活动。(B) 根据我们的分析,根据代谢途径对酶丰度进行分组,包括健康小鼠十二指肠中上皮 (EP) 和固有层 (LP) 的需氧糖酵解、氧化磷酸化/厌氧糖酵解以及氧化爆发。(C,D)与 (A) 和 (B) 类似,对急性感染过程中获得的宿主组织进行了分析。第 6 天宿主组织的平均代谢活性确定为 74.0% ± 7.6%,第 10 天为 72.4% ± 10.5%,第 14 天为 73.5% ± 11.4%。(东、女)与 (C) 和 (D) 一样,在疾病的急性期过程中分析了寄生虫组织。寄生虫在第 6 天的平均代谢活性为 (58.0% ± 2%,第 10 天为 61.1% ± 9.9%,第 12 天为 64.5% ± 5.8%,第 14 天为 73.1% ± 5.9%。使用方差分析检验进行统计分析(ns p > 0.05,*p < 0.05,**p < 0.01,***p < 0.001)。请单击此处查看此图的较大版本。
补充图 1:NAD(P)H 结合状态依赖性荧光寿命。 (A I) 未结合(游离)NAD(P)H 在时间轴上的时间点 0 被光子吸收激发时的示意图及其在 450 ps 时的结合态特异性荧光寿命 (τ)。(A II) NADH 与 LDH 和 (A III) PDH 结合,荧光寿命分别为 1,600 ps 和 2,470 ps,NADPH 与 NADPH 氧化酶 (NOX) 结合 (A IV),其荧光寿命为 3,650 ps。(B) NAD(P)H 与最丰富的酶结合的特征荧光寿命表。 请点击此处下载此文件。
讨论
方案中的关键步骤发生在准备期间和查找 ROI 时。部分消化食品的纤维对成像提出了挑战,主要是由于纤维的内源性发光与 NAD(P)H 荧光重叠,但也由于它们的谐波产生信号。找到没有粪便的 ROI 非常重要。我们的目标是避免测量含有粪便的区域。避免洗涤,因为这会影响脆弱绒毛的完整性并影响粘液粘度。此外,每个样品最好在整个制备过程中持续约 5-10 分钟,以避免组织变性和干燥。此外,使用的低浓度琼脂糖模拟肠粘液的粘度34。因此,可以确保在测量过程中蜗杆自由移动时绒毛不会塌陷。测量持续时间不得超过 45 分钟(图 1A、B 和图 1A、B、C),以保持细胞代谢成像的最佳条件。因此,感兴趣区域 (ROI) 搜索的时间不应超过 15 分钟。然而,为了确保保存在冰上的组织在加热板上加热到所需的 37 °C,应在 ~10 分钟后进行成像。成像通常持续 45 分钟(参见 TCSPC 数据分析以在测量前和测量期间控制组织活力;图 1C 和图 4)。在寻找合适的投资回报率时,确保物镜下十二指肠组织的管腔侧至少有 20%(感染后第 10-14 天)被蠕虫组织覆盖,并且寄生虫的运动是可见的。对于第 6 天和健康的个体,合适的 ROI 定义为在制备后保持组织完整性(通常远离样品边界)。这确保了分割模型的最佳训练条件,并为进一步分析提供了足够的数据点。对于从第 10 天开始的感染阶段,当蠕虫突破到管腔并附着在绒毛上时,数据仅根据背景和具有高酶活性的寄生虫和肠道组织进行分割,因为在蠕虫存在的情况下,绒毛由于筑巢而大多是非结构化的,绒毛的组织完整性不再存在。对于第 6 天移植的组织和来自健康小鼠的组织,组织完整性允许分割固有层以及上皮与背景。因此,强烈建议针对这些条件使用单独训练的模型。该信号使高度自发荧光材料的检测器过载,偶尔会在测量体积内累积。在这项工作中,在软件的整个成像深度上使用了动态激光功率控制。
该技术的局限性与激发体积的物理限制有关。先前确定物镜的轴向分辨率在 850 nm 处为 1.3 μm28,但由于它通常会在组织中降解,我们发现 2 μm 的步长是合适的。FOV 为 0.5 mm x 0.5 mm,每个切片为 505 像素 x 505 像素。物镜的横向分辨率为 ~ 350 nm;然而,由于分辨率下降24,28,发现 ~ 1 μm 的像素尺寸就足够了。这意味着测量遵循随机方法。获得的衰变曲线由所有衰变曲线的总和组成,这意味着 ~1 x 1 x 2 μm3 的退出体积内的所有荧光寿命。随时间记录的每个体素(测得的荧光寿命或 TCSPC 堆栈)的光子计数遵循一条多指数衰减曲线或多指数衰减曲线的线性组合的形式,这很难预测。使用无模型的归一化基于傅里叶的分析来分析每个体素中的复杂荧光衰减曲线,称为相量分析。预先使用高斯滤光片 (σ2) 对堆栈进行卷积,以减少时间分辨荧光图像中的噪声。空间分辨率的损失是可以接受的,并且是为了获得更好的荧光寿命分辨率而进行的权衡。
帧采集时间在 2.5 秒到 7 秒(平均)的范围内,典型 z 堆栈的采集时间为 500 x 500 x 200 μm³(505 x 505 x 101 体素)需要 ~ 450 秒(7.5 分钟)。这些时间通常会导致在一次测量期间寄生虫导致移动伪影。在这种情况下,数据仍然可用。有时,寄生虫在测量过程中会移动到测量体积之外。
使用感染线虫 H. polygyrus 的小鼠切除的小鼠十二指肠组织的 NADH 和 NADPH 荧光寿命成像以及我们之前发表的分析框架,使用 NAD(P)H 与频繁的 NAD(P)H 依赖性酶结合的参考荧光寿命,可视化和分析宿主肠道组织和寄生虫组织中的一般代谢活性和不同的代谢途径;该分析揭示了两者之间的代谢串扰。或者,如果对有关整体代谢活动或 NADH 和 NADPH 之间平衡的信息感兴趣,则可以使用普遍适用的稳健算法和软件例程22,23。
此处介绍的方案和已建立的分析程序代表了一种适用于其他器官和其他类型的病理学的测量代谢和代谢途径的通用方法 13,15,16,17,19,20。
例如,与使用细胞术进行单细胞分析相比,该方法的优雅之处在于可以在生物系统中以最少的干预和无标记方式获得有关代谢途径的亚细胞空间信息,并且可以额外使用标记物或染料以获得更高的信息密度。由于选择了啮齿动物中天然存在的寄生虫,以及对肠道组织的初步检查及其在活力方面与活组织的可比性,因此对生物组织和与之相关的感染过程的干扰是最小的。通过适当的分割和标准化,可以在第四个维度(时间)上生成可比较的测量值。
披露声明
作者声明没有竞争性的经济利益。
致谢
我们感谢 Robert Günther 的出色技术支持。德国研究委员会 (DFG) 根据 Grant SPP2332 HA2542/12-1 (S.H.)、NI1167/7-1 (R.A.N.)、HA5354/11-1 (A.E.H.) 和 RA2544/1-1 (S.R.)、Grant SFB1444、P14 (R.A.N., A.E.H.)、Grant HA5354/8-2 (A.E.H.) 以及 Grant GRK2046 B4 和 B5 (S.H., S.R.)和 HA2542/8-1 (S.H.) 得到高度认可。W.L. 获得了柏林应用科学学院医学物理/物理工程博士学位。
材料
| Name | Company | Catalog Number | Comments |
| Agarose | Thermo fisher | J32802.22 | ultra pure |
| Blunt scissors | FST fine science tools | 14108-09 | blund-blund 14 cm |
| Bodipy c12 | thermo fisher | D3822 | 1 mg solid |
| Control units, diode, TCSPC | LaVision Biontech | custom | TrimScope II |
| DMSO | Thermo fisher | D12345 | 3 mL |
| Filters | Chroma | 755 | 466 ± 20, 525 ± 25, 593 ± 20, 655 ± 20 nm |
| Foliodrape sheet | Hartmann | 277500 | |
| Gloves | Sigma-Aldrich | Z423262 | nitril |
| Halogen torch | Leica | This item has been phased out and is no longer available | KL 1500 LCD |
| hPMT | Hamamatsu, Germany | H7422 | GaAsP |
| Ilastik | Netlify | free Software | Java Backend |
| ImageJ | National Institutes of health | free Software | FIJI - standard plugins |
| Imspector | LaVision Biontech | - | Vers. 208 |
| Intravital stage | LaVision Biontech | custom | TrimScope II |
| Lens system 20x | Zeiss | custom | W-plan-apochom 20x Waterimmersion NA 1.05 |
| Mercury vapor torch | LaVision Biontech | custom | |
| microbrush | Fisher scientific | 22-020-002 | 85 mm |
| Microscope | LaVision Biontech | custom | TrimScope II |
| Oscilloscope | Rhode & Schwarz | 1326.2000.22 | |
| PBS | Sigma-Aldrich | AM9624 | 0.5 L |
| Petri dish | Sigma aldrich | P5606 | 40 x 15 mm |
| Pipette | thermo fisher | 4651280N | Einkanalpipette |
| Pipette tips | thermo fisher | 94056980 | Spitzen mit Filter |
| PMT | Hamamatsu, Japan | H7422 | GaAsP |
| Python | Python Software foundation | free Software | Anaconda 3.7 Spyder IDE, standard librarys with KYTE |
| Sterio microscope | Leica | This item has been phased out and is no longer available | M26, 6.3x zoom |
| Ti:Sa LASER CHAMELION ULTRA II | Coherent, APE | - | 690-1080 nm tunable, 80MHz |
| Tissueglue | 3M | 51115053603 | 3 mL |
| Tweezers | FST fine science tools | 11049-10 | blund, graefe, angeled |
| Tweezers | FST fine science tools | 91197-00 | Dumont, curved |
参考文献
- Hotez, P. J., Fenwick, A., Savioli, L., Molyneux, D. H. Rescuing the bottom billion through control of neglected tropical diseases. Lancet. 373 (9674), 1570-1575 (2009).
- Sartorius, B., et al. Prevalence and intensity of soil-transmitted helminth infections of children in sub-Saharan Africa, 2000-18: A geospatial analysis. The Lancet Global Health. 9 (1), e52-e60 (2021).
- Affinass, N., et al. Manipulation of the balance between Th2 and Th2/1 hybrid cells affects parasite nematode fitness in mice. European Journal of Immunology. 48 (12), 1958-1964 (2018).
- Hartmann, S., et al. Gastrointestinal nematode infection interferes with experimental allergic airway inflammation but not atopic dermatitis. Clinical & Experimental Allergy. 39 (10), 1585-1596 (2009).
- Hepworth, M. R., et al. Mast cells orchestrate type 2 immunity to helminths through regulation of tissue-derived cytokines. Proceedings of the National Academy of Sciences of the United States of America. 109 (17), 6644-6649 (2012).
- Rausch, S., et al. Establishment of nematode infection despite increased Th2 responses and immunopathology after selective depletion of Foxp3+ cells. European Journal of Immunology. 39 (11), 3066-3077 (2009).
- Rausch, S., et al. Parasitic nematodes exert antimicrobial activity and benefit from microbiota-driven support for host immune regulation. Frontiers in Immunology. 9, 2282 (2018).
- Steinfelder, S., Rausch, S., Michael, D., Kuhl, A. A., Hartmann, S. Intestinal helminth infection induces highly functional resident memory CD4(+) T cells in mice. European Journal of Immunology. 47 (2), 353-363 (2017).
- Whelan, R. A., et al. A transgenic probiotic secreting a parasite immunomodulator for site-directed treatment of gut inflammation. Molecular Therapy. 22 (10), 1730-1740 (2014).
- Ziegler, T., et al. A novel regulatory macrophage induced by a helminth molecule instructs IL-10 in CD4+ T cells and protects against mucosal inflammation. The Journal of Immunology. 194 (4), 1555-1564 (2015).
- Grantham, B. D., Barrett, J. Amino acid catabolism in the nematodes Heligmosomoides polygyrus and Panagrellus redivivus. 2. Metabolism of the carbon skeleton. Parasitology. 93 (Pt 3), 495-504 (1986).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. Journal of Cell Science. 134 (9), 1-17 (2021).
- Mossakowski, A., et al. Tracking CNS and systemic sources of oxidative stress during the course of chronic neuroinflammation. Acta Neuropathologica. 130 (6), 799-814 (2015).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nature Communications. 12, 4580 (2021).
- Bayerl, S., et al. Time lapse in vivo microscopy reveals distinct dynamics of microglia-tumor environment interactions-a new role for the tumor perivascular space as highway for trafficking microglia. Glia. 64 (7), 1210-1226 (2016).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. Multiphoton microscopy of endogenous fluorescence differentiates normal, precancerous, and cancerous squamous epithelial tissues. Cancer Research. 65 (4), 1180-1186 (2005).
- Leben, R., Kohler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free NAD(P)H fluorescence lifetime imaging. International Journal of Molecular Sciences. 20 (22), 5565 (2019).
- Chacko, J. V., Eliceiri, K. W. Autofluorescence lifetime imaging of cellular metabolism: Sensitivity toward cell density, pH, intracellular, and intercellular heterogeneity. Cytometry. 95 (1), 56-69 (2019).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5 (1), 3936 (2014).
- Blacker, T. S., Duchen, M. R. NAD(P)H binding configurations revealed by time-resolved fluorescence and two-photon absorption. Biophys. J. 122 (7), 1240-1253 (2023).
- Niesner, R., et al. Selective detection of NADPH oxidase in polymorphonuclear cells by means of NAD(P)H-based fluorescence lifetime imaging. Journal of Biophysics. 2008, 602639 (2008).
- Vishwasrao, H. D., Heikal, A. A., Kasischke, K. A., Webb, W. W. Conformational dependence of intracellular NADH on metabolic state revealed by associated fluorescence anisotropy. Journal of Biology and Chemistry. 280 (26), 25119-25126 (2005).
- Babior, B. M. NADPH oxidase: An update. Blood. 93 (5), 1464-1476 (1999).
- Hewitson, J. P., et al. Proteomic analysis of secretory products from the model gastrointestinal nematode Heligmosomoides polygyrus reveals dominance of venom allergen-like (VAL) proteins. Journal of Proteomics. 74 (9), 1573-1594 (2011).
- Rakhymzhan, A., et al. Synergistic strategy for multicolor two-photon microscopy: Application to the analysis of germinal center reactions in vivo. Scientific Reports. 7, 7101 (2017).
- Bremer, D., et al. Method to detect the cellular source of over-activated NADPH oxidases using NAD(P)H fluorescence lifetime imaging. Current Protocols in Cytometry. 80, 9.52.1-9.52.14 (2017).
- Leben, R., et al. Phasor-based endogenous NAD(P)H fluorescence lifetime imaging unravels specific enzymatic activity of neutrophil granulocytes preceding NETosis. International Journal of Molecular Sciences. 19 (4), 1018 (2018).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysics Journal. 94 (2), L14-L16 (2008).
- Lindquist, R. L., Bayat-Sarmadi, J., Leben, R., Niesner, R., Hauser, A. E. NAD(P)H oxidase activity in the small intestine is predominantly found in enterocytes, not professional phagocytes. International Journal of Molecular Sciences. 19 (5), 1365 (2018).
- Liublin, W., et al. NAD(P)H fluorescence lifetime imaging of live intestinal nematodes reveals metabolic crosstalk between parasite and host. Scientific Reports. 12, 7264 (2022).
- Nhu, N. T. Q., et al. Alkaline pH increases swimming speed and facilitates mucus penetration for Vibrio cholerae. Journal of Bacteriology. 203 (7), e00607 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。