Method Article
Generazione e caratterizzazione di astrociti derivati da cellule staminali pluripotenti indotte umane prive di ribonucleoproteina messaggero X fragile
In questo articolo
Riepilogo
Qui descriviamo un protocollo che facilita la differenziazione di cellule staminali pluripotenti indotte umane in astrociti funzionali specifici per il proencefalo. Ciò consente di studiare il ruolo delle cellule gliali nella patogenesi dei disturbi dello sviluppo neurologico, come la sindrome dell'X fragile, e di modellare altri disturbi cerebrali.
Abstract
La sindrome dell'X fragile (FXS), una delle principali cause ereditarie del disturbo dello spettro autistico e della disabilità intellettiva, è stata ampiamente studiata utilizzando modelli di roditori. Più recentemente, sono stati utilizzati anche sistemi modello derivati da cellule staminali umane per ottenere informazioni meccanicistiche sulla fisiopatologia della FXS. Tuttavia, questi studi si sono concentrati quasi esclusivamente sui neuroni. Inoltre, nonostante la crescente evidenza di un ruolo chiave della glia nella funzione neuronale nella salute e nella malattia, si sa poco su come gli astrociti umani siano influenzati dalla FXS.
Pertanto, in questo studio, abbiamo sviluppato con successo un protocollo che cattura le tappe spaziotemporali chiave dello sviluppo cerebrale e si allinea anche con il processo di gliogenesi. Insieme, questo offre un quadro utile per studiare i disturbi del neurosviluppo. In primo luogo, abbiamo modellato le cellule staminali pluripotenti indotte umane nel lignaggio neuroectodermico con doppio soppressore delle madri contro l'inibizione decapopentaplegica (SMAD) e piccole molecole. Successivamente, abbiamo utilizzato specifici fattori di crescita e citochine per generare cellule progenitrici astrocitiche (APC) di controllo (CTRL) e FXS derivate da pazienti. Il trattamento delle APC con fattore neurotrofico ciliare, una citochina differenziante, ha regolato e guidato le cellule progenitrici verso la maturazione astrocitica, producendo astrociti che esprimono proteine acide fibrillari gliali specifiche per il prosencefalo.
Abbiamo scoperto che questi astrociti sono funzionali, come evidenziato dalle loro risposte di calcio all'applicazione di ATP, e mostrano un metabolismo glicolitico e mitocondriale disregolato nella FXS. Nel loro insieme, questi risultati forniscono un'utile piattaforma sperimentale di origine umana per lo studio delle conseguenze cellulo-autonome e non-cellulo-autonome delle alterazioni della funzione astrocitaria causate da disturbi del neurosviluppo.
Introduzione
La sindrome dell'X fragile (FXS), una forma ereditaria comune di disabilità intellettiva e disturbo dello spettro autistico (ASD), è causata dalla mancanza di ribonucleoproteina messaggero dell'X fragile (FMRP) prodotta dal gene della ribonucleoproteina 1 del messaggero dell'X fragile (FMR1) (OMIM: #300624, https://www.omim.org/entry/300624). L'FMRP svolge un ruolo nella regolazione della traduzione dell'mRNA, nella formazione e nel trasporto dei granuli di mRNA e nella regolazione dell'espressione genica1 mediata dai microRNA. Pertanto, la perdita di FMRP influisce non solo sullo sviluppo del cervello, ma anche sulla funzione cerebrale adulta. Sia i livelli di trascrizione dell'mRNA di FMR1 che l'immunocolorazione per FMRP nel cervello hanno mostrato un'elevata espressione neuronale, insieme a un'espressione significativa anche nelle cellule gliali2. Tuttavia, la stragrande maggioranza degli studi precedenti su modelli animali di FXS si è concentrata principalmente sui neuroni e sulle aberrazioni nella loro funzione. Di conseguenza, si sa poco sul ruolo della glia in FXS3. Tradizionalmente considerate come cellule di "supporto passivo"4, si stanno accumulando prove che gli astrociti sono fondamentali nella mediazione di un'ampia gamma di funzioni neuronali 5,6, tra cui la promozione della sinaptogenesi7, il perfezionamento dei circuiti neurali in via di sviluppo8 e il riciclaggio dei neurotrasmettitori9. Parallelamente, vi è una crescente evidenza del ruolo degli astrociti nella patogenesi della malattia e molte condizioni neurologiche sono state associate alla disfunzione astrocitaria10.
Mentre gran parte del lavoro precedente che utilizzava modelli animali di FXS si concentrava sull'identificazione e la convalida di vari bersagli molecolari nei neuroni per il trattamento di FXS, questi risultati preclinici non hanno sempre portato a risultati clinici di successo. Inoltre, le battute d'arresto nei recenti studi clinici sottolineano anche la necessità di sistemi modello basati sull'uomo. I modelli di disturbi neurologici basati su cellule cerebrali derivate da cellule staminali umane offrono una potente strategia per colmare questo divario tra le intuizioni meccanicistiche degli studi sugli animali e il successo limitato con i risultati clinici per i pazienti. Tuttavia, solo una manciata di questi studi si è concentrata sugli astrociti e anche su astrociti di origine spinale. Questo, a sua volta, è rilevante alla luce degli studi che dimostrano che la struttura e la funzione degli astrociti variano tra le regioni del cervello11,12. Pertanto, una migliore comprensione dei cambiamenti indotti dalla malattia negli astrociti umani deve anche tenere conto di queste differenze specifiche della regione del cervello negli astrociti. Tuttavia, i modelli di disturbi dello sviluppo neurologico che utilizzano astrociti derivati da cellule staminali umane che sono specifici per il proencefalo rimangono relativamente poco esplorati13. Quindi, per iniziare a colmare queste lacune, descriviamo i protocolli per la generazione di astrociti specifici per il proencefalo da cellule staminali pluripotenti indotte (iPSC) derivate da pazienti portatori di mutazioni FXS; Inoltre, dimostriamo che gli astrociti sono funzionali e mostrano un metabolismo alterato.
Protocollo
Tutti gli esperimenti con cellule staminali pluripotenti indotte umane (hiPSC) (Tabella 1) sono stati eseguiti dopo aver ottenuto le appropriate approvazioni normative istituzionali. La Figura 1A rappresenta il protocollo di differenziazione completo delle hiPSC verso astrociti maturi specifici del proencefalo.
1. Manutenzione ed espansione delle hiPSC
- Un giorno prima di placcare le iPSC, rivestire una piastra a 6 pozzetti con Matrigel diluito 1:60 (una matrice extracellulare [ECM]) in Advanced Dulbecco's Modified Eagle Medium/Ham's F-12 (DMEM/F12) e conservare a 2-8 °C.
NOTA: Evitare di conservare le piastre rivestite con Matrigel per >5 giorni a 2-8 °C, poiché ciò potrebbe causare la degradazione delle proteine nella MEC nel tempo. Per evitare l'essiccazione della piastra, rivestire un minimo di 1 mL di Matrigel diluito 1:60 per un pozzetto di una piastra a 6 pozzetti e distribuirla uniformemente sulla piastra. Se si utilizza lo stesso giorno, conservare la piastra in un'incubatrice umidificata a 37 °C e 5% di CO2 per 1 ora. - Il giorno della coltura delle hiPSC, rimuovere il materiale di rivestimento e aggiungere 1 mL di terreno completo Essential 8 (terreno basale E8 con integratore Essential 8) insieme all'inibitore ROCK a una concentrazione finale pari a 1x e conservare la piastra in un incubatore a 37 °C e CO2 al 5% prima di aggiungere le hiPSC.
- Risospendere le hiPSC in terreno E8 completo insieme all'inibitore ROCK a una concentrazione finale pari a 1x per una migliore aderenza delle colonie.
NOTA: Non preparare grandi quantità di terreno di coltura. Preparare solo per 3-4 giorni al massimo e conservare a 2-8 °C. - Il giorno successivo, reintegrare l'intero terreno E8 senza l'inibitore ROCK fino a quando le cellule non sono confluenti all'80% (circa 4-5 giorni).
- Quando raggiungono l'80% di confluenza, staccano le colonie enzimaticamente.
- Per far passare le iPSC, rimuovere il terreno esaurito dalla piastra e aggiungere 1 mL per pozzetto di miscela preriscaldata (37 °C) di collagenasi (2 mg/mL) e dispasi (1 mg/mL) in un rapporto di 1:1 e incubare a 37 °C per più di 20-30 minuti per consentire alle colonie di iPSC di sollevarsi.
NOTA: Non lasciare le stoviglie a 37 °C per più di 20-30 min; Oltre questo periodo, le colonie si disintegreranno e le cellule moriranno.
- Per far passare le iPSC, rimuovere il terreno esaurito dalla piastra e aggiungere 1 mL per pozzetto di miscela preriscaldata (37 °C) di collagenasi (2 mg/mL) e dispasi (1 mg/mL) in un rapporto di 1:1 e incubare a 37 °C per più di 20-30 minuti per consentire alle colonie di iPSC di sollevarsi.
- Quando le colonie iniziano a sollevarsi, estrarre le piastre dall'incubatrice e neutralizzare l'attività enzimatica aggiungendo 2 ml di soluzione salina tamponata con fosfato di Dulbco (DPBS).
- Raschiare le colonie con DPBS e raccogliere la sospensione in una provetta conica da 15 mL utilizzando una pipetta sierologica da 10 mL a foro largo.
- Triturare delicatamente la sospensione 2-3 volte per rompere le colonie utilizzando una pipetta sierologica da 10 ml e lasciare che le colonie si stabilizzino.
NOTA: Non trasformare le colonie in singole cellule; Ciò può comportare la mancata adesione delle cellule al piatto e causare una maggiore morte cellulare. Inoltre, non lasciarli come grandi colonie; Questo porterà a colonie più differenziate in seguito. - Una volta che le colonie si sono stabilizzate (circa dopo 2 minuti), aspirare la miscela enzimatica DPBS lasciando circa 1 mL nella provetta.
- Aggiungere 2 ml di DPBS alla provetta, mescolare le colonie picchiettando e lasciarle depositare. Ripeti l'operazione 2 volte per rimuovere tutto l'enzima residuo dalle colonie. Rimuovere quanto più surnatante possibile dopo il 2° lavaggio, risospendere le colonie in 1 mL di E8 Medium completo e impiattare in un piatto appena preparato come indicato nei passaggi 1.2-1.3.
- In alternativa, crioconservare una parte delle colonie utilizzando il 10% di dimetilsolfossido (DMSO) come soluzione crioprotettiva per l'espansione e l'uso futuri.
- Preparare il terreno di crioconservazione appena miscelando il 90% di terreno E8 completo e il 10% di DMSO come soluzione crioprotettiva e posizionarlo a 2-8 °C fino all'uso.
- Per la crioconservazione, seguire i passaggi 1.5-1.10.
- Durante il passaggio 1.10, dopo il 2° lavaggio, lasciare che le colonie si depositino e rimuovere il surnatante. Alle colonie, aggiungere 1 mL di terreno di crioconservazione a freddo appena preparato e trasferire in crioviali.
NOTA: Dopo aver aggiunto il terreno di crioconservazione alle colonie, trasferire rapidamente il contenuto nei crioviali. Il ritardo può comportare un tasso di rianimazione inferiore, poiché il DMSO è un crioprotettore che può danneggiare le cellule. - Spostare immediatamente i crioviali in una crioscatola e conservarli in un congelatore a -80 °C per una notte.
- Il giorno successivo, sposta tutti i crioviali in un serbatoio di azoto liquido (serbatoio LN2 ) per un uso futuro.
NOTA: le hiPSC devono essere regolarmente cariotipizzate utilizzando il G-banding (Figura S1 supplementare) per eventuali anomalie, caratterizzate utilizzando l'immunocitochimica per la pluripotenza (Figura 1B) e testate per il micoplasma.- Per caratterizzare le hiPSC utilizzando l'immunocitochimica, placcare le colonie su vetrini coprioggetto da 13 mm rivestiti in Matrigel e autoclavati in scala 1:60. Una volta che sono confluenti al 40%, lavare le cellule con PBS-T (PBS-0,1% Tween 20), fissare con paraformaldeide al 4% per 10 minuti, permeabilizzare con Triton X-100 allo 0,3% in PBS per 10 minuti e bloccare con albumina sierica bovina (BSA) al 3% in PBS per 1 ora per prevenire il legame non specifico.
- Dopo il blocco, incubare le colture con anticorpi primari per 1 ora e quindi lavare 3 volte con PBS-T per 5 minuti ciascuna, seguite dai corrispondenti anticorpi secondari (Tabella dei materiali) al buio per 1 ora.
- Monta i vetrini coprioggetti su vetrini con mezzo di montaggio e acquisisci immagini mediante scansione laser confocale a 405 nm, 488 nm, 561 nm e 633 nm. Cattura immagini a 512 x 512 pixel; impostare la dimensione del passo Z a 0,5 μm con 1 unità di diametro arioso del foro stenopeico.
2. Generazione e caratterizzazione di cellule progenitrici astrocitiche (APCs)
- Sollevare enzimaticamente le hiPSC come indicato nei passaggi 1.5-1.10 e piastrelarle su un piatto di coltura in sospensione non aderente (100 mm) con terreno chimicamente definito14 contenente il 50% di terreno di Dulbecco modificato di Iscove (IMDM), il 50% di miscela di nutrienti F-12 di Ham (F12), 5 mg/mL di BSA, l'1% di concentrato lipidico chimicamente definito (CD-Lipid), 450 μM di monotioglicerolo, 7 μg/mL di insulina, 15 μg/mL di transferrina, Penicillina-streptomicina all'1% integrata con mitogeni di patterning del proencefalo N-acetil cisteina (1 mM), LDN 193189 (0,1 μM) e SB431542 (10 μM) per 7 giorni.
NOTA: Gli inibitori di piccole molecole SB431542 e LDN-193189 (LDN) sono inibitori delle proteine morfogenetiche ossee e delle vie di segnalazione beta del fattore di crescita trasformante.
Da questo momento in poi, il mezzo è stato rifornito una volta ogni 2 giorni o seguendo il protocollo lunedì/mercoledì/venerdì. - Posizionare la sospensione cellulare su un agitatore orbitale a 40 giri/min per 7 giorni in condizioni normossiche per favorire lo sviluppo delle corticosfere (Figura 1A).
- Il giorno 8, trasferire le corticosfere in un terreno di proliferazione cellulare contenente DMEM/F12 avanzato con l'1% di antibiotico-antimicotico, l'1% di integratore di N2, l'1% di sostituto della glutammina, lo 0,1% di integratore di B27 e 2,5 ng/mL di fattore di crescita basico dei fibroblasti (bFGF) per 7 giorni.
- Indurre le sfere per la specificazione gliale sottoponendole a un terreno di arricchimento gliale contenente DMEM/F12 avanzato con l'1% di antibiotico-antimicotico, l'1% di N2, l'1% di sostituto della glutammina, lo 0,1% di integratore di B27, 20 ng/mL di fattore di crescita epidermico (EGF), bFGF-H (20 ng/mL di bFGF-5 mg/mL di eparina) per 2 settimane per ottenere gliosfere precoci (Figura 1A).
- Per la maturazione delle gliosfere precoci, sostituire bFGF-H con 20 ng/mL di fattore inibitorio della leucemia (LIF) e mantenere le sfere per 4 settimane.
- Dopo 4 settimane nel terreno di maturazione, mantenere le sfere nel terreno di arricchimento gliale per periodi prolungati. Per prevenire l'aggregazione e la perdita di vitalità, ogni 2 settimane, tritare meccanicamente le gliosfere utilizzando una lama industriale sterile e sostituire l'intero terreno con DNasi I per rimuovere i frammenti di DNA generati dal taglio.
- Dissociare le gliosfere in monostrati di APC utilizzando un kit di dissociazione con papaina e dissociare su un piatto aderente trattato con coltura cellulare con diluizione 1:80 del rivestimento Matrigel.
- Propagare le APC nel terreno di arricchimento gliale fino all'80% di confluenza e farle passare enzimaticamente utilizzando il terreno di distacco cellulare enzimatico (vedi Tabella dei Materiali).
- Al passaggio:
- Rimuovere il terreno esaurito e raccoglierlo in un tubo conico. Alle cellule, aggiungere il terreno di distacco cellulare enzimatico e attendere 1-2 minuti. Una volta che le cellule iniziano a staccarsi, aggiungere il terreno esaurito per neutralizzare l'attività enzimatica.
- Raccogliere la sospensione cellulare e centrifugare a 800 x ɡ per 2 min.
- Aspirare il surnatante, risospendere le cellule in un terreno di arricchimento gliale e impiattare circa 1 x 106 cellule/pozzetto su una piastra a 6 pozzetti rivestita di Matrigel 1:80.
- Per la crioconservazione:
- Risospendere le cellule in una miscela fredda di 90% di terreno di proliferazione corticosfera (senza bFGF) e 10% di crioprotettore. Trasferire le cellule risospese in crioviali.
- Spostare immediatamente i crioviali in una crioscatola e conservarli in congelatore a -80 °C per una notte.
- Il giorno successivo, sposta tutti i crioviali in un serbatoio di azoto liquido (serbatoio LN2 ) per un uso futuro.
NOTA: Gli APC sono stati crioconservati utilizzando il 10% di DMSO fino al passaggio numero 6.
- Caratterizzare le APC dissociate mediante immunocolorazione (come menzionato nei passaggi 1.15.1-1.15.3) con marcatori di vimentina e fattore nucleare IA (NFIA) (Figura 1C, D) (vedere la tabella dei materiali per le diluizioni).
- Per confermare la specificità regionale del proencefalo delle APC derivate da hiPSC, testare le cellule per un marcatore del proencefalo come la scatola a forchetta umana G1 (hFOXG1) (positivo) e il marcatore del rombencefalo come l'omeobox B4 umano (hHOXB4) (negativo) utilizzando la qPCR in tempo reale (Figura 1E, F).
NOTA: Le sequenze di primer utilizzate per questi esperimenti sono state elencate nella Tabella Supplementare S1.
3. Generazione e caratterizzazione di una popolazione omogenea di astrociti prosencefalo-specifici
- Differenziazione degli astrociti dalle APC
- Utilizzare il mezzo di differenziazione astrocitaria (ADM) per 14 giorni per differenziare le APC in astrociti. Il terreno di differenziazione astrocitaria contiene Neurobasal, 1% antibiotico-antimicotico, 1% sostituto della glutammina, 1% integratore di N2, 0,2% integratore di B27, 1% mezzo di aminoacidi non essenziali (NEAA) e 10 ng/mL di fattore neurotrofico ciliare (CNTF).
- Confermare l'identità astrocitaria utilizzando l'immunocolorazione (come menzionato nei passaggi 1.15.1-1.15.3.) con la proteina acida fibrillare gliale (GFAP) e i marcatori di espressione S100β (Figura 2A, B).
- Sintesi proteica de novo in APC e astrociti
- Mantenere le APC del prosencefalo derivate da hiPSC e gli astrociti su vetrini coprioggetti sterili autoclavati da 13 mm e immunocolorarli come indicato nei passaggi 1.15.1-1.15.3.
- Per la sintesi proteica de novo , utilizzare il metodo FUNCAT (fluorescente non canonical amminoacid tagging), spiegato brevemente nei passaggi 3.2.3-3.2.6 (Figura 3A).
- Per garantire la carenza di metionina, rimuovere il terreno di coltura dalle colture e sostituirlo con un terreno privo di metionina e cisteina mescolato con 1 mM di L-azidoomoalanina (AHA) per 30 minuti a 37 °C e 5% di CO2.
- Lavare le colture con PBS-T, fissare con paraformaldeide al 4% per 10 minuti, permeabilizzare con Triton X-100 allo 0,3% in PBS per 10 minuti e bloccare con BSA al 3% in PBS per 1 ora per evitare il legame non specifico.
- Incubare le colture al buio per 1 ora a temperatura ambiente con la miscela di reazione chimica delle cellule Click e Alkyne Alexa Fluor 647, seguita da anticorpi primari per 1 ora e anticorpi secondari corrispondenti (Tabella dei materiali) per 1 ora (Figura 3B).
- Monta i vetrini coprioggetti su vetrini con supporto di montaggio e utilizzali per ulteriori analisi delle immagini.
- Acquisisci immagini mediante scansione laser confocale a 405 nm, 488 nm, 561 nm e 633 nm.
- Cattura immagini a 512 x 512 pixel; impostare la dimensione del passo Z a 0,5 μm con 1 unità di diametro arioso del foro stenopeico.
- Mantenere i parametri di microscopia e imaging a un'impostazione costante tra i tipi di cellule. Cattura immagini per ogni replica biologica nella stessa sessione.
- Esegui misurazioni dell'intensità utilizzando qualsiasi software di analisi delle immagini standard (ad es. Fiji o Imaris). In Imaris, utilizzare il modulo SURFACES per produrre misurazioni volumetriche dei corpi cellulari positivi per il segnale fluorescente FUNCAT (Non-Canonical Amino Acid Tagging) con Vimentina (APC) e GFAP (astrociti) che limitano l'intervallo di voxel (250-350) per rimuovere i detriti.
NOTA: Mantenere i parametri in tutte le repliche biologiche per entrambi i tipi di cellule.
- Onde di calcio oscillatorie indotte da adenosina 5′-trifosfato (ATP)
- Piastra gli astrociti su una piastra con fondo di vetro (35 mm) a 5 × 103 cellule/piastra per misurare la risposta cellulare all'ATP.
- Lasciare che le cellule aderiscano al fondo di vetro per 24 ore, lavarle 3 volte con HBSS (20 mM HEPES, 137 mM NaCl, 5 mM KCl, 10 mM Glucosio, 1 mM MgCl2, pH = 7,3) senza calcio e incubare in terreno di coltura con colorante metrico Fura-2AM con rapporto 5 μM e Pluronics F127 0,02% per 1 ora a temperatura ambiente.
NOTA: Fura-2AM è una sostanza chimica sensibile alla luce e alla temperatura; conservare a -20 °C. - Dopo l'incubazione, lavare 2x con terreno di coltura e sostituire con 2 mM di Ca2+ contenente HBSS e visualizzare le cellule a una velocità di 2 FPS utilizzando un obiettivo a olio 60x (1,35 NA) in un microscopio invertito a compensazione della deriva della messa a fuoco.
- Registrare le risposte di calcio indotte da ATP mediante l'applicazione di ATP in bagno alla concentrazione finale di 5 mM a 25° s (Figura 4A).
- Disegna le regioni di interesse attorno a ciascuna cella utilizzando Fiji/ImageJ e calcola il rapporto F340/F380 in tutti i punti temporali (Figura 4B).
- Saggi metabolici cellulari
NOTA: Il tasso di consumo di ossigeno (OCR) e il tasso di acidificazione extracellulare (ECAR) delle cellule vive sono stati misurati secondo il protocollo del produttore (vedere la Tabella dei materiali).- Un giorno prima del saggio, lasciare che gli astrociti aderiscano alla micropiastra (1,5 × 104 densità di semina) in terreno CNTF e incubare a 37 °C con il 5% di CO2.
- Per il test dello stress da glicolisi:
- Sostituire il terreno di coltura cellulare con un terreno di base integrato con 2 mM di glutammina (pH regolato a 7,4) e incubare in una camera non CO2 a 37 °C per 1 ora.
NOTA: Il pH deve essere regolato a bagnomaria a 37 °C. - Inserire la piastra della cartuccia nello strumento per calibrare il sensore. Dopo la calibrazione, sostituire la piastra della cartuccia con la piastra di coltura cellulare a una concentrazione finale dei seguenti componenti del kit: 10 mM di glucosio, 1 μM di oligomicina e 50 mM di 2-desossiglucosio (2-DG) (Figura 5A).
- Al termine del test, lisare le cellule e stimare il contenuto proteico totale.
- Sostituire il terreno di coltura cellulare con un terreno di base integrato con 2 mM di glutammina (pH regolato a 7,4) e incubare in una camera non CO2 a 37 °C per 1 ora.
- Per il test di stress dei mitocondri:
- Sostituire il terreno CNTF con un terreno basico integrato con 2 mM di glutammina, 1 mM di piruvato, 10 mM di glucosio (pH regolato a 7,4) a bagnomaria a 37 °C.
- Incubare la piastra in una camera non contenente CO2 a 37 °C per 1 ora. Nel frattempo, inserire la piastra della cartuccia nello strumento per calibrare il sensore.
- Dopo la calibrazione, sostituire la piastra della cartuccia con la piastra di coltura cellulare con una concentrazione finale dei seguenti componenti del kit: 1,5 μM di oligomicina, 1 μM di cianuro di carbonile-4 (trifluorometossi) fenilidrazone (FCCP) e 0,5 μM di rotenone/antimicina A (Figura 5C).
- Al termine del test, lisare le cellule e stimare il contenuto proteico totale.
Risultati
Le colonie di cellule staminali pluripotenti indotte umane (hiPSC) sono state mantenute utilizzando un terreno definito disponibile in commercio e immunocolorate per i marcatori di pluripotenza, Oct4 e Nanog (Figura 1B). Le APC FXS e CTRL hanno mostrato proporzioni comparabili altamente arricchite di cellule immunopositive per vimentina e NFIA (Figura 1C, D). Abbiamo scoperto che le APC derivate da hiPSC sane e FXS hanno mostrato una sostanziale sovraregolazione di FOXG1 rispetto a HOXB4 (Figura 1F), coerentemente con un'identità predominante del proencefalo. Abbiamo osservato una riduzione significativa del numero di astrociti che esprimono GFAP nei gruppi FXS rispetto agli astrociti CTRL senza influenzare il numero di astrociti che esprimono S100β (Figura 2B). L'analisi del western blot ha mostrato che gli astrociti generati da entrambe le linee FXS mancavano di espressione di FMRP (Figura 2C, D). La quantificazione delle proteine di nuova sintesi non ha rivelato alcuna differenza significativa tra le linee CTRL e FXS né nelle APC né negli astrociti (Figura 3C). Tuttavia, abbiamo riscontrato che la sintesi proteica è costantemente più alta nelle APC che nei rispettivi astrociti per ciascuna linea (Figura 3C). Questi risultati suggeriscono una riduzione stadio-specifica della sintesi proteica de novo dalle APC agli astrociti in entrambe le linee FXS e CTRL.
Il controllo, così come gli astrociti FXS, mostravano transitori di calcio indotti da ATP (Figura 4A). Tuttavia, un'analisi più dettagliata ha rivelato diverse differenze chiave tra i singoli transienti di calcio registrati dagli astrociti CTRL, FXS1 e FXS2 (Figura 4B). Abbiamo classificato gli astrociti come responder e non-responder all'ATP in base alla presenza o all'assenza di una risposta al calcio, rispettivamente (Figura 4C). Il numero di responder in FXS1 e FXS2 era significativamente inferiore a CTRL. In particolare, abbiamo osservato una significativa riduzione dell'ampiezza di picco del primo transiente di calcio evocato dopo l'applicazione di ATP in entrambe le linee astrocitarie FXS (Figura 4D). Inoltre, la durata totale del primo transitorio di calcio era significativamente più breve nelle linee FXS1 e FXS2 rispetto a CTRL (Figura 4E). Tuttavia, il numero di eventi di calcio (quantificato come il numero totale di eventi per 250 s; Figura 4F), era comparabile tra le linee. Nel loro insieme, questi risultati rivelano alterazioni nelle risposte al calcio suscitate dall'ATP negli astrociti FXS derivati da hiPSC. I risultati dell'ECAR suggeriscono un tasso più elevato di glicolisi, capacità glicolitica e riserva glicolitica negli astrociti FXS derivati da hiPSC (Figura 5A, B). Non abbiamo trovato differenze significative nella respirazione basale negli astrociti CTRL e FXS. La produzione cellulare di ATP non ha mostrato differenze significative tra gli astrociti CTRL e FXS. La stimolazione FCCP ha suscitato una respirazione massimale significativamente più bassa negli astrociti FXS rispetto agli astrociti CTRL (Figura 5C, D).

Figura 1: Derivazione di cellule progenitrici astrocitiche specifiche del prosencefalo da hiPSC. (A) Flusso di lavoro illustrativo per la generazione di astrociti da hiPSC. (B) Immagini rappresentative di hiPSC che mostrano un'espressione comparabile di Oct4/Nanog nelle linee CTRL e FXS. (C) La popolazione omogenea di APC derivate da hiPSC esprimeva un numero simile di cellule Vimentina e NFIA-positive. (D) Nessuna differenza significativa nell'espressione di Vimentina e NFIA tra CTRL e FXS APCs. L'analisi statistica è stata eseguita utilizzando l'ANOVA a due fattori seguita dal confronto a coppie di Tukey. (E) Schema che illustra l'espressione region-specific di FOXG1 e HOXB4 durante lo sviluppo in vivo. (F) Rappresentazione grafica dell'espressione di FOXG1 rispetto a HOXB4 (utilizzando qRT-PCR) attraverso genotipi che suggeriscono una propensione verso il lignaggio del proencefalo. Analisi statistica mediante ANOVA a due fattori seguita dal test di confronto multiplo di Sidak. Per tutti gli esperimenti N = 3 repliche biologiche. p < 0,001. Barra di scala = 50 μm. Le barre di errore rappresentano SEM. Abbreviazioni: hiPSCs = cellule staminali pluripotenti indotte umane; CTRL = Controllo; FXS = Sindrome dell'X Fragile; APCs = cellule progenitrici degli astrociti. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Numero ridotto di astrociti FXS derivati da hiPSC positivi alla proteina acida fibrillare gliale. (A) Immagini rappresentative di astrociti derivati da hiPSC che mostrano l'espressione di S100β e GFAP. (B) La percentuale di astrociti GFAP-positivi era significativamente più bassa negli astrociti derivati da FXS rispetto a CTRL, sebbene entrambi mostrassero un'espressione simile di S100β. Significatività statistica determinata dall'ANOVA a due fattori seguita dal confronto a coppie di Tukey. (C) Immunoblot che rappresenta l'espressione di FMRP negli astrociti da CTRL e FXS. (D) Grafici che mostrano l'assenza di FMRP negli astrociti FXS derivati da hiPSC. Analisi statistica effettuata mediante ANOVA a fattore singolo con confronto a coppie di Tukey. Per tutti gli esperimenti di cui sopra N = 3 repliche biologiche. Barra di scala = 50 μm. ***p < 0,001. Le barre di errore rappresentano SEM. Abbreviazioni: hiPSCs = cellule staminali pluripotenti indotte umane; CTRL = Controllo; FXS = Sindrome dell'X Fragile; GFAP = proteina acida fibrillare gliale; FMRP = Ribonucleoproteina messaggero dell'X fragile. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Riduzione della sintesi proteica dovuta alla differenziazione da APC ad astrociti. (A) Flusso di lavoro schematico che illustra l'etichettatura e la visualizzazione della sintesi proteica in APC e astrociti derivati da hiPSC utilizzando la marcatura metabolica FUNCAT. (B) Immagini rappresentative di (in alto) APC derivate da hiPSC Vimentina-positive e (in basso) astrociti GFAP-positivi con etichetta FUNCAT/Human Nuclei Antibody (hNA) in cellule derivate da CTRL e FXS. (C) FUNCAT/volume di APC derivate da hiPSC era significativamente più alto rispetto ai corrispondenti astrociti derivati. Per tutti gli esperimenti N = 3 repliche biologiche. Barra della scala = 50 μm. **p < 0,01, ***p < 0,001. I baffi rappresentano 1,5 × IQR. Abbreviazioni: hiPSCs = cellule staminali pluripotenti indotte umane; CTRL = Controllo; FXS = Sindrome dell'X Fragile; GFAP = proteina acida fibrillare gliale; FUNCAT = marcatura fluorescente non canonica degli amminoacidi. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Segnalazione carente di Ca2+ evocata dall'ATP negli astrociti FXS derivati da hiPSC. (A) Tracce rappresentative di transienti di Ca2+ registrate da singoli astrociti dopo applicazione esterna di ATP a 25sec . (B) Rapporti medi F340/F380 che descrivono transienti di Ca2+ oltre 250 s dopo l'applicazione di ATP. (C) Dati raggruppati che mostrano una percentuale più elevata di non-responder all'ATP negli astrociti FXS derivati da hiPSC. Significatività statistica determinata dall'ANOVA a due fattori con il confronto a coppie di Tukey. (D-F) Quantificazione della risposta al primo picco (ampiezza e durata) e della frequenza degli eventi. (D) Mostra una significativa riduzione dell'ampiezza. Significatività statistica determinata dall'ANOVA a fattore singolo con il confronto a coppie di Tukey e la durata (E) negli astrociti FXS derivati da hiPSC. Significatività statistica determinata dal test di Kruskal-Wallis con il test di confronto multiplo di Dunn; set di dati rappresentato come ranghi medi con SEM. (F) Rappresentazione grafica della frequenza transitoria di Ca2+ negli astrociti FXS derivati da hiPSC. Analisi statistica mediante ANOVA a fattore singolo con confronto a coppie di Tukey. Per tutti gli esperimenti N = 3 repliche biologiche; n = 19 celle per ogni linea cellulare. *p < 0,05, **p < 0,01, ***p < 0,001. Le barre di errore rappresentano SEM. # = Numero. Abbreviazioni: hiPSCs = cellule staminali pluripotenti indotte umane; CTRL = Controllo; FXS = Sindrome dell'X Fragile; GFAP = proteina acida fibrillare gliale. Clicca qui per visualizzare una versione più grande di questa figura.
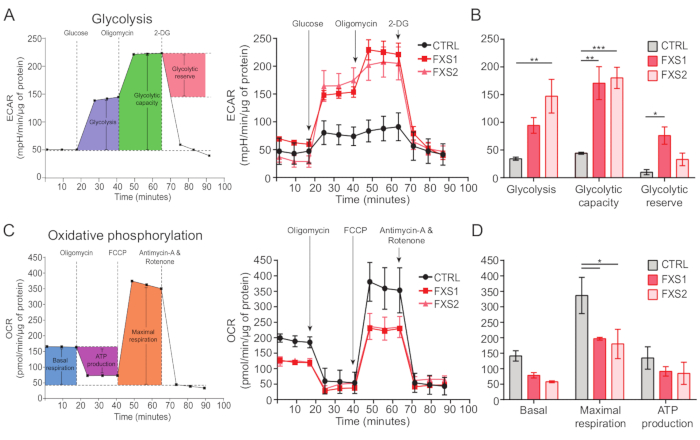
Figura 5: Deficit bioenergetici negli astrociti FXS derivati da hiPSC. (A) Grafico a linee che rappresenta il tasso di acidificazione extracellulare degli astrociti derivati da hiPSC (CTRL e FXS) dopo l'aggiunta di 10 mM di glucosio, 1 μM di oligomicina e 50 mM 2-DG in sequenza e tracciato su un grafico a linee. (B) La glicolisi, la capacità glicolitica e la quantificazione della riserva glicolitica hanno mostrato un aumento degli astrociti FXS derivati da hiPSC. Analisi statistica effettuata mediante ANOVA a due fattori con il confronto a coppie di Tukey. (C) Grafico a linee che rappresenta la misurazione del tasso di consumo di ossigeno mediante l'aggiunta sequenziale di 1,5 μM di oligomicina, 1 μM di FCCP e 0,5 μM di antimicina A e rotenone. (D) La respirazione basale, la respirazione massimale e la produzione di ATP sono state quantificate dal grafico a linee e gli astrociti FXS derivati da hiPSC hanno mostrato una diminuzione significativa della respirazione massimale rispetto agli astrociti CTRL. Analisi statistica effettuata mediante ANOVA a due fattori con il confronto a coppie di Tukey. Per tutti gli esperimenti N = 2 repliche biologiche. *p < 0,05, **p < 0,01, ***p < 0,001. Le barre di errore rappresentano SEM. Abbreviazioni: hiPSCs = cellule staminali pluripotenti indotte umane; CTRL = Controllo; FXS = Sindrome dell'X Fragile; ECAR = tasso di acidificazione extracellulare; 2-DG = 2-desossiglucosio; OCR = tasso di consumo di ossigeno; FCCP = cianuro di carbonile-4 (trifluorometossi) fenilidrazone. Clicca qui per visualizzare una versione più grande di questa figura.
| ID nel manoscritto | ID alla fonte | Età | Sesso | Nome della linea cellulare riprogrammata | Metodo di riprogrammazione | Tipo di cella iniziale | Cariotipo banda G |
| (anni) | |||||||
| CTRL | ND30625 | 76 | M | CS25iCTR-18nxx | Vettori episomiali | Fibroblasto | Normale |
| FXS1 | GM07072 | 22 | M | CS072iFXS-n4 | Vettori episomiali | Fibroblasto | Normale |
| FXS2 | GM05848 | 4 | M | CS848iFXS-n5 | Vettori episomiali | Fibroblasto | Normale |
Tabella 1: Linee cellulari utilizzate in questo studioper generare astrociti specifici per il proencefalo.
Figura supplementare S1: Rapporto di analisi cromosomica. Cariotipo normale di bande GTG di (A) CTRL 46, XY, (B) FXS1 46, XY e (C) FXS2 46, XY. Clicca qui per scaricare questa figura.
Tabella supplementare S1: Primer utilizzati in questo studio per la caratterizzazione delle cellule. Clicca qui per scaricare questa tabella.
Discussione
Qui, descriviamo un metodo per generare astrociti umani derivati da iPSC che fungono da piattaforma di analisi per caratterizzare i cambiamenti funzionali indotti da FXS. Questi astrociti sono funzionalmente vitali in coltura e mostrano varie proprietà, come evidenziato da una serie di misurazioni effettuate nel presente studio. Un passaggio critico in questo protocollo è la conversione iniziale delle iPSC in corticosfere utilizzando il metodo del lifting enzimatico. In questa fase, è fondamentale ottimizzare il tempo di incubazione della collagenasi di tipo IV e della dispasi. Se questo non viene ottimizzato, può causare la disintegrazione delle iPSC o la formazione di singole cellule, il che impedirà la formazione di corticosfere. Queste corticosfere sono modellate verso la specificità del proencefalo utilizzando la doppia inibizione SMAD15 e sono ulteriormente differenziate in cellule progenitrici degli astrociti (APC). Per ottenere un'elevata popolazione di APC, il taglio meccanico delle gliosfere è fondamentale. La qPCR in tempo reale può essere utilizzata per confermare la specificità del proencefalo; abbiamo osservato un aumento significativo della variazione di piega in FOXG1, un marcatore per il proencefalo, rispetto a HOXB4, un marcatore per il midollo spinale.
Le APC vengono quindi differenziate in astrociti utilizzando CNTF, un attivatore della via di segnalazione JAK-STAT16. I risultati di questo studio indicano che gli astrociti terminalmente differenziati derivati dalle iPSC FXS hanno livelli di GFAP più bassi, il che è indicativo di una maturità compromessa. Sebbene le nostre analisi non abbiano riscontrato alcuna differenza nella sintesi proteica de novo negli astrociti FXS e nelle APC rispetto ai controlli, abbiamo riscontrato una riduzione della sintesi proteica de novo nella transizione dalle APC agli astrociti sia nelle FXS che nelle linee di controllo. La significativa riduzione della traduzione tra APC e astrociti è coerente con una traduzione più snella e curata che è stata dimostrata per i neuroni rispetto alle cellule precursori neurali17. Lavori precedenti che hanno esaminato la sintesi proteica aberrante hanno utilizzato interi tessuti cerebrali, neuroni e fibroblasti derivati da pazienti, non astrociti o APC18. Gli unici rapporti pubblicati sulla sintesi proteica da linee derivate da pazienti non sono negli astrociti, ma nelle cellule linfoblastoidi19 e nei fibroblasti 20,21. Alla luce di queste variazioni tra gli studi, gli studi futuri trarrebbero vantaggio dall'utilizzo delle misurazioni FUNCAT negli astrociti derivati da ulteriori linee derivate da pazienti FXS.
Abbiamo valutato la funzionalità degli astrociti misurando la dinamica intracellulare del calcio22 e la bioenergetica. Gli astrociti FXS hanno mostrato una riduzione dell'ampiezza del picco e della durata dei transienti di calcio indotti da ATP, con un minor numero di cellule che rispondono all'ATP rispetto agli astrociti di controllo. Insieme, queste alterazioni suggeriscono che la FXS interrompe l'omeostasi del calcio negli astrociti umani. Questi risultati sono coerenti con uno studio precedente 23,24 che riportava un'attività compromessa del recettore IP3 nei fibroblasti FXS. Inoltre, abbiamo osservato un aumento della glicolisi, della capacità glicolitica e della riserva glicolitica negli astrociti FXS, insieme a una riduzione dei tassi di consumo di ossigeno mitocondriale.
Il presente studio descrive la generazione e la caratterizzazione di un nuovo modello in vitro per astrociti FXS sfruttando la potenza delle iPSC derivate da pazienti. In particolare, è solo attraverso uno studio precedente che ha utilizzato astrociti umani co-coltivati con neuroni umani che abbiamo scoperto il ruolo critico degli astrociti nel determinare il fenotipo elettrofisiologico dei neuroni14. Questi studi, tuttavia, si sono concentrati quasi interamente sui cambiamenti indotti dalla malattia nell'attività neuronale e nulla è stato esplorato negli astrociti. Qui, questo protocollo offre un nuovo quadro per spostare la nostra attenzione sulle alterazioni funzionali negli astrociti. Questo, a sua volta, farà luce su interazioni astrocita-neurone precedentemente inesplorate, come la segnalazione del glutammato e le risposte elettriche. I nostri risultati in questa fase sottolineano la necessità di dare priorità agli astrociti nella ricerca futura.
È fondamentale riconoscere i limiti di questo protocollo. In primo luogo, comporta il taglio meccanico, che porta anche alla frammentazione del DNA. Questo può causare l'aggregazione delle gliosfere tritate, portando infine alla morte cellulare. In secondo luogo, la densità di semina delle APC durante la fase terminale di differenziazione in astrociti è essenziale per prevenire l'inibizione cellula-cellula mediata dal contatto. Si consiglia di ottimizzare questo aspetto su tutte le linee cellulari per evitare il distacco dalla superficie del recipiente di coltura.
Qui, i nostri risultati sono la prima analisi di questo tipo negli astrociti umani poiché tutti gli studi precedenti si basavano su altri tipi di cellule come le linee cellulari linfoblastoidi e i fibroblasti umani25. Il metabolismo energetico disfunzionale è un attore noto nell'eziologia dei disturbi dello spettro autistico, come dimostrato dall'aumento dei livelli di lattato plasmatico umano, dal tasso di consumo di ossigeno significativamente più basso nei granulociti26 e dalla ridotta espressione dei geni della fosforilazione ossidativa mitocondriale nella corteccia cingolata anteriore, nella corteccia motoria, nel talamo e nel cervelletto dei bambini con ASD27. Cambiamenti simili nel metabolismo energetico si osservano in malattie neurodegenerative come il morbo di Parkinson28 e il morbo di Alzheimer29, suggerendo che l'aumento della glicolisi può compensare la ridotta funzione funzionale30. Sono necessari studi futuri per esplorare se meccanismi simili sono in gioco nel metabolismo energetico disfunzionale osservato negli astrociti derivati da pazienti FXS.
Al momento, la generazione di astrociti specifici per il proencefalo è un risultato significativo, ma i progressi futuri potrebbero consentire la creazione di glia specifici per regione, come gli astrociti corticali/ippocampali. Ciò fornirebbe informazioni più approfondite sui ruoli distinti svolti da questi astrociti nelle funzioni cerebrali regionali, nella plasticità sinaptica e nelle interazioni neuronali-gliali. La capacità di generare tali astrociti specifici per regione potrebbe far avanzare modelli di neurosviluppo e malattie neurodegenerative. In definitiva, questo potrebbe portare a strategie terapeutiche più precise che mirano alla disfunzione degli astrociti in specifiche regioni del cervello.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo il Prof. Sumantra Chattarji per i fondi intramurali. Ringraziamo il Prof. Gaiti Hasan per l'accesso alla configurazione di imaging del calcio, al Central Imaging and Flow Facility-National Centre for Biological Sciences, a Padmanabh Singh e Prangya Hota per la correzione di bozze e i suggerimenti, e al team di Labmate Asia per l'assistenza nell'esecuzione dei saggi Seahorse XF.
Materiali
| Name | Company | Catalog Number | Comments |
| 1-Thioglycerol | Sigma-Aldrich | M6145 | |
| Accutase solution | Sigma-Aldrich | A6964 | Enzyme cell detachment medium |
| Adenosine 5′-triphosphate magnesium salt | Sigma-Aldrich | A9187 | |
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634010 | |
| Antibiotic-Antimycotic (100x) | ThermoFisher Scientific | 15240062 | |
| B-27 Supplement (50x), serum-free | ThermoFisher Scientific | 17504044 | |
| Bovine Serum Albumin | Sigma-Aldrich | A9418 | |
| Chemically Defined Lipid Concentrate | ThermoFisher Scientific | 11905031 | |
| Collagenase, Type IV, powder | ThermoFisher Scientific | 17104019 | |
| Deoxyribonuclease I | Worthington Biochemical Corporation | LK003170 | |
| Dimethyl sulfoxide | Sigma-Aldrich | D2650 | |
| Dispase II, powder | ThermoFisher Scientific | 17105041 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| Essential 8 Medium | ThermoFisher Scientific | A1517001 | |
| FXS1, FXS2 | Coriell Institute of Medical Research | GM07072, GM05848 | FXS patient cells |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | glutamine substitute |
| Ham's F-12 Nutrient Mix | ThermoFisher Scientific | 11765054 | |
| Healthy control cells | Cedars-Sinai Medical Center | ND30625 | healthy control cells |
| Heparin sodium salt from porcine intestinal mucosa | Sigma-Aldrich | H3149 | |
| IMDM | ThermoFisher Scientific | 12440053 | |
| Insulin, human | Roche | 11376497001 | |
| LDN 193189 | Stratech Scientific | S2618-SEL | |
| Leukemia Inhibitory Factor human | Sigma-Aldrich | L5283 | |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 354230 | |
| MEM Non-Essential Amino Acids Solution (100x) | ThermoFisher Scientific | 11140050 | |
| Mouse FGF-basic (FGF-2/bFGF) Recombinant Protein | Peprotech | 450-33 | |
| Mr. Frosty freezing container | ThermoFisher Scientific | 5100-0001 | cryobox |
| N-2 Supplement (100x) | ThermoFisher Scientific | 17502048 | |
| N-Acetyl-L-cysteine | Sigma-Aldrich | A9165 | |
| Neurobasal Medium | ThermoFisher Scientific | 21103049 | |
| Nunc Biobanking and Cell Culture Cryogenic Tubes | ThermoFisher Scientific | 377267 | |
| Nunc Cell-Culture Treated 6 well dish | ThermoFisher Scientific | 140675 | |
| Papain Dissociation System | Worthington Biochemical Corporation | LK003150 | |
| Penicillin-Streptomycin | ThermoFisher Scientific | 15140122 | |
| Recombinant Human CNTF Protein, CF | R&D Systems | 257-NT-010 | |
| Recombinant Human EGF Protein, CF | R&D Systems | 236-EG-01M | |
| RevitaCell Supplement (100x) | ThermoFisher Scientific | A2644501 | |
| SB431542 | Tocris | 1614 | |
| Seahorse XFe24 Analyzer | Agilent Technologies | ||
| Seahorse XF Cell Mito Stress Test Kit | Agilent Technologies | 103015-100 | |
| Seahorse XF Glycolysis Stress Test Kit | Agilent Technologies | 103020-100 | |
| Tissue Culture Dishes-100 cm | Biostar Lifetech | TCD000100 | |
| Transferrin | Roche | 10652202001 | |
| VWR Razor Blades | VWR International | 55411-050 | |
| Antibodies | |||
| Primary antibody | Company | Catalog number | Dilution |
| Oct4 (C-10) | Santa Cruz Biotechnology | sc-5279 | Dilution: 1:250 Secondary antibody: Goat anti-Mouse IgG, Alexa Fluor 568 |
| Nanog | R & D Systems | AF1997 | Dilution: 1:100 Secondary antibody: Donkey anti-Goat IgG, Alexa Fluor 488 |
| Vimentin | Abcam | Ab5733 | Dilution: 1:500 Secondary antibody: Goat anti-Chicken IgY, Alexa Fluor 488 |
| NFIA | Abcam | Ab41851 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| GFAP-cy3 | Sigma | C9205 | Dilution: 1:500 Secondary antibody: NA |
| GFAP | DAKO | Z0334 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| S100β | DAKO | IR504 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 488 |
| Anti-Nuclei Antibody, clone 235-1 | Merck Millipore | MAB1281 | Dilution: 1:1000 Secondary antibody: Goat anti-Mouse IgG1, Alexa Fluor 555 |
| Secondary antibodies | Dilution | ||
| Goat anti-Mouse IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11004 | 1:1000 |
| Donkey anti-Goat IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11055 | 1:1000 |
| Goat anti-Chicken IgY, Alexa Fluor 488 | Thermo Fisher Scientific | A11039 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11011 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11034 | 1:1000 |
| Goat anti-Mouse IgG1, Alexa Fluor 555 | Thermo Fisher Scientific | A21127 | 1:1000 |
Riferimenti
- Santoro, M. R., Bray, S. M., Warren, S. T. Molecular of fragile X syndrome: A twenty-year perspective. Annu Rev Pathol. 7, 219-245 (2012).
- Wang, H., et al. Developmentally-programmed FMRP expression in oligodendrocytes: A potential role of FMRP in regulating translation in oligodendroglia progenitors. Hum Mol Genet. 13 (1), 79-89 (2004).
- Pacey, L. K. K., Doering, L. C. Developmental expression of FMRP in the astrocyte lineage: Implications for fragile X syndrome. Glia. 55 (15), 1601-1609 (2007).
- Zhang, Y., et al. Purification and characterization of progenitor and mature human astrocytes reveals transcriptional and functional differences with mouse. Neuron. 89 (1), 37-53 (2016).
- Allen, N. J., Barres, B. A. Neuroscience: Glia - more than just brain glue. Nature. 457 (7230), 675-677 (2009).
- Khakh, B. S., McCarthy, K. D. Astrocyte calcium signaling: From observations to functions and the challenges therein. Cold Spring Harb Perspect Biol. 7 (4), a020404(2015).
- Allen, N. J., et al. Astrocyte glypicans 4 and 6 promote formation of excitatory synapses via GluA1 AMPA receptors. Nature. 486 (7403), 410-414 (2012).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Rothstein, J. D., et al. Knockout of glutamate transporters reveals a major role for astroglial transport in excitotoxicity and clearance of glutamate. Neuron. 16 (3), 675-686 (1996).
- Almad, A., Maragakis, N. J. A stocked toolbox for understanding the role of astrocytes in disease. Nat Rev Neurol. 14 (6), 351-362 (2018).
- Zhang, Y., Barres, B. A. Astrocyte heterogeneity: An underappreciated topic in neurobiology. Curr Opin Neurobiol. 20 (5), 588-594 (2010).
- Clarke, L. E., Barres, B. A. Emerging roles of astrocytes in neural circuit development. Nat Rev Neurosci. 14 (5), 311-321 (2013).
- Bradley, R. A., et al. Regionally specified human pluripotent stem cell-derived astrocytes exhibit different molecular signatures and functional properties. Development. 146 (13), dev170910(2019).
- Das Sharma, S., et al. Astrocytes mediate cell non-autonomous correction of aberrant firing in human FXS neurons. Cell Rep. 42 (4), 112344(2023).
- Chambers, S. M., et al. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Hu, X., et al. The JAK/STAT signaling pathway: from bench to clinic. Sig Transduct Target Ther. 6, 402(2021).
- Baser, A., et al. Onset of differentiation is post-transcriptionally controlled in adult neural stem cells. Nature. 566 (7742), 100-104 (2019).
- Pal, R., Bhattacharya, A. Modelling protein synthesis as a biomarker in fragile x syndrome patient-derived cells. Brain Sci. 9 (3), 1-12 (2019).
- Gross, C., Bassell, G. J. Excess protein synthesis in FXS patient lymphoblastoid cells can be rescued with a p110β-selective inhibitor. J Mol Med. 18 (3), 336-345 (2012).
- Kumari, D., et al. Identification of fragile X syndrome specific molecular markers in human fibroblasts: A useful model to test the efficacy of therapeutic drugs. Hum Mutat. 35 (12), 1485-1494 (2014).
- Jacquemont, S., et al. Protein synthesis levels are increased in a subset of individuals with fragile X syndrome. Hum Mol Genet. 27 (12), 2039-2051 (2018).
- Bowser, D. N., Khakh, B. S. ATP excites interneurons and astrocytes to increase synaptic inhibition in neuronal networks. J Neurosci. 24 (39), 8606-8620 (2004).
- Schmunk, G., Boubion, B. J., Smith, I. F., Parker, I., Gargus, J. J. Shared functional defect in IP3R-mediated calcium signaling in diverse monogenic autism syndromes. Transl Psychiatry. 5 (9), e643-e710 (2015).
- Peteri, U. K., et al. Generation of the human pluripotent stem-cell-derived astrocyte model with forebrain identity. Brain Sci. 11 (2), 209(2021).
- Alvarez-Mora, M. I., et al. Impaired mitochondrial function and dynamics in the pathogenesis of FXTAS. Mol Neurobiol. 54 (9), 6896-6902 (2017).
- Giulivi, C., et al. Mitochondrial dysfunction in autism. JAMA. 304 (21), 2389-2396 (2010).
- Anitha, A., et al. Brain region-specific altered expression and association of mitochondria-related genes in autism. Mol Autism. 3 (1), 12(2012).
- Teves, J. M. Y., et al. Parkinson's disease skin fibroblasts display signature alterations in growth, redox homeostasis, mitochondrial function, and autophagy. Front Neurosci. 11, 737(2018).
- Yao, J., et al. Mitochondrial bioenergetic deficit precedes Alzheimer's pathology in female mouse model of Alzheimer's disease. Proc Natl Acad Sci USA. 106 (34), 14670-14675 (2009).
- Sonntag, K. C., et al. Late-onset Alzheimer's disease is associated with inherent changes in bioenergetics profiles. Sci Rep. 7 (1), 14038(2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon