Method Article
Génération et caractérisation d’astrocytes dérivés de cellules souches pluripotentes induites humaines dépourvus de ribonucléoprotéine messagère X fragile
Dans cet article
Résumé
Nous décrivons ici un protocole qui facilite la différenciation des cellules souches pluripotentes induites humaines en astrocytes fonctionnels spécifiques du cerveau antérieur. Cela permet d’étudier le rôle des cellules gliales dans la pathogenèse des troubles neurodéveloppementaux, tels que le syndrome de l’X fragile, et de modéliser d’autres troubles cérébraux.
Résumé
Le syndrome de l’X fragile (FXS), l’une des principales causes héréditaires de troubles du spectre autistique et de déficience intellectuelle, a été étudié de manière approfondie à l’aide de modèles de rongeurs. Plus récemment, des systèmes modèles dérivés de cellules souches humaines ont également été utilisés pour obtenir des informations mécanistes sur la physiopathologie du FXS. Cependant, ces études se sont concentrées presque exclusivement sur les neurones. De plus, malgré les preuves croissantes d’un rôle clé de la glie dans la fonction neuronale dans la santé et la maladie, on sait peu de choses sur la façon dont les astrocytes humains sont affectés par le FXS.
Par conséquent, dans cette étude, nous avons réussi à développer un protocole qui capture les étapes spatio-temporelles clés du développement du cerveau et s’aligne également sur le processus de gliogenèse. Ensemble, ces éléments offrent un cadre utile pour l’étude des troubles neurodéveloppementaux. Tout d’abord, nous avons modelé les cellules souches pluripotentes induites humaines dans la lignée neuroectodermique avec un double suppresseur de mères contre l’inhibition décapentaplégique (SMAD) et de petites molécules. Par la suite, nous avons utilisé des facteurs de croissance et des cytokines spécifiques pour générer des cellules progénitrices astrocytaires (APC) de contrôle (CTRL) et de patients FXS. Le traitement des CPA avec le facteur neurotrophique ciliaire, une cytokine différenciante, a régulé et conduit les cellules progénitrices vers la maturation astrocytaire, produisant des astrocytes exprimant la protéine acide fibrillaire gliale spécifique du cerveau antérieur.
Nous avons constaté que ces astrocytes sont fonctionnels, comme en témoignent leurs réponses calciques à l’application d’ATP, et qu’ils présentent un métabolisme glycolytique et mitochondrial dérégulé dans le FXS. Pris ensemble, ces résultats fournissent une plate-forme expérimentale utile d’origine humaine pour l’étude des conséquences autonomes et non cellulaires des altérations de la fonction astrocytaire causées par des troubles neurodéveloppementaux.
Introduction
Le syndrome de l’X fragile (FXS), une forme héréditaire courante de déficience intellectuelle et de trouble du spectre autistique (TSA), est causé par l’absence de ribonucléoprotéine messagère de l’X fragile (FMRP) produite par le gène de la ribonucléoprotéine messagère de l’X fragile 1 (FMR1) (OMIM : #300624, https://www.omim.org/entry/300624). FMRP joue un rôle dans la régulation de la traduction de l’ARNm, la formation et le transport des granules d’ARNm, et la régulation de l’expression des gènes par les microARN1. Ainsi, la perte de FMRP affecte non seulement le développement du cerveau, mais aussi le fonctionnement du cerveau adulte. Les niveaux de transcription de l’ARNm de FMR1 et l’immunomarquage pour FMRP dans le cerveau ont montré une expression neuronale élevée, ainsi qu’une expression significative dans les cellules gliales ainsi que2. Cependant, une grande majorité des études antérieures sur des modèles animaux de FXS se sont principalement concentrées sur les neurones et les aberrations de leur fonction. Par conséquent, on sait peu de choses sur le rôle de la glie dans FXS3. Traditionnellement considérées comme des cellules de « soutien passif »4, il existe de plus en plus de preuves que les astrocytes sont essentiels à la médiation d’un large éventail de fonctions neuronales 5,6, notamment la promotion de la synaptogenèse7, le raffinement des circuits neuronaux en développement8 et le recyclage des neurotransmetteurs9. En parallèle, il existe de plus en plus de preuves du rôle des astrocytes dans la pathogenèse de la maladie et de nombreuses affections neurologiques ont été associées à la dysfonction astrocytaire10.
Alors qu’une grande partie des travaux antérieurs utilisant des modèles animaux de FXS se concentraient sur l’identification et la validation de diverses cibles moléculaires dans les neurones pour le traitement du FXS, ces résultats précliniques n’ont pas toujours conduit à des résultats cliniques positifs. De plus, les revers des récents essais cliniques soulignent également la nécessité de systèmes modèles basés sur l’homme. Les modèles de troubles neurologiques basés sur des cellules cérébrales dérivées de cellules souches humaines offrent une stratégie puissante pour combler ce fossé entre les connaissances mécanistes des études animales et le succès limité des résultats cliniques pour les patients. Cependant, seule une poignée de ces études se sont concentrées sur les astrocytes et cela aussi principalement sur les astrocytes d’origine spinale. Ceci, à son tour, est pertinent à la lumière des études montrant que la structure et la fonction des astrocytes varient entre les régions du cerveau11,12. Ainsi, une meilleure compréhension des changements induits par la maladie dans les astrocytes humains doit également prendre en compte ces différences spécifiques à la région du cerveau dans les astrocytes. Cependant, les modèles de troubles neurodéveloppementaux utilisant des astrocytes dérivés de cellules souches humaines spécifiques au cerveau antérieur restent relativement sous-explorés13. Par conséquent, pour commencer à combler ces lacunes, nous décrivons des protocoles pour générer des astrocytes spécifiques au cerveau antérieur à partir de cellules souches pluripotentes induites (iPSC) dérivées de patients portant des mutations FXS ; De plus, nous montrons que les astrocytes sont fonctionnels et présentent un métabolisme altéré.
Protocole
Toutes les expériences utilisant des cellules souches pluripotentes induites humaines (hiPSC) (tableau 1) ont été réalisées après l’obtention des approbations réglementaires institutionnelles appropriées. La figure 1A représente le protocole de différenciation complet des hiPSC vers les astrocytes matures spécifiques du cerveau antérieur.
1. Maintenance et extension des hiPSC
- Un jour avant de plaquer les iPSC, enduire un plat à 6 puits de Matrigel dilué 1:60 (une matrice extracellulaire [ECM]) dans Advanced Dulbecco’s Modified Eagle Medium/Ham’s F-12 (DMEM/F12) et conserver à 2-8 °C.
REMARQUE : Évitez de stocker des boîtes enrobées de Matrigel pendant >5 jours à 2-8 °C, car cela peut entraîner une dégradation des protéines dans l’ECM au fil du temps. Pour éviter le dessèchement du plat, enrobez un minimum de 1 mL de Matrigel dilué 1:60 par puits d’un plat à 6 puits et étalez-le uniformément sur le plat. En cas d’utilisation le même jour, conservez la plaque dans un incubateur humidifié à 37 °C et 5 % de CO2 pendant 1 h. - Le jour de la culture des hiPSC, retirez le matériau d’enrobage et ajoutez 1 mL de milieu Essential 8 complet (milieu basal E8 avec supplément Essential 8) avec l’inhibiteur de ROCK à une concentration finale de 1x et conservez la boîte dans un incubateur à 37 °C et 5 % de CO2 avant d’ajouter les hiPSC.
- Remettre en suspension les hiPSC dans le milieu E8 complet avec l’inhibiteur de ROCK à une concentration finale de 1x pour une meilleure adhérence des colonies.
REMARQUE : Ne préparez pas de grandes quantités de milieu de culture. Ne préparez que pendant 3-4 jours maximum et conservez-le à 2-8 °C. - Le lendemain, réapprovisionnez le milieu E8 complet sans l’inhibiteur de ROCK jusqu’à ce que les cellules soient confluentes à 80 % (environ 4 à 5 jours).
- Lorsqu’elles atteignent 80 % de confluence, détachez les colonies par voie enzymatique.
- Pour faire passer les CSPi, retirez les milieux épuisés de la boîte et ajoutez 1 mL par puits d’un mélange préchauffé (37 °C) de collagénase (2 mg/mL) et de dispase (1 mg/mL) dans un rapport de 1:1 et incubez à 37 °C pendant plus de 20 à 30 minutes pour permettre aux colonies de CSPi de se soulever.
REMARQUE : Ne laissez pas la vaisselle à 37 °C pendant plus de 20-30 min ; Au-delà de cette période, les colonies se désintégreront et les cellules mourront.
- Pour faire passer les CSPi, retirez les milieux épuisés de la boîte et ajoutez 1 mL par puits d’un mélange préchauffé (37 °C) de collagénase (2 mg/mL) et de dispase (1 mg/mL) dans un rapport de 1:1 et incubez à 37 °C pendant plus de 20 à 30 minutes pour permettre aux colonies de CSPi de se soulever.
- Lorsque les colonies commencent à se soulever, sortez les plats de l’incubateur et neutralisez l’activité enzymatique en ajoutant 2 ml de solution saline tamponnée au phosphate de Dulbecco (DPBS).
- Grattez les colonies à l’aide de DPBS et prélevez la suspension dans un tube conique de 15 mL à l’aide d’une pipette sérologique de 10 mL à gros calibre.
- Triturez doucement la suspension 2 à 3 fois pour briser les colonies à l’aide d’une pipette sérologique de 10 mL, et laissez les colonies se stabiliser.
REMARQUE : Ne faites pas des colonies des cellules uniques ; Cela peut empêcher les cellules d’adhérer à la boîte et provoquer une plus grande mort cellulaire. De plus, ne les laissez pas comme de grandes colonies ; Cela conduira à des colonies plus différenciées plus tard. - Une fois que les colonies se sont stabilisées (environ après 2 min), aspirez le mélange d’enzymes DPBS en laissant environ 1 mL dans le tube.
- Ajoutez 2 ml de DPBS dans le tube, mélangez les colonies en tapotant et laissez-les se déposer. Répétez cette opération 2 fois pour éliminer toute l’enzyme résiduelle des colonies. Retirer autant de surnageant que possible après le 2elavage, remettre les colonies en suspension dans 1 mL de milieu E8 complet et les déposer dans un plat fraîchement préparé comme mentionné aux étapes 1.2-1.3.
- Alternativement, cryoconservez une proportion de colonies en utilisant 10 % de diméthylsulfoxyde (DMSO) comme solution cryoprotectrice pour une expansion et une utilisation futures.
- Préparez fraîchement le milieu de cryoconservation en mélangeant 90 % de milieu E8 complet et 10 % de DMSO comme solution cryoprotectrice et placez-le à 2-8 °C jusqu’à utilisation.
- Pour cryoconserver, suivez les étapes 1.5 à 1.10.
- Lors de l’étape 1.10, après le 2èmelavage, laissez les colonies se déposer et retirez le surnageant. Aux colonies, ajouter 1 mL de milieu de cryoconservation froid fraîchement préparé et transférer dans des flacons cryogéniques.
REMARQUE : Après avoir ajouté le milieu de cryoconservation aux colonies, transférez rapidement le contenu dans des cryoflacons. Retarder peut entraîner un taux de réactivation plus faible, car le DMSO est un cryoprotecteur qui peut endommager les cellules. - Placez immédiatement les flacons cryogéniques dans une cryoboîte et conservez-les au congélateur à -80 °C pendant la nuit.
- Le lendemain, placez tous les cryoflacons dans un réservoir d’azote liquide (réservoir LN2 ) pour une utilisation future.
REMARQUE : Les hiPSC doivent être systématiquement caryotypées à l’aide de la bande G (figure supplémentaire S1) pour toute anomalie, caractérisées par immunocytochimie pour la pluripotence (figure 1B) et testées pour les mycoplasmes.- Pour caractériser les hiPSC à l’aide de l’immunocytochimie, plaquez les colonies sur des lamelles en verre de 13 mm recouvertes d’un Matrigel 1:60 et autoclavées. Une fois qu’elles sont confluentes à 40 %, laver les cellules avec du PBS-T (PBS-0,1 % Tween 20), fixer avec du paraformaldéhyde à 4 % pendant 10 min, perméabiliser avec du Triton X-100 à 0,3 % dans du PBS pendant 10 min, et bloquer avec de l’albumine sérique bovine (BSA) à 3 % dans du PBS pendant 1 h pour éviter la liaison non spécifique.
- Après le blocage, incubez les cultures avec des anticorps primaires pendant 1 h, puis lavez-les 3 fois avec du PBS-T pendant 5 minutes chacune, puis avec les anticorps secondaires correspondants (tableau des matériaux) dans l’obscurité pendant 1 h.
- Montez les lamelles sur des lames de verre avec un support de montage et acquérez des images par balayage laser confocal à 405 nm, 488 nm, 561 nm et 633 nm. Capturer des images à 512 x 512 pixels ; réglez la taille du pas Z à 0,5 μm avec 1 unité aérée de diamètre de sténopé.
2. Génération et caractérisation de cellules progénitrices astrocytaires (CPA)
- Soulever par voie enzymatique les hiPSC comme mentionné aux étapes 1.5 à 1.10 et les déposer sur une boîte de culture en suspension non adhérente (100 mm) avec un milieu chimiquement défini14 contenant 50 % de milieu Dulbecco modifié d’Iscove (IMDM), 50 % de mélange de nutriments F-12 de Ham (F12), 5 mg/mL de BSA, 1 % de concentré de lipides chimiquement définis (CD-Lipid), 450 μM de monothioglycérol, 7 μg/mL d’insuline, 15 μg/mL de transferrine, Pénicilline-Streptomycine à 1 % supplémenté en mitogènes de structuration du cerveau antérieur N-acétyl cystéine (1 mM), LDN 193189 (0,1 μM) et SB431542 (10 μM) pendant 7 jours.
REMARQUE : Les inhibiteurs de petites molécules SB431542 et LDN-193189 (LDN) sont des inhibiteurs des protéines morphogénétiques osseuses et des voies de signalisation du facteur de croissance transformant bêta.
À partir de ce moment, le support a été réapprovisionné une fois tous les 2 jours ou selon le protocole lundi/mercredi/vendredi. - Placez la suspension cellulaire sur un agitateur orbital à 40 tr/min pendant 7 jours dans des conditions normoxiques pour favoriser le développement des corticosphères (Figure 1A).
- Le jour 8, transférez les corticosphères dans un milieu de prolifération cellulaire contenant du DMEM/F12 avancé avec 1 % d’antibiotique-antimycosique, 1 % de supplément de N2, 1 % de substitut de glutamine, 0,1 % de supplément de B27 et 2,5 ng/mL de facteur de croissance des fibroblastes basiques (bFGF) pendant 7 jours.
- Induire la spécification gliale des sphères en les soumettant à un milieu d’enrichissement glial contenant du DMEM/F12 avancé avec 1 % d’antibiotique-antimycosique, 1 % de N2, 1 % de substitut de glutamine, 0,1 % de supplément de B27, 20 ng/mL de facteur de croissance épidermique (EGF), bFGF-H (20 ng/mL bFGF-5 mg/mL d’héparine) pendant 2 semaines pour obtenir des gliosphères précoces (Figure 1A).
- Pour la maturation des gliosphères précoces, remplacez bFGF-H par 20 ng/mL de facteur inhibiteur de la leucémie (LIF) et maintenez les sphères pendant 4 semaines.
- Après 4 semaines dans le milieu de maturation, maintenez les sphères dans le milieu d’enrichissement glial pendant des périodes prolongées. Pour éviter l’agrégation et la perte de viabilité, toutes les 2 semaines, hachez mécaniquement les gliosphères à l’aide d’une lame industrielle stérile et remplacez l’ensemble du milieu par de la DNase I pour éliminer les fragments d’ADN générés par le hachage.
- Dissociez les gliosphères en monocouches d’APC à l’aide d’un kit de dissociation Papain et plaquez-les sur une boîte adhérente traitée par culture cellulaire avec dilution 1:80 de revêtement Matrigel.
- Propager les APC dans un milieu d’enrichissement glial jusqu’à ce qu’ils confluent à 80 % et les faire passer enzymatiquement à l’aide du milieu de détachement de cellules enzymatiques (voir le tableau des matériaux).
- Vers le passage :
- Retirez le support usagé et collectez-le dans un tube conique. Aux cellules, ajoutez le milieu enzymatique de décollement cellulaire et attendez 1 à 2 min. Une fois que les cellules commencent à se détacher, ajoutez le milieu épuisé pour neutraliser l’activité enzymatique.
- Recueillir la suspension cellulaire et centrifuger à 800 x ɡ pendant 2 min.
- Aspirez le surnageant, remettez les cellules en suspension dans un milieu d’enrichissement glial et placez environ 1 x 106 cellules/puits dans une boîte à 6 puits recouverte de Matrigel 1:80.
- Pour la cryoconservation :
- Remettre les cellules en suspension dans un mélange froid de milieu de prolifération de la corticosphère à 90 % (sans bFGF) et de 10 % de cryoprotecteur. Transférez les cellules remises en suspension dans des cryoflacons.
- Déplacez immédiatement les cryoflacons dans une cryoboîte et conservez-les au congélateur à -80 °C pendant la nuit.
- Le lendemain, placez tous les cryoflacons dans un réservoir d’azote liquide (réservoir LN2 ) pour une utilisation future.
REMARQUE : Les APC ont été cryoconservés avec 10 % de DMSO jusqu’au passage numéro 6.
- Caractériser les CPA dissociés par immunomarquage (comme mentionné aux étapes 1.15.1-1.15.3) avec des marqueurs de vimentine et de facteur nucléaire IA (NFIA) (Figure 1C,D) (Voir le tableau des matériaux pour les dilutions).
- Pour confirmer la spécificité régionale du cerveau antérieur des APC dérivées de hiPSC, testez les cellules pour un marqueur de cerveau antérieur tel que la boîte de tête de fourche humaine G1 (hFOXG1) (positif) et un marqueur de cerveau postérieur tel que l’Homeobox B4 humain (hHOXB4) (négatif) en utilisant la qPCR en temps réel (Figure 1E,F).
REMARQUE : Les séquences d’amorces utilisées pour ces expériences ont été énumérées dans le tableau supplémentaire S1.
3. Génération et caractérisation d’une population homogène d’astrocytes spécifiques du cerveau antérieur
- Différenciation des astrocytes des APC
- Utilisez le milieu de différenciation astrocytaire (ADM) pendant 14 jours pour différencier les APC en astrocytes. Le milieu de différenciation astrocytaire contient du neurobasal, 1 % d’antibiotique-antimycosique, 1 % de substitut de glutamine, 1 % de supplément N2, 0,2 % de supplément B27, 1 % de milieu d’acides aminés non essentiels (NEAA) et 10 ng/mL de facteur neurotrophique ciliaire (CNTF).
- Confirmer l’identité astrocytaire en utilisant l’immunomarquage (comme mentionné aux étapes 1.15.1-1.15.3.) avec la protéine acide fibrillaire gliale (GFAP) et les marqueurs d’expression S100β (Figure 2A,B).
- Synthèse protéique de novo dans les APC et les astrocytes
- Maintenir les APC et les astrocytes du cerveau antérieur dérivés de hiPSC sur des lamelles en verre stériles autoclavées de 13 mm et les immunocolorer comme mentionné aux étapes 1.15.1-1.15.3.
- Pour la synthèse de novo des protéines, utilisez la méthode de marquage fluorescent non canonical des acides aminés (FUNCAT), expliquée brièvement aux étapes 3.2.3 à 3.2.6 (Figure 3A).
- Pour éviter une carence en méthionine, retirer le milieu de croissance des cultures et le remplacer par un milieu exempt de méthionine et de cystéine mélangé à 1 mM de L-azidohomoalanine (AHA) pendant 30 min à 37 °C et 5 % de CO2.
- Laver les cultures avec du PBS-T, fixer avec du paraformaldéhyde à 4 % pendant 10 min, perméabiliser avec du Triton X-100 à 0,3 % dans du PBS pendant 10 min, et bloquer avec du BSA à 3 % dans du PBS pendant 1 h pour éviter une liaison non spécifique.
- Incuber les cultures dans l’obscurité pendant 1 h à température ambiante avec un mélange réactionnel de chimie cellulaire Click et d’Alcyne Alexa Fluor 647, suivi d’anticorps primaires pendant 1 h et d’anticorps secondaires correspondants (tableau des matériaux) pendant 1 h (Figure 3B).
- Montez les lamelles sur des lames de verre à l’aide d’un support de montage et utilisez-les pour une analyse d’image plus approfondie.
- Acquérez des images par balayage laser confocal à 405 nm, 488 nm, 561 nm et 633 nm.
- Capturer des images à 512 x 512 pixels ; réglez la taille du pas Z à 0,5 μm avec 1 unité aérée de diamètre de sténopé.
- Maintenez les paramètres de microscopie et d’imagerie à un réglage constant sur tous les types de cellules. Capturez des images pour chaque répétition biologique au cours de la même session.
- Effectuez des mesures d’intensité à l’aide de n’importe quel logiciel d’analyse d’images standard (par exemple, Fiji ou Imaris). Dans Imaris, utilisez le module SURFACES pour obtenir des mesures volumétriques des corps cellulaires positifs pour le signal FUNCAT (fluorescent non-canonical amino acid markging) avec des Vimentines (APCs) et des GFAP (astrocytes) limitant la plage de voxels (250-350) pour éliminer les débris.
REMARQUE : Maintenez les paramètres sur toutes les réplicats biologiques pour les deux types de cellules.
- Ondes calciques oscillatoires induites par l’adénosine 5′-triphosphate (ATP)
- Déposer les astrocytes sur une boîte à fond de verre (35 mm) à 5 × 103 cellules/boîte pour mesurer la réponse cellulaire à l’ATP.
- Laissez les cellules adhérer au fond du verre pendant 24 h, lavez-les 3 fois avec HBSS (20 mM HEPES, 137 mM NaCl, 5 mM KCl, 10 mM Glucose, 1 mM MgCl2, pH = 7,3) sans calcium, et incubez dans un milieu de culture avec un colorant métrique de rapport 5 μM Fura-2AM et 0,02 % de Pluronics F127 pendant 1 h à température ambiante.
REMARQUE : Fura-2AM est un produit chimique sensible à la lumière et à la température ; Conserver à -20 °C. - Après l’incubation, laver 2x avec un milieu de culture et remplacer par 2 mM de Ca2+ contenant HBSS et imager les cellules à une vitesse de 2 FPS à l’aide d’un objectif à huile 60x (1,35 NA) dans un microscope inversé à compensation de dérive de focale.
- Enregistrer les réponses calciques induites par l’ATP par l’application en bain d’ATP à la concentration finale de 5 mM à la 25e s (Figure 4A).
- Dessinez des régions d’intérêt autour de chaque cellule à l’aide de Fiji/ImageJ et calculez le rapport F340/F380 à tous les points temporels (Figure 4B).
- Essais métaboliques cellulaires
REMARQUE : Le taux de consommation d’oxygène (OCR) et le taux d’acidification extracellulaire (ECAR) des cellules vivantes ont été mesurés conformément au protocole du fabricant (voir le tableau des matériaux).- Un jour avant le dosage, laisser les astrocytes adhérer à la microplaque (densité d’ensemencement de 1,5 × 104 ) dans le milieu CNTF et incuber à 37 °C avec 5 % de CO2.
- Pour le test de stress de glycolyse :
- Remplacer le milieu de culture cellulaire par un milieu de base complété par 2 mM de glutamine (pH ajusté à 7,4) et incuber dans une chambre sans CO2 à 37 °C pendant 1 h.
REMARQUE : Le pH doit être ajusté dans un bain d’eau à 37 °C. - Insérez la plaque de la cartouche dans l’instrument pour calibrer le capteur. Après l’étalonnage, remplacez la plaque de la cartouche par la plaque de culture cellulaire à la concentration finale des composants suivants du kit : 10 mM de glucose, 1 μM d’oligomycine et 50 mM de 2-désoxy-glucose (2-DG) (Figure 5A).
- À la fin du test, lyser les cellules et estimer la teneur totale en protéines.
- Remplacer le milieu de culture cellulaire par un milieu de base complété par 2 mM de glutamine (pH ajusté à 7,4) et incuber dans une chambre sans CO2 à 37 °C pendant 1 h.
- Pour le dosage du stress mitochondrique :
- Remplacer le milieu CNTF par un milieu de base complété par 2 mM de glutamine, 1 mM de pyruvate, 10 mM de glucose (pH ajusté à 7,4) dans un bain-marie à 37 °C.
- Incuber la plaque dans une chambre sans CO2 à 37 °C pendant 1 h. Pendant ce temps, insérez la plaque de la cartouche dans l’instrument pour calibrer le capteur.
- Après l’étalonnage, remplacez la plaque de cartouche par la plaque de culture cellulaire avec une concentration finale des composants suivants du kit : 1,5 μM d’oligomycine, 1 μM de cyanure de carbonyle-4 (trifluorométhoxy) phénylhydrazone (FCCP) et 0,5 μM de roténone/antimycine A (Figure 5C).
- À la fin du test, lyser les cellules et estimer la teneur totale en protéines.
Résultats
Des colonies de cellules souches pluripotentes induites humaines (hiPSC) ont été maintenues à l’aide d’un milieu défini disponible dans le commerce et immunomarqués pour les marqueurs de pluripotence, Oct4 et Nanog (Figure 1B). Les APC FXS et CTRL ont montré des proportions comparables et fortement enrichies de cellules immunopositives pour la vimentine et la NFIA (Figure 1C, D). Nous avons constaté que les APC dérivées d’hiPSC sains et FXS présentaient une régulation positive substantielle de FOXG1 par rapport à HOXB4 (Figure 1F), ce qui correspond à une identité prédominante du cerveau antérieur. Nous avons observé une réduction significative du nombre d’astrocytes exprimant GFAP dans les groupes FXS par rapport aux astrocytes CTRL sans affecter le nombre d’astrocytes exprimant S100β (Figure 2B). L’analyse par transfert Western a montré que les astrocytes générés par les deux lignées FXS n’exprimaient pas FMRP (Figure 2C,D). La quantification des protéines nouvellement synthétisées n’a pas révélé de différence significative entre les lignées CTRL et FXS dans les APC ou les astrocytes (Figure 3C). Cependant, nous avons constaté que la synthèse des protéines était systématiquement plus élevée dans les APC que dans leurs astrocytes respectifs pour chaque lignée (Figure 3C). Ces résultats suggèrent une réduction spécifique à l’étape de la synthèse des protéines de novo des APC aux astrocytes dans les lignées FXS et CTRL.
Les astrocytes témoins, ainsi que les astrocytes FXS, ont présenté des transitoires calciques induits par l’ATP (Figure 4A). Cependant, une analyse plus détaillée a révélé plusieurs différences clés entre les transitoires calciques individuels enregistrés à partir des astrocytes CTRL, FXS1 et FXS2 (Figure 4B). Nous avons classé les astrocytes en deux catégories : répondeurs et non-répondeurs à l’ATP en fonction de la présence ou de l’absence d’une réponse calcique, respectivement (Figure 4C). Le nombre de répondeurs dans FXS1 et FXS2 était significativement inférieur à CTRL. Plus précisément, nous avons observé une réduction significative de l’amplitude maximale du premier transitoire calcique évoqué après l’application d’ATP dans les deux raies astrocytaires FXS (Figure 4D). De plus, la durée totale du premier transitoire calcique était significativement plus courte dans les lignées FXS1 et FXS2 que dans les lignées CTRL (Figure 4E). Cependant, le nombre d’événements calciques (quantifié comme le nombre total d’événements par 250 s ; Figure 4F), était comparable entre les lignes. Pris ensemble, ces résultats révèlent des altérations des réponses calciques induites par l’ATP dans les astrocytes FXS dérivés de hiPSC. Les résultats de l’ECAR suggèrent un taux plus élevé de glycolyse, de capacité glycolytique et de réserve glycolytique dans les astrocytes FXS dérivés de hiPSC (Figure 5A,B). Nous n’avons trouvé aucune différence significative dans la respiration basale dans les astrocytes CTRL et FXS. La production cellulaire d’ATP n’a montré aucune différence significative entre les astrocytes CTRL et FXS. La stimulation FCCP a induit une respiration maximale significativement plus faible dans les astrocytes FXS que dans les astrocytes CTRL (Figure 5C,D).

Figure 1 : Dérivation de cellules progénitrices astrocytaires spécifiques du cerveau antérieur à partir de hiPSC. (A) Flux de travail illustratif pour la génération d’astrocytes à partir de hiPSC. (B) Images représentatives de hiPSC montrant une expression comparable d’Oct4/Nanog dans les raies CTRL et FXS. (C) La population homogène d’APC dérivées de hiPSC a exprimé un nombre similaire de cellules positives à la vimentine et à la NFIA. (D) Aucune différence significative dans l’expression de la vimentine et de la NFIA entre les APC CTRL et FXS. L’analyse statistique a été effectuée à l’aide d’une ANOVA à deux facteurs suivie d’une comparaison par paires de Tukey. (E) Schéma illustrant l’expression spécifique à la région de FOXG1 et HOXB4 au cours du développement in vivo. (F) Représentation graphique de l’expression de FOXG1 par rapport à HOXB4 (à l’aide de la qRT-PCR) à travers les génotypes, suggérant une propension à la lignée du cerveau antérieur. Analyse statistique par ANOVA à deux facteurs suivie du test de comparaison multiple de Sidak. Pour toutes les expériences, N = 3 répétitions biologiques. p < 0,001. Barre d’échelle = 50 μm. Abréviations : hiPSCs = cellules souches pluripotentes induites par l’homme ; CTRL = Contrôle ; FXS = Syndrome de l’X fragile ; APCs = cellules progénitrices des astrocytes. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Réduction du nombre d’astrocytes FXS dérivés de la protéine hiPSC positive à l’acide glial. (A) Images représentatives d’astrocytes dérivés de hiPSC présentant une expression de S100β et GFAP. (B) Le pourcentage d’astrocytes positifs au GFAP était significativement plus faible dans les astrocytes dérivés de FXS par rapport à CTRL, bien que les deux aient présenté une expression similaire de S100β. Signification statistique déterminée par l’ANOVA à deux facteurs suivie de la comparaison par paires de Tukey. (C) Immunoblot représentant l’expression de FMRP dans les astrocytes de CTRL et FXS. (D) Graphiques montrant l’absence de FMRP dans les astrocytes FXS dérivés de hiPSC. Analyse statistique réalisée par ANOVA à facteur unique avec comparaison par paires de Tukey. Pour toutes les expériences ci-dessus, N = 3 répétitions biologiques. Barre d’échelle = 50 μm. ***p < 0,001. Abréviations : hiPSCs = cellules souches pluripotentes induites par l’homme ; CTRL = Contrôle ; FXS = Syndrome de l’X fragile ; GFAP = protéine acide fibrillaire gliale ; FMRP = ribonucléoprotéine messagère de l’X fragile. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Réduction de la synthèse des protéines en raison de la différenciation des APC en astrocytes. (A) Flux de travail schématique décrivant le marquage et la visualisation de la synthèse des protéines dans les APC et les astrocytes dérivés des hiPSC à l’aide du marquage métabolique FUNCAT. (B) Images représentatives d’APC dérivées de hiPSC positives à la vimentine (en haut) et d’astrocytes positifs à GFAP (en bas) avec marquage FUNCAT/anticorps anti-noyau humain (hNA) dans des cellules dérivées de CTRL et de FXS. (C) Le FUNCAT/volume des APC dérivés des hiPSC était significativement plus élevé que celui des astrocytes dérivés correspondants. Pour toutes les expériences, N = 3 répétitions biologiques. Barre d’échelle = 50 μm. **p < 0,01, ***p < 0,001. Les moustaches représentent 1,5 × IQR. Abréviations : hiPSCs = cellules souches pluripotentes induites humaines ; CTRL = Contrôle ; FXS = Syndrome de l’X fragile ; GFAP = protéine acide fibrillaire gliale ; FUNCAT = marquage fluorescent non canonical des acides aminés. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Signalisation Ca2+ déficiente évoquée par l’ATP dans les astrocytes FXS dérivés de hiPSC. (A) Traces représentatives de transitoires Ca2+ enregistrées à partir d’astrocytes individuels lors de l’application externe d’ATP à 25e s. (B) Rapports F340/F380 moyens représentant des transitoires Ca2+ sur 250 s après l’application d’ATP. (C) Données groupées montrant un pourcentage plus élevé de non-répondeurs à l’ATP dans les astrocytes FXS dérivés de hiPSC. Signification statistique déterminée par l’ANOVA à deux facteurs avec comparaison par paires de Tukey. (D-F) Quantification de la réponse du premier pic (amplitude et durée) et de la fréquence des événements. (D) Montre une réduction significative de l’amplitude. Signification statistique déterminée par l’ANOVA à facteur unique avec comparaison par paires de Tukey et durée (E) dans les astrocytes FXS dérivés de hiPSC. Signification statistique déterminée par le test de Kruskal-Wallis avec le test de comparaison multiple de Dunn ; ensemble de données représenté sous forme de rangs moyens avec SEM. (F) Représentation graphique de la fréquence transitoire Ca2+ dans les astrocytes FXS dérivés de hiPSC. Analyse statistique par ANOVA à facteur unique avec comparaison par paires de Tukey. Pour toutes les expériences, N = 3 répétitions biologiques ; n = 19 cellules pour chaque lignée cellulaire. *p < 0,05, **p < 0,01, ***p < 0,001. Les barres d’erreur représentent le MEB. # = Nombre. Abréviations : hiPSCs = cellules souches pluripotentes induites humaines ; CTRL = Contrôle ; FXS = Syndrome de l’X fragile ; GFAP = protéine acide fibrillaire gliale. Veuillez cliquer ici pour voir une version agrandie de cette figure.
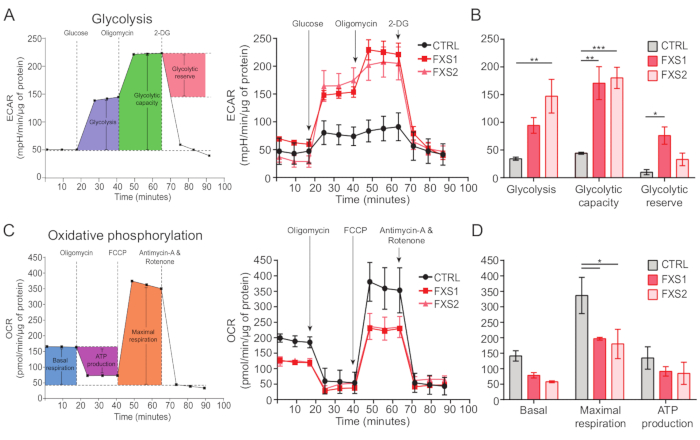
Figure 5 : Déficits bioénergétiques dans les astrocytes FXS dérivés de hiPSC. (A) Graphique linéaire représentant le taux d’acidification extracellulaire des astrocytes dérivés des hiPSC (CTRL et FXS) après ajout séquentiel de 10 mM de glucose, 1 μM d’oligomycine et 50 mM de 2-DG et tracé sur un graphique linéaire. (B) La glycolyse, la capacité glycolytique et la quantification de la réserve glycolytique ont montré une augmentation des astrocytes FXS dérivés de hiPSC. Analyse statistique réalisée par ANOVA à deux facteurs avec comparaison par paires de Tukey. (C) Graphique linéaire illustrant la mesure du taux de consommation d’oxygène par addition séquentielle de 1,5 μM d’oligomycine, de 1 μM de FCCP et de 0,5 μM d’antimycine A et de roténone. (D) La respiration basale, la respiration maximale et la production d’ATP ont été quantifiées à partir du graphique linéaire et les astrocytes FXS dérivés de hiPSC ont montré une diminution significative de la respiration maximale par rapport aux astrocytes CTRL. Analyse statistique réalisée par ANOVA à deux facteurs avec comparaison par paires de Tukey. Pour toutes les expériences, N = 2 répétitions biologiques. *p < 0,05, **p < 0,01, ***p < 0,001. Abréviations : hiPSCs = cellules souches pluripotentes induites par l’homme ; CTRL = Contrôle ; FXS = Syndrome de l’X fragile ; ECAR = taux d’acidification extracellulaire ; 2-DG = 2-désoxyglucose ; OCR = taux de consommation d’oxygène ; FCCP = cyanure de carbonyle-4 (trifluorométhoxy) phénylhydrazone. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| ID dans le manuscrit | Identification à la source | Âge | Sexe | Nom de la lignée cellulaire reprogrammé | Méthode de reprogrammation | Type de cellule de départ | Caryotype de la bande G |
| (années) | |||||||
| CTRL | ND30625 | 76 | M | Référence CS25iCTR-18nxx | Vecteurs épisomaux | Fibroblaste | Normal |
| Le FXS1 | GM07072 | 22 | M | Référence CS072iFXS-n4 | Vecteurs épisomaux | Fibroblaste | Normal |
| Le FXS2 | GM05848 | 4 | M | CS848iFXS-n5 | Vecteurs épisomaux | Fibroblaste | Normal |
Tableau 1 : Lignées cellulaires utilisées dans cette étudepour générer des astrocytes spécifiques au cerveau antérieur.
Figure supplémentaire S1 : Rapport d’analyse chromosomique. Caryotype de bandes GTG normales de (A) CTRL 46, XY, (B) FXS1 46, XY, et (C) FXS2 46, XY. Veuillez cliquer ici pour télécharger cette figure.
Tableau supplémentaire S1 : Amorces utilisées dans cette étude pour la caractérisation des cellules. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Ici, nous décrivons une méthode pour générer des astrocytes humains dérivés d’iPSC qui servent de plate-forme de test pour caractériser les changements fonctionnels induits par FXS. Ces astrocytes sont fonctionnellement viables en culture et présentent diverses propriétés, comme en témoigne une série de mesures effectuées dans la présente étude. Une étape critique de ce protocole est la conversion initiale des CSPi en corticosphères à l’aide de la méthode de lifting enzymatique. À ce stade, l’optimisation du temps d’incubation de la collagénase de type IV et de la dispase est cruciale. Si cela n’est pas optimisé, cela peut provoquer la désintégration des iPSC ou la formation de cellules uniques, ce qui empêchera la formation de corticosphères. Ces corticosphères sont modelées en fonction de la spécificité du cerveau antérieur à l’aide de la double inhibitionSMAD 15 et sont ensuite différenciées en cellules progénitrices des astrocytes (APC). Pour obtenir une population élevée d’APC, le découpage mécanique des gliosphères est crucial. La qPCR en temps réel peut être utilisée pour confirmer la spécificité du cerveau antérieur ; nous avons observé une augmentation significative du changement de pli de FOXG1, un marqueur du cerveau antérieur, par rapport à HOXB4, un marqueur de la moelle épinière.
Les APC sont ensuite différenciés en astrocytes à l’aide de CNTF, un activateur de la voie de signalisation JAK-STAT16. Les résultats de cette étude indiquent que les astrocytes différenciés en phase terminale dérivés des iPSC FXS ont des niveaux de GFAP plus faibles, ce qui suggère une maturité altérée. Bien que nos analyses n’aient pas trouvé de différence dans la synthèse des protéines de novo dans les astrocytes FXS et les APC par rapport aux témoins, nous avons constaté une réduction de la synthèse des protéines de novo dans la transition des APC aux astrocytes dans les lignes FXS et de contrôle. La réduction significative de la traduction entre les APC et les astrocytes est cohérente avec une traduction plus rationalisée et organisée qui a été démontrée pour les neurones par rapport aux cellules précurseurs neurales17. Des travaux antérieurs qui ont examiné la synthèse aberrante des protéines ont utilisé des tissus cérébraux entiers, des neurones et des fibroblastes dérivés de patients, et non des astrocytes ou des APC18. Les seuls rapports publiés sur la synthèse des protéines à partir de lignées dérivées de patients ne concernent pas les astrocytes, mais les cellules lymphoblastoïdes19 et les fibroblastes 20,21. À la lumière de ces variations d’une étude à l’autre, les études futures bénéficieraient de l’utilisation de mesures FUNCAT dans les astrocytes dérivés de lignées supplémentaires dérivées de patients FXS.
Nous avons évalué la fonctionnalité des astrocytes en mesurant la dynamique calcique intracellulaire22 et la bioénergétique. Les astrocytes FXS ont montré une amplitude maximale et une durée réduites des transitoires calciques induits par l’ATP, avec moins de cellules répondant à l’ATP par rapport aux astrocytes témoins. Ensemble, ces altérations suggèrent que le FXS perturbe l’homéostasie du calcium dans les astrocytes humains. Ces résultats sont cohérents avec une étude antérieure23,24 faisant état d’une altération de l’activité du récepteur IP3 dans les fibroblastes FXS. De plus, nous avons observé une augmentation de la glycolyse, de la capacité glycolytique et de la réserve glycolytique dans les astrocytes FXS, ainsi qu’une réduction des taux de consommation d’oxygène mitochondrial.
La présente étude décrit la génération et la caractérisation d’un nouveau modèle in vitro pour les astrocytes FXS en exploitant la puissance des iPSC dérivées de patients. Notamment, ce n’est que grâce à une étude antérieure utilisant des astrocytes humains co-cultivés avec des neurones humains que nous avons découvert le rôle critique des astrocytes déterminant le phénotype électrophysiologique des neurones14. Cependant, ces études se sont concentrées presque entièrement sur les changements induits par la maladie dans l’activité neuronale, et rien n’a été exploré dans les astrocytes. Ici, ce protocole offre un nouveau cadre pour nous concentrer sur les altérations fonctionnelles dans les astrocytes. Ceci, à son tour, mettra en lumière des interactions astrocytes-neurones jusqu’alors inexplorées, telles que la signalisation du glutamate et les réponses électriques. Nos résultats à ce stade soulignent la nécessité de donner la priorité aux astrocytes dans les recherches futures.
Il est essentiel de reconnaître les limites de ce protocole. Tout d’abord, il s’agit d’un hachage mécanique, qui conduit également à la fragmentation de l’ADN. Cela peut provoquer l’agglutination des gliosphères hachées, conduisant finalement à la mort cellulaire. Deuxièmement, la densité d’ensemencement des APC pendant l’étape de différenciation terminale vers les astrocytes est essentielle pour prévenir l’inhibition cellule-cellule médiée par contact. Il est recommandé d’optimiser cela sur toutes les lignées cellulaires pour éviter le détachement de la surface du récipient de culture.
Ici, nos résultats sont la première analyse de ce type dans les astrocytes humains, car toutes les études précédentes étaient basées sur d’autres types de cellules telles que les lignées cellulaires lymphoblastoïdes et les fibroblastes humains25. Le métabolisme énergétique dysfonctionnel est un acteur connu dans l’étiologie des troubles du spectre autistique, comme le montrent l’augmentation des niveaux de lactate plasmatique humain, le taux de consommation d’oxygène significativement plus faible dans les granulocytes26 et la réduction de l’expression des gènes de phosphorylation oxydative mitochondriale dans le cortex cingulaire antérieur, le cortex moteur, le thalamus et le cervelet des enfants atteints de TSA27. Des changements similaires dans le métabolisme énergétique sont observés dans les troubles neurodégénératifs comme la maladie de Parkinson28 et la maladie d’Alzheimer29, ce qui suggère que l’augmentation de la glycolyse peut compenser la réduction de la fonction fonctionnelle30. Des études futures sont nécessaires pour explorer si des mécanismes similaires sont en jeu dans le métabolisme énergétique dysfonctionnel observé dans les astrocytes dérivés de patients FXS.
À l’heure actuelle, la génération d’astrocytes spécifiques au cerveau antérieur est une réalisation importante, mais les progrès futurs pourraient permettre la création de cellules gliales spécifiques à une région, telles que les astrocytes corticaux/hippocampiques. Cela permettrait de mieux comprendre les rôles distincts joués par ces astrocytes dans les fonctions cérébrales régionales, la plasticité synaptique et les interactions neuronales-gliales. La capacité de générer de tels astrocytes spécifiques à une région pourrait faire progresser les modèles de neurodéveloppement et de maladies neurodégénératives. À terme, cela pourrait conduire à des stratégies thérapeutiques plus précises ciblant le dysfonctionnement des astrocytes dans des régions cérébrales spécifiques.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions le professeur Sumantra Chattarji pour les fonds intra-muros. Nous remercions le professeur Gaiti Hasan pour l’accès à l’installation d’imagerie calcique, le Centre central d’imagerie et d’écoulement du Centre national des sciences biologiques, Padmanabh Singh et Prangya Hota pour la relecture et les suggestions, et l’équipe de Labmate Asia pour leur aide dans la réalisation des tests Seahorse XF.
matériels
| Name | Company | Catalog Number | Comments |
| 1-Thioglycerol | Sigma-Aldrich | M6145 | |
| Accutase solution | Sigma-Aldrich | A6964 | Enzyme cell detachment medium |
| Adenosine 5′-triphosphate magnesium salt | Sigma-Aldrich | A9187 | |
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634010 | |
| Antibiotic-Antimycotic (100x) | ThermoFisher Scientific | 15240062 | |
| B-27 Supplement (50x), serum-free | ThermoFisher Scientific | 17504044 | |
| Bovine Serum Albumin | Sigma-Aldrich | A9418 | |
| Chemically Defined Lipid Concentrate | ThermoFisher Scientific | 11905031 | |
| Collagenase, Type IV, powder | ThermoFisher Scientific | 17104019 | |
| Deoxyribonuclease I | Worthington Biochemical Corporation | LK003170 | |
| Dimethyl sulfoxide | Sigma-Aldrich | D2650 | |
| Dispase II, powder | ThermoFisher Scientific | 17105041 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| Essential 8 Medium | ThermoFisher Scientific | A1517001 | |
| FXS1, FXS2 | Coriell Institute of Medical Research | GM07072, GM05848 | FXS patient cells |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | glutamine substitute |
| Ham's F-12 Nutrient Mix | ThermoFisher Scientific | 11765054 | |
| Healthy control cells | Cedars-Sinai Medical Center | ND30625 | healthy control cells |
| Heparin sodium salt from porcine intestinal mucosa | Sigma-Aldrich | H3149 | |
| IMDM | ThermoFisher Scientific | 12440053 | |
| Insulin, human | Roche | 11376497001 | |
| LDN 193189 | Stratech Scientific | S2618-SEL | |
| Leukemia Inhibitory Factor human | Sigma-Aldrich | L5283 | |
| Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | Corning | 354230 | |
| MEM Non-Essential Amino Acids Solution (100x) | ThermoFisher Scientific | 11140050 | |
| Mouse FGF-basic (FGF-2/bFGF) Recombinant Protein | Peprotech | 450-33 | |
| Mr. Frosty freezing container | ThermoFisher Scientific | 5100-0001 | cryobox |
| N-2 Supplement (100x) | ThermoFisher Scientific | 17502048 | |
| N-Acetyl-L-cysteine | Sigma-Aldrich | A9165 | |
| Neurobasal Medium | ThermoFisher Scientific | 21103049 | |
| Nunc Biobanking and Cell Culture Cryogenic Tubes | ThermoFisher Scientific | 377267 | |
| Nunc Cell-Culture Treated 6 well dish | ThermoFisher Scientific | 140675 | |
| Papain Dissociation System | Worthington Biochemical Corporation | LK003150 | |
| Penicillin-Streptomycin | ThermoFisher Scientific | 15140122 | |
| Recombinant Human CNTF Protein, CF | R&D Systems | 257-NT-010 | |
| Recombinant Human EGF Protein, CF | R&D Systems | 236-EG-01M | |
| RevitaCell Supplement (100x) | ThermoFisher Scientific | A2644501 | |
| SB431542 | Tocris | 1614 | |
| Seahorse XFe24 Analyzer | Agilent Technologies | ||
| Seahorse XF Cell Mito Stress Test Kit | Agilent Technologies | 103015-100 | |
| Seahorse XF Glycolysis Stress Test Kit | Agilent Technologies | 103020-100 | |
| Tissue Culture Dishes-100 cm | Biostar Lifetech | TCD000100 | |
| Transferrin | Roche | 10652202001 | |
| VWR Razor Blades | VWR International | 55411-050 | |
| Antibodies | |||
| Primary antibody | Company | Catalog number | Dilution |
| Oct4 (C-10) | Santa Cruz Biotechnology | sc-5279 | Dilution: 1:250 Secondary antibody: Goat anti-Mouse IgG, Alexa Fluor 568 |
| Nanog | R & D Systems | AF1997 | Dilution: 1:100 Secondary antibody: Donkey anti-Goat IgG, Alexa Fluor 488 |
| Vimentin | Abcam | Ab5733 | Dilution: 1:500 Secondary antibody: Goat anti-Chicken IgY, Alexa Fluor 488 |
| NFIA | Abcam | Ab41851 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| GFAP-cy3 | Sigma | C9205 | Dilution: 1:500 Secondary antibody: NA |
| GFAP | DAKO | Z0334 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 568 |
| S100β | DAKO | IR504 | Dilution: 1:500 Secondary antibody: Goat anti-Rabbit IgG, Alexa Fluor 488 |
| Anti-Nuclei Antibody, clone 235-1 | Merck Millipore | MAB1281 | Dilution: 1:1000 Secondary antibody: Goat anti-Mouse IgG1, Alexa Fluor 555 |
| Secondary antibodies | Dilution | ||
| Goat anti-Mouse IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11004 | 1:1000 |
| Donkey anti-Goat IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11055 | 1:1000 |
| Goat anti-Chicken IgY, Alexa Fluor 488 | Thermo Fisher Scientific | A11039 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 568 | Thermo Fisher Scientific | A11011 | 1:1000 |
| Goat anti-Rabbit IgG, Alexa Fluor 488 | Thermo Fisher Scientific | A11034 | 1:1000 |
| Goat anti-Mouse IgG1, Alexa Fluor 555 | Thermo Fisher Scientific | A21127 | 1:1000 |
Références
- Santoro, M. R., Bray, S. M., Warren, S. T. Molecular of fragile X syndrome: A twenty-year perspective. Annu Rev Pathol. 7, 219-245 (2012).
- Wang, H., et al. Developmentally-programmed FMRP expression in oligodendrocytes: A potential role of FMRP in regulating translation in oligodendroglia progenitors. Hum Mol Genet. 13 (1), 79-89 (2004).
- Pacey, L. K. K., Doering, L. C. Developmental expression of FMRP in the astrocyte lineage: Implications for fragile X syndrome. Glia. 55 (15), 1601-1609 (2007).
- Zhang, Y., et al. Purification and characterization of progenitor and mature human astrocytes reveals transcriptional and functional differences with mouse. Neuron. 89 (1), 37-53 (2016).
- Allen, N. J., Barres, B. A. Neuroscience: Glia - more than just brain glue. Nature. 457 (7230), 675-677 (2009).
- Khakh, B. S., McCarthy, K. D. Astrocyte calcium signaling: From observations to functions and the challenges therein. Cold Spring Harb Perspect Biol. 7 (4), a020404(2015).
- Allen, N. J., et al. Astrocyte glypicans 4 and 6 promote formation of excitatory synapses via GluA1 AMPA receptors. Nature. 486 (7403), 410-414 (2012).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Rothstein, J. D., et al. Knockout of glutamate transporters reveals a major role for astroglial transport in excitotoxicity and clearance of glutamate. Neuron. 16 (3), 675-686 (1996).
- Almad, A., Maragakis, N. J. A stocked toolbox for understanding the role of astrocytes in disease. Nat Rev Neurol. 14 (6), 351-362 (2018).
- Zhang, Y., Barres, B. A. Astrocyte heterogeneity: An underappreciated topic in neurobiology. Curr Opin Neurobiol. 20 (5), 588-594 (2010).
- Clarke, L. E., Barres, B. A. Emerging roles of astrocytes in neural circuit development. Nat Rev Neurosci. 14 (5), 311-321 (2013).
- Bradley, R. A., et al. Regionally specified human pluripotent stem cell-derived astrocytes exhibit different molecular signatures and functional properties. Development. 146 (13), dev170910(2019).
- Das Sharma, S., et al. Astrocytes mediate cell non-autonomous correction of aberrant firing in human FXS neurons. Cell Rep. 42 (4), 112344(2023).
- Chambers, S. M., et al. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Hu, X., et al. The JAK/STAT signaling pathway: from bench to clinic. Sig Transduct Target Ther. 6, 402(2021).
- Baser, A., et al. Onset of differentiation is post-transcriptionally controlled in adult neural stem cells. Nature. 566 (7742), 100-104 (2019).
- Pal, R., Bhattacharya, A. Modelling protein synthesis as a biomarker in fragile x syndrome patient-derived cells. Brain Sci. 9 (3), 1-12 (2019).
- Gross, C., Bassell, G. J. Excess protein synthesis in FXS patient lymphoblastoid cells can be rescued with a p110β-selective inhibitor. J Mol Med. 18 (3), 336-345 (2012).
- Kumari, D., et al. Identification of fragile X syndrome specific molecular markers in human fibroblasts: A useful model to test the efficacy of therapeutic drugs. Hum Mutat. 35 (12), 1485-1494 (2014).
- Jacquemont, S., et al. Protein synthesis levels are increased in a subset of individuals with fragile X syndrome. Hum Mol Genet. 27 (12), 2039-2051 (2018).
- Bowser, D. N., Khakh, B. S. ATP excites interneurons and astrocytes to increase synaptic inhibition in neuronal networks. J Neurosci. 24 (39), 8606-8620 (2004).
- Schmunk, G., Boubion, B. J., Smith, I. F., Parker, I., Gargus, J. J. Shared functional defect in IP3R-mediated calcium signaling in diverse monogenic autism syndromes. Transl Psychiatry. 5 (9), e643-e710 (2015).
- Peteri, U. K., et al. Generation of the human pluripotent stem-cell-derived astrocyte model with forebrain identity. Brain Sci. 11 (2), 209(2021).
- Alvarez-Mora, M. I., et al. Impaired mitochondrial function and dynamics in the pathogenesis of FXTAS. Mol Neurobiol. 54 (9), 6896-6902 (2017).
- Giulivi, C., et al. Mitochondrial dysfunction in autism. JAMA. 304 (21), 2389-2396 (2010).
- Anitha, A., et al. Brain region-specific altered expression and association of mitochondria-related genes in autism. Mol Autism. 3 (1), 12(2012).
- Teves, J. M. Y., et al. Parkinson's disease skin fibroblasts display signature alterations in growth, redox homeostasis, mitochondrial function, and autophagy. Front Neurosci. 11, 737(2018).
- Yao, J., et al. Mitochondrial bioenergetic deficit precedes Alzheimer's pathology in female mouse model of Alzheimer's disease. Proc Natl Acad Sci USA. 106 (34), 14670-14675 (2009).
- Sonntag, K. C., et al. Late-onset Alzheimer's disease is associated with inherent changes in bioenergetics profiles. Sci Rep. 7 (1), 14038(2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon