Method Article
Nocaute de gene mediado por CRISPR-Cas9 baseado em eletroporação em células THP-1 e isolamento de clone de célula única
Neste Artigo
Resumo
A linhagem celular THP-1 é amplamente utilizada como modelo para investigar as funções de monócitos/macrófagos humanos em várias áreas de pesquisa relacionadas à biologia. Este artigo descreve um protocolo para engenharia eficiente baseada em CRISPR-Cas9 e isolamento de clone de célula única, permitindo a produção de dados fenotípicos robustos e reprodutíveis.
Resumo
A linhagem celular THP-1 de leucemia monocítica aguda humana (LMA) é amplamente utilizada como modelo para estudar as funções de macrófagos derivados de monócitos humanos, incluindo sua interação com patógenos humanos significativos, como o vírus da imunodeficiência humana (HIV). Em comparação com outras linhagens celulares imortalizadas de origem mieloide, as células THP-1 retêm muitas vias de sinalização inflamatória intactas e exibem características fenotípicas que se assemelham mais às dos monócitos primários, incluindo a capacidade de se diferenciar em macrófagos quando tratadas com forbol-12-miristato 13-acetato (PMA). O uso da tecnologia CRISPR-Cas9 para projetar células THP-1 por meio de nocaute genético direcionado (KO) fornece uma abordagem poderosa para caracterizar melhor os mecanismos relacionados ao sistema imunológico, incluindo interações vírus-hospedeiro. Este artigo descreve um protocolo para engenharia eficiente baseada em CRISPR-Cas9 usando eletroporação para fornecer ribonucleoproteínas Cas9:sgRNA pré-montadas no núcleo da célula. O uso de vários sgRNAs direcionados ao mesmo locus em posições ligeiramente diferentes resulta na deleção de grandes fragmentos de DNA, aumentando assim a eficiência da edição, conforme avaliado pelo ensaio de endonuclease I T7. A edição mediada por CRISPR-Cas9 no nível genético foi validada por sequenciamento de Sanger seguido de análise de inferência de edições CRISPR (ICE). A depleção de proteínas foi confirmada por immunoblotting acoplado a um ensaio funcional. Usando este protocolo, foram alcançados até 100% de indels no locus alvo e uma diminuição de mais de 95% na expressão da proteína. A alta eficiência de edição torna conveniente isolar clones de célula única, limitando a diluição.
Introdução
O THP-1 é uma linhagem celular derivada de monócitos humanos isolada de um paciente que sofre de leucemia aguda (LMA), que apresenta características fenotípicas muito semelhantes às dos monócitos primários1. Em comparação com os macrófagos primários derivados de monócitos, que não se dividem e exibem vida útil limitada e variabilidade inter/intradoador no fenótipo, as células THP-1 podem ser cultivadas virtualmente para sempre e ter um comportamento mais homogêneo que favorece a reprodutibilidade dos resultados 2,3,4,5,6 . Notavelmente, as células THP-1 podem ser diferenciadas em direção a um fenótipo semelhante a macrófagos com forbol-12-miristato 13-acetato (PMA), tornando-as um modelo in vitro amplamente utilizado para investigar as respostas de monócitos / macrófagos a sinais inflamatórios 7,8,9,10,11,12,13 ou infecção por patógenos humanos clinicamente relevantes, incluindo HIV 14,15,16. A possibilidade de projetar geneticamente células THP-1 é de interesse em muitas áreas de pesquisa relacionadas à biologia.
A proteína 9 associada a CRISPR (CRISPR-Cas9) é um sistema imunológico adaptativo procariótico que retransmite nuclease guiada por RNA para degradar genomas virais invasores, que foi reprogramado como uma ferramenta de engenharia genética17. O processo de edição do genoma prossegue em três etapas: reconhecimento, clivagem e reparo. Um RNA de guia único (sgRNA) recruta a nuclease Cas9 para um locus genômico específico por meio do emparelhamento de bases com sua sequência guia de 20 pb. A presença de uma sequência de Motivo Adjacente ao Protoespaçador (PAM) diretamente 3 'da sequência alvo genômica de 20 pb desencadeia o desenrolamento e a clivagem mediados por Cas9 em ambas as fitas de DNA entre as posições 17 e 18 (3 pb 5' do PAM). A quebra de fita dupla (DSB) resultante é processada por duas vias principais de reparo. Na ausência de um modelo de reparo com homologia com o locus danificado, a via de junção final não homóloga (NHEJ) propensa a erros introduzirá inserções e/ou deleções aleatórias de nucleotídeos (indels), potencialmente levando a mutações frameshift e/ou a introdução de códons de terminação prematura (PTC). Por sua vez, os mRNAs contendo PTC são alvo de degradação pela via de decaimento de mRNA mediada por nonsense (NMD), interrompendo a expressão / função da proteína18 , 19 , 20 . Alternativamente, a via de reparo dirigido por homologia (HDR) dependente do modelo pode operar e reparar fielmente o DSB. Esse mecanismo foi aproveitado para obter uma edição precisa de genes, incluindo knock-ins e substituições de bases. Vale ressaltar que o status do ciclo celular é um fator importante que influencia a escolha da via de reparo do DSB. De fato, o NHEJ é ativo em todos os estágios do ciclo celular, enquanto o HDR é restrito principalmente às fases S / G221.
As células THP-1 crescem em suspensão e são notoriamente difíceis de transfectar com DNA plasmidial, procedimento que possivelmente também altera sua viabilidade e/ou capacidade de diferenciação22,23. A transdução com vetores lentivirais baseados em HIV-1 que codificam Cas9 e o sgRNA é frequentemente empregada para nocautear (KO) um gene de interesse24. A integração do Cas9 / sgRNA no genoma celular garante expressão prolongada e KO eficiente, mas também é uma fonte persistente de efeitos fora do alvo25. Alternativamente, as ribonucleoproteínas Cas9:sgRNA (RNPs) pré-montadas são entregues por eletroporação, um método que envolve a formação temporária de poros nas membranas plasmática e nuclear após a aplicação de impulsos elétricos. Preservar a viabilidade celular é um desafio importante ao empreender essa abordagem.
Aqui, uma linhagem celular THP-1 expressando GFP (THP-1_GFP) de forma estável foi produzida para servir como uma ferramenta para estabelecer um protocolo para obter uma edição eficiente baseada em CRISPR-Cas9. Depois de projetar uma estratégia para inativar o gene EGFP usando três sgRNAs simultaneamente (abordagem multiguia), a eficiência do KO entre várias condições de eletroporação foi determinada usando a expressão de GFP como leitura. A proliferação celular foi monitorada em paralelo. A edição gênica foi confirmada por um ensaio de endonuclease T7 I (T7EI) e sequenciamento de Sanger, seguido de análise com o algoritmo Inference of CRISPR Edits (ICE)26. Parâmetros que produziram até 95% de redução na expressão de GFP, com células THP-1 recuperando taxas de crescimento normais após eletroporação, foram empregados com sucesso para inativar um gene endógeno (SAMHD1) e produzir clones de THP-1 de célula única.
Protocolo
Os detalhes dos reagentes e dos equipamentos utilizados neste estudo estão listados na Tabela de Materiais.
1. Desenho do guia com CRISPOR (Figura 1.1)
NOTA: O software SnapGene Viewer pode ser usado nas etapas 4, 7 e 10 para anotar o local de destino de edição e a localização da hibridização do primer de PCR dentro do gene de interesse.
- Acesse o site da Ensembl (www.ensembl.org). Na caixa Pesquisar , selecione uma espécie e digite o nome do gene de interesse. Clique em Ir. Selecione o resultado que corresponde ao gene (não um transcrito).
- Clique em Mostrar tabela de transcrição e selecione o ID de transcrição que corresponde ao código de proteína rotulado em ouro na coluna Biotipo . Uma vez na página de transcrição, clique em Exons no menu à esquerda.
- Role para baixo e clique em Baixar sequência. Certifique-se de que o formato do arquivo seja FASTA. Em Configurações - Sequências incluídas, desmarque tudo, exceto Sequência genômica.
Digite o número "500" em Sequência de flanqueamento em cada extremidade da caixa de transcrição . Clique em Visualizar, selecione toda a sequência (apenas os nucleotídeos sem o cabeçalho) e copie-a (Ctrl+C). - Abra o SnapGene Viewer e clique em Novo > arquivo de DNA. Cole a sequência (Ctrl+V) na caixa Criar uma sequência . Desmarque Detectar recursos comuns e selecione Linear na topologia.
Renomeie o arquivo e clique em Criar. No menu à esquerda, desmarque Mostrar enzimas (primeiro ícone). Na parte inferior da janela, selecione a guia Sequência .
NOTA: Esta etapa permite a recuperação da sequência completa do gene, incluindo éxons, íntrons e sequências flanqueadoras de 500 pb (opcional). Esta última informação é útil para projetar primers de PCR para a amplificação de um local alvo localizado dentro do primeiro éxon. - De volta ao site do Ensembl, role para cima na visualização do arquivo e clique em Voltar. Agora, altere o formato do arquivo para RTF. Em Configurações - Sequências incluídas, desmarque tudo, exceto Exons. Em Mostrar variantes, selecione Não. Clique em Download na parte superior da página.
- Abra o arquivo baixado (com o Word), que contém a sequência de éxons e mostra a sequência de codificação em azul, começando com o ATG inicial. Escolha o éxon a ser direcionado pela edição direcionada ao CRISPR-Cas9 (veja abaixo as recomendações), selecione-o e copie-o (Ctrl+C).
- O éxon alvo é um éxon inicial ou um éxon que codifica um domínio funcionalmente importante da proteína. Vale a pena notar que o estabelecimento de um PTC em um éxon tardio próximo ao 3 'UTR provavelmente não conseguirá eliciar NMD, levando à expressão de uma proteína truncada C-terminalmente. Por outro lado, a introdução de um PTC em um éxon inicial proximal ao local de iniciação nativo está associada a um risco de tradução ilegítima (ITL, também conhecida como iniciação de tradução alternativa (ATI)), produzindo a expressão inesperada de uma proteína truncada N-terminal que começa em um local de iniciação de tradução (TIS) a jusante do primeiro códon ATG. Para mitigar este último risco, recomenda-se avaliar a ocorrência de TIS alternativo usando ATGpr27 (atgpr.dbcls.jp) e/ou NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/).
- Certifique-se de que o éxon de destino contenha uma sequência de codificação. No entanto, pode ser útil escolher um recozimento de sgRNA com a região 5'UTR a montante do ATG inicial para incluí-lo no fragmento excluído.
- Idealmente, o éxon alvo deve ser comum a todas as variantes de transcrição codificadoras de proteínas do gene. Verifique se esse é o caso no Genome Data Viewer (www.ncbi.nlm.nih.gov/gdv/) do NCBI pesquisando o gene de interesse.
Clicar no nome do gene (em verde) na janela de exibição mostrará as variantes de transcrição (em roxo). Os éxons são representados por um retângulo.
- No SnapGene Viewer, pressione Ctrl+F, Ctrl+V e Enter para pesquisar a sequência do éxon. Pressione Ctrl+T para adicionar um novo recurso, nomeie-o, altere o tipo para "exon" e clique em OK.
- Acesse o site do CRISPOR (http://crispor.gi.ucsc.edu/) e cole a sequência de exons na Etapa 1. Primeiro, selecione um genoma de referência na Etapa 2 e, em seguida, o tipo de PAM a ser direcionado na Etapa 3, normalmente 20bp-NGG para SpCas9. Clique em ENVIAR.
- Selecione dois sgRNAs para que fiquem separados por até 150 pb e, em seguida, selecione um terceiro sgRNA entre eles. Aqui estão algumas diretrizes para a seleção de sgRNA:
- O escore de especificidade do MIT está relacionado a efeitos fora do alvo. Uma pontuação mais alta indica menos potenciais fora dos alvos. A coluna da direita exibe três locais fora do alvo previstos classificados do mais para o menos provável, juntamente com suas localizações (em um éxon, um íntron ou uma região intergênica). A lista completa de sites fora do alvo previstos pode ser acessada clicando em mostrar tudo. Se possível, selecione sgRNAs com uma pontuação MIT >80, priorizando aqueles sem alvos para 0, 1 ou 2 incompatibilidades. Além disso, sgRNAs com alvos fora de um éxon, que têm o maior potencial de afetar o fenótipo, devem ser evitados.
- Para eficácia prevista, consulte a pontuação Doench '16. Observe que uma pontuação alta de Doench '16 simplesmente indica que o sgRNA tem maior probabilidade de ser eficaz. A eficácia real deve ser determinada experimentalmente. Por esse motivo, é sempre útil selecionar vários sgRNAs, mesmo que não se destinem a ser usados juntos.
- Um conteúdo de GC muito alto ou muito baixo, bem como certos motivos, podem ser prejudiciais à eficiência do sgRNA e devem ser evitados. Esses parâmetros são destacados pelo CRISPOR.
- Repita a etapa 1.7 para adicionar a sequência de sgRNA e a sequência PAM associada à sequência do gene no SnapGene Viewer. Ao mesmo tempo, cole a sequência de sgRNA (sem o PAM) em um arquivo de texto ou Excel para manter a orientação 5'-3' necessária ao solicitar o oligonucleotídeo.
- Para amplificação baseada em PCR do local alvo, projete alguns primers ao redor dele. O tamanho do amplicon deve estar entre 800 e 1000 bp. O PrimerQuest foi usado para projetar os primers para este protocolo (https://eu.idtdna.com/pages/tools/primerquest). Como alternativa, o CRISPOR fornece uma lista de primers para amplificar a região genômica alvo, bem como os locais fora do alvo. Depois de exibir a lista completa de possíveis locais fora do alvo (etapa 1.9.1), clique em Primers fora do alvo no canto inferior direito.
2. Reagente e preparação de células para eletroporação (Figura 1.2)
- Prepare uma placa de cultura de 24 poços para recuperar as células após a eletroporação, enchendo os poços com 500 μL de meio RPMI 1640 suplementado com 20% de soro fetal bovino (FBS) inativado por calor (56 ° C, 30 min) filtrado (0,20 μm). Não adicione antibióticos. Mantenha a placa em uma incubadora umidificada a 37 ° C e 5% de CO2 por 24 h até a eletroporação.
- Se os sgRNAs forem enviados secos, reidrateie-os com tampão TE (10 mM Tris, 1 mM EDTA, pH 8,0) até uma concentração final de 100 μM (ou seja, 10 μL de tampão TE por 1 nmol de sgRNA). Vortex por 30 s, incubar a 4 ° C durante a noite para permitir a reidratação completa e, após uma breve homogeneização da pipeta, armazenar a solução estoque de sgRNA a -20 ° C. Dependendo do volume final, faça alíquotas para evitar vários ciclos de congelamento e descongelamento.
- Preparar uma solução de sgRNA funcional a uma concentração final de 30 μM, diluindo a solução-mãe de 100 μM em água isenta de nuclease. Vortex por 30 s e incubar 5 min em temperatura ambiente.
- Monte os RNPs Cas9:sgRNA em uma proporção molar de 1:9 diluindo simultaneamente 1 μL de cada um dos três sgRNAs de 30 μM e 0,5 μL de soluções Cas9 de 20 μM em 3,5 μL de tampão de ressuspensão R, incluído no kit de eletroporação (volume final de 7 μL, para uma condição experimental; dimensione de acordo). Vortex brevemente e incubar por 5 min em temperatura ambiente.
- Enquanto isso, prepare um controle não editado adicionando 0,5 μL de 20 μM Cas9 a 6,5 μL de tampão de ressuspensão R. Vórtice brevemente e incube por 5 min em temperatura ambiente.
- Adicione 5 μL de tampão de ressuspensão R a todas as amostras para um volume final de 12 μL por condição de eletroporação.
- Prepare as células THP-1 para eletroporação.
- Avaliar a concentração e a viabilidade pelo teste de exclusão do azul de tripano. Dilua as células 1:2 em uma solução de coloração azul de tripano a 0,4%. Após 30 s de incubação, homogeneizar bem e adicionar 10 μL em uma parede de uma câmara de contagem com um estilo de grade aprimorado por Neubauer. Conte três quadrados grandes e divida a contagem por 100 para obter a concentração celular (x106 células/mL).
NOTA: A saúde das células influencia sua sensibilidade à eletroporação. Deve-se ter cuidado para realizar a cultura de células em condições ideais. O tempo fora da incubadora deve ser limitado e todos os reagentes e soluções devem ser preparados e aquecidos com antecedência. - Para cada condição, colete um volume equivalente a 0,2 x 106 células e centrifugue (336 x g, 5 min, 20 ° C).
- Aspirar o sobrenadante com uma pipeta e ressuspender o sedimento em 500 μL de PBS. Centrifugue novamente (336 x g, 5 min, 20 °C).
- Aspire o sobrenadante cuidadosamente usando uma pipeta e ressuspenda o pellet de células THP-1 com 12 μL de solução RNP (etapa 2.6).
- Avaliar a concentração e a viabilidade pelo teste de exclusão do azul de tripano. Dilua as células 1:2 em uma solução de coloração azul de tripano a 0,4%. Após 30 s de incubação, homogeneizar bem e adicionar 10 μL em uma parede de uma câmara de contagem com um estilo de grade aprimorado por Neubauer. Conte três quadrados grandes e divida a contagem por 100 para obter a concentração celular (x106 células/mL).
3. Configuração do sistema de eletroporação e nucleofecção (Figura 1.3)
- Coloque a estação de pipeta sob um gabinete de biossegurança, coloque um tubo de eletroporação no suporte e adicione 3 mL de tampão E, incluído no kit de eletroporação.
- Depois de ligar o dispositivo de eletroporação, use a tela sensível ao toque para definir os seguintes parâmetros de eletroporação: Voltage = 1 500 V, Duração = 10 ms, Número = 3.
- Equipar a pipeta de eletroporação com uma ponteira e aspirar 10 μL da solução RNP/THP-1 (passo 2.7.4). Insira a pipeta no tubo de eletroporação e pressione Iniciar na tela do dispositivo de eletroporação. Aguarde até que a mensagem Concluído apareça e remova a pipeta do tubo.
- Transfira as células para um poço da placa pré-aquecida de 24 poços e homogeneize suavemente. Coloque a placa de volta na incubadora umidificada e deixe-a descansar sem ser perturbada por 72 h.
NOTA: Tome cuidado para não fazer bolhas ao pipetar a suspensão RNP/THP-1, pois elas interferirão no procedimento de eletroporação. Se uma mensagem de erro aparecer na ausência de um arco elétrico visível, remova a pipeta de eletroporação do tubo e pressione Iniciar novamente. No entanto, se um arco elétrico na forma de uma breve faísca brilhante for observado, isso pode indicar a presença de bolhas. O procedimento de eletroporação provavelmente falhará, mesmo na ausência de uma mensagem de erro.
4. Recuperação de THP-1 72 h após a eletroporação (Figura 1.4)
- Contar as células para avaliar a concentração (passo 2.7.1) 72 h após a electroporação.
- Se houver células suficientes (ou seja, ≥0,6 x 106 células/mL), dilua-as pelo menos um fator 2 com RPMI suplementado com 20% de FBS e 1% de Penicilina-Estreptomicina e traga a concentração entre 0,3-0,5 x 106 células por mL. Caso contrário, permita que a célula se recupere por mais 72 h.
- Passe e amplifique as células até que haja o suficiente para a validação do KO. Enquanto isso, o isolamento de clones de célula única pode ser iniciado (etapa 7).
5. Validação da edição gênica pelo ensaio de incompatibilidade T7EI (Figura 1.5)
NOTA: O ensaio pode subestimar a eficiência de edição, uma vez que o T7EI reconhece incompatibilidades maiores que 1 pb. Assim, o ensaio T7EI não é útil para rastrear populações de células homozigóticas (ou seja, clones de células únicas), a menos que seja modificado adequadamente (etapa 5.7).
- Avalie a concentração celular (etapa 2.7.1) e retire um volume equivalente a 0,1 x 106 células em um tubo de 1,5 mL. Centrifugar (336 x g, 5 min, 20 °C), aspirar o sobrenadante e ressuspender o pellet em 500 μL de PBS. Centrifugar novamente e, com uma pipeta, aspirar o máximo de sobrenadante possível sem perturbar o sedimento. Congelar rapidamente a amostra e conservá-la a -20 °C.
- Extraia o DNA genômico para servir como matriz para amplificação por PCR.
- Ressuspenda o pellet com 50 μL de solução de extração de DNA, homogeneize e transfira todo o volume em um tubo de PCR de 0,2 mL. Vórtice e centrífuga brevemente (pulsar por 3 s).
- Coloque o tubo em um termociclador e aqueça-o a 65 °C por 15 min, seguido de 98 °C por 10 min.
- Diluir o ADN extraído com 90 μL de água ultrapura. Vórtice e centrífuga brevemente (5.000 x g, 3 s).
- Dilua o primer de PCR (consulte a Tabela de Materiais) em água ultrapura para uma concentração final de 10 μM (ou seja, 10 pmol/μL).
- Prepare a mistura de PCR (seguindo a NOTA abaixo) em um tubo de PCR de 0,2 mL (volume final = 50 μL, para uma condição). Normalmente, haverá pelo menos duas condições: o KO e o controle não editado apenas com Cas9.
NOTA: DNA genômico purificado: 10 μL; Primer direto e reverso (10 μM): 2,5 μL cada, a concentração final de 500 nM; Tampão de reação (5x): 10 μL. Vórtice bem antes de adicionar. Misture dNTP (25 mM de cada): 0,6 μL, concentração final de 0,3 mM para cada dNTP. DNA polimerase: 0,5 μL; Água ultrapura: 23,9 μL. Vórtice e centrífuga brevemente (pulso por 3 s). - Coloque os tubos no termociclador e execute o programa de PCR com as seguintes configurações:
NOTA: Um ciclo a 95 °C durante 5 min, seguido de 30 ciclos [98 °C durante 20 s (fase de desnaturação), X °C durante 15 s (etapa de recozimento), 72 °C durante 45 s (etapa de alongamento)] e, em seguida, um ciclo final a 72 °C durante 2 min. No final da amplificação, remova os tubos, o vórtice e a centrífuga brevemente (pulso por 3 s). A temperatura de recozimento (X) é a temperatura de fusão (Tm) dos primers menos 5 °C. - Para uma população policlonal editada: em um novo tubo de 0,2 mL, adicione 17,5 μL do amplicon de PCR e 2 μL de NEBuffer 2 (10x) para um volume final de 19,5 μL. Vórtice e centrifugue brevemente (pulso por 3 s). Alternativamente, para rastrear clones de célula única, misture 1:1 dos produtos de PCR das células de controle editadas e não editadas (etapa 5.6) (Figura Suplementar 1).
- Para a formação heteroduplex, coloque os tubos no termociclador e execute o seguinte programa: um ciclo a 95 °C por 10 min, um com rampa de -2 °C/s de 95 a 85 °C, um com rampa de -0,3 °C/s de 85 a 25 °C e um ciclo final de resfriamento a 10 °C em HOLD.
- Após a formação do heteroduplex, adicione 0,5 μL de solução de T7EI no tubo. Incubar a 37 °C durante 30 min.
- Prepare um gel de agarose a 1,2%:
- Pese a agarose em um frasco de vidro e adicione o volume apropriado de 1x tampão TAE (40 mM Tris-acetato, 1 mM EDTA, pH 8,3). Coloque-o no micro-ondas prestando atenção para não enroscar a tampa com firmeza. Aqueça várias vezes até que os cristais de agarose estejam completamente dissolvidos.
NOTA: Se necessário, misture a solução. Não deixe ferver, ou o volume diminuirá, alterando a concentração de agarose. - Adicione a mancha de gel de DNA diluída a 1/20.000 e misture bem.
- Despeje a solução de agarose em um molde, adicione um pente e deixe solidificar em temperatura ambiente.
- Pese a agarose em um frasco de vidro e adicione o volume apropriado de 1x tampão TAE (40 mM Tris-acetato, 1 mM EDTA, pH 8,3). Coloque-o no micro-ondas prestando atenção para não enroscar a tampa com firmeza. Aqueça várias vezes até que os cristais de agarose estejam completamente dissolvidos.
- Prepare amostras para eletroforese em gel misturando 5 μL dos produtos de digestão T7EI (etapa 5.9) ou o produto de PCR não digerido com 5 μL de água e 2 μL de corante de carga de DNA 6x. Carregue as amostras e a escada de tamanho e migre a 80 V por 45 min (o tempo pode ser adaptado dependendo do tamanho esperado dos fragmentos de DNA). Terminada a migração, adquira uma imagem do gel com um sistema de imagem apropriado.
6. Validação da edição gênica por análise de sequenciamento de Sanger (Figura 1.6)
- Para caracterizar a modificação genética, purificar os produtos da PCR (etapa 5.4) e, após o sequenciamento de Sanger, analisar os resultados com a ferramenta ICE (https://www.synthego.com/products/bioinformatics/crispr-analysis).
- Controle se uma proteína pode ser produzida apesar das modificações.
- Baixe os resultados do ICE e abra o arquivo contribs.txt.
- Compare as sequências editadas com a contraparte WT. Mapeie os indels e verifique se eles surgiram na região genômica alvo. Avalie seu tamanho. Se o comprimento indel não for um múltiplo de três, ocorrerá um frameshift e um PTC possivelmente será introduzido. Espera-se que o mRNA mutado sofra degradação mediada por NMD.
7. Isolamento de clone de célula única limitando a diluição (Figura 1.7)
NOTA: O isolamento de clones de célula única não é obrigatório. No entanto, se optar por fazê-lo, é importante caracterizar vários clones e comparar seu fenótipo com a população policlonal original.
- Colher uma amostra da suspensão celular, diluir 1/2 com meio de cultura e avaliar a concentração (passo 2.7.1). A diluição aumenta a precisão da contagem.
- Execute 2 a 3 etapas de diluição em série para atingir uma concentração de 7 células / mL. Dispense 100 μL da suspensão celular por poço em uma placa de fundo redondo de 96 poços (ou seja, 0,7 células por poço). Deixe a célula decantar por algumas horas antes de observar as placas no microscópio para identificar poços contendo uma célula.
- Monitore regularmente a formação e o crescimento de colônias e transfira células para uma placa ou frasco de cultura maior quando necessário.
8. Caracterização funcional de células THP-1 KO SAMHD1 por ensaio de restrição de HIV-1
- Semear THP-1 em uma placa de cultura de 24 poços com 0,25 x 106 células por poço em 300 μL de RPMI suplementado com 10% de FBS, 1% de Penicilina-Estreptomicina (RPMI completo) e contendo 300 ng/mL de PMA para diferenciação. Mantenha a placa em uma incubadora umidificada (37 °C, 5% CO2) por 24 h.
- Substitua o meio por um meio RPMI completo sem PMA e coloque a placa de volta na incubadora por mais 24 horas.
- Usando uma bomba de vácuo, aspire todo o meio de cultura. Adicione 250 μL de solução contendo estoque viral HIV-1-GFP pseudotipada com VSVg (MOI = 0,5 IFU / célula). Inclua um controle não infectado. Coloque a placa a 4 °C por 2 h para sincronizar a infecção.
- Lave as células uma vez com RPMI. Adicione 500 μL de RPMI completo a cada poço e incube por 48 h em condições padrão (o tempo pode ser ajustado).
- Depois de remover o meio de cultura, lave as células uma vez com PBS 1x frio. Em seguida, adicione 100 μL de tripsina-EDTA a 0,05% em cada poço. Colocar a placa numa incubadora a 37 °C até que as células estejam completamente separadas (5 min devem ser suficientes) antes de adicionar 200 μL de RPMI completo para inativar a enzima. Transfira 250 μL de cada poço para uma placa de fundo redondo de 96 poços (certifique-se de que o citômetro de fluxo aceite placas de cultura).
- Centrifugar a placa (757 x g, 3 min, desaceleração = 6, 20 °C) e aspirar o sobrenadante com uma pipeta multicanal. Ressuspenda o pellet com 100 μL de PFA a 4% e incube a 4 °C por 10 min. Adicione 100 μL de PBS 1X frio.
- Analise a taxa de infecção, representada por células positivas para GFP, por citometria de fluxo (consulte a Figura 2 suplementar para uma estratégia de gating sugerida).
Resultados
Uma linhagem celular THP-1 foi gerada expressando de forma estável a proteína repórter GFP (THP-1_GFP) (Figura 2A) e usada como ferramenta para estabelecer um protocolo para uma edição eficiente do gene mediada por CRISPR-Cas9. Para este objetivo, 3 sgRNA direcionados ao gene EGFP foram projetados com a ferramenta CRISPOR web29 (Figura 2B), que foram simultaneamente complexados com Cas9 em uma proporção molar de 9: 1 para formar RNPs antes da entrega nas células por eletroporação usando diferentes configurações. Em seguida, as células foram cultivadas em meio RPMI contendo 20% de FBS sozinho (Figura 2C, painel superior) ou diluído 1:1 com meio condicionado (Figura 2C, painel inferior). A proliferação celular e a expressão de GFP, como uma leitura da eficiência do EGFP KO, foram monitoradas ao longo do tempo. Para várias condições, a porcentagem de células positivas para GFP diminuiu drasticamente, atingindo >90% de redução no dia 7 pós-eletroporação (pe). No dia 3 pe, o número de células caiu pela metade, provavelmente devido ao impacto negativo da eletroporação na viabilidade celular. No entanto, o número de células aumentou novamente e o tempo de duplicação voltou à taxa normal no dia 7 pe (Figura 2C, painel superior). O uso de meios condicionados não favoreceu a recuperação celular em nossas condições experimentais (Figura 2C, painel inferior). Com base nesses resultados, três pulsos de 10 ms de 1500 V foram escolhidos para experimentos de acompanhamento.
O EGFP KO mediado por CRISPR-Cas9 foi então caracterizado no nível genômico. Para isso, o DNA genômico das células THP-1_GFP foi extraído, eletroporado com os RNPs Cas9:sgRNA (editado) ou Cas9 sozinho (não editado) e usado como modelo para amplificar por PCR uma região de 891 pb contendo o local alvo. As células parentais THP-1 sem EGFP também foram incluídas como controle negativo. Em seguida, o produto de PCR de células THP-1_GFP editadas foi preparado para detecção de incompatibilidade pelo ensaio T7EI, seguido pela separação dos fragmentos de DNA por eletroforese em gel de agarose. Uma banda de ~ 900 pb correspondente ao tamanho esperado do amplicon WT foi prontamente visualizada para células THP-1_GFP não editadas, mas não para o controle parental THP-1 (Figura 2D, compare as linhas 2 e 3). No caso das células THP-1_GFP editadas, a banda de ~ 900 pb tornou-se indetectável e substituída por uma de migração mais rápida, cujo tamanho correspondia à perda de um fragmento de ~ 75 pb conseqüente à edição de CRISPR (Figura 2D, linha 4). Este padrão de bandas foi alterado após a digestão do T7EI, causando o aparecimento de fragmentos de DNA menores, confirmando ainda mais a edição bem-sucedida do local alvo (Figura 2D, linha 5). Para obter informações sobre a eficiência de edição e os genótipos específicos da população editada, o amplicon de PCR foi sequenciado usando o método de Sanger, seguido de análise com a ferramenta ICE26. A amostra de controle não editada foi processada em paralelo. Recuperamos escores indel e KO de 100%, correspondendo à porcentagem de sequências não-WT na amostra editada e à proporção de indels que provavelmente levam a um KO funcional, respectivamente (Figura 2E, esquerda). A análise detalhada do espectro de tamanho e frequência indel mostrou a presença de duas populações dominantes com uma deleção de 62 pb ou 76 pb e representando 57% ou 37% do total de sequências, respectivamente (Figura 2E, à direita). Esses achados sugerem que o sgRNA # 1, em combinação com o sgRNA # 2 ou # 3, desencadeou DSB simultâneo no gene alvo (Figura 2B).
Este protocolo foi então aplicado para inativar o gene codificador endógeno de SAMHD1 (Figura 3A). A caracterização da linha celular policlonal SAMHD1 KO THP-1 incluiu o monitoramento da proliferação celular ao longo do tempo, que correspondia à das células THP-1 não editadas (Figura 3B). O resultado da edição gênica foi avaliado por amplificação baseada em PCR da sequência alvo, que revelou uma banda menor para as células SAMHD1 KO em comparação com o controle somente Cas9 (Figura 3C, compare as linhas 2 e 4), indicativo de uma perda de sequência de DNA. Essas observações foram confirmadas pelo ensaio T7EI, mostrando o aparecimento de produtos de clivagem para as células SAMHD1 KO, mas não inéditas (Figura 3C, compare as linhas 3 e 5). A análise dos dados de sequenciamento de Sanger com a ferramenta ICE retornou escores indel e KO de 100% e 97%, respectivamente. As principais modificações no locus SAMHD1 foram deleções de sequência de 170 pb, 93 pb ou 104 pb em 46%, 13% e 10% das sequências, respectivamente (Figura 3D). Os níveis de expressão proteica no lisado de células brutas também foram avaliados por immunoblotting. A banda de ~ 70 kDa consistente com o tamanho do SAMHD1 endógeno torna-se virtualmente indetectável após a edição, correspondendo a uma redução estimada de >97% (Figura 3E). Finalmente, a caracterização fenotípica das linhagens celulares policlonais SAMHD1 KO THP-1 também foi avaliada testando a suscetibilidade à infecção pelo HIV-1. De acordo com seu conhecido papel antiviral30,31, a inativação do locus SAMHD1 foi acompanhada por um aumento da taxa de infecção pelo HIV-1 (Figura 3F).
Paralelamente, clones unicelulares limitando a diluição foram gerados. A amplificação do local-alvo genômico dos clones SAMHD1 KO produziu produtos de PCR com maior mobilidade eletroforética em comparação com os de células de controle não editadas ( Figura 4A ). A análise baseada em ICE dos dados de sequenciamento de Sanger retornou uma pontuação indel de 100% para todos os clones de célula única selecionados, exceto um (clone A2) (Figura 4B, à esquerda). A caracterização dos resultados da edição no nível genômico retornou uma única sequência para o clone A3 com uma deleção de 98 pb localizada 28 pb a jusante do ATG inicial, levando a uma mudança fora do quadro (Figura 4B, à direita). Os outros clones continham três (clone B1) ou mais sequências, indicativas da presença de alelos que sofreram diferentes eventos de edição e/ou mais de uma célula por poço na etapa de diluição limite (Figura 4B, à direita). Notavelmente, 11% das sequências dos clones A4 e A7 apresentam indels a montante do ATG inicial, deixando a sequência de codificação inalterada. Além disso, 1% das sequências do clone A7 não tinham um fragmento de 123 pb 9 pb a jusante do ATG inicial, resultando em uma deleção no quadro de 41 aminoácidos. Consistente com esses achados, estudos funcionais revelaram que clones com um locus SAMHD1 interrompido (A3, A8, B1, B3), portanto, sem expressão de SAMHD1, eram altamente permissivos à infecção pelo HIV-1 (Figura 4C). Por outro lado, os clones A4 e A7, onde o SAMHD1 está intacto, eram tão refratários quanto os clones THP-1 de controle parental ou não editado (Figura 4C). Em conclusão, foi confirmada a edição bem-sucedida do locus SAMHD1 no clone A3.

Figura 1: Esboço experimental. (1) O CRISPOR é usado para gerar sequências de sgRNA contra o éxon alvo. Três deles são selecionados de acordo com a alta eficácia no alvo e a fraca previsão fora do alvo. (2) Os três gRNAs são misturados com Cas9 para montar a ribonucleoproteína (RNP), resultando em uma mistura de três RNPs diferentes. (3) As células são eletroporadas para permitir a entrada dos RNPs. (4) As células são transferidas para uma placa de cultura de 24 poços e deixadas em repouso por pelo menos 72 h. (5) A edição de genes é testada qualitativamente pela primeira vez por um ensaio de digestão T7EI, e (6) para as condições validadas, ainda caracterizadas por sequenciamento de Sanger e análise ICE. (7) Se necessário, as células podem ser semeadas após limitar a diluição para produzir populações clonais. Clique aqui para ver uma versão maior desta figura.

Figura 2: Configuração das configurações de eletroporação para EGFP KO em células THP-1 e validação de edição de genes. (A) As células THP-1 foram transduzidas com vetores lentivirais que codificam o gene repórter EGFP. Os clones unicelulares foram obtidos por meio de diluição limitante seguida de expansão em cultura por 30 dias. A análise por citometria de fluxo confirmou que praticamente toda a população celular expressou GFP. A intensidade média de fluorescência (MFI) da população positiva para GFP, indicativa do número de cópias de EGFP integradas, variou entre vários clones celulares. O clone G5 foi escolhido para estudos de acompanhamento. (B) Representação esquemática do gene EGFP, incluindo os locais de ligação para sgRNA e primers. Os locais de corte Cas9 são indicados pelas linhas tracejadas. (C) Células THP-1_GFP indiferenciadas (2 x 105) foram misturadas com RNPs Cas9:sgRNA pré-montadas antes da eletroporação com as configurações indicadas. Em seguida, as células foram transferidas em uma placa de 24 poços contendo 500 μL de meio RPMI pré-aquecido contendo 20% de FBS sozinho ou diluído 1:1 com meio condicionado produzido por uma cultura de células THP-1 de 48 h. A proliferação (barras laranja) e a porcentagem de células positivas para GFP (linha verde) foram monitoradas ao longo do tempo. Os dados representam a média para n = 2 réplicas biológicas. A moldura vermelha destaca a condição selecionada para experimentos subsequentes. (D) O DNA genômico de células THP-1_GFP editadas (RNP 3 sgRNA) ou não editadas (Cas9) foi extraído e a região ao redor do locus alvo amplificada por PCR. As células parentais THP-1 foram usadas como controle negativo. Os amplicons passaram por um ciclo de desnaturação/renaturação, levando à formação de um heteroduplex. Em seguida, foram detectadas incompatibilidades pelo ensaio T7EI. Os fragmentos de DNA foram separados por eletroforese em gel de agarose a 1,2%. (E) Os produtos de PCR purificados foram sequenciados pelo método de Sanger, seguido de análise com o algoritmo ICE. Os escores Indel e KO indicam a porcentagem de sequências não WT na amostra e a proporção de células com frameshift ou indel de 21+ pb, respectivamente. Clique aqui para ver uma versão maior desta figura.
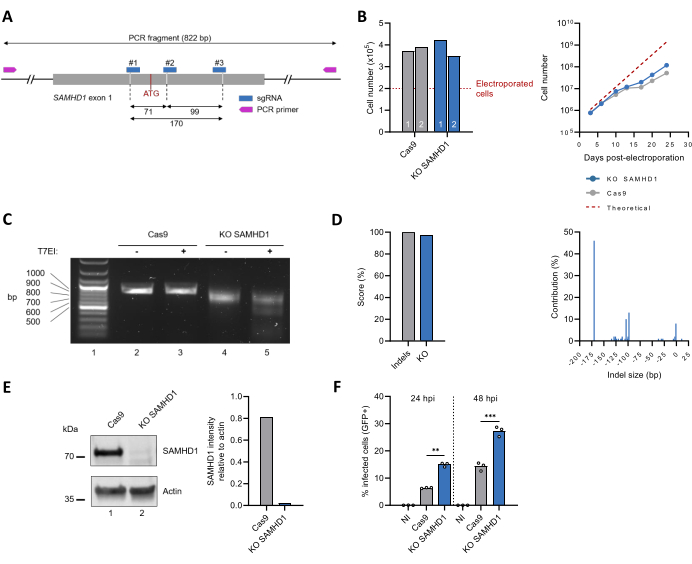
Figura 3: Produção e caracterização de uma população de células policlonais SAMHD1 KO THP-1. (A) Representação esquemática do éxon 1 de SAMHD1, incluindo os locais de ligação para sgRNA e primers. Os locais de corte Cas9 são indicados pelas linhas tracejadas e o códon ATG inicial é mostrado em vermelho. (B) Três dias após a eletroporação, as células viáveis (n = 2) foram quantificadas usando o teste de exclusão do azul de Trypan (diagrama à esquerda). O crescimento celular foi monitorado por 25 dias (diagrama à direita). A linha tracejada corresponde ao número teórico de células com base em um tempo estimado de duplicação de 49 h. (C) Os resultados da edição no nível genômico foram avaliados, conforme mostrado na Figura 2D. (D) Os genótipos após a edição CRISPR foram identificados e quantificados por análise baseada em ICE de sequências de Sanger. (E) A proteína contida no lisado bruto de células THP-1 editadas e não editadas foi separada por migração em gel SDS-PAGE e, em seguida, visualizada por immunoblotting usando anticorpos contra SAMHD1. A actina foi usada como controle de carga. A intensidade das bandas SAMHD1 e Actina foi quantificada por densitometria com o software ImageJ. (F) As células THP-1 não editadas (Cas9) e KO SAMHD1 foram diferenciadas por tratamento com PMA (300 ng / mL, 24 h) e, em seguida, desafiadas com um vírus HIV-1 pseudotipado VSVg expressando GFP como repórter. A proporção de células positivas para GFP foi pontuada por citometria de fluxo em 24 h e 48 h pós-infecção (hpi). NI: não infectado. **P < 0,01; P < 0,001 pelo teste t não pareado com correção de Welch. Clique aqui para ver uma versão maior desta figura.
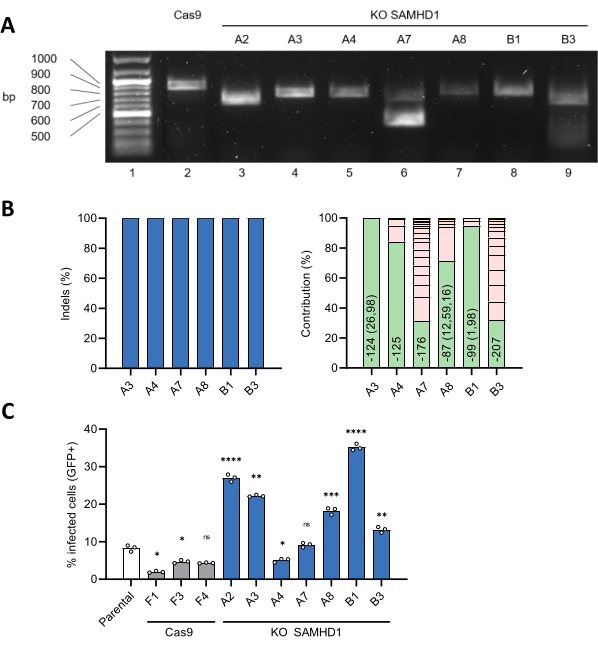
Figura 4: Produção e caracterização de clones de célula única SAMHD1 KO. (A) O resultado da edição para clones de célula única SAMHD1 KO selecionados foi avaliado monitorando a mobilidade dos fragmentos de DNA produzidos por amplificação por PCR da região que contém o local alvo por eletroforese em gel de agarose. (B) Os dados de sequenciamento de Sanger foram analisados com a ferramenta ICE, como na Figura 2E. As barras no gráfico da direita representam a diversidade de indels em cada população de células. A barra verde representa a sequência dominante e o tamanho total da exclusão associada. Os números entre parênteses indicam o tamanho das lacunas não contínuas. (C) A permissividade de clones de célula única não editados e SAMHD1 KO para infecção pelo HIV-1 foi testada como na Figura 3F e comparada com a de células THP-1 parentais não eletroporadas ou clones de células não editadas (Cas9). A taxa de infecção foi pontuada em 48 hpi. *P < 0,05; **P < 0,01; P < 0,001; P < 0,0001; ns P≥ 0,05 por Brown-Forsythe e Welch ANOVA com teste de comparações múltiplas de Dunnett. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Avaliação do resultado da edição para clones de célula única KO por teste T7EI adaptado. O DNA genômico de clones unicelulares KO ou células não editadas foi purificado e a região ao redor do locus alvo foi amplificada por PCR. Os amplicons de cada clone de célula única foram misturados na proporção de 1:1 com os de células de controle não editadas. Após a formação do heteroduplex e digestão do T7EI, o produto foi analisado por eletroforese em gel de agarose. Os clones editados produzem múltiplos fragmentos de digestão (clones A, B, C, D e E), enquanto os não editados mostram uma única banda WT (clones F e G). Clique aqui para baixar este arquivo.
Figura suplementar 2: Estratégia de bloqueio de citometria de fluxo para quantificar a porcentagem de células THP-1 tratadas com PMA infectadas pelo HIV-1. Usando a área de dispersão direta (FSC-A) contra a área de dispersão lateral (SSC-A), uma primeira porta é definida para eliminar detritos e selecionar as células THP-1 tratadas com PMA. Em seguida, células únicas são selecionadas com um SSC-A em relação a um gráfico SSC-H. Finalmente, células GFP-positivas são detectadas com GFP-A contra FSC-H. Clique aqui para baixar este arquivo.
Discussão
Aqui, um protocolo é descrito para obter uma edição mediada por CRISPR bem-sucedida da linha celular THP-1. A abordagem baseia-se na transferência de RNPs sgRNA/Cas9 pré-montados por eletroporação/nucleofecção. Essa estratégia foi escolhida para limitar os efeitos fora do alvo que potencialmente surgem na integração mediada por lentivírus do sgRNA/Cas9, produzindo expressão persistente da nuclease. Vários sgRNAs direcionados ao gene de interesse foram selecionados para obter uma edição confiável e eficiente, o que aumenta a probabilidade de criar indels genômicos, levando à perda de expressão de proteínas e KO32 funcional. Foi dado especial cuidado às configurações de eletroporação para obter a permeabilização ideal da membrana celular, o que foi necessário para garantir a entrega eficiente dos RNPs sgRNA / Cas9, evitando a morte celular excessiva33. Nesse sentido, um parâmetro crucial é o vigor da cultura de células antes da eletroporação (ou seja, baixo número de passagens).
A edição mediada por CRISPR-Cas9 foi validada em nível genético com o ensaio de detecção de incompatibilidade T7EI, que fornece uma avaliação rápida e econômica de várias amostras para pré-seleção antes do sequenciamento de Sanger. Este método foi inicialmente desenvolvido para estimar a eficiência de edição usando um único sgRNA, comparando a intensidade de amplicons de comprimento total e produtos de clivagem34,35. Digno de nota, os grandes indels (>70 bp) produzidos ao usar múltiplos sgRNA podem ser detectados comparando a mobilidade dos fragmentos de DNA obtidos por amplificação por PCR dos loci alvo de células editadas versus não editadas por eletroforese em gel padrão. No entanto, o ensaio T7EI pode ser útil para verificar a presença de sequências que abrigam pequenos indels (>1bp) em uma população de células policlonais ou, usando o protocolo adaptado, monoclonal.
A presença de mutações fora do quadro no alvo, que interrompem a sequência de codificação, foi confirmada por uma abordagem direta, onde o genoma de toda a população de células é extraído e usado como matriz para amplificação por PCR. Em seguida, os amplicons purificados passam por sequenciamento de Sanger em massa, seguido de análise de bioinformática com a ferramenta ICE. No geral, esse método supera a necessidade de subclonar os produtos de PCR em um plasmídeo e transformar os produtos de ligação em bactérias para isolar colônias unicelulares, que são sequenciadas individualmente.
Uma vez validada a inativação do locus alvo, é necessária uma caracterização completa da linhagem celular editada para atribuir com segurança um determinado fenótipo à falta da proteína de interesse e descartar efeitos imprevistos decorrentes de alterações fora do alvo, mas também no alvo que podem afetar a estrutura e/ou função do mRNA.
A ausência de expressão proteica deve ser confirmada por immunoblotting e/ou imunofluorescência usando possivelmente dois anticorpos que reconheçam epítopos diferentes. De fato, a introdução de um PTC em um éxon inicial próximo ao códon ATG canônico está associada ao risco de eventos ITL, que produzem variantes de proteína truncadas N-terminalmente36. Digno de nota, Tuladhar et al. relataram a detecção de espécies de proteínas aberrantes em uma proporção significativa (~ 50%) de células editadas por CRISPR de origem comercial ou interna18,19. A ocorrência de novos produtos proteicos foi atribuída a processos diversos e potencialmente específicos do tipo celular, como pular o éxon contendo indel ou o surgimento de locais de entrada de ribossomos internos (IRES), causando o início da síntese de proteínas em TIS alternativo a jusante do códon ATG canônico. Também há indícios de que a ITL está associada a uma fuga do NMD em certas circunstâncias37,38. Da mesma forma, as estratégias de edição do genoma direcionadas a um éxon tardio podem falhar em desencadear o NMD do mRNA mutado subjacente à ablação ineficaz da expressão gênica20. Finalmente, se Cas9 for guiado por múltiplos sgRNAs, podem surgir rearranjos cromossômicos complexos, incluindo inversões ou grandes deleções de fragmentos de DNA39, o que pode alterar a estrutura e a expressão do genoma, particularmente quando os elementos reguladores são direcionados40.
Para mitigar ainda mais o impacto dos resultados não intencionais de edição do genoma, estudos funcionais são necessários para confirmar a relação entre a perda de expressão da proteína e o fenótipo resultante (ou seja, atividade antiviral). Se as células editadas forem subclonadas, o comportamento de vários clones entre si e com a população policlonal deve ser comparado. A realização de experimentos de resgate, que consistem em restaurar a expressão gênica para reverter o fenótipo das células editadas para um fenótipo WT, também fortalecerá a especificidade e excluirá possíveis eventos fora do alvo.
Embora este protocolo tenha sido concebido para a edição de células THP-1, ele fornece uma estrutura geral para o fluxo de trabalho CRISPR-Cas9 KO que pode ser adaptada a outras linhagens celulares. Pode ser necessário ajustar os parâmetros de eletroporação e, se for aplicada uma tensão superior a 1800 V, deve ser utilizado um tampão de ressuspensão T, de acordo com as instruções do fabricante. Outra variável a ser considerada é a razão de concentração entre Cas9 e sgRNA durante a montagem da RNP. Embora a proporção de 1:9 tenha funcionado bem neste caso, modificá-la pode melhorar a eficiência da edição em outras circunstâncias. Finalmente, embora não seja benéfico aqui, o uso de meios condicionados para recuperação celular após eletroporação deve ser testado, pois o efeito pode ser diferente com diferentes linhagens celulares.
Divulgações
Todos os autores não têm conflitos de interesse.
Agradecimentos
Agradecemos ao JP Concordet (MNHN, U1154/UMR7196, Paris), G. Bossis (IGMM, Montpellier) e D. Schlüter (Hannover Medical School, Alemanha) pelo compartilhamento de protocolos e pela discussão. Este projeto recebeu financiamento do programa de investigação e inovação Horizonte 2020 da União Europeia (acordo de subvenção n.º 101017572 à AZ) e da ANRS (subvenção ECTZ162721 à AZ). A infraestrutura de investigação do Modelo de Doenças Infecciosas e Terapias Inovadoras (IDMIT) é apoiada pelo "programa investissement d'avenir (PIA)" sob a referência ANR_11_INSB_0008.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
Referências
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados