Method Article
Knockout génico mediado por CRISPR-Cas9 basado en electroporación en células THP-1 y aislamiento de clones de una sola célula
En este artículo
Resumen
La línea celular THP-1 se utiliza ampliamente como modelo para investigar las funciones de los monocitos/macrófagos humanos en diversas áreas de investigación relacionadas con la biología. Este artículo describe un protocolo para la ingeniería eficiente basada en CRISPR-Cas9 y el aislamiento de clones de una sola célula, lo que permite la producción de datos fenotípicos robustos y reproducibles.
Resumen
La línea celular THP-1 de leucemia monocítica aguda (LMA) humana se utiliza ampliamente como modelo para estudiar las funciones de los macrófagos derivados de monocitos humanos, incluida su interacción con patógenos humanos importantes, como el virus de la inmunodeficiencia humana (VIH). En comparación con otras líneas celulares inmortalizadas de origen mieloide, las células THP-1 conservan muchas vías de señalización inflamatoria intactas y muestran características fenotípicas que se asemejan más a las de los monocitos primarios, incluida la capacidad de diferenciarse en macrófagos cuando se tratan con forbol-12-miristato 13-acetato (PMA). El uso de la tecnología CRISPR-Cas9 para diseñar células THP-1 a través de la eliminación de genes (KO) específicos proporciona un enfoque poderoso para caracterizar mejor los mecanismos relacionados con el sistema inmunitario, incluidas las interacciones virus-huésped. Este artículo describe un protocolo para la ingeniería eficiente basada en CRISPR-Cas9 que utiliza la electroporación para administrar ribonucleoproteínas Cas9:sgRNA preensambladas en el núcleo de la célula. El uso de múltiples sgRNAs dirigidos al mismo locus en posiciones ligeramente diferentes da como resultado la deleción de grandes fragmentos de ADN, lo que aumenta la eficiencia de la edición, según lo evaluado por el ensayo de endonucleasa T7 I. La edición mediada por CRISPR-Cas9 a nivel genético se validó mediante secuenciación de Sanger seguida de un análisis de inferencia de ediciones CRISPR (ICE). La depleción proteica se confirmó mediante inmunotransferencia junto con un ensayo funcional. Con este protocolo, se lograron hasta un 100% de indels en el locus objetivo y una disminución de más del 95% en la expresión de proteínas. La alta eficiencia de edición hace que sea conveniente aislar clones de una sola célula limitando la dilución.
Introducción
THP-1 es una línea celular derivada de monocitos humanos aislada de un paciente que padece leucemia aguda (LMA), que muestra características fenotípicas muy parecidas a las de los monocitos primarios1. En comparación con los macrófagos primarios derivados de monocitos, que no se dividen y muestran una vida útil limitada y una variabilidad entre donantes e intradonantes en el fenotipo, las células THP-1 pueden cultivarse prácticamente para siempre y tienen un comportamiento más homogéneo que favorece la reproducibilidad de los resultados 2,3,4,5,6 . En particular, las células THP-1 se pueden diferenciar hacia un fenotipo similar al de los macrófagos con forbol-12-miristato 13-acetato (PMA), lo que las convierte en un modelo in vitro ampliamente utilizado para investigar las respuestas de monocitos/macrófagos a las señales inflamatorias 7,8,9,10,11,12,13 o la infección por patógenos humanos clínicamente relevantes, incluido el VIH 14,15,16. La posibilidad de modificar genéticamente las células THP-1 es de interés en muchas áreas de investigación relacionadas con la biología.
La proteína 9 asociada a CRISPR (CRISPR-Cas9) es un sistema inmunitario adaptativo procariota que se basa en la nucleasa guiada por ARN para degradar los genomas virales invasores, que ha sido reprogramada como una herramienta de ingeniería genética17. El proceso de edición del genoma se desarrolla en tres etapas: reconocimiento, escisión y reparación. Un ARN de una sola guía (sgRNA) recluta la nucleasa Cas9 a un locus genómico específico a través del emparejamiento de bases con su secuencia guía de 20 pb. La presencia de una secuencia de motivo adyacente al protoespaciador (MAP) directamente a 3' de la secuencia objetivo genómica de 20 pb desencadena el desenrollado y la escisión mediados por Cas9 en ambas hebras de ADN entre las posiciones 17 y 18 (3-pb 5' del PAM). La rotura de doble cadena (DSB) resultante es procesada por dos vías de reparación principales. En ausencia de una homología de rodamiento de plantilla de reparación con el locus dañado, la vía de unión de extremos no homólogos (NHEJ) propensa a errores introducirá inserciones y/o deleciones aleatorias de nucleótidos (indels), lo que podría conducir a mutaciones de cambio de marco y/o la introducción de codones de terminación prematura (PTC). A su vez, los ARNm que contienen PTC son objeto de degradación por la vía de desintegración del ARNm (NMD) mediada por sinsentido, lo que en última instancia interrumpe la expresión/función de la proteína 18,19,20. Alternativamente, la vía de reparación dirigida por homología (HDR) dependiente de la plantilla puede operar y reparar fielmente el DSB. Este mecanismo se ha aprovechado para lograr una edición genética precisa, incluidos los knock-ins y las sustituciones de bases. Vale la pena señalar que el estado del ciclo celular es un factor importante que influye en la elección de la vía de reparación de DSB. De hecho, NHEJ está activo en todas las etapas del ciclo celular, mientras que HDR se restringe principalmente a las fases S/G221.
Las células THP-1 crecen en suspensión y son notoriamente difíciles de transfectar con ADN plasmídico, procedimiento que posiblemente también altere su viabilidad y/o capacidad de diferenciación22,23. La transducción con vectores lentivirales basados en el VIH-1 que codifican tanto Cas9 como el sgRNA se emplea a menudo para eliminar (KO) un gen de interés24. La integración del casete Cas9/sgRNA en el genoma celular garantiza una expresión prolongada y un KO eficiente, pero también es una fuente persistente de efectos fuera del objetivo25. Alternativamente, las ribonucleoproteínas (RNP) Cas9:sgRNA preensambladas se administran por electroporación, un método que implica la formación temporal de poros tanto en el plasma como en las membranas nucleares mediante la aplicación de impulsos eléctricos. Preservar la viabilidad celular es un reto importante a la hora de emprender este enfoque.
Aquí, se produjo una línea celular THP-1 que expresa GFP de manera estable (THP-1_GFP) para que sirva como herramienta para establecer un protocolo para lograr una edición eficiente basada en CRISPR-Cas9. Después de diseñar una estrategia para inactivar el gen EGFP utilizando tres sgRNAs simultáneamente (enfoque multiguía), se determinó la eficiencia de KO entre varias condiciones de electroporación utilizando la expresión de GFP como lectura. La proliferación celular se monitoreó en paralelo. La edición génica se confirmó mediante un ensayo de endonucleasa T7 I (T7EI) y secuenciación de Sanger, seguido de un análisis con el algoritmo de inferencia de ediciones CRISPR (ICE)26. Los parámetros que produjeron una disminución de la expresión de GFP de hasta el 95%, con células THP-1 recuperando tasas de crecimiento normales después de la electroporación, se emplearon con éxito para inactivar un gen endógeno (SAMHD1) y producir clones de THP-1 de una sola célula.
Protocolo
Los detalles de los reactivos y el equipo utilizado en este estudio se enumeran en la Tabla de Materiales.
1. Diseño de guías con CRISPOR (Figura 1.1)
NOTA: El software SnapGene Viewer se puede utilizar en los pasos 4, 7 y 10 para anotar el sitio objetivo de edición y la ubicación de la hibridación del cebador de PCR dentro del gen de interés.
- Ir al sitio web de Ensembl (www.ensembl.org). En el cuadro de búsqueda , seleccione una especie y escriba el nombre del gen de interés. Haga clic en Ir. Seleccione el resultado que corresponda al gen (no una transcripción).
- Haga clic en Mostrar tabla de transcripción y luego seleccione el ID de transcripción que corresponda a la codificación de proteínas con etiqueta dorada en la columna Biotipo . Una vez en la página de transcripción, haga clic en Exones en el menú de la izquierda.
- Desplácese hacia abajo y haga clic en Descargar secuencia. Asegúrese de que el formato de archivo sea FASTA. En Configuración - Secuencias incluidas, anule la selección de todo excepto Secuencia genómica.
Introduzca el número "500" en Secuencia de flanqueo en cualquiera de los extremos del cuadro de transcripción . Haga clic en Vista previa, seleccione la secuencia completa (solo los nucleótidos sin el encabezado) y cópiela (Ctrl + C). - Abra SnapGene Viewer y haga clic en Nuevo archivo de ADN >. Pegue la secuencia (Ctrl+V) en el cuadro Crear una secuencia . Desactive Detectar entidades comunes y seleccione Lineal en Topología.
Cambie el nombre del archivo y haga clic en Crear. En el menú de la izquierda, anule la selección de Mostrar enzimas (primer icono). En la parte inferior de la ventana, seleccione la pestaña Secuencia .
NOTA: Este paso permite la recuperación de la secuencia completa del gen, incluidos los exones, los intrones y las secuencias flanqueantes de 500 pb (opcional). Esta última información es útil para el diseño de cebadores de PCR para la amplificación de un sitio objetivo ubicado dentro del primer exón. - De vuelta en el sitio web de Ensmbl, desplácese hacia arriba en la vista previa del archivo y haga clic en Atrás. Ahora, cambie el formato de archivo a RTF. En Configuración - Secuencias incluidas, anule la selección de todo excepto Exones. En Mostrar variantes, seleccione No. Haga clic en Descargar en la parte superior de la página.
- Abra el archivo descargado (con Word), que contiene la secuencia de exones y muestra la secuencia de codificación en azul, comenzando con el ATG inicial. Elija el exón al que se dirigirá la edición dirigida por CRISPR-Cas9 (consulte las recomendaciones a continuación), selecciónelo y cópielo (Ctrl+C).
- El exón objetivo es un exón temprano o un exón que codifica un dominio funcionalmente importante de la proteína. Vale la pena señalar que el establecimiento de una PTC en un exón tardío cerca de la UTR 3' probablemente no provocará NMD, lo que conducirá a la expresión de una proteína C-terminal truncada. Por el contrario, la introducción de una PTC en un exón temprano proximal al sitio de iniciación nativo se asocia con un riesgo de traducción ilegítima (ITL, también conocida como iniciación de traducción alternativa (ATI)), que produce la expresión inesperada de una proteína truncada N-terminal que comienza en un sitio de iniciación de traducción (TIS) en el marco aguas abajo del primer codón ATG. Para mitigar este último riesgo, se recomienda evaluar la aparición de TIS alternativos utilizando ATGpr27 (atgpr.dbcls.jp) y/o NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/).
- Asegúrese de que el exón de destino contenga una secuencia de codificación. Sin embargo, podría resultar útil elegir un recocido de sgRNA con la región 5'UTR aguas arriba del ATG inicial para incluirlo en el fragmento delecionado.
- Idealmente, el exón objetivo debería ser común a todas las variantes de transcripción del gen que codifican proteínas. Verifique si ese es el caso en el Visor de Datos del Genoma (www.ncbi.nlm.nih.gov/gdv/) del NCBI buscando el gen de interés.
Al hacer clic en el nombre del gen (en verde) en la ventana de visualización, se mostrarán las variantes de la transcripción (en púrpura). Los exones están representados por un rectángulo.
- En SnapGene Viewer, presione Ctrl+F, Ctrl+V y, a continuación, Intro para buscar la secuencia del exón. Presione Ctrl + T para agregar una nueva característica, asígnele un nombre, cambie el tipo a "exón" y haga clic en Aceptar.
- Vaya al sitio web de CRISPOR (http://crispor.gi.ucsc.edu/) y pegue la secuencia de exones en el paso 1. En primer lugar, seleccione un genoma de referencia en el paso 2 y, a continuación, el tipo de PAM al que se dirigirá en el paso 3, normalmente 20 pb-NGG para SpCas9. Haga clic en ENVIAR.
- Seleccione dos sgRNAs para que estén separados por hasta 150 pb, luego seleccione un tercer sgRNA en el medio. Estas son algunas pautas para la selección de sgRNA:
- La puntuación de especificidad del MIT se relaciona con los efectos fuera del objetivo. Una puntuación más alta indica menos posibles objetivos desviados. La columna de la derecha muestra tres sitios fuera del objetivo pronosticados clasificados de mayor a menor probabilidad, junto con sus ubicaciones (en un exón, un intrón o una región intergénica). Se puede acceder a la lista completa de sitios fuera del objetivo previstos haciendo clic en Mostrar todo. Si es posible, seleccione sgRNAs con una puntuación MIT >80, dando prioridad a aquellos sin objetivos desviados para 0, 1 o 2 discrepancias. Además, deben evitarse los sgRNAs con objetivos fuera de un exón, que tienen el mayor potencial para afectar al fenotipo.
- Para conocer la eficacia prevista, consulte la puntuación de Doench '16. Tenga en cuenta que una puntuación alta de Doench '16 simplemente indica que es más probable que el sgRNA sea efectivo. La eficacia real debe determinarse experimentalmente. Por esta razón, siempre es útil seleccionar varios sgRNAs, incluso si no están destinados a ser utilizados juntos.
- Un contenido de GC demasiado alto o demasiado bajo, así como ciertos motivos, pueden ser perjudiciales para la eficiencia del sgRNA y deben evitarse. Estos parámetros son resaltados por CRISPOR.
- Repita el paso 1.7 para agregar la secuencia de sgRNA y la secuencia PAM asociada a la secuencia génica en SnapGene Viewer. Al mismo tiempo, pegue la secuencia de sgRNA (sin el PAM) en un archivo de texto o Excel para conservar la orientación 5'-3' requerida al pedir el oligonucleótido.
- Para la amplificación basada en PCR del sitio objetivo, diseñe un par de cebadores a su alrededor. El tamaño del amplicón debe estar entre 800 y 1000 pb. Se utilizó PrimerQuest para diseñar los cebadores para este protocolo (https://eu.idtdna.com/pages/tools/primerquest). Alternativamente, CRISPOR proporciona una lista de cebadores para amplificar la región genómica objetivo, así como los sitios fuera del objetivo. Después de mostrar la lista completa de posibles sitios fuera del objetivo (paso 1.9.1), haga clic en Cartillas fuera del objetivo en la esquina inferior derecha.
2. Preparación de reactivos y celdas para electroporación (Figura 1.2)
- Prepare una placa de cultivo de 24 pocillos para recuperar las células después de la electroporación llenando los pocillos con 500 μL de medio RPMI 1640 suplementado con un 20% de suero fetal bovino (FBS) inactivado por calor (56 °C, 30 min), filtrado (0,20 μm). No agregue antibióticos. Mantener la placa en una incubadora humidificada a 37 °C y 5% de CO2 durante 24 h hasta la electroporación.
- Si los sgRNAs se envían secos, rehidratarlos con tampón TE (10 mM de Tris, 1 mM de EDTA, pH 8,0) hasta una concentración final de 100 μM (es decir, 10 μL de tampón TE por 1 nmol de sgRNA). Vortex durante 30 s, incubar a 4 °C durante la noche para permitir una rehidratación completa y, después de una breve homogeneización de la pipeta, almacenar la solución madre de sgRNA a -20 °C. Dependiendo del volumen final, haga alícuotas para evitar múltiples ciclos de congelación y descongelación.
- Prepare una solución de sgRNA de trabajo a una concentración final de 30 μM diluyendo la solución madre de 100 μM en agua libre de nucleasas. Vórtice durante 30 s e incube 5 min a temperatura ambiente.
- Ensamble los RNP Cas9:sgRNA en una proporción molar de 1:9 diluyendo simultáneamente 1 μL de cada uno de los tres sgRNAs de 30 μM y 0,5 μL de soluciones Cas9 de 20 μM en 3,5 μL de tampón de resuspensión R, incluido en el kit de electroporación (volumen final de 7 μL, para una condición experimental; escala correspondiente). Agite brevemente e incube durante 5 minutos a temperatura ambiente.
- Mientras tanto, prepare un control sin editar añadiendo 0,5 μL de 20 μM de Cas9 a 6,5 μL de tampón de resuspensión R. Vortex brevemente e incube durante 5 minutos a temperatura ambiente.
- Añada 5 μL de tampón de resuspensión R a todas las muestras para obtener un volumen final de 12 μL por condición de electroporación.
- Prepare las celdas THP-1 para la electroporación.
- Evaluar la concentración y viabilidad mediante la prueba de exclusión de azul de tripano. Diluir las células 1:2 en una solución de tinción con azul de tripano al 0,4%. Después de 30 s de incubación, homogeneizar bien y añadir 10 μL en la pared de una cámara de recuento con un estilo de rejilla mejorado por Neubauer. Cuente tres cuadrados grandes y divida la cuenta por 100 para obtener la concentración de células (x106 células/mL).
NOTA: La salud de las células influye en su sensibilidad a la electroporación. Se debe tener cuidado de realizar el cultivo celular en condiciones óptimas. El tiempo fuera de la incubadora debe ser limitado, y todos los reactivos y soluciones deben prepararse y calentarse con anticipación. - Para cada condición, recoja un volumen equivalente a 0,2 x 106 celdas y centrifugue (336 x g, 5 min, 20 °C).
- Aspirar el sobrenadante con una pipeta y volver a suspender el pellet en 500 μL de PBS. Vuelva a centrifugar (336 x g, 5 min, 20 °C).
- Aspirar el sobrenadante cuidadosamente con una pipeta y resuspender el pellet de la célula THP-1 con los 12 μL de solución de RNP (paso 2.6).
- Evaluar la concentración y viabilidad mediante la prueba de exclusión de azul de tripano. Diluir las células 1:2 en una solución de tinción con azul de tripano al 0,4%. Después de 30 s de incubación, homogeneizar bien y añadir 10 μL en la pared de una cámara de recuento con un estilo de rejilla mejorado por Neubauer. Cuente tres cuadrados grandes y divida la cuenta por 100 para obtener la concentración de células (x106 células/mL).
3. Configuración del sistema de electroporación y nucleofección (Figura 1.3)
- Coloque la estación de pipetas debajo de una cabina de bioseguridad, coloque un tubo de electroporación en el soporte y agregue 3 mL de tampón E, incluido en el kit de electroporación.
- Después de encender el dispositivo de electroporación, utilice la pantalla táctil para configurar los siguientes parámetros de electroporación: Voltaje = 1 500 V, Duración = 10 ms, Número = 3.
- Equipar la pipeta de electroporación con una punta y aspirar 10 μL de la solución RNP/THP-1 (paso 2.7.4). Inserte la pipeta en el tubo de electroporación y pulse Start en la pantalla del dispositivo de electroporación. Espere a que aparezca el mensaje Completado y retire la pipeta del tubo.
- Transfiera las celdas a un pocillo de la placa precalentada de 24 pocillos y homogeneice suavemente. Vuelva a colocar la placa en la incubadora humidificada y déjela reposar sin ser molestada durante 72 h.
NOTA: Tenga cuidado de no hacer burbujas al pipetear la suspensión RNP/THP-1, ya que interferirán con el procedimiento de electroporación. Si aparece un mensaje de error en ausencia de un arco eléctrico visible, retire la pipeta de electroporación del tubo y vuelva a pulsar Iniciar . Sin embargo, si se observa un arco eléctrico en forma de una breve chispa brillante, podría indicar la presencia de burbujas. Es probable que el procedimiento de electroporación falle, incluso en ausencia de un mensaje de error.
4. Recuperación de THP-1 72 h después de la electroporación (Figura 1.4)
- Cuente las células para evaluar la concentración (paso 2.7.1) 72 h después de la electroporación.
- Si hay suficientes células (es decir, ≥0,6 x 106 células/mL), diluirlas al menos un factor 2 con RPMI suplementado con 20% de FBS y 1% de penicilina-estreptomicina y llevar la concentración entre 0,3-0,5 x 106 células por mL. De lo contrario, deje que la celda se recupere durante otras 72 horas.
- Pase y amplifique las celdas hasta que haya suficientes para la validación de KO. Mientras tanto, se puede iniciar el aislamiento de clones unicelulares (paso 7).
5. Validación de la edición génica mediante el ensayo de desajuste T7EI (Figura 1.5)
NOTA: El ensayo podría subestimar la eficiencia de edición dado que T7EI reconoce discrepancias mayores que 1 pb. Por lo tanto, el ensayo T7EI no es útil para el cribado de poblaciones de células homocigóticas (es decir, clones de células individuales) a menos que se modifique adecuadamente (paso 5.7).
- Evalúe la concentración de células (paso 2.7.1) y extraiga un volumen equivalente a 0,1 x 106 células en un tubo de 1,5 mL. Centrífuga (336 x g, 5 min, 20 °C), aspire el sobrenadante y vuelva a suspender el pellet en 500 μL de PBS. Centrifugar de nuevo y, con una pipeta, aspirar la mayor cantidad posible de sobrenadante sin alterar el pellet. Congele la muestra y guárdela a -20 °C.
- Extraer el ADN genómico para que sirva de matriz para la amplificación por PCR.
- Resuspender el pellet con 50 μL de solución de extracción de ADN, homogeneizar y transferir todo el volumen en un tubo de PCR de 0,2 mL. Vórtice y centrífuga brevemente (pulso durante 3 s).
- Coloque el tubo en un termociclador y caliéntelo a 65 °C durante 15 minutos, seguido de 98 °C durante 10 minutos.
- Diluir el ADN extraído con 90 μL de agua ultrapura. Vórtice y centrífuga brevemente (5.000 x g, 3 s).
- Diluya el cebador de PCR (consulte la tabla de materiales) en agua ultrapura hasta obtener una concentración final de 10 μM (es decir, 10 pmol/μL).
- Prepare la mezcla de PCR (siguiendo la NOTA a continuación) en un tubo de PCR de 0,2 mL (volumen final = 50 μL, para una condición). Por lo general, habrá al menos dos condiciones: el KO y el control sin editar solo con Cas9.
NOTA: ADN genómico purificado: 10 μL; Cebador directo e inverso (10 μM): 2,5 μL cada uno, la concentración final de 500 nM; Tampón de reacción (5x): 10 μL. Vortex bien antes de añadir. Mezcla de dNTP (25 mM de cada uno): 0,6 μL, concentración final de 0,3 mM para cada dNTP. ADN polimerasa: 0,5 μL; Agua ultrapura: 23,9 μL. Vórtice y centrífuga brevemente (pulso durante 3 s). - Coloque los tubos en el termociclador y ejecute el programa de PCR con los siguientes ajustes:
NOTA: Un ciclo a 95 °C durante 5 min, seguido de 30 ciclos [98 °C durante 20 s (etapa de desnaturalización), X °C durante 15 s (etapa de recocido), 72 °C durante 45 s (etapa de elongación)], luego un ciclo final a 72 °C durante 2 min. Al final de la amplificación, retire los tubos, el vórtice y centrifugue brevemente (pulse durante 3 s). La temperatura de recocido (X) es la temperatura de fusión (Tm) de los cebadores menos 5 °C. - Para una población policlonal editada: en un nuevo tubo de 0,2 mL, añadir 17,5 μL del amplicón de PCR y 2 μL de NEBuffer 2 (10x) para un volumen final de 19,5 μL. Vórtice y centrífuga brevemente (pulso durante 3 s). Alternativamente, para examinar clones de una sola célula, mezcle 1:1 de los productos de PCR de las células de control editadas y no editadas (paso 5.6) (Figura complementaria 1).
- Para la formación heterodúplex, coloque los tubos en el termociclador y ejecute el siguiente programa: un ciclo a 95 °C durante 10 min, uno con una rampa de -2 °C/s de 95 a 85 °C, uno con una rampa de -0,3 °C/s de 85 a 25 °C y un ciclo de enfriamiento final a 10 °C en HOLD.
- Después de la formación heterodúplex, agregue 0,5 μL de solución T7EI en el tubo. Incubar a 37 °C durante 30 min.
- Prepara un gel de agarosa al 1,2%:
- Pesar la agarosa en un frasco de vidrio y añadir el volumen adecuado de 1x tampón TAE (40 mM de Tris-acetato, 1 mM EDTA, pH 8,3). Colócalo en un microondas prestando atención a no enroscar la tapa firmemente. Calentar varias veces hasta que los cristales de agarosa se disuelvan por completo.
NOTA: Si es necesario, mezcle la solución. No dejes que hierva, o el volumen disminuirá, cambiando la concentración de agarosa. - Añadir la tinción de gel de ADN diluida a 1/20.000 y mezclar bien.
- Vierta la solución de agarosa en un molde, agregue un peine y deje que se solidifique a temperatura ambiente.
- Pesar la agarosa en un frasco de vidrio y añadir el volumen adecuado de 1x tampón TAE (40 mM de Tris-acetato, 1 mM EDTA, pH 8,3). Colócalo en un microondas prestando atención a no enroscar la tapa firmemente. Calentar varias veces hasta que los cristales de agarosa se disuelvan por completo.
- Prepare las muestras para la electroforesis en gel mezclando 5 μL de los productos de digestión T7EI (paso 5.9) o el producto de PCR no digerido con 5 μL de agua y 2 μL de colorante de carga de ADN 6x. Cargue las muestras y la escala de tamaño y migre a 80 V durante 45 min (el tiempo se puede adaptar en función del tamaño esperado de los fragmentos de ADN). Una vez finalizada la migración, adquiera una imagen del gel con un sistema de imagen adecuado.
6. Validación de la edición génica mediante análisis de secuenciación de Sanger (Figura 1.6)
- Para caracterizar la modificación genética, purificar los productos de PCR (paso 5.4) y, después de la secuenciación de Sanger, analizar los resultados con la herramienta ICE (https://www.synthego.com/products/bioinformatics/crispr-analysis).
- Controle si una proteína se puede producir a pesar de las modificaciones.
- Descargue los resultados de ICE y abra el archivo contribs.txt.
- Compare las secuencias editadas con la contraparte de WT. Mapea los indels y verifica que surgieron en la región genómica objetivo. Evalúe su tamaño. Si la longitud indel no es múltiplo de tres, se producirá un cambio de marco y posiblemente se introducirá un PTC. Se espera que el ARNm mutado sufra una degradación mediada por NMD.
7. Aislamiento de clones unicelulares mediante dilución limitada (Figura 1.7)
NOTA: El aislamiento de clones unicelulares no es obligatorio. Sin embargo, si se opta por hacerlo, es importante caracterizar múltiples clones y comparar su fenotipo con la población policlonal original.
- Tome una muestra de suspensión celular, dilúyala hasta la mitad con medio de cultivo y evalúe la concentración (paso 2.7.1). La dilución aumenta la precisión del recuento.
- Realice de 2 a 3 pasos de dilución en serie para alcanzar una concentración de 7 células/mL. Dispensar 100 μL de la suspensión celular por pocillo en una placa de 96 pocillos de fondo redondo (es decir, 0,7 celdas por pocillo). Deje que la célula decante durante unas horas antes de observar las placas en el microscopio para identificar los pocillos que contienen una célula.
- Monitoree regularmente la formación y el crecimiento de colonias y transfiera las células a una placa de cultivo o matraz más grande cuando sea necesario.
8. Caracterización funcional de células THP-1 KO SAMHD1 mediante ensayo de restricción de VIH-1
- Siembra THP-1 en una placa de cultivo de 24 pocillos con 0,25 x 106 células por pocillo en 300 μL de RPMI suplementada con 10% de FBS, 1% de penicilina-estreptomicina (RPMI completa) y que contiene 300 ng/mL de PMA para la diferenciación. Mantener la placa en una incubadora humidificada (37 °C, 5% CO2) durante 24 h.
- Sustituya el medio por un medio RPMI completo sin PMA y vuelva a colocar la placa en la incubadora durante otras 24 horas.
- Con una bomba de vacío, aspire todo el medio de cultivo. Añadir 250 μL de solución que contiene stock viral de VIH-1-GFP pseudotipado por VSVg (MOI = 0,5 IFU/célula). Incluya un control no infectado. Coloque la placa a 4 °C durante 2 h para sincronizar la infección.
- Lave las celdas una vez con RPMI. Agregue 500 μL de RPMI completo a cada pocillo e incube durante 48 h en condiciones estándar (el tiempo se puede ajustar).
- Después de retirar el medio de cultivo, lave las células una vez con 1x PBS frío. A continuación, añadir 100 μL de tripsina-EDTA al 0,05% en cada pocillo. Coloque la placa en una incubadora a 37 °C hasta que las células se desprendan por completo (5 min deberían ser suficientes) antes de agregar 200 μL de RPMI completo para inactivar la enzima. Transfiera 250 μL de cada pocillo a una placa de fondo redondo de 96 pocillos (asegúrese de que el citómetro de flujo acepte placas de cultivo).
- Centrifugar la placa (757 x g, 3 min, deceleración = 6, 20 °C) y aspirar el sobrenadante con una pipeta multicanal. Vuelva a suspender el pellet con 100 μL de PFA al 4% e incube a 4 °C durante 10 min. Añadir 100 μL de PBS frío 1X.
- Analice la tasa de infección, representada por las células GFP positivas, mediante citometría de flujo (consulte la Figura complementaria 2 para obtener una estrategia de activación sugerida).
Resultados
Se generó una línea celular THP-1 que expresa de forma estable la proteína reportera GFP (THP-1_GFP) (Figura 2A) y se utilizó como herramienta para establecer un protocolo para una edición génica eficiente mediada por CRISPR-Cas9. Para ello, se diseñaron 3 sgRNA dirigidos al gen EGFP con la herramienta web CRISPOR29 (Figura 2B), que se complejaron simultáneamente con Cas9 en una proporción molar de 9:1 para formar RNPs antes de su entrega en las células por electroporación utilizando diferentes configuraciones. A continuación, las células se cultivaron en un medio RPMI que contenía un 20% de FBS solo (Figura 2C, panel superior) o se diluyeron 1:1 con medios acondicionados (Figura 2C, panel inferior). La proliferación celular y la expresión de GFP, como una lectura de la eficiencia de EGFP KO , se monitorearon a lo largo del tiempo. En varias condiciones, el porcentaje de células positivas para GFP disminuyó drásticamente, alcanzando una reducción del >90 % el día 7 después de la electroporación (pe). En el día 3 pe, el número de células se había reducido a la mitad, probablemente debido al impacto negativo de la electroporación en la viabilidad de las células. Sin embargo, el número de células volvió a aumentar y el tiempo de duplicación volvió a una tasa normal a los 7 días (Figura 2C, panel superior). El uso de medios condicionados no favoreció la recuperación celular en nuestras condiciones experimentales (Figura 2C, panel inferior). Sobre la base de estos resultados, se eligieron tres pulsos de 10 ms de 1500 V para los experimentos de seguimiento.
A continuación, se caracterizó a nivel genómico el EGFP KO mediado por CRISPR-Cas9. Para ello, se extrajo el ADN genómico de las células THP-1_GFP, se electroporó con los RNPs Cas9:sgRNA (editados) o Cas9 solos (sin editar), y se utilizó como molde para amplificar por PCR una región de 891 pb que contenía el sitio objetivo. Las células parentales THP-1 que carecían de EGFP también se incluyeron como control negativo. A continuación, se preparó el producto de PCR de células THP-1_GFP editadas para la detección de desajustes mediante el ensayo T7EI, seguido de la separación de los fragmentos de ADN mediante electroforesis en gel de agarosa. Se visualizó fácilmente una banda de ~900 pb correspondiente al tamaño esperado del amplicón WT para las células THP-1_GFP no editadas, pero no para el control parental THP-1 (Figura 2D, compare las líneas 2 y 3). En el caso de las células THP-1_GFP editadas, la banda de ~900 pb se volvió indetectable y fue reemplazada por una de migración más rápida, cuyo tamaño coincidía con la pérdida de un fragmento de ~75 pb como consecuencia de la edición CRISPR (Figura 2D, línea 4). Este patrón de bandas se alteró durante la digestión de T7EI, causando la aparición de fragmentos de ADN más pequeños, lo que confirma aún más la edición exitosa del sitio objetivo (Figura 2D, línea 5). Para obtener información sobre la eficiencia de la edición y los genotipos específicos de la población editada, el amplicón de PCR se secuenció utilizando el método de Sanger, seguido de un análisis con la herramienta ICE26. La muestra de control no editada se procesó en paralelo. Recuperamos puntuaciones indel y KO del 100%, correspondientes al porcentaje de secuencias no WT en la muestra editada y la proporción de indels que probablemente conducen a un KO funcional, respectivamente (Figura 2E, izquierda). El análisis detallado del espectro de tamaños y frecuencias del indel mostró la presencia de dos poblaciones dominantes con una deleción de 62 pb o 76 pb y que representan el 57% o el 37% de las secuencias totales, respectivamente (Figura 2E, derecha). Estos hallazgos sugieren que el sgRNA #1, en combinación con el sgRNA #2 o #3, desencadenó DSB concurrente en el gen diana (Figura 2B).
Este protocolo se aplicó para inactivar el gen endógeno que codifica para SAMHD1 (Figura 3A). La caracterización de la línea celular policlonal SAMHD1 KO THP-1 incluyó el monitoreo de la proliferación celular a lo largo del tiempo, que coincidió con la de las células THP-1 no editadas (Figura 3B). El resultado de la edición génica se evaluó mediante la amplificación basada en PCR de la secuencia diana, que reveló una banda más pequeña para las células SAMHD1 KO en comparación con el control solo Cas9 (Figura 3C, comparar líneas 2 y 4), lo que indica una pérdida de secuencia de ADN. Estas observaciones fueron confirmadas por el ensayo T7EI, mostrando la apariencia de productos de escisión para las células SAMHD1 KO, pero no sin editar (Figura 3C, comparar líneas 3 y 5). El análisis de los datos de secuenciación de Sanger con la herramienta ICE arrojó puntuaciones indel y KO del 100% y el 97%, respectivamente. Las principales modificaciones dentro del locus SAMHD1 fueron deleciones de secuencia de 170 pb, 93 pb o 104 pb en el 46%, 13% y 10% de las secuencias, respectivamente (Figura 3D). Los niveles de expresión de proteínas en el lisado de células crudas también se evaluaron mediante inmunotransferencia. La banda de ~70 kDa consistente con el tamaño de la SAMHD1 endógena se vuelve prácticamente indetectable después de la edición, lo que corresponde a una reducción estimada del >97 % (Figura 3E). Por último, también se evaluó la caracterización fenotípica de las líneas celulares policlonales SAMHD1 KO THP-1 mediante pruebas de susceptibilidad a la infección por VIH-1. De acuerdo con su conocida función antiviral30,31, la inactivación del locus SAMHD1 se acompañó de un aumento de la tasa de infección por VIH-1 (Figura 3F).
Paralelamente, se generaron clones unicelulares limitando la dilución. La amplificación del sitio objetivo genómico de los clones SAMHD1 KO produjo productos de PCR con una mayor movilidad electroforética en comparación con los de las células de control no editadas (Figura 4A). El análisis basado en ICE de los datos de secuenciación de Sanger arrojó una puntuación indel del 100% para todos los clones de una sola célula seleccionados, excepto uno (clon A2) (Figura 4B, izquierda). La caracterización de los resultados de edición a nivel genómico arrojó una sola secuencia para el clon A3 con una deleción de 98 pb ubicada a 28 pb aguas abajo del ATG inicial, lo que conduce a un cambio fuera de marco (Figura 4B, derecha). Los otros clones contenían tres (clon B1) o más secuencias, indicativas de la presencia de alelos que habían sufrido diferentes eventos de edición y/o más de una célula por pocillo en la etapa de dilución limitante (Figura 4B, derecha). En particular, el 11% de las secuencias de los clones A4 y A7 llevan indels aguas arriba del ATG inicial, dejando la secuencia de codificación inalterada. Además, el 1% de las secuencias del clon A7 carecían de un fragmento de 123 pb 9 pb aguas abajo del ATG inicial, lo que resultó en una deleción en el marco de 41 aminoácidos. De acuerdo con estos hallazgos, los estudios funcionales revelaron que los clones con un locus SAMHD1 alterado (A3, A8, B1, B3), por lo que carecían de expresión de SAMHD1, eran altamente permisivos a la infección por VIH-1 (Figura 4C). Por el contrario, los clones A4 y A7, donde SAMHD1 está intacto, fueron tan refractarios como los clones parentales o de control THP-1 sin editar (Figura 4C). En conclusión, se confirmó la edición exitosa del locus SAMHD1 en el clon A3.

Figura 1: Esquema experimental. (1) CRISPOR se utiliza para generar secuencias de sgRNA contra el exón objetivo. Tres de ellos se seleccionan de acuerdo con la alta eficacia en el objetivo y la débil predicción fuera del objetivo. (2) Los tres ARNg se mezclan con Cas9 para ensamblar la ribonucleoproteína (RNP), lo que da como resultado una mezcla de tres RNP diferentes. (3) Las células se electroporan para permitir la entrada de las RNP. (4) Las células se transfieren a una placa de cultivo de 24 pocillos y se dejan reposar durante al menos 72 h. (5) La edición de genes se prueba primero cualitativamente mediante un ensayo de digestión T7EI, y (6) para las condiciones validadas, caracterizadas además por secuenciación de Sanger y análisis ICE. (7) Si es necesario, las células pueden sembrarse después de limitar la dilución para producir poblaciones clonales. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Configuración de los ajustes de electroporación para EGFP KO en células THP-1 y validación de la edición de genes. (A) Las células THP-1 se transducieron con vectores lentivirales que codifican el gen reportero EGFP . Los clones unicelulares se obtuvieron limitando la dilución seguida de una expansión en cultivo durante 30 días. El análisis por citometría de flujo confirmó que prácticamente toda la población celular expresaba GFP. La mediana de la intensidad de fluorescencia (MFI) de la población GFP positiva, indicativa del número de copias de EGFP integrado, varió entre varios clones de células. Se eligió el clon G5 para los estudios de seguimiento. (B) Representación esquemática del gen EGFP , incluyendo los sitios de unión tanto para el sgRNA como para los cebadores. Los sitios de corte de Cas9 se indican mediante líneas discontinuas. (C) Las células THP-1_GFP indiferenciadas (2 x 105) se mezclaron con RNP Cas9:sgRNA preensambladas antes de la electroporación con los ajustes indicados. A continuación, las células se transfirieron en una placa de 24 pocillos que contenía 500 μL de medio RPMI precalentado al 20% que contenía FBS solo o diluido 1:1 con medio acondicionado producido por un cultivo celular THP-1 de 48 h. La proliferación (barras naranjas) y el porcentaje de células GFP positivas (línea verde) se monitorizaron a lo largo del tiempo. Los datos representan la media de n = 2 réplicas biológicas. El marco rojo resalta la condición seleccionada para experimentos posteriores. (D) Se extrajo el ADN genómico de células THP-1_GFP editadas (RNP 3 sgRNA) o no editadas (Cas9), y se amplificó la región que rodea el locus objetivo mediante PCR. Se utilizaron células parentales THP-1 como control negativo. Los amplicones se sometieron a un ciclo de desnaturalización/renaturalización, que condujo a la formación de un heterodúplex. A continuación, se detectaron desajustes mediante el ensayo T7EI. Los fragmentos de ADN se separaron mediante electroforesis en gel de agarosa al 1,2%. (E) Los productos de PCR purificados se secuenciaron por el método Sanger, seguido de un análisis con el algoritmo ICE. Las puntuaciones Indel y KO indican el porcentaje de secuencias no WT en la muestra y la proporción de células con un indel de cambio de marco o de 21+ pb, respectivamente. Haga clic aquí para ver una versión más grande de esta figura.
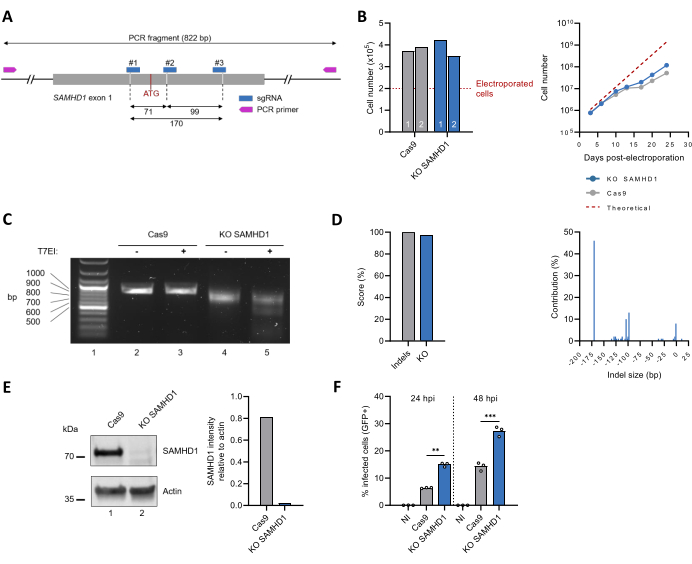
Figura 3: Producción y caracterización de una población de células policlonales SAMHD1 KO THP-1. (A) Representación esquemática del exón 1 de SAMHD1, incluidos los sitios de unión para sgRNA y cebadores. Los sitios de corte Cas9 se indican mediante líneas discontinuas y el codón ATG inicial se muestra en rojo. (B) Tres días después de la electroporación, las células viables (n = 2) se cuantificaron utilizando la prueba de exclusión de azul de tripano (diagrama de la izquierda). El crecimiento celular se monitoreó durante 25 días (diagrama de la derecha). La línea discontinua corresponde al número teórico de células basado en un tiempo de duplicación estimado de 49 h. (C) Se evaluaron los resultados de edición a nivel genómico, como se muestra en la Figura 2D. (D) Los genotipos después de la edición CRISPR se identificaron y cuantificaron mediante análisis de secuencias de Sanger basado en ICE. (E) La proteína contenida en el lisado crudo de las células THP-1 editadas y no editadas se separó por migración en el gel SDS-PAGE y, a continuación, se visualizó mediante inmunotransferencia utilizando anticuerpos contra SAMHD1. Se utilizó actina como control de carga. La intensidad de las bandas de SAMHD1 y actina se cuantificó mediante densitometría con el software ImageJ. (F) Las células THP-1 no editadas (Cas9) y KO SAMHD1 se diferenciaron mediante tratamiento con PMA (300 ng/mL, 24 h) y luego se desafiaron con un virus VIH-1 pseudotipado VSVg que expresaba GFP como reportero. La proporción de células GFP positivas se puntuó mediante citometría de flujo a las 24 h y 48 h después de la infección (hpi). NI: no infectado. **P < 0,01; P < 0,001 mediante la prueba t desapareada con corrección de Welch. Haga clic aquí para ver una versión más grande de esta figura.
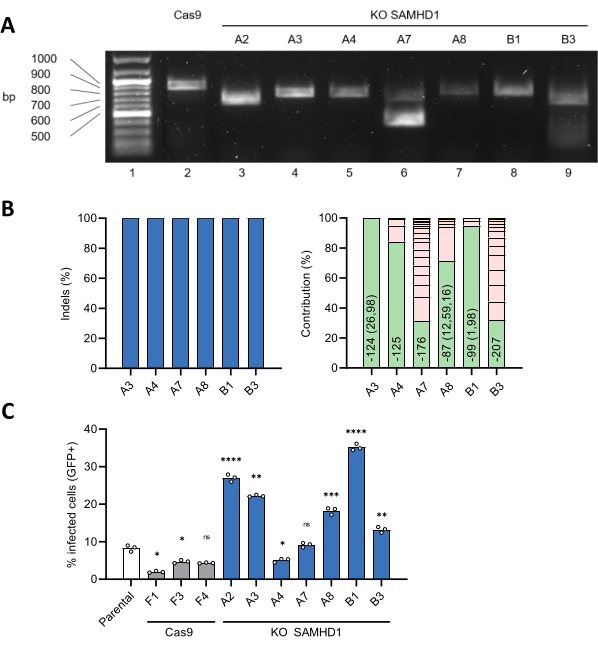
Figura 4: Producción y caracterización de clones unicelulares SAMHD1 KO. (A) El resultado de la edición para clones unicelulares seleccionados de SAMHD1 KO se evaluó mediante el monitoreo de la movilidad de los fragmentos de ADN producidos por la amplificación por PCR de la región que contiene el sitio objetivo mediante electroforesis en gel de agarosa. (B) Los datos de secuenciación de Sanger se analizaron con la herramienta ICE, como se muestra en la Figura 2E. Las barras en el gráfico de la derecha representan la diversidad de indels en cada población celular. La barra verde representa la secuencia dominante y el tamaño total de la eliminación asociada. Los números entre paréntesis indican el tamaño de los espacios no continuos. (C) La permisividad de los clones unicelulares no editados y SAMHD1 KO a la infección por VIH-1 se probó como se muestra en la Figura 3F, y se comparó con la de las células parentales THP-1 no electroporadas o clones de células no editadas (Cas9). La tasa de infección se puntuó en 48 hpi. *P < 0,05; **P < 0,01; P < 0,001; P < 0,0001; ns P≥ 0.05 por el ANOVA de Brown-Forsythe y Welch con la prueba de comparaciones múltiples de Dunnett. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Evaluación del resultado de la edición de clones unicelulares KO mediante la prueba T7EI adaptada. Se purificó el ADN genómico de clones unicelulares KO o células no editadas, y la región que rodea el locus objetivo se amplificó mediante PCR. Los amplicones de cada clon de una sola célula se mezclaron en una proporción de 1:1 con los de las células de control no editadas. Después de la formación de heterodúplex y la digestión de T7EI, el producto se analizó mediante electroforesis en gel de agarosa. Los clones editados producen múltiples fragmentos de digestión (clones A, B, C, D y E), mientras que los no editados muestran una sola banda WT (clones F y G). Haga clic aquí para descargar este archivo.
Figura complementaria 2: Estrategia de activación por citometría de flujo para cuantificar el porcentaje de células THP-1 tratadas con PMA infectadas por VIH-1. Utilizando el área de dispersión directa (FSC-A) contra el área de dispersión lateral (SSC-A), se define una primera puerta para eliminar los desechos y seleccionar las celdas THP-1 tratadas con PMA. A continuación, se seleccionan celdas individuales con un SSC-A frente a un gráfico SSC-H. Por último, las células GFP positivas se detectan con GFP-A frente a FSC-H. Haga clic aquí para descargar este archivo.
Discusión
Aquí, se describe un protocolo para obtener una edición exitosa mediada por CRISPR de la línea celular THP-1. El enfoque se basa en la transferencia de RNPs sgRNA/Cas9 preensamblados por electroporación/nucleofección. Esta estrategia se eligió para limitar los efectos fuera del objetivo que potencialmente surgen de la integración mediada por lentivirales del casete sgRNA/Cas9, lo que produce una expresión persistente de la nucleasa. Se seleccionaron múltiples sgRNAs dirigidos al gen de interés para lograr una edición confiable y eficiente, lo que aumenta la probabilidad de crear indels genómicos, lo que lleva a la pérdida de la expresión de proteínas y KO32 funcional. Se prestó especial atención a los ajustes de electroporación para lograr una permeabilización óptima de la membrana celular, lo que se requería para garantizar la entrega eficiente de los RNP de sgRNA/Cas9 y evitar la muerte celular excesiva33. En este sentido, un parámetro crucial es el vigor del cultivo celular antes de la electroporación (es decir, un número de paso bajo).
La edición mediada por CRISPR-Cas9 se validó a nivel genético con el ensayo de detección de desajustes T7EI, que proporciona una evaluación rentable y rápida de múltiples muestras para la preselección antes de la secuenciación de Sanger. Este método se desarrolló inicialmente para estimar la eficiencia de la edición utilizando un solo sgRNA comparando la intensidad de los amplicones de longitud completa y los productos de escisión34,35. Cabe destacar que los grandes indels (>70 pb) producidos cuando se utilizan múltiples sgRNA se pueden detectar comparando la movilidad de los fragmentos de ADN obtenidos por amplificación por PCR de los loci dirigidos de células editadas frente a no editadas mediante electroforesis en gel estándar. Sin embargo, el ensayo T7EI podría ser útil para determinar la presencia de secuencias que albergan indels pequeños (>1bp) en una población de células policlonales o, utilizando el protocolo adaptado, en una población de células monoclonales.
La presencia de mutaciones fuera de marco en el objetivo, que interrumpen la secuencia codificante, se confirmó mediante un enfoque sencillo, en el que se extrae el genoma de toda la población celular y se utiliza como matriz para la amplificación de PCR. A continuación, los amplicones purificados se someten a una secuenciación Sanger a granel, seguida de un análisis bioinformático con la herramienta ICE. En general, este método supera la necesidad de subclonar los productos de PCR en un plásmido y transformar los productos de ligadura en bacterias para aislar colonias unicelulares, que se secuencian individualmente.
Una vez validada la inactivación del locus objetivo, se requiere una caracterización exhaustiva de la línea celular editada para atribuir con confianza un fenotipo determinado a la falta de la proteína de interés y descartar efectos imprevistos derivados de cambios fuera del objetivo, pero también dentro del objetivo que podrían afectar a la estructura y/o función del ARNm.
La ausencia de expresión de proteínas debe confirmarse mediante inmunotransferencia y/o inmunofluorescencia utilizando posiblemente dos anticuerpos que reconozcan epítopos diferentes. De hecho, la introducción de una PTC en un exón temprano cerca del codón ATG canónico se asocia con el riesgo de eventos ITL, que producen variantes de proteínas truncadas N-terminales36. Cabe destacar que Tuladhar et al. informaron de la detección de especies de proteínas aberrantes en una proporción significativa (~50%) de células editadas con CRISPR de origen comercial o interno18,19. La aparición de nuevos productos proteicos se atribuyó a diversos procesos potencialmente específicos del tipo de célula, como la omisión del exón que contiene indel o la aparición de sitios de entrada de ribosoma internos (IRES), lo que provoca el inicio de la síntesis de proteínas en la alternativa TIS aguas abajo del codón ATG canónico. También hay indicios de que el ITL se combina con una fuga de NMD bajo ciertas circunstancias37,38. De manera similar, las estrategias de edición del genoma dirigidas a un exón tardío podrían no desencadenar la NMD del ARNm mutado que subyace a la ablación ineficaz de la expresión génica20. Por último, si Cas9 es guiado por múltiples sgRNAs, podrían surgir reordenamientos cromosómicos complejos, incluyendo inversiones o grandes deleciones de fragmentos de ADN39, que podrían alterar la estructura y la expresión del genoma, particularmente cuando se dirigen elementos reguladores40.
Para mitigar aún más el impacto de los resultados no deseados de la edición del genoma, se necesitan estudios funcionales para confirmar la relación entre la pérdida de expresión de proteínas y el fenotipo resultante (es decir, la actividad antiviral). Si se subclonan células editadas, se debe comparar el comportamiento de múltiples clones entre sí y con la población policlonal. La realización de experimentos de rescate, que consisten en restaurar la expresión génica para revertir el fenotipo de las células editadas a un fenotipo WT, también fortalecerá la especificidad y excluirá posibles eventos fuera del objetivo.
Aunque este protocolo fue concebido para la edición de células THP-1, proporciona una estructura general para el flujo de trabajo CRISPR-Cas9 KO que se puede adaptar a otras líneas celulares. Es posible que sea necesario ajustar los parámetros de electroporación, y si se aplica un voltaje superior a 1800 V, se debe usar un tampón de resuspensión T, de acuerdo con las instrucciones del fabricante. Otra variable a considerar es la relación de concentración entre Cas9 y sgRNA durante el ensamblaje de RNP. Aunque la proporción 1:9 funcionó bien en este caso, modificarla puede mejorar la eficiencia de la edición en otras circunstancias. Finalmente, aunque no es beneficioso aquí, se debe probar el uso de medios acondicionados para la recuperación celular después de la electroporación, ya que el efecto podría ser diferente con diferentes líneas celulares.
Divulgaciones
Todos los autores no tienen conflictos de intereses.
Agradecimientos
Agradecemos a JP Concordet (MNHN, U1154/UMR7196, París), G. Bossis (IGMM, Montpellier) y D. Schlüter (Facultad de Medicina de Hannover, Alemania) por compartir los protocolos y por la discusión. Este proyecto ha recibido financiación del programa de investigación e innovación Horizonte 2020 de la Unión Europea (acuerdo de subvención nº 101017572 a AZ) y ANRS (subvención ECTZ162721 a AZ). La infraestructura de investigación del Modelo de Enfermedades Infecciosas y Terapias Innovadoras (IDMIT) está respaldada por el "programme investissement d'avenir (PIA)" bajo el ANR_11_INSB_0008 de referencia.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
Referencias
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados