Method Article
Inactivation du gène CRISPR-Cas9 médiée par électroporation dans les cellules THP-1 et isolement de clones unicellulaires
Dans cet article
Résumé
La lignée cellulaire THP-1 est largement utilisée comme modèle pour étudier les fonctions des monocytes/macrophages humains dans divers domaines de recherche liés à la biologie. Cet article décrit un protocole pour une ingénierie efficace basée sur CRISPR-Cas9 et l’isolement de clones unicellulaires, permettant la production de données phénotypiques robustes et reproductibles.
Résumé
La lignée cellulaire THP-1 de la leucémie monocytaire aiguë humaine (LMA) est largement utilisée comme modèle pour étudier les fonctions des macrophages dérivés des monocytes humains, y compris leur interaction avec des agents pathogènes humains importants tels que le virus de l’immunodéficience humaine (VIH). Par rapport à d’autres lignées cellulaires immortalisées d’origine myéloïde, les cellules THP-1 conservent de nombreuses voies de signalisation inflammatoires intactes et présentent des caractéristiques phénotypiques qui ressemblent davantage à celles des monocytes primaires, y compris la capacité de se différencier en macrophages lorsqu’elles sont traitées avec du phorbol-12-myristate 13-acétate (PMA). L’utilisation de la technologie CRISPR-Cas9 pour concevoir des cellules THP-1 par inactivation génique ciblée (KO) offre une approche puissante pour mieux caractériser les mécanismes immunitaires, y compris les interactions virus-hôte. Cet article décrit un protocole d’ingénierie efficace basé sur CRISPR-Cas9 utilisant l’électroporation pour délivrer des ribonucléoprotéines Cas9 :sgRNA pré-assemblées dans le noyau cellulaire. L’utilisation de plusieurs ARNsg ciblant le même locus à des positions légèrement différentes entraîne la délétion de grands fragments d’ADN, augmentant ainsi l’efficacité de l’édition, comme évalué par le test de l’endonucléase I T7. L’édition médiée par CRISPR-Cas9 au niveau génétique a été validée par le séquençage de Sanger suivi de l’analyse ICE (Inference of CRISPR Edits). La déplétion protéique a été confirmée par immunoblot couplé à un test fonctionnel. En utilisant ce protocole, jusqu’à 100 % d’indels dans le locus ciblé et une diminution de plus de 95 % de l’expression des protéines ont été obtenues. L’efficacité d’édition élevée permet d’isoler facilement les clones unicellulaires en limitant la dilution.
Introduction
THP-1 est une lignée cellulaire dérivée de monocytes humains isolée d’un patient souffrant de leucémie aiguë (LMA), qui présente des caractéristiques phénotypiques ressemblant étroitement à celles des monocytes primaires1. Par rapport aux macrophages primaires dérivés de monocytes, qui ne se divisent pas et présentent à la fois une durée de vie limitée et une variabilité inter-/intra-donneur dans le phénotype, les cellules THP-1 peuvent être cultivées pratiquement indéfiniment et ont un comportement plus homogène qui favorise la reproductibilité des résultats 2,3,4,5,6 . Notamment, les cellules THP-1 peuvent être différenciées vers un phénotype de type macrophage avec le phorbol-12-myristate 13-acétate (PMA), ce qui en fait un modèle in vitro largement utilisé pour étudier les réponses des monocytes/macrophages aux signaux inflammatoires 7,8,9,10,11,12,13 ou à l’infection par des agents pathogènes humains cliniquement pertinents, y compris le VIH 14,15,16. La possibilité de modifier génétiquement les cellules THP-1 présente un intérêt dans de nombreux domaines de recherche liés à la biologie.
La protéine associée à CRISPR 9 (CRISPR-Cas9) est un système immunitaire adaptatif procaryote qui s’appuie sur une nucléase guidée par l’ARN pour dégrader les génomes viraux envahisseurs, qui a été reprogrammé comme un outil de génie génétique17. Le processus d’édition du génome se déroule en trois étapes : la reconnaissance, le clivage et la réparation. Un ARN à guide unique (ARNsg) recrute la nucléase Cas9 dans un locus génomique spécifique par appariement de bases avec sa séquence guide de 20 pb. La présence d’une séquence PAM (Protospacer Adjacent Motif) directement 3' de la séquence cible génomique de 20 pb déclenche le déroulement et le clivage médiés par Cas9 sur les deux brins d’ADN entre les positions 17 et 18 (3-bp 5' du PAM). La rupture double brin (DSB) résultante est traitée par deux voies de réparation principales. En l’absence d’un modèle de réparation présentant une homologie avec le locus endommagé, la voie NHEJ (Non-Homologous End Joindre), sujette aux erreurs, introduira des insertions et/ou des délétions aléatoires de nucléotides (indels), conduisant potentiellement à des mutations de décalage de cadre et/ou à l’introduction de codons de terminaison prématurée (PTC). À leur tour, les ARNm contenant du PTC sont ciblés par la dégradation par la voie de désintégration de l’ARNm médiée par le non-sens (NMD), perturbant finalement l’expression/la fonction des protéines 18,19,20. Alternativement, le chemin HDR (Homology-Directed Repair) dépendant du modèle peut fonctionner et réparer fidèlement le DSB. Ce mécanisme a été exploité pour réaliser une édition génétique précise, y compris des knock-ins et des substitutions de bases. Il convient de noter que l’état du cycle cellulaire est un facteur important influençant le choix de la voie de réparation des CDB. En effet, NHEJ est actif à tous les stades du cycle cellulaire, tandis que le HDR est principalement limité aux phases S/G221.
Les cellules THP-1 se développent en suspension et sont notoirement difficiles à transfecter avec l’ADN plasmidique, une procédure qui peut également modifier leur viabilité et/ou leur capacité de différenciation22,23. La transduction avec des vecteurs lentiviraux basés sur le VIH-1 codant à la fois pour Cas9 et l’ARNsg est souvent utilisée pour inactiver (KO) un gène d’intérêt24. L’intégration de la cassette Cas9/sgRNA dans le génome cellulaire garantit une expression prolongée et une KO efficace, mais constitue également une source persistante d’effets hors cible25. Alternativement, les ribonucléoprotéines (RNP) Cas9 :sgRNA pré-assemblées sont délivrées par électroporation, une méthode impliquant la formation temporaire de pores dans les membranes plasmique et nucléaire lors de l’application d’impulsions électriques. La préservation de la viabilité cellulaire est un défi important lorsque l’on entreprend cette approche.
Ici, une lignée cellulaire THP-1 exprimant de manière stable la GFP (THP-1_GFP) a été produite pour servir d’outil à l’établissement d’un protocole permettant d’obtenir une édition efficace basée sur CRISPR-Cas9. Après avoir conçu une stratégie pour inactiver le gène EGFP à l’aide de trois ARNsg simultanément (approche multi-guides), l’efficacité de KO dans plusieurs conditions d’électroporation a été déterminée en utilisant l’expression de la GFP comme lecture. La prolifération cellulaire a été surveillée en parallèle. L’édition génomique a été confirmée à la fois par un test d’endonucléase I T7 (T7EI) et un séquençage de Sanger, suivis d’une analyse avec l’algorithme ICE (Inference of CRISPR Edits)26. Des paramètres qui ont permis d’obtenir une diminution de l’expression de la GFP allant jusqu’à 95 %, les cellules THP-1 retrouvant des taux de croissance normaux après l’électroporation, ont été utilisés avec succès pour inactiver un gène endogène (SAMHD1) et produire des clones de THP-1 sur cellule unique.
Protocole
Les détails des réactifs et de l’équipement utilisés dans cette étude sont énumérés dans la table des matériaux.
1. Conception du guide avec CRISPOR (Figure 1.1)
REMARQUE : Le logiciel SnapGene Viewer peut être utilisé aux étapes 4, 7 et 10 pour annoter le site cible d’édition et l’emplacement de l’hybridation de l’amorce PCR dans le gène d’intérêt.
- Rendez-vous sur le site d’Ensembl (www.ensembl.org). Dans la zone de recherche , sélectionnez une espèce et entrez le nom du gène d’intérêt. Cliquez sur Go. Sélectionnez le résultat qui correspond au gène (pas un transcrit).
- Cliquez sur Afficher le tableau des transcriptions , puis sélectionnez l’ID de transcription qui correspond au codage des protéines étiqueté en or dans la colonne Biotype . Une fois sur la page de transcription, cliquez sur Exons dans le menu de gauche.
- Faites défiler vers le bas et cliquez sur Télécharger la séquence. Assurez-vous que le format de fichier est FASTA. Dans Paramètres - Séquences incluses, désélectionnez tout sauf Séquence génomique.
Entrez le nombre « 500 » dans la séquence latérale à l’une ou l’autre des extrémités de la boîte de transcription . Cliquez sur Aperçu, sélectionnez la séquence entière (uniquement les nucléotides sans l’en-tête) et copiez-la (Ctrl+C). - Ouvrez SnapGene Viewer et cliquez sur Nouveau fichier > ADN. Collez la séquence (Ctrl+V) dans la zone Créer une séquence . Décochez la case Détecter les fonctionnalités communes et sélectionnez Linéaire dans la topologie.
Renommez le fichier et cliquez sur Créer. Dans le menu de gauche, désélectionnez Afficher les enzymes (première icône). En bas de la fenêtre, sélectionnez l’onglet Séquence .
REMARQUE : Cette étape permet de récupérer la séquence complète du gène, y compris les exons, les introns et les séquences flanquantes de 500 pb (facultatif). Cette dernière information est utile pour concevoir des amorces PCR pour l’amplification d’un site cible situé dans le premier exon. - De retour sur le site Web d’Ensembl, faites défiler vers le haut jusqu’à l’aperçu du fichier et cliquez sur Retour. Maintenant, changez le format de fichier en RTF. Dans Paramètres - Séquences incluses, désélectionnez tout sauf Exons. Dans Afficher les variantes, sélectionnez Non. Cliquez sur Télécharger en haut de la page.
- Ouvrez le fichier téléchargé (avec Word), qui contient la séquence des exons et affiche la séquence de codage en bleu, en commençant par l’ATG initial. Choisissez l’exon à cibler par l’édition dirigée par CRISPR-Cas9 (voir ci-dessous pour les recommandations), sélectionnez-le et copiez-le (Ctrl+C).
- L’exon ciblé est soit un exon précoce, soit un exon codant pour un domaine fonctionnellement important de la protéine. Il convient de noter que l’établissement d’une PTC dans un exon tardif proche de l’UTR 3' ne provoquera probablement pas de NMD, conduisant à l’expression d’une protéine tronquée C-terminale. À l’inverse, l’introduction d’un PTC dans un exon précoce proximal au site d’initiation natif est associée à un risque de traduction illégitime (ITL, alias initiation de traduction alternative (ATI)), produisant l’expression inattendue d’une protéine tronquée N-terminale qui commence à un site d’initiation de traduction (TIS) en aval du premier codon ATG. Pour atténuer ce dernier risque, il est conseillé d’évaluer l’occurrence d’autres TIS à l’aide d’ATGpr27 (atgpr.dbcls.jp) et/ou de NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/).
- Assurez-vous que l’exon cible contient une séquence codante. Cependant, il peut s’avérer utile de choisir un recuit d’ARNsg avec la région 5'UTR en amont de l’ATG initial pour l’inclure dans le fragment supprimé.
- Idéalement, l’exon ciblé devrait être commun à toutes les variantes de transcription codant pour les protéines du gène. Vérifiez si c’est le cas sur le Genome Data Viewer (www.ncbi.nlm.nih.gov/gdv/) du NCBI en recherchant le gène d’intérêt.
En cliquant sur le nom du gène (en vert) dans la fenêtre d’affichage, les variantes de transcription (en violet) s’afficheront. Les exons sont représentés par un rectangle.
- Dans SnapGene Viewer, appuyez sur Ctrl+F, Ctrl+V, puis sur Entrée pour rechercher la séquence de l’exon. Appuyez sur Ctrl+T pour ajouter une nouvelle fonctionnalité, nommez-la, changez le type en « exon » et cliquez sur OK.
- Allez sur le site Web de CRISPOR (http://crispor.gi.ucsc.edu/) et collez la séquence d’exons à l’étape 1. Tout d’abord, sélectionnez un génome de référence à l’étape 2 , puis le type de PAM à cibler à l’étape 3, généralement 20bp-NGG pour SpCas9. Cliquez sur SOUMETTRE.
- Sélectionnez deux ARNsg de manière à ce qu’ils soient séparés jusqu’à 150 pb, puis sélectionnez un troisième ARNsg entre les deux. Voici quelques directives pour la sélection de l’ARNsg :
- Le score de spécificité du MIT est lié aux effets hors cible. Un score plus élevé indique qu’il y a moins de hors-cibles potentiels. La colonne de droite affiche trois sites hors cible prédits, classés du plus probable au moins probable, ainsi que leur emplacement (dans un exon, un intron ou une région intergénique). La liste complète des sites hors cible prévus est accessible en cliquant sur Afficher tout. Si possible, sélectionnez des ARNsg avec un score MIT >80, en donnant la priorité à ceux qui n’ont pas de cibles externes pour 0, 1 ou 2 mésappariements. De plus, les ARNsg avec des cibles secondaires dans un exon, qui ont le plus grand potentiel d’affecter le phénotype, doivent être évités.
- Pour l’efficacité prévue, reportez-vous au score Doench '16. Notez qu’un score élevé de Doench '16 indique simplement que l’ARNsg est plus susceptible d’être efficace. L’efficacité réelle doit être déterminée expérimentalement. Pour cette raison, il est toujours utile de sélectionner plusieurs ARNsg, même s’ils ne sont pas destinés à être utilisés ensemble.
- Une teneur en GC trop élevée ou trop faible, ainsi que certains motifs, peuvent nuire à l’efficacité de l’ARNsg et doivent être évités. Ces paramètres sont mis en évidence par CRISPOR.
- Répétez l’étape 1.7 pour ajouter la séquence d’ARNsg et la séquence PAM associée à la séquence de gène dans SnapGene Viewer. En même temps, collez la séquence d’ARNsg (sans le PAM) dans un fichier texte ou Excel pour conserver l’orientation 5'-3' requise lors de la commande de l’oligonucléotide.
- Pour l’amplification du site cible par PCR, concevez quelques amorces autour de celui-ci. La taille de l’amplicon doit être comprise entre 800 et 1000 pb. PrimerQuest a été utilisé pour concevoir les amorces de ce protocole (https://eu.idtdna.com/pages/tools/primerquest). Alternativement, CRISPOR fournit une liste d’amorces pour amplifier la région génomique ciblée ainsi que les sites hors cible. Après avoir affiché la liste complète des sites hors cible potentiels (étape 1.9.1), cliquez sur Amorces hors cible dans le coin inférieur droit.
2. Préparation du réactif et de la cellule pour l’électroporation (Figure 1.2)
- Préparez une plaque de culture à 24 puits pour récupérer les cellules après électroporation en remplissant les puits avec 500 μL de milieu RPMI 1640 complété par 20 % de sérum fœtal bovin (FBS) inactivé à la chaleur (56 °C, 30 min), filtré (0,20 μm). N’ajoutez pas d’antibiotiques. Maintenir la plaque dans un incubateur humidifié à 37 °C et 5 % de CO2 pendant 24 h jusqu’à l’électroporation.
- Si les ARNsg sont expédiés secs, réhydratez-les avec un tampon TE (10 mM Tris, 1 mM EDTA, pH 8,0) jusqu’à une concentration finale de 100 μM (c’est-à-dire 10 μL de tampon TE pour 1 nmol d’ARNsg). Agiter pendant 30 s, incuber à 4 °C pendant la nuit pour permettre une réhydratation complète et, après une brève homogénéisation à la pipette, conserver la solution mère d’ARNsg à -20 °C. Selon le volume final, faites des aliquotes pour éviter plusieurs cycles de gel-dégel.
- Préparez une solution fonctionnelle d’ARNsg à une concentration finale de 30 μM en diluant la solution mère de 100 μM dans de l’eau exempte de nucléases. Agiter pendant 30 s et incuber 5 min à température ambiante.
- Assemblez les RNP Cas9 :sgRNA à un rapport molaire de 1:9 en diluant simultanément 1 μL de chacun des trois sgRNA de 30 μM et 0,5 μL de solutions Cas9 de 20 μM dans 3,5 μL de tampon de résuspension R, inclus dans le kit d’électroporation (volume final de 7 μL, pour une condition expérimentale ; mise à l’échelle en conséquence). Agitez brièvement et laissez incuber pendant 5 minutes à température ambiante.
- Entre-temps, préparez un contrôle non édité en ajoutant 0,5 μL de 20 μM de Cas9 à 6,5 μL de tampon de remise en suspension R. Vortex brièvement et incubez pendant 5 min à température ambiante.
- Ajouter 5 μL de tampon de remise en suspension R à tous les échantillons pour un volume final de 12 μL par condition d’électroporation.
- Préparez les cellules THP-1 pour l’électroporation.
- Évaluer la concentration et la viabilité par le test d’exclusion au bleu de trypan. Diluez les cellules 1:2 dans une solution de coloration au bleu de trypan à 0,4 %. Après 30 s d’incubation, bien homogénéiser et ajouter 10 μL dans une paroi d’une chambre de comptage avec un style de grille amélioré par Neubauer. Comptez trois grands carrés et divisez le nombre par 100 pour obtenir la concentration cellulaire (x10,6 cellules/ml).
REMARQUE : La santé des cellules influence leur sensibilité à l’électroporation. Des précautions doivent être prises pour réaliser la culture cellulaire dans des conditions optimales. Le temps passé à l’extérieur de l’incubateur doit être limité, et tous les réactifs et solutions doivent être préparés et réchauffés à l’avance. - Pour chaque condition, prélever un volume équivalent à 0,2 x 106 cellules et centrifuger (336 x g, 5 min, 20 °C).
- Aspirer le surnageant à l’aide d’une pipette et remettre la pastille en suspension dans 500 μL de PBS. Centrifuger à nouveau (336 x g, 5 min, 20 °C).
- Aspirez soigneusement le surnageant à l’aide d’une pipette et mettez en suspension la pastille de cellule THP-1 avec les 12 μL de solution RNP (étape 2.6).
- Évaluer la concentration et la viabilité par le test d’exclusion au bleu de trypan. Diluez les cellules 1:2 dans une solution de coloration au bleu de trypan à 0,4 %. Après 30 s d’incubation, bien homogénéiser et ajouter 10 μL dans une paroi d’une chambre de comptage avec un style de grille amélioré par Neubauer. Comptez trois grands carrés et divisez le nombre par 100 pour obtenir la concentration cellulaire (x10,6 cellules/ml).
3. Configuration du système d’électroporation et nucléofection (Figure 1.3)
- Placez la station de pipette sous une armoire de biosécurité, placez un tube d’électroporation dans le support et ajoutez 3 ml de tampon E, inclus dans le kit d’électroporation.
- Après avoir allumé l’appareil d’électroporation, utilisez l’écran tactile pour régler les paramètres d’électroporation suivants : Tension = 1 500 V, Durée = 10 ms, Nombre = 3.
- Équiper la pipette d’électroporation d’une pointe et aspirer 10 μL de la solution RNP/THP-1 (étape 2.7.4). Insérez la pipette dans le tube d’électroporation et appuyez sur Start sur l’écran de l’appareil d’électroporation. Attendez que le message Terminé s’affiche et retirez la pipette du tube.
- Transférez les cellules dans un puits de la plaque préchauffée à 24 puits et homogénéisez doucement. Remettez la plaque dans l’incubateur humidifié et laissez-les reposer sans être dérangés pendant 72 h.
REMARQUE : Veillez à ne pas faire de bulles lors du pipetage de la suspension RNP/THP-1, car elles interféreraient avec la procédure d’électroporation. Si un message d’erreur s’affiche en l’absence d’un arc électrique visible, retirez la pipette d’électroporation du tube et appuyez à nouveau sur Démarrer . Cependant, si un arc électrique sous la forme d’une brève étincelle brillante est observé, cela pourrait indiquer la présence de bulles. La procédure d’électroporation échouera probablement, même en l’absence d’un message d’erreur.
4. Récupération du THP-1 72 h après l’électroporation (Figure 1.4)
- Compter les cellules pour évaluer la concentration (étape 2.7.1) 72 h après l’électroporation.
- S’il y a suffisamment de cellules (c.-à-d. ≥0,6 x 106 cellules/mL), diluez-les au moins un facteur 2 avec du RPMI complété par 20 % de FBS et 1 % de pénicilline-streptomycine et portez la concentration entre 0,3 et 0,5 x 106 cellules par mL. Sinon, laissez la cellule récupérer pendant 72 heures supplémentaires.
- Passez et amplifiez les cellules jusqu’à ce qu’il y en ait assez pour la validation KO. Entre-temps, l’isolement de clones unicellulaires peut être initié (étape 7).
5. Validation de l’édition génomique par test de mésappariement T7EI (Figure 1.5)
REMARQUE : Le test peut sous-estimer l’efficacité de l’édition étant donné que T7EI reconnaît les discordances supérieures à 1 pb. Par conséquent, le test T7EI n’est pas utile pour le criblage de populations de cellules homozygotes (c.-à-d. des clones unicellulaires), à moins qu’il ne soit modifié de manière appropriée (étape 5.7).
- Évaluer la concentration cellulaire (étape 2.7.1) et prélever un volume équivalent à 0,1 x 106 cellules dans un tube de 1,5 mL. Centrifuger (336 x g, 5 min, 20 °C), aspirer le surnageant et remettre la pastille en suspension dans 500 μL de PBS. Centrifugez à nouveau et, à l’aide d’une pipette, aspirez autant de surnageant que possible sans perturber la pastille. Congelez l’échantillon et conservez-le à -20 °C.
- Extraire l’ADN génomique pour servir de matrice pour l’amplification par PCR.
- Remettez la pastille en suspension avec 50 μL de solution d’extraction d’ADN, homogénéisez et transférez tout le volume dans un tube PCR de 0,2 mL. Vortex et centrifugeuse brièvement (impulsion pendant 3 s).
- Placez le tube dans un thermocycleur et chauffez-le à 65 °C pendant 15 min, puis à 98 °C pendant 10 min.
- Diluez l’ADN extrait avec 90 μL d’eau ultrapure. Vortex et centrifugeuse brièvement (5 000 x g, 3 s).
- Diluer l’amorce de PCR (voir le tableau des matériaux) dans de l’eau ultrapure pour une concentration finale de 10 μM (c’est-à-dire 10 pmol/μL).
- Préparez le mélange PCR (en suivant la REMARQUE ci-dessous) dans un tube PCR de 0,2 mL (volume final = 50 μL, pour une condition). Habituellement, il y aura au moins deux conditions : le KO et le contrôle non édité avec Cas9 uniquement.
REMARQUE : ADN génomique purifié : 10 μL ; Amorce directe et inverse (10 μM) : 2,5 μL chacune, la concentration finale étant de 500 nM ; Tampon de réaction (5x) : 10 μL. Bien vortex avant d’ajouter. Mélanger le dNTP (25 mM de chaque) : 0,6 μL, concentration finale de 0,3 mM pour chaque dNTP. ADN polymérase : 0,5 μL ; Eau ultrapure : 23,9 μL. Vortex et centrifugeuse brièvement (impulsion pendant 3 s). - Placez les tubes dans le thermocycleur et exécutez le programme PCR avec les paramètres suivants :
REMARQUE : Un cycle à 95 °C pendant 5 min, suivi de 30 cycles [98 °C pendant 20 s (étape de dénaturation), X °C pendant 15 s (étape de recuit), 72 °C pendant 45 s (étape d’allongement)], puis un dernier cycle à 72 °C pendant 2 min. À la fin de l’amplification, retirez brièvement les tubes, le vortex et la centrifugeuse (impulsion pendant 3 s). La température de recuit (X) est la température de fusion (Tm) des amorces moins 5 °C. - Pour une population polyclonale modifiée : dans un nouveau tube de 0,2 mL, ajouter 17,5 μL de l’amplicon PCR et 2 μL de NEBuffer 2 (10x) pour un volume final de 19,5 μL. Vortex et centrifugeuse brièvement (impulsion pendant 3 s). Alternativement, pour cribler des clones unicellulaires, mélanger 1:1 des produits PCR à partir des cellules de contrôle modifiées et non modifiées (étape 5.6) (figure supplémentaire 1).
- Pour la formation hétéroduplex, placez les tubes dans le thermocycleur et exécutez le programme suivant : un cycle à 95 °C pendant 10 min, un avec une rampe de -2 °C/s de 95 à 85 °C, un avec une rampe de -0,3 °C/s de 85 à 25 °C et un dernier cycle de refroidissement à 10 °C en HOLD.
- Après la formation de l’hétéroduplex, ajoutez 0,5 μL de solution T7EI dans le tube. Incuber à 37 °C pendant 30 min.
- Préparez un gel d’agarose à 1,2 % :
- Pesez l’agarose dans une bouteille en verre et ajoutez le volume approprié de 1 tampon TAE (40 mM de tris-acétate, 1 mM d’EDTA, pH 8,3). Placez-le au micro-ondes en faisant attention à ne pas visser fermement le bouchon. Chauffer plusieurs fois jusqu’à ce que les cristaux d’agarose soient complètement dissous.
REMARQUE : Si nécessaire, mélangez la solution. Ne le laissez pas bouillir, sinon le volume diminuera, modifiant la concentration d’agarose. - Ajouter la coloration gel ADN diluée à 1/20 000 et bien mélanger.
- Versez la solution d’agarose dans un moule, ajoutez un peigne et laissez se solidifier à température ambiante.
- Pesez l’agarose dans une bouteille en verre et ajoutez le volume approprié de 1 tampon TAE (40 mM de tris-acétate, 1 mM d’EDTA, pH 8,3). Placez-le au micro-ondes en faisant attention à ne pas visser fermement le bouchon. Chauffer plusieurs fois jusqu’à ce que les cristaux d’agarose soient complètement dissous.
- Préparez les échantillons pour l’électrophorèse sur gel en mélangeant 5 μL des produits de digestion T7EI (étape 5.9) ou le produit PCR non digéré avec 5 μL d’eau et 2 μL de colorant de charge d’ADN 6x. Chargez les échantillons et dimensionnez l’échelle et migrez à 80 V pendant 45 min (le temps peut être adapté en fonction de la taille attendue des fragments d’ADN). Une fois la migration terminée, acquérez une image du gel avec un système d’imagerie approprié.
6. Validation de l’édition génomique par analyse de séquençage de Sanger (Figure 1.6)
- Pour caractériser la modification génétique, purifier les produits de PCR (étape 5.4) et, après séquençage de Sanger, analyser les résultats avec l’outil ICE (https://www.synthego.com/products/bioinformatics/crispr-analysis).
- Contrôler si une protéine peut être produite malgré les modifications.
- Téléchargez les résultats de l’ICE et ouvrez le fichier contribs.txt.
- Comparez les séquences éditées avec l’homologue WT. Cartographiez les indels et vérifiez qu’ils sont apparus dans la région génomique ciblée. Évaluez leur taille. Si la longueur indel n’est pas un multiple de trois, un décalage d’image se produira et un PTC sera éventuellement introduit. L’ARNm muté subira probablement une dégradation médiée par NMD.
7. Isolement d’un clone unicellulaire par dilution limitée (Figure 1.7)
REMARQUE : L’isolement des clones unicellulaires n’est pas obligatoire. Cependant, si vous choisissez de le faire, il est important de caractériser plusieurs clones et de comparer leur phénotype avec la population polyclonale d’origine.
- Prélever un échantillon de suspension cellulaire, le diluer 1/2 avec un milieu de culture et évaluer la concentration (étape 2.7.1). La dilution augmente la précision du comptage.
- Effectuer 2 à 3 étapes de dilution en série pour atteindre une concentration de 7 cellules/mL. Distribuer 100 μL de suspension cellulaire par puits dans une plaque à fond rond de 96 puits (c.-à-d. 0,7 cellule par puits). Laissez la cellule décanter pendant quelques heures avant d’observer les plaques au microscope pour identifier les puits contenant une cellule.
- Surveillez régulièrement la formation et la croissance des colonies et transférez les cellules dans une plaque ou un flacon de culture plus grand au besoin.
8. Caractérisation fonctionnelle des cellules THP-1 KO SAMHD1 par test de restriction du VIH-1
- Semez le THP-1 dans une plaque de culture à 24 puits avec 0,25 x 106 cellules par puits dans 300 μL de RPMI complété par 10 % de FBS, 1 % de pénicilline-streptomycine (RPMI complet) et contenant 300 ng/mL de PMA pour la différenciation. Conservez la plaque dans un incubateur humidifié (37 °C, 5 % CO2) pendant 24 h.
- Remplacez le milieu par un milieu RPMI complet exempt de PMA et remettez la plaque dans l’incubateur pendant encore 24 heures.
- À l’aide d’une pompe à vide, aspirez tout le milieu de culture. Ajouter 250 μL de solution virale contenant du VIH-1-GFP pseudotypé VSVg (MOI = 0,5 IFU/cellule). Incluez un témoin non infecté. Placez la plaque à 4 °C pendant 2 h pour synchroniser l’infection.
- Lavez les cellules une fois avec du RPMI. Ajouter 500 μL de RPMI complet dans chaque puits et incuber pendant 48 h dans des conditions standard (le temps peut être ajusté).
- Après avoir retiré le milieu de culture, lavez une fois les cellules avec du PBS froid. Ensuite, ajoutez 100 μL de trypsine-EDTA à 0,05 % dans chaque puits. Placez la plaque dans un incubateur à 37 °C jusqu’à ce que les cellules soient complètement détachées (5 min devraient suffire) avant d’ajouter 200 μL de RPMI complet pour inactiver l’enzyme. Transvaser 250 μL de chaque puits dans une plaque à fond rond de 96 puits (s’assurer que le cytomètre en flux accepte les plaques de culture).
- Centrifuger la plaque (757 x g, 3 min, décélération = 6, 20 °C) et aspirer le surnageant à l’aide d’une pipette multicanaux. Remettre la pastille en suspension avec 100 μL de PFA à 4 % et incuber à 4 °C pendant 10 min. Ajouter 100 μL de PBS froid.
- Analyser le taux d’infection, représenté par les cellules GFP positives, par cytométrie en flux (voir la figure supplémentaire 2 pour une stratégie de contrôle suggérée).
Résultats
Une lignée cellulaire THP-1 a été générée exprimant de manière stable la protéine rapporteure GFP (THP-1_GFP) (Figure 2A) et utilisée comme outil pour établir un protocole pour une édition efficace du gène médiée par CRISPR-Cas9. Dans ce but, 3 sgRNA ciblant le gène EGFP ont été conçus avec l’outil webCRISPOR 29 (Figure 2B), qui ont été simultanément complexés avec Cas9 à un rapport molaire de 9:1 pour former des RNP avant d’être livrés dans les cellules par électroporation en utilisant différents réglages. Ensuite, les cellules ont été cultivées dans un milieu RPMI contenant 20 % de FBS seul (figure 2C, panneau supérieur) ou diluées 1:1 avec un milieu conditionné (figure 2C, panneau inférieur). La prolifération cellulaire et l’expression de la GFP, en tant que lecture de l’efficacité de l’EGFP KO, ont été surveillées au fil du temps. Pour plusieurs affections, le pourcentage de cellules positives à la GFP a fortement diminué, atteignant une réduction de >90 % au jour 7 après l’électroporation (pe). Au jour 3 pe, le nombre de cellules avait diminué de moitié, probablement en raison de l’impact négatif de l’électroporation sur la viabilité des cellules. Cependant, le nombre de cellules a de nouveau augmenté et le temps de doublement est revenu à un taux normal au jour 7 (figure 2C, panneau supérieur). L’utilisation de milieux conditionnés n’a pas favorisé la récupération cellulaire dans nos conditions expérimentales (Figure 2C, panneau inférieur). Sur la base de ces résultats, trois impulsions de 10 ms de 1500 V ont été choisies pour les expériences de suivi.
L’EGFP KO médié par CRISPR-Cas9 a ensuite été caractérisé au niveau génomique. Pour cela, l’ADN génomique des cellules THP-1_GFP a été extrait, électroporé avec les RNP Cas9 :sgRNA (édité) ou Cas9 seul (non édité), et utilisé comme modèle pour amplifier par PCR une région de 891 pb contenant le site cible. Les cellules parentales THP-1 dépourvues d’EGFP ont également été incluses comme témoins négatifs. Ensuite, le produit PCR des cellules THP-1_GFP modifiées a été préparé pour la détection des mésappariements par le test T7EI, suivi de la séparation des fragments d’ADN par électrophorèse sur gel d’agarose. Une bande de ~900 pb correspondant à la taille attendue de l’amplicon WT a été facilement visualisée pour les cellules THP-1_GFP non modifiées, mais pas pour le témoin parental THP-1 (figure 2D, comparer les lignes 2 et 3). Dans le cas des cellules THP-1_GFP modifiées, la bande de ~900 pb est devenue indétectable et remplacée par une bande de migration plus rapide, dont la taille correspondait à la perte d’un fragment de ~75 pb résultant de l’édition CRISPR (Figure 2D, ligne 4). Ce modèle de bandes a été modifié lors de la digestion de T7EI, provoquant l’apparition de fragments d’ADN plus petits, confirmant davantage la réussite de l’édition du site cible (Figure 2D, ligne 5). Pour mieux comprendre l’efficacité de l’édition et les génotypes spécifiques de la population modifiée, l’amplicon PCR a été séquencé à l’aide de la méthode de Sanger, suivi d’une analyse avec l’outil ICE26. L’échantillon de contrôle non édité a été traité en parallèle. Nous avons récupéré des scores d’indel et de KO de 100 %, correspondant respectivement au pourcentage de séquences non-WT dans l’échantillon édité et à la proportion d’indels susceptibles de conduire à un KO fonctionnel (Figure 2E, à gauche). L’analyse détaillée du spectre de taille et de la fréquence de l’indel a montré la présence de deux populations dominantes portant une délétion de 62 pb ou 76 pb et représentant respectivement 57 % ou 37 % des séquences totales (figure 2E, à droite). Ces résultats suggèrent que l’ARNsg #1, en combinaison avec l’ARNg #2 ou #3, a déclenché une DSB concomitante dans le gène cible (Figure 2B).
Ce protocole a ensuite été appliqué pour inactiver le gène endogène codant pour SAMHD1 (Figure 3A). La caractérisation de la lignée cellulaire polyclonale SAMHD1 KO THP-1 comprenait le suivi de la prolifération cellulaire au fil du temps, qui correspondait à celle des cellules THP-1 non modifiées (Figure 3B). Le résultat de l’édition génomique a été évalué par amplification de la séquence cible par PCR, qui a révélé une bande plus petite pour les cellules KO SAMHD1 par rapport au contrôle Cas9 uniquement (Figure 3C, comparer les lignées 2 et 4), ce qui indique une perte de séquence d’ADN. Ces observations ont été confirmées par le test T7EI, montrant l’apparition de produits de clivage pour les cellules SAMHD1 KO, mais non non modifiées (Figure 3C, comparer les lignées 3 et 5). L’analyse des données de séquençage de Sanger à l’aide de l’outil ICE a donné des scores indel et KO de 100 % et 97 %, respectivement. Les principales modifications au sein du locus SAMHD1 étaient des délétions de séquence de 170 pb, 93 pb ou 104 pb dans 46 %, 13 % et 10 % des séquences, respectivement (Figure 3D). Les niveaux d’expression protéique dans le lysat de cellules brutes ont également été évalués par immunoblottage. La bande de ~70 kDa correspondant à la taille de SAMHD1 endogène devient pratiquement indétectable après modification, ce qui correspond à une réduction estimée de >97 % (Figure 3E). Enfin, la caractérisation phénotypique des lignées cellulaires polyclonales SAMHD1 KO THP-1 a également été évaluée en testant la sensibilité à l’infection par le VIH-1. En accord avec son rôle antiviral bien connu30,31, l’inactivation du locus SAMHD1 s’est accompagnée d’une augmentation du taux d’infection par le VIH-1 (Figure 3F).
En parallèle, des clones unicellulaires par dilution limite ont été générés. L’amplification du site cible génomique à partir des clones KO de SAMHD1 a donné des produits de PCR avec une mobilité électrophorétique accrue par rapport à ceux des cellules témoins non modifiées (Figure 4A). L’analyse des données de séquençage de Sanger basée sur l’ICE a donné un score indel de 100 % pour tous les clones unicellulaires sélectionnés, sauf un (clone A2) (figure 4B, à gauche). La caractérisation des résultats de l’édition au niveau génomique a renvoyé une seule séquence pour le clone A3 avec une délétion de 98 pb située à 28 pb en aval de l’ATG initial, conduisant ainsi à un changement hors cadre (Figure 4B, à droite). Les autres clones contenaient trois séquences (clone B1) ou plus, ce qui indique la présence d’allèles ayant subi différents événements d’édition et/ou plus d’une cellule par puits à l’étape de dilution limite (figure 4B, à droite). Notamment, 11 % des séquences des clones A4 et A7 portent des indels en amont de l’ATG initial, laissant la séquence codante inaltérée. De plus, 1 % des séquences du clone A7 étaient dépourvues d’un fragment de 123 pb à 9 pb en aval de l’ATG initial, ce qui a entraîné une délétion dans le cadre de 41 acides aminés. En accord avec ces résultats, des études fonctionnelles ont révélé que les clones avec un locus SAMHD1 perturbé (A3, A8, B1, B3), donc dépourvus d’expression de SAMHD1, étaient très permissifs à l’infection par le VIH-1 (Figure 4C). À l’inverse, les clones A4 et A7, où SAMHD1 est intact, étaient aussi réfractaires que les clones parentaux ou témoins non modifiés de THP-1 (Figure 4C). En conclusion, l’édition réussie du locus SAMHD1 dans le clone A3 a été confirmée.

Figure 1 : Schéma expérimental. (1) CRISPOR est utilisé pour générer des séquences d’ARNsg contre l’exon ciblé. Trois d’entre eux sont sélectionnés en fonction de leur efficacité élevée sur la cible et de leur faible prédiction hors cible. (2) Les trois ARNg sont mélangés avec Cas9 pour assembler la ribonucléoprotéine (RNP), ce qui donne un mélange de trois RNP différents. (3) Les cellules sont électroporées pour permettre l’entrée des RNP. (4) Les cellules sont transférées sur une plaque de culture à 24 puits et laissées au repos pendant au moins 72 h. (5) L’édition génomique est d’abord testée qualitativement par un test de digestion T7EI, et (6) pour les conditions validées, caractérisées par le séquençage de Sanger et l’analyse ICE. (7) Si nécessaire, les cellules peuvent être ensemencées après dilution limitée pour produire des populations clonales. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Configuration des paramètres d’électroporation pour EGFP KO dans les cellules THP-1 et validation de l’édition de gènes. (A) Les cellules THP-1 ont été transduites avec des vecteurs lentiviraux codant pour le gène rapporteur EGFP. Des clones unicellulaires ont été obtenus en limitant la dilution suivie d’une expansion en culture pendant 30 jours. L’analyse par cytométrie en flux a confirmé que la quasi-totalité de la population cellulaire exprimait la GFP. L’intensité médiane de fluorescence (MFI) de la population GFP positive, indicative du nombre de copies EGFP intégrées, variait entre plusieurs clones cellulaires. Le clone G5 a été choisi pour les études de suivi. (B) Représentation schématique du gène EGFP, y compris les sites de liaison pour l’ARNsg et les amorces. Les sites de coupe Cas9 sont indiqués par des lignes pointillées. (C) Des cellules THP-1_GFP indifférenciées (2 x 105) ont été mélangées avec des RNP Cas9 :sgRNA pré-assemblés avant électroporation avec les paramètres indiqués. Ensuite, les cellules ont été transférées dans une plaque à 24 puits contenant 500 μL de milieu RPMI préchauffé contenant 20 % de FBS seul ou dilué 1:1 avec un milieu conditionné produit par une culture cellulaire THP-1 de 48 heures. La prolifération (barres oranges) et le pourcentage de cellules positives à la GFP (ligne verte) ont été suivis au fil du temps. Les données représentent la moyenne pour n = 2 réplicats biologiques. Le cadre rouge met en évidence la condition sélectionnée pour les expériences ultérieures. (D) L’ADN génomique des cellules THP-1_GFP modifiées (RNP 3) ou non modifiées (Cas9) a été extrait, et la région entourant le locus cible amplifiée par PCR. Les cellules parentales THP-1 ont été utilisées comme contrôle négatif. Les amplicons ont subi un cycle de dénaturation/renaturation, conduisant à la formation d’un hétéroduplex. Ensuite, des discordances ont été détectées par le test T7EI. Les fragments d’ADN ont été séparés par électrophorèse sur gel d’agarose à 1,2 %. (E) Les produits PCR purifiés ont été séquencés par la méthode de Sanger, suivis d’une analyse avec l’algorithme ICE. Les scores Indel et KO indiquent le pourcentage de séquences non-WT dans l’échantillon, et la proportion de cellules avec un décalage de cadre ou un indel de 21+ pb, respectivement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
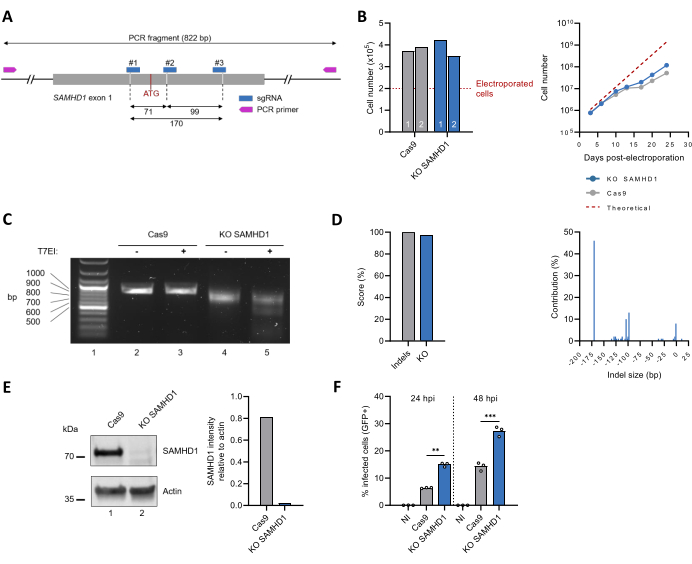
Figure 3 : Production et caractérisation d’une population polyclonale de cellules SAMHD1 KO THP-1. (A) Représentation schématique de l’exon 1 de SAMHD1, y compris les sites de liaison pour l’ARNsg et les amorces. Les sites de coupe Cas9 sont indiqués par des lignes pointillées, et le codon ATG initial est indiqué en rouge. (B) Trois jours après l’électroporation, les cellules viables (n = 2) ont été quantifiées à l’aide du test d’exclusion du bleu de trypan (schéma de gauche). La croissance cellulaire a été surveillée pendant 25 jours (schéma de droite). La ligne pointillée correspond au nombre de cellules théorique basé sur un temps de doublement estimé de 49 h. (C) Les résultats de l’édition au niveau génomique ont été évalués, comme le montre la figure 2D. (D) Les génotypes après édition CRISPR ont été identifiés et quantifiés par l’analyse des séquences de Sanger basée sur l’ICE. (E) La protéine contenue dans le lysat brut des cellules THP-1 modifiées et non modifiées a été séparée par migration sur gel SDS-PAGE et, ensuite, visualisée par immunoblot à l’aide d’anticorps contre SAMHD1. L’actine a été utilisée comme contrôle de la charge. L’intensité des bandes SAMHD1 et Actin a été quantifiée par densitométrie avec le logiciel ImageJ. (F) Les cellules THP-1 SAMHD1 non modifiées (Cas9) et KO ont été différenciées par un traitement par PMA (300 ng/mL, 24 h) et ensuite testées par un virus VIH-1 pseudotypé VSVg exprimant la GFP en tant que rapporteur. La proportion de cellules GFP positives a été notée par cytométrie en flux 24 h et 48 h après l’infection (hpi). NI : non infecté. **P < 0,01 ; P < 0,001 par test t non apparié avec correction de Welch. Veuillez cliquer ici pour voir une version agrandie de cette figure.
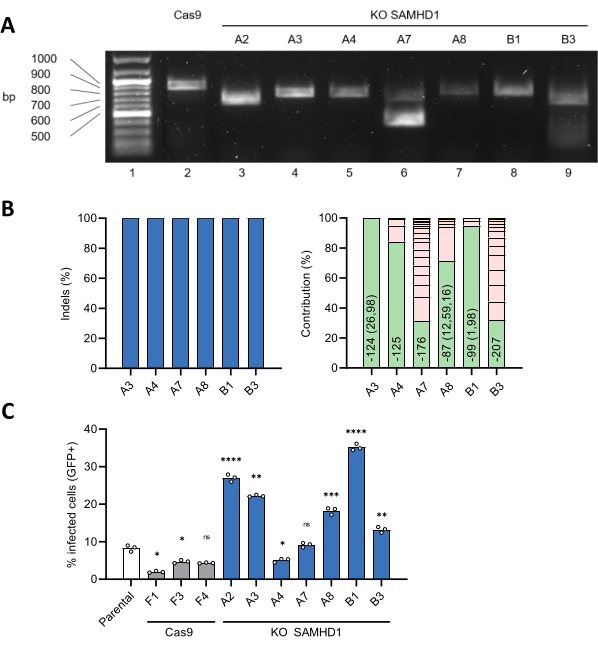
Figure 4 : Production et caractérisation de clones unicellulaires de SAMHD1 KO. (A) Le résultat de l’édition de clones unicellulaires de SAMHD1 KO sélectionnés a été évalué en surveillant la mobilité des fragments d’ADN produits par amplification par PCR de la région contenant le site cible par électrophorèse sur gel d’agarose. (B) Les données de séquençage de Sanger ont été analysées avec l’outil ICE, comme dans la figure 2E. Les barres du graphique de droite représentent la diversité des indels dans chaque population cellulaire. La barre verte représente la séquence dominante et la taille totale de la suppression associée. Les chiffres entre parenthèses indiquent la taille des espaces non continus. (C) La permissivité des clones unicellulaires non modifiés et de SAMHD1 KO à l’infection par le VIH-1 a été testée comme dans la figure 3F, et comparée à celle des cellules parentales THP-1 non électroporées ou des clones cellulaires non édités (Cas9). Le taux d’infection a été évalué à 48 hpi. *P < 0,05 ; **P < 0,01 ; P < 0,001 ; P < 0,0001 ; ns P≥ 0,05 par Brown-Forsythe et Welch ANOVA avec le test de comparaisons multiples de Dunnett. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Évaluation du résultat de l’édition pour les clones unicellulaires KO par le test T7EI adapté. L’ADN génomique de clones unicellulaires KO ou de cellules non modifiées a été purifié, et la région entourant le locus cible a été amplifiée par PCR. Les amplicons de chaque clone unicellulaire ont été mélangés selon un rapport de 1:1 avec ceux des cellules témoins non modifiées. Après formation d’hétéroduplex et digestion T7EI, le produit a été analysé par électrophorèse sur gel d’agarose. Les clones modifiés produisent plusieurs fragments de digestion (clones A, B, C, D et E), tandis que les clones non modifiés présentent une seule bande WT (clones F et G). Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Stratégie de contrôle de la cytométrie en flux pour quantifier le pourcentage de cellules THP-1 infectées par le VIH-1 traitées par PMA. En utilisant la zone de diffusion vers l’avant (FSC-A) contre la zone de diffusion latérale (SSC-A), une première porte est définie pour éliminer les débris et sélectionner les cellules THP-1 traitées par PMA. Ensuite, des cellules individuelles sont sélectionnées avec un SSC-A contre un tracé SSC-H. Enfin, les cellules GFP-positives sont détectées avec la GFP-A contre le FSC-H. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Ici, un protocole est décrit pour obtenir une édition réussie de la lignée cellulaire THP-1 médiée par CRISPR. L’approche repose sur le transfert de sgRNA/Cas9 RNP pré-assemblés par électroporation/nucléofection. Cette stratégie a été choisie pour limiter les effets hors cible qui peuvent survenir lors de l’intégration lentivirale de la cassette sgRNA/Cas9, produisant une expression persistante de la nucléase. Plusieurs ARNsg ciblant le gène d’intérêt ont été sélectionnés pour obtenir une édition fiable et efficace, ce qui augmente la probabilité de créer des indels génomiques, entraînant une perte d’expression protéique et de KO32 fonctionnel. Une attention particulière a été accordée aux paramètres d’électroporation pour obtenir une perméabilisation optimale de la membrane cellulaire, ce qui était nécessaire pour assurer une livraison efficace des RNP sgRNA/Cas9 tout en empêchant une mort cellulaire excessive33. À cet égard, un paramètre crucial est la vigueur de la culture cellulaire avant l’électroporation (c’est-à-dire un faible nombre de passages).
L’édition médiée par CRISPR-Cas9 a été validée au niveau génétique avec le test de détection des mésappariements T7EI, qui fournit une évaluation rentable et rapide de plusieurs échantillons pour la présélection avant le séquençage de Sanger. Cette méthode a été initialement développée pour estimer l’efficacité de l’édition à l’aide d’un seul ARNsg en comparant l’intensité d’amplicons pleine longueur et de produits de clivage34,35. Il convient de noter que les grands indels (>70 pb) produits lors de l’utilisation de plusieurs sgRNA peuvent être détectés en comparant la mobilité des fragments d’ADN obtenus par amplification PCR des loci ciblés à partir de cellules modifiées par rapport à des cellules non modifiées par électrophorèse sur gel standard. Néanmoins, le test T7EI pourrait être utile pour déterminer la présence de séquences hébergeant de petits indels (>1bp) dans une population cellulaire polyclonale ou, en utilisant le protocole adapté, une population cellulaire monoclonale.
La présence de mutations hors cadre ciblées, qui interrompent la séquence codante, a été confirmée par une approche simple, où le génome de l’ensemble de la population cellulaire est extrait et utilisé comme matrice pour l’amplification par PCR. Ensuite, les amplicons purifiés subissent un séquençage de Sanger en vrac, suivi d’une analyse bio-informatique avec l’outil ICE. Dans l’ensemble, cette méthode permet de surmonter la nécessité de sous-cloner les produits de PCR dans un plasmide et de transformer les produits de ligature en bactéries pour isoler des colonies de cellules uniques, qui sont séquencées individuellement.
Une fois que l’inactivation du locus ciblé est validée, une caractérisation approfondie de la lignée cellulaire modifiée est nécessaire pour attribuer avec certitude un phénotype donné à l’absence de la protéine d’intérêt et exclure les effets imprévus découlant de changements hors cible, mais aussi sur la cible qui pourraient avoir un impact sur la structure et/ou la fonction de l’ARNm.
L’absence d’expression protéique doit être confirmée par immunobuvardage et/ou immunofluorescence en utilisant éventuellement deux anticorps reconnaissant des épitopes différents. En effet, l’introduction d’une PTC dans un exon précoce proche du codon ATG canonique est associée au risque d’événements ITL, qui produisent des variants protéiques tronqués N-terminalement36. Il convient de noter que Tuladhar et al. ont signalé la détection d’espèces protéiques aberrantes dans une proportion significative (~50 %) de cellules éditées par CRISPR d’origine commerciale ou interne18,19. L’apparition de nouveaux produits protéiques a été attribuée à divers processus potentiellement spécifiques au type cellulaire, tels que le saut de l’exon contenant l’indel ou l’émergence de sites d’entrée internes du ribosome (IRES) provoquant l’initiation de la synthèse de protéines à d’autres TIS en aval du codon ATG canonique. Il y a aussi des indices que l’ITL est couplé à une évasion de la NMD dans certaines circonstances37,38. De même, les stratégies d’édition du génome ciblant un exon tardif pourraient ne pas déclencher la NMD de l’ARNm muté sous-jacent à l’ablation inefficace de l’expression génique20. Enfin, si Cas9 est guidé par plusieurs ARNsg, des réarrangements chromosomiques complexes peuvent survenir, y compris des inversions ou de grandes délétions de fragments d’ADN39, qui pourraient modifier la structure et l’expression du génome, en particulier lorsque les éléments régulateurs sont ciblés40.
Afin d’atténuer davantage l’impact des résultats involontaires de l’édition du génome, des études fonctionnelles sont nécessaires pour confirmer la relation entre la perte d’expression des protéines et le phénotype qui en découle (c’est-à-dire l’activité antivirale). Si les cellules modifiées sont sous-clonées, le comportement de plusieurs clones entre eux et avec la population polyclonale doit être comparé. La réalisation d’expériences de sauvetage, qui consistent à restaurer l’expression génique pour inverser le phénotype des cellules modifiées en un phénotype WT, renforcera également la spécificité et exclura d’éventuels événements hors cible.
Bien que ce protocole ait été conçu pour l’édition de cellules THP-1, il fournit une structure générale pour le flux de travail CRISPR-Cas9 KO qui peut être adaptée à d’autres lignées cellulaires. Il peut être nécessaire d’ajuster les paramètres d’électroporation, et si une tension supérieure à 1800 V est appliquée, un tampon de remise en suspension T doit être utilisé, conformément aux instructions du fabricant. Une autre variable à prendre en compte est le rapport de concentration entre Cas9 et l’ARNsg lors de l’assemblage des RNP. Bien que le rapport 1:9 ait bien fonctionné dans ce cas, sa modification peut améliorer l’efficacité du montage dans d’autres circonstances. Enfin, bien que cela ne soit pas bénéfique ici, l’utilisation de milieux conditionnés pour la récupération des cellules après l’électroporation doit être testée, car l’effet pourrait être différent avec différentes lignées cellulaires.
Déclarations de divulgation
Tous les auteurs n’ont aucun conflit d’intérêts.
Remerciements
Nous remercions JP Concordet (MNHN, U1154/UMR7196, Paris), G. Bossis (IGMM, Montpellier) et D. Schlüter (Faculté de médecine de Hanovre, Allemagne) pour le partage des protocoles et la discussion. Ce projet a reçu un financement du programme de recherche et d’innovation Horizon 2020 de l’Union européenne (convention de subvention n° 101017572 à l’AZ) et de l’ANRS (subvention ECTZ162721 à l’AZ). L’infrastructure de recherche IDMIT (Infectious Disease Model and Innovative Therapies) est soutenue par le programme d’investissement d’avenir (PIA) sous référence ANR_11_INSB_0008.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
Références
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon