Method Article
الضربة القاضية الجينية بوساطة CRISPR-Cas9 القائمة على التثقيب الكهربائي في خلايا THP-1 وعزل استنساخ الخلية المفردة
In This Article
Summary
يستخدم خط خلايا THP-1 على نطاق واسع كنموذج للتحقيق في وظائف الخلايا الوحيدة / الضامة البشرية عبر مختلف مجالات البحث المتعلقة بعلم الأحياء. توضح هذه المقالة بروتوكولا للهندسة الفعالة القائمة على CRISPR-Cas9 وعزل الاستنساخ أحادي الخلية ، مما يتيح إنتاج بيانات نمطية قوية وقابلة للتكرار.
Abstract
يستخدم خط خلايا ابيضاض الدم وحيد الخلية البشري (AML) THP-1 على نطاق واسع كنموذج لدراسة وظائف البلاعم البشرية المشتقة من الخلايا الوحيدة ، بما في ذلك تفاعلها مع مسببات الأمراض البشرية المهمة مثل فيروس نقص المناعة البشرية (HIV). بالمقارنة مع خطوط الخلايا الخالدة الأخرى ذات الأصل النخاعي ، تحتفظ خلايا THP-1 بالعديد من مسارات الإشارات الالتهابية السليمة وتعرض خصائص النمط الظاهري التي تشبه إلى حد كبير تلك الموجودة في الخلايا الوحيدة الأولية ، بما في ذلك القدرة على التمايز إلى ضامة عند معالجتها بفوربول -12-ميريستات 13-أسيتات (PMA). يوفر استخدام تقنية CRISPR-Cas9 لهندسة خلايا THP-1 من خلال الضربة القاضية الجينية المستهدفة (KO) نهجا قويا لتوصيف الآليات المتعلقة بالمناعة بشكل أفضل ، بما في ذلك تفاعلات الفيروس مع المضيف. تصف هذه المقالة بروتوكولا للهندسة الفعالة القائمة على CRISPR-Cas9 باستخدام التثقيب الكهربائي لتوصيل البروتينات النووية الريبية Cas9: sgRNA المجمعة مسبقا في نواة الخلية. يؤدي استخدام العديد من sgRNAs الذي يستهدف نفس الموضع في مواضع مختلفة قليلا إلى حذف شظايا الحمض النووي الكبيرة ، وبالتالي زيادة كفاءة التحرير ، كما تم تقييمه بواسطة اختبار نوكلياز داخلي T7 I. تم التحقق من صحة التحرير بوساطة CRISPR-Cas9 على المستوى الجيني عن طريق تسلسل Sanger متبوعا بتحليل استنتاج تعديلات CRISPR (ICE). تم تأكيد استنفاد البروتين عن طريق النشاف المناعي إلى جانب الفحص الوظيفي. باستخدام هذا البروتوكول ، تم تحقيق ما يصل إلى 100٪ indels في الموضع المستهدف وانخفاض يزيد عن 95٪ في التعبير عن البروتين. تجعل كفاءة التحرير العالية من السهل عزل المستنسخة أحادية الخلية عن طريق الحد من التخفيف.
Introduction
THP-1 هو خط خلوي مشتق من الخلايا الأحادية البشرية معزول من مريض يعاني من سرطان الدم الحاد (AML) ، والذي يعرض سمات نمطية تشبه إلى حد كبير تلك الموجودة في الخلايا الوحيدة الأولية1. بالمقارنة مع الضامة الأولية المشتقة من الخلايا الوحيدة ، والتي لا تنقسم وتعرض كلا من العمر المحدود والتباين بين / داخل المتبرعين في النمط الظاهري ، يمكن زراعة خلايا THP-1 إلى الأبد تقريبا ولها سلوك أكثر تجانسا يفضل استنساخ النتائج2،3،4،5،6. والجدير بالذكر أنه يمكن تمييز خلايا THP-1 نحو نمط ظاهري يشبه البلاعم مع فوربول -12-ميريستات 13-أسيتات (PMA) ، مما يجعلها نموذجا مستخدما على نطاق واسع في المختبر للتحقيق في استجابات الخلايا الوحيدة / البلاعم للإشارات الالتهابية7،8،9،10،11،12،13 أو العدوى بمسببات الأمراض البشرية ذات الصلة سريريا ، بما في ذلك فيروس نقص المناعة البشرية14، 15 ، 16. تعد إمكانية الهندسة الوراثية لخلايا THP-1 ذات أهمية في العديد من مجالات البحث المتعلقة بعلم الأحياء.
البروتين 9 المرتبط ب Clustered Rotating Short Palindromic Repeats-CRISPR (CRISPR-Cas9) هو نظام مناعي تكيفي بدائية النواة يعتمد على النوكلياز الموجه بالحمض النووي الريبي لتحلل الجينومات الفيروسية الغازية ، والتي أعيد برمجتها كأداة هندسة وراثية17. تستمر عملية تحرير الجينوم في ثلاث خطوات: التعرف والانقسام والإصلاح. يقوم الحمض النووي الريبي أحادي التوجيه (sgRNA) بتجنيد نوكلياز Cas9 إلى موضع جيني معين من خلال الاقتران الأساسي مع تسلسل دليل 20 نقطة أساس. يؤدي وجود تسلسل الشكل المجاور Protospacer (PAM) مباشرة 3 'من التسلسل الجينومي المستهدف 20 نقطة أساس إلى الفك والانقسام بوساطة Cas9 على كل من خيوط الحمض النووي بين الموضعين 17 و 18 (3-bp 5' من PAM). تتم معالجة كسر الخيط المزدوج الناتج (DSB) بواسطة مسارين رئيسيين للإصلاح. في حالة عدم وجود قالب إصلاح يحمل التماثل مع الموضع التالف ، فإن مسار الانضمام النهائي غير المتماثل (NHEJ) المعرض للخطأ سيقدم عمليات إدخال و / أو حذف عشوائية للنيوكليوتيدات (indels) ، مما قد يؤدي إلى طفرات في تغيير الإطار و / أو إدخال أكواد الإنهاء المبكرة (PTC). في المقابل ، يتم استهداف mRNAs المحتوية على PTC عن طريق التحلل بواسطة مسار اضمحلال mRNA بوساطة هراء (NMD) ، مما يؤدي في النهاية إلى تعطيل تعبير / وظيفة البروتين18،19،20. بدلا من ذلك، يمكن لمسار الإصلاح الموجه بالتماثل (HDR) المعتمد على القالب تشغيل DSB وإصلاحه بأمانة. تم تسخير هذه الآلية لتحقيق تحرير جيني دقيق ، بما في ذلك الضربات وبدائل القاعدة. تجدر الإشارة إلى أن حالة دورة الخلية هي عامل مهم يؤثر على اختيار مسار إصلاح DSB. في الواقع ، ينشط NHEJ في جميع مراحل دورة الخلية ، بينما يقتصر HDR بشكل أساسي على S / G2 المراحل21.
تنمو خلايا THP-1 في معلق ومن المعروف أنه يصعب نقلها باستخدام الحمض النووي البلازميد ، وهو إجراء من المحتمل أن يغير أيضا قابليتها للبقاء و / أو قدرتها على التمايز22،23. غالبا ما يتم استخدام التنبيغ مع نواقل الفيروسات العدسية القائمة على فيروس نقص المناعة البشرية -1 التي تشفر كل من Cas9 و sgRNA للضربة القاضية (KO) ، وهو جين مهم24. يضمن دمج كاسيت Cas9 / sgRNA في الجينوم الخلوي التعبير المطول وكفاءة KO ، ولكنه أيضا مصدر مستمر للتأثيرات غير المستهدفة25. بدلا من ذلك ، يتم تسليم البروتينات النووية الريبية Cas9: sgRNA المجمعة مسبقا (RNPs) عن طريق التثقيب الكهربائي ، وهي طريقة تنطوي على تكوين مؤقت للمسام في كل من البلازما والأغشية النووية عند تطبيق النبضات الكهربائية. يعد الحفاظ على صلاحية الخلية تحديا مهما عند اتباع هذا النهج.
هنا ، تم إنتاج خط خلية THP-1 يعبر بثبات عن GFP (THP-1_GFP) ليكون بمثابة أداة لإنشاء بروتوكول لتحقيق تحرير فعال قائم على CRISPR-Cas9. بعد تصميم استراتيجية لتعطيل جين EGFP باستخدام ثلاثة sgRNAs في وقت واحد (نهج متعدد الإرشادات) ، تم تحديد كفاءة KO بين العديد من ظروف التثقيب الكهربائي باستخدام تعبير GFP كقراءة. تم رصد تكاثر الخلايا بالتوازي. تم تأكيد تحرير الجينات من خلال كل من اختبار نوكلياز داخلي T7 I (T7EI) وتسلسل Sanger ، متبوعا بالتحليل باستخدام خوارزمية Inference of CRISPR Edits (ICE)26. تم استخدام المعلمات التي أسفرت عن انخفاض في تعبير GFP بنسبة تصل إلى 95٪ ، مع استعادة خلايا THP-1 لمعدلات النمو الطبيعية بعد التثقيب الكهربائي ، بنجاح لتعطيل جين داخلي (SAMHD1) وإنتاج استنساخ THP-1 أحادي الخلية.
Protocol
تفاصيل الكواشف والمعدات المستخدمة في هذه الدراسة مدرجة في جدول المواد.
1. دليل التصميم باستخدام CRISPOR (الشكل 1.1)
ملاحظة: يمكن استخدام برنامج SnapGene Viewer في الخطوات 4 و 7 و 10 للتعليق على موقع هدف التحرير وموقع تهجين التمهيدي PCR داخل الجين محل الاهتمام.
- انتقل إلى موقع Ensembl (www.ensembl.org). في مربع البحث ، حدد نوعا وأدخل اسم الجين محل الاهتمام. انقر فوق Go. حدد النتيجة التي تتوافق مع الجين (وليس نسخة نصية).
- انقر فوق إظهار جدول النص ثم حدد معرف النص الذي يتوافق مع ترميز البروتين المسمى بالذهب في عمود النمط الحيوي . بمجرد الوصول إلى صفحة النص ، انقر فوق Exons في القائمة اليسرى.
- قم بالتمرير لأسفل وانقر على تنزيل التسلسل. تأكد من أن تنسيق الملف هو FASTA. في الإعدادات - التسلسلات المضمنة، قم بإلغاء تحديد كل شيء باستثناء التسلسل الجيني.
أدخل الرقم "500" في تسلسل الجناح في أي من طرفي مربع النص . انقر فوق معاينة ، وحدد التسلسل الكامل (فقط النيوكليوتيدات بدون الرأس) ، وانسخه (Ctrl + C). - افتح SnapGene Viewer وانقر على ملف جديد > DNA. الصق التسلسل (Ctrl+V) في المربع إنشاء تسلسل . قم بإلغاء تحديد اكتشاف الميزات الشائعة وحدد خطي في المخطط.
أعد تسمية الملف وانقر فوق إنشاء. في القائمة اليمنى، قم بإلغاء تحديد إظهار الإنزيمات (الرمز الأول). في أسفل النافذة، حدد علامة التبويب تسلسل .
ملاحظة: تسمح هذه الخطوة باسترجاع تسلسل الجينات الكامل ، بما في ذلك exons و introns و 500 bp allanking sequences (اختياري). هذه المعلومات الأخيرة مفيدة لتصميم بادئات PCR لتضخيم الموقع المستهدف الموجود داخل exon الأول. - مرة أخرى على موقع Ensembl على الويب ، قم بالتمرير لأعلى في معاينة الملف وانقر فوق رجوع. الآن ، قم بتغيير تنسيق الملف إلى RTF. في الإعدادات - التسلسلات المضمنة، قم بإلغاء تحديد كل شيء باستثناء Exons. في إظهار المتغيرات، حدد لا. انقر فوق تنزيل في أعلى الصفحة.
- افتح الملف الذي تم تنزيله (باستخدام Word) ، والذي يحتوي على تسلسل exons ويظهر تسلسل الترميز باللون الأزرق ، بدءا من ATG الأولي. اختر exon المراد استهدافه بواسطة التحرير الموجه ب CRISPR-Cas9 (انظر أدناه للحصول على توصيات) ، وحدده وانسخه (Ctrl + C).
- الإكسون المستهدف هو إما إكسون مبكر أو إكسون يشفر مجالا مهما وظيفيا للبروتين. تجدر الإشارة إلى أن إنشاء PTC في exon متأخر قريب من 3 'UTR من المحتمل أن يفشل في استنباط NMD ، مما يؤدي إلى التعبير عن بروتين مقطوع بشكل C. على العكس من ذلك ، فإن إدخال PTC في exon مبكر قريب من موقع البدء الأصلي يرتبط بخطر الترجمة غير المشروعة (ITL ، المعروف أيضا باسم بدء الترجمة البديلة (ATI)) ، مما ينتج عنه تعبير غير متوقع عن بروتين مبتورة N الطرفية يبدأ في مواقع بدء الترجمة في الإطار (TIS) في اتجاه مجرى أول كودون ATG. وللتخفيف من هذا الخطر الأخير، ينصح بتقييم حدوث نظام معلومات الترجمة التكنولوجية البديل باستخدام ATGpr27 (atgpr.dbcls.jp) و/أو NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/).
- تأكد من أن exon الهدف يحتوي على تسلسل تشفير. ومع ذلك ، قد يكون من المفيد اختيار تلدين sgRNA مع منطقة 5'UTR في المنبع من ATG الأولي لتضمينه في الجزء المحذوف.
- من الناحية المثالية ، يجب أن يكون exon المستهدف مشتركا في جميع متغيرات نسخ ترميز البروتين للجين. تحقق مما إذا كان هذا هو الحال في عارض بيانات الجينوم (www.ncbi.nlm.nih.gov/gdv/) التابع ل NCBI من خلال البحث عن الجين محل الاهتمام.
سيؤدي النقر فوق اسم الجين (باللون الأخضر) في نافذة العرض إلى إظهار متغيرات النسخ (باللون الأرجواني). يتم تمثيل Exons بمستطيل.
- في SnapGene Viewer، اضغط على Ctrl+F، Ctrl+V، ثم Enter للبحث عن تسلسل exon. اضغط على Ctrl + T لإضافة ميزة جديدة ، وقم بتسميته ، وقم بتغيير النوع إلى "exon" وانقر فوق موافق.
- انتقل إلى موقع CRISPOR على الويب (http://crispor.gi.ucsc.edu/) والصق تسلسل exon في الخطوة 1. أولا ، حدد جينوم مرجعي في الخطوة 2 ثم نوع PAM المراد استهدافه في الخطوة 3 ، عادة 20bp-NGG ل SpCas9. انقر فوق إرسال.
- حدد اثنين من sgRNAs بحيث يتم فصلهما بما يصل إلى 150 نقطة أساس ، ثم حدد sgRNA ثالثا بينهما. فيما يلي بعض الإرشادات لاختيار sgRNA:
- ترتبط درجة خصوصية معهد ماساتشوستس للتكنولوجيا بالتأثيرات خارج الهدف. تشير الدرجة الأعلى إلى عدد أقل من الأهداف المحتملة. يعرض العمود الأيمن ثلاثة مواقع متوقعة خارج الهدف مرتبة من الأكثر إلى الأقل احتمالا ، جنبا إلى جنب مع مواقعها (في exon أو intron أو منطقة بين الجينات). يمكن الوصول إلى القائمة الكاملة للمواقع المتوقعة غير المستهدفة بالنقر فوق عرض الكل. إذا أمكن ، حدد sgRNAs بدرجة >80 في معهد ماساتشوستس للتكنولوجيا ، مع إعطاء الأولوية لأولئك الذين ليس لديهم أهداف خارج الأهداف ل 0 أو 1 أو 2 من عدم التطابق. بالإضافة إلى ذلك ، يجب تجنب sgRNAs ذات الأهداف غير المستهدفة في exon ، والتي لديها أكبر إمكانية للتأثير على النمط الظاهري.
- للحصول على الفعالية المتوقعة ، راجع درجة Doench '16. لاحظ أن درجة Doench '16 العالية تشير ببساطة إلى أن sgRNA من المرجح أن يكون فعالا. يجب تحديد الفعالية الفعلية تجريبيا. لهذا السبب ، من المفيد دائما تحديد العديد من sgRNAs ، حتى لو لم يكن المقصود استخدامها معا.
- يمكن أن يكون محتوى GC المرتفع جدا أو المنخفض جدا ، بالإضافة إلى زخارف معينة ، ضارا بكفاءة sgRNA ويجب تجنبه. يتم تمييز هذه المعلمات بواسطة CRISPOR.
- كرر الخطوة 1.7 لإضافة تسلسل sgRNA وتسلسل PAM المرتبط به إلى تسلسل الجينات في SnapGene Viewer. في الوقت نفسه ، الصق تسلسل sgRNA (بدون PAM) في ملف نصي أو Excel للاحتفاظ بالاتجاه 5'-3' المطلوب عند طلب قليل النوكليوتيد.
- للتضخيم المستند إلى تفاعل البوليميراز المتسلسل للموقع المستهدف ، صمم اثنين من الاشعال المحيطة به. يجب أن يتراوح حجم الأمبليكون بين 800 و 1000 نقطة أساس. تم استخدام PrimerQuest لتصميم البادئات لهذا البروتوكول (https://eu.idtdna.com/pages/tools/primerquest). بدلا من ذلك ، يوفر CRISPOR قائمة بالبادئات لتضخيم المنطقة الجينومية المستهدفة بالإضافة إلى المواقع غير المستهدفة. بعد عرض القائمة الكاملة للمواقع المحتملة خارج الهدف (الخطوة 1.9.1) ، انقر فوق البادئات خارج الهدف في الزاوية اليمنى السفلية.
2. تحضير الكاشف والخلية للتثقيب الكهربائي (الشكل 1.2)
- قم بإعداد صفيحة استزراع 24 بئرا لاستعادة الخلايا بعد التثقيب الكهربائي عن طريق ملء الآبار ب 500 ميكرولتر من وسط RPMI 1640 مكمل بنسبة 20٪ من المعطل الحراري (56 درجة مئوية ، 30 دقيقة) ، مصل بقري الجنين (FBS). لا تضيف المضادات الحيوية. احتفظ باللوحة في حاضنة مرطبة عند 37 درجة مئوية و 5٪ ثاني أكسيد الكربون2 لمدة 24 ساعة حتى التثقيب الكهربائي.
- إذا تم شحن sgRNAs جافة ، فأعد ترطيبها باستخدام المخزن المؤقت TE (10 ملي مولار تريس ، 1 ملي مولار EDTA ، درجة الحموضة 8.0) إلى تركيز نهائي يبلغ 100 ميكرومتر (أي 10 ميكرولتر من المخزن المؤقت TE لكل 1 نانومول من sgRNA). دوامة لمدة 30 ثانية، تحتضن عند 4 درجات مئوية طوال الليل للسماح بالإماهة الكاملة، وبعد تجانس قصير للماصة، قم بتخزين محلول مخزون sgRNA عند -20 درجة مئوية. اعتمادا على الحجم النهائي ، قم بعمل حصص لتجنب دورات التجميد والذوبان المتعددة.
- قم بإعداد محلول sgRNA عامل بتركيز نهائي قدره 30 ميكرومتر عن طريق تخفيف محلول المخزون 100 ميكرومتر في ماء خال من النوكلياز. دوامة لمدة 30 ثانية واحتضانها لمدة 5 دقائق في درجة حرارة الغرفة.
- قم بتجميع Cas9: sgRNA RNPs بنسبة 1: 9 مولارية عن طريق تخفيف 1 ميكرولتر في وقت واحد من كل من محاليل sgRNA الثلاثة 30 ميكرومتر و 0.5 ميكرولتر من 20 ميكرومتر من محاليل Cas9 في 3.5 ميكرولتر من محلول إعادة التعليق R ، المدرجة في مجموعة التثقيب الكهربائي (الحجم النهائي 7 ميكرولتر ، لحالة تجريبية واحدة ؛ مقياس وفقا لذلك). دوامة لفترة وجيزة واحتضانها لمدة 5 دقائق في درجة حرارة الغرفة.
- في غضون ذلك ، قم بإعداد عنصر تحكم غير معدل عن طريق إضافة 0.5 ميكرولتر من 20 ميكرومتر Cas9 إلى 6.5 ميكرولتر من المخزن المؤقت لإعادة التعليق R. Vortex لفترة وجيزة واحتضانه لمدة 5 دقائق في درجة حرارة الغرفة.
- أضف 5 ميكرولتر من المخزن المؤقت لإعادة التعليق R إلى جميع العينات للحصول على حجم نهائي قدره 12 ميكرولتر لكل حالة تثقيب كهربائي.
- تحضير خلايا THP-1 للتثقيب الكهربائي.
- لتقييم التركيز والجدوى عن طريق اختبار استبعاد Trypan blue. خفف الخلايا 1: 2 في محلول تلطيخ أزرق 0.4٪. بعد حضانة 30 ثانية ، قم بالتجانس جيدا وأضف 10 ميكرولتر في جدار غرفة العد بنمط شبكة محسن من Neubauer. عد ثلاثة مربعات كبيرة واقسم العد على 100 للحصول على تركيز الخلية (x106 خلايا / مل).
ملاحظة: تؤثر صحة الخلايا على حساسيتها للتثقيب الكهربائي. يجب توخي الحذر لإجراء زراعة الخلايا في الظروف المثلى. يجب أن يكون الوقت خارج الحاضنة محدودا ، ويجب تحضير جميع الكواشف والحلول وتسخينها مسبقا. - لكل حالة ، اجمع حجما يعادل 0.2 × 106 خلايا وأجهزة الطرد المركزي (336 × جم ، 5 دقائق ، 20 درجة مئوية).
- قم بشفط المادة الطافية باستخدام ماصة وأعد تعليق الحبيبات في 500 ميكرولتر من PBS. جهاز طرد مركزي مرة أخرى (336 × جم ، 5 دقائق ، 20 درجة مئوية).
- قم بشفط المادة الطافية بعناية باستخدام ماصة وأعد تعليق حبيبات خلية THP-1 ب 12 ميكرولتر من محلول RNP (الخطوة 2.6).
- لتقييم التركيز والجدوى عن طريق اختبار استبعاد Trypan blue. خفف الخلايا 1: 2 في محلول تلطيخ أزرق 0.4٪. بعد حضانة 30 ثانية ، قم بالتجانس جيدا وأضف 10 ميكرولتر في جدار غرفة العد بنمط شبكة محسن من Neubauer. عد ثلاثة مربعات كبيرة واقسم العد على 100 للحصول على تركيز الخلية (x106 خلايا / مل).
3. إعداد نظام التثقيب الكهربائي والنوة (الشكل 1.3)
- ضع محطة الماصة تحت خزانة السلامة الحيوية، ووضع أنبوب التثقيب الكهربائي في الدعامة، وأضف 3 مل من المخزن المؤقت E، المضمن في مجموعة التثقيب الكهربائي.
- بعد تشغيل جهاز التثقيب الكهربائي ، استخدم شاشة اللمس لضبط معلمات التثقيب الكهربائي التالية: الجهد = 1 500 فولت ، المدة = 10 مللي ثانية ، الرقم = 3.
- جهز ماصة التثقيب الكهربائي بطرف واستنشق 10 ميكرولتر من محلول RNP/THP-1 (الخطوة 2.7.4). أدخل الماصة في أنبوب التثقيب الكهربائي واضغط على Start (البدء) على شاشة جهاز التثقيب الكهربائي. انتظر حتى تظهر الرسالة اكتمل وقم بإزالة الماصة من الأنبوب.
- انقل الخلايا إلى بئر من اللوحة المسخنة مسبقا المكونة من 24 بئرا وتجانسها برفق. ضع الطبق مرة أخرى في الحاضنة المرطبة واتركها ترتاح دون إزعاج لمدة 72 ساعة.
ملاحظة: احرص على عدم عمل أي فقاعات عند سحب تعليق RNP/THP-1، لأنها ستتداخل مع إجراء التثقيب الكهربائي. إذا ظهرت رسالة خطأ في حالة عدم وجود قوس كهربائي مرئي، فقم بإزالة ماصة التثقيب الكهربائي من الأنبوب واضغط على البدء مرة أخرى. ومع ذلك ، إذا لوحظ قوس كهربائي على شكل شرارة ساطعة قصيرة ، فقد يشير ذلك إلى وجود فقاعات. من المحتمل أن يفشل إجراء التثقيب الكهربائي ، حتى في حالة عدم وجود رسالة خطأ.
4. استرداد THP-1 بعد 72 ساعة من التثقيب الكهربائي (الشكل 1.4)
- عد الخلايا لتقييم التركيز (الخطوة 2.7.1) بعد 72 ساعة من التثقيب الكهربائي.
- إذا كان هناك عدد كاف من الخلايا (أي ≥0.6 × 106 خلايا / مل) ، فقم بتخفيفها على الأقل بعامل 2 مع RPMI مكمل بنسبة 20٪ FBS و 1٪ بنسلين ستربتومايسين واجعل التركيز بين 0.3-0.5 × 106 خلايا لكل مل. خلاف ذلك ، اسمح للخلية بالتعافي لمدة 72 ساعة أخرى.
- قم بتمرير الخلايا وتضخيمها حتى يكون هناك ما يكفي للتحقق من صحة KO. في غضون ذلك ، يمكن البدء في عزل المستنسخة أحادية الخلية (الخطوة 7).
5. التحقق من صحة تحرير الجينات عن طريق مقايسة عدم تطابق T7EI (الشكل 1.5)
ملاحظة: قد يقلل الاختبار من كفاءة التحرير نظرا لأن T7EI يتعرف على عدم التطابق الأكبر من 1 نقطة أساس. وبالتالي ، فإن اختبار T7EI ليس مفيدا لفحص مجموعات الخلايا متماثلة اللواقح (أي المستنسخة أحادية الخلية) ما لم يتم تعديلها بشكل مناسب (الخطوة 5.7).
- قم بتقييم تركيز الخلية (الخطوة 2.7.1) واسحب حجما يعادل 0.1 × 106 خلايا في أنبوب 1.5 مل. جهاز الطرد المركزي (336 × جم ، 5 دقائق ، 20 درجة مئوية) ، يشط المادة الطافية ، ويعيد تعليق الحبيبات في 500 ميكرولتر من PBS. جهاز الطرد المركزي مرة أخرى ، وباستخدام الماصة ، يستنشق أكبر قدر ممكن من المواد الطافية دون إزعاج الحبيبات. قم بتجميد العينة وتخزينها في درجة حرارة -20 درجة مئوية.
- استخرج الحمض النووي الجيني ليكون بمثابة مصفوفة لتضخيم تفاعل البوليميراز المتسلسل (PCR).
- أعد تعليق الحبيبات ب 50 ميكرولتر من محلول استخراج الحمض النووي, تجانس, ونقل الحجم بالكامل في 0.2 مل أنبوب PCR. دوامة وجهاز طرد مركزي لفترة وجيزة (نبض لمدة 3 ثوان).
- ضع الأنبوب في جهاز تدوير حراري وقم بتسخينه عند 65 درجة مئوية لمدة 15 دقيقة ، متبوعا ب 98 درجة مئوية لمدة 10 دقائق.
- خفف الحمض النووي المستخرج ب 90 ميكرولتر من الماء عالي النقاء. دوامة وجهاز طرد مركزي لفترة وجيزة (5,000 × [غ], 3 ث).
- قم بتخفيف برايمر PCR (انظر جدول المواد) في ماء فائق النقاء للحصول على تركيز نهائي يبلغ 10 ميكرومتر (أي 10 ميكرومول / ميكرولتر).
- قم بإعداد مزيج PCR (باتباع الملاحظة أدناه) في أنبوب PCR سعة 0.2 مل (الحجم النهائي = 50 ميكرولتر ، لحالة واحدة). عادة ، سيكون هناك شرطان على الأقل: KO وعنصر التحكم غير المحرر مع Cas9 فقط.
ملاحظة: الحمض النووي الجيني المنقى: 10 ميكرولتر ؛ التمهيدي الأمامي والخلفي (10 ميكرومتر): 2.5 ميكرولتر لكل منهما ، التركيز النهائي 500 نانومتر ؛ المخزن المؤقت للتفاعل (5x): 10 ميكرولتر. دوامة جيدا قبل الإضافة. امزج dNTP (25 ملي مولار لكل منهما): 0.6 ميكرولتر ، التركيز النهائي 0.3 ملي مولار لكل dNTP. بوليميراز الحمض النووي: 0.5 ميكرولتر ؛ ماء فائق النقاء: 23.9 ميكرولتر. دوامة وجهاز طرد مركزي لفترة وجيزة (نبضة لمدة 3 ثوان). - ضع الأنابيب في جهاز التدوير الحراري وقم بتشغيل برنامج PCR بالإعدادات التالية:
ملاحظة: دورة واحدة عند 95 درجة مئوية لمدة 5 دقائق ، تليها 30 دورة [98 درجة مئوية لمدة 20 ثانية (خطوة التمسخ) ، X درجة مئوية لمدة 15 ثانية (خطوة التلدين) ، 72 درجة مئوية لمدة 45 ثانية (خطوة استطالة)] ، ثم دورة أخيرة واحدة عند 72 درجة مئوية لمدة دقيقتين. في نهاية التضخيم ، قم بإزالة الأنابيب والدوامة وجهاز الطرد المركزي لفترة وجيزة (نبض لمدة 3 ثوان). درجة حرارة التلدين (X) هي درجة حرارة الانصهار (Tm) للبادئات ناقص 5 درجات مئوية. - بالنسبة للسكان المحررين متعدد النسيلة: في أنبوب جديد سعة 0.2 مل ، أضف 17.5 ميكرولتر من أمبليكون تفاعل البوليميراز المتسلسل و 2 ميكرولتر من NEBuffer 2 (10x) لحجم نهائي يبلغ 19.5 ميكرولتر. دوامة وجهاز طرد مركزي لفترة وجيزة (نبضة لمدة 3 ثوان). بدلا من ذلك ، لفحص المستنسخة أحادية الخلية ، امزج 1: 1 من منتجات تفاعل البوليميراز المتسلسل من كل من خلايا التحكم المعدلة وغير المعدلة (الخطوة 5.6) (الشكل التكميلي 1).
- لتشكيل heteroduplex ، ضع الأنابيب في جهاز التدوير الحراري وقم بتشغيل البرنامج التالي: دورة واحدة عند 95 درجة مئوية لمدة 10 دقائق ، وواحدة بمنحدر -2 درجة مئوية / ثانية من 95 إلى 85 درجة مئوية ، وواحدة بمنحدر -0.3 درجة مئوية / ثانية من 85 إلى 25 درجة مئوية ودورة تبريد نهائية واحدة عند 10 درجات مئوية عند الانتظار.
- بعد تكوين heteroduplex ، أضف 0.5 ميكرولتر من محلول T7EI إلى الأنبوب. احتضان عند 37 درجة مئوية لمدة 30 دقيقة.
- تحضير جل الاغاروز 1.2٪:
- قم بوزن الاغاروز في زجاجة زجاجية وأضف الحجم المناسب من 1x TAE buffer (40 ملي أسيتات ثلاثية ، 1 ملي EDTA ، درجة الحموضة 8.3). ضعه في الميكروويف مع الانتباه إلى عدم شد الغطاء بإحكام. سخنيها عدة مرات حتى تذوب بلورات الاغاروز تماما.
ملاحظة: إذا لزم الأمر ، امزج المحلول. لا تدعه يغلي ، وإلا سينخفض الحجم ، مما يؤدي إلى تغيير تركيز الاغاروز. - أضف بقعة هلام الحمض النووي المخففة عند 1 / 20,000 واخلطها جيدا.
- يسكب محلول الاغاروز في قالب ، ويضاف مشطا ، ويترك في درجة حرارة الغرفة.
- قم بوزن الاغاروز في زجاجة زجاجية وأضف الحجم المناسب من 1x TAE buffer (40 ملي أسيتات ثلاثية ، 1 ملي EDTA ، درجة الحموضة 8.3). ضعه في الميكروويف مع الانتباه إلى عدم شد الغطاء بإحكام. سخنيها عدة مرات حتى تذوب بلورات الاغاروز تماما.
- قم بإعداد عينات للرحلان الكهربائي للهلام عن طريق خلط 5 ميكرولتر من منتجات الهضم T7EI (الخطوة 5.9) أو منتج تفاعل البوليميراز المتسلسل غير المهضوم مع 5 ميكرولتر من الماء و 2 ميكرولتر من صبغة تحميل 6x DNA. قم بتحميل العينات وسلم الحجم وانتقل عند 80 فولت لمدة 45 دقيقة (يمكن تكييف الوقت اعتمادا على الحجم المتوقع لشظايا الحمض النووي). بمجرد انتهاء الهجرة ، احصل على صورة للهلام باستخدام نظام تصوير مناسب.
6. التحقق من صحة تحرير الجينات عن طريق تحليل تسلسل سانجر (الشكل 1.6)
- لتوصيف التعديل الجيني ، قم بتنقية منتجات تفاعل البوليميراز المتسلسل (الخطوة 5.4) ، وبعد تسلسل Sanger ، قم بتحليل النتائج باستخدام أداة ICE (https://www.synthego.com/products/bioinformatics/crispr-analysis).
- التحكم في إمكانية إنتاج البروتين على الرغم من التعديلات.
- قم بتنزيل نتائج ICE وافتح ملف contribs.txt.
- قارن التسلسلات المعدلة مع نظير WT. قم بتعيين indels وتحقق من أنها نشأت في المنطقة الجينومية المستهدفة. تقييم حجمها. إذا لم يكن طول indel مضاعفا لثلاثة ، فسيحدث إزاحة إطار ، وربما يتم إدخال PTC. من المتوقع أن يخضع mRNA المتحور للتدهور بوساطة NMD.
7. عزل استنساخ الخلية الواحدة عن طريق الحد من التخفيف (الشكل 1.7)
ملاحظة: عزل المستنسخة أحادية الخلية ليس إلزاميا. ومع ذلك ، إذا اخترت القيام بذلك ، فمن المهم توصيف المستنسخة المتعددة ومقارنة نمطها الظاهري مع المجموعة الأصلية متعددة النسيلة
- خذ عينة من تعليق الخلية ، وقم بتخفيفها 1/2 بوسط الثقافة ، وقم بتقييم التركيز (الخطوة 2.7.1). التخفيف يزيد من دقة العد.
- قم بتنفيذ 2 إلى 3 خطوات تخفيف تسلسلي للوصول إلى تركيز 7 خلايا / مل. قم بتوزيع 100 ميكرولتر من تعليق الخلية لكل بئر في صفيحة مستديرة القاع 96 بئرا (أي 0.7 خلية لكل بئر). دع الخلية تصب لبضع ساعات قبل مراقبة الصفائح في المجهر لتحديد الآبار التي تحتوي على خلية واحدة.
- راقب تكوين المستعمرة ونموها بانتظام وانقل الخلايا إلى صفيحة ثقافة أكبر أو قارورة عند الحاجة.
8. التوصيف الوظيفي لخلايا THP-1 KO SAMHD1 عن طريق فحص تقييد فيروس نقص المناعة البشرية -1
- بذرة THP-1 في صفيحة استزراع 24 بئرا مع 0.25 × 106 خلايا لكل بئر في 300 ميكرولتر من RPMI مكملة بنسبة 10٪ FBS ، 1٪ بنسلين - ستربتومايسين (RPMI كامل) وتحتوي على 300 نانوغرام / مل PMA للتمايز. احتفظ باللوحة في حاضنة رطبة (37 درجة مئوية ، 5٪ ثاني أكسيدالكربون 2) لمدة 24 ساعة.
- استبدل الوسيط بوسيط RPMI كامل خال من PMA وأعد اللوحة إلى الحاضنة لمدة 24 ساعة أخرى.
- باستخدام مضخة تفريغ ، قم بشفط كل وسط الثقافة. أضف 250 ميكرولتر من محلول يحتوي على مخزون فيروسي VSVg-pseudotype HIV-1-GFP (وزارة الداخلية = 0.5 IFU/خلية). قم بتضمين عنصر تحكم غير مصاب. ضع اللوحة على حرارة 4 درجات مئوية لمدة ساعتين لمزامنة العدوى.
- اغسل الخلايا مرة واحدة باستخدام RPMI. أضف 500 ميكرولتر من RPM الكامل إلى كل بئر واحتضانه لمدة 48 ساعة في الظروف القياسية (يمكن ضبط الوقت).
- بعد إزالة وسط الثقافة ، اغسل الخلايا مرة واحدة باستخدام 1x PBS بارد. ثم أضف 100 ميكرولتر من 0.05٪ Trypsin-EDTA في كل بئر. ضع اللوحة في حاضنة عند 37 درجة مئوية حتى تنفصل الخلايا تماما (يجب أن تكون 5 دقائق كافية) قبل إضافة 200 ميكرولتر من RPMI الكامل لتعطيل الإنزيم. انقل 250 ميكرولتر من كل بئر إلى صفيحة مستديرة القاع سعة 96 بئرا (تأكد من أن مقياس التدفق الخلوي يقبل ألواح الاستزراع).
- قم بالطرد المركزي للوحة (757 × جم، 3 دقائق، التباطؤ = 6، 20 درجة مئوية) وشفط المادة الطافية باستخدام ماصة متعددة القنوات. أعد تعليق الحبيبات ب 100 ميكرولتر من 4٪ PFA واحتضانها عند 4 درجات مئوية لمدة 10 دقائق. أضف 100 ميكرولتر من 1X PBS البارد.
- تحليل معدل الإصابة ، ممثلا بالخلايا الإيجابية ل GFP ، عن طريق قياس التدفق الخلوي (انظر الشكل التكميلي 2 للحصول على استراتيجية بوابة مقترحة).
النتائج
تم إنشاء خط خلية THP-1 بثبات يعبر عن بروتين مراسل GFP (THP-1_GFP) (الشكل 2 أ) ويستخدم كأداة لإنشاء بروتوكول لإصدار جيني فعال بوساطة CRISPR-Cas9. لهذا الهدف ، تم تصميم 3 sgRNA تستهدف جين EGFP باستخدام أداة الويب CRISPOR29 (الشكل 2 ب) ، والتي تم تعقيدها في وقت واحد مع Cas9 بنسبة مولية تبلغ 9: 1 لتشكيل RNPs قبل تسليمها إلى الخلايا عن طريق التثقيب الكهربائي باستخدام إعدادات مختلفة. بعد ذلك ، نمت الخلايا إما في وسط RPMI يحتوي على FBS بنسبة 20٪ وحده (الشكل 2C ، اللوحة العلوية) أو مخفف بنسبة 1: 1 بوسائط مكيفة (الشكل 2 ج ، اللوحة السفلية). تمت مراقبة تكاثر الخلايا وتعبير GFP ، كقراءة لكفاءة EGFP KO ، بمرور الوقت. في العديد من الحالات ، انخفضت النسبة المئوية للخلايا الإيجابية ل GFP بشكل حاد ، حيث وصلت إلى انخفاض بنسبة >90٪ في اليوم السابع بعد التثقيب الكهربائي (PE). في اليوم 3 pe ، انخفض عدد الخلايا إلى النصف ، ويرجع ذلك على الأرجح إلى التأثير السلبي للتثقيب الكهربائي على بقاء الخلية. ومع ذلك ، ارتفع عدد الخلية مرة أخرى ، وعاد وقت المضاعفة إلى المعدل الطبيعي بحلول اليوم 7 pe (الشكل 2C ، اللوحة العلوية). لم يفضل استخدام الوسائط المشروطة استعادة الخلايا في ظل ظروفنا التجريبية (الشكل 2 ج ، اللوحة السفلية). بناء على هذه النتائج ، تم اختيار ثلاث نبضات 10 مللي ثانية من 1500 فولت لتجارب المتابعة.
ثم تم تمييز EGFP KO بوساطة CRISPR-Cas9 على المستوى الجيني. لهذا الغرض ، تم استخراج الحمض النووي الجيني لخلايا THP-1_GFP ، وتغذيته بالكهرباء إما باستخدام Cas9: sgRNA RNPs (تم تحريره) أو Cas9 وحده (غير محرر) ، واستخدامه كقالب لتضخيم PCR منطقة 891 نقطة أساس تحتوي على الموقع المستهدف. كما تم تضمين خلايا THP-1 الأبوية التي تفتقر إلى EGFP كتحكم سلبي. بعد ذلك ، تم إعداد منتج تفاعل البوليميراز المتسلسل لخلايا THP-1_GFP المعدلة للكشف عن عدم التطابق بواسطة مقايسة T7EI ، متبوعا بفصل شظايا الحمض النووي عن طريق الرحلان الكهربائي لهلام الاغاروز. تم تصور نطاق ~ 900 نقطة أساس المقابل للحجم المتوقع لأمبليكون WT بسهولة لخلايا THP-1_GFP غير المعدلة ولكن ليس للتحكم الأبوي THP-1 (الشكل 2D ، قارن السطرين 2 و 3). في حالة خلايا THP-1_GFP المعدلة ، أصبح النطاق ~ 900 نقطة أساس غير قابل للاكتشاف واستبداله بنطاق أسرع ، يتطابق حجمه مع فقدان جزء ~ 75 نقطة أساس نتيجة لتحرير كريسبر (الشكل 2 د ، السطر 4). تم تغيير نمط النطاقات هذا عند هضم T7EI ، مما تسبب في ظهور شظايا أصغر من الحمض النووي ، مما يؤكد التحرير الناجح للموقع المستهدف (الشكل 2D ، السطر 5). لاكتساب نظرة ثاقبة لكفاءة التحرير والأنماط الجينية المحددة للسكان المحررين ، تم تسلسل أمبليكون تفاعل البوليميراز المتسلسل باستخدام طريقة سانجر ، متبوعا بالتحليل باستخدام أداة ICE26. تمت معالجة عينة التحكم غير المحررة بالتوازي. استرجعنا درجات indel و KO بنسبة 100٪ ، المقابلة للنسبة المئوية للتسلسلات غير WT في العينة المعدلة ونسبة indels التي من المحتمل أن تؤدي إلى KO وظيفي ، على التوالي (الشكل 2E ، على اليسار). أظهر التحليل التفصيلي للطيف والتردد بحجم indel وجود مجموعتين من السكان المهيمنة تحملان حذفا 62 نقطة أساس أو 76 نقطة أساس ويمثلان 57٪ أو 37٪ من إجمالي التسلسلات ، على التوالي (الشكل 2E ، على اليمين). تشير هذه النتائج إلى أن sgRNA # 1 ، بالاشتراك مع sgRNA # 2 أو # 3 ، أدى إلى حدوث DSB متزامن في الجين المستهدف (الشكل 2 ب).
ثم تم تطبيق هذا البروتوكول لتعطيل جين ترميز SAMHD1 الداخلي (الشكل 3 أ). تضمن توصيف خط الخلايا متعددة النسيلة SAMHD1 KO THP-1 مراقبة تكاثر الخلايا بمرور الوقت ، والتي تطابقت مع خلايا THP-1 غير المعدلة (الشكل 3 ب). تم تقييم نتيجة تحرير الجينات عن طريق التضخيم القائم على تفاعل البوليميراز المتسلسل المستهدف ، والذي كشف عن نطاق أصغر لخلايا SAMHD1 KO مقارنة بعنصر التحكم Cas9 فقط (الشكل 3C ، قارن الخطين 2 و 4) ، مما يدل على فقدان تسلسل الحمض النووي. تم تأكيد هذه الملاحظات من خلال اختبار T7EI ، الذي يظهر ظهور منتجات الانقسام لخلايا SAMHD1 KO ، ولكن ليس غير المحررة ، (الشكل 3C ، قارن الخطين 3 و 5). أعاد تحليل بيانات تسلسل Sanger باستخدام أداة ICE درجات indel و KO بنسبة 100٪ و 97٪ على التوالي. كانت التعديلات الرئيسية داخل موضع SAMHD1 هي عمليات حذف تسلسل 170 نقطة أساس أو 93 نقطة أساس أو 104 نقطة أساس في 46٪ و 13٪ و 10٪ من التسلسلات على التوالي (الشكل 3 د). كما تم تقييم مستويات التعبير عن البروتين في محللة الخلايا الخام عن طريق النشاف المناعي. يصبح النطاق ~ 70 كيلو دالتون المتوافق مع حجم SAMHD1 الداخلي غير قابل للاكتشاف تقريبا بعد التحرير ، وهو ما يتوافق مع انخفاض يقدر بنسبة >97٪ (الشكل 3E). أخيرا ، تم أيضا تقييم التوصيف الظاهري لخطوط الخلايا متعددة النسيلة SAMHD1 KO THP-1 من خلال اختبار القابلية للإصابة بعدوى فيروس نقص المناعة البشرية -1. بالاتفاق مع دوره المعروف المضاد للفيروسات30،31 ، كان تعطيل موضع SAMHD1 مصحوبا بزيادة معدل الإصابة بفيروس نقص المناعة البشرية -1 (الشكل 3F).
في موازاة ذلك ، تم إنشاء استنساخ أحادي الخلية عن طريق الحد من التخفيف. أدى تضخيم الموقع المستهدف الجيني من استنساخ SAMHD1 KO إلى إنتاج منتجات تفاعل البوليميراز المتسلسل مع زيادة الحركة الكهربية مقارنة بتلك الموجودة في خلايا التحكم غير المحررة (الشكل 4 أ). أعاد التحليل المستند إلى ICE لبيانات تسلسل Sanger درجة indel بنسبة 100٪ لجميع المستنسخة أحادية الخلية المختارة باستثناء واحدة (استنساخ A2) (الشكل 4 ب ، على اليسار). أدى توصيف نتائج التحرير على المستوى الجينومي إلى إرجاع تسلسل واحد لاستنساخ A3 مع حذف 98 نقطة أساس يقع في اتجاه مجرى ATG الأولي ، مما يؤدي إلى تغيير خارج الإطار (الشكل 4 ب ، على اليمين). احتوت المستنسخة الأخرى على ثلاثة (استنساخ B1) أو أكثر من التسلسلات ، مما يشير إلى وجود الأليلات التي خضعت لأحداث تحرير مختلفة و / أو أكثر من خلية واحدة لكل بئر في خطوة التخفيف المحددة (الشكل 4 ب ، على اليمين). والجدير بالذكر أن 11٪ من تسلسلات المستنسخة A4 و A7 تحمل indels في المنبع من ATG الأولي ، مما يترك تسلسل الترميز دون تغيير. علاوة على ذلك ، فإن 1٪ من تسلسلات A7 المستنسخة تفتقر إلى جزء 123 نقطة أساس 9 نقاط أساس في اتجاه مجرى ATG الأولي ، مما أدى إلى حذف 41 من الأحماض الأمينية في الإطار. تمشيا مع هذه النتائج ، كشفت الدراسات الوظيفية أن المستنسخة ذات موضع SAMHD1 المعطل (A3 ، A8 ، B1 ، B3) ، وبالتالي تفتقر إلى تعبير SAMHD1 ، كانت متساهلة للغاية للإصابة بفيروس نقص المناعة البشرية -1 (الشكل 4 ج). على العكس من ذلك ، كانت المستنسخة A4 و A7 ، حيث يكون SAMHD1 سليما ، مقاومة للحرارة مثل استنساخ THP-1 للتحكم الأبوي أو غير المحرر (الشكل 4 ج). في الختام ، تم تأكيد التحرير الناجح لموضع SAMHD1 في استنساخ A3.

الشكل 1: المخطط التجريبي. (1) يستخدم CRISPOR لإنشاء تسلسلات sgRNA ضد exon المستهدف. يتم اختيار ثلاثة منهم وفقا لفعالية عالية على الهدف والتنبؤ الضعيف خارج الهدف. (2) يتم خلط gRNAs الثلاثة مع Cas9 لتجميع البروتين النووي الريبي (RNP) ، مما ينتج عنه مزيج من ثلاثة RNPs مختلفة. (3) يتم تزويد الخلايا بالكهرباء للسماح بدخول RNPs. (4) يتم نقل الخلايا إلى صفيحة مزرعة مكونة من 24 بئرا وتترك لترتاح لمدة 72 ساعة على الأقل. (5) يتم اختبار تحرير الجينات نوعيا لأول مرة بواسطة مقايسة الهضم T7EI ، و (6) للظروف التي تم التحقق من صحتها ، والتي تتميز أيضا بتسلسل سانجر وتحليل ICE. (7) إذا لزم الأمر ، يمكن زرع الخلايا بعد الحد من التخفيف لإنتاج مجموعات نسيلية. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2: إعداد إعدادات التثقيب الكهربائي ل EGFP KO في خلايا THP-1 والتحقق من صحة تحرير الجينات. (أ) تم نقل خلايا THP-1 بنواقل الفيروسية العدسية التي تشفر جين مراسل EGFP. تم الحصول على استنساخ وحيد الخلية عن طريق الحد من التخفيف متبوعا بالتوسع في الثقافة لمدة 30 يوما. أكد التحليل عن طريق قياس التدفق الخلوي أن جميع الخلايا تقريبا عبرت عن GFP. تباين متوسط شدة التألق (MFI) للسكان الإيجابيين ل GFP ، مما يدل على رقم نسخة EGFP المتكامل ، بين العديد من نسخ الخلايا. تم اختيار Clone G5 لدراسات المتابعة. (ب) التمثيل التخطيطي لجين EGFP ، بما في ذلك مواقع الارتباط لكل من sgRNA والبادئات. يشار إلى مواقع قطع Cas9 بالخطوط المتقطعة. (ج) تم خلط خلايا THP-1_GFP غير المتمايزة (2 × 105) مع Cas9: sgRNA RNPs المجمعة مسبقا قبل التثقيب الكهربائي بالإعدادات المشار إليها. بعد ذلك ، تم نقل الخلايا في صفيحة من 24 بئرا تحتوي على 500 ميكرولتر من وسط RPMI المحتوي على FBS بنسبة 20٪ مسخن مسبقا بمفرده أو مخفف بنسبة 1: 1 مع وسط مكيف ينتج عن مزرعة خلية THP-1 لمدة 48 ساعة. تم رصد التكاثر (القضبان البرتقالية) والنسبة المئوية للخلايا الإيجابية ل GFP (الخط الأخضر) بمرور الوقت. تمثل البيانات متوسط n = 2 تكرار بيولوجي. يسلط الإطار الأحمر الضوء على الحالة المحددة للتجارب اللاحقة. (د) تم استخراج الحمض النووي الجيني لخلايا THP-1_GFP المعدلة (RNP 3 sgRNA) أو غير المعدلة (Cas9) ، وتم تضخيم المنطقة المحيطة بالموضع المستهدف بواسطة تفاعل البوليميراز المتسلسل (PCR). تم استخدام خلايا THP-1 الأبوية كعنصر تحكم سلبي. خضعت الأمبليكونات لدورة تمسخ / إعادة تمسخ ، مما أدى إلى تكوين ثنائي الأحجاء. بعد ذلك ، تم اكتشاف عدم التطابق بواسطة اختبار T7EI. تم فصل شظايا الحمض النووي عن طريق الرحلان الكهربائي لهلام الاغاروز بنسبة 1.2٪. (ه) تم تسلسل منتجات تفاعل البوليميراز المتسلسل المنقى بطريقة سانجر ، متبوعا بالتحليل باستخدام خوارزمية ICE. تشير درجات Indel و KO إلى النسبة المئوية للتسلسلات غير WT في العينة ، ونسبة الخلايا ذات إزاحة الإطار أو 21+ نقطة أساس ، على التوالي. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
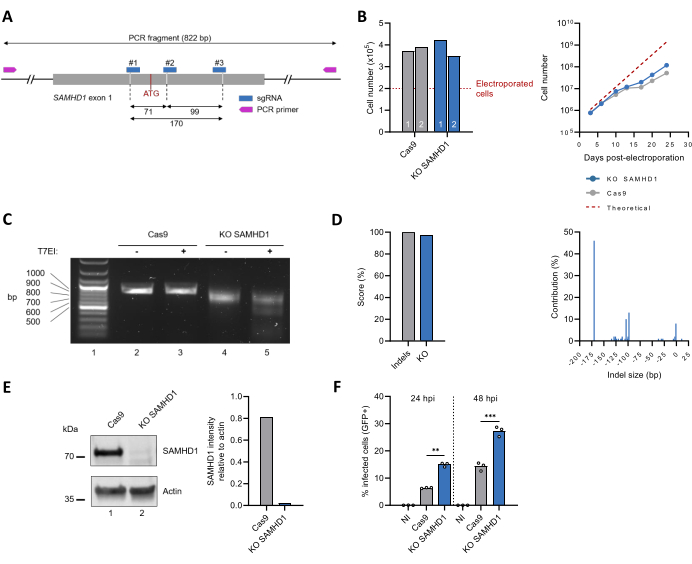
الشكل 3: إنتاج وتوصيف مجموعة خلايا SAMHD1 KO THP-1 متعددة النسيحة. (أ) التمثيل التخطيطي ل SAMHD1 exon 1 ، بما في ذلك مواقع الربط ل sgRNA والبادئات. يشار إلى مواقع قطع Cas9 بالخطوط المتقطعة ، ويظهر كودون ATG الأولي باللون الأحمر. (ب) بعد ثلاثة أيام من التثقيب الكهربائي ، تم قياس الخلايا القابلة للحياة (ن = 2) باستخدام اختبار استبعاد Trypan blue (الرسم البياني الأيسر). تمت مراقبة نمو الخلايا على مدار 25 يوما (الرسم البياني الأيمن). يتوافق الخط المتقطع مع رقم الخلية النظرية بناء على وقت مضاعفة يقدر ب 49 ساعة. (ج) تم تقييم نتائج التحرير على المستوى الجينومي ، كما هو موضح في الشكل 2 د. (د) تم تحديد الأنماط الجينية بعد تحرير كريسبر وقياسها كميا من خلال التحليل المستند إلى ICE لتسلسلات سانجر. (ه) تم فصل البروتين الموجود في المحللة الخام لكل من خلايا THP-1 المعدلة وغير المعدلة عن طريق الهجرة على هلام SDS-PAGE ، وبعد ذلك ، تم تصوره عن طريق النشاف المناعي باستخدام الأجسام المضادة ضد SAMHD1. تم استخدام الأكتين كعنصر تحكم في التحميل. تم قياس كثافة نطاقات SAMHD1 و Actin عن طريق قياس الكثافة باستخدام برنامج ImageJ. (F) تم تمييز الخلايا غير المعدلة (Cas9) و KO SAMHD1 THP-1 عن طريق العلاج باستخدام PMA (300 نانوغرام / مل ، 24 ساعة) وتم تحديها بعد ذلك بفيروس HIV-1 ذو النمط الزائف VSVg الذي يعبر عن GFP كمراسل. تم تسجيل نسبة الخلايا الإيجابية ل GFP عن طريق قياس التدفق الخلوي في 24 ساعة و 48 ساعة بعد الإصابة (hpi). NI: غير مصاب. ** ص < 0.01 ؛ P < 0.001 عن طريق اختبار t غير المزاوج مع تصحيح ويلش. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
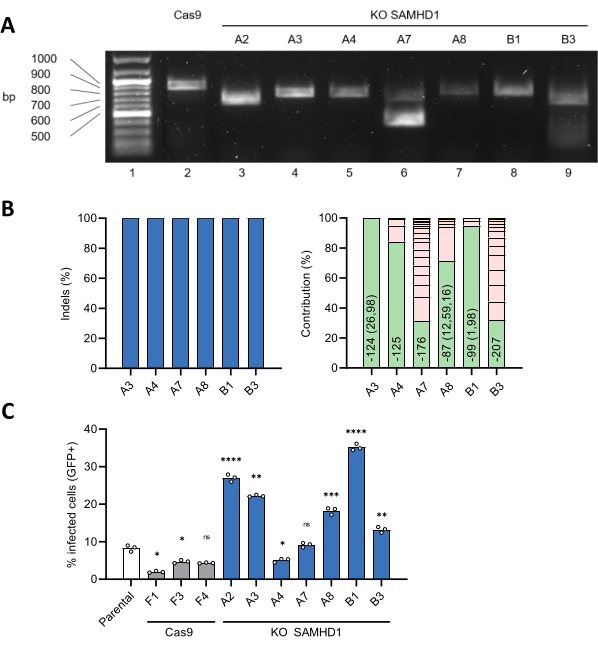
الشكل 4: إنتاج وتوصيف المستنسخة أحادية الخلية SAMHD1 KO. (أ) تم تقييم نتيجة التحرير لاستنساخ SAMHD1 KO أحادي الخلية المختارة من خلال مراقبة حركة شظايا الحمض النووي الناتجة عن تضخيم تفاعل البوليميراز المتسلسل للمنطقة التي تحتوي على الموقع المستهدف بواسطة الرحلان الكهربائي لهلام الاغاروز. (ب) تم تحليل بيانات تسلسل Sanger باستخدام أداة ICE ، كما في الشكل 2E. تمثل الأشرطة الموجودة في الرسم البياني الأيمن تنوع indels في كل مجموعة من الخلايا. يمثل الشريط الأخضر التسلسل السائد والحجم الإجمالي للحذف المرتبط. تشير الأرقام الموجودة بين قوسين إلى حجم الفجوات غير المستمرة. (ج) تم اختبار تساهل المستنسخة أحادية الخلية غير المحررة و SAMHD1 KO لعدوى فيروس نقص المناعة البشرية -1 كما في الشكل 3F ، ومقارنتها بخلايا THP-1 الأبوية غير المثقلة بالكهرباء أو استنساخ الخلايا غير المحررة (Cas9). تم تسجيل معدل الإصابة عند 48 hpi. * P < 0.05 ؛ ** ص < 0.01 ؛ P < 0.001; P < 0.0001; ns P≥ 0.05 بواسطة Brown-Forsythe و Welch ANOVA مع اختبار المقارنات المتعددة ل Dunnett. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل التكميلي 1: تقييم نتائج التحرير لاستنساخ خلية KO أحادية الخلية عن طريق اختبار T7EI المعدل. تم تنقية الحمض النووي الجيني لاستنساخ KO أحادي الخلية أو الخلايا غير المعدلة ، وتم تضخيم المنطقة المحيطة بالموضع المستهدف بواسطة تفاعل البوليميراز المتسلسل (PCR). تم خلط أمبليكونات كل استنساخ أحادي الخلية بنسبة 1: 1 مع تلك الموجودة في خلايا التحكم غير المعدلة. بعد تكوين heteroduplex وهضم T7EI ، تم تحليل المنتج عن طريق الرحلان الكهربائي لهلام الاغاروز. تنتج المستنسخة المعدلة أجزاء هضم متعددة (استنساخ A و B و C و D و E) ، بينما تظهر النسخ غير المعدلة نطاق WT واحد (استنساخ F و G). الرجاء النقر هنا لتنزيل هذا الملف.
الشكل التكميلي 2: استراتيجية بوابات قياس التدفق الخلوي لتحديد النسبة المئوية لخلايا PMA المعالجة بفيروس نقص المناعة البشرية -1 بفيروس نقص المناعة البشرية -1. باستخدام منطقة الانتثار الأمامي (FSC-A) مقابل منطقة التشتت الجانبي (SSC-A) ، يتم تحديد بوابة أولى للتخلص من الحطام وتحديد خلايا THP-1 المعالجة ب PMA. بعد ذلك ، يتم تحديد الخلايا المفردة باستخدام SSC-A مقابل مخطط SSC-H. أخيرا ، يتم الكشف عن الخلايا الإيجابية ل GFP باستخدام GFP-A مقابل FSC-H. الرجاء النقر هنا لتنزيل هذا الملف.
Discussion
هنا ، يتم وصف بروتوكول للحصول على تحرير ناجح بوساطة كريسبر لخط خلية THP-1. يعتمد النهج على نقل sgRNA / Cas9 RNPs المجمعة مسبقا عن طريق التثقيب الكهربائي / النواة. تم اختيار هذه الإستراتيجية للحد من التأثيرات غير المستهدفة التي يحتمل أن تنشأ عند التكامل بوساطة الفيروسات لشريط sgRNA / Cas9 ، مما ينتج عنه تعبير مستمر عن النوكلياز. تم اختيار العديد من sgRNAs التي تستهدف الجين محل الاهتمام لتحقيق تحرير موثوق وفعال ، مما يزيد من احتمالية إنشاء indels جينومية ، مما يؤدي إلى فقدان التعبير عن البروتين و KO32 الوظيفي. تم إيلاء عناية خاصة لإعدادات التثقيب الكهربائي لتحقيق النفاذية المثلى لغشاء الخلية ، وهو أمر مطلوب لضمان التوصيل الفعال ل sgRNA / Cas9 RNPs مع منع الموت المفرط للخلايا33. في هذا الصدد ، فإن المعلمة الحاسمة هي قوة ثقافة الخلية قبل التثقيب الكهربائي (أي رقم المرور المنخفض).
تم التحقق من صحة التحرير بوساطة CRISPR-Cas9 على المستوى الجيني باستخدام مقايسة الكشف عن عدم التطابق T7EI ، والتي توفر تقييما فعالا من حيث التكلفة وسريعا لعينات متعددة للاختيار المسبق قبل تسلسل Sanger. تم تطوير هذه الطريقة في البداية لتقدير كفاءة التحرير باستخدام sgRNA واحد من خلال مقارنة شدة الأمبليكونات كاملة الطول ومنتجات الانقسام34،35. وتجدر الإشارة إلى أنه يمكن اكتشاف indels الكبيرة (>70 نقطة أساس) التي يتم إنتاجها عند استخدام sgRNA متعدد من خلال مقارنة حركة شظايا الحمض النووي التي تم الحصول عليها عن طريق تضخيم تفاعل البوليميراز المتسلسل للمواقع المستهدفة من الخلايا المعدلة مقابل الخلايا غير المعدلة بواسطة الرحلان الكهربائي الهلامي القياسي. ومع ذلك ، قد يكون اختبار T7EI مفيدا للتأكد من وجود تسلسلات تحتوي على إندلات صغيرة (>1 نقطة أساس) إما في مجموعة خلايا أحادية النسيلة أو باستخدام البروتوكول المعدل ، في مجموعة خلايا أحادية النسيلة.
تم تأكيد وجود طفرات خارج الإطار على الهدف ، والتي تقاطع تسلسل الترميز ، من خلال نهج مباشر ، حيث يتم استخراج جينوم مجموعة الخلايا بأكملها واستخدامه كمصفوفة لتضخيم تفاعل البوليميراز المتسلسل (PCR). بعد ذلك ، تخضع الأمبليكون المنقى لتسلسل Sanger بكميات كبيرة ، يليه تحليل المعلوماتية الحيوية باستخدام أداة ICE. بشكل عام ، تتغلب هذه الطريقة على الحاجة إلى استنساخ منتجات تفاعل البوليميراز المتسلسل إلى بلازميد وتحويل منتجات الربط إلى بكتيريا لعزل مستعمرات خلية واحدة ، والتي يتم تسلسلها بشكل فردي.
بمجرد التحقق من صحة تعطيل الموضع المستهدف ، يلزم توصيف شامل لخط الخلية المحرر لإسناد نمط ظاهري معين بثقة إلى نقص البروتين محل الاهتمام واستبعاد التأثيرات غير المتوقعة الناجمة عن خارج الهدف ، ولكن أيضا التغييرات المستهدفة التي قد تؤثر على بنية و / أو وظيفة mRNA.
يجب تأكيد عدم وجود تعبير البروتين عن طريق النشاف المناعي و / أو التألق المناعي باستخدام جسمين مضادين يتعرفان على حواتم مختلفة. في الواقع ، يرتبط إدخال PTC في exon مبكر بالقرب من كودون ATG المتعارف عليه بخطر أحداث ITL ، والتي تنتج متغيرات بروتين مقطوعة بشكل نهائيN 36. وتجدر الإشارة إلى أن Tuladhar et al. أبلغ عن اكتشاف أنواع البروتين الشاذة في نسبة كبيرة (~ 50٪) من الخلايا المعدلة بواسطة CRISPR من أصل تجاري أو داخلي18،19. يعزى حدوث منتجات بروتينية جديدة إلى عمليات متنوعة ويحتمل أن تكون خاصة بنوع الخلية مثل تخطي الإكسون المحتوي على indel أو ظهور مواقع دخول الريبوسوم الداخلية (IRES) مما يتسبب في بدء تخليق البروتين في TIS البديل في اتجاه مجرى كودون ATG الكنسي. وهناك أيضا تلميحات إلى أن سجل المعاملات الدولي يقترن بالهروب من NMD في ظل ظروف معينة37،38. وبالمثل ، قد تفشل استراتيجيات تحرير الجينوم التي تستهدف إكسون متأخرا في تحفيز NMD لل mRNA المتحور الكامن وراء الاستئصال غير الفعال للتعبير الجيني20. أخيرا ، إذا كان Cas9 مسترشدا بالعديد من sgRNAs ، فقد تنشأ إعادة ترتيب كروموسومية معقدة ، بما في ذلك الانقلابات أو عمليات حذف شظايا الحمض النووي الكبيرة39 ، والتي قد تغير بنية الجينوم والتعبير عنه ، خاصة عندما يتم استهداف العناصر التنظيمية40.
لمزيد من التخفيف من تأثير نتائج تحرير الجينوم غير المقصودة ، هناك حاجة إلى دراسات وظيفية لتأكيد العلاقة بين فقدان التعبير عن البروتين والنمط الظاهري الذي يترتب على ذلك (أي النشاط المضاد للفيروسات). إذا تم استنساخ الخلايا المحررة ، فيجب مقارنة سلوك المستنسخة المتعددة فيما بينها ومع السكان متعدد النسيلة. سيؤدي إجراء تجارب الإنقاذ ، والتي تتكون من استعادة التعبير الجيني لعكس النمط الظاهري للخلايا المعدلة إلى نمط ظاهري WT ، أيضا إلى تعزيز الخصوصية واستبعاد الأحداث المحتملة خارج الهدف.
على الرغم من أن هذا البروتوكول قد تم تصميمه لتحرير خلايا THP-1 ، إلا أنه يوفر بنية عامة لسير عمل CRISPR-Cas9 KO الذي يمكن تكييفه مع خطوط الخلايا الأخرى. قد تكون هناك حاجة إلى ضبط معلمات التثقيب الكهربائي ، وإذا تم تطبيق جهد أعلى من 1800 فولت ، فيجب استخدام عازل إعادة التعليق T ، وفقا لتعليمات الشركة المصنعة. متغير آخر يجب مراعاته هو نسبة التركيز بين Cas9 و sgRNA أثناء تجميع RNP. على الرغم من أن نسبة 1: 9 تعمل بشكل جيد في هذه الحالة ، إلا أن تعديلها قد يحسن كفاءة التحرير في ظروف أخرى. أخيرا ، على الرغم من أنه ليس مفيدا هنا ، يجب اختبار استخدام الوسائط المشروطة لاستعادة الخلايا بعد التثقيب الكهربائي ، حيث يمكن أن يكون التأثير مختلفا مع خطوط الخلايا المختلفة.
Disclosures
جميع المؤلفين ليس لديهم تضارب في المصالح.
Acknowledgements
نحن ممتنون ل JP Concordet (MNHN ، U1154 / UMR7196 ، باريس) ، G. Bossis (IGMM ، مونبلييه) ، و D. Schlüter (كلية الطب في هانوفر ، ألمانيا) لمشاركة البروتوكولات والمناقشة. تلقى هذا المشروع تمويلا من برنامج Horizon 2020 للبحث والابتكار التابع للاتحاد الأوروبي (اتفاقية المنحة رقم 101017572 إلى AZ) و ANRS (منحة ECTZ162721 إلى AZ). يتم دعم البنية التحتية البحثية لنموذج الأمراض المعدية والعلاجات المبتكرة (IDMIT) من قبل "برنامج الاستثمار (PIA)" تحت ANR_11_INSB_0008 المرجعية.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
References
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved