Method Article
Generazione di colture primarie per l'imaging di cellule vive di cheratinociti
In questo articolo
Riepilogo
Questo protocollo delinea la coltura di cheratinociti appena ottenuti (passaggio 0) per l'uso nell'imaging di cellule vive.
Abstract
Il monitoraggio delle cellule staminali e del comportamento dei progenitori impegnati a livello di singola cellula nella pelle umana è stato impegnativo sia in vivo che in vitro. L'imaging di cellule vive ha permesso progressi significativi nella capacità di identificare le differenze tra le cellule staminali dei cheratinociti e il comportamento dei progenitori impegnati. L'imaging di cellule vive è un metodo in evoluzione e alquanto impegnativo per studiare il comportamento dei cheratinociti in vitro. Questo protocollo è stato sviluppato per coltivare cheratinociti a bassa densità di semina, consentendo la fotografia time-lapse a lungo termine e il monitoraggio del comportamento delle singole cellule. I cheratinociti del Passaggio 0 vengono coltivati a densità clonale e la fotografia time-lapse consente di documentare le singole divisioni cellulari e il momento in cui si sono verificate. Per la massima rilevanza biologica, i cheratinociti umani appena isolati vengono posti in vitro. Questo approccio si concentra sulla proliferazione. Tuttavia, questo protocollo può essere adattato per l'uso in altre applicazioni di imaging di cellule vive che misurano il comportamento delle singole cellule, come la misurazione della migrazione cellulare, della guarigione delle ferite e della motilità.
Introduzione
L'obiettivo di questo protocollo è quello di fornire la capacità di tracciare i singoli cheratinociti in coltura per un periodo relativamente lungo (diverse settimane) grazie alle basse densità di semina e alla fotografia time-lapse. L'imaging di cellule vive a bassa densità offre ai ricercatori l'opportunità di visualizzare il comportamento cellulare in vitro a livello di singola cellula. Sebbene non sia possibile nelle colture cellulari convenzionali, l'imaging su cellule vive consente di sviluppare alberi di lignaggio in tempo reale, senza l'uso di marcatura, da cui è possibile accertare informazioni riguardanti la cinetica di divisione, come la durata del ciclo cellulare e la proporzione di divisioni di proliferazione e differenziazione in una colonia. Consente inoltre lo studio della migrazione e della motilità cellulare. Può essere tecnicamente impegnativo, con aspetti che necessitano di ottimizzazione, a seconda del tipo di cella e dei dati che devono essere acquisiti. Altri laboratori hanno utilizzato cheratinociti neonatali e/o cheratinociti passati che sono più facili da coltivare1.
Tuttavia, le linee cellulari che subiscono passaggi ripetuti sono significativamente diverse nel comportamento e nella morfologia dal loro stato in vivo 2. Per la massima rilevanza biologica al fine di ottenere il comportamento più vicino a quello in vivo , vengono utilizzate cellule di passaggio 0. Nelle popolazioni di cheratinociti, è possibile distinguere tra la cellula staminale e il progenitore impegnato senza smistamento o marcature perché ci sono differenze distinte nel comportamento di divisione che possono essere osservate tramite l'imaging di cellule vive3.
Utilizziamo l'imaging di cellule vive per studiare la cinetica di proliferazione dei cheratinociti. Un approccio simile è stato utilizzato per studiare la motilità delle cellule di melanoma cutaneo cocoltivate con cheratinociti4, la migrazione cellulare aderente dei saggi scratch5 e il modo in cui gli inibitori ROCK influenzano la proliferazione dei cheratinociti6. Questo protocollo è specificamente progettato per la coltura di cellule per facilitare il tracciamento del lignaggio mediante imaging di cellule vive, sebbene possa essere adattato per altri scopi.
Questo protocollo delinea l'isolamento dei cheratinociti del passaggio 0 ai fini dell'imaging di cellule vive, discute le complicanze che possono insorgere e fornisce considerazioni che possono richiedere modifiche al protocollo (Figura 1).
Protocollo
Per l'uso di tessuti umani è necessaria l'approvazione del Comitato per la ricerca umana dell'istituto. I campioni umani per la ricerca sono spesso ottenuti da fonti commerciali (ad esempio, ATCC, tecnologia cellulare Lifeline e Zen-Bio). I nostri studi utilizzano tessuti scartati al momento degli interventi chirurgici o campioni di biopsia cutanea prelevati appositamente per scopi di studio. Queste situazioni richiedono diverse approvazioni e procedure di autorizzazione. Tutti i tessuti sono ottenuti dopo l'approvazione del Comitato per la ricerca umana dell'UCSF, utilizzando il consenso informato scritto e seguendo i principi della Dichiarazione di Helsinki.
1. Separazione dell'epidermide dal derma
- Assicurarsi che siano ottenute le approvazioni appropriate per utilizzare tessuti umani per lo studio.
- Prelevare il campione di pelle e trasportarlo in un contenitore isolato con un impacco di ghiaccio. Collocare il campione in un terreno di raccolta contenente una concentrazione di 5 volte superiore a quella di penicillina, streptomicina e amfotericina.
NOTA: La quantità richiesta dipenderà dall'applicazione. Nella nostra esperienza, circa 1.000.000 di cheratinociti per cm2 sono ottenuti da pelle adulta e invecchiata. Tuttavia, questo può variare in modo significativo per ogni campione.- Posizionare i prepuzi neonatali in terreni di coltura cellulare e iniziare la procedura di isolamento dei cheratinociti non più tardi di 48 ore dopo la circoncisione per ridurre al minimo la morte cellulare. Conservare il fazzoletto a 4 °C fino alla lavorazione. L'elaborazione di campioni adulti e invecchiati inizia in genere entro poche ore dalla raccolta.
- Ottieni l'accesso a una cabina di biosicurezza con apparecchiature di livello BS2 progettate per lavorare con agenti che causano malattie per ingestione, mucosa o esposizione percutanea7. Assicurarsi che la cabina di biosicurezza sia sterilizzata ai raggi UV per almeno 15 minuti e che tutte le superfici siano spruzzate con alcol al 70% prima dell'uso.
- Indossare i guanti, spruzzare il contenitore di raccolta con alcol al 70% e posizionare il contenitore nella cappa aspirante. Posiziona un tampone di raffreddamento sotto il fazzoletto per evitare che si riscaldi a temperatura ambiente e per mantenerne l'integrità. Se il fazzoletto è estremamente grande, tagliarne una parte per la lavorazione e mantenere il resto umido nel contenitore di raccolta.
NOTA: Poiché molti degli esemplari invecchiati sono grandi strati di pelle addominale o mammaria da interventi chirurgici, ci vuole tempo per tagliare ed elaborare il tessuto. Il fazzoletto non deve mai essere posizionato direttamente sul cuscinetto, poiché il freddo estremo può causarne il congelamento. Invece, dovrebbe essere posizionato su una superficie intermedia, come una lastra di marmo. - Posizionare due piastre di Petri da 100 mm x 15 mm e un bisturi chirurgico sterile #23 nel cappuccio.
- Aprire le piastre di Petri e posizionarle sulla superficie dell'armadio in modo che le superfici interne di ciascuna piastra siano rivolte verso l'alto. Aggiungere 10 ml di clorexidina gluconato al 10% in una delle piastre di Petri. Aggiungere 10 ml di 5x penicillina/streptomicina/amfotericina/gentamicina nella soluzione salina bilanciata di Hank (HBSS; 5x) ad altre 2 metà della piastra di Petri (vedi Tabella dei materiali).
- Preparare una piastra a 6 pozzetti per la fase Dispase. Per il tessuto neonatale, utilizzare 3 mL di Dispasi per pozzetto. Per campioni di tessuto invecchiati, utilizzare 4 ml. L'uso di quantità inadeguate di Dispase per il volume del tessuto può causare difficoltà a separare l'epidermide dal derma.
- Usa un bisturi per tagliare il tessuto in pezzi più piccoli in modo che possa essere meglio penetrato dalla Dispae. La dispasi penetra solo fino a 5 mm dai bordi del tessuto, quindi tagliare lunghe strisce di tessuto larghe 0,5-1 cm. Se non tagliato abbastanza piccolo, non si verificherà la separazione dell'epidermide dal derma.
- Taglia il tessuto più spesso in pezzi più piccoli. Tagliare i prepuzi neonatali in 3 o 4 pezzi (~5 mm x 7 mm). Utilizzare un massimo di due prepuzi interi per pozzetto di Dispase. I lembi addominali superficiali tendono ad essere molto più sottili, con tessuto sottocutaneo minimo o assente, tagliarli in strisce più lunghe (~5 mm x 15 mm).
- Posizionare un massimo di quattro strisce per pozzetto di Dispase. Se il tessuto ottenuto ha molto grasso sottocutaneo attaccato, tagliare il tessuto sottocutaneo per aiutare Dispase a penetrare completamente nel tessuto. Se il tessuto è tagliato troppo piccolo, potrebbe essere necessario un microscopio da dissezione per determinare quale lato è epidermico e quale lato è dermico mentre si stacca l'epidermide. Tirare il lato sbagliato della pelle può provocare un ulteriore trauma tissutale.
- Posizionare il tessuto con il lato epidermico rivolto verso il basso nella prima piastra di Petri preparata al punto 1.6, contenente il 10% di clorexidina gluconato, per 10-20 s Quindi trasferire il tessuto con il lato epidermico verso il basso nella seconda piastra di Petri, contenente 5x penicillina/streptomicina/amfotericina (PSA) con HBSS, e agitare per 10-20 s. Trasferire il tessuto con il lato epidermico rivolto verso il basso nella terza piastra di Petri, sempre contenente 5x PSA con HBSS, e agitare per altri 10 s. È accettabile l'elaborazione di più campioni con la stessa soluzione.
- Trasferire il tessuto con il lato epidermico verso il basso nella piastra Dispase a 6 pozzetti preparata al punto 1.7.
NOTA: Consigliamo il lato epidermico rivolto verso il basso secondo la nostra convenzione. Altri protocolli raccomandano il derma verso il basso8. Non tutte le Dispase sono ugualmente efficaci. Il peeling dell'epidermide dalla pelle invecchiata si è rivelato difficile con alcuni prodotti, anche a concentrazioni fino a 4 volte il livello raccomandato dal produttore. Ciò ha comportato la perdita di tempo e di campioni. Le aziende utilizzano unità non standardizzate per Dispase, rendendo difficile identificare le differenze tra le concentrazioni tra le marche. A causa di queste sfide, si consiglia vivamente l'uso di uno dei marchi Dispase elencati nella Tabella dei materiali . - Etichettare la piastra a 6 pozzetti contenente la Dispasi e i campioni e trasferirla a 4 °C. Incubare campioni neonatali per 16-18 ore in Dispase e campioni adulti/anziani per 24 ore.
NOTA: Esiste un protocollo alternativo di 2 ore che può essere utilizzato per i campioni, in cui il campione viene posto in Dispasi a 37 °C per 2 ore, quindi l'epidermide viene sbucciata. Sfortunatamente, questo non sempre funziona e l'integrità del tessuto può essere compromessa dopo l'incubazione, con conseguente perdita dell'esemplare. L'incubazione notturna è preferita quando possibile.
2. Isolamento dei cheratinociti
- Mettere lo 0,05% di tripsina-EDTA (o TrypLE), la soluzione neutralizzante della tripsina e i terreni per cheratinociti in un bagno d'acqua a 37 °C 20 minuti prima di iniziare la serie successiva di passaggi
- Rimuovere una piastra a 6 pozzetti da 4 °C, spruzzarla con etanolo al 70% e collocarla in una cappa di biosicurezza preparata (vedere il passaggio 1.3).
- Posizionare le pinze dentate sterili e le pinze non dentate in una provetta conica da 50 mL contenente 20 mL di etanolo al 70%. Se inizialmente si trova all'esterno della cappa, spruzzare una pinza con etanolo al 70% prima di inserirla nella cappa.
- Usando una pinza sterilizzata chimicamente, afferrare un pezzo di tessuto e metterlo su una superficie sterile con il lato epidermico rivolto verso l'alto (utilizzare una capsula di Petri o l'interno della metà superiore della piastra a 6 pozzetti). Tenere la parte dermica del tessuto con una pinza dentata usando la mano non dominante e contemporaneamente afferrare saldamente l'epidermide con la pinza non dentata ed estrarla da un bordo all'altro. Dovrebbe staccarsi senza intoppi come un unico pezzo.
- Posizionare l'epidermide all'interno del labbro di una provetta conica da 15 mL. Ogni provetta conica da 15 ml deve avere un'epidermide di massimo due prepuzi.
- Lavora in modo efficiente; Se il tessuto si secca, la resa sarà compromessa. Se la Dispasi non penetra adeguatamente nel tessuto, potrebbero esserci difficoltà a staccare l'epidermide. In tal caso, tirare/raschiare l'epidermide dal derma sottostante. Ciò si traduce anche in una resa molto più bassa e non è raccomandato.
- Aggiungere 4 mL di tripsina-EDTA allo 0,05% nella provetta conica da 15 mL, quindi chiudere e capovolgere la provetta assicurandosi che tutta l'epidermide sia sospesa nella tripsina e non bloccata sul lato della provetta/del coperchio. Mettere a bagnomaria a 37 °C per 10 min. Agitare le cellule ogni 2 minuti a mano.
NOTA: La tripsinizzazione per più di 10 minuti può comportare una minore resa della cella. Inoltre, se l'integrità delle proteine della superficie cellulare è un problema, utilizzare il TrypLE in quanto è più specifico per il suo sito di scissione, con conseguente minor danno alle cellule e ha dimostrato di non ridurre significativamente l'espressione dell'antigene di superficie rispetto alla tripsina-EDTA9. Inoltre, il bagnomaria è una fonte comune di contaminazione, quindi assicurati che venga pulito regolarmente. Cambiare regolarmente l'acqua con acqua deionizzata contenente la soluzione aquaguard-2. - Inserire una provetta conica da 50 mL nella camera di biosicurezza. Svitare il coperchio e posizionare un filtro cellulare da 100 μm sull'apertura del tubo conico.
- Rimuovere la provetta conica da 15 ml contenente il campione digerito dal bagnomaria, asciugarla accuratamente e spruzzarla con alcol al 70% prima di riporla nella camera di biosicurezza. Battere il tubo conico 3 volte contro la superficie della cappa aspirante, quindi capovolgerlo 6 volte. Ripetere il ciclo di maschiatura-inversione 3 volte.
- Aggiungere 6 mL di soluzione neutralizzante della tripsina nella provetta conica. Versare attraverso il colino cellulare nella provetta conica da 50 mL dal passaggio 2.7.
- Sciacquare la provetta conica da 15 mL contenente il campione con 5 mL di soluzioni neutralizzanti la tripsina (TNS). Versare attraverso un colino cellulare nella provetta conica da 50 mL. Centrifugare a 300 x g per 5 min.
- Estrarre immediatamente dalla centrifuga e pipettare il surnatante per evitare la perdita di cellule nel terreno. Dovrebbe rimanere solo il pellet con una piccola raccolta di liquido. Può essere difficile vedere il pellet da campioni di tessuto più piccoli.
NOTA: C'è tripsina presente nel surnatante, quindi è meglio rimuoverne il più possibile senza perdere il pellet. - Risospendere il pellet di cheratinociti in 2 mL di terreno di coltura per cheratinociti.
NOTA: Ci sono due considerazioni qui. Innanzitutto, quali media utilizzare. A tale scopo, si consiglia un terreno privo di siero e collagene. Storicamente, lo standard per la coltura cellulare di cheratinociti è stato il Modified Eagle's Medium (DMEM) di Dulbecco (DMEM) +10% di siero fetale bovino (FBS) + F-12 di Ham su uno strato di alimentazione di fibroblasti (3t3-J2s)10. Tuttavia, con l'avvento dei terreni privi di siero e la disponibilità di collagene, molte altre possibilità rimangono praticabili (vedi discussione)11. La seconda considerazione è se l'uso di antibiotici nel terreno di coltura sia appropriato per l'applicazione di imaging di cellule vive. Ciò dipenderà dal tempo di imaging, dalla durata e dall'esperienza con il sistema di imaging. Le colonie vengono visualizzate per più settimane utilizzando un sistema di imaging condiviso, rendendo l'uso di antibiotici un approccio preferito nonostante il potenziale impatto sulle colture (vedi discussione). Durante la risospensione vengono utilizzati penicillina, streptomicina, amfotericina B e terreno cheratinocitario contenente gentamicina, seguito da un passaggio a terreni contenenti penicillina/streptomicina dopo il cambio iniziale del terreno. Questo metodo ha efficacemente impedito la contaminazione durante l'imaging esteso di cellule vive. - Contare le celle e seminare la piastra (vedere la discussione per i commenti sulla selezione della densità di semina). Per garantire una distribuzione uniforme della semina, prima della piastratura, risospendere le cellule dei cheratinociti nella quantità totale di terreno da piastrare. Dopo una leggera agitazione per inversione, agitare leggermente per 1-2 s e impiattare. Quindi, spostare la micropiastra a croce (su-giù, sinistra-destra) 3 volte nell'incubatrice.
- Dopo 24 ore, posizionare la piastra nell'incubatore del sistema di imaging di cellule vive. Esegui l'imaging di cellule vive a 10x con immagini scattate ogni 20 minuti. Posizionare i campioni a 37 °C e 5% di CO2 nell'incubatore collegato al microscopio.
Risultati
Date le molteplici variabili che possono essere manipolate in questo metodo e che alla fine si traducono in una varietà di risultati, qui descriviamo i passi falsi comuni e i risultati che ne derivano, oltre a fornire risultati esemplari e le circostanze dietro di essi.
Come con qualsiasi tecnica di coltura cellulare, la contaminazione è una possibilità (Video supplementare 1). Insieme a un'attenta tecnica di sterilizzazione, prendere in considerazione l'uso di antibiotici nei terreni, soprattutto se si coltiva per lunghi periodi di tempo. Una contaminazione frequente è stata osservata quando le cellule coltivate sono state trasportate in un sistema di imaging in un'altra posizione senza l'uso di antibiotici. Si prega di consultare la sezione di discussione per gli aspetti negativi dell'uso di antibiotici.
Come indicato nel protocollo, la selezione dei media è fondamentale. Inizialmente è stato utilizzato DMEM con il 10% di FBS e F-12 di Ham su uno strato di alimentazione di fibroblasti. Tuttavia, ci sono stati problemi con questa selezione (Video supplementare 2). La densità di semina iniziale dei fibroblasti utilizzati era troppo alta e, con il passare del tempo, i fibroblasti sembravano espandersi, oscurando la vista dei cheratinociti. La riduzione della densità di semina dei fibroblasti ha risolto il problema dell'oscuramento dei fibroblasti. Tuttavia, in aggiunta, i cheratinociti sembravano differenziarsi più rapidamente rispetto al terreno privo di siero, e questo ha portato ad aree non uniformi di cheratinociti, che erano difficili da tracciare ai fini del tracciamento del lignaggio (Video supplementare 3). Altre questioni relative ai media basati su DMEM vengono approfondite nella discussione.
Dopo aver riscontrato problemi con l'F-12 tradizionale di DMEM/FBS/Ham, è stato scelto come alternativa un terreno privo di siero. Con terreni di coltura integrati con HKGS e cheratinociti a densità clonale, c'erano colonie uniformi che erano tracciabili (Video supplementare 4, Video supplementare 5). Con i terreni privi di siero, la differenziazione avviene più lentamente (probabilmente a causa del calcio più basso) e molte cellule aderenti non si dividono mai ma non si staccano (come fanno in altri mezzi; Video supplementare 4, Video supplementare 3).
La densità di semina può influire sulla capacità di tracciare le cellule. Troppe cellule possono comportare l'incapacità di tracciare le cellule in modo accurato e troppo poche cellule, al contrario, provocano una crescita insufficiente. Molti produttori di terreni privi di siero raccomandano di abbinare il terreno definito con il collagene, consentendo densità di semina inferiori (Figura 2). Vedi la discussione per ulteriori informazioni su questo argomento.
La differenziazione dei cheratinociti è evidente morfologicamente dalle cellule grandi e appiattite e dalla morfologia impilata. Vedere il video supplementare 2. La valutazione della differenziazione terminale è una mancanza di divisione su 48 h 3,12,13.
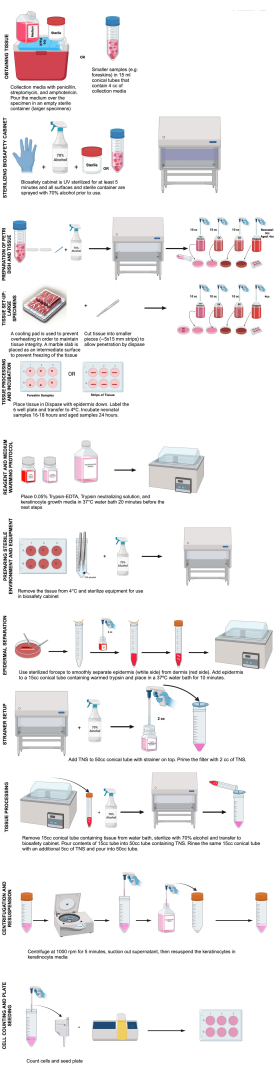
Figura 1: Fasi di isolamento dei cheratinociti. Questo diagramma illustra le fasi dell'isolamento dei cheratinociti come descritto in questo protocollo. Creato nel BioRender.com Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Aderenza cellulare e formazione di colonie. La differenza nell'aderenza cellulare e nella formazione di colonie a 2 giorni e 23 ore con e senza collagene. Il collagene migliora significativamente l'aderenza iniziale dei cheratinociti. Creato nel BioRender.com Clicca qui per visualizzare una versione più grande di questa figura.
Video supplementare 1: Contaminazione della colonia di cheratinociti. Immagini scattate ogni 20 minuti mediante imaging di cellule vive raccolte in video. Clicca qui per scaricare questo file.
Video supplementare 2: Crescita rapida dei cheratinociti utilizzando l'alimentatore tradizionale e i terreni basati su DMEM. Immagini scattate ogni 20 minuti mediante imaging di cellule vive raccolte in video. Clicca qui per scaricare questo file.
Video supplementare 3: Aumento della differenziazione dei cheratinociti utilizzando alimentatori tradizionali e terreni basati su DMEM. Immagini scattate ogni 20 minuti mediante imaging di cellule vive raccolte in video. Clicca qui per scaricare questo file.
Video supplementare 4: Colonia di cheratinociti tracciabile. Immagini scattate ogni 20 minuti mediante imaging di cellule vive raccolte in video. Clicca qui per scaricare questo file.
Video supplementare 5: Colonia di cheratinociti tracciabile. Immagini scattate ogni 20 minuti mediante imaging di cellule vive raccolte in video. Clicca qui per scaricare questo file.
Discussione
Qui, abbiamo dettagliato un metodo per la coltura primaria di cheratinociti umani ai fini dell'imaging di cellule vive. Questo metodo adatta le tecniche di coltura cellulare esistenti che utilizzano la placcatura a bassa densità e la coltura a lungo termine per consentire il successo degli studi di imaging di cellule vive. Il metodo preferito per la dissociazione di questo tipo di cellule prevede una digestione enzimatica in due fasi, che ha dimostrato di portare a cheratinociti, di cui il 3%-4% è in grado di diventare unità formanti colonie14. La crescita dei cheratinociti adulti e invecchiati in vitro è impegnativa, poiché molti campioni semplicemente non riescono a crescere.
Più passaggi sono fondamentali per l'obiettivo di massimizzare i raccolti per migliorare la crescita. Si dovrebbero isolare queste cellule il prima possibile dopo la raccolta dei tessuti. Viene mantenuta una linea di comunicazione diretta con i chirurghi e i campioni vengono raccolti immediatamente dopo essere stati ottenuti dal paziente. Sono state formulate diverse altre raccomandazioni sui modi per preservare l'integrità dei tessuti utilizzando questo metodo. Mantieni i tessuti freschi, mantieni i tessuti nei terreni di raccolta ed elabora i tessuti in modo efficiente. Prendi in considerazione la possibilità di raggruppare campioni, se piccoli, per massimizzare la crescita in vitro. Utilizzare sempre i reagenti per almeno 20 minuti. Utilizzare Dispase di provata efficacia e considerare il taglio di strisce di tessuto più piccole o l'estensione del periodo di incubazione se ci sono problemi a staccare l'epidermide. Utilizzare il minor tempo possibile per la digestione con un trattamento enzimatico a scelta e neutralizzare immediatamente dopo l'incubazione. Risospendere il pellet il prima possibile dopo la centrifugazione. Anche dopo aver fatto tutto questo, aspettatevi che ci saranno occasionalmente campioni umani adulti e anziani che non crescono, anche in quelle che sembrano essere le circostanze più ideali.
L'obiettivo è quello di placcare i cheratinociti a densità clonale o vicino a quella e tracciarli utilizzando l'imaging di cellule vive; Questo metodo si allinea bene con questi obiettivi. Se l'obiettivo è isolare i cheratinociti e osservare la motilità in diverse condizioni o per un test funzionale come un test scratch, un metodo alternativo da considerare è la tecnica dell'espianto cutaneo. Questo approccio privo di enzimi per isolare i cheratinociti può portare a rese cellulari più elevate 14,15,16.
Le modifiche nei terreni utilizzati possono essere importanti per l'imaging di cellule vive. Il terreno selezionato per promuovere la crescita dei cheratinociti avrà un impatto su tutte le fasi dell'esperimento. DMEM, 10% FBS e F-12 di Ham con celle di alimentazione 3T3-J2 hanno consentito densità di semina inferiori, ma hanno comportato diversi problemi. Le cellule di alimentazione, a meno che non vengano utilizzate a bassa densità, oscuravano il piano visivo su cui si trovavano i cheratinociti, rendendo più difficile il tracciamento. Utilizzando il DMEM, i fibroblasti sono rimasti per tutta la durata dell'imaging di cellule vive (~2 settimane). Inoltre, ci sono altri problemi con i terreni a base di siero che sono importanti, tra cui la promozione della differenziazione (che impedisce l'uso del software IncuCyte per l'imaging di cellule vive a causa della mancanza di un aspetto uniforme delle cellule) e il rischio di possibile contaminazione dal siero17. Nei terreni privi di siero, è necessaria una densità di semina più elevata rispetto al DMEM, poiché aderiscono meno cellule. Le combinazioni di 154 CF + HKGS, Epilife + HKGS ed Epilife + S7 si sono rivelate efficaci. Come promemoria, usa sempre un terreno fresco quando usi integratori per la crescita poiché si degradano nel tempo. Testare sempre il terreno con almeno tre campioni quando si inizia un nuovo protocollo per verificare che possa supportare le cellule in coltura. I produttori di terreni privi di siero raccomandano spesso l'uso di collagene di coda di ratto (tipo 1)18. Il collagene aiuta l'attaccamento iniziale (consentendo una minore densità di semina) e la proliferazione19.
Insieme al tipo di terreno, un altro fattore che può influenzare i risultati è la densità di semina e la distribuzione cellulare. La coltura dei cheratinociti è aiutata dall'interazione cellula-cellula per la proliferazione e, quindi, densità di semina più elevate si traducono in una crescita più prolifica20. Terreni ed età cellulari diversi richiedono densità di semina variabili per ottenere la crescita clonale. Gli esperimenti vengono solitamente eseguiti con più densità di semina per ottenere tassi di crescita/dimensioni delle colonie appropriati per l'applicazione di imaging di cellule vive desiderata. Le densità per la crescita clonale e il tracciamento del lignaggio possono essere diverse da quelle necessarie per altre applicazioni. Assicurarsi che la distribuzione della semina sia uniforme prima di consentire alle cellule di aderire.
L'uso di antibiotici durante l'isolamento e la crescita è controverso. In precedenza è stato dimostrato che gli antibiotici di uso comune possono avere un effetto inibitorio sulla proliferazione dei cheratinociti21. Detto questo, molti scelgono di non usare antibiotici dopo aver pulito la pelle inizialmente prima della lavorazione. Questa applicazione di imaging di cellule vive comporta diverse settimane di crescita delle colonie, aumentando il rischio di contaminazione. Il rischio di perdere settimane di tempo/campioni preziosi a causa della contaminazione deve essere soppesato con il rischio di una possibile diminuzione della proliferazione dei cheratinociti. Le applicazioni più brevi potrebbero non richiedere antibiotici, mentre periodi prolungati di imaging di cellule vive lo fanno.
Una limitazione della coltura di cellule per l'imaging di cellule vive, come per altre tecniche in vitro, è l'incognita rilevanza in vivo . Inoltre, l'uso di cellule appena ottenute rispetto a quelle passate può produrre risultati diversi man mano che le caratteristiche cellulari si evolvono all'aumentare del numero di passaggidi 20. Inoltre, i risultati possono essere influenzati dall'ambiente estrinseco uniforme. Questo ambiente uniforme può rappresentare un punto di forza o un limite, a seconda degli obiettivi dello studio, in particolare quando si distinguono tra cambiamenti intrinseci ed estrinseci nel comportamento cellulare.
C'è una pletora di fattori da considerare quando si coltivano cheratinociti ai fini dell'imaging di cellule vive. Il metodo presentato può aiutare a semplificare il processo. Essere in grado di eseguire con successo l'imaging di cellule vive di cheratinociti in studi futuri consentirà una comprensione più granulare del comportamento proliferativo e differenziativo dei cheratinociti e dei meccanismi di mantenimento epidermico.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Un ringraziamento speciale a T. Richard Parenteau, MD/PhD, e Alexandra Charruyer, PharmD/PhD, per avermi insegnato come coltivare i cheratinociti. Grazie a Merisa Piper MD per averci fornito campioni da cui isolare i cheratinociti. Un ringraziamento finale a Michael Rosenblum, MD, per averci permesso di accedere al suo sistema di imaging di cellule vive per completare gli esperimenti.
Materiali
| Name | Company | Catalog Number | Comments |
| .05% Trypsin-EDTA (1X), 100 mL | Gibco | 25300-054 | |
| 100 um cell filter | Corning | 352360 | |
| 15 cc Falcon | Corning | 352096 | |
| 50 cc conical Falcon | Corning | 352070 | |
| 6 well microplate | Corning | 3516 | |
| 70% reagent alcohol, 4 L | VWR Chemicals | BDH1164-4LP | Can alternatively dilute your own |
| Amphotericin B, 50 ml | Corning | 30-003-CF | Dilute to 5X (50 ug/mL) |
| Dipase, 100 ml | Corning | 354235 | Dilute 1:1 with HBSS, add 1x gentamycin, filter sterilize |
| Epilife, 50 mL | Gibco | MEP1500CA | Add HKGS, consider antibiotics |
| Fetal Bovine Serum Value Heat Inactivated FBS, 500 ml | Gibco | A52568-01 | FBS |
| Forceps | |||
| Gentamicin, 10 ml | Gibco | 15750-060 | Dilute to 50 ug/ml for 5X |
| Hank's Balanced Salt Solution (1X), 500 ml | Gibco | 14170-112 | (HBSS) |
| HBSS 5X PSAG | This is HBSS + 5X concentrations of Pen/Strep/Ampho/Gent | ||
| Hibiclens, Gallon | Molnlycke | 57591 | Dilute to 10% using deionized H20 |
| Human Keratinocyte Growth Serum, 5 mL | Gibco | S-001-5 | Added to epilife |
| Penicillin/Streptomycin, 100 ml | Corning | 30-002-Cl | Dilute to 5X (comes in 100x stock) for 5X PSA - 1X for media changes |
| Petri Dish 100 mm x 15 mm | Fisher Scientific | FB0875713 | |
| Scalpel Blade NO 23 | VWR | 76457-480 | |
| TrypLE | Gibco | 12604021 | A less caustic alternative to regular Trypsin |
| Trypsin Neutralizing Solution | (TNS), this is HBSS with 10% FBS (some use less serum, 5%) |
Riferimenti
- Mateu, R., et al. Functional differences between neonatal and adult fibroblasts and keratinocytes: Donor age affects epithelial-mesenchymal crosstalk in vitro. Int J Mol Med. 38 (4), 1063-1074 (2016).
- Handl, J., Čapek, J., Majtnerová, P., Báčová, J., Roušar, T. The effect of repeated passaging on the susceptibility of human proximal tubular HK-2 cells to toxic compounds. Phy Res Aca Sci Boh. 69 (4), 731-738 (2020).
- Xiao, T., et al. Short cell cycle duration is a phenotype of human epidermal stem cells. Stem Cell Res The. 15 (1), 76 (2024).
- Noujarède, J., et al. Sphingolipid paracrine signaling impairs keratinocyte adhesion to promote melanoma invasion. Cell Rep. 42 (12), 113586 (2023).
- Sun, M., et al. An image-based dynamic high-throughput analysis of adherent cell migration. Bio-prol. 11 (6), e3957 (2021).
- Chapman, S., McDermott, D. H., Shen, K., Jang, M. K., McBride, A. A. The effect of Rho kinase inhibition on long-term keratinocyte proliferation is rapid and conditional. Stem Cell Res The. 5 (2), 60 (2014).
- U.S. Department of Health and Human Services. . Biosafety in microbiological and biomedical laboratories. , (2020).
- Poumay, Y., Roland, I. H., Leclercq-Smekens, M., Leloup, R. Basal detachment of the epidermis using dispase: tissue spatial organization and fate of integrin alpha 6 beta 4 and hemidesmosomes. J Inv Der. 102 (1), 111-117 (1994).
- Tsuji, K., et al. Effects of different cell-detaching methods on the viability and cell surface antigen expression of synovial mesenchymal stem cells. CellTra. 26 (6), 1089-1102 (2017).
- Rheinwald, J. G., Green, H. Serial cultivation of strains of human epidermal keratinocytes: the formation of keratinizing colonies from single cells. Cell. 6 (3), 331-343 (1975).
- Boisseau, A. M., et al. Production of epidermal sheets in a serum free culture system: a further appraisal of the role of extracellular calcium. J Der Sci. 3 (2), 111-120 (1992).
- Roshan, A., et al. Human keratinocytes have two interconvertible modes of proliferation. Nat Cell Bio. 18 (2), 145-156 (2016).
- Nanba, D., et al. EGFR-mediated epidermal stem cell motility drives skin regeneration through COL17A1 proteolysis. J Cell Bio. 220 (11), e202012073 (2021).
- Ścieżyńska, A., et al. Isolation and culture of human primary keratinocytes-a methods review. Exp Der. 28 (2), 107-112 (2019).
- Guo, A., Jahoda, C. A. B. An improved method of human keratinocyte culture from skin explants: cell expansion is linked to markers of activated progenitor cells. Exp Derm. 18 (8), 720-726 (2009).
- Orazizadeh, M., Hashemitabar, M., Bahramzadeh, S., Dehbashi, F. N., Saremy, S. Comparison of the enzymatic and explant methods for the culture of keratinocytes isolated from human foreskin. Bio Rep. 3 (3), 304-308 (2015).
- Usta, S. N., Scharer, C. D., Xu, J., Frey, T. K., Nash, R. J. Chemically defined serum-free and xeno-free media for multiple cell lineages. Ann of tra med. 2 (10), 97 (2014).
- Lenihan, C., Rogers, C., Metcalfe, A. D., Martin, Y. H. The effect of isolation and culture methods on epithelial stem cell populations and their progeny-toward an improved cell expansion protocol for clinical application. Cyt. 16 (12), 1750-1759 (2014).
- Bernstam, L. I., Vaughan, F. L., Bernstein, I. A. Keratinocytes grown at the air-liquid interface. In Vitro Cell Dev Biol. 22 (12), 695-705 (1986).
- Ponce, L., et al. Isolation and cultivation of primary keratinocytes from piglet skin for compartmentalized co-culture with dorsal root ganglion neurons. J Cell Bio. 2 (2), 93-115 (2017).
- Nygaard, U. H., et al. Antibiotics in cell culture: friend or foe? Suppression of keratinocyte growth and differentiation in monolayer cultures and 3D skin models. Exp Der. 24 (12), 964-965 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon