Method Article
Imagerie de cellules vivantes avec photographie en accéléré pour étudier la cinétique de prolifération des kératinocytes épidermiques
Dans cet article
Résumé
Ici, nous fournissons une méthode d’analyse d’imagerie cellulaire vivante qui peut être utilisée pour suivre manuellement les lignées de kératinocytes de passage 0 et qui permet la collecte de paramètres de prolifération, y compris le devenir de la division cellulaire et la durée du cycle cellulaire.
Résumé
L’imagerie de cellules vivantes est une méthode évolutive et quelque peu difficile pour étudier le comportement des kératinocytes in vitro. Historiquement, le comportement de division des kératinocytes a été étudié à l’aide de méthodes telles que l’analyse clonale, l’immunocoloration et l’analyse du cycle cellulaire. Aucune de ces méthodes ne permet d’analyser en temps réel le comportement des kératinocytes au niveau de la cellule unique. Au cours de la dernière décennie, des groupes ont utilisé l’imagerie de cellules vivantes pour identifier les cellules souches kératinocytaires et les progéniteurs engagés sans qu’il soit nécessaire de les marquer. Des différences ont été identifiées dans le comportement de division de chaque groupe respectif, le taux de différenciation terminale et la durée du cycle cellulaire. Ici, une méthode d’imagerie de cellules vivantes de kératinocytes avec photographie en accéléré et son analyse est décrite. L’utilisation de kératinocytes non passés est recommandée pour que cette méthode imite le plus fidèlement possible le comportement in vivo . L’imagerie des cellules vivantes offre une capacité unique d’étudier le comportement des cellules souches et des progéniteurs engagés au niveau de la cellule unique et de déterminer le destin de division, la durée du cycle cellulaire, ainsi que d’autres paramètres de prolifération.
Introduction
La possibilité de visualiser les populations cellulaires in vitro en temps réel pendant de longues périodes est un avantage unique de l’imagerie des cellules vivantes. L’imagerie des cellules vivantes permet d’évaluer la motilité, la migration et la prolifération des cellules au niveau de la cellule unique. L’objectif de ce protocole est d’optimiser la visualisation des cultures de kératinocytes via la photographie en accéléré, en produisant des vidéos qui peuvent ensuite être suivies manuellement pour obtenir des données granulaires sur le comportement cellulaire.
Nous nous concentrons sur la cinétique de prolifération. À partir de l’analyse des vidéos d’imagerie de cellules vivantes, des arbres de lignage peuvent être élucidés et le temps entre les divisions (un indicateur de la durée du cycle cellulaire), ainsi que les proportions de divisions qui conduisent à une division ultérieure par rapport à la différenciation des cellules filles peuvent être évalués.
Il existe une variabilité substantielle d’un donneur à l’autre lorsqu’il s’agit de kératinocytes primaires et de fréquentes tentatives infructueuses de propagation cellulaire. Pour cette raison, de nombreux chercheurs choisissent d’utiliser des kératinocytes hautement prolifératifs tels que les cellules HaCaT ou les kératinocytes néonatals, souvent après qu’ils aient subi plusieurs passages in vitro1. La culture de kératinocytes primaires à partir de peau adulte ou âgée à des fins de traçage de la lignée peut s’avérer difficile. Cependant, il existe des problèmes avec l’utilisation de cellules passées à partir de lignées cellulaires ou de prépuce masculin. Le passage répété aboutit à des cellules qui sont significativement différentes de leur état in vivo 2. De plus, il a été démontré que les cellules HaCaT réagissent différemment des kératinocytes primaires dans plusieurs dosages 3,4,5. Pour utiliser les cellules qui ressemblent le plus à leurs homologues in vivo, on utilise des kératinocytes de passage 0 provenant de donneurs humains adultes. Les cellules souches kératinocytaires et les progéniteurs engagés présentent des différences distinctes de comportement, ce qui permet de distinguer les colonies de l’une ou l’autre population par imagerie de cellules vivantes6. Cette capacité relativement nouvelle à visualiser le comportement de kératinocytes uniques à long terme n’a été utilisée que dans quelques études précédentes utilisant des techniques similaires 6,7,8. Ce protocole décrit l’imagerie de cellules vivantes de kératinocytes primaires à l’aide du système d’analyse de cellules vivantes IncuCyte S3. À partir des arbres de lignage construits, il est possible de déterminer le type de colonie (cellule souche ou progéniteur engagé), ainsi que la durée du cycle cellulaire et la proportion de divisions de différenciation.
Protocole
Cette étude a été réalisée conformément à la Déclaration d’Helsinki. Tous les tissus humains ont été obtenus après l’approbation du comité d’examen institutionnel (IRB) de l’Université de Californie à San Francisco (UCSF), et le consentement a été obtenu pour tous les tissus utilisés.
1. Photographie en accéléré du passage 0 kératinocytes humains
REMARQUE : Ce protocole est spécifique aux IncuCyte S3 et SX5.
- S’assurer que les approbations appropriées du comité de recherche sur les êtres humains de l’établissement ont été obtenues pour utiliser des tissus humains pour l’étude.
- Isolez les kératinocytes de la peau fraîche comme décrit précédemment9.
- Déterminer la densité de semis pour l’essai. Mener des études pilotes avec des échantillons à dilutions multiples provenant de différents donneurs pour comprendre la densité de cellules nécessaires afin d’assurer des colonies adéquates pour l’étude, mais d’éviter les colonies excessives qui entraînent un chevauchement des colonies au cours de la période d’observation. Exécutez ces pilotes dans des conditions expérimentales identiques (mêmes plaques, réactifs, etc.), car ces facteurs peuvent altérer les résultats.
REMARQUE : Une densité d’ensemencement trop faible entraîne une croissance insuffisante, tandis qu’une densité d’ensemencement trop élevée empêche de suivre avec précision les cellules en raison de l’encombrement des cellules dans le champ.- Pour le passage 0 des kératinocytes provenant de prépuces néonatals de moins de 48 h à partir d’une collection stockée à 4 °C dans un milieu de collecte, assurez-vous que la densité d’ensemencement est de 1000-5000/cm2. Pour les kératinocytes P1, utilisez 500-2000 cellules/cm2. Plus le passage est long, plus la densité de semis est faible.
- Cellules de plaque sur la taille de plaque sélectionnée.
REMARQUE : Les plaques à 96 puits et les microplaques ont un effet ménisque, ce qui entraîne une distribution cellulaire non uniforme, une grande partie de la croissance se faisant en dehors du champ de l’imageur. Les plaques avec des puits plus grands (plaques de 24 puits par exemple) permettent de capturer plus de données. Les plaques à 24 puits permettent de visualiser jusqu’à 36 champs, tandis que les plaques à 96 puits permettent de visualiser jusqu’à 5 champs. Cependant, le nombre de trames capturées sur un instrument est limité, et plus il y a de trames, plus la capacité de l’imageur est consommée. En règle générale, une plaque à 24 puits est utilisée.- Pour obtenir une répartition uniforme des cellules sur la plaque, différentes techniques peuvent être utilisées. Tout d’abord, plutôt que d’ensemencer individuellement chaque puits, aliquote le milieu total nécessaire pour tous les puits à une dilution spécifique dans un microtube. Ensuite, pipetez le nombre total de cellules nécessaires pour que l’aliquote atteigne la densité souhaitée et retournez doucement le tube pour répartir uniformément les cellules.
- Après le placage, déplacez la plaque en croix trois fois (haut-bas, gauche-droite), puis transférez-la avec précaution dans l’incubateur.
ATTENTION : Si vous utilisez une microplaque, évitez les rangées et les colonnes extérieures des puits, car le microscope time-lapse génère de la chaleur lors de l’imagerie et peut provoquer l’évaporation du média dans ces puits. Les puits expérimentaux doivent être regroupés au centre de la plaque et entourés de puits contenant du PBS ou un autre liquide stérile à leur capacité maximale pour réduire les effets d’évaporation et de bord.
- Incuber les cellules pendant 24 h à 37 °C et 5 % de CO2 pour permettre l’observance.
- Changez de support après 24 h.
REMARQUE : Ne dérangez pas la monocouche en croissance lors de l’aspiration. Utilisez toujours un média chauffé (37 °C) et versez doucement le média dans les puits à l’aide de la paroi latérale. Le support utilisé ici est Epilife/Supplement S7/Primocin. L’utilisation d’antibiotiques dans les milieux pour l’imagerie à long terme de cellules vivantes est recommandée pour cette application. La culture prolongée dans une machine utilisée pour plusieurs expériences simultanées présente un risque élevé de contamination. La pénicilline et la streptomycine sont couramment utilisées. - Ouvrez l’incubateur en appuyant sur le gros bouton triangulaire en bas à gauche pour ouvrir le plateau lorsque le voyant est vert. Placez le récipient dans une baie ouverte. Ensuite, fermez le plateau à l’aide du même bouton en bas à gauche. N’ouvrez jamais la barre d’état lorsque le bouton d’ouverture est rouge, car cela signifie qu’elle est en train de numériser activement et que la numérisation sera interrompue.
ATTENTION : Assurez-vous qu’il n’y a pas de balayages en commençant par regarder l’écran du module de contrôleur. Il indique l’heure jusqu’au prochain balayage et la durée de l’examen. Si l’analyse a commencé, il vous donnera le temps jusqu’à ce que l’analyse soit terminée. - Ouvrez l’application sur l’ordinateur et connectez-vous à l’aide de l’ID utilisateur et du mot de passe appropriés. Sélectionnez Planifier.
- Appuyez sur le bouton + dans le coin supérieur gauche sous l’horaire pour ajouter l’assiette à l’horaire.
- Sélectionnez Numériser selon la planification, puis cliquez sur Suivant.
- Sélectionnez Nouveau, puis cliquez sur Suivant.
REMARQUE : Si cette plaque est identique à une expérience précédente ou en cours, les options Copier précédent ou Copier actuel peuvent être utilisées pour accélérer le formatage de l’expérience. - Sélectionnez Standard pour le type de balayage, puis cliquez sur Suivant.
REMARQUE : Image Lock est une plaque propriétaire à 96 puits qui aide à minimiser l’occurrence de perte de mise au point/saut d’image, ce qui peut être utile si ces problèmes sont rencontrés. Les autres types de balayage ne sont pas particulièrement utiles pour le traçage de lignée. - Pour les paramètres de balayage, sélectionnez Adhérent cellule par cellule à l’aide du canal de phase à l’objectif 10x, puis cliquez sur Suivant.
REMARQUE : Tant que le canal de phase est sélectionné, il n’est pas nécessaire de sélectionner une option d’analyse à ce stade, car l’analyse peut être lancée après la capture des données. - Sélectionnez le navire, puis cliquez sur Suivant. La plupart des récipients courants sont compatibles avec la machine, bien que les non-microplaques puissent nécessiter des attaches spéciales pour s’adapter aux baies du microscope time-lapse.
- Sélectionnez une baie vide pour placer le navire, puis cliquez sur Suivant.
- Sélectionnez les puits à analyser ainsi que les images par puits, puis cliquez sur Suivant. La durée estimée de l’analyse sera indiquée en bas à gauche de l’écran.
- Nommez l’expérience à l’aide de la convention de dénomination souhaitée. Créez une carte de plaque de l’expérience pour référence future, puis appuyez sur OK. Enfin, cliquez sur Suivant.
- Reportez l’analyse jusqu’à la collecte des données, car l’analyse cellule par cellule doit être lancée une fois le balayage terminé. Cliquez sur Suivant.
- Enfin, réglez la fréquence d’imagerie. Afin de suivre de manière fiable les mitoses, qui se produisent sur une période de 30 minutes, balayez les kératinocytes à intervalles de 20 minutes. Appuyez sur Suivant et confirmez les paramètres pour commencer l’expérience.
REMARQUE : Les temps de numérisation ne peuvent pas se chevaucher et les fabricants recommandent que la machine soit au repos aussi longtemps qu’elle numérise. Avec des intervalles de 20 minutes, cela signifie que 10 minutes maximum doivent être consacrées à la numérisation. Si 12 puits d’une plaque de 24 puits sont imagés à 36 vues par puits, il faut 7 minutes pour que le balayage se termine, alors gardez toujours à l’esprit les ressources disponibles pour l’expérience lors de la planification.- Il y a une notification si la machine n’a pas assez de temps pour se reposer, et si les heures de balayage se chevauchent, le navire ne peut pas être ajouté à l’horaire. Si le récipient ne peut pas s’adapter à l’horaire à intervalles réguliers, cliquez sur Réserver l’emplacement du plateau et appuyez sur Suivant.
- Double-cliquez sur l’horaire en haut de l’écran et sélectionnez le navire en réserve, puis cliquez manuellement pour ajouter des images sur les créneaux horaires ouverts. Vous pouvez également cliquer avec le bouton droit de la souris une fois le programme ouvert pour supprimer toutes les heures de balayage programmées et cliquer avec le bouton droit de la souris pour définir de nouvelles heures de balayage pour un groupe de balayage à intervalles réguliers.
REMARQUE : Cela interromprait toutes les expériences actuellement sur la machine jusqu’à ce que de nouvelles heures soient réglées. N’oubliez pas d’appuyer sur l’icône de la disquette pour enregistrer ou sur le X rouge pour annuler les modifications apportées à la planification. La sélection des puits ou des vues souhaités est un processus simple, car la machine fournit immédiatement le temps de balayage estimé lors de la sélection des paramètres. La durée du balayage dépend du type et de la marque de la plaque sélectionnée, ainsi que du nombre de puits ou de vues à scanner. Cela permet aux utilisateurs d’ajuster les paramètres de balayage en fonction de la capacité de la machine.
- Pour les kératinocytes, changer de milieu toutes les 48 heures. Pour garantir l’orientation correcte de la plaque, attendez que le premier balayage soit terminé chaque fois qu’un récipient est placé dans la machine (si le temps le permet). C’est une simple erreur à commettre. Placez la plaque dans la machine de manière à ce que les lettres indiquant les rangées se trouvent sur le côté gauche de la baie.
- Pour afficher les numérisations, appuyez sur le bouton Afficher avec l’icône en forme d’œil , puis double-cliquez sur l’expérience. Envisagez de changer de milieu tôt s’il y a un changement soudain de couleur du milieu (les milieux contenant du rouge de phénol deviennent jaunes en raison de l’acidification), s’il y a un excès de cellules mortes (peut interférer avec la prolifération des colonies vivantes) ou si une morphologie anormale est observée.
- Une fois que tous les puits sont devenus calmes ou ont atteint la confluence, exportez l’expérience. Suivre les colonies d’adultes pendant ~2 semaines et les néonataux pendant ~10 jours, période à laquelle l’encombrement entraîne des rendements décroissants pour les analyses.
- Pour exporter, cliquez sur l’onglet Affichage et double-cliquez sur l’expérience sous la liste des analyses récentes (par défaut, elles doivent être classées à partir de la plus récente vers l’arrière). Cliquez sur l’icône Paysage avec la flèche et attendez que l’outil d’exportation se lance.
- Cliquez sur Tel qu’affiché, puis sur Suivant.
- Cliquez manuellement sur chaque champ de vision à exporter, puis cliquez sur Suivant.
- Indiquez si vous souhaitez importer une vidéo ou une série d’images, puis sélectionnez les durées de numérisation à exporter. Pour le traçage de la lignée des kératinocytes, utilisez l’option film et sélectionnez tous les balayages (il y a une icône Sélectionner tout rapide avec une ligne pointillée pour accélérer cela). Cliquez sur Suivant.
- Exportez à 1 image par seconde en qualité maximale (réglez-la à l’aide de la barre à côté de la qualité). Cliquez sur Suivant.
- Choisissez le dossier cible et le type de fichier. Nommez le fichier, puis cliquez sur Exporter.
REMARQUE : L’exportation de vidéos peut prendre beaucoup de temps, en fonction de la vitesse de la connexion Internet et de l’ordinateur concerné. Préparez-vous à attendre plusieurs jours avant d’exporter tous les fichiers vidéo pour des expériences plus importantes. Avoir la possibilité de se connecter au microscope time-lapse à distance est extrêmement pratique.
2. Utilisation de l’imagerie time-lapse pour construire des arbres de lignage et générer des fiches techniques
- Ouvrez le fichier vidéo à l’aide d’un lecteur multimédia. Le lecteur multimédia VLC est recommandé.
- Faites défiler les vidéos et identifiez les colonies en croissance. Avec VLC, utilisez la touche fléchée pour sauter les images en avant et en arrière. Faites une capture d’écran de la colonie à suivre et étiquetez la colonie.
- Identifiez une colonie d’intérêt à la fin ou après quelques jours d’enregistrement vidéo, puis rembobinez la vidéo et identifiez la cellule formant la colonie.
REMARQUE : C’est là qu’il est important d’avoir la densité de semis appropriée. Si trop de colonies se développent les unes à côté des autres, elles s’étendent et fusionnent les unes dans les autres, ce qui rend impossible le suivi précis des divisions cellulaires. - Lorsqu’une cellule est sur le point de se diviser, elle semble se condenser (Figure 1). Mettez la vidéo en pause lorsque la division se produit. Enregistrez l’heure de la division (un horodatage se trouve dans le coin inférieur gauche). Faites une capture d’écran de cette division initiale et donnez-lui la même étiquette que la colonie. Notez les divisions dans un diagramme de lignage dessiné à la main (voir Résultats représentatifs). Continuez à suivre et à documenter autant de générations que possible.
REMARQUE : Le suivi automatisé des cellules a été mis au point ou est en cours d’élaboration afin d’accélérer ce processus et de faire un suivi plus précis des cellules7. Même dans des conditions de culture à faible teneur en calcium avec une différenciation minimale, les modèles d’apprentissage automatique n’ont actuellement pas la sensibilité nécessaire pour suivre avec précision les divisions des kératinocytes. - Transcrire manuellement l’arbre de lignage en feuilles de données - appelées « feuilles vertes » en raison de la couleur de la feuille de calcul (fichier supplémentaire 1).
- Utilisez les feuilles vertes pour calculer les proportions de divisions prolifératives et de différenciation, identifier les colonies de cellules souches et de progéniteurs engagés, ainsi que la durée du cycle cellulaire (voir les résultats représentatifs).
REMARQUE : La machine est capable d’analyser les données via ses propres analyseurs de base et cellule par cellule pour exécuter plusieurs autres tests (confluence, scratch). Cependant, cela dépasse le champ d’application de ce protocole.
Résultats
Les kératinocytes primaires se développent de manière stéréotypée, qui peut être suivie par l’imagerie cellulaire en direct. Le lecteur multimédia VLC est utilisé pour examiner les enregistrements. Le temps jusqu’à la première division est variable et peut être de plusieurs jours en fonction des caractéristiques du donneur, telles que l’âge, l’état de santé ou les facteurs de croissance présents dans l’environnement in vitro . Lors de l’ensemencement initial, les kératinocytes ont un aspect petit et arrondi (figure 1). Après l’ensemencement, les kératinocytes formant des colonies s’aplatissent généralement (Figure 1). Ces kératinocytes aplatis ont tendance à être plus mobiles que leurs homologues non diviseurs (Vidéo supplémentaire 1). Immédiatement avant la division, le kératinocyte aplati semble se condenser au centre (Figure 1).
À l’exception de la division initiale, 95 % des kératinocytes qui vont se diviser (prolifératif - P) le font dans les 48 heures suivant la division6 précédente. Ceux qui ne le sont pas sont considérés comme différenciés en phase terminale (D) (Figure 1)6. Ces cellules différenciées restent collées jusqu’à la fin de la période d’observation ou jusqu’à ce qu’elles se détachent de la plaque et soient retirées lors du changement de milieu ultérieur. Les cellules qui se différencient ont tendance à se dilater avec le temps, ce qui entraîne une morphologie non uniforme de la colonie de kératinocytes (Figure 1). Exportez les vidéos une fois la période d’observation terminée et commencez l’analyse (Figure 2).
Les colonies sont documentées à l’aide d’un processus normalisé. Le préfixe vidéo dans lequel se trouvait la colonie (A1, A2..., B1, B2...) est utilisé, suivi du numéro de la colonie. Par exemple, A2-6 serait la vidéo A2, colonie 6. L’analyse commence par le traçage de la lignée. Avance rapide jusqu’à la fin d’une vidéo pour identifier les colonies, puis retour au début de la période d’observation pour identifier la cellule d’origine formant la colonie. Suivez les horodatages de toutes les divisions au fur et à mesure qu’elles se produisent et créez un diagramme ramifié à la main, en suivant autant de générations que possible (Figure 3). Finalement, il n’est plus possible de suivre avec précision les divisions cellulaires en raison de la densité cellulaire (cela se produit généralement vers la génération 5 à 7, selon que la colonie provient d’une cellule souche ou d’un progéniteur engagé). Dans les générations suivantes, la colonie fusionne souvent avec une autre colonie, ou la colonie sort de l’écran. À ce stade, marquez la dernière cellule traçable comme U (intraçable). Prenez toujours une capture d’écran de la colonie suivie (cela peut être fait en utilisant la fonction d’instantané sur VLC) et marquez la colonie en utilisant la nomenclature décrite ci-dessus. Assurez-vous d’étiqueter la capture d’écran avec une nomenclature standardisée, y compris la vidéo spécifique analysée et le numéro de colonie qui peut être associé à la capture d’écran.
Une fois le diagramme de branche construit, les données peuvent être transférées sur une feuille de calcul ou une « feuille verte » (fichier supplémentaire 1). La feuille verte contient la même étiquette pour chaque colonie suivie par un diagramme de branche. Pour identifier facilement les générations, les reflets de couleur sont alternés entre les générations. Le temps 1 de la génération 1 fait référence à la période entre le placage des cellules et leur placement sur le microscope time-lapse. Dans le fichier supplémentaire 1, le temps 1 de la génération 1 est de 24 h, car les cellules sont placées sur la machine 24 h après le placage. Le temps 2 représente la durée jusqu’à ce que la cellule subisse sa première division. N’oubliez pas que l’horodatage dans les vidéos ne tient pas compte des 24 heures supplémentaires, de sorte que les heures doivent être ajoutées manuellement à chaque génération. Lors de la transcription des données du diagramme de branche sur la feuille verte, ajoutez toujours 24 h au temps des divisions car l’horodatage fourni par la machine ne tient pas compte du temps avant le début de l’enregistrement. ΔT est la durée du cycle cellulaire. La plupart des études 6,8 n’incluent pas le ΔT de la première génération dans les analyses, car il est toujours beaucoup plus long que celui des générations suivantes et extrêmement variable, ce qui entraîne des analyses biaisées.
Une fois la feuille verte construite, on peut déterminer quelles colonies proviennent de cellules souches et lesquelles proviennent de progéniteurs engagés. Les colonies qui présentent des divisions principalement prolifératives (Figure 1) et qui durent jusqu’à la fin de la période d’observation sont considérées comme des colonies de cellules souches6. Les colonies qui se différencient en phase terminale (figure 1) pendant la période d’observation sont considérées comme des colonies progénitrices engagées6. Le ΔT moyen des colonies de cellules souches et des colonies de progéniteurs engagés, respectivement, peut alors être calculé. En divisant la proportion de divisions D par le nombre total de divisions, on peut calculer la proportion de divisions de différenciation, soit avec toutes les divisions regroupées de cellules souches/colonies de progéniteurs engagés, respectivement, soit par génération. La feuille verte est un moyen utile d’organiser les données pour obtenir des informations efficacement et elle évolue en fonction des données et des statistiques nécessaires pour les objectifs de l’étude.
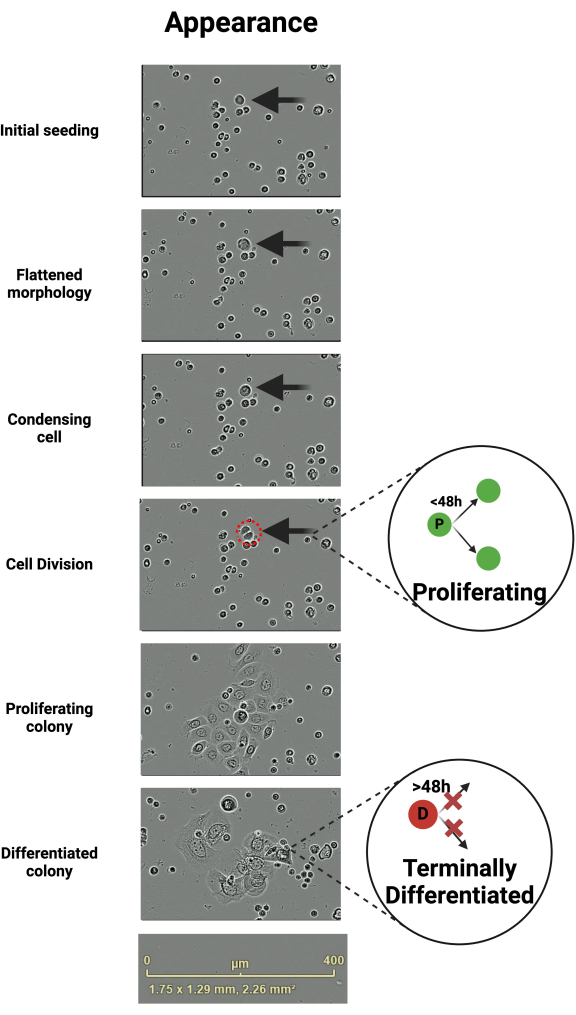
Figure 1 : Changements dans la morphologie des kératinocytes menant à la division cellulaire et à la terminologie de division. Les kératinocytes progressent à travers une séquence stéréotypée de changements morphologiques menant à la division cellulaire. Cette figure représente un kératinocyte subissant cette séquence d’événements au fil du temps. Les cellules individuelles qui sont sur le point de se diviser sont mobiles et prennent d’abord une morphologie aplatie et se condensent au centre immédiatement avant de se diviser. Les cellules filles qui continueront à proliférer se divisent dans les 48 heures suivant leur génération, sinon elles sont considérées comme différenciées en phase terminale. Barre d’échelle 400 μm. Créé dans BioRender. Ghadially, R. (2025) https://BioRender.com/k40e714 Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Exportation de données à partir du microscope time-lapse. Un guide sur la façon d’exporter des données à partir de la machine. Créé dans BioRender. Ghadially, R. (2024) BioRender.com/x99d452 Veuillez cliquer ici pour voir une version agrandie de cette figure.
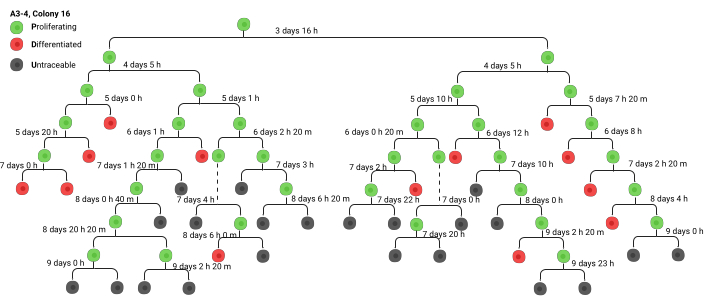
Graphique 3. Diagramme de branche précurseur (arbre de lignage) pour informer la création de feuilles vertes. Un exemple d’arbre de lignage, généralement dessiné à la main. Abréviations : h : heures, m : min. Créé dans BioRender. Ghadially, R. (2025) https://BioRender.com/wbt7z8x Veuillez cliquer ici pour voir une version agrandie de cette figure.
Dossier supplémentaire 1. Exemples de fiches techniques (feuilles vertes) de données d’imagerie de cellules vivantes non publiées. Un exemple de feuille verte contenant des données non publiées. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire 1. Exemple de vidéo d’imagerie de cellules vivantes de kératinocytes. Une vidéo d’imagerie cellulaire en direct avec une densité cellulaire appropriée pour suivre les colonies de kératinocytes Veuillez cliquer ici pour télécharger ce fichier.
Discussion
L’imagerie de cellules vivantes de kératinocytes est une méthode sans marquage pour suivre le comportement de division des cellules souches et des progéniteurs engagés. Étant donné que le maintien de l’épiderme dépend de la cinétique de prolifération des cellules souches et des progéniteurs engagés10, une compréhension granulaire des changements dans ces populations de kératinocytes et de la façon dont elles sont affectées dans diverses conditions facilite le développement de thérapies pour améliorer les défauts découverts.
En fin de compte, le traçage de la lignée via l’imagerie de cellules vivantes dépend de l’obtention de données utilisables. Les unités formant colonie doivent être clairement visibles dans les vidéos générées par la photographie en accéléré. Avec des cellules de passage 0 fraîchement isolées, il est difficile d’obtenir une véritable densité clonale. Seulement 3 à 4 % des kératinocytes en plaques forment finalement des colonies11. Trop de cellules peuvent rendre impossible le suivi des divisions ou même l’identification de la cellule initiale formant la colonie. Trop peu de cellules, et les colonies peuvent ne pas se former. De même, tout ce qui obscurcit le champ visuel rend impossible le suivi des cellules via le traçage de lignée. L’utilisation de mangeurs de fibroblastes peut rendre difficile le suivi des divisions, car les fibroblastes obscurcissent le champ visuel. De même, les précipités, les débris cellulaires et même la condensation sur le couvercle de la plaque peuvent masquer les colonies en croissance. Même quelque chose d’aussi petit que de ne pas s’assurer que la microplaque est correctement insérée dans la baie sélectionnée peut finalement entraîner l’échec de l’expérience, car l’image ne sera pas focalisée sur les cellules. C’est pourquoi il est très important de surveiller les colonies quotidiennement pendant qu’elles se développent dans la machine et de toujours rester jusqu’après le premier balayage, chaque fois que la microplaque est retirée de la machine pour des changements de support, afin de s’assurer que les cellules sont capturées par la photographie en accéléré. Un autre problème que les débris peuvent causer est le saut d’image. En règle générale, le microscope a des champs visuels fixes qui ne bougent pas, mais lorsque les débris s’accumulent ou que la densité cellulaire devient trop élevée, la machine peut perdre son champ de vision et sauter vers une autre partie de la plaque. Pour éviter cela, les fabricants de la machine ont développé un type spécial de plaque à 96 puits avec une grille qui empêche la perte de mise au point.
La machine génère de la chaleur tout en capturant des images. Il est important de réduire la température de l’incubateur contenant le microscope à 36,5 °C pour que l’appareil s’équilibre à 37 °C lors de la capture d’images. Lorsque vous utilisez des plaques de plus petite surface (plaques à 96 puits), tenez compte de la chaleur supplémentaire générée. N’utilisez pas les puits périphériques pour les expériences (première et dernière rangées/colonnes) et, envisagez de remplir au maximum les puits entourant les puits expérimentaux avec d’autres fluides (PBS stérile), et utilisez du ruban respirant pour réduire la perte par évaporation du milieu. Il existe des plaques commercialisées qui pourraient être étudiées pour minimiser les effets de bord12. Cependant, les méthodes susmentionnées ont permis l’utilisation de microplaques compatibles recommandées.
Lors de l’analyse des vidéos, les enquêteurs doivent être conscients des modèles de division anormaux de leurs colonies. Par exemple, s’il n’y a pas de divisions dans une vue pendant 10 jours et qu’il y a ensuite une formation de colonie rampante provenant du bord du champ de vision, il est peu probable qu’il s’agisse de la première division d’une nouvelle colonie. Il est probable qu’une colonie en croissance à partir d’un champ visuel environnant ait empiété sur la partie de la plaque capturée plutôt que d’être une nouvelle colonie. Cela peut être vérifié en se connectant à la suite logicielle, qui peut représenter l’ensemble de la plaque (contenant toutes les vues enregistrées) à des moments précis et montrer la migration des cellules d’un champ de vision à un autre.
La principale limitation de cette méthode est sa nature laborieuse. De plus, le suivi des divisions cellulaires au-delà de la 5egénération est difficile et nécessite des heures de travail, en revisionnant des vidéos pour capturer avec précision ce qui se divise et quand. De nombreux algorithmes de suivi automatisé des cellules par apprentissage profond sont en cours de développement, ce qui aboutira à terme à une analyse purement basée sur l’IA dans les années à venir 7,13,14. D’ici là, le suivi manuel, tel que détaillé dans ce protocole, est une méthode réalisable pour développer des données de lignage.
Déclarations de divulgation
Aucun.
Remerciements
Ce travail a été soutenu par le Merit Review Award Number I01 CX001816 des États-Unis (U.S.) Service de R&D en sciences cliniques du ministère des Anciens Combattants. Le contenu ne représente pas les opinions du ministère américain des Anciens combattants ou du gouvernement des États-Unis. Nous remercions le Dr Michael Rosenblum de nous avoir donné accès à son microscope time-lapse pour mener nos expériences.
matériels
| Name | Company | Catalog Number | Comments |
| 96 Well Imagelock plate | Sartorius | BA-04856 | Suggested microplate compatible with machine if using a 96 well plate. |
| 24 well plate | Corning | 3524 | Suggested microplate compatible with machine if using a 24 well plate. |
| Amphotericin B, 50 mL | Corning | 30-003-CF | Dilute to 5x (comes in 100x stock) for 5x PSA - 1x for media changes |
| Epilife, 50 mL | Gibco | MEP1500CA | Add S7, consider primocin |
| IncuCyte S3 | Sartorius | 4637 | Imager (Zoom/SX5 acceptable alternatives) |
| Penicillin/Streptomycin, 100 mL | Corning | 30-002-Cl | Dilute to 5x (comes in 100x stock) |
| Primocin | Invivogen | ant-pm-05 | 1 mL per 500 mL media |
| Supplement S7 | Gibco | S0175 | Added to epilife |
Références
- Mateu, R., et al. Functional differences between neonatal and adult fibroblasts and keratinocytes: Donor age affects epithelial-mesenchymal crosstalk in vitro. Int J Mol Med. 38 (4), 1063-1074 (2016).
- Handl, J., Čapek, J., Majtnerová, P., Báčová, J., Roušar, T. The effect of repeated passaging on the susceptibility of human proximal tubular HK-2 cells to toxic compounds. Physiol Res. 69 (4), 731-738 (2020).
- Moran, M. C., et al. Characterization of human keratinocyte cell lines for barrier studies. JID Innov. 1 (2), 100018(2021).
- Seo, M. -D., Kang, T. J., Lee, C. H., Lee, A. -Y., Noh, M. HaCaT keratinocytes and primary epidermal keratinocytes have different transcriptional profiles of cornified envelope-associated genes to T helper cell cytokines. Biomol Ther. 20 (2), 171-176 (2012).
- Jahn, M., et al. Different immortalized keratinocyte cell lines display distinct capabilities to differentiate and reconstitute an epidermis in vitro. Exp Dermatol. 33 (1), e14985(2024).
- Xiao, T., et al. Short cell cycle duration is a phenotype of human epidermal stem cells. Stem Cell Res Ther. 15 (1), 76(2024).
- Hirose, T., Kotoku, J., Toki, F., Nishimura, E. K., Nanba, D. Label-free quality control and identification of human keratinocyte stem cells by deep learning-based automated cell tracking. Stem Cells. 39 (8), 1091-1100 (2021).
- Roshan, A., Murai, K., Fowler, J., Simons, B. D., Nikolaidou-Neokosmidou, V., Jones, P. H. Human keratinocytes have two interconvertible modes of proliferation. Nat Cell Biol. 18 (2), 145-156 (2016).
- Abegaze, B., Ijeh, N., Vittimberga, B., Ghadially, R. Generating primary cultures for the purpose of keratinocyte live cell imaging. J Vis Exp. , In Press (2024).
- Piedrafita, G., et al. A single-progenitor model as the unifying paradigm of epidermal and esophageal epithelial maintenance in mice. Nat Commun. 11 (1), 1429(2020).
- Ścieżyńska, A., et al. Isolation and culture of human primary keratinocytes-a methods review. Exp Dermatol. 28 (2), 107-112 (2019).
- Mansoury, M., Hamed, M., Karmustaji, R., Al Hannan, F., Safrany, S. T. The edge effect: A global problem. The trouble with culturing cells in 96-well plates. Biochem Biophys Rep. 26, 100987(2021).
- Malin-Mayor, C., et al. Automated reconstruction of whole-embryo cell lineages by learning from sparse annotations. Nat Biotechnol. 41 (1), 44-49 (2023).
- Waliman, M., et al. Automated cell lineage reconstruction using label-free 4D microscopy. Genetics. 228 (2), iyae135(2024).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon