Method Article
Imágenes de células vivas con fotografía de lapso de tiempo para estudiar la cinética de proliferación de queratinocitos epidérmicos
En este artículo
Resumen
Aquí, proporcionamos un método para el análisis de imágenes de células vivas que se puede utilizar para rastrear manualmente los linajes de los queratinocitos de paso 0 y que permite la recopilación de métricas de proliferación, incluido el destino de la división celular y la duración del ciclo celular.
Resumen
La obtención de imágenes de células vivas es un método en evolución y algo desafiante para estudiar el comportamiento de los queratinocitos in vitro. Históricamente, el comportamiento de la división de los queratinocitos se investigó mediante métodos como el análisis clonal, la inmunotinción y el análisis del ciclo celular. Ninguno de estos métodos permite analizar el comportamiento de los queratinocitos a nivel de una sola célula en tiempo real. Durante la última década, los grupos han utilizado imágenes de células vivas para identificar células madre de queratinocitos y progenitores comprometidos sin necesidad de etiquetado. Se han identificado diferencias en el comportamiento de división, la tasa de diferenciación terminal y la duración del ciclo celular de cada grupo respectivo. En este trabajo se describe un método para la obtención de imágenes de células vivas de queratinocitos con fotografía time-lapse y su análisis. Se recomienda el uso de queratinocitos no transitados para que este método imite más de cerca el comportamiento in vivo . Las imágenes de células vivas proporcionan una capacidad única para estudiar el comportamiento de las células madre y los progenitores comprometidos a nivel de una sola célula y para determinar los destinos de la división, la duración del ciclo celular, así como otras métricas de proliferación.
Introducción
La capacidad de visualizar poblaciones de células in vitro en tiempo real a medida que se expanden durante largos períodos de tiempo es un beneficio único de las imágenes de células vivas. Las imágenes de células vivas permiten evaluar la motilidad, la migración y la proliferación de las células a nivel de una sola célula. El objetivo de este protocolo es optimizar la visualización de los cultivos de queratinocitos a través de la fotografía de lapso de tiempo, produciendo videos que luego se pueden rastrear manualmente para obtener datos granulares sobre el comportamiento celular.
Nos centramos en la cinética de la proliferación. A partir del análisis de los videos de imágenes de células vivas, se pueden dilucidar los árboles de linaje y se puede evaluar el tiempo entre divisiones (un indicador de la duración del ciclo celular), así como las proporciones de divisiones que conducen a una mayor división frente a la diferenciación de las células hijas.
Existe una variabilidad sustancial de donante a donante cuando se trata de queratinocitos primarios y frecuentes intentos fallidos de propagación celular. Debido a esto, muchos investigadores optan por utilizar queratinocitos altamente proliferativos, como las células HaCaT o los queratinocitos neonatales, a menudo después de haber pasado por múltiples deposiciones in vitro1. El cultivo de queratinocitos primarios de piel adulta o envejecida con el fin de rastrear el linaje puede ser un desafío. Sin embargo, existen problemas con el uso de células pasadas de líneas celulares o del prepucio masculino. El paso repetido da como resultado células que son significativamente diferentes de su estado in vivo 2. Además, se ha demostrado que las células HaCaT reaccionan de manera diferente a los queratinocitos primarios en múltiples ensayos 3,4,5. Para utilizar las células que más se parecen a sus contrapartes in vivo, se utilizan queratinocitos de paso 0 de donantes humanos adultos. Las células madre de queratinocitos y los progenitores comprometidos exhiben claras diferencias en el comportamiento, que permiten distinguir las colonias de cualquiera de las poblaciones a través de imágenes de células vivas6. Esta capacidad relativamente novedosa para visualizar el comportamiento de los queratinocitos individuales a largo plazo se ha utilizado solo en unos pocos estudios previos que utilizan técnicas similares 6,7,8. Este protocolo describe la obtención de imágenes de células vivas de queratinocitos primarios utilizando el sistema de análisis de células vivas IncuCyte S3. A partir de los árboles de linaje que se construyen, se puede determinar el tipo de colonia (célula madre frente a progenitora comprometida), así como la duración del ciclo celular y la proporción de divisiones de diferenciación.
Protocolo
Este estudio se realizó de acuerdo con la Declaración de Helsinki. Todo el tejido humano se obtuvo después de la aprobación de la junta de revisión institucional (IRB) de la Universidad de California, San Francisco (UCSF), y se obtuvo el consentimiento para todo el tejido utilizado.
1. Fotografía de lapso de tiempo de queratinocitos humanos de pasaje 0
NOTA: Este protocolo es específico para IncuCyte S3 y SX5.
- Asegurarse de que se hayan obtenido las aprobaciones apropiadas del comité de investigación en seres humanos de la institución para utilizar tejido humano para el estudio.
- Aislar los queratinocitos de la piel fresca como se describió anteriormente9.
- Determine la densidad de siembra para el ensayo. Llevar a cabo estudios piloto con muestras en múltiples diluciones de diferentes donantes para comprender la densidad de células necesaria con el fin de garantizar colonias adecuadas para el estudio, pero evitar colonias excesivas que resulten en una superposición de colonias durante el período de observación. Ejecute estos pilotos en condiciones experimentales idénticas (mismas placas, reactivos, etc.), ya que estos factores pueden alterar los resultados.
NOTA: Una densidad de siembra demasiado baja da como resultado un crecimiento insuficiente, mientras que una densidad de siembra demasiado alta da como resultado la incapacidad de rastrear con precisión las células debido al hacinamiento de células en el campo.- Para los queratinocitos de paso 0 de prepucios neonatales de menos de 48 h de una colección almacenada a 4 °C en medios de recolección, asegúrese de que la densidad de siembra sea de 1000-5000/cm2. Para los queratinocitos P1, utilizar 500-2000 células/cm2. Cuanto más lejos esté el paso, menor será la densidad de siembra.
- Celdas de placa en el tamaño de placa seleccionado.
NOTA: Las placas y microplacas de 96 pocillos tienen un efecto de menisco, lo que da como resultado una distribución celular no uniforme, con gran parte del crecimiento fuera del campo del generador de imágenes. Las placas con pocillos más grandes (placas de 24 pocillos, por ejemplo) permiten capturar más datos. Las placas de 24 pocillos permiten la visualización de hasta 36 campos, mientras que las placas de 96 pocillos permiten la visualización de solo 5 campos. Sin embargo, el número de campos capturados en un instrumento es finito y más campos consumirán más capacidad del generador de imágenes. Por lo general, se utiliza una placa de 24 pocillos.- Para lograr una distribución uniforme de las células en la placa, se pueden utilizar varias técnicas. En primer lugar, en lugar de sembrar individualmente cada pocillo, alícuota el medio total necesario para todos los pocillos en una dilución específica en un microtubo. A continuación, pipetee el número total de células necesarias para que la alícuota alcance la densidad deseada e invierta suavemente el tubo para distribuir homogéneamente las células.
- Después de colocar el plato, mueva la placa en un patrón de cruz tres veces (arriba-abajo, izquierda-derecha) y luego transfiérala a la incubadora con cuidado.
PRECAUCIÓN: Si utiliza una microplaca, evite las filas y columnas exteriores de los pocillos, ya que el microscopio de lapso de tiempo genera calor al obtener imágenes y puede causar la evaporación de los medios en esos pocillos. Los pozos experimentales deben agruparse en el centro de la placa y rodearse de pocillos que contengan PBS u otro fluido estéril a su máxima capacidad para reducir los efectos de evaporación y borde.
- Incubar las células durante 24 h a 37 °C y 5% de CO2 para permitir la adherencia.
- Cambie el soporte después de 24 h.
NOTA: No moleste la monocapa en crecimiento al aspirar. Utilice siempre medios calentados (37 °C) y haga fluir suavemente los medios hacia los pocillos utilizando la pared lateral. Los medios utilizados aquí son Epilife/Supplement S7/Primocin. Se recomienda el uso de antibióticos en medios para la obtención de imágenes de células vivas a largo plazo para esta aplicación. El cultivo extendido en una máquina que se utiliza para múltiples experimentos simultáneos tiene un alto riesgo de contaminación. La penicilina y la estreptomicina se usan comúnmente. - Abra la incubadora presionando el botón triangular grande en la parte inferior izquierda para abrir la bandeja cuando la luz esté verde. Coloque la embarcación en una bahía abierta. Luego, cierre la bandeja usando el mismo botón en la parte inferior izquierda. Nunca abra la bandeja cuando el botón para abrir esté rojo, ya que eso significa que está escaneando activamente y el escaneo se interrumpirá.
PRECAUCIÓN: Asegúrese de que no haya escaneos que comiencen mirando la pantalla en el módulo del controlador. Indica el tiempo hasta el próximo escaneo y la duración del escaneo. Si el escaneo ha comenzado, le dará el tiempo hasta que se complete el escaneo. - Abra la aplicación en la computadora e inicie sesión con el ID de usuario y la contraseña adecuados. Seleccione Programar.
- Presione el botón + en la esquina superior izquierda debajo del horario para agregar el plato al horario.
- Seleccione Analizar según programación y, a continuación, haga clic en Siguiente.
- Seleccione Nuevo y, a continuación, haga clic en Siguiente.
NOTA: Si esta placa es idéntica a un experimento anterior o en ejecución, se puede utilizar Copiar anterior o Copiar actual para acelerar el formato del experimento. - Seleccione Estándar para el tipo de análisis y, a continuación, haga clic en Siguiente.
NOTA: Image Lock es una placa patentada de 96 pocillos que ayuda a minimizar la ocurrencia de pérdida de enfoque / salto de imagen, lo que puede ser útil si se enfrentan estos problemas. Los otros tipos de escaneo no son particularmente útiles para el rastreo de linaje. - Para la configuración de escaneo, seleccione Adherent Cell-by-Cell usando el canal de fase en el objetivo 10x y, a continuación, haga clic en Siguiente.
NOTA: Siempre que se elija el canal de fase, no es necesario seleccionar ninguna opción de análisis en este punto, ya que el análisis se puede iniciar después de capturar los datos. - Seleccione la embarcación y, a continuación, haga clic en Siguiente. La mayoría de los recipientes comunes son compatibles con la máquina, aunque las no microplacas pueden necesitar accesorios especiales para encajar en las bahías del microscopio de lapso de tiempo.
- Seleccione una bahía vacía para colocar la embarcación y, a continuación, haga clic en Siguiente.
- Seleccione los pocillos que desea escanear, así como las imágenes por pocillo y, a continuación, haga clic en Siguiente. La duración estimada del escaneo se proporcionará en la parte inferior izquierda de la pantalla.
- Asigne un nombre al experimento con la convención de nomenclatura deseada. Crea un mapa de placas del experimento para futuras referencias, luego presiona Aceptar. Finalmente, haga clic en Siguiente.
- Aplace el análisis hasta después de la recopilación de datos, ya que el análisis celda por celda debe iniciarse después de que se haya completado el escaneo. Haga clic en Siguiente.
- Por último, establezca la frecuencia de las imágenes. Con el fin de realizar un seguimiento fiable de las mitosis, que se producen durante 30 minutos, se realiza una exploración a intervalos de 20 minutos en busca de queratinocitos. Presione Siguiente y confirme la configuración para comenzar el experimento.
NOTA: Los tiempos de escaneo no pueden superponerse, y los fabricantes recomiendan que la máquina esté en reposo durante todo el tiempo que esté escaneando. Con intervalos de 20 minutos, eso significa que se deben dedicar un máximo de 10 minutos a escanear. Si se toman imágenes de 12 pocillos de una placa de 24 pocillos a 36 vistas por pocillo, el escaneo tarda 7 minutos en completarse, por lo que siempre debe tener en cuenta los recursos disponibles para el experimento al planificar.- Hay una notificación si no hay tiempo suficiente para que la máquina descanse, y si los tiempos de escaneo se superponen, entonces el recipiente no se puede agregar al horario. Si la embarcación no puede caber en el horario a intervalos regulares, haga clic en Reservar ubicación de la bandeja y presione Siguiente.
- Haga doble clic en el horario en la parte superior de la pantalla y seleccione el barco en reserva, luego haga clic manualmente para agregar imágenes en las franjas horarias abiertas. Como alternativa, haga clic con el botón derecho una vez que la programación esté abierta para eliminar todas las horas de análisis programadas y haga clic con el botón derecho para establecer nuevas horas de análisis para un grupo de análisis a intervalos regulares.
NOTA: Esto interrumpiría todos los experimentos actualmente en la máquina hasta que se establezcan nuevos tiempos. No olvide presionar el ícono del disquete para guardar o la X roja para cancelar los cambios en el horario. La selección de los pozos o vistas deseados es un proceso sencillo, ya que la máquina proporciona inmediatamente el tiempo estimado de escaneo tras la selección de parámetros. La duración del escaneo depende del tipo y la marca de placa seleccionada, así como del número de pocillos o vistas que se van a escanear. Esto permite a los usuarios ajustar los parámetros de escaneo para que coincidan con la capacidad de la máquina.
- En el caso de los queratinocitos, cambiar de medio cada 48 h. Para garantizar la orientación correcta de la placa, espere a que se complete el primer escaneo cada vez que se coloque un recipiente en la máquina (si el tiempo lo permite). Es un error sencillo de cometer. Coloque la placa en la máquina de modo que las letras que denotan las filas estén en el lado izquierdo de la bahía.
- Para ver los exámenes, presione el botón Ver con el icono del ojo y, a continuación, haga doble clic en el experimento. Considere cambiar el medio temprano si hay un cambio repentino en el color del medio (los medios que contienen rojo de fenol se vuelven amarillos debido a la acidificación), si hay un exceso de células muertas presentes (puede interferir con la proliferación de colonias vivas) o si se observa una morfología anormal.
- Una vez que todos los pozos se hayan vuelto inactivos o hayan alcanzado la confluencia, exporte el experimento. Seguimiento de las colonias adultas durante ~2 semanas y neonatales durante ~10 días, momento en el que el hacinamiento provoca rendimientos decrecientes para los análisis.
- Para exportar, haga clic en la pestaña Vista y haga doble clic en el experimento en la lista de exámenes recientes (el valor predeterminado es que se ordenen desde el más reciente hacia atrás). Haga clic en el icono Horizontal con la flecha y espere a que se inicie la herramienta de exportación.
- Haga clic en "Como se muestra" y, a continuación, haga clic en Siguiente.
- Haga clic manualmente en cada campo de vista para exportar y, a continuación, haga clic en Siguiente.
- Seleccione si desea una película o una serie de imágenes y, a continuación, seleccione los tiempos de escaneo que desea exportar. Para el rastreo del linaje de queratinocitos, use la opción de película y seleccione todos los escaneos (hay un ícono rápido de Seleccionar todo con una línea discontinua para acelerar esto). Haga clic en Siguiente.
- Exporte a 1 fotograma por segundo con la máxima calidad (configúrelo usando la barra junto a calidad). Haga clic en Siguiente.
- Elija la carpeta de destino y el tipo de archivo. Asigne un nombre al archivo y, a continuación, haga clic en Exportar.
NOTA: La exportación de vídeos puede llevar mucho tiempo, dependiendo de la velocidad de la conexión a Internet y del ordenador de que se trate. Prepárese para esperar varios días para exportar todos los archivos de video para experimentos más grandes. Tener la capacidad de iniciar sesión en el microscopio de lapso de tiempo de forma remota es extremadamente conveniente.
2. Uso de imágenes de lapso de tiempo para construir árboles de linaje y generar hojas de datos
- Abra el archivo de video con un reproductor multimedia. Se recomienda el reproductor multimedia VLC.
- Desplácese por los videos e identifique las colonias en crecimiento. Con VLC, usa la tecla de flecha para saltar fotogramas hacia adelante y hacia atrás. Tome una captura de pantalla de la colonia que se va a rastrear y etiquete la colonia.
- Identifique una colonia de interés al final o después de algunos días de grabación de video, luego rebobine el video e identifique la célula formadora de colonias.
NOTA: Aquí es donde es importante tener la densidad de siembra adecuada. Si se desarrollan demasiadas colonias una al lado de la otra, se expanden y se fusionan entre sí, lo que hace imposible realizar un seguimiento preciso de las divisiones celulares. - Cuando una célula está a punto de dividirse, parece condensarse (Figura 1). Pausa el vídeo cuando se produzca la división. Registre la hora de la división (hay una marca de tiempo en la esquina inferior izquierda). Haz una captura de pantalla de esta división inicial y ponle la misma etiqueta que a la colonia. Registre las divisiones en un diagrama de linaje dibujado a mano (consulte Resultados representativos). Continúe rastreando y documentando tantas generaciones como sea posible.
NOTA: Se ha desarrollado/se está desarrollando el seguimiento automatizado de células para acelerar este proceso y realizar un seguimiento más preciso de las células7. Incluso en condiciones de cultivo con bajo contenido de calcio con una diferenciación mínima, los modelos de aprendizaje automático carecen actualmente de la sensibilidad necesaria para realizar un seguimiento preciso de las divisiones de los queratinocitos. - Transcriba el árbol de linaje manual en hojas de datos, llamadas "hojas verdes" debido al color de la hoja de cálculo (Archivo complementario 1).
- Utilice las hojas verdes para calcular las proporciones de divisiones proliferativas y de diferenciación, identificar las colonias de células madre y progenitoras comprometidas, y la duración del ciclo celular (ver resultados representativos).
NOTA: La máquina es capaz de analizar datos a través de sus propios analizadores básicos y célula por celda para ejecutar muchos otros ensayos (confluencia, rasguño). Sin embargo, esto está más allá del alcance de este protocolo.
Resultados
Los queratinocitos primarios crecen de forma estereotipada, lo que se puede rastrear a través de imágenes de células vivas. El reproductor multimedia VLC se utiliza para inspeccionar grabaciones. El tiempo hasta la primera división es variable y puede ser de varios días dependiendo de las características del donante, como la edad, el estado de salud o los factores de crecimiento presentes en el ambiente in vitro . Tras la siembra inicial, los queratinocitos tienen un aspecto pequeño y redondeado (Figura 1). Después de la siembra, los queratinocitos formadores de colonias generalmente se aplanan (Figura 1). Estos queratinocitos aplanados tienden a ser más móviles que sus contrapartes no divisorias (Video Suplementario 1). Inmediatamente antes de dividirse, el queratinocito aplanado parece condensarse centralmente (Figura 1).
A excepción de la división inicial, el 95% de los queratinocitos que se van a dividir (proliferativos - P) lo hacen dentro de las 48 h de la división anterior6. Las que no lo hacen se consideran terminalmente diferenciadas (D) (Figura 1)6. Estas células diferenciadas permanecen adheridas hasta el final del período de observación o hasta que se levantan de la placa y se retiran en el cambio de medio posterior. Las células que se diferencian tienden a expandirse con el tiempo, lo que da lugar a una morfología no uniforme de la colonia de queratinocitos (Figura 1). Exporte los videos una vez que concluya el período de observación y comience el análisis (Figura 2).
Las colonias se documentan mediante un proceso estandarizado. Se utiliza el prefijo de vídeo en el que se encontraba la colonia (A1, A2..., B1, B2...), seguido del número de colonia. Por ejemplo, A2-6 sería el vídeo A2, colonia 6. El análisis comienza con el rastreo del linaje. Avance rápido hasta el final de un video para identificar las colonias, luego retroceda hasta el comienzo del período de observación para identificar la célula formadora de colonias original. Realice un seguimiento de las marcas de tiempo de todas las divisiones a medida que ocurren y cree un diagrama ramificado a mano, rastreando tantas generaciones como se pueda hacer con precisión (Figura 3). Eventualmente, ya no es posible rastrear con precisión las divisiones celulares debido a la densidad celular (esto generalmente sucede alrededor de la generación 5-7, dependiendo de si la colonia se originó a partir de una célula madre o de un progenitor comprometido). En generaciones posteriores, la colonia a menudo se fusiona con otra colonia, o la colonia sale de la pantalla. En este punto, marque la última celda rastreable como una U (no rastreable). Siempre tome una captura de pantalla de la colonia que se está rastreando (esto se puede hacer usando la función de instantánea en VLC) y marque la colonia usando la nomenclatura descrita anteriormente. Asegúrese de etiquetar la captura de pantalla con una nomenclatura estandarizada, incluido el video específico analizado y el número de colonia que se puede hacer coincidir con la captura de pantalla.
Una vez que se ha construido el diagrama de rama, los datos se pueden transferir a una hoja de cálculo o "hoja verde" (Archivo Complementario 1). La hoja verde contiene la misma etiqueta para cada colonia rastreada a través de un diagrama de ramas. Para identificar fácilmente las generaciones, los resaltados de color se alternan entre generaciones. El tiempo 1 de la generación 1 se refiere al período que transcurre desde la siembra de las células hasta su colocación en el microscopio de lapso de tiempo. En el Archivo Suplementario 1, el tiempo 1 de la generación 1 es de 24 h, ya que las celdas se colocan en la máquina 24 h después del enchapado. El tiempo 2 representa la duración hasta que la célula pasa por su primera división. Recuerde que la marca de tiempo en los videos no tiene en cuenta las 24 horas adicionales, por lo que las horas deben agregarse manualmente en cada generación. Al transcribir los datos del diagrama de ramificaciones a la hoja verde, agregue siempre 24 h al tiempo de las divisiones, ya que la marca de tiempo proporcionada por la máquina no tiene en cuenta el tiempo antes de que comience la grabación. ΔT es la duración del ciclo celular. La mayoría de los estudios 6,8 no incluyen el ΔT de la primera generación en los análisis, ya que siempre es mucho más largo que el de las generaciones posteriores y extremadamente variable, lo que da lugar a análisis sesgados.
Una vez que se construye la hoja verde, se puede determinar qué colonias se originan a partir de células madre y cuáles se originan a partir de progenitores comprometidos. Las colonias que muestran divisiones predominantemente proliferativas (Figura 1) y duran hasta el final del período de observación se consideran colonias de células madre6. Las colonias que se diferencian terminalmente (Figura 1) durante el período de observación se consideran colonias progenitoras comprometidas6. A continuación, se puede calcular el ΔT medio de las colonias de células madre y de las colonias de progenitores comprometidos, respectivamente. Al dividir la proporción de divisiones D sobre las divisiones totales, se puede calcular la proporción de divisiones de diferenciación, ya sea con todas las divisiones agrupadas de colonias de células madre / progenitores comprometidos, respectivamente, o por generación. La hoja verde es una forma útil de organizar los datos para obtener información de manera eficiente y evoluciona en función de los datos y estadísticas que se necesitan para los objetivos del estudio.
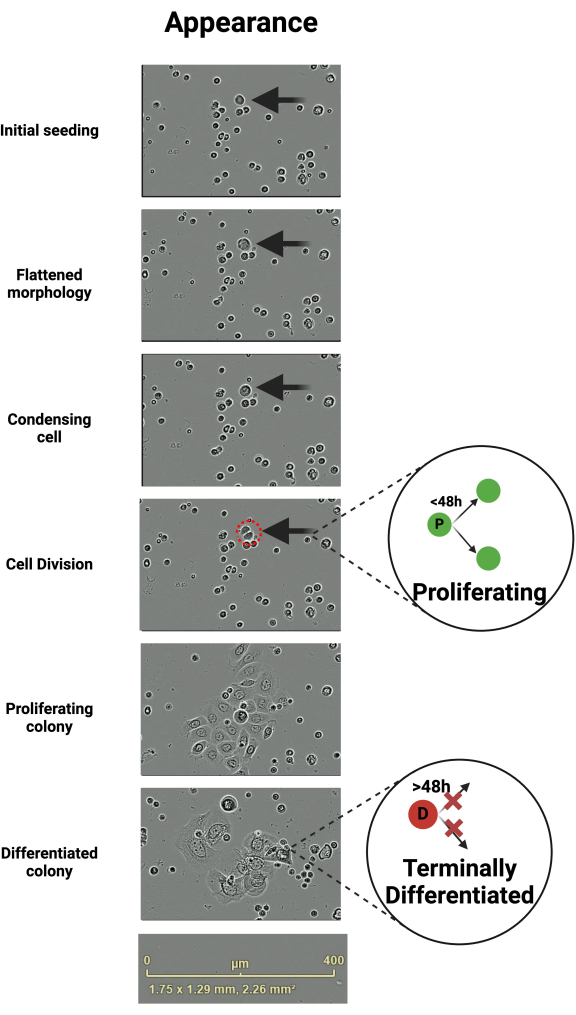
Figura 1: Cambios en la morfología de los queratinocitos que conducen a la división celular y terminología de división. Los queratinocitos progresan a través de una secuencia estereotipada de cambios morfológicos que conducen a la división celular. Esta figura representa un queratinocito sometido a esta secuencia de eventos a lo largo del tiempo. Las células individuales que están a punto de dividirse son móviles e inicialmente adquieren una morfología aplanada y se condensan centralmente inmediatamente antes de dividirse. Las células hijas que continuarán proliferando se dividen dentro de las 48 horas posteriores a su generación, de lo contrario, se consideran terminalmente diferenciadas. Barra de escala 400 μm. Creado en BioRender. Ghadially, R. (2025) https://BioRender.com/k40e714 Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Exportación de datos desde el microscopio time-lapse. Una guía sobre cómo exportar datos desde la máquina. Creado en BioRender. Ghadially, R. (2024) BioRender.com/x99d452 Haga clic aquí para ver una versión más grande de esta figura.
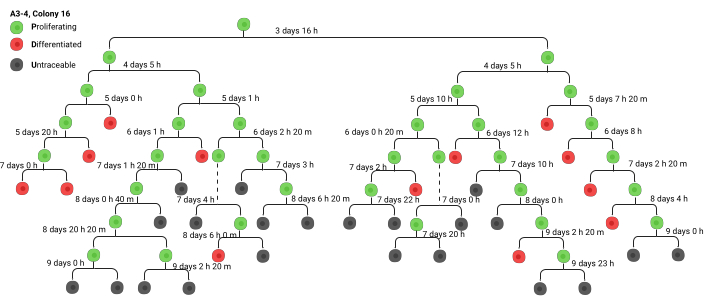
Figura 3. Diagrama de rama precursora (árbol de linaje) para informar la creación de hojas verdes. Un ejemplo de un árbol de linaje, normalmente dibujado a mano. Abreviaturas: h: horas, m: min. Creado en BioRender. Ghadially, R. (2025) https://BioRender.com/wbt7z8x Haga clic aquí para ver una versión más grande de esta figura.
Legajo Complementario 1. Ejemplos de hojas de datos (hojas verdes) de datos de imágenes de células vivas no publicadas. Un ejemplo de hoja verde que contiene datos no publicados. Haga clic aquí para descargar este archivo.
Video complementario 1. Ejemplo de vídeo de imágenes de células vivas de queratinocitos. Un video de imágenes de células vivas con una densidad celular adecuada para rastrear colonias de queratinocitos Haga clic aquí para descargar este archivo.
Discusión
La obtención de imágenes de células vivas de queratinocitos es un método sin etiquetas para rastrear el comportamiento de división de las células madre y los progenitores comprometidos. Dado que el mantenimiento de la epidermis depende de la cinética de proliferación de las células madre y de los progenitores comprometidos10, tener una comprensión granular de los cambios en estas poblaciones de queratinocitos y cómo se ven afectados en diversas condiciones facilita el desarrollo de terapias para mejorar los defectos que se descubren.
En última instancia, el rastreo del linaje a través de imágenes de células vivas depende de la obtención de datos utilizables. Las unidades formadoras de colonias deben ser claramente visibles en los videos generados por la fotografía de lapso de tiempo. Con células de pasaje 0 recién aisladas, es difícil obtener una verdadera densidad clonal. Solo el 3%-4% de los queratinocitos plateados finalmente forman colonias11. Demasiadas células pueden hacer que sea imposible rastrear las divisiones o incluso identificar la célula formadora de colonias inicial. Muy pocas células y es posible que no se formen colonias. Del mismo modo, cualquier cosa que oscurezca el campo visual hace que sea imposible rastrear las células a través del rastreo de linaje. El uso de alimentadores de fibroblastos puede dificultar el seguimiento de las divisiones, ya que los fibroblastos oscurecen el campo visual. Del mismo modo, los precipitados, los desechos celulares e incluso la condensación en la tapa de la placa pueden oscurecer las colonias en crecimiento. Incluso algo tan pequeño como no asegurarse de que la microplaca esté insertada de forma segura en la bahía seleccionada puede resultar en un experimento fallido, ya que la imagen no se enfocará en las células. Es por eso que es muy importante monitorear las colonias diariamente mientras crecen en la máquina y siempre permanecer hasta después del primer escaneo, cuando se retira la microplaca de la máquina para ver si hay cambios de medios, para asegurarse de que las células estén siendo capturadas por la fotografía de lapso de tiempo. Otro problema que pueden causar los escombros es el salto de imagen. Por lo general, el microscopio tiene campos visuales fijos que no se mueven, pero cuando se acumulan desechos o la densidad celular es demasiado alta, la máquina puede perder su campo de visión y saltar a una parte diferente de la placa. Para evitar esto, los fabricantes de la máquina desarrollaron un tipo especial de placa de 96 pocillos con una rejilla que evita la pérdida de enfoque.
La máquina genera calor mientras captura imágenes. Es importante reducir la temperatura de la incubadora que contiene el microscopio a 36,5 °C para que el dispositivo se equilibre a 37 °C al capturar imágenes. Cuando utilice placas con áreas de superficie más pequeñas (placas de 96 pocillos), considere el calor adicional generado. No utilice los pozos periféricos para experimentos (primera y última fila/columna) y considere llenar al máximo los pozos que rodean los pozos experimentales con otro fluido (PBS estéril), y utilice cinta adhesiva transpirable para reducir la pérdida de medios por evaporación. Hay placas comercializadas que podrían ser investigadas para minimizar los efectos de borde12. Sin embargo, los métodos antes mencionados han permitido el uso de microplacas compatibles recomendadas.
Al analizar videos, los investigadores deben ser conscientes de los patrones de división anormales de sus colonias. Por ejemplo, si no hay divisiones dentro de una vista durante 10 días y luego hay una formación desenfrenada de colonias que se origina desde el borde del campo de visión, es poco probable que sea la primera división de una nueva colonia. Es probable que una colonia en crecimiento de un campo visual circundante invadiera la porción de la placa capturada, en lugar de ser una colonia nueva. Esto se puede verificar iniciando sesión en el paquete de software, que puede representar la placa completa (que contiene todas las vistas registradas) en puntos de tiempo específicos y mostrar la migración de las celdas de un campo de visión a otro.
La principal limitación de este método es su naturaleza intensiva en mano de obra. Además, el seguimiento de las divisiones celulares más allá de la5ª generación es difícil y requiere horas de trabajo, volviendo a ver videos para capturar con precisión qué se está dividiendo y cuándo. Se están desarrollando múltiples algoritmos de seguimiento celular automatizados de aprendizaje profundo, que eventualmente resultarán en un análisis puramente basado en IA en los próximos años 7,13,14. Hasta entonces, el seguimiento manual, tal y como se detalla en este protocolo, es un método factible para desarrollar datos de linaje.
Divulgaciones
Ninguno.
Agradecimientos
Este trabajo fue apoyado por el Premio de Revisión al Mérito Número I01 CX001816 de los Estados Unidos (EE. UU.) Servicio de Investigación y Desarrollo de Ciencias Clínicas (CSRD) del Departamento de Asuntos de Veteranos. El contenido no representa los puntos de vista del Departamento de Asuntos de Veteranos de EE. UU. ni del Gobierno de los Estados Unidos. Agradecemos al Dr. Michael Rosenblum por proporcionarnos acceso a su microscopio de lapso de tiempo para realizar nuestros experimentos.
Materiales
| Name | Company | Catalog Number | Comments |
| 96 Well Imagelock plate | Sartorius | BA-04856 | Suggested microplate compatible with machine if using a 96 well plate. |
| 24 well plate | Corning | 3524 | Suggested microplate compatible with machine if using a 24 well plate. |
| Amphotericin B, 50 mL | Corning | 30-003-CF | Dilute to 5x (comes in 100x stock) for 5x PSA - 1x for media changes |
| Epilife, 50 mL | Gibco | MEP1500CA | Add S7, consider primocin |
| IncuCyte S3 | Sartorius | 4637 | Imager (Zoom/SX5 acceptable alternatives) |
| Penicillin/Streptomycin, 100 mL | Corning | 30-002-Cl | Dilute to 5x (comes in 100x stock) |
| Primocin | Invivogen | ant-pm-05 | 1 mL per 500 mL media |
| Supplement S7 | Gibco | S0175 | Added to epilife |
Referencias
- Mateu, R., et al. Functional differences between neonatal and adult fibroblasts and keratinocytes: Donor age affects epithelial-mesenchymal crosstalk in vitro. Int J Mol Med. 38 (4), 1063-1074 (2016).
- Handl, J., Čapek, J., Majtnerová, P., Báčová, J., Roušar, T. The effect of repeated passaging on the susceptibility of human proximal tubular HK-2 cells to toxic compounds. Physiol Res. 69 (4), 731-738 (2020).
- Moran, M. C., et al. Characterization of human keratinocyte cell lines for barrier studies. JID Innov. 1 (2), 100018(2021).
- Seo, M. -D., Kang, T. J., Lee, C. H., Lee, A. -Y., Noh, M. HaCaT keratinocytes and primary epidermal keratinocytes have different transcriptional profiles of cornified envelope-associated genes to T helper cell cytokines. Biomol Ther. 20 (2), 171-176 (2012).
- Jahn, M., et al. Different immortalized keratinocyte cell lines display distinct capabilities to differentiate and reconstitute an epidermis in vitro. Exp Dermatol. 33 (1), e14985(2024).
- Xiao, T., et al. Short cell cycle duration is a phenotype of human epidermal stem cells. Stem Cell Res Ther. 15 (1), 76(2024).
- Hirose, T., Kotoku, J., Toki, F., Nishimura, E. K., Nanba, D. Label-free quality control and identification of human keratinocyte stem cells by deep learning-based automated cell tracking. Stem Cells. 39 (8), 1091-1100 (2021).
- Roshan, A., Murai, K., Fowler, J., Simons, B. D., Nikolaidou-Neokosmidou, V., Jones, P. H. Human keratinocytes have two interconvertible modes of proliferation. Nat Cell Biol. 18 (2), 145-156 (2016).
- Abegaze, B., Ijeh, N., Vittimberga, B., Ghadially, R. Generating primary cultures for the purpose of keratinocyte live cell imaging. J Vis Exp. , In Press (2024).
- Piedrafita, G., et al. A single-progenitor model as the unifying paradigm of epidermal and esophageal epithelial maintenance in mice. Nat Commun. 11 (1), 1429(2020).
- Ścieżyńska, A., et al. Isolation and culture of human primary keratinocytes-a methods review. Exp Dermatol. 28 (2), 107-112 (2019).
- Mansoury, M., Hamed, M., Karmustaji, R., Al Hannan, F., Safrany, S. T. The edge effect: A global problem. The trouble with culturing cells in 96-well plates. Biochem Biophys Rep. 26, 100987(2021).
- Malin-Mayor, C., et al. Automated reconstruction of whole-embryo cell lineages by learning from sparse annotations. Nat Biotechnol. 41 (1), 44-49 (2023).
- Waliman, M., et al. Automated cell lineage reconstruction using label-free 4D microscopy. Genetics. 228 (2), iyae135(2024).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados