Method Article
Construcción de mutantes homocigotos de langostas migratorias utilizando tecnología CRISPR/Cas9
* Estos autores han contribuido por igual
En este artículo
Resumen
Este estudio proporciona un procedimiento sistemáticamente optimizado de la construcción basada en ribonucleasa CRISPR / Cas9 de mutantes de langosta homocigota, así como un método detallado para la criopreservación y reanimación de los huevos de langosta.
Resumen
La langosta migratoria, Locusta migratoria, no solo es una de las langostas de peste mundial que causó enormes pérdidas económicas a los seres humanos, sino también un importante modelo de investigación para la metamorfosis de insectos. El sistema CRISPR / Cas9 puede localizar con precisión un locus de ADN específico y escindirlo dentro del sitio objetivo, introduciendo eficientemente roturas de doble cadena para inducir la eliminación del gen objetivo o integrar nuevos fragmentos de genes en el locus específico. La edición del genoma mediada por CRISPR/Cas9 es una herramienta poderosa para abordar las preguntas encontradas en la investigación de la langosta, así como una tecnología prometedora para el control de la langosta. Este estudio proporciona un protocolo sistemático para la eliminación de genes mediados por CRISPR / Cas9 con el complejo de proteína Cas9 y ARN guía única (sgRNA) en langostas migratorias. La selección de los sitios diana y el diseño del sgRNA se describen en detalle, seguidos de la síntesis in vitro y la verificación de los sgRNAs. Los procedimientos posteriores incluyen la recolección de balsas de huevos y la separación de huevos curtidos para lograr una microinyección exitosa con baja tasa de mortalidad, cultivo de huevos, estimación preliminar de la tasa de mutación, reproducción de langostas, así como detección, preservación y paso de los mutantes para garantizar la estabilidad de la población de las langostas editadas. Este método se puede utilizar como referencia para aplicaciones de edición de genes basadas en CRISPR / Cas9 en langostas migratorias, así como en otros insectos.
Introducción
Las tecnologías de edición de genes podrían utilizarse para introducir inserciones o deleciones en un locus específico del genoma para modificar artificialmente el gen objetivo a propósito1. En los últimos años, la tecnología CRISPR/Cas9 se ha desarrollado rápidamente y tiene un alcance creciente de aplicaciones en diversos campos de las ciencias de la vida 2,3,4,5,6. El sistema CRISPR/Cas9 fue descubierto en 19877, y se encuentra ampliamente en bacterias y arqueas. Investigaciones posteriores indicaron que se trataba de un sistema inmune adaptativo procariota que depende de la nucleasa Cas9 guiada por ARN para luchar contra los fagos8. El sistema CRISPR/Cas9 modificado artificialmente consta principalmente de dos componentes, un único ARN guía (sgRNA) y la proteína Cas9. El sgRNA está formado por un ARN CRISPR (crRNA) complementario a la secuencia diana y un crRNA transactivador auxiliar (tracrRNA), que está relativamente conservado. Cuando se activa el sistema CRISPR/Cas9, el sgRNA forma una ribonucleoproteína (RNP) con la proteína Cas9 y guía a Cas9 a su sitio objetivo a través del emparejamiento de bases de las interacciones ARN-ADN. Luego, el ADN de doble cadena puede ser escindido por la proteína Cas9 y, como resultado, la ruptura de doble cadena (DSB) emerge cerca del motivo adyacente al protoespaciador (PAM) del sitio objetivo 9,10,11,12. Para mitigar el daño causado por los DSB, las células activarían respuestas integrales al daño del ADN para detectar eficientemente los daños genómicos e iniciar el procedimiento de reparación. Hay dos mecanismos de reparación distintos en la célula: unión de extremos no homólogos (NHEJ) y reparación dirigida por homología (HDR). NHEJ es la vía de reparación más común que puede reparar las roturas de doble cadena del ADN rápidamente y previene la apoptosis celular. Sin embargo, es propenso a errores debido a dejar pequeños fragmentos de inserciones y deleciones (indeles) cerca de los DSB, lo que generalmente resulta en un cambio de marco de lectura abierto y, por lo tanto, puede conducir a la eliminación de genes. En contraste, la reparación homóloga es un evento bastante raro. Con la condición de que haya una plantilla de reparación con secuencias homólogas al contexto del DSB, las células ocasionalmente repararían la ruptura genómica de acuerdo con la plantilla cercana. El resultado de HDR es que el DSB se repara con precisión. Especialmente, si hay una secuencia adicional entre las secuencias homólogas en la plantilla, se integrarían en el genoma a través de HDR, y de esta manera, las inserciones genéticas específicas podrían realizarse13.
Con la optimización y el desarrollo de las estructuras sgRNA y las variantes de la proteína Cas9, el sistema de edición genética basado en CRISPR / Cas9 también se ha aplicado con éxito en la investigación de insectos, incluidos, entre otros, Drosophila melanogaster, Aedes aegypti, Bombyx mori, Helicoverpa Armigera, Plutella xylostella y Locusta migratoria 14,15,16,17,18 ,19. Hasta donde saben los autores, aunque las RNP que consisten en la proteína Cas9 y el sgRNA transcrito in vitro se han utilizado para la edición del genoma de la langosta20,21,22, todavía falta un protocolo sistemático y detallado para la construcción mediada por ribonucleasa CRISPR / Cas9 de mutantes homocigotos de la langosta migratoria.
La langosta migratoria es una plaga agrícola importante que tiene una distribución mundial y representa amenazas sustanciales para la producción de alimentos, siendo especialmente dañina para las plantas gramíneas, como el trigo, el maíz, el arroz y el mijo23. El análisis de la función génica basado en tecnologías de edición del genoma puede proporcionar nuevos objetivos y nuevas estrategias para el control de las langostas migratorias. Este estudio propone un método detallado para eliminar genes de langostas migratorias a través del sistema CRISPR / Cas9, incluida la selección de sitios objetivo y el diseño de sgRNAs, síntesis in vitro y verificación de los sgRNAs, microinyección y cultivo de óvulos, estimación de la tasa de mutación en la etapa embrionaria, detección de mutantes, así como paso y preservación de los mutantes. Este protocolo podría usarse como referencia basal para la manipulación de la gran mayoría de los genes de langostas y puede proporcionar referencias valiosas para la edición del genoma de otros insectos.
Protocolo
1. Selección del sitio objetivo y diseño de sgRNA
- Recopilar tanta información de secuencia como sea posible para el gen de interés a través de la investigación bibliográfica y/o la búsqueda del ARNm o la secuencia de ADN codificante (CDS) del gen en la base de datos NCBI y langosta24.
- Compare la secuencia del gen interesado con su secuencia de ADN genómico para distinguir las regiones de exón e intronio.
- Seleccione una región candidata para el diseño del sitio de destino en función del propósito de la investigación. Diseñe pares de cebadores para amplificar el fragmento de la región candidata y verificar su secuencia de tipo salvaje mediante el análisis de secuenciación del producto de PCR.
NOTA: Existen diferentes estrategias para la selección de la región objetivo candidata. Por ejemplo, en la mayoría de las investigaciones sobre la función de genes / proteínas, use el fragmento de exón cerca de su codón de inicio como la región candidata para asegurarse de que toda la función de proteína / ARN se pierda después de la edición del genoma (es decir, eliminación de genes). Mientras que para el análisis de funciones de un dominio específico, seleccione la región candidata al principio (o ambos extremos) del dominio. - Utilice recursos en línea como E-CRISP Design25 para buscar posibles sitios objetivo en la región objetivo candidata.
- Seleccione "Drosophila melanogaster BDGP6" (o cualquier otro insecto) en la lista desplegable de genomas de referencia y elija "La entrada es la secuencia FASTA" seguido de pegar la secuencia de la región objetivo en el cuadro de texto (en formato FASTA).
- Inicie la aplicación presionando "Iniciar búsqueda de sgRNA" con "medio" y "diseño único" para obtener los posibles sitios de destino. Seleccione 1-3 objetivos potenciales de los resultados en función de sus puntuaciones previstas y diseñe los sgRNAs correspondientes en consecuencia.
NOTA: Hay muchas más plataformas en línea para el diseño CRISPR, como CRISPOR, CHOPCHOP, CRISPRdirect, ZiFiT, etc. (consulte la Tabla de materiales). Algunos de ellos tienen la función de verificación de especificidad para las especies cuyos datos de secuencia genómica están en sus bases de datos. Sin embargo, la secuencia genómica de las langostas no se ha encontrado en ningún sitio web en línea. Es mejor usar más de una herramienta en línea para el diseño CRISPR en langostas. De manera exhaustiva, considere los resultados de diferentes sitios web y seleccione el sgRNA de los resultados según el principio de mayor especificidad, mayor eficiencia y menor tasa de desajuste (Figura 1A, B).
2. Síntesis y verificación del sgRNA in vitro
- Sintetice el sgRNA utilizando kits de síntesis de sgRNA de acuerdo con el manual del fabricante (Tabla de materiales). Este procedimiento generalmente incluye tres pasos: amplificación de la plantilla de ADN, transcripción in vitro y purificación de sgRNA21. Diluir el sgRNA sintetizado con agua libre de nucleasas a una concentración de almacenamiento de 300 ng/μL para su uso posterior.
- Amplificar y purificar el fragmento del gen diana para que sirva como sustrato del ensayo de escisión in vitro del sistema CRISPR/Cas9. Realice estos pasos siguiendo las instrucciones del fabricante.
- Diluir la proteína Cas9 comprada a 300 ng/μL con agua libre de nucleasas. Incubar 1 μL de sgRNA (300 ng/μL) y 1 μL de proteína Cas9 (300 ng/μL) con 200 ng de fragmento diana purificado en el tampón de escisión a 37 °C durante 1 h (10 μL de volumen total; ver Tabla 1). Estimar la eficiencia de la escisión de Cas9 inducida por sgRNA mediante electroforesis en gel de agarosa (Figura 1C). Seleccione los sgRNAs con alta actividad para la siguiente microinyección.
NOTA: Los resultados de la electroforesis en gel de agarosa se pueden convertir en imágenes en escala de grises con software de procesamiento de imágenes y la eficiencia de la escisión se estima de acuerdo con la escala de grises de las bandas de tamaños especulados.
3. Microinyección y cultivo de los huevos
- Prepare las agujas de inyección tirando de los capilares de vidrio con un extractor de micropipetas (Tabla de materiales). Establezca los parámetros de la siguiente manera: Calor a 588, Tirar a 90, Velocidad a 60 y Tiempo a 40. Moler la punta de la aguja de inyección con un micro molinillo (Tabla de materiales).
NOTA: Las puntas ideales de las agujas son abiertas y de bordes afilados (Figura 2A). Se recomienda encarecidamente preparar agujas adicionales para los experimentos porque las agujas a veces se bloquean o se rompen accidentalmente durante las inyecciones. - Mezclar 1 μL de proteína Cas9 (300 ng/μL) con 1 μL del sgRNA verificado (300 ng/μL) para obtener los RNPs para inyección. Agregue 8 μL de agua estéril libre de RNasa para que el volumen final de la mezcla sea de 10 μL. Mezcle bien la solución y manténgala en hielo.
NOTA: Las concentraciones finales de la proteína Cas9 y los sgRNAs se pueden optimizar de acuerdo con los resultados de edición. Evite la congelación y descongelación repetitiva de la solución de RNP preparada y se recomienda su uso inmediato. - Criar las langostas adultas macho y hembra juntas a 30 °C con un fotoperiodo 16 (claro):8 (oscuro) y suministrarles suficientes plántulas de trigo fresco como alimento. Observe estas langostas diariamente y coloque una maceta de oviposición (una maceta de plástico o una taza llena de arena estéril húmeda) en la jaula de cría para la oviposición una vez que las langostas se aparearon.
NOTA: Por lo general, 100 parejas de langostas adultas criadas en una jaula de cría (40 cm x 40 cm x 40 cm) son suficientes para generar huevos para eliminar un solo gen. Cultive pares adicionales de langostas si se necesitan más huevos. - Recoja las vainas de huevo recién puestas de la olla de oviposición y espere unos 30 minutos para el bronceado del huevo. Aísle suavemente los huevos de las vainas de huevo en agua con un cepillo fino y lávelos con agua estéril tres veces. Mantenga los huevos en una placa de Petri y agregue agua estéril para mantenerlos húmedos.
NOTA: El curtido de los huevos recién puestos puede mejorar significativamente la eficiencia mutante21. - Llene una aguja de inyección con la solución de RNP preparada y cárguela en el micromanipulador (Tabla de materiales).
- Ajuste los parámetros de microinyección de la siguiente manera: 300 hPa de la presión de inyección (pi), 0,5 s del tiempo de inyección (ti) y 25 hPa de la presión de compensación (pc).
- Presione el pedal para evaluar el volumen de la solución inyectada. Ajuste la presión de inyección y el tiempo de inyección para asegurarse de que el volumen de inyección sea de aproximadamente 50-100 nL.
NOTA: Expulse el aire residual en la aguja para que la solución de inyección sea continua y controlable tanto como sea posible. La conexión de la aguja y el micromanipulador debe ser estrecha.
- Coloque los huevos estériles regularmente en la almohadilla de inyección (Figura 2B, C) y coloque la almohadilla en la mesa de objetos del microscopio (Figura 3A). Ajuste el aumento del microscopio hasta que se observen los huevos. Ajuste la aguja de microinyección bajo el microscopio hasta que se pueda ver la punta de inyección y colóquela cerca del huevo que se inyectará.
- Comience la inyección a una altura adecuada y un ángulo de 30-45 grados. Inserte la punta en el huevo cuidadosamente cerca de los micropyles del huevo (Figura 3B) y presione el pedal para realizar la inyección. Retraiga la aguja rápidamente y mueva la almohadilla de inyección para inyectar el siguiente óvulo.
NOTA: Se debe observar una ligera expansión del huevo durante la microinyección. Una pequeña cantidad de fuga citoplasmática en el agujero de alfiler es aceptable. Reemplace la aguja de inyección por una nueva o ajuste el ángulo de inyección si la salida de líquido es demasiado. - Transfiera los huevos inyectados a una placa de cultivo (una placa de Petri con un trozo de papel de filtro húmedo) y colóquelos en una incubadora a 30 °C.
NOTA: Pasarán unos 13-14 días hasta que las ninfas eclosionen de estos huevos inyectados (a condición de que la mutación del gen diana no afecte el desarrollo de los embriones) (Figura 3C). Inyecte al menos 100 huevos para asegurar una cantidad suficiente de eclosión y eclosión para eliminar un gen específico.
4. Estimación de la tasa de mutación y cribado de mutantes
- Verifique el desarrollo de los óvulos inyectados todos los días después de la inyección. Transfiera los huevos inyectados a una nueva placa de cultivo cada 24 h en los primeros 5 días.
- En el6º día (o más tarde) después de la inyección, recoja y transfiera al azar 10 huevos a un tubo y muele adecuadamente los huevos con dos bolas de acero a 60 Hz durante 6 minutos usando un molinillo (consulte la Tabla de materiales). Resuspender los desechos con 1 ml de PBS. Transfiera 5 μL de la mezcla a 45 μL de NaOH a 50 mM y lílea a 95 °C durante 5 min. Añadir 5 μL de 1 M Tris-HCl (pH=8,0) en el sistema de lisis para terminar la reacción de lisis alcalina.
NOTA: Los huevos se pueden seleccionar para la lisis alcalina en cualquier día después de la última transferencia y se pueden usar múltiples óvulos como una muestra dependiendo de su situación de desarrollo (por ejemplo, cinco embriones de 6 días se pueden usar como una muestra, y dos embriones de 10 días se pueden usar como una muestra). A veces, no es factible obtener el ADN genómico de los huevos utilizando el método de lisis alcalina. Los kits comerciales de extracción de ADN genómico se pueden utilizar como un método alternativo para aislar el ADN genómico de los óvulos. - Tome 1 μL del producto de lisis como plantilla de PCR para amplificar el fragmento del gen diana (Tabla 2 y Tabla 3) y envíe los productos de PCR para su secuenciación. Compare los resultados de la secuenciación con la secuencia de tipo salvaje para evaluar preliminarmente si el sistema CRISPR/Cas9 escindió el gen diana in vivo (Figura 4A). Permitir el resto de los óvulos para su posterior desarrollo a condición de que los indeles se detecten en la etapa embrionaria.
NOTA: De esta manera, la tasa de mutación se puede estimar preliminarmente en la etapa embrionaria. Si no se encuentran indeles en este paso, prepare algunos nuevos sgRNAs para el gen diana y repita el protocolo knock-out del paso 2.1. - Transfiera las ninfas eclosionadas a una jaula de cría y críelas como se describe en el paso 3.3. Cuando las ninfas se desarrollaron a la quinta etapa de estadio, sepárelas con vasos de cultivo de plástico (1 ninfa / taza).
NOTA: Por lo general, las ninfas tardan entre 25 y 35 días en desarrollarse hasta su quinto estadio y el fenotipo más obvio es que las yemas de las alas se extienden hasta el cuarto o quinto segmento abdominal. Además, se recomienda registrar los fenotipos de ninfas muertas para predecir la relación entre el gen objetivo y los fenotipos en futuras investigaciones. - Cortar aproximadamente 2 mm de longitud de las antenas con tijeras de disección y lisarlo utilizando el método de lisis alcalina (paso 4.2). Analice la secuencia del fragmento del gen objetivo de estas ninfas como se describió anteriormente (paso 4.3) para identificar los mutantes G0 (Figura 4B).
NOTA: Las antenas cortadas para la lisis pueden ser un poco más largas y moler o picar las antenas es útil para la lisis, aunque las antenas se pueden digerir directamente. Los individuos con múltiples picos cerca del sitio objetivo en el resultado de la secuenciación se identifican como mutantes positivos y se les permite el desarrollo y el apareamiento posteriores.
5. Establecimiento y paso de líneas mutantes
- Realizar cruzamientos utilizando los mutantes G0 y langostas de tipo salvaje (Figura 4B). Recoger los ooquistes e incubar estos ooquistes por separado a 30 °C. Use 3-6 óvulos desarrollados en cada ooquiste para detectar mutaciones como se describe en los pasos 4.2 y 4.3. Mantener los ooquistes con mutación positiva para el desarrollo posterior y abandonar los ooquistes con mutación negativa.
NOTA: La estimación de la tasa de mutación en la etapa embrionaria puede acelerar en gran medida la detección de mutantes G1 . Por lo general, 3-6 óvulos desarrollados se pueden mezclar como una muestra para el genotipado mediado por PCR como se describió anteriormente. - Criar las ninfas G1 como se describe en el paso 4.4. Corte aproximadamente 2 mm de longitud de las antenas para realizar el genotipado basado en PCR como se describe en el paso 4.5 para detectar heterocigotos G1 . Realice la clonación de TA de acuerdo con las instrucciones del fabricante (consulte la Tabla de materiales) para identificar sus mutaciones. Realizar in-cross utilizando heterocigotosG1 con las mismas mutaciones para obtener ninfasG2 .
NOTA: La secuenciación de Sanger de los productos de PCR solo puede proporcionar información para descubrir los heterocigotos, pero no suficiente información para identificar las mutaciones exactas. Por lo tanto, la clonación de TA utilizando los productos de PCR es necesaria para identificar claramente las mutaciones y puede promover el establecimiento de líneas mutantes estables. - Cría a las ninfas G2 hasta su quinto estadio. Utilice el genotipado basado en PCR descrito en el paso 5.2 para identificar los homocigotos y/o heterocigotos que son adecuados para futuras investigaciones y pases estables (Figura 4B).
NOTA: Preste atención para evitar la mezcla de homocigotos y heterocigotos. Se recomienda realizar genotipado basado en PCR en cada generación para confirmar la homocigosidad y/o heterocigosidad de cada población. El número de langostas utilizadas para este control depende del tamaño de la población. Además, la población de langostas puede ampliarse mediante la estrategia de cruce.
6. Criopreservación y reanimación de óvulos
- Lave los huevos a criopreservar con agua estéril e incubarlos en una placa de cultivo como se describe anteriormente (paso 3.8) durante 5-6 días. Reúna estos huevos en la placa de cultivo y cúbralos con fragmentos de papel de filtro. Envuelva todo el plato de cultivo con una película de parafina.
- Mantener el plato a 25 °C durante 2 días seguidos de otros 2 días a una temperatura relativamente más baja (13-16 °C). A continuación, transfiera el plato a una nevera a 6 °C. Agregue agua cada 2 semanas para proporcionar un ambiente húmedo para los huevos (Figura 5A).
NOTA: Se especula que los embriones están en la etapa de katatrepsis después de haber sido desarrollados durante 5-6 días a 30 ° C26. El enfriamiento por gradiente es útil para la criopreservación de óvulos (Tabla 5). - Para resucitar los huevos criopreservados, saque la placa de Petri del refrigerador y manténgala a 25 °C durante 2 días. Coloque estos huevos en una incubadora a 30 °C hasta que las ninfas eclosionen (Figura 5B).
NOTA: Es necesario poner los huevos criopreservados a 25 °C antes de transferirlos a una incubadora a 30 °C. Los embriones pueden ser criopreservados durante al menos cinco meses, aunque la tasa de eclosión y la tasa de eclosión pueden disminuir debido a la criopreservación (Tabla 5).
Resultados
Este protocolo contiene los pasos detallados para generar mutantes homocigotos de las langostas migratorias con el RNP constituido por la proteína Cas9 y sgRNA sintetizado in vitro . Los siguientes son algunos resultados representativos de la eliminación del gen diana mediado por CRISPR/Cas9 en langostas, incluida la selección de dianas, la síntesis y verificación de sgRNA (Figura 1A), la recolección e inyección de óvulos, la detección y el paso de mutantes, la criopreservación y la reanimación de los huevos homocigotos.
En este estudio, el sitio objetivo para el sistema CRISPR / Cas9 se selecciona de acuerdo con los resultados de tres programas en línea (E-CRISP, CRISPOR y ZiFit) y se encuentra en el primer exón (Figura 1B). De acuerdo con el ensayo de escisión Cas9 in vitro (Tabla 1), el RNP CRISPR/Cas9 podría digerir el fragmento de PCR (que contiene el sitio objetivo) con una tasa de escisión de aproximadamente el 55% (Figura 1C). Luego, este RNP fue microinyectado en 120 óvulos fertilizados en su etapa unicelular utilizando el sistema de microinyección estándar (Figura 2 y Figura 3). Los resultados de la secuenciación para la estimación de la tasa de mutación en estadio embrionario sugirieron una edición eficiente del genoma en el sitio objetivo (Figura 4A). Además, este estudio resultó en una tasa de eclosión de ninfas del 52,73% y el 66,7% de los adultos G0 eran mutantes mosaico (Tabla 4).
Además, las quimeras G0 se cruzaron con langostas de tipo salvaje para obtener individuos heterocigotos G 1 y los heterocigotos G1 con las mismas mutaciones (cribados por clonación TA) se cruzaron para generar los animales G2. Se utilizó fenotipado basado en PCR para detectar mutantes y los homocigotos obtenidos se cruzaron para establecer líneas mutantes estables (Figura 4B).
Mientras tanto, el exceso de huevos homocigotos fueron criopreservados para mejorar la tasa de utilización de los homocigotos. Aunque la tasa de eclosión de los huevos criopreservados se redujo con la extensión del tiempo de criopreservación (Tabla 5), las acciones correctivas que incluyen la aplicación de fragmentos de papel de filtro para mantener los huevos húmedos y recuperar estos huevos preservados con un gradiente de aumento de la temperatura fueron útiles para la reanimación (Figura 5 y Tabla 5). Finalmente, la población homocigótica de mutantes se mantuvo con éxito para investigaciones posteriores.
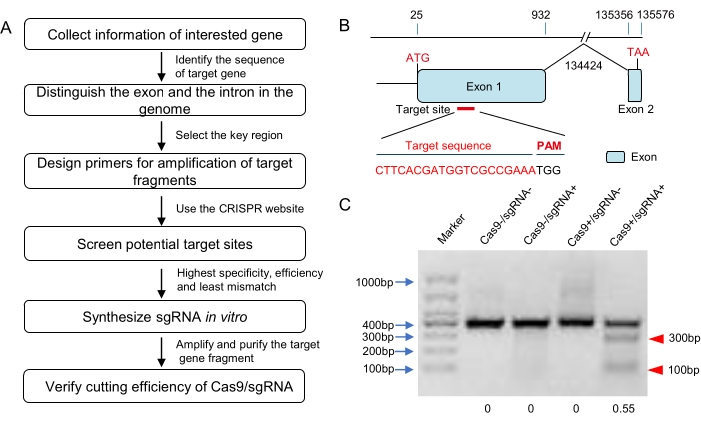
Figura 1: Diseño del objetivo y verificación in vitro del sgRNA. (A) El diagrama de flujo para la selección del objetivo, la síntesis de sgRNA y la verificación de la bioactividad in vitro (incluidas, entre otras, las langostas). (B) Diagrama esquemático de la estructura del gen y el sitio objetivo. Los exones de este gen diana se muestran como dominios azules y el sitio objetivo se seleccionó aguas abajo del codón de inicio en el exón 1. La secuencia de destino se resalta en rojo. (C) El resultado de la electroforesis en gel de agarosa del ensayo de escisión in vitro Cas9. Un fragmento de ADN que albergaba la secuencia objetivo (alrededor de 400 pb de longitud) se amplificó y se utilizó como sustrato para la digestión de Cas9. Las bandas pequeñas esperadas (alrededor de 100 pb y 300 pb aquí; marcadas con triángulos rojos) sugirieron que el sgRNA sintetizado podría inducir una escisión efectiva de Cas9. La tasa de escisión fue de alrededor del 55% según el análisis de escala de grises. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Preparación de la aguja y la almohadilla de microinyección para huevos de langosta. (A) Punta de una aguja preparada para microinyección de huevos de langosta. Barra de escala: 1 mm. (B) Imagen de una almohadilla de microinyección con huevos (indicados con un triángulo rojo) dispuestos en ella. La barra de escala representa 1 cm. (C) El tamaño de la almohadilla de microinyección. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Microinyecciones de huevo. (A) Un sistema representativo de microinyección etiquetado con cajas rojas que indican un microscopio (centro) y un micromanipulador (derecha) conectado a un microinyector (izquierda). La computadora se utiliza para el almacenamiento de datos y para proporcionar observación auxiliar. B) Inyección del huevo de langosta. El lugar de inyección está marcado con un triángulo rojo. (C) Una ninfa eclosionada bajo el microscopio. Las barras de escala representan 1 mm. Haga clic aquí para ver una versión más grande de esta figura.
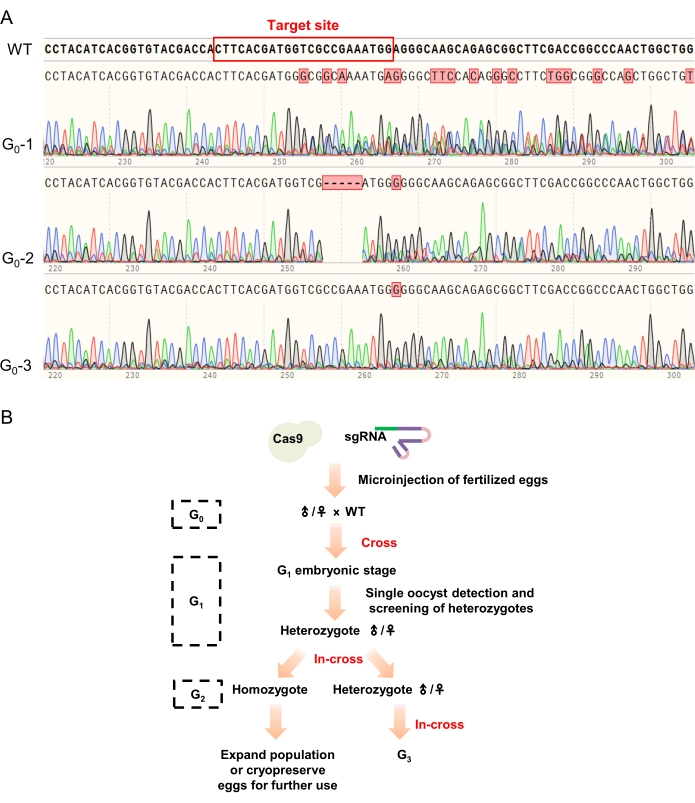
Figura 4: Los resultados de secuenciación de los animales G0 y la estrategia de paso de las líneas mutantes. (A) La secuenciación típica da como resultado el cribado G0. Múltiples picos cerca del sitio objetivo (marcados con un rectángulo rojo en la secuencia de tipo salvaje) indicaron que el huevo / langosta probado fue editado con éxito por el sistema CRISPR / Cas9 (como se muestra en G 0-1 y G0-2), mientras que el individuo sin ningún cambio en el resultado de la secuenciación se consideró como no editado y abandonado (por ejemplo, G0-3). (B) La estrategia de paso de las líneas mutantes. Los animales G0 fueron examinados primero por genotipado mediado por PCR y aquellos con múltiples picos en sus resultados de secuenciación se cruzaron con langostas de tipo salvaje para generar los animales G1. Se utilizó genotipado mediado por PCR y clonación de AT para identificar los heterocigotosG1. Luego, heterocigotos con las mismas mutaciones se cruzaron para generar los animales G2 (homocigotos y heterocigotos) para una mayor investigación y paso. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Criopreservación y reanimación de huevos. (A) El procedimiento de criopreservación: Aislar los huevos de las vainas de huevo e incubarlos a 30 °C durante 5-6 días después del lavado con agua estéril. Luego, reúna los huevos desarrollados en la placa de Petri y cúbralos con pequeños trozos de papel de filtro húmedo. Envuelva toda la placa de Petri con una película de parafina y manténgala a 25 °C durante 2 días, seguido de otros 2 días a una temperatura relativamente más baja (por ejemplo, 13-16 °C). Finalmente, refrigerar a 6 °C. (B) El procedimiento de reanimación: Saque las placas de Petri con huevos criopreservados del refrigerador y manténgalas a 25 ° C durante 2 días después de retirar los restos de papel de filtro. Luego, los huevos pueden ser cultivados para su posterior desarrollo a 30 °C hasta que las ninfas eclosionen. Haga clic aquí para ver una versión más grande de esta figura.
| Reactivo | Volumen (μL) |
| Proteína Cas9 (300 ng/μL) | 1 |
| sgRNA (300 ng/μL) | 1 |
| Producto de PCR (200 ng/μL) | 1 |
| 10×NEBuffer r3.1 | 1 |
| Agua libre de nucleasas | 6 |
Tabla 1: Sistema de digestión in vitro . Se mezclaron 200 ng de fragmento diana purificado (producto de PCR) con el sistema CRISPR/Cas9 (300 ng de cada componente) para probar la bioactividad de los sgRNAs sintetizados. Se utilizó un tampón comercial y se agregó agua libre de nucleasas para completar el volumen total a 10 μL.
| Reactivo | Volumen (μL) |
| 2xEs Taq MasterMix | 12.5 |
| Cebador hacia adelante | 0.5 |
| Cebador inverso | 0.5 |
| Plantilla (producto de lisis) | 1 |
| Agua libre de nucleasas | 10.5 |
Tabla 2: Sistema de PCR para amplificación de fragmentos de genes diana. Para amplificar el fragmento genómico del gen diana, se utilizó una mezcla comercial de Taq y se agregaron cebadores de acuerdo con las instrucciones del fabricante. Se utilizó 1 μL del producto de lisis como plantilla y agua libre de nucleasas para hacer el volumen total a 25 μL.
| Temperatura (°C) | Hora | Ciclos |
| 95 | 5 minutos | 1 |
| 95 | 30 s | 35 |
| 55 | 30 s | |
| 72 | 40 s | |
| 72 | 10 minutos | 1 |
Tabla 3: Programa de PCR para amplificación del gen diana. El programa de PCR para la amplificación del gen diana fue: 95 °C durante 5 min; 35 ciclos de 95 °C durante 30 s, 55 °C durante 30 s, 72 °C durante 40 s; 72 °C durante 10 min.
| Artículos | Datos |
| Nº de huevos inyectados | 120 |
| Embriones probados | 10 |
| Nº de ninfas eclosionadas | 58 |
| Tasa de eclosión | 52.73% |
| Nº de G0 adulto | 12 |
| Nº de mutantes G0 | 8 |
| Eficacia de la mutación en adultos G0 | 66.67% |
Tabla 4: Resumen de la eficiencia de edición. Se inyectaron 120 óvulos para eliminar el gen objetivo y se tomaron 10 óvulos para realizar pruebas durante la etapa embrionaria. 58 ninfas nacieron del resto de embriones (la tasa de eclosión fue del 52,73%). Finalmente, 12 langostas G0 se desarrollaron hasta la etapa adulta y 8 de ellas fueron editadas con éxito en el sitio objetivo. La tasa de mutación en adultos G0 fue del 66,67%.
| Grupo de control | Grupo 1 | Grupo 2 | Grupo 3 | Grupo 4 | |
| Nº de huevos | 120 | 120 | 120 | 120 | 120 |
| Tratamiento de temperatura para almacenamiento | 30 °C | 30°C (5-6 d)→6 °C | 30°C (5-6 d)→25°C (2 d)→13-16°C (2 d)→6 °C | 30°C (5-6 d)→25°C (2 d)→13-16°C (2 d)→6 °C | 30°C (5-6 d)→25°C (2 d)→13-16°C (2 d)→6 °C |
| Tiempo de almacenamiento | 14 días | 14 días | 1 mes | 3 meses | 5 meses |
| Tratamiento de temperatura para reanimación | 30 °C | 6°C→30 °C | 6°C→25°C (2 d)→30 °C | 6°C→25°C (2 d)→30 °C | 6°C→25°C (2 d)→30 °C |
| Nº de langostas eclosionadas | 108 | 0 | 96 | 86 | 78 |
| Tasa de eclosión | 90.00% | 0 | 80.00% | 71.67% | 65.00% |
| Nº de langostas adultas | 81 | 0 | 60 | 46 | 31 |
| Tasa de eclosión | 75.00% | 0 | 62.50% | 53.49% | 39.74% |
Tabla 5: Resumen de la criopreservación y reanimación de óvulos. Se dividieron 600 óvulos en cinco grupos para estudio de criopreservación y reanimación. El primer grupo (control) de 120 huevos siempre se incubó a 30 °C y se almacenó durante 14 días. El segundo grupo (Grupo 1) de 120 huevos se transfirió a un refrigerador después de la incubación a 30 °C durante 5-6 días y se almacenó a 6 °C durante 14 días. Luego, estos huevos se transfirieron a 30 ° C para su reanimación. Los otros grupos (grupo 2, grupo 3 y grupo 4, cada grupo contenía 120 huevos) experimentaron un tratamiento de enfriamiento y recuperación de temperatura de gradiente como se describe en el protocolo (pasos 6.1-6.3) con un tiempo de almacenamiento diferente (1 mes para el grupo 2, 3 meses para el grupo 3 y 5 meses para el grupo 4). Finalmente, 108 ninfas nacieron del grupo de control (a una tasa de eclosión del 90%) y 81 de ellas se desarrollaron hasta la etapa adulta (75% de la tasa de eclosión). No nacieron langostas en el segundo grupo (Grupo 1). La tasa de eclosión y la tasa de eclosión de los otros grupos fueron menores en comparación con el grupo control y disminuyeron con el tiempo de almacenamiento. 96 ninfas eclosionaron en el grupo 2 y 60 de ellas se desarrollaron hasta la etapa adulta. 86 ninfas eclosionaron en el grupo 3 y 46 de ellas se desarrollaron hasta la etapa adulta. En el grupo 4, 78 ninfas eclosionaron y 31 de ellas se desarrollaron hasta la etapa adulta.
Discusión
Las langostas han sido una de las plagas más devastadoras para la agricultura desde la civilización de los seres humanos23. La tecnología de edición del genoma basada en CRISPR/Cas9 es una herramienta poderosa para proporcionar un mejor conocimiento de los mecanismos biológicos en las langostas, así como una estrategia prometedora de control de plagas. Por lo tanto, es de gran beneficio desarrollar un método eficiente y fácil de usar para la construcción mediada por CRISPR / Cas9 de mutantes de langosta homocigota. Aunque se han reportado algunos grandes trabajos y proporcionado un flujo de trabajo básico para la edición del genoma en langostas 18,20,21,22, todavía falta una optimización sistemática de todo el procedimiento. En general, el mutante de langosta homocigótica generado por inyección embrionaria del sistema CRISPR/Cas9 se puede dividir en tres pasos principales: a) diseño, síntesis y cribado de sgRNA in vitro, b) recolección, preparación e inyección de óvulos; y c) cribado mutante y mantenimiento homocigoto. Este protocolo proporciona un flujo de trabajo optimizado y los pasos de selección del sitio objetivo, detección in vitro de sgRNA, microinyección de los óvulos y detección de mutantes son especialmente críticos para generar langostas mutantes homocigotas de manera efectiva con el sistema CRISPR / Cas9.
En primer lugar, el sitio objetivo, la forma del sgRNA correspondiente y la concentración de sgRNA han sido sugeridos como los factores críticos de la eficiencia de edición del genoma en insectos18,27,28. Para resolver este problema, se recomienda analizar primero la estructura del gen y diseñar más de un sitio objetivo en el exón 1 o cerca del codón de inicio utilizando los recursos en línea. Luego, sintetice sgRNA in vitro y mézclelo con la proteína Cas9 a diferentes concentraciones para optimizar la eficiencia de corte de RNP. Este procedimiento ayudará a identificar un sgRNA con alta bioactividad y optimizar la composición del complejo RNP para el gen diana candidato.
En segundo lugar, recolectar tantos huevos frescos como sea posible en un tiempo limitado y mejorar la tasa de eclosión, así como la tasa de mutación, también son grandes desafíos para las operaciones de edición del genoma. Las langostas adultas gregarias criadas en la condición de 16 (claro): 8 (oscuro) fotoperíodo se utilizan para recolectar suficientes huevos en un corto período durante las 9:00-11:00 am. Es importante aplicar suavemente presión hacia abajo entre los huevos con un pincel o pinzas para aislar cuidadosamente los huevos de la vaina. Con suficiente práctica, múltiples usuarios pueden separar de manera confiable los huevos individuales de la vaina. Después de que cada huevo se aísla y se lava con agua estéril, los huevos se alinean en las almohadillas de inyección diseñadas (Figura 2B, C) para estabilizar los huevos durante la microinyección. El huevo se inyecta con el RNP optimizado cerca de los micropilos. Para limitar el daño mecánico y minimizar la contaminación de los huevos, se permite que los huevos frescos permanezcan en las vainas durante unos 30 minutos para asegurarse de que el curtido de la cáscara del huevo sea ideal para la microinyección21, debido a su estructura condensada y alta capacidad de defensa en la invasión después de un bronceado suficiente29,30. Mientras tanto, un informe publicado indicó que la división sincitial31 de los huevos de langosta ocurre en algún momento entre 0 h y 4 h después de la puesta. Con el protocolo de microinyección estándar actual, la inyección de aproximadamente 100 huevos se puede lograr en 40 minutos. Tomados en conjunto, el procedimiento de curtido y microinyección de 100 óvulos fertilizados se puede completar en 2 h. Por lo tanto, quedan al menos 2 h para la escisión mediada por CRISRP / Cas9 y la reparación de genes diana para lograr una mutación eficiente en los óvulos inyectados y futuras langostas adultas.
Por último, las estrategias efectivas para el paso y la identificación de mutantes son importantes para generar homocigotos en la mayoría de los animales mutantes. En langostas, para genes que no son letales después de la edición, la estrategia cruzada sugerida aquí es que los mutantes G0 se cruzan con langostas de tipo ancho y se pueden realizar más cruces utilizando los heterocigotos G1 con las mismas mutaciones para generar mutantes homocigotos en la siguiente generación. Para verificar la mutación en cada langosta durante el proceso de herencia mutante, se recomienda utilizar el método de lisis alcalina para digerir un segmento corto de antenas como plantilla para el genotipado basado en PCR. Finalmente, utilice los adultos homocigotos obtenidos para ampliar aún más la población. Limitado por el tamaño de la población y las diferencias en la etapa de desarrollo entre las langostas homocigóticas, el exceso de huevos se recomienda para la criopreservación a 6 °C y la reanimación por aumento de la temperatura del gradiente (Figura 5), que se basa en el principio de diapausa a baja temperatura y liberación de diapausa a alta temperatura de huevos de langosta migratoria32,33.
En resumen, el sistema CRISPR/Cas9 ha demostrado ser una herramienta fiable para facilitar el estudio de la genómica funcional de las langostas migratorias. Este protocolo detallado se puede utilizar como referencia para aplicaciones de edición de genes basadas en CRISPR / Cas9 en langostas migratorias, así como en otros insectos.
Divulgaciones
Los autores declaran que no tienen conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por la Fundación Nacional de Ciencias Naturales de China (32070502, 31601697, 32072419 y la Fundación de Ciencias Postdoctorales de China (2020M672205).
Materiales
| Name | Company | Catalog Number | Comments |
| 10×NEBuffer r3.1 | New England Biolabs | B7030S | The buffer of in vitro Cas9 cleavage assays |
| 2xEs Taq MasterMix (Dye) | Cwbio | CW0690 | For gene amplification |
| 2xPfu MasterMix (Dye) | Cwbio | CW0686 | For gene amplification |
| CHOPCHOP | Online website for designing sgRNAs, http://chopchop.cbu.uib.no/. | ||
| CRISPOR | Online website for designing sgRNAs, http://crispor.org. | ||
| CRISPRdirect | Online website for designing sgRNAs, http://crispr.dbcls.jp/. | ||
| Electrophoresis power supply | LIUYI BIOLOGY | DYY-6D | Separation of nucleic acid molecules of different sizes |
| Eppendorf Tube | Eppendorf | 30125177 | For sample collection, etc. |
| Fine brushes | Annigoni | 1235 | For cleaning and isolating eggs. Purchased online. |
| Flaming/brown micropipette puller | Sutter Instrument | P97 | For making the microinjection needles |
| Gel Extraction Kit | Cwbio | CW2302 | DNA recovery and purification |
| Gel Imaging Analysis System | OLYMPUS | Gel Doc XR | Observe the electrophoresis results |
| GeneTouch Plus | Bioer | B-48DA | For gene amplification |
| Glass electrode capillary | Gairdner | GD-102 | For making injection needles with a micropipette Puller |
| Incubator | MEMMERT | INplus55 | For migratory locust embryo culture |
| Metal bath | TIANGEN | AJ-800 | For heating the sample |
| Micro autoinjector | Eppendorf | 5253000068 | Microinjection of embryos early in development |
| Micro centrifuge | Allsheng | MTV-1 | Used for mixing reagents |
| Microgrinder | NARISHIGE | EG-401 | To ground the tip of injection needle |
| Microloader | Eppendorf | 5242956003 | For loading solutions into the injection needles. |
| Micromanipulation system | Eppendorf | TransferMan 4r | An altinative manipulation system for microinjection |
| Microscope | cnoptec | SZ780 | For microinjection |
| Motor-drive Manipulator | NARISHIGE | MM-94 | For controling the position of the micropipette during the microinjection precedure |
| Multi-Sample Tissue Grinder | jingxin | Tissuelyser-64 | Grind and homogenize the eggs |
| ovipisition pot | ChangShengYuanYi | CS-11 | Filled with wet sterile sand for locust ovipositing in it. The oocysts are collected from this container. Purchased online. |
| Parafilm | ParafilmM | PM996 | For wrapping the petri dishes. |
| pEASY-T3 Cloning Kit | TransGen Biotech | CT301-01 | For TA cloning |
| Petri dish | NEST | 752001 | For culture and preservation of the eggs. |
| Pipettor | Eppendorf | Research plus | For sample loading |
| plastic culture cup | For rearing locusts seperately and any plastic cup big enough (not less than 1000 mL in volume) will do. Purchased online. | ||
| Precision gRNA Synthesis Kit | Thermo | A29377 | For sgRNA synthesis |
| Primer Premier | PREMIER Biosoft | Primer Premier 5.00 | For primer design |
| SnapGene | Insightful Science | SnapGene®4.2.4 | For analyzing sequences |
| Steel balls | HuaXinGangQiu | HXGQ60 | For sample grinding.Purchased online. |
| Tips | bioleaf | D781349 | For sample loading |
| Trans DNA Marker II | TransGen Biotech | BM411-01 | Used to determine gene size |
| TrueCut Cas9 Protein v2 | Thermo | A36496 | Cas9 protein |
| UniversalGen DNA Kit | Cwbio | CWY004 | For genomic DNA extraction |
| VANNAS Scissors | Electron Microscopy Sciences | 72932-01 | For cutting off the antennae |
| Wheat | To generate wheat seedlings as the food for locusts. Bought from local farmers. | ||
| ZiFiT | Online website for designing sgRNAs, http://zifit.partners.org/ZiFiT/ChoiceMenu.aspx. |
Referencias
- Doudna, J. A. The promise and challenge of therapeutic genome editing. Nature. 578 (7794), 229-236 (2020).
- van Haasteren, J., Li, J., Scheideler, O. J., Murthy, N., Schaffer, D. V. The delivery challenge: fulfilling the promise of therapeutic genome editing. Nature Biotechnology. 38 (7), 845-855 (2020).
- McCarty, N. S., Graham, A. E., Studena, L., Ledesma-Amaro, R. Multiplexed CRISPR technologies for gene editing and transcriptional regulation. Nature Communications. 11 (1), 1281 (2020).
- Manghwar, H., et al. CRISPR/Cas systems in genome editing: Methodologies and tools for sgRNA design, off-target evaluation, and strategies to mitigate off-target effects. Advanced Science. 7 (6), 1902312 (2020).
- Anzalone, A. V., Koblan, L. W., Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nature Biotechnology. 38 (7), 824-844 (2020).
- Rees, H. A., Liu, D. R. Base editing: precision chemistry on the genome and transcriptome of living cells. Nature Reviews Genetics. 19 (12), 770-788 (2018).
- Ishino, Y., Shinagawa, H., Makino, K., Amemura, M., Nakata, A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology. 169 (12), 5429-5433 (1987).
- Barrangou, R., et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 315 (5819), 1709-1712 (2007).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. eLife. 2, 00471 (2013).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Scully, R., Panday, A., Elango, R., Willis, N. A. DNA double-strand break repair-pathway choice in somatic mammalian cells. Nature Reviews Molecular Cell Biology. 20 (11), 698-714 (2019).
- Ai, D., et al. Embryo microinjection and knockout mutant identification of CRISPR/Cas9 genome-edited Helicoverpa Armigera (Hubner). Journal of Visualized Experiments: JoVE1. (173), e62068 (2021).
- Huang, Y., et al. CRISPR/Cas9 mediated knockout of the abdominal-A homeotic gene in the global pest, diamondback moth (Plutella xylostella). Insect Biochemistry and Molecular Biology. 75, 98-106 (2016).
- Gratz, S. J., et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics. 196 (4), 961-971 (2014).
- Kistler, K. E., Vosshall, L. B., Matthews, B. J. Genome engineering with CRISPR-Cas9 in the mosquito Aedes aegypti. Cell Reports. 11 (1), 51-60 (2015).
- Li, Y., et al. CRISPR/Cas9 in locusts: Successful establishment of an olfactory deficiency line by targeting the mutagenesis of an odorant receptor co-receptor (Orco). Insect Biochemistry and Molecular Biology. 79, 27-35 (2016).
- Xu, X., et al. BmHpo mutation induces smaller body size and late stage larval lethality in the silkworm, Bombyx mori. Insect Science. 25 (6), 1006-1016 (2018).
- Chen, D., et al. CRISPR/Cas9-mediated genome editing induces exon skipping by complete or stochastic altering splicing in the migratory locust. BMC Biotechnology. 18 (1), 60 (2018).
- Zhang, T., et al. Egg tanning improves the efficiency of CRISPR/Cas9-mediated mutant locust production by enhancing defense ability after microinjection. Journal of Integrative Agriculture. 20 (10), 2716-2726 (2021).
- Guo, X., et al. 4-Vinylanisole is an aggregation pheromone in locusts. Nature. 584 (7822), 584-588 (2020).
- Zhang, L., Lecoq, M., Latchininsky, A., Hunter, D. Locust and grasshopper management. Annual Review of Entomology. 64, 15-34 (2019).
- Pétavy, G. Origin and development of the vitellophags during embryogenesis of the migratory locust, Locusta migratoria L. (Orthoptera : Acrididae). International Journal of Insect Morphology and Embryology. 14 (6), 361-379 (1985).
- Barry, S. K., et al. Injecting Gryllus bimaculatus eggs. Journal of Visualized Experiments: JoVE. (150), e59726 (2019).
- Watanabe, T., Noji, S., Mito, T. Genome editing in the cricket, Gryllus bimaculatus. Methods in Molecular Biology. 1630, 219-233 (2017).
- Du, M. H., et al. Suppression of Laccase 2 severely impairs cuticle tanning and pathogen resistance during the pupal metamorphosis of Anopheles sinensis (Diptera: Culicidae). Parasites & Vectors. 10 (1), 171 (2017).
- Eisner, T., Shepherd, J., Happ, G. M. Tanning of grasshopper eggs by an exocrine secretion. Science. 152 (3718), 95-97 (1966).
- Ho, K., Dunin-Borkowski, O. M., Akam, M. Cellularization in locust embryos occurs before blastoderm formation. Development. 124 (14), 2761-2768 (1997).
- Wang, X., et al. Interactive effect of photoperiod and temperature on the induction and termination of embryonic diapause in the migratory locust. Pest Managment Science. 77 (6), 2854-2862 (2021).
- Jarwar, A. R., et al. Comparative transcriptomic analysis reveals molecular profiles of central nervous system in maternal diapause induction of Locusta migratoria. G3-Genes Genomes Genetics. 9 (10), 3287-3296 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados