Method Article
Schnelle hochauflösende Bildgebung des Gefäßsystems von Kleintieren für quantitative Studien
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt eine schnelle Methode für die quantitative dreidimensionale Gefäßbildgebung mit Hilfe der Lichtblatt-Fluoreszenzmikroskopie vor. Die Wirksamkeit der Methode wird anhand des pharyngealen Bogenarteriensystems des Hühnerembryomodells demonstriert, wobei die hämodynamischen Kräfte mittels Computational Fluid Dynamics quantifiziert werden.
Zusammenfassung
In Kleintiermodellen der kardiovaskulären Entwicklung und Erkrankungen ermöglichen subjektspezifische Computersimulationen des Blutflusses quantitative Bewertungen hämodynamischer Metriken, die experimentell schwer zu messen sind. Numerische Strömungssimulationen geben Aufschluss über die entscheidende Rolle der Mechanik bei der kardiovaskulären Funktion und dem Fortschreiten der Krankheit. Die Aufnahme hochwertiger volumetrischer Bilder der interessierenden Gefäße ist von zentraler Bedeutung für die Genauigkeit und Reproduzierbarkeit morphologischer Mess- und Durchflussquantifizierungsergebnisse. Diese Studie stellt eine schnelle, kostengünstige und zugängliche Methode für die hochauflösende Bildgebung von Kleintiergefäßen mittels Lichtblatt-Fluoreszenzmikroskopie vor. Das modifizierte iDISCO+-Protokoll (immunolabeling-enabled three-dimensional imaging of solvent-cleared organs) umfasst (1) die Markierung des Gefäßsystems mit einem Fluoreszenzmittel, (2) die Konservierung der Probe und (3) die Transparenz der Probe. Im Gegensatz zum klassischen iDISCO+, das eine immunhistochemische Färbung verwendet, markieren die Autoren das vaskuläre Endothel mit FITC-markiertem Poly-L-Lysin, einem erschwinglichen unspezifischen Fluoreszenzfarbstoff, der sehr widerstandsfähig gegen Photobleiche ist, in einem Prozess, der als "Endo-Painting" bezeichnet wird. Die schnelle Etikettierung reduziert die Probenvorbereitungszeit von ca. vier Wochen auf weniger als 3 Tage. Darüber hinaus macht die Verwendung von minimal gefährlichem Lösungsmittelethylcinnamat (ECi) als Clearingmittel und Bildgebungslösung die Proben sicherer in der Handhabung und kompatibel mit einer breiteren Palette von Bildgebungseinrichtungen. Das vorgeschlagene Protokoll wird angewendet, um hochaufgelöste Lichtblatt-Fluoreszenzmikroskopie-Bildstapel des Herz-Kreislauf-Systems in Hühnerembryonen von Tag 3 (HH18) bis Tag 8 (HH34) zu erhalten. Diese Studie zeigt außerdem die Eignung dieser Methode für die vaskuläre Quantifizierung durch 3D-Rekonstruktion und computergestützte hämodynamische Modellierung eines Hühnerembryos an Tag 5 (HH 26).
Einleitung
Die volumetrische Bildgebung ist für genaue Untersuchungen der kardiovaskulären Physiologie und Erkrankungen erforderlich. Die quantitative Bildgebung erzeugt hochauflösende Bildstapel mit intakten volumetrischen Abmessungen. Die Proben müssen sowohl konserviert werden, um ihre In-vivo-Morphologie und ihr Lumenvolumen zu erhalten, als auch in einer einheitlich hohen Auflösungskapazität abgebildet werden. Aus hochauflösenden Bildgebungsstapeln kann der Benutzer dreidimensionale Gefäß-Renderings mit hoher Wiedergabetreue erstellen, die eine vollständige Darstellung von Gefäßformen, -struktur und -konnektivität ermöglichen1.
Kardiovaskuläre Strukturen besitzen komplexe dreidimensionale anatomische Merkmale, die nicht genau erfasst werden können, wenn man sie durch eine zweidimensionale, unzusammenhängende Linse untersucht. Stereoskop, die morphologische Bildgebung und histologische Schnitte sind unzureichend, um komplexe dreidimensionale Variationen zu erfassen 1,2,3. Mikro- und Nano-Computertomographie-Bilder sind der Goldstandard für die quantitative volumetrische Bildgebung von Kleintieren 1,4, sind aber in der biologischen Gemeinschaft nicht allgemein zugänglich oder werden nicht übernommen. Jüngste Innovationen in der Gewebereinigung und der Ganzorgan-/Kleintiermikroskopie haben quantitative Anwendungen von Whole-Mount-Clearing- und Gefäßmarkierungstechniken ermöglicht 5,6,7. Tissue-Clearing dient der Homogenisierung der Lichtstreuung in Gewebeproben und reduziert so Verzögerungen bei der Lichtausbreitung durch das Medium, indem die Wahrscheinlichkeit von Lichtstreuung oder -absorption verringert wird. Eine hohe Transparenz erfordert eine stringente Gewebeverarbeitung, die sich auf die Antigenität oder Helligkeit der Fluoreszenzsignalmarkierung auswirken kann8. Die Lichtblattmikroskopie hat sich zu einem schnellen, leistungsstarken Bildgebungswerkzeug entwickelt, das von Biologen weit verbreitet ist9 und einen Geschwindigkeitsgewinn um mehrere Größenordnungen gegenüber Rastermikroskopen bietet und die Möglichkeit bietet, Proben mit einer Größe von mehr als 1 cm abzubilden. Bei der Lichtblatt-Fluoreszenzmikroskopie (LSFM) beleuchtet ein Laser einen Probenquerschnitt mit erhöhter Geschwindigkeit und Tiefe im Vergleich zur konfokalen Mikroskopie; Aus diesem Grund erfordert die Methode eine hohe Probentransparenz.
Hier adaptieren die Autoren aktuelle iDISCO+-Clearing-Methoden und kombinieren sie mit Endo-Painting10 im Tiermodell des Hühnerembryos, um die Wirksamkeit der Methode von der frühen bis zur späten kardiovaskulären Entwicklung zu demonstrieren. iDISCO (immunolabeling-enabled three-dimensional imaging of solvent-cleared organs) ist ein organisches lösungsmittelbasiertes Clearingverfahren, das im Gegensatz zu wässrigen Clearing-basierten Methoden keinen bildgebenden Artefakten durch Lösungsmittelverdampfung unterliegt. iDISCO unterscheidet sich von iDISCO+ dadurch, dass der Tetrahydrofuran-Dehydratisierungsschritt des ersteren (iDISCO) durch eine mildere Methanol-Dehydratisierung gefolgt von einem Lipidextraktionsschritt (iDISCO+) ersetzt wird. Zu den Vorteilen der iDISCO+ Clearing-Methode gehören die Immunmarkierung großer adulter Proben und Embryonen, die geringe Gewebeschrumpfung und die hohe Transparenz 8,11. Wichtig ist, dass iDISCO+ die Erzeugung hochauflösender Bildstapel ermöglicht und die traditionellen Immunmarkierungstechniken der Biologie erweitert, um Informationen über große Organproben oder einen ganzen Embryo zu gewinnen, anstatt auf die Probenahme kleiner Regionen beschränkt zu sein, denen Informationen über die gesamte Organisation auf Gewebeebene fehlen, wie bei der traditionellen Histologie9. Zu den Nachteilen von iDISCO+ gehört, dass genetisch kodierte fluoreszierende Proteine nicht konserviert werden11. Die Gewebemarkierungsmethode des Endo-Painting wurde zunächst als Hochdurchsatz-Screening auf kardiovaskuläre Defekte unter Verwendung von HH31-HH36-Hühnerembryonen eingeführt, die mit 0,5 mg/ml FITC-poly-L-lysin in der linksventrikulären Spitze perfundiert wurden. Der Farbstoff wurde 4 Minuten lang binden gelassen, bevor er fixiert undgelagert wurde 10.
Die vorliegende Studie ergab, dass die gleiche FITC-Poly-L-Lysin-Konzentration für ein breiteres Spektrum von Embryonen (HH18 - HH34) verwendet werden konnte, fand aber die ideale Fixierungszeit zu variieren (von 5-10 Minuten), um hell markierte Gefäße zu gewährleisten. Benutzer der vorliegenden endo-DISCO-Technik möchten möglicherweise die Farbstoffkonzentration anpassen (jeweils um 0,1 mg/ml verringern), falls sich die Lösung als zu viskos erweist, um alle gewünschten Gefäße zu markieren, werden jedoch ermutigt, zuerst die Fixierungszeit anzupassen und die Muskelkontraktion des linken Ventrikels zu optimieren, bevor sie die Farbstoffkonzentration anpassen. Die Autoren versuchten es mit Endo-Painting mit einer Konzentration von 0,1 mg/ml und stellten fest, dass sich der Farbstoff zwar leichter durch kleine Gefäße ausbreitete, aber bei der PFA-Perfusion leichter weggespült wurde. Die Autoren zeigen, dass die hochauflösenden Bildgebungsstacks, die mit der vorliegenden Technik erzeugt werden, von ausreichender Qualität für die computergestützte hämodynamische Modellierung sind. Blutströmungswege und entsprechende hämodynamische Kräfte, einschließlich Druck- und Wandscherspannungsverteilungen, treten in komplexen lokalisierten Mustern auf, die nur durch computergestützte Strömungssimulationen aufgelöst werden können 1,12. Diese biomechanischen Kräfte beeinflussen das Verhalten benachbarter kardiovaskulärer Gewebe und lösen die Anpassung, das Wachstum und den Umbau der Gefäßeaus 13. Das Verständnis der lokalen hämodynamischen Kraftwerte wirft ein kritisches Licht auf die mechanistischen Regulatoren der kardiovaskulären Funktion und der Krankheitsentstehung oder -progression2.
Protokoll
Das Office of Laboratory Animal Welfare interpretiert die Politik des öffentlichen Gesundheitsdienstes so, dass sie erst nach dem Schlüpfen auf das Kükenmodell als "Wirbeltier" anwendbar ist. Diese Embryonen sind in ähnlicher Weise von der Zuständigkeit des Institutional Animal Care and Use Committee (IACUC) ausgenommen. Die relevanten häufig gestellten Fragen der National Institutes of Health finden Sie unter: http://grants.nih.gov/grants/olaw/faqs.htm#ApplicabilityofthePHSPolicy.
1. Entnahme, Kennzeichnung und Fixierung der Embryonen
- Fashion zog Glaskapillarstäbe mit einem Innendurchmesservon 0,75 mit einer microforge 1,12 in geschnittene Mikronadeln.
HINWEIS: Die Größe der erforderlichen Spitze hängt vom gewünschten Alter/der gewünschten Durchflussrate des Tieres ab. Die Mikronadel kann alternativ mit einer Mikrodissektionszange oder einer Schere mit stark reduzierter Präzision geschnitten werden. - Die Tyrode-Lösung auf ca. 38 °C erwärmen.
- FITC Poly-L-Lysin-Feststoff in Tyrode-Lösung auflösen, um eine Stammlösung von 0,5 mg/ml herzustellen.
HINWEIS: Diese Konzentration ist für die Markierung der Arterien von Hühnerembryonen optimiert. Die optimale Konzentration für andere Gewebetypen kann variieren. Die überschüssige Stammlösung sollte bei -20 °C gelagert werden. - Der Vogelembryo (im Bereich von HH18 (Tag 3) bis HH34 (Tag 8) für die vorliegende Hühnerherzstudie) aus dem Eigelb sezieren, indem der Embryo mit einer Schere mit gebogener Spitze umschnitten und mit einer Transferpipette oder einem Spatel in eine 35-mm-Petrischale übertragen wurde, die mit warmer Tyrodelösung gefüllt war.
- Entfernen Sie vorsichtig die Chorion- und Allantoismembranen, die den Embryo umhüllen, und die Perikardmembran um das Herz herum mit einer Pinzette mit feiner Spitze, indem Sie winzige (~0,1 mm) Schnitte in die Membranen machen und sie vom Embryo wegziehen.
- Übertragen Sie den Embryo in eine neue, saubere Petrischale, die mit warmer Tyrode-Lösung gefüllt ist, um das Herz am Schlagen zu halten.
- Füllen Sie eine 5-ml-Kunststoffspritze mit warmer Tyrode-Lösung. Schneiden Sie das breite Ende einer 0-20 μl Kunststoff-Pipettenspitze ab und befestigen Sie sie an der flachen Spitze der Spritze. Die Spritze wurde nun auf den Durchmesser der feinen Pipettenspitze verlängert.
- Konstruieren Sie eine "Injektionsleitung", indem Sie ein Segment eines Silikonschlauchs mit einem Innendurchmesser von 0,03 Zoll an den neu befestigten Pipettenspitzenspritzenapparat anschließen (Schritt 1,7). Befestigen Sie eine Glaskapillar-Mikronadel am gegenüberliegenden Ende des Schlauchs.
- Montieren Sie die Mikronadel auf dem Mikroinjektionshalter, der an einem Mikromanipulator befestigt ist. Reinigen Sie den Silikonschlauch und die Mikronadel von Luftblasen.
- Führen Sie die Nadel mit dem Mikromanipulator in die Herzspitze ein und perfundieren Sie den Embryo 1,12, indem Sie die Tyrode-Lösung langsam in das Herz injizieren. Fahren Sie mit der Perfusion des Embryos fort, bis er weitgehend von Blut befreit ist.
- Ziehen Sie mit dem Mikromanipulator die Nadel und die Injektionsleitung von ihrer Einstichstelle zurück und richten Sie die Nadel so aus, dass sie bei der Vorbereitung der nächsten Injektionsleitung nicht gestört wird. Entfernen Sie den Silikonschlauch von der Nadel und der Spritze.
HINWEIS: Einige Benutzer finden es möglicherweise optimal, die Nadel am Herzen zu halten, in diesem Fall sollte der Benutzer den Silikonschlauch zwischen Nadel und Spritze entfernen, während die Nadel an Ort und Stelle bleibt. - Führen Sie ein Endo-Painting durch, um das vaskuläre Endothel mit grüner Fluoreszenz zu markieren.
- Laden Sie mit einer Mikropipette 20-40 μl FITC-Poly-L-Lysin-Stammlösung in den Silikonschlauch und achten Sie darauf, dass keine Luftblasen vor der FITC-Flüssigkeitsbarriere entstehen.
- Bringen Sie die mit Tyrode-Lösung gefüllte Spritze wieder an und injizieren Sie mit dem Mikromanipulator langsam in die Herzspitze, wobei Sie die Nadel bei Bedarf wieder einführen. Entfernen Sie die Nadel, nachdem das FITC-Poly-L-Lysin diffundiert wurde und bevor Luftblasen in das Herz gelangen.
- Lassen Sie FITC Poly-L-Lysin 5-10 Minuten im Embryo einwirken.
HINWEIS: (optionale Qualitätsprüfung) Um die Fluoreszenz zu überprüfen, nehmen Sie ein Bild mit einem fluoreszierenden Stereo- oder Makroskop auf. Das Bild kann verwendet werden, um einen Dehydrationsfaktor zu bestimmen, nachdem die Embryonenreinigung abgeschlossen ist.
- Füllen Sie eine 5-ml-Spritze mit 4 % Paraformaldehyd (PFA) und befestigen Sie sie für einen dritten Injektionsschritt an den Silikon-Injektionsschlauch.
- Perfundieren Sie den Embryo 1,12 mit 4% PFA, bis alle kardiovaskulären Strukturen ihre volle Volumenkapazität erreicht haben und das Gewebe undurchsichtiger wird.
- Laden Sie den Embryo vorsichtig in ein 2-5-ml-Fläschchen, das mit ausreichend 4% PFA gefüllt ist, um die Probe abzudecken. Achten Sie darauf, den Embryo nicht übermäßig zu deformieren.
HINWEIS: Wählen Sie ein Fläschchen, das groß genug für die zu untersuchende Probe ist, um eine Quetschung oder Verformung der Probe zu vermeiden. - Inkubieren Sie den Embryo über Nacht bei 4 °C mit einem Shaker.
2. Dehydrierung und Reinigung des Embryos
- Pipettieren Sie die 4% PFA vorsichtig heraus.
HINWEIS: Vermeiden Sie ab diesem Zeitpunkt den direkten Kontakt mit dem Embryo, um Gewebeschäden oder -verformungen zu vermeiden. - Um den Embryo zu waschen, inkubieren Sie den Embryo in frischer Phosphatpufferlösung (PBS) bei Raumtemperatur für 30 Minuten unter leichtem Schütteln. Wiederholen Sie dies zwei weitere Male für insgesamt 3 Wäschen.
- Beginnen Sie mit der Dehydrierung des Embryos in einem Abzug. Pipettieren Sie das PBS vorsichtig aus und inkubieren Sie den Embryo in einer abgestuften Serie von 5 Methanol-Inkubationen bei Raumtemperatur für jeweils 1 Stunde: 20 %, 40 %, 60 %, 80 % und 100 % Methanolkonzentration.
- Lassen Sie den Embryo über Nacht in frischem 100%igem Methanol bei Raumtemperatur.
HINWEIS: (Optionaler Haltepunkt). Embryonen können bei -20 °C in 100 % Methanol für eine spätere Verwendung von bis zu 6 Monaten gelagert werden. - Beginnen Sie im Abzug mit der Lipidentfernung. Inkubieren Sie den Embryo in einer 2:1 (Volumen zu Volumen) DCM: Methanollösung bei Raumtemperatur für 3 Stunden unter leichtem Schütteln.
ACHTUNG: DCM ist giftig und muss im Rauchabzug gehandhabt werden. Treffen Sie zusätzliche Vorsichtsmaßnahmen, wenn Sie mit DCM arbeiten. Doppelte Handschuhe können helfen, eine zusätzliche Barriere zu schaffen. - Nehmen Sie Ethylzimt (ECi) aus 4 °C und lassen Sie es bei Raumtemperatur auftauen.
- Waschen Sie die Embryonen 15 Minuten lang in frischem 100% DCM bei Raumtemperatur und schütteln Sie sie. Wiederholen Sie die 100 % DCM-Wäsche, um insgesamt 2 Wäschen zu erreichen.
- Pipettieren Sie DCM aus und inkubieren Sie den Embryo in 100% ECi. Lassen Sie den Embryo in ECi, bis er in ca. 1 h klar ist. Schütteln Sie die Tube bei Bedarf vorsichtig.
- (Optionaler Verifizierungsschritt) Untersuchen Sie die Qualität der Reinigung und Gefäßfärbung mit einem fluoreszierenden Stereo oder Makroskop. Machen Sie ein Foto des Embryos, um den stadien-/altersspezifischen Dehydrationsskalierungsfaktor zu bestimmen.
- Lagern Sie den gereinigten Embryo bis zu 6 Monate lang bei 4 °C und halten Sie ihn vor Licht geschützt.
3. Erfassung von Daten
- Um Embryonen abzubilden, verwenden Sie ein Lichtblatt-Fluoreszenzmikroskop. Befestigen Sie den Kopf eines optisch gereinigten Embryos mit Sekundenkleber auf einer Glaskapillare.
- Montieren Sie die Glaskapillare auf dem Probenhalter, füllen Sie die Bildgebungskammer mit ECi und senken Sie den Embryo in die Bildgebungskammer ab.
- Passen Sie die Parameter an und führen Sie ein Imaging durch.
4. Quantitative Anwendung: 3D-Rekonstruktion und Computational Fluid Dynamic Modeling
HINWEIS: In diesen Schritten werden die aus Lichtblättern generierten hochauflösenden Bildstapel in die Open-Source-Software SimVascular14 für die anatomische 3D-Rekonstruktion und die numerische Strömungsmodellierung geladen. Detaillierte Tutorials gibt es auf der SimVascular Website (siehe Materialtabelle). Die Rekonstruktion besteht aus der Erstellung von Pfadlinien in den interessierenden Gefäßen, der Erstellung von 2D-Segmentierungen entlang der Pfadlinien und der Kombination von erhabenen Segmentierungen zu einem 3D-Volumenmodell. Die computergestützte Modellierung besteht aus der Vorbereitung einer vernetzten Geometrie, der Definition von Randbedingungen und der Durchführung von Simulationen.
- Erstellen Sie ein neues SVProject unter Datei und laden Sie einen hochauflösenden Lightsheet-Imaging-Stack hoch, indem Sie mit der rechten Maustaste auf Bilder und dann auf Bild hinzufügen/ersetzen klicken.
- Befolgen Sie die Schritte des Software-Tutorials, um die Gefäßanatomie in silico zu rekonstruieren.
- Erstellen Sie eine Pfadlinie, indem Sie mit der rechten Maustaste auf Pfade klicken und Pfad erstellen auswählen. Platzieren Sie Wegmarkierungen entlang eines interessanten Gefäßes. Wiederholen Sie den Vorgang für jedes Gefäß, das Sie interessiert.
- Verfolgen Sie für jede erstellte Pfadlinie 2D-Gefäßquerschnitte, indem Sie mit der rechten Maustaste auf Segmentierungen klicken und die Konturgruppe erstellen auswählen. Wählen Sie den Schiffspfad aus und doppelklicken Sie unter Segmentierung auf den Schiffsnamen, um die manuelle Segmentierung zu starten.
- Nachdem alle Schiffe segmentiert wurden, erstellen Sie ein Modell, indem Sie mit der rechten Maustaste auf Modelle klicken und dann Modell erstellen auswählen. Wählen Sie den PolyData-Typ aus, geben Sie einen Modellnamen ein, klicken Sie auf Volumenmodell erstellen, und wählen Sie alle Segmentierungen aus, die Teil des Modells sein sollen.
- Vernetzen Sie die Geometrie, indem Sie den in SimVascular SVMesher integrierten Tetgen-Mesher ausführen. Definieren Sie eine maximale Kantengröße, und führen Sie sowohl die Flächen- als auch die Volumenvernetzung durch.
HINWEIS: Wählen Sie eine Maschenweite, die feine geometrische Details und lokalisierte hämodynamische Variabilitäten im Gefäß auflösen kann. Beginnen Sie mit der Schaltfläche Globale Netzgröße schätzen . Bei der Fertigstellung von Simulationen kann eine Netzkonvergenzstudie erforderlich sein. - Richten Sie eine rechnerische Blutflusssimulation mit dem SimVascular Solver ein. Erstellen Sie Simulationsdateien, indem Sie mit der rechten Maustaste auf Simulationen klicken und Simulationsauftrag erstellen auswählen. Passen Sie die Grundparameter an und wählen Sie Einlass- und Auslass-BCs für bestimmte Randbedingungen aus.
- Passen Sie die Solver-Parameter an, navigieren Sie zur Registerkarte "Dateien erstellen" und "Simulation ausführen", wählen Sie die Netzdatei aus und klicken Sie auf "Datendateien für Simulation erstellen".
- Starten Sie die Simulation mit einer High-Performance-Computing-Workstation oder einem Supercomputer.
Ergebnisse
Das hier vorgestellte hochauflösende Bildgebungsprotokoll für den schnellen Whole-Mount (Abbildung 1, Tabelle 1) erzeugt klar umrissene Gefäßlumen, wie in Abbildung 2, Abbildung 3 und Abbildung 4 gezeigt, wobei das Gefäßendothel des Hühnerembryos GFP-fluoreszierend ist und daher über die Embryostadien von der frühen bis zur reifen Herzentwicklung grün umrandet ist (Abbildung 4). Es ist wichtig, die richtige Kombination aus Poly-L-Lysin-Konzentration, Farbstofffixierungszeit und Verwendung von ventrikulären Kontraktionen (falls zutreffend) zu finden, wenn das Gefäßsystem der Probe nicht eindeutig gekennzeichnet erscheint (siehe Abbildung 2 für das Aussehen von Embryonen unter einem fluoreszierenden Stereoskop oder Makroskop vor der LSFM-Bildgebung). Die Aufrechterhaltung eines schlagenden Herzens durch warme Tyrode-Lösung und die schnelle Dissektion des Embryos weg vom Dottersack hilft bei der Diffusion der Lösung in der Probe/im Kleintiermodell. Die Viskosität des Poly-L-Lysins kann durch die Konzentration der Stammlösung gesteuert werden. Eine weniger viskose Lösung kann die Fluoreszenzpenetranz in der gesamten Probe verbessern, obwohl die Robustheit der Markierung nach der PFA-Perfusion überprüft werden sollte. Die Anwenderin kann eine niedrigere Konzentration der Poly-L-Lysin-Stammlösung ausgleichen, indem sie das injizierte Volumen vergrößert und eine längere Inkubationszeit zulässt, bevor der Embryo mit PFA fixiert wird.
Abbildung 5 zeigt die Eignung der vorgestellten Methode für die anatomische 3D-Rekonstruktion und computergestützte Modellierung. Die Werte für die Wandschubspannung stimmen mit den früheren Studien der Autoren überein, die auf Nano-Computertomographie-Rekonstruktionenbasieren 1.

Abbildung 1: Aufbau der Embryomarkierung und -fixierung (oben) und Aufbau der Lichtblattmikroskopie-Datenerfassung (unten). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Embryonen vor und nach der Klärung. Repräsentative Hellfeld- und GFP-Kanal-beleuchtete Embryonen für Proben, die die Schritte 1 (vor der Clearing) und 2 (nach der Clearing) des Protokolls durchlaufen haben, wie sie mit einem Fluoreszenzmakroskop gesehen wurden. Beachten Sie, wie der gesamte Embryo im GFP-Kanal leuchtet, bevor er sich lichtet, insbesondere im Herzen, für das die äußeren Membranen entfernt wurden. Alle gewünschten Gefäße sind im GFP-freigegebenen Embryo eindeutig gekennzeichnet. Maßstabsleisten = 1 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
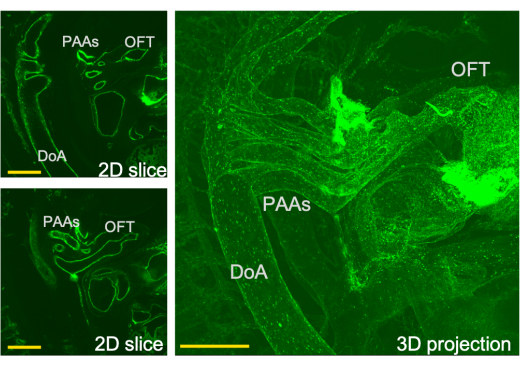
Abbildung 3: Dreidimensionale Ansicht des kardialen Abflusssystems und entsprechende Schnitte durch diese Ansicht, wie sie von LSFM erhalten wurde. PAA - Arteria pharyngeus arch, OFT-Ausflusstrakt, DoA-dorsale Aorta. Maßstabsleisten = 0,5 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
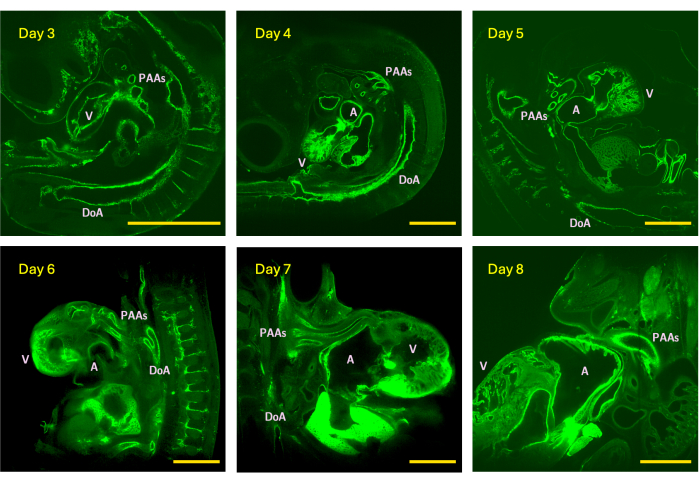
Abbildung 4: Beispiel-Z-Slices von LSFM-Bildstapeln, die von Tag 3 (Stadium HH18), 4 (HH24), 5 (HH26), 6 (HH29), 7 (HH31) und 8 (HH34) Hühnerembryonen aufgenommen wurden. Die Bildgröße stellt das maximale (ohne Kacheln) Sichtfeld des Zielfernrohrs dar, das mit einem 5-fach-Detektionsobjektiv ausgestattet ist. V - Ventrikel; A - Atrium; DoA - dorsale Aorta; PAAs - pharyngeale Bogenarterien. Maßstabsleisten = 1 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Repräsentative in silico anatomische Rekonstruktionen des Aortenbogens von Tag 5 (Stadium HH26) und wichtige hämodynamische Simulationsergebnisse. Links: Ein rekonstruiertes Modell der Gefäßanatomie, das den Ausflusstrakt (OFT), die Pharyngealbogenarterien (PAAs) und die dorsale Aorta (DoA) zeigt. Mitte: Der Blutfluss rationalisiert sich bei maximaler Systole, die mit Hilfe der Blutflusssimulation erhalten wurde. Rote Stromlinien bedienen hauptsächlich kraniale Regionen, während blaue kaudale Regionen dienen. Rechts: Maximale systolische Wandscherspannungsverteilung (WSS) über die Gefäßwand, die mit Hilfe der Blutflusssimulation erhalten wurde Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Tage | Schritte | Zeit | Temperatur |
| Tag 1 | Entnahme von Embryonen | 5-10 min/Embryo | RT (Tischgerät) |
| FITC Endo-Farbe | 10-30 min/Embryo | RT (Tischgerät) | |
| PFA-Perfusionskorrektur | Perfusion: 5-15 min/Embryo | Perfusion: RT (Benchtop) Inkubation: 4 °C | |
| Inkubation: O/N | |||
| Tag 2 | PBS-Wäsche | 90 Minuten | RT (Tischgerät) |
| Austrocknung | 5 Std. + O/N | RT (Rauchabzug) | |
| Tag 3 | Entfernung von Lipiden | 3,5 h | RT (Rauchabzug) |
| Lichtung | ≥1 h | RT (Tischgerät) |
Tabelle 1: Überblick über die Probenvorbereitung für die schnelle quantitative Bildgebung von Lichtschichten.
Diskussion
Die Fähigkeit, Biologie in 3D zu studieren, ist entscheidend für ein genaues Verständnis der morphologischen Komplexität, der inneren Organstruktur und der Gefäßverbindungen. Genaue und zuverlässige 3D-Gefäßbilder sind auch von zentraler Bedeutung für subjektspezifische hämodynamische Computersimulationen, die oft das einzige zuverlässige Mittel zur Quantifizierung wichtiger hämodynamischer Parameter wie Wandscherspannung und Druckverteilung sind. Hier stellen die Autoren eine schnelle und zugängliche Probenvorbereitungsmethode für die hochauflösende 3D-Gefäßbildgebung bei Kleintieren mittels LSFM vor. Die Methode erzeugt zuverlässig hochauflösende Bildstapel von HH18 (Tag 3) bis HH34 (Tag 8) Hühnerembryonen, die eine kritische Phase der Herzentwicklung darstellen. Von HH24 (Tag 4) bis HH34 (Tag 8) nimmt die Gesamtgröße des Embryos von ~30-550 mm3 zu, eine 2-fache durchschnittliche Wachstumssteigerung pro 24-Stunden-Periode, wobei die Herzmyokardgröße von ~1 mm3 (Tag 4) auf ~17 mm3 (Tag 8) zunimmt15. Aufgrund des schnellen Embryonen- und kardiovaskulären Wachstums wurden Bilder für die aktuelle Studie mit einer In-Plane-Auflösung von 0,61 μm bei HH18 (Tag 3) bis 2,28 μm bei HH34 (Tag 8) mit einem Z-Schritt-Bereich von 1,92-2,4 μm bei der Fokussierung auf das Rachenbogen-Arteriensystem und von 1,22 μm (HH18) bis 2,53 μm (HH34) mit einem Z-Schritt-Bereich von 1,94-3,5 μm bei der Erfassung des gesamten Embryos aufgenommen (Abbildung 4). Die erzeugten hochauflösenden Bildstapel eignen sich für die anatomische In-silico-Gefäßrekonstruktion und subjektspezifische hämodynamische Simulationen, wie HH26 (Tag 5) Modelle zeigen, die mit einer In-Plane-Auflösung von 1,30 μm und einer Z-Schrittgröße von 1,92 μm16 abgebildet wurden. Wie bei anderen Whole-Mount-Techniken kann die vorgeschlagene Methode in allen Tiermodellen eingesetzt werden, insbesondere bei der Durchführung von Ganzorgan-Bildgebungen 5,6,7. Die iDISCO+-Technik wurde auf neugeborene und erwachsene Mäuseherzen angewendet5.
Der große Vorteil der vorgeschlagenen Methode liegt in der Verwendung von FITC-markiertem Poly-L-Lysin für die Fluoreszenzmarkierung und ECi für die Clearing. Poly-L-Lysin ist unter physiologischem pH-Wert positiv geladen, so dass es unspezifisch an das vaskuläre Endothel bindet, während es durch die Probe perfundiert wird. Das Verfahren befestigt FITC schnell und zuverlässig an den Gefäßwänden und markiert sie mit einer hellen, stabilen Fluoreszenz, die sehr widerstandsfähig gegen Photobleiche ist. Traditionell erfordert die volumetrische Bildgebung der gesamten Halterung, einschließlich LSFM, die Markierung von Proben mit immunhistochemischer Färbung. Dieses Verfahren erfordert den Einsatz teurer Antikörper und verlängert die Probenvorbereitungszeit auf bis zu 4 Wochen 1,8,9. Frühe Embryonen exprimieren keine traditionellen Gefäßmarker wie Elastin oder glatte Gefäßmuskelzellen und sind daher schwieriger mit spezifischen Antikörpern zu erreichen. Darüber hinaus werden geklärte Proben oft abgebildet, während sie in die Reinigungslösung getaucht sind. Das klassische iDISCO+ Clearing-Verfahren erfordert die gefährliche Verbindung Dibenzylether, die in bildgebenden Einrichtungen oft nicht zugelassen ist8. ECi ist eine minimal gefährliche Verbindung, die außerhalb von Laborumgebungen sicher zu handhaben ist und mit geringerer Wahrscheinlichkeit optische Geräte beschädigt. Falls gewünscht, kann die vorgeschlagene Technik mit Immunfärbung kombiniert oder gemultiplext werden, vorausgesetzt, dass die gewählten spektralen Emissionseigenschaften Koexpressionsexperimente zulassen. Multiplexing kann die Probenmarkierung und die Vorbereitungszeit verlängern.
Das vorgeschlagene Protokoll weist einige Einschränkungen auf. Das Verfahren ist nur auf entwertete Embryonen und exzidiertes Gewebe anwendbar, so dass Längsschnittstudien ausgeschlossen sind. Die FITC-Poly-L-Lysin-Endo-Lackierung ist zwar schnell und wirtschaftlich, erfordert aber ein hohes Maß an Fingerfertigkeit und Präzision. Da der Farbstoff nicht ohne weiteres durch das Gewebe dringt, markiert er außerdem nur die Gefäßwände, die er erreichen kann, was eine Herausforderung für die Untersuchung kleiner Kapillarnetzwerke darstellt. Solche Herausforderungen können bewältigt werden, indem das Herz bei der Verabreichung des Poly-L-Lysins am Schlagen gehalten und die Viskosität/Konzentration der injizierten Lösung angepasst wird. Die Unspezifität des Farbstoffs erschwert die Unterscheidung zwischen den Gefäßtypen1. Überschüssiges FITC-Poly-L-Lysin, das während des Injektionsprozesses aus dem Gefäßsystem austritt, kann sich auch an nicht-vaskuläre Gewebe wie oberflächliche Haut und Membran binden, was das leuchtende Lichtblatt während der Bildgebung beeinträchtigen und die Bildqualität beeinträchtigen kann. Der Benutzer sollte bei der Injektion des Poly-L-Lysins wachsam sein, um ein Auslaufen zu vermeiden. Das aktuelle Protokoll wird umfassend validiert, indem Hühnerembryonen im Frühstadium für die Bildgebung der großen systemischen Arterien verwendet werden. Die Konzentration, das Volumen und die Fixierungszeit der FITC-Poly-L-Lysin-Injektion müssen möglicherweise für verschiedene Anwendungsfälle weiter optimiert werden.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Arbeit wurde durch einen American Heart Association Career Development Award, den Burroughs Wellcome Fund Career Award an der Scientific Interface, den Additional Ventures Single Ventricle Research Fund und den UCSD School of Medicine Microscopy Core (Grant P30 NS047101) unterstützt. Die Autoren danken Dr. Bobby Thompson für seine Einführung in die Endo-Malerei, dem Microscopy Core der UCSD School of Medicine und Robert Porter (UCSD) für die experimentelle Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| #5 forceps | Fine Science Tools | 11252-30 | |
| #55-forceps | Fine Science Tools | 11295-51 | |
| 0.03 inch inner diameter silicone tubing | VWR | 32829-182 | |
| 20 μL pipette tips | VWR | 76322-134 | |
| 35 mm Petri dish | VWR | 10799-192 | |
| 5 mL plastic syringe | VWR | BD 309646 | |
| Dichloromethane (DCM) | Sigma-Aldrich | 270997 | Refer to MSDS. Stored in side cabinet under fume hood |
| Ethyl cinnamate (ECi) | Sigma-Aldrich | 112372 | Stored at 4 °C |
| Fine Curved scissors | Fine Science Tools | 14061-09 | |
| FITC-poly-L-lysine | Sigma-Aldrich | P3069 | Store at -20 °C (powder, stock solution), 4ºC (working solution) |
| Fluoresent microscope | EVIDENT SCIENTIFIC | MVX10 | |
| Glass capillary tubes (0.75 mm ID) | Sutter Instrument | FG-GB100-75-10 | |
| Lightsheet microscope | Zeiss | Z.1 system | |
| Methanol | Sigma-Aldrich | M1775 | Refer to MSDS. Stored in flammable cabinet under fume hood |
| Microforge | Narishige International USA, Inc. | MF2 | |
| Micromanipulator | World Percision Instrrument | M3301R | |
| Paraformaldehyde (PFA) 4% | Thermo Scientific | J19943.K2 | Refer to MSDS. Stored at -20 °C (powder), 4 °C (4% working solution) |
| Phosphate buffered saline (PBS) | Cytiva | SH30256.01 | Stored on benchtop |
| SimVascular | open source software www.simvascular.org | ||
| Tyrode’s Solution | Made in-house |
Referenzen
- Lindsey, S. E., Butcher, J. T., Vignon-Clementel, I. E. Cohort-based multiscale analysis of hemodynamic-driven growth and remodeling of the embryonic pharyngeal arch arteries. Development. 145 (20), dev162578(2018).
- Lindsey, S. E., Vignon-Clementel, I. E., Butcher, J. T. Assessing early cardiac outflow tract adaptive responses through combined experimental-computational manipulations. Ann Biomed Eng. 49 (12), 3227-3242 (2021).
- Salman, H. E., et al. Effect of left atrial ligation-driven altered inflow hemodynamics on embryonic heart development: Clues for prenatal progression of hypoplastic left heart syndrome. Biomech Model Mechanobiol. 20 (2), 733-750 (2021).
- Henning, A. L., Jiang, M. X., Yalcin, H. C., Butcher, J. T. Quantitative three-dimensional imaging of live avian embryonic morphogenesis via micro-computed tomography. Dev Dyn. 240 (8), 1949-1957 (2011).
- Anbazhakan, S., et al. Blood flow modeling reveals improved collateral artery performance during the regenerative period in mammalian hearts. Nat Cardiovasc Res. 1 (8), 775-790 (2022).
- Pan, C., et al. Shrinkage-mediated imaging of entire organs and organisms using uDISCO. Nat Methods. 13 (10), 859-867 (2016).
- Renier, N., et al. iDISCO: a simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159 (4), 896-910 (2016).
- Vieites-Prado, A., Renier, N. Tissue clearing and 3D imaging in developmental biology. Development. 148 (18), dev199369(2021).
- Rios Coronado, P. E., Red-Horse, K. Enhancing cardiovascular research with whole-organ imaging. Curr Opin Hematol. 28 (3), 214-220 (2021).
- Miller, C. E., et al. Confocal imaging of the embryonic heart: How deep. Microsc Microanal. 11 (3), 216-223 (2005).
- Renier, N., et al. Mapping of brain activity by automated volume analysis of immediate early genes. Cell. 165 (7), 1789-1802 (2016).
- Lindsey, S. E., et al. Growth and hemodynamics after early embryonic aortic arch occlusion. Biomech Model Mechanobiol. 14 (4), 735-751 (2015).
- Humphrey, J. D. Constrained mixture models of soft tissue growth and remodeling-twenty years after. J Elast. 145 (1), 49-75 (2021).
- Updegrove, A., et al. SimVascular: An open-source pipeline for cardiovascular simulation. Ann Biomed Eng. 45 (3), 525-541 (2017).
- Kim, J. S., Min, J., Recknagel, A. K., Riccio, M., Butcher, J. T. Quantitative three-dimensional analysis of embryonic chick morphogenesis via microcomputed tomography. Anat Rec. 294 (1), 1-10 (2011).
- Zhang, D., Lindsey, S. Evaluation of high-resolution image accuracy for small animal vascular flow quantitation. Bull Am Phys Soc. , https://meetings.aps.org/Meeting/DFD24/Session/X03.8 (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten