Method Article
CRISPR-Cas9 시스템을 사용한 RIP1 Knockout U937 세포주 생성
요약
CRISPR 관련 프로토콜이 점점 더 유용해지고 접근성이 높아짐에 따라 특정 실험 조건에서 복잡성과 장애물이 여전히 발생할 수 있습니다. 이 프로토콜은 CRISPR/Cas9을 사용하여 수용체 상호 작용 세린/트레오닌-단백질 키나아제 1(RIPK1/RIP1) 녹아웃 인간 세포주를 생성하는 방법을 간략하게 설명하고 이 과정에서 직면할 수 있는 잠재적인 문제를 강조합니다.
초록
이 프로토콜은 인간 단핵구 U937 세포주에서 CRISPR/Cas9을 사용하여 RIP1 유전자를 knockout하는 절차를 간략하게 설명합니다. 이 방법은 지정된 가이드 RNA 플라스미드와 렌티바이러스 패키징 플라스미드를 사용하여 RIP1 유전자 knockout을 달성합니다. 이 프로토콜은 기존 CRISPR 방법의 문제점과 개선 사항을 해결하여 향후 세포 사멸 연구를 위한 복제를 가능하게 합니다. 그 결과로 생성된 돌연변이 세포는 기능적 RIP1 단백질이 다른 역할을 하는 세포 사멸의 기계론적 변화를 조사하는 데 사용할 수 있습니다. 생존력 분석은 괴사 유도 후 녹아웃 세포에서 세포 사멸을 현저히 감소시키는 것으로 나타났습니다. 형광 현미경 검사는 동일한 조건에서 녹아웃 세포에서 미토콘드리아 활성 산소 종(ROS)의 현저한 감소를 보여주었습니다. 이러한 기능 분석법은 함께 RIP1 단백질의 손실을 확인합니다. U937 인간 단핵구와 함께 사용하도록 최적화된 이 절차는 다른 주요 세포 사멸 조절자를 표적으로 하도록 조정하여 기능적이고 치명적이지 않은 돌연변이를 생성할 수도 있습니다. 돌연변이 생성 중에 발생할 수 있는 문제에 대한 통찰력을 제공하기 위해 잠재적인 함정이 전체적으로 해결됩니다.
서문
CRISPR/Cas9 유전자 편집 기술의 사용은발견 1,2,3 이후 빠르게 발전해 왔습니다. 세포주 또는 박테리아 내에서 유전자를 knock-in하거나 knockout하는 능력은 연구를 진행하고 세포 내 메커니즘에 대한 이해를 촉진하는 데 매우 중요합니다 1,2,3,4,5,6. CRISPR-Cas9 시스템은 유전자 특이성 엔지니어링을 단순화하여 전사 활성제 유사 효과기 뉴클레아제(TALEN)와 같은 이전 유전자 편집 방법을 개선합니다. 이 절차에는 두 가지 기본 구성 요소가 포함됩니다: 의도된 유전자 표적을 찾는 데 사용되는 가이드 RNA(gRNA)와 이중 가닥 DNA 단절 3,4로 의도된 게놈 위치를 수정하는 엔도뉴클레아제인 Cas9. gRNA는 Cas9 엔도뉴클레아제가 왓슨-크릭 염기쌍을 통해 의도한 유전자 염기서열에서 이중 가닥 절단을 찾고 시작하기 위한 가이드 역할을 합니다. CRISPR/Cas9 시스템에 의한 게놈 편집의 전체 프로세스에는 비상동 말단 결합(NHEJ) 또는 상동 재조합(homologous recombination)을 통해 DNA의 이중 가닥 절단을 복구하는 세포 기계가 포함됩니다 3,7. NHEJ가 발생할 가능성이 더 높으며, 게놈에 돌연변이를 효과적으로 생성하여 표적 유전자의 발현 상실을 초래합니다 3,4.
상용 소스는 박테리아 성장 및 분리를 통해 발현될 수 있는 gRNA 타겟 라이브러리를 생성할 수 있었으며, 이는 사용 편의성을 크게 향상시킵니다. 그러나 CRISPR/Cas9 시스템의 주요 한계는 gRNA 및 Cas9 복합체를 타겟 세포주에 전달하기 어렵다는 것입니다. 이러한 한계는 일반적으로 hard-to-transfection으로 지칭되는 현탁 세포주에서 발생합니다8. 일반적인 transfection 방법은 일반적으로 CRISPR/Cas9 시스템을 suspension cell에 전달하는 데 효율적이지 않기 때문에 lentiviral transfection 및 transduction과 같은 바이러스 전달 방법이 이러한 유형의 세포주에 더 적합합니다 8,9.
이러한 유형의 transfection에는 gRNA 및 Cas9 엔도뉴클레아제를 인코딩하는 렌티바이러스 벡터와 렌티바이러스 패키징 플라스미드가 필요하며, 이는 렌티바이러스 입자를 제조할 수 있는 세포주로 transfection됩니다. 이 과정에서 일반적으로 선택되는 세포주는 HEK293T세포인데, 이는 세포가 형질주입이 쉽고 gRNA와 Cas9 9,10의 조립에서 매우 효율적으로 작용하기 때문입니다. 그런 다음 이러한 입자는 렌티바이러스로 상층액으로 방출되며, 이는 gRNA 및 Cas9를 U937 인간 단핵구와 같은 의도된 현탁 세포주로 형질전환시키는 데 사용할 수 있습니다. 이와 같이, 여기에 설명된 절차는 확립된 방법과 비교하여 다음과 같은 변경 사항이 있습니다: (1) transfection이 어려운 세포주에 대한 대체 transfection 방법; (2) CRISPR 플라스미드 DNA를 농축하거나 초원심분리기를 사용할 필요가 없습니다. (3) 단일 세포 클로닝의 필요성을 제거합니다.
이 논문의 직접적인 초점은 U937 인간 단핵구에서 RIP1 유전자를 녹아웃하는 것이었습니다. 염증성이 높은 세포 사멸 경로 괴사(cell death pathway necroptosis)의 표준 형태는 세포 사멸 연구의 중추적인 표적 역할을 하는 RIP1에 의해 제어됩니다. 11,12,13,14 RIP1이 자가인산화를 통해 활성화되면, 수용체 상호작용 세린/트레오닌-단백질 키나아제 3(RIPK3/RIP3) 및 혼합 계통 키나아제 도메인 유사(MLKL) 슈도키나아제를 모집하여 직접 인산화 및 활성화를 일으켜 네크로솜을 형성합니다. 이 형성에 따라 괴사체는 세포 전체를 자유롭게 이동하여 미토콘드리아12,13와 같은 세포 기관과 상호 작용합니다. 미토콘드리아에서 RIP1은 세포 대사와 함께 긍정적 인 피드백 루프를 강화하여 미토콘드리아 ROS의 생성에 직접적인 영향을 미치고, 이는 차례로 RIP1의 추가 자가인산화, 괴사 형성 및 괴사증 11,12,13,14의 다운스트림 실행을 촉진합니다.
현재 연구 그룹의 초점은 세포 사멸에서 RIP1의 역할이지만, RIP1을 연구하는 다른 이유에는 염증 및 감염에 대한 역할도 포함됩니다. TNF 수용체와 같은 사멸 수용체에 의해 활성화되면, RIP1은 NF-κB 신호전달 경로의 활성화를 촉진하여 면역세포 동원에 필수적인 전염증성 사이토카인, 케모카인 및 기타 분자의 전사를 유발하고 염증 반응의 증폭을 촉진합니다15. NF-κB 활성화 외에도 RIPK1은 MAPK 신호 경로에 관여하여 염증을 더욱 강화할 수 있습니다15,16. 감염에 대한 반응에서의 역할과 관련하여, RIP1은 숙주 염증 반응의 중추적인 매개체 역할을 하며, 특히 톨 유사 수용체(TLR)와 같은 패턴 인식 수용체(PRR)에 의해 인식되는 병원체 관련 분자 패턴(PAMP)에 대한 반응으로 작용합니다17. 더욱이, 패혈증이 진행되는 동안 RIP1은 TNF 수용체와 같은 사멸 수용체를 통한 신호에 의해 활성화되어 전염증성 폭포(pro-inflammatory cascades)를 시작합니다. RIPK1은 NF-κB 및 MAPK 경로의 활성화를 매개하여 패혈증18의 전신 염증 반응의 핵심 동인인 TNF-α, IL-1β 및 IL-6와 같은 전염증성 사이토카인의 생성을 촉진합니다.
프로토콜
절차의 개략도는 그림 1에 나와 있습니다. 가이드 RNA(gRNA) 및 타겟 염기서열은 표 1에 나와 있습니다. 시약 및 사용된 장비에 대한 자세한 내용은 재료 표에 나열되어 있습니다.
1. 대장균에서 Cas9 엔도뉴클레아제 및 퓨로마이신 내성을 포함하는 CRISPR gRNA 렌티바이러스 발현 벡터를 표적으로 하는 RIP1 수확
- 100 μg/mL 암피실린이 보충된 LB 한천 플레이트에 렌티바이러스 벡터 gRNA를 품고 있는 사분면 줄무늬 대장균.
- 플레이트의 가장 희석된 영역에서 개별 콜로니가 보일 때까지 37°C에서 1-2일 동안 플레이트를 배양합니다.
- 멸균 루프가 있는 단일 콜로니를 선택하고 각 콜로니를 50mL 원뿔형 튜브에 100μg/mL 암피실린이 보충된 5mL의 LB 육수에 개별적으로 추가합니다. 균질화를 보장하기 위해 단일 콜로니를 추가한 후 위아래로 철저히 피펫팅하십시오.
- 50mL 코니컬 튜브의 캡을 환기시키고 테이프로 감은 다음 37°C 및 225rpm의 오비탈 셰이커에서 8시간 동안 배양합니다.
- 37°C에서 8시간 배양 동안 100μg/mL 암피실린이 보충된 LB 육수 40mL를 4개의 개별 50mL 원뿔형 튜브 각각에 추가합니다.
- 37°C에서 8시간 배양 후 성장한 E 40μL를 취합니다 . 대장균 배양액을 넣고 4개의 50mL 코니컬 튜브 모두에 추가합니다.
- 50mL 코니컬 튜브의 캡을 환기시키고 테이프로 감아 37°C 및 225rpm의 오비탈 셰이커에서 12-16시간 동안 배양합니다.
- 37°C에서 배양한 후 스윙 버킷 로터에서 3220 x g 의 모든 튜브를 20분 동안 회전시켜 성장한 대장균 박테리아를 펠릿화합니다.
- 상등액을 폐기물 비커에 디캔팅한 다음 100μg/mL 암피실린이 보충된 10mL의 LB 육수에 4개의 펠릿을 모두 결합합니다.
- 20분 동안 흔들리는 버킷 로터에서 결합된 펠릿을 최대 속도로 다시 회전시켜 단일 펠릿을 얻습니다.
- 가능한 한 많은 상등액을 폐기물 비커에 버리고 다음 옵션 중 하나를 진행하십시오.: (1) 젖은 펠릿을 -80°에서 최대 1개월 동안 그대로 동결합니다. (2) 렌티바이러스 벡터 플라스미드 정제를 진행한다.
참고: 옵션 1을 선택하면 여기에서 프로토콜이 일시 중지될 수 있습니다.
2. RIP1을 타겟으로 하는 정제된 CRISPR gRNA 렌티바이러스 발현 벡터를 사용한 HEK293T세포 transfection
- 3 ×10 6 × 5 106 세포의 농도로 HEK293T 세포를 4.5 g/L D-글루코오스, L-글루타민, 110 mg/L 피루브산 나트륨 및 10 % 열 비활성화 FBS가 포함된 DMEM이 포함된 10cm 플레이트에 하룻밤 동안 파딩합니다.
- 다음 날, 플레이트의 밀도가 약 70%-90%인지 확인한 다음 프로토콜을 진행합니다.
- 1:1:1:1 비율의 플라스미드(실험용 CRISPR 플라스미드: pLP1: pLP2: pLP/VSVG)와 함께 3:1 비율(3 시약: 1 파트 플라스미드 DNA)과 함께 환원된 혈청 배지를 사용하여 세포의 transfection을 설정합니다.
참고: 비율의 pLP1: pLP2: pLP/VSVG 부분은 렌티바이러스 패키징 혼합물입니다.- transfection 설정 전에 transfection 시약, 환원된 혈청 배지, CRISPR 플라스미드 DNA 및 렌티바이러스 패키징 혼합물을 실온으로 평형을 이룹니다.
- 75 μL의 transfection 시약, 2500 μL의 환원된 혈청 배지 및 25 μg의 총 DNA를 함께 혼합합니다(CRISPR 플라스미드 DNA에 대한 분광광도계 분석 및 렌티바이러스 패키징 혼합물을 사용한 1:1:1:1 비율에 기반한 계산).
- 이 혼합물을 실온에서 15-30분 동안 배양합니다. 이 혼합물의 전체 부피를 배지에 바로 합류 HEK293T 세포 플레이트로 조심스럽게 피펫팅합니다(배지를 변경할 필요 없음).
- 플레이트를 부드럽게 소용돌이쳐 transfection mix와 플레이트 배지의 적절한 균질화를 보장합니다. 플레이트를 37 ° C에서 48 시간 동안 배양합니다.
3. U937 세포 또는 표적 세포의 렌티바이러스 형질도입
- 37°C 에서 48시간 배양이 완료되면 생산자 HEK293T 세포 플레이트의 배지를 15mL 코니컬 튜브로 옮깁니다.
- 실온에서 800 x g 의 부피를 5분 동안 원심분리하여 남아 있는 HEK293T 세포를 펠릿화합니다. 펠릿화된 HEK293T 세포를 방해하지 않도록 주의하면서 바이러스를 함유한 모든 상등액을 제거하고 별도의 15mL 원뿔형 튜브에 보관하십시오. 그런 다음 오염을 제거하고 펠릿 함유 튜브를 적절한 생물학적 위험 폐기물 용기에 버리십시오.
참고: HIV 기반 렌티바이러스 상등액은 -80°C에서 보관할 수 있습니다. 그러나 그렇게 하면 첫 번째 동결/해동 주기19 이후 바이러스 안정성이 최대 55%까지 손실될 위험이 있습니다. - 실온에서 400 x g에서 10분 동안 적절한 부피를 원심분리하고 상등액을 디캔팅하여 15mL 코니컬 튜브에서 2 × 106 U937 세포의 펠릿을 계수하고 얻습니다.
- U937 세포 펠릿에 바이러스가 함유된 모든 상등액을 다시 부유시킨 다음 290 x g 에서 60분 동안 튜브를 원심분리합니다.
- 원심분리 후 피펫을 사용하여 이미 튜브에 있는 바이러스 함유 상등액과 함께 펠릿을 재현탁합니다.
- 튜브를 종단 간 회전자(또는 유사한 회전 장치)에 60분 동안 놓습니다.
- 이 배양 후 실온에서 10분 동안 400 x g 의 튜브를 원심분리하여 세포를 펠릿화합니다.
- 바이러스 함유 상등액과 10% 열 불활성화 FBS가 보충된 완전한 RPMI-1640 배지의 1:1 비율 배지 혼합물로 세포 펠릿을 재현탁합니다.
- 세포 혼합물을 10cm 조직 배양 플레이트에 옮기고 37°C에서 48시간 동안 배양합니다.
- 이 배양 후 U937 세포를 400 x g 에서 10분 동안 원심분리하고 상층액을 제거합니다.
- 10% 열 비활성화 FBS와 5μg/mL의 퓨로마이신이 보충된 완전한 RPMI-1640 배지로 펠릿을 재현탁하고 세포를 T25 플라스크로 옮깁니다.
- 37 °C에서 2-3주 동안 손대지 않은 상태로 세포를 배양하고 1-2일마다 세포 성장 징후를 확인합니다.
참고: 세포가 합류하고 초기 통과 후 건강한 전환 시간을 갖게 되면 완료된 프로토콜의 효과를 테스트할 수 있습니다.
4. 웨스턴 블롯 분석을 이용한 RIP1 CRISPR 돌연변이 세포 생성에서 완성된 프로토콜의 효과 테스트
- 야생형(WT) U937 단핵구, 비표적 대조군(NTC) CRISPR 세포 및 RIP1 CRISPR 돌연변이 세포 11,12,13의 세포 단백질 용해물을 생성합니다.
- SDS-PAGE 겔에서 이러한 용해물을 실행하고 웨스턴 블롯 분석14을 진행합니다.
- 먼저 샘플을 하우스키핑 단백질로 정규화합니다.
- 정규화 후 RIP1 단백질 발현 수준에 대한 샘플을 분석하여 RIP1 CRISPR 돌연변이 세포주14의 성공 여부를 적절하게 판단합니다.
- 웨스턴 블롯 분석 형태에 따라 세포를 실험 목적으로 사용할 수 있습니다.
결과
RIP1 CRISPR 돌연변이 U937 세포의 합류 집단을 생산한 후 SDS-PAGE 및 웨스턴 블롯 분석을 수행했습니다. 웨스턴 블롯 분석은 RIP1 단백질 발현 수준의 손실을 평가하여 RIP1 CRISPR 돌연변이 세포주의 성공적인 생성을 결정하는 데 사용되었습니다. 이 결정은 WT U937 단핵구와 NTC 세포의 비교 결과를 기반으로 이루어졌습니다. 그림 2에서 RIP1의 발현은 5 μg/mL puromycin RIP1 CRISPR 돌연변이 세포에서는 검출되지 않았지만 WT U937 및 NTC 세포 모두에서 검출되었습니다. 이 결과는 이 프로토콜이 실행 가능한 RIP1 CRISPR 돌연변이를 선택하기 위한 신뢰할 수 있고 효과적인 방법임을 보여줍니다.
RIP1 CRISPR 돌연변이 세포에서 RIP1 단백질 발현이 성공적으로 소실됨에 따라 이것이 RIP1의 기능적 손실과 동일한지 확인했습니다. 이전에 확립된 연구에서 종양괴사인자-알파(TNF-α), 시클로헥시미드 및 판카스파제 억제제 zVAD-fmk의 조합은 TCZ11,14로 약칭되는 괴사의 직접적인 자극입니다. 그림 3에서 볼 수 있듯이, TCZ 처리로 인한 괴사 세포 사멸 유도는 WT U937 단핵구와 NTC 세포주 모두에서 알려진 RIP1 억제제인 necrostatin-1s(nec-1s)로 치료한 후 되돌려집니다. 그러나 RIP1 CRISPR 돌연변이 세포의 경우 TCZ 처리로 인해 WT 및 NTC 세포에 비해 세포 사멸이 크게 감소했습니다. 이후 nec-1을 사용한 처리는 RIP1 CRISPR 돌연변이 세포에 영향을 미치지 않았으며, 이는 괴사와 RIP1 기능이 없음을 나타냅니다.
이러한 RIP1 CRISPR 돌연변이 세포 내 RIP1 기능 손실을 추가로 평가하기 위해 DNA Hoecsht 염색(청색 형광) 및 미토콘드리아 과산화물 염색(적색 형광)으로 생세포 형광 현미경 검사를 수행했습니다. 이러한 실험을 통해 우리는 이전에 확립된 괴사 중 미토콘드리아 과산화물 생산의 증가가 이러한 세포에서 손실되는지 확인할 수 있었습니다. RIP1은 세포 대사와 직접 상호 작용하고 미토콘드리아 과산화물 생산을 크게 증가시키는 것으로 알려져 있습니다. 세포 내에 이러한 과산화물이 풍부하면 RIP1이 활성화되고 괴사(necroptosis)가 다운스트림으로 실행되면서 긍정적인 피드백 루프가 생성됩니다. 그림 4에서 볼 수 있듯이 TCZ를 사용한 처리는 WT U937 단핵구와 NTC 세포 모두에서 미토콘드리아 과산화물 생성이 풍부했지만 RIP1 CRISPR 돌연변이 세포에서는 그렇지 않았습니다. 이후 nec-1을 처리한 결과 WT U937 단핵구와 NTC 세포 모두에서 적색 형광 염색이 크게 감소했으며, RIP1 CRISPR 돌연변이 세포에서는 변화가 관찰되지 않았습니다.

그림 1: CRISPR/Cas9 프로토콜의 개략도 모델. 이해를 돕기 위해 이 프로토콜 내에서 제공되는 다양한 단계를 시각적으로 표현합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
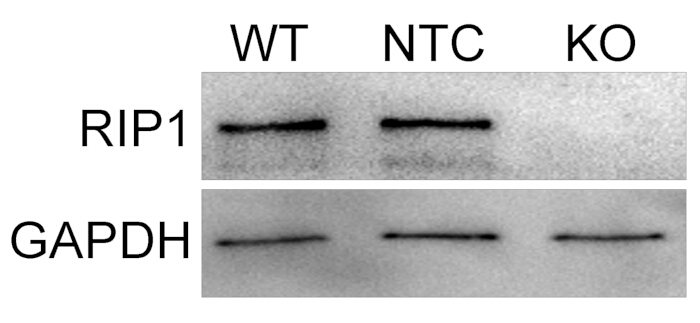
그림 2: 웨스턴 블롯 분석은 RIP1 CRISPR 돌연변이 세포에서 RIP1 단백질 발현 소실을 확인했습니다.RIP1 단백질의 발현은 WT U937 세포 및 NTC 세포와 비교할 때 RIP1 CRISPR 돌연변이 U937 세포에서 5μg/mL 퓨로마이신 선택에서 손실되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: 세포 생존율 분석은 RIP1 CRISPR 돌연변이 세포에서 RIP1의 기능 상실을 보여줍니다.TCZ 처리를 통해 괴사 세포 사멸을 유도하고 NEC-1을 사용한 후속 처리로 분석했습니다. WT U937 단핵구와 NTC 세포 모두 nec-1 치료 후 세포 사멸이 현저히 감소한 것으로 나타났습니다. RIP1 CRISPR 돌연변이 세포는 TCZ로 처리했을 때 세포 사멸이 훨씬 적었고 이후 nec-1로 처리했을 때 변화가 없었습니다. 표시된 결과는 3개의 독립적인 실험에서 얻은 것입니다. 오차 막대는 표준 편차를 나타냅니다. Bonferroni 사후 테스트를 사용한 양방향 분산 분석 ***p < 0.001. ns = 통계적으로 유의하지 않음. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 형광 현미경을 사용한 RIP1 CRISPR 돌연변이 세포의 RIP1에 대한 다운스트림 기능 분석.(A) 미토콘드리아 과산화물 염색(적색 형광) 측정은 nec-1s 처리 후 WT U937 단핵구와 NTC 세포 모두에서 과산화물 생성의 감소를 보여주었습니다. RIP1 CRISPR 돌연변이 세포는 TCZ로 처리했을 때 미토콘드리아 과산화물의 생성을 보여주지 않았으며 이후 nec-1로 처리했을 때 변화가 없었습니다. 스케일 바: 100μm. (B) (A)에 표시된 결과의 평균 형광 강도 정량화. 표시된 결과는 3개의 독립적인 실험에서 얻은 것입니다. 오차 막대는 표준 편차를 나타냅니다. Bonferroni 사후 테스트를 사용한 양방향 분산 분석, ***p < 0.001. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 유전자 표적 | 유전자 ID | 지RNA | 유전자의 해당 뉴클레오티드 | 팸 | Cas9 컷사이트 | ||
| 립1 | 8737 | GCTTTGCGTTGACGTCATTC | 88-841 | 아그 | 816 | ||
표 1: 가이드 RNA(gRNA) 및 타겟 염기서열.
토론
이 프로토콜은 RIP1 knockout U937 세포주를 생성하기 위해 렌티바이러스 transfection 및 transduction의 효율성과 신뢰성에 대한 잠재적 함정에 대한 자세한 지침과 분석을 제공하는 것을 목표로 합니다. 이러한 transfection 및 transduction 방법은 힘들고 시간이 많이 걸리지만, 일반적으로 선택한 gRNA 및 Cas9 endonuclease를 transfection이 어려운 세포주에 통합하는 효율적인 방법으로 간주됩니다 8,9,20. U937 세포 및 다른 면역 세포와 같은 단핵구의 경우와 마찬가지로, 외부 핵산을 인식하고 반응하는 선천적 센서를 가지고 있기 때문에 transfection을 수행하는 데 본질적으로 어려움이 있습니다. 이는 핵산 파괴/과돌연변이, 전사 억제 및/또는 외부 핵산에 대한 반응으로 세포 사멸을 유발할 수 있습니다21.
이 전달 방법은 렌티바이러스 입자, HEK293T 세포를 제조할 수 있는 세포주를 사용해야만 가능하며, 이는 transfection이 쉬운 것으로 널리 받아들여지는 부착 인간 배아 신장 세포주입니다 9,10. 이 정보를 통해 이 절차에서 주의해서 처리해야 하는 가장 중요한 단계가 간략하게 설명되어 있습니다. 세포 배양 및 모니터링이 가장 중요합니다. 이 연구는 U937 세포를 빠르게 성장하고 사용하기 쉬운 현탁 인간 단핵구인 gRNA 및 Cas9의 표적 세포주로 사용했습니다22. 빠르게 성장하는 대부분의 세포와 마찬가지로, 세포가 과도하게 자라서 실험에서 예상대로 수행되지 않는 것을 방지하기 위해 세포의 정기적인 유지 관리 및 통과가 중요합니다. 이 프로토콜의 HEK293T 셀에도 동일한 개념이 적용됩니다. 이러한 세포주가 프로토콜에서 사용되는 동안 적절하게 유지되고 건강하고 융합된 상태로 유지되도록 하는 것은 효율적인 렌티바이러스 생산에 필수적입니다. 이 절차의 또 다른 중요한 단계는 적절한 농도의 transfection 시약과 CRISPR plasmid DNA와 lentiviral 패키징 혼합물의 비율을 사용하는 것입니다. 이러한 수학적 비율은 이 방법의 여러 다른 농도에서 경험적으로 테스트되었으며, 나열된 값은 HEK293T 세포의 transfection에 중요합니다.
대부분의 고안된 방법 및 절차와 마찬가지로 확립된 작업 실험 프로세스에 들어가는 상당한 양의 문제 해결이 있습니다. 다른 승인된 프로토콜과 함께 여기에서 결정된 한 가지 문제는 U937 세포에 대한 더 짧은 형질도입 단계 후 퓨로마이신의 즉각적인 도전입니다. transduction step은 2 × 106 U937 세포만 사용하기 때문에 배지의 절반이 완전한 RPMI-1640에 10% 열 비활성화 FBS가 보충된 전체 48시간의 배양이 이 RIP1 CRISPR 돌연변이 세포주의 생성을 촉진하는 것으로 경험적으로 확인되었습니다. 세포에 개입 없이 5μg/mL의 퓨로마이신을 지속적으로 투여했을 때, 2-3주 후에 살아있고 건강한 U937 세포가 배양액에 나타나기 시작했습니다. 이 프로토콜은 시간이 많이 걸리기 때문에 이 transfection 및 transduction 단계를 여러 번 반복하면 여러 가지 가능한 복구 가능한 배양이 있습니다. 배양이 융합되면 SDS-PAGE 및 Western blotting 기술을 통해 프로토콜의 효과를 확인할 수 있습니다. RIP1 CRISPR 돌연변이 세포주를 성공적으로 분리하고 확인한 후에도 실험에서 이러한 세포를 지속적으로 사용하기 위해 RIP1 단백질 발현의 세포 손실에 대한 단백질 용해물 기반 SDS-PAGE 및 웨스턴 블롯 분석을 정기적으로 모니터링하는 것이 좋습니다.
경험적 문제 해결이 필요한 또 다른 이유는 면역 세포주와 함께 transfection 시약을 사용하는 데 사용할 수 있는 간단한 정보가 부족하기 때문입니다. 회사와 프로토콜에서 사용할 수 있는 정보 사이에는 이 프로토콜에 사용되는 transfection 시약과 DNA의 농도 기반 비율에 관한 상충되는 정보가 있습니다. 시도 범위는 각각 5:1에서 3:1 사이이지만, 3:1이 사용할 최대 비율이며, 그렇지 않으면 HEK293T 세포에 대한 transfection이 효율적으로 완료되지 않는 것으로 판단되었습니다. 이러한 선택과 함께, 더 낮은 부피에서 CRISPR 플라스미드 DNA를 단일하게 반복하는 것은 10cm 플레이트의 HEK293T 세포를 완전히 transfection하기에 충분하지 않다는 것이 확인되었습니다. plasmid purification kit는 E. coli에서 많은 양의 CRISPR plasmid DNA를 생성하므로 모든 transfection 성분의 부피가 증가하고, transduction 후 U937 세포를 회수하기 위한 transfection 효율이 5배 향상되었습니다. 프로토콜의 이러한 구성 요소에 대해 나열된 볼륨은 허용된 프로토콜의 단일 반복에서 5배 증가를 나타냅니다. transfection 시약 비율의 이러한 변화와 이러한 구성 요소에 사용되는 총 부피 사이에서 프로토콜은 CRISPR 플라스미드 DNA를 농축하거나 초원심분리기를 사용할 필요 없이 고안되었습니다. 그러나, 이러한 물질을 이용할 수 있는 경우, 확립된 방법을 사용하여 분리된 CRISPR 플라스미드 DNA를 농축할 수 있으며, 이에 따라 이 프로토콜의 다른 구성 요소의 필요한 부피를 줄일 수 있습니다23,24.
렌티바이러스 transfection 방법을 사용할 때 CRISPR/Cas9 유전자 편집에 대한 사용에 대한 한계가 있을 수 있습니다. 가장 주목할 만한 제한 사항은 이 프로토콜 9,20에 대한 시간 및 노동 요구 사항입니다. 이 절차는 일시 중지할 수 있지만 transfection이 어려운 세포주에 최적화된 두 단계만 포함합니다. U937 단핵구와 같이 transfection이 어려운 세포주의 경우, 플라스미드의 화학적 매개 도입을 포함한 일반적인 transfection 방법은 렌티바이러스 기반 전달만큼 효과적이지 않습니다25.
보다 광범위한 연구 관심은 U937 단핵구에서 세포사멸에서 괴사(necroptosis)로의 고혈당 이동에 초점을 맞추고 있으며, 이 과정에서 기계론적 이해를 위해 RIP1의 기능적 녹아웃(knockout)의 필요성을 강조합니다. RIP1은 괴사(necroptosis)의 실행에 중요한 단백질이기 때문에 세포 내의 모든 기능과 단백질 상호 작용을 이해하는 것이 필수적입니다 11,12,13,14. 이 프로토콜의 향후 방향에는 세포사멸에서 괴사화로의 고혈당 전환에 관한 추가 조사 및 실험에 이러한 방법을 적용하는 것이 포함됩니다. 이 프로토콜은 U937 세포와 RIP1의 기능적 knockout을 넘어 CRISPR/Cas9 시스템을 사용하여 다양한 gRNA 표적에 대한 부유 세포를 사용한 lentiviral transfection 및 transduction 방법에 광범위하게 적용할 수 있을 것으로 예상됩니다.
공개
없음.
감사의 말
이 연구는 미국 국립보건원(NIH)의 국립심장폐혈액연구소(NHLBI)의 자금 지원을 받았으며, 허가 번호 NIH 2R15-HL135675-02는 T.J.L.에 수여했습니다.
자료
| Name | Company | Catalog Number | Comments |
| Adjusted DMEM Medium | Gibco | 11995-040 | |
| Ampicillin | Sigma | A1593 | |
| bisBenzimide Hoechst 33342 trihydrochloride | Sigma | B2261 | |
| Complete RPMI-1640 Medium | Sigma | R6504 | |
| CRISPR NTC gRNA E.coli Strain | transOMIC | TELA1011 | |
| CRISPR RIP1 gRNA E.coli Strain | transOMIC | TEVH-1162203 | |
| End-over-end Rotator | Thermo Scientific | ||
| EVOS FL Fluorescence Microscope | Life Technologies | ||
| GenElute Plasmid Maxiprep Kit | Sigma | PLX15 | |
| Goat Anti-Rabbit IgG Antibody, (H+L) HRP conjugate | Sigma | AP307P | |
| HEK293T Cells | ATCC | ||
| Incubator Shaker | New Brunswick Scientific | ||
| LB Agar | BD | 244520 | |
| LB Broth | BD | 244610 | |
| LV-MAX Lentiviral Packaging Mix | Gibco | A43237 | |
| MitoSOX Red | MedChemExpress | HY-D1055 | |
| NanoDrop Spectrophotometer | Thermo Scientific | ||
| Necrostatin-1 | MedChemExpress | HY-14622A | |
| OPTI-MEM | Gibco | 31985-062 | |
| Puromycin | Sigma | P7255 | |
| Rabbit anti-human RIP1 mAb | Cell Signaling Technology | 3493 | |
| SDS-PAGE and western blot equipment | BioRad | ||
| TNF-α | MedChemExpress | HY-P7058 | |
| U937 Human Monocytes | ATCC | ||
| WST-1 Cell Proliferation Assay System | TaKaRa | MK400 | |
| X-tremeGENE 9 DNA Transfection Reagent | Roche Diagnostics | 6365779001 | |
| z-VAD-FMK | APExBIO | A1902 |
참고문헌
- Gostimskaya, I. CRISPR–Cas9: A History of its discovery and ethical considerations of its use in genome editing. Biochem (Mosc.). 87 (8), 777-788 (2022).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Redman, M., King, A., Watson, C., King, D. What is CRISPR/Cas9. Arch Dis Child Educ Pract Ed. 101 (4), 213-215 (2016).
- Ebrahimi, S., et al. In vitro evaluation of CRISPR PX-LmGP63 vector effect on pathogenicity of Leishmania major as a primary step to control leishmaniasis. Microb Pathog. 161 (Pt A), 105281 (2021).
- Ebrahimi, S., Alipour, H., Azizi, K., Kalantari, M. Construction of px-lmgp63 using crispr-cas9 as primary goal for gp63 gene knockout in Leishmania major and leishmanization. Jundishapur J Microbiol. 14 (1), 112965 (2021).
- Lim, J. M., Kim, H. H. Basic principles and clinical applications of CRISPR-based genome editing. Yonsei Med J. 63 (2), 105-113 (2022).
- Han, X., et al. CRISPR-Cas9 delivery to hard-to-transfect cells via membrane deformation. Sci Adv. 1 (7), 1500454 (2015).
- Sano, S., et al. Lentiviral CRISPR/Cas9-mediated genome editing for the study of hematopoietic cells in disease models. J Vis Exp. (152), e59977 (2019).
- Tan, E., Chin, C. S. H., Lim, Z. F. S., Ng, S. K. HEK293 cell line as a platform to produce recombinant proteins and viral vectors. Front Bioeng Biotechnol. 9, 796991 (2021).
- Mccaig, W. D., et al. Hyperglycemia potentiates a shift from apoptosis to RIP1-dependent necroptosis. Cell Death Dis. , 1-14 (2018).
- Deragon, M. A., et al. Mitochondrial ROS prime the hyperglycemic shift from apoptosis to necroptosis. Cell Death Dis. 6 (1), (2020).
- Deragon, M. A., et al. Mitochondrial trafficking of MLKL, Bak/Bax, and Drp1 Is mediated by RIP1 and ROS which leads to decreased mitochondrial membrane integrity during the hyperglycemic shift to necroptosis. Int J Mol Sci. 24 (10), 8609 (2023).
- LaRocca, T. J., Sosunov, S. A., Shakerley, N. L., Ten, V. S., Ratner, A. J. Hyperglycemic conditions prime cells for RIP1-dependent necroptosis. J Biol Chem. 291 (26), 13753-13761 (2016).
- Pasparakis, M., Vandenabeele, P. Necroptosis and its role in inflammation. Nature. 517 (7534), 311-320 (2015).
- Yang, Y., et al. A Cytosolic ATM/NEMO/RIP1 complex recruits TAK1 To mediate the NF-κB and p38 Mitogen-Activated Protein Kinase (MAPK)/MAPK-activated protein 2 responses to DNA damage. Mol Cell Biol. 31 (14), 2774-2786 (2011).
- Eng, V. V., Wemyss, M. A., Pearson, J. S. The diverse roles of RIP kinases in host-pathogen interactions. Semin Cell Dev Biol. 109, 125-143 (2021).
- Liu, X., et al. RIPK1 in the inflammatory response and sepsis: Recent advances, drug discovery and beyond. Front Immunol. 14, 1114103 (2023).
- Kowolik, C. M., Yee, J. -. K. Preferential transduction of human hepatocytes with lentiviral vectors pseudotyped by Sendai virus F protein. Mol Ther. 5 (6), 762-769 (2002).
- Chong, Z. X., Yeap, S. K., Ho, W. Y. Transfection types, methods and strategies: A technical review. PeerJ. 9, 11165 (2021).
- Keller, A. A., Maeß, M. B., Schnoor, M., Scheiding, B., Lorkowski, S. Transfecting Macrophages. Methods Mol Biol (Clifton, N.J). 1784, 187-195 (2018).
- Chanput, W., Peters, V., Wichers, H. THP-1 and U937 cells. The impact of food bioactives on health: In vitro and ex vivo. models. 159 (147), (2015).
- Kutner, R. H., Zhang, X. Y., Reiser, J. Production, concentration and titration of pseudotyped HIV-1-based lentiviral vectors. Nat Protoc. 4 (4), 495-505 (2009).
- Ricks, D. M., Kutner, R., Zhang, X. Y., Welsh, D. A., Reiser, J. Optimized lentiviral transduction of mouse bone marrow-derived mesenchymal stem cells. Stem Cells Dev. 17 (3), 441-450 (2008).
- Swainson, L., Mongellaz, C., Adjali, O., Vicente, R., Taylor, N. Lentiviral transduction of immune cells. Methods Mol Biol (Clifton, N.J.). 415, 301-320 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유