Method Article
使用 CRISPR-Cas9 系统生成 RIP1 敲除 U937 细胞系
摘要
随着 CRISPR 相关方案变得越来越有用和可及,在特定的实验条件下仍然会出现并发症和障碍。该方案概述了使用 CRISPR/Cas9 创建受体相互作用的丝氨酸/苏氨酸蛋白激酶 1 (RIPK1/RIP1) 敲除人细胞系,并强调了在此过程中遇到的潜在挑战。
摘要
该方案概述了在人单核细胞 U937 细胞系中使用 CRISPR/Cas9 敲除 RIP1 基因的程序。该方法利用指定的向导 RNA 质粒和慢病毒包装质粒来实现 RIP1 基因敲除。该方案解决了对传统 CRISPR 方法的挑战和改进,使其能够在未来的细胞死亡研究中复制。所得突变细胞可用于研究细胞死亡的机制变化,否则功能性 RIP1 蛋白将发挥作用。活力测定表明,坏死性凋亡诱导后敲除细胞的细胞死亡显著减少。荧光显微镜显示,在相同条件下,敲除细胞中的线粒体活性氧 (ROS) 显着降低。这些功能测定共同证实了 RIP1 蛋白的丢失。该程序针对 U937 人单核细胞进行了优化,也可用于靶向其他关键细胞死亡调节因子,产生功能性、非致命突变体。自始至终都解决了潜在的陷阱,以提供对突变体生成过程中可能出现的挑战的见解。
引言
自发现以来,CRISPR/Cas9 基因编辑技术的使用一直在迅速发展 1,2,3。敲入或敲除细胞系或细菌内基因的能力对于推进研究和理解细胞内机制非常宝贵 1,2,3,4,5,6。CRISPR-Cas9 系统通过简化基因特异性的工程设计,改进了以前的基因编辑方法,例如转录激活因子样效应子核酸酶 (TALEN)。该程序包括两个基本组分:用于定位预期基因靶标的向导 RNA (gRNA) 和 Cas9,Cas9 是一种核酸内切酶,可通过双链 DNA 断裂修饰预期基因组位置 3,4。gRNA 将作为 Cas9 核酸内切酶的向导,通过 Watson-Crick 碱基配对定位并启动预期基因序列的双链断裂。CRISPR/Cas9 系统进行基因组编辑的整个过程包括细胞机制通过非同源末端连接 (NHEJ) 或同源重组修复 DNA 中的这些双链断裂 3,7。NHEJ 更有可能发生,从而有效地在基因组中产生突变,导致靶基因表达缺失 3,4。
商业来源已经能够创建可通过细菌生长和分离表达的 gRNA 靶标文库,这显著提高了它们的易用性。然而,CRISPR/Cas9 系统的主要局限性是难以将 gRNA 和 Cas9 复合物递送到靶细胞系中。这些限制出现在悬浮细胞系中,因为它们通常被称为难以转染8。典型的转染方法通常不能有效地将 CRISPR/Cas9 系统递送到悬浮细胞中,这就是为什么慢病毒转染和转导等病毒递送方法更适合这种类型的细胞系的原因 8,9。
这种类型的转染需要编码 gRNA 和 Cas9 核酸内切酶的慢病毒载体以及添加的慢病毒包装质粒,这些质粒被转染到能够制造慢病毒颗粒的细胞系中。该过程通常选择的细胞系是 HEK293T 细胞,因为它们更容易转染,并且在 gRNA 和 Cas9 9,10 的组装中工作非常高效。然后,这些颗粒以慢病毒的形式释放到上清液中,可用于将 gRNA 和 Cas9 转导到预期的悬浮细胞系中,例如 U937 人单核细胞。因此,与已建立的方法相比,此处描述的程序具有以下变化:(1) 难转染细胞系的替代转染方法;(2) 无需浓缩 CRISPR 质粒 DNA 或使用超速离心机;(3) 它消除了对单细胞克隆的需求。
本文的直接重点是敲除 U937 人单核细胞中的 RIP1 基因。高度炎性细胞死亡途径坏死性凋亡的经典形式受 RIP1 控制,RIP1 是细胞死亡研究的关键靶标。 11,12,13,14 当 RIP1 通过自磷酸化变得活跃时,它会募集并导致受体相互作用的丝氨酸/苏氨酸蛋白激酶 3 (RIPK3/RIP3) 和混合谱系激酶结构域样 (MLKL) 假激酶的直接磷酸化和激活,从而形成坏死小体。形成后,坏死体可以在整个细胞中自由移动,与线粒体等细胞器相互作用12,13。在线粒体中,RIP1 增强细胞代谢的正反馈回路,直接影响线粒体 ROS 的产生,进而促进 RIP1 的进一步自磷酸化、坏死体形成和坏死性凋亡的下游执行 11,12,13,14。
虽然当前研究小组的重点是 RIP1 在细胞死亡中的作用,但研究 RIP1 的其他原因包括其在炎症和感染中的作用。在被 TNF 受体等死亡受体激活后,RIP1 促进 NF-κB 信号通路的激活,从而触发促炎细胞因子、趋化因子和其他对免疫细胞募集和炎症反应放大至关重要的分子的转录15。除了 NF-κB 激活外,RIPK1 还可以参与 MAPK 信号通路,进一步增强炎症15,16。关于其在感染反应中的作用,RIP1 是宿主炎症反应的关键介质,特别是响应模式识别受体 (PRR)(如 Toll 样受体 (TLR))识别的病原体相关分子模式 (PAMP)17。此外,在脓毒症期间,RIP1 通过 TNF 受体等死亡受体的信号传导激活,导致促炎级联反应的启动。RIPK1 介导 NF-κB 和 MAPK 通路的激活,促进促炎细胞因子(如 TNF-α、IL-1β 和 IL-6)的产生,这些细胞因子是脓毒症全身炎症反应特征的关键驱动因素18。
研究方案
图 1 提供了该过程的示意图。向导 RNA (gRNA) 和靶序列如表 1 所示。材料表中列出了所用试剂和所用设备的详细信息。
1. 从大肠杆菌中收获靶向 RIP1 的 CRISPR gRNA 慢病毒表达载体,其中包含 Cas9 核酸内切酶和嘌呤霉素耐药性
- 在补充有 100 μg/mL 氨苄青霉素的LB 琼脂平板上含有慢病毒载体 gRNA 的象限条纹大肠杆菌。
- 在 37°C 下孵育板 1-2 天,直到在板最稀释的区域可以看到单个菌落。
- 选择带有无菌定量环的单个菌落,并将每个菌落单独添加到 5 mL 补充有 100 μg/mL 氨苄青霉素的 LB 肉汤中,放入 50 mL 锥形管中。确保在添加单个菌落后彻底上下移液以确保均质化。
- 放空并贴上 50 mL 锥形管的盖子,并在 37°C 和 225 rpm 的定轨振荡器中孵育 8 小时。
- 在 37°C 下孵育 8 小时期间,将 40 mL 补充有 100 μg/mL 氨苄青霉素的 LB 肉汤添加到四个单独的 50 mL 锥形管中。
- 在 37°C 下孵育 8 小时后,取 40 μL 生长的 E。 大肠杆菌培养物,并将其添加到所有四个 50 mL 锥形管中。
- 放空并贴上 50 mL 锥形管的盖子,并在 37°C 和 225 rpm 的定轨振荡器中孵育 12-16 小时。
- 在 37°C 下孵育后,在摆动桶转子中以 3220 x g 的速度离心所有试管 20 分钟,以沉淀生长的大 肠杆菌 。
- 将上清液倒入废烧杯中,然后将所有四个沉淀混合在 10 mL 补充有 100 μg/mL 氨苄青霉素的 LB 肉汤中。
- 在摆动的桶转子中以最大速度再次旋转组合的颗粒 20 分钟,以获得单个颗粒。
- 将尽可能多的上清液丢弃到废液烧杯中,然后进行以下选项之一:(1) 将湿沉淀在 -80° 下冷冻长达 1 个月。(2) 进行慢病毒载体质粒纯化。
注意:如果选择了选项 1,则协议可能会在此处暂停。
2. 用靶向 RIP1 的纯化 CRISPR gRNA 慢病毒表达载体转染 HEK293T 细胞
- 将浓度为 3 ×10 6 至 5 × 106 个细胞的 HEK293T 细胞接种过夜,接种到含有 4.5 g/L D-葡萄糖、L-谷氨酰胺、110 mg/L 丙酮酸钠和 10% 热灭活 FBS 的 DMEM 的 10 cm 板中。
- 第二天,确保平板具有大约 70%-90% 的汇合度,然后继续执行方案。
- 使用转染试剂以 3:1 的比例(3 份试剂:1 份质粒 DNA)和还原血清培养基,以 1:1:1:1 比例的质粒(实验 CRISPR 质粒:pLP1:pLP2:pLP/VSVG)设置细胞转染。
注:该比率的 pLP1:pLP2:pLP/VSVG 部分是慢病毒包装混合物。- 在设置转染之前,将转染试剂、减量血清培养基、CRISPR 质粒 DNA 和慢病毒包装混合物平衡至室温。
- 将 75 μL 转染试剂、2500 μL 减血清培养基和 25 μg 总 DNA 混合在一起(根据 CRISPR 质粒 DNA 的分光光度计分析和 1:1:1:1 的比例与慢病毒包装混合物混合计算)。
- 让这种混合物在室温下孵育 15-30 分钟。小心地将该混合物的全部体积移液到汇合HEK293T细胞板中,直接移液到培养基中(无需更换培养基)。
- 轻轻旋转板,以确保转染混合物和板培养基充分匀浆。将板在 37 °C 下孵育 48 小时。
3. U937 细胞或靶细胞的慢病毒转导
- 在 37°C 下完成 48 小时孵育后,将培养基从生产HEK293T细胞板转移到 15 mL 锥形管中。
- 在室温下以 800 x g 离心体积 5 分钟,以沉淀任何剩余的 HEK293T 细胞。去除所有含病毒的上清液,注意不要干扰沉淀的 HEK293T 细胞,并将其保存在单独的 15 mL 锥形管中。然后,对含颗粒的试管进行消毒并丢弃在适当的生物危害废物容器中。
注:基于 HIV 的慢病毒上清液可储存在 -80°C 下;然而,这样做会在第一次冻融循环后病毒稳定性损失高达55% 19。 - 通过在室温下以 400 x g 离心适当体积 10 分钟,在 15 mL 锥形管中计数并获得 2 ×6 个 U937 细胞沉淀,然后倒出上清液,将细胞沉淀留在管中。
- 用所有含病毒的上清液重悬 U937 细胞沉淀,然后以 290 x g 离心管 60 分钟。
- 离心后,使用移液管用管中已有的含病毒的上清液重悬沉淀。
- 将管子放在端到端旋转器(或类似的旋转装置)上 60 分钟。
- 孵育后,在室温下以 400 x g 离心试管 10 分钟以沉淀细胞。
- 用含病毒的上清液和补充有 10% 热灭活 FBS 的完全 RPMI-1640 培养基的 1:1 比例培养基混合物重悬细胞沉淀。
- 将细胞混合物转移到 10 cm 组织培养板中,并在 37°C 下孵育 48 小时。
- 孵育后,将 U937 细胞以 400 x g 离心 10 分钟并去除上清液。
- 用补充有 10% 热灭活 FBS 和 5 μg/mL 嘌呤霉素的完全 RPMI-1640 培养基重悬沉淀,并将细胞转移到 T25 培养瓶中。
- 将细胞在 37 °C 下原封不动地孵育 2-3 周,确保每 1-2 天检查一次细胞生长迹象。
注:一旦细胞在初始传代后汇合并具有健康的周转时间,就可以测试它们完成方案的有效性。
4. 使用 Western blot 分析测试已完成方案在创建 RIP1 CRISPR 突变细胞中的有效性
- 生成野生型 (WT) U937 单核细胞、非靶向对照 (NTC) CRISPR 细胞和 RIP1 CRISPR 突变细胞的细胞蛋白裂解物 11,12,13。
- 在 SDS-PAGE 凝胶上运行这些裂解物,然后进行 Western 印迹分析14。
- 首先将样品标准化为管家蛋白。
- 标准化后,分析样品的 RIP1 蛋白表达水平,以充分确定创建 RIP1 CRISPR 突变细胞系的成功14。
- 在蛋白质印迹分析构象之后,细胞可用于实验目的。
结果
在产生 RIP1 CRISPR 突变体 U937 细胞的汇合群体后,进行 SDS-PAGE 和 Western blot 分析。Western blot 分析用于通过评估 RIP1 蛋白表达水平的损失来确定 RIP1 CRISPR 突变细胞系的成功创建。该确定是基于 WT U937 单核细胞和 NTC 细胞的比较结果做出的。 在图 2 中,未检测到 5 μg/mL 嘌呤霉素 RIP1 CRISPR 突变细胞的 RIP1 表达,但在 WT U937 和 NTC 细胞中均检测到 RIP1 的表达。该结果表明,该方案是选择可行的 RIP1 CRISPR 突变体的可靠且有效的方法。
随着 RIP1 CRISPR 突变细胞中 RIP1 蛋白表达的成功缺失,我们随后确定了这是否也等同于 RIP1 的功能缺失。在先前建立的研究中,肿瘤坏死因子-α (TNF-α)、放线菌酮和泛半胱天冬酶抑制剂 zVAD-fmk 的组合是坏死性凋亡的直接刺激,缩写为 TCZ11,14。如图 3 所示,在 WT U937 单核细胞和 NTC 细胞系中,用已知的 RIP1 抑制剂坏死抑制素-1 (nec-1s) 处理后,TCZ 处理诱导的坏死性细胞死亡的诱导恢复。然而,对于 RIP1 CRISPR 突变细胞,与 WT 和 NTC 细胞相比,TCZ 处理导致细胞死亡的显着损失。随后用 nec-1s 处理对 RIP1 CRISPR 突变细胞没有影响,表明不存在坏死性凋亡和 RIP1 功能。
为了进一步评估这些 RIP1 CRISPR 突变细胞中 RIP1 功能的丧失,使用 DNA Hoecsht 染色(蓝色荧光)和线粒体超氧化物染色(红色荧光)进行活细胞荧光显微镜检查。这些实验使我们能够确定先前确定的坏死性凋亡期间线粒体超氧化物产生的增加是否在这些细胞中丢失。已知 RIP1 直接与细胞代谢相互作用并显着增加线粒体超氧化物的产生。细胞内这种丰富的超氧化物反过来又随着 RIP1 的激活和坏死性凋亡的下游执行而形成一个正反馈回路。如图 4 所示,TCZ 处理显示 WT U937 单核细胞和 NTC 细胞中都有丰富的线粒体超氧化物产生,但在 RIP1 CRISPR 突变细胞中没有。随后用 nec-1s 处理导致 WT U937 单核细胞和 NTC 细胞的红色荧光染色显着降低,在 RIP1 CRISPR 突变细胞中未观察到变化。

图 1:CRISPR/Cas9 协议的示意图模型。 此协议中提供的各种步骤的可视化表示,以便于理解。 请单击此处查看此图的较大版本。
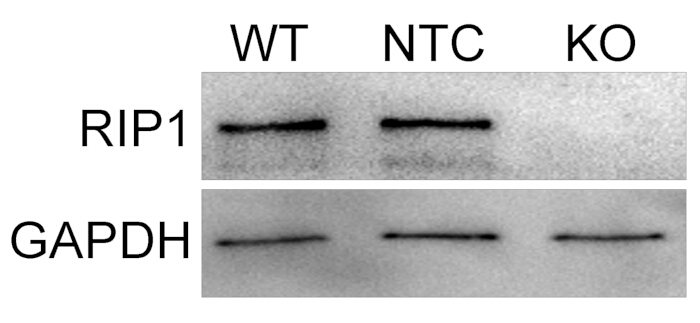
图 2:Western blot 分析证实 RIP1 CRISPR 突变细胞中 RIP1 蛋白表达缺失 。 与 WT U937 细胞和 NTC 细胞相比, RIP1 CRISPR 突变型 U937 细胞中 RIP1 蛋白在 5 μg/mL 嘌呤霉素选择中丢失。 请单击此处查看此图的较大版本。

图 3:细胞活力测定显示 RIP1 CRISPR 突变细胞中 RIP1 的功能丧失。通过 TCZ 处理诱导坏死性细胞凋亡,并随后用 nec-1s 处理进行分析。WT U937 单核细胞和 NTC 细胞在用 nec-1s 处理后均显示细胞死亡显着减少。RIP1 CRISPR 突变细胞在用 TCZ 处理时表现出更少的细胞死亡,并且在随后用 nec-1s 处理时没有变化。显示的结果来自 3 个独立实验。误差线表示标准差。Bonferroni 后测 ***p 的双向方差分析< 0.001。ns = 无统计学意义。请单击此处查看此图的较大版本。

图 4:使用荧光显微镜对 RIP1 CRISPR 突变细胞中的 RIP1 进行下游功能分析。(A) 线粒体超氧化物染色(红色荧光)的测量显示,NEC-1s 处理后,WT U937 单核细胞和 NTC 细胞中的超氧化物产生减少。RIP1 CRISPR 突变细胞在用 TCZ 处理时未显示线粒体超氧化物的产生,随后用 nec-1s 处理时未显示变化。比例尺:100 μm。(B) (A) 中表示的结果的平均荧光强度定量。显示的结果来自 3 个独立实验。误差线表示标准差。Bonferroni 后测的双向方差分析,***p < 0.001。请单击此处查看此图的较大版本。
| 基因靶标 | 基因 ID | gRNA | 基因中的相应核苷酸 | PAM 系列 | Cas9 切割点 | ||
| RIPK1 | 8737 | GCTTTGCGTTGACGTCATTC | 88-841 | AGG | 816 | ||
表 1:向导 RNA (gRNA) 和靶序列。
讨论
该协议旨在提供慢病毒转染和转导效率和可靠性潜在陷阱的详细说明和分析,以创建 RIP1 敲除 U937 细胞系。虽然这种转染和转导方法费力且耗时,但通常认为是将所选 gRNA 和 Cas9 核酸内切酶掺入难转染细胞系的有效方法 8,9,20。与 U937 细胞和其他免疫细胞等单核细胞一样,进行转染存在固有的困难,因为它们具有识别并响应外源核酸的先天传感器。这可能导致核酸破坏/超突变、转录抑制和/或响应外源核酸的细胞死亡21。
这种递送方法只能使用能够制造慢病毒颗粒(HEK293T细胞)的细胞系,慢病毒颗粒是一种贴壁人胚胎肾细胞系,被广泛认为易于转染 9,10。有了这些信息,概述了此程序中最关键的步骤,必须小心处理。细胞培养和监测至关重要。本研究使用 U937 细胞作为 gRNA 和 Cas9 的靶细胞系,它们是快速生长且易于使用的悬浮人单核细胞22。与大多数快速生长的细胞一样,细胞的定期维护和传代对于防止它们过度生长和无法按预期进行实验非常重要。相同的概念适用于本协议中的 HEK293T 细胞。确保这些细胞系在方案中使用期间得到适当维护并保持健康、融合状态对于高效的慢病毒生产至关重要。该程序的另一个关键步骤是使用适当浓度的转染试剂以及 CRISPR 质粒 DNA 与慢病毒包装混合物的比例。在该方法中,已在几种不同浓度下对这些数学比例进行了经验测试,列出的值对于HEK293T细胞的转染至关重要。
与大多数设计的方法和程序一样,在已建立的工作实验过程中需要进行相当多的故障排除。这里通过其他公认的方案确定的一个问题是嘌呤霉素在 U937 细胞的较短转导步骤后的直接挑战。由于转导步骤仅使用 2 × 10个 6 个 U937 细胞,因此根据经验确定,在一半培养基完全 RPMI-1640 补充有 10% 热灭活 FBS 的情况下孵育整整 48 小时,以促进这种 RIP1 CRISPR 突变细胞系的产生。当细胞在没有干预的情况下连续用 5 μg/mL 嘌呤霉素攻击时,活的和健康的 U937 细胞在 2-3 周后开始出现在培养物中。由于该方案非常耗时,因此该转染和转导步骤的多次迭代具有几种可能的可回收培养物。一旦培养物汇合,就可以通过 SDS-PAGE 和 Western blotting 技术检查方案的有效性。即使在成功分离和确认 RIP1 CRISPR 突变细胞系后,最好对基于蛋白质裂解物的 SDS-PAGE 进行常规监测和蛋白质印迹分析,以发现 RIP1 蛋白表达的细胞丢失,以便在实验中继续使用这些细胞。
经验性故障排除的另一个原因是缺乏可用于免疫细胞系的转染试剂的直接信息。在来自公司和方案的可用信息之间,关于本方案中使用的转染试剂与 DNA 的基于浓度的比率的信息存在冲突。虽然尝试范围分别为 5:1 至 3:1,但它确定 3:1 是要使用的最大比例,否则,HEK293T细胞无法高效完成转染。除了这种选择,还确定了较低体积的 CRISPR 质粒 DNA 的单一迭代不足以完全转染 10 cm 板的 HEK293T 细胞。由于质粒纯化试剂盒从大肠杆菌中产生大量 CRISPR 质粒 DNA,增加了所有转染组分的体积,因此 5 倍的转染效率更高,可在转导后回收 U937 细胞。协议中为这些组件列出的体积代表了与接受协议的单次迭代相比增加了 5 倍。在转染试剂比例和用于这些组分的总体积的这些变化之间,设计方案时无需浓缩 CRISPR 质粒 DNA 或需要使用超速离心机。然而,如果这些材料可用,可以使用既定方法浓缩分离的 CRISPR 质粒 DNA,从而减少该方案中其他组分的所需体积23,24。
当使用慢病毒转染方法时,可以注意到它们在 CRISPR/Cas9 基因编辑中的应用存在局限性。最显着的限制是该协议的时间和劳动力要求 9,20。该程序仅包括两个可以暂停的步骤,但针对难以转染的细胞系进行了优化。对于难以转染的细胞系(如 U937 单核细胞),典型的转染方法(包括化学介导的质粒引入)不如基于慢病毒的递送有效25。
更广泛的研究兴趣集中在 U937 单核细胞从细胞凋亡到坏死性凋亡的高血糖转变,强调了在此过程中 RIP1 功能敲除对于机制理解的必要性。由于 RIP1 是坏死性凋亡执行的关键蛋白,因此了解其所有功能和细胞内的蛋白质相互作用至关重要 11,12,13,14。该方案的未来方向包括将这些方法应用于有关从细胞凋亡到坏死性凋亡的高血糖转变的进一步调查和实验。预计该方案将广泛适用于使用 CRISPR/Cas9 系统对悬浮细胞进行各种 gRNA 靶标的慢病毒转染和转导方法,超越 U937 细胞和 RIP1 的功能性敲除。
披露声明
没有。
致谢
这项研究由美国国立卫生研究院 (NIH) 的国家心肺血液研究所 (NHLBI) 资助,批准号 NIH 2R15-HL135675-02 给 TJ。
材料
| Name | Company | Catalog Number | Comments |
| Adjusted DMEM Medium | Gibco | 11995-040 | |
| Ampicillin | Sigma | A1593 | |
| bisBenzimide Hoechst 33342 trihydrochloride | Sigma | B2261 | |
| Complete RPMI-1640 Medium | Sigma | R6504 | |
| CRISPR NTC gRNA E.coli Strain | transOMIC | TELA1011 | |
| CRISPR RIP1 gRNA E.coli Strain | transOMIC | TEVH-1162203 | |
| End-over-end Rotator | Thermo Scientific | ||
| EVOS FL Fluorescence Microscope | Life Technologies | ||
| GenElute Plasmid Maxiprep Kit | Sigma | PLX15 | |
| Goat Anti-Rabbit IgG Antibody, (H+L) HRP conjugate | Sigma | AP307P | |
| HEK293T Cells | ATCC | ||
| Incubator Shaker | New Brunswick Scientific | ||
| LB Agar | BD | 244520 | |
| LB Broth | BD | 244610 | |
| LV-MAX Lentiviral Packaging Mix | Gibco | A43237 | |
| MitoSOX Red | MedChemExpress | HY-D1055 | |
| NanoDrop Spectrophotometer | Thermo Scientific | ||
| Necrostatin-1 | MedChemExpress | HY-14622A | |
| OPTI-MEM | Gibco | 31985-062 | |
| Puromycin | Sigma | P7255 | |
| Rabbit anti-human RIP1 mAb | Cell Signaling Technology | 3493 | |
| SDS-PAGE and western blot equipment | BioRad | ||
| TNF-α | MedChemExpress | HY-P7058 | |
| U937 Human Monocytes | ATCC | ||
| WST-1 Cell Proliferation Assay System | TaKaRa | MK400 | |
| X-tremeGENE 9 DNA Transfection Reagent | Roche Diagnostics | 6365779001 | |
| z-VAD-FMK | APExBIO | A1902 |
参考文献
- Gostimskaya, I. CRISPR–Cas9: A History of its discovery and ethical considerations of its use in genome editing. Biochem (Mosc.). 87 (8), 777-788 (2022).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Redman, M., King, A., Watson, C., King, D. What is CRISPR/Cas9. Arch Dis Child Educ Pract Ed. 101 (4), 213-215 (2016).
- Ebrahimi, S., et al. In vitro evaluation of CRISPR PX-LmGP63 vector effect on pathogenicity of Leishmania major as a primary step to control leishmaniasis. Microb Pathog. 161 (Pt A), 105281 (2021).
- Ebrahimi, S., Alipour, H., Azizi, K., Kalantari, M. Construction of px-lmgp63 using crispr-cas9 as primary goal for gp63 gene knockout in Leishmania major and leishmanization. Jundishapur J Microbiol. 14 (1), 112965 (2021).
- Lim, J. M., Kim, H. H. Basic principles and clinical applications of CRISPR-based genome editing. Yonsei Med J. 63 (2), 105-113 (2022).
- Han, X., et al. CRISPR-Cas9 delivery to hard-to-transfect cells via membrane deformation. Sci Adv. 1 (7), 1500454 (2015).
- Sano, S., et al. Lentiviral CRISPR/Cas9-mediated genome editing for the study of hematopoietic cells in disease models. J Vis Exp. (152), e59977 (2019).
- Tan, E., Chin, C. S. H., Lim, Z. F. S., Ng, S. K. HEK293 cell line as a platform to produce recombinant proteins and viral vectors. Front Bioeng Biotechnol. 9, 796991 (2021).
- Mccaig, W. D., et al. Hyperglycemia potentiates a shift from apoptosis to RIP1-dependent necroptosis. Cell Death Dis. , 1-14 (2018).
- Deragon, M. A., et al. Mitochondrial ROS prime the hyperglycemic shift from apoptosis to necroptosis. Cell Death Dis. 6 (1), (2020).
- Deragon, M. A., et al. Mitochondrial trafficking of MLKL, Bak/Bax, and Drp1 Is mediated by RIP1 and ROS which leads to decreased mitochondrial membrane integrity during the hyperglycemic shift to necroptosis. Int J Mol Sci. 24 (10), 8609 (2023).
- LaRocca, T. J., Sosunov, S. A., Shakerley, N. L., Ten, V. S., Ratner, A. J. Hyperglycemic conditions prime cells for RIP1-dependent necroptosis. J Biol Chem. 291 (26), 13753-13761 (2016).
- Pasparakis, M., Vandenabeele, P. Necroptosis and its role in inflammation. Nature. 517 (7534), 311-320 (2015).
- Yang, Y., et al. A Cytosolic ATM/NEMO/RIP1 complex recruits TAK1 To mediate the NF-κB and p38 Mitogen-Activated Protein Kinase (MAPK)/MAPK-activated protein 2 responses to DNA damage. Mol Cell Biol. 31 (14), 2774-2786 (2011).
- Eng, V. V., Wemyss, M. A., Pearson, J. S. The diverse roles of RIP kinases in host-pathogen interactions. Semin Cell Dev Biol. 109, 125-143 (2021).
- Liu, X., et al. RIPK1 in the inflammatory response and sepsis: Recent advances, drug discovery and beyond. Front Immunol. 14, 1114103 (2023).
- Kowolik, C. M., Yee, J. -. K. Preferential transduction of human hepatocytes with lentiviral vectors pseudotyped by Sendai virus F protein. Mol Ther. 5 (6), 762-769 (2002).
- Chong, Z. X., Yeap, S. K., Ho, W. Y. Transfection types, methods and strategies: A technical review. PeerJ. 9, 11165 (2021).
- Keller, A. A., Maeß, M. B., Schnoor, M., Scheiding, B., Lorkowski, S. Transfecting Macrophages. Methods Mol Biol (Clifton, N.J). 1784, 187-195 (2018).
- Chanput, W., Peters, V., Wichers, H. THP-1 and U937 cells. The impact of food bioactives on health: In vitro and ex vivo. models. 159 (147), (2015).
- Kutner, R. H., Zhang, X. Y., Reiser, J. Production, concentration and titration of pseudotyped HIV-1-based lentiviral vectors. Nat Protoc. 4 (4), 495-505 (2009).
- Ricks, D. M., Kutner, R., Zhang, X. Y., Welsh, D. A., Reiser, J. Optimized lentiviral transduction of mouse bone marrow-derived mesenchymal stem cells. Stem Cells Dev. 17 (3), 441-450 (2008).
- Swainson, L., Mongellaz, C., Adjali, O., Vicente, R., Taylor, N. Lentiviral transduction of immune cells. Methods Mol Biol (Clifton, N.J.). 415, 301-320 (2008).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。