Method Article
Analisi quantitativa della stabilizzazione della membrana del reticolo endoplasmatico (MAM) associata ai mitocondri in un modello neurale della malattia di Alzheimer (AD)
In questo articolo
Riepilogo
Qui, descriviamo la misurazione della velocità di trasporto assonale degli stabilizzatori costitutivi delle membrane del reticolo endoplasmatico (ER) associate ai mitocondri (MAM) aumentando o mantenendo la generazione neurotossica di β-amiloide (Aβ) dai neuroni della malattia di Alzheimer (AD) in tempo reale per fungere da metrica diretta e quantitativa per misurare la stabilizzazione MAM e aiutare lo sviluppo di terapie per l'AD.
Abstract
Qui viene presentato un metodo per quantificare la stabilizzazione delle membrane del reticolo endoplasmatico associate ai mitocondri (MAM) in un modello neurale tridimensionale (3D) della malattia di Alzheimer (AD). Per cominciare, le cellule ReN neuroprogenitrici umane fresche che esprimono la proteina precursore dell'amiloide-β (APP) contenente la malattia di Alzheimer familiare (FAD) o le cellule ReN naive vengono coltivate in piastre di coltura tissutale sottili (1:100) rivestite di Matrigel. Dopo che le cellule hanno raggiunto la confluenza, queste vengono elettroporate con plasmidi di espressione che codificano la sequenza di legame dei mitocondri coniugata con la proteina di fluorescenza rossa (RFP) di AKAP1(34-63) (Mito-RFP) che rileva i mitocondri o gli stabilizzatori costitutivi MAM MAM 1X o MAM 9X che stabilizzano rispettivamente MAM stretti (6 nm ± 1 nm di larghezza del gap) o sciolti (24 nm ± 3 nm di larghezza del gap). Dopo 16-24 ore, le cellule vengono raccolte e arricchite da un selezionatore cellulare attivato dalla fluorescenza (FACS). Un numero uguale di cellule arricchite con FACS viene seminato nella matrice tridimensionale (Matrigel 1:1) e lasciato differenziare in neuroni maturi per 10 giorni. Le immagini delle cellule differenziate a 10 giorni che esprimono gli stabilizzatori MAM coniugati con RFP vengono acquisite al microscopio a fluorescenza dotato di una camera di coltura per l'imaging di cellule vive che mantiene la CO2 (5%), la temperatura (37 °C) e l'umidità (~90%). A tal fine, abbiamo eseguito l'imaging di cellule vive e analisi chimografiche per misurare la motilità dei mitocondri liberi marcati con Mito-RFP o mitocondri legati all'ER di larghezze di gap strette o larghe stabilizzate da MAM 1X o MAM 9X, rispettivamente, nel processo neuronale più esteso di ciascun neurone ReN GA che è lungo almeno 500 nm, considerandoli come assoni.
Introduzione
Prove emergenti suggeriscono che i contatti specializzati del reticolo endoplasmatico associati ai mitocondri (MERC), raccolti biochimicamente come membrane ER associate ai mitocondri, spesso indicati come MAM 1,2, svolgono un ruolo in diverse malattie neurodegenerative, tra cui AD 3,4. Questi MAM sono composti da microdomini lipidici ricchi di colesterolo nell'ER e nella membrana esterna dei mitocondri legati da una serie di proteine che creano diversità strutturali e funzionali tra i MAM 5,6,7. L'ipotesi MAM, coniata di recente, postula che l'aumento dei MAM porti a un aumento della produzione di Aβ e alla cascata patogena dell'AD, tra cui la formazione di grovigli neurofibrillari (NFT), la disomeostasi del calcio e la neuroinfiammazione 3,8. Circa il 5%-20% dei mitocondri entra in contatto fisico con l'ER per formare le MAM9. La larghezza della fessura delle MAM è determinata dall'ER liscio e ruvido (sER e rER, rispettivamente). La larghezza variabile del gap tra i mitocondri sER (10-50 nm) e i mitocondri rER (50-80 nm) suggerisce che l'ampiezza del gap dei MAM ha un lungo spettro che varia da stretto (~10 nm) a sciolto (~80 nm)10,11,12,13. L'ampiezza dello spazio MAM determina le funzioni MAM, come l'omeostasi del calcio e il trasporto dei lipidi 1,14. Un recente rapporto ha dimostrato che le MAM formate tra ER strettamente connessi (~10 nm) e mitocondri, chiamate MAM complete, sono apoptotiche. Al contrario, le MAM formate tra ER debolmente connesse (~25 nm) e mitocondri, denominate MAM difettose o medie, sono anti-apoptotiche 14,15,16. La stabilizzazione delle MAM con una larghezza di gap di 6 nm ± 1 nm ha aumentato la generazione di Aβ da un nuovo modello di coltura neurale tridimensionale (3D) di AD. Al contrario, la stabilizzazione delle MAM con una larghezza di gap di 24 nm ± 3 nm non ha alcun effetto sulla generazioneAβ 17. Questa scoperta suggerisce per la prima volta che la regolazione del grado di stabilizzazione dei MAM, ma non la destabilizzazione dei MAM, è la chiave per regolare la generazione di Aβ. Un tentativo di destabilizzare completamente i MAM può avere conseguenze indesiderate perché i MAM mantengono diversi eventi cellulari critici per la sopravvivenza cellulare12.
La modulazione dei MAM è un'area di ricerca emergente con potenziali implicazioni per vari disturbi, tra cui il cancro, i disturbi metabolici e le malattie neurodegenerative18. Nonostante la disponibilità di molti modulatori MAM, finora non è stato fatto alcun tentativo importante per testare le loro capacità di destabilizzare i MAM e abbassare la patologia AD, principalmente perché le diversità strutturali dei MAM li rendono un sistema altamente complesso da mirare per la scoperta di farmaci. Tuttavia, la farmacologia dei sistemi strutturali di recente sviluppo, che considera le proprietà specifiche dei bersagli farmacologici e del loro ambiente18,19, dovrebbe superare le difficoltà e sviluppare farmaci altamente potenti che prendono di mira i MAM o le proteine associate ai MAM nell'AD. Tuttavia, la ricerca di un modulatore efficace della stabilizzazione MAM richiede metodi per quantificare con precisione il grado di stabilizzazione MAM. Le tecniche tradizionali come la microscopia elettronica (EM) o la microscopia a super-risoluzione hanno limitazioni nel determinare la stabilizzazione MAM. Il superamento di queste sfide richiederebbe probabilmente lo sviluppo di nuove tecniche di imaging più dinamiche o di saggi biochimici in grado di fornire misure quantitative della stabilizzazione MAM nelle cellule viventi. La microscopia elettronica a scansione a fascio ionico focalizzato (FIB-SEM) dei neuroni primari ha rivelato che l'ER tende a formare una rete attorno ai mitocondri che probabilmente limita la motilità mitocondriale20,21. L'interruzione dei sistemi di trasporto mitocondriale, retrogrado, anterogrado o entrambi, ha avuto un profondo impatto sulla funzione sinaptica e neuronale22. Pertanto, il nuovo imaging di cellule vive e l'analisi basata sulla chimografia della velocità assonale dei mitocondri legati all'ER qui descritti come metrica per misurare quantitativamente la stabilizzazione MAM faciliteranno l'identificazione di modulatori MAM in grado di commutare la soglia di stabilizzazione MAM in una che mantiene o possibilmente abbassa invece di aumentare la generazione di Aβ.
Protocollo
Modelli di cultura neurale AD: Questo studio ha utilizzato neuroni derivati da cellule ReN progenitrici neurali umane [ReN naive (Millipore)] o cellule ReN che esprimono mutazioni AD familiari (fAD) nel gene della proteina precursore dell'amiloide (APP) (APPSwe / Lon), cellule ReN GA. Il sistema di coltura tridimensionale (3D) ReN-GA ricapitola la patologia dell'AD, in particolare i grovigli neurofibrillari (NFT) guidati da oligomeri Aβ 23,24. Le cellule Naive ReN sono disponibili in commercio. Le linee ReN GA sono state ottenute dal Dr. Doo Y. Kim, Professore Associato, Massachusetts General Hospital (MGH)23,24,25.
Plasmidi di espressione: AKP1 (34-63) e la sequenza ER bersaglio delle proteine Ubc 6 (283-303) si legano direttamente con RFP (Mito-RFP-ER indicato come MAM 1X) o contengono un linker di 9 aminoacidi (Mito-9X-RFP-ER indicato come MAM 9X) progettato per stabilizzare MAM di 6 nm ± 1 nm o 24 nm + 3 nm di gap larghezze, rispettivamente15,26 (Figura 1A).
1. Elettroporazione
- Trasfezione delle cellule con stabilizzatori MAM (1-2 h)
NOTA: Seguire il protocollo descritto dal produttore del kit del nucleolettore (Tabella dei materiali). Prima di iniziare, preparare piastre di coltura a 6 pozzetti con fondo di vetro pre-rivestite con DMEM/F12 contenente l'1% di Matrigel (d'ora in poi denominato matrice a membrana basale [BMM]). Utilizzare 2 ml di BMM per ricoprire ogni pozzetto. Incubare per almeno 1 ora a 37 °C. BMM, terreno, puntali per pipette e pipette devono essere preraffreddati prima della miscelazione per evitare che il BMM si solidifichi.- Aspirare la miscela BMM. Sostituire con 2 mL di DMEM/F12 a temperatura ambiente (RT) in ciascun pozzetto.
- Combinare Nucleofector con l'integratore 1 in un rapporto 4,5:1 (82 μL:18 μL di rapporto Nucleofector/Integratore per 100 μL di soluzione). Per ogni trasfezione desiderata, sono necessari 100 μL di Nucleofector.
- DNA a vortice. Quindi, aggiungere 1-5 μg di DNA (per trattamento) in provette da microcentrifuga da 1 ml per ogni trasfezione.
- Usa l'acustasi per raccogliere una piastra di cellule ReN-GA sane.
NOTA: Piastra le cellule nel terreno di espansione (Tabella 1) in piastre di coltura tissutale da 100 mm e attendi che la confluenza raggiunga il 70-80%.- Aspirare il terreno esistente e lavare una volta con 10 ml di PBS.
- Aggiungere 1 mL di acutasi direttamente alle cellule e incubare per 5-10 minuti a 37 °C.
- Staccare le cellule dal piatto picchiettando delicatamente il lato del piatto. Controlla al microscopio per assicurarti che le cellule siano allentate e scorrano liberamente.
- Neutralizzare l'acutasi con 10 mL di DMEM/F12 e trasferire in una provetta pulita da 15 mL.
- Centrifugare il numero richiesto di cellule a 274 x g per 5 minuti e aspirare il surnatante per rimuovere le cellule morte. Quindi, risospendere il pellet in 10 mL di DMEM/F12.
- Conta le celle per determinare la densità.
NOTA: In questo studio, è stato utilizzato un contatore automatico di celle per contare il numero di cellule. Per elettroporazione, sono necessarie 3-5 X 106 celle (ad esempio, 5 trattamenti richiederebbero 15-25 X 106 cellule).- Prelevare 10 μl di celle sospese e aggiungerle al lato A di una camera di conteggio delle cellule. Quindi aggiungere 10 μl al lato B.
NOTA: Aggiungere la sospensione cellulare inclinando la pipetta lateralmente. Evitare la formazione di bolle evitando di spingere oltre il primo arresto sullo stantuffo della pipetta. - Portare la camera di conteggio piena al contacelle e inserire il lato A nella fessura principale sulla parte anteriore.
- Dopo aver premuto Misura, annotare il numero di celle per ml.
- Estrarre la camera, capovolgerla sul lato B e ripetere i passaggi 1.1.5.2-1.1.5.3.
- Somma questi due numeri, quindi dividi per 4 (se non usi il blu di tripano), quindi moltiplica il numero per i millilitri di liquido in cui sono sospese le celle per ottenere il numero totale di celle nella sospensione.
- Prelevare 10 μl di celle sospese e aggiungerle al lato A di una camera di conteggio delle cellule. Quindi aggiungere 10 μl al lato B.
- Centrifugare il numero richiesto di cellule a 274 x g per 5 minuti e aspirare il surnatante.
- Risospendere il pellet in 100 μL di miscela di nucleofector per elettroporazione. (ad esempio, 500 μl per 5 trattamenti).
NOTA: Lasciare le cellule nella soluzione di Nucleofector per più di 15 minuti potrebbe ridurre la vitalità cellulare e l'efficacia complessiva. - Aggiungere 100 μl di sospensione cellulare a una delle provette contenenti DNA e mescolare mediante pipettaggio.
- Trasferire la miscela di DNA in una cuvetta per elettroporazione e sigillare con il coperchio in dotazione. Per evitare la formazione di bolle, inclinare la cuvetta verso il basso e pipettare lentamente.
- Selezionare il programma Nucleofector appropriato per il dispositivo in uso. Per il dispositivo utilizzato in questo studio, utilizzare il programma A-033 per la trasfezione. Per ottimizzare, prova tutti e 5 i programmi Nucleofector per determinare quello più appropriato per ogni tipo di cellula.
NOTA: Una conferma di successo dell'elettroporazione è la schiuma visibile nella parte superiore della miscela. - Immediatamente, utilizzando le pipette sterili fornite, aggiungere ~500 μL di DMEM dalla piastra preriempita a 6 pozzetti nella cuvetta. Mescolare delicatamente una volta e poi trasferire le celle elettroporate e il terreno nel pozzetto corrispondente.
NOTA: Dopo l'elettroporazione, le cellule sono estremamente sensibili, quindi è essenziale trasferire rapidamente il terreno e pipettare con cura. - Ripetere i passaggi 1.1.8-1.1.11 per tutti i restanti trattamenti del DNA.
- Incubare le cellule a 37 °C in presenza di CO2 (5%) per una notte.
- Il giorno successivo, scambiare i terreni con i mezzi di differenziazione freschi (Tabella 2) e lasciare che le cellule si differenzino per 10 giorni. Ogni 2-3 giorni, sostituire con un nuovo supporto.
2. Imaging di cellule vive
- Preparazione delle cellule per la microscopia su cellule vive (30 min)
- Preparazione della camera delle cellule vive (prima di spostare le cellule nella camera)
- Assicurarsi che il serbatoio di CO2 e l'umidificatore siano collegati alla camera, che le valvole siano aperte e che i serbatoi siano pieni.
- Impostare la temperatura a 37 °C, la CO2 al 5% e l'umidità al 95%. (Questo potrebbe richiedere del tempo prima che i livelli si equilibrino).
- Posizionare una piastra a 6 pozzetti contenente le cellule nella camera e regolare la messa a fuoco del microscopio fino a quando le cellule non diventano visibili.
- Accendere il laser (Tabella dei materiali).
- Per catturare il segnale RFP, eccitare il fluoroforo utilizzando un laser a 594 nm e utilizzare un'emissione di 570-640 nm. Per la GFP, utilizzare un laser a 488 nm per l'eccitazione e l'emissione a 510-540 nm.
- Utilizzando il filtro fluorescente incorporato, regolare l'intensità del segnale fino a quando il segnale di fondo non si è dissipato (dovrebbe essere quasi nero fisso).
- Acquisizione di video in diretta di assoni (10 h-15 h in totale)
NOTA: Un microscopio confocale invertito Nikon C2 Eclipse Ti2 è stato utilizzato per acquisire immagini fluorescenti utilizzando il software NIS Element AR. Utilizza l'ingrandimento 60x con una risoluzione di 512 pixel, riprendendo video a 1 fotogramma al secondo per 3 minuti di chimografi più puliti prodotti.- Trova una cellula che esprime biosensori RFP. Esporta un'immagine della cella per riferimento successivo prima di registrare un video.
- Ritagliare l'area di scansione in modo che si adatti all'assone. L'utilizzo di un'area di scansione più piccola riduce i tempi di elaborazione del microscopio e rende la generazione della chimografia molto più semplice.
- Spegni tutti i laser per aiutare il software a funzionare in modo più fluido. Si prega di notare che quando il programma viene eseguito con tutti i laser attivi, non tutti i fotogrammi vengono acquisiti.
NOTA: Il segnale fluorescente rosso dal soma è molto più luminoso che nell'assone. Pertanto, il soma viene escluso per aumentare l'intensità complessiva del segnale nell'assone. Passare alla scheda Misurazione tempo . - Impostare l'intervallo su 1 fotogramma al secondo e impostare il tempo complessivo su 181 s.
- Fare clic su Esegui.
- Salva questo file come file .nds (correttamente etichettato) e ripeti il processo ~10 volte per stabilizzatori MAM (MAM 1X o MAM 9X).
NOTA: La potenza del laser a volte produceva un notevole effetto di sbiancamento sul segnale RFP se scansionato troppo a lungo. Lavorare rapidamente per catturare video è importante da considerare.
3. Post-elaborazione (7 giorni)
NOTA: Per analizzare il trasporto e generare chimografi, sono state utilizzate le macro Fiji ImageJ. Le vescicole che si muovevano meno di 0,1 mm/s sono state classificate come stazionarie. La frequenza di movimento delle particelle è stata calcolata dividendo il numero di particelle che si muovono in una data direzione (anterograda, retrograda) o non in movimento (stazionarie) per il numero totale di particelle analizzate nel chimografo. Il tempo trascorso da ciascuna vescicola in pausa o in movimento è stato calcolato calcolando la media della percentuale di tempo trascorso in ciascuna condizione per tutte le vescicole in ciascun neurone analizzato. La distribuzione di frequenza per la velocità e la lunghezza della corsa è stata calcolata utilizzando solo vescicole mobili per ogni condizione sperimentale. L'analisi è stata eseguita su tratti assonali di 100 mm per 3 min.
- Generazione di un chimografo
- Aprire il file .nds in Fiji ImageJ.
- Vai alla scheda Immagine e fai clic su Proprietà. Registra il rapporto pixel/micron. Questo è necessario per il calcolo successivo.
- Fare clic su File > Salva come > Tiff per salvare il file come .tiff nella relativa cartella (la macro salverà automaticamente tutto ciò che è stato generato in questa cartella).
- Trascina la macro Kymo in ImageJ. Il codice è fornito nel file di codifica supplementare 1. Fare clic su Esegui.
- Non premere OK. Passa alla finestra MAX_raw. Fare clic manualmente con il pulsante sinistro del mouse lungo l'assone e fare doppio clic per terminare il tracciamento.
NOTA: Assicurarsi di tracciare l'assone dal soma in modo che il terminale dell'assone. In questo modo i calcoli anterogradi e retrogradi saranno corretti. - Premi il comando T o Aggiungi su ROI manager.
- Ora fai clic su OK. Verrà generato un chimografo nella cartella creata in precedenza. (L'asse X è la lunghezza dell'assone in micron e l'asse Y è il tempo in secondi).
- Tracciamento del chimografo
- Trascina il chimografo in Fiji Image J (dovrebbe essere posizionato automaticamente nella cartella creata in precedenza).
- Trascinare la macro Traccia nell'immagine J.
- Al prompt seguente, fare clic su OK. Se si riprende il lavoro da prima, inserire il punto numerico da cui continuare e quindi premere Ok.
- Utilizzando lo strumento Rettangolo nel pannello Seleziona , selezionare un'area vicino al centro del chimografo che sia alta 60 pixel e abbia sempre un diametro di 100 μm. A tale scopo, dividere 100 per il rapporto pixel/micro registrato nel passaggio 3.1.2.
- Dopo aver selezionato un'area, premere Ctrl T per aggiungere i t alla ROI e salvare la ROI nella cartella in cui si sta lavorando (premere Altro nel menu ROI e quindi Salva).
NOTA: Invertendo l'area selezionata premendo Ctrl+Maiusc+I , sarà più facile tracciare. - Per tracciare il movimento di una singola vescicola, tieni premuto Ctrl mentre fai clic con il pulsante sinistro del mouse dall'alto verso il basso.
- Per fare ciò, tieni premuto Ctrl mentre fai clic con il pulsante destro del mouse e apparirà una finestra che mostra dove vengono tracciati gli assoni. Se l'aspetto è buono, premere Ok.
- Per continuare e selezionare un'altra vescicola, fare clic su Sì e ripetere la procedura dai passaggi 3.2.6-3.2.8. Una volta tracciate tutte le vescicole visibili, premere No. In questo modo la sovrapposizione tracciata a mano verrà salvata automaticamente nella stessa cartella.
- Misurazione dei dati del chimografo
- Creare una nuova cartella. Trascina tutti i file di testo generati nella cartella.
- Apri il file del chimografo e registra le dimensioni.
- Trascinare la macro Misura nell'immagine J.
- Immettere il rapporto micron/pixel registrato in precedenza in PixelScale.
- Modificare il limite di velocità minimo a 0,1. (Le vescicole che si muovono al di sotto di questo limite sono considerate stazionarie).
- Inserire le dimensioni del chimografo precedentemente registrate. Il file di riepilogo verrà generato automaticamente nella cartella di testo creata. Ogni riempimento di riepilogo conterrà la percentuale di tempo percorso, la velocità complessiva (μm/s), la distanza totale, il segmento medio percorso, il numero di volte in cui si è fermato e il numero di volte in cui è stato invertito. Il codice per la generazione, il tracciamento e la misurazione dei dati del chimografo è fornito come file di codifica supplementare 1.
Risultati
Sono state eseguite analisi chimografiche e di imaging su cellule vive per misurare la motilità dei mitocondri liberi marcati con Mito-RFP o dei mitocondri legati all'ER di larghezze di contatto strette (6 nm ± 1 nm) o sciolte (24 nm ± 3 nm) stabilizzate da MAM 1X o MAM 9X, rispettivamente, nel processo neuronale più lungo di ciascun neurone ReN GA (AD) o ReN (naïve) lungo almeno 500 nm, considerando questo come un assone (Figura 1 e Figura 2). Le frequenze dei movimenti (complessivi, retrogradi e anterogradi) sono state calcolate dividendo il numero di puncta marcati RFP (MAM) in movimento o stazionari per il numero totale nei chimografi (Figura 1A-E). La velocità assonale complessiva dei mitocondri legati all'ER marcati con MAM 1X è diminuita drasticamente di ~50% rispetto ai mitocondri liberi da ER marcati con Mito-RFP o ai mitocondri legati all'ER marcati con MAM 9X (Figura 1B). L'analisi quantitativa ha anche rivelato differenze drammatiche tra i movimenti complessivi e retrogradi dei mitocondri legati all'ER stabilizzati MAM 1X rispetto ai mitocondri legati all'ER liberi (Mito-RFP) o stabilizzati con MAM 9X. Mentre il 53,82% ± il 3,3% dei mitocondri liberi da ER (Mito-RFP) erano mobili, solo il 26,6% ± il 3,4% dei mitocondri legati all'ER marcati con MAM 1X erano mobili, suggerendo che la stabilizzazione dei MAM ha ridotto significativamente la mobilità assonale complessiva dei mitocondri, strettamente associati all'ER, rispetto ai mitocondri non legati o debolmente legati all'ER (44,79% ± 2,6% di MAM 9X rispetto al 53,82% ± 3,3% di Mito RFP, (Figura 1C). Coerentemente, sia i movimenti retrogradi che anterogradi dei mitocondri legati all'ER marcati con MAM 1X erano significativamente più bassi rispetto ai mitocondri marcati con MAM 9X o liberi (Mito-RFP) (retrogrado: 12,33% ± 2,55% per MAM 1X rispetto al 25,78% ± 2,31% per Mito RFP; anterogrado: 14,27% ± 2,81% per MAM 1X rispetto al 28,04% ± 2,48% per Mito RFP) (Figura 1D ed E). La tabella 3 fornisce le precise velocità assonali dei mitocondri liberi o di quelli strettamente o debolmente legati all'ER. Questi valori possono essere utilizzati come un notevole mezzo quantitativo per valutare il grado di stabilizzazione della MAM che varia tra le MAM strette e quelle allentate, portando alla riduzione della generazione di Ab. Le velocità di trasporto assonale mitocondriale dopo la stabilizzazione delle MAM strette e libere nelle cellule ReN naive rispecchiavano i modelli di trasporto osservati nei neuroni ReN GA (Figura 1F-G). I risultati coerenti tra i neuroni ReN naive e i neuroni ReN GA AD che esprimono APPSwe/Lon suggeriscono che l'effetto sul trasporto assonale è prevalentemente attribuito allo stato di stabilizzazione MAM, indipendentemente dalla presenza di APPSwe/Lon o dalla conseguente produzione di Aβ.
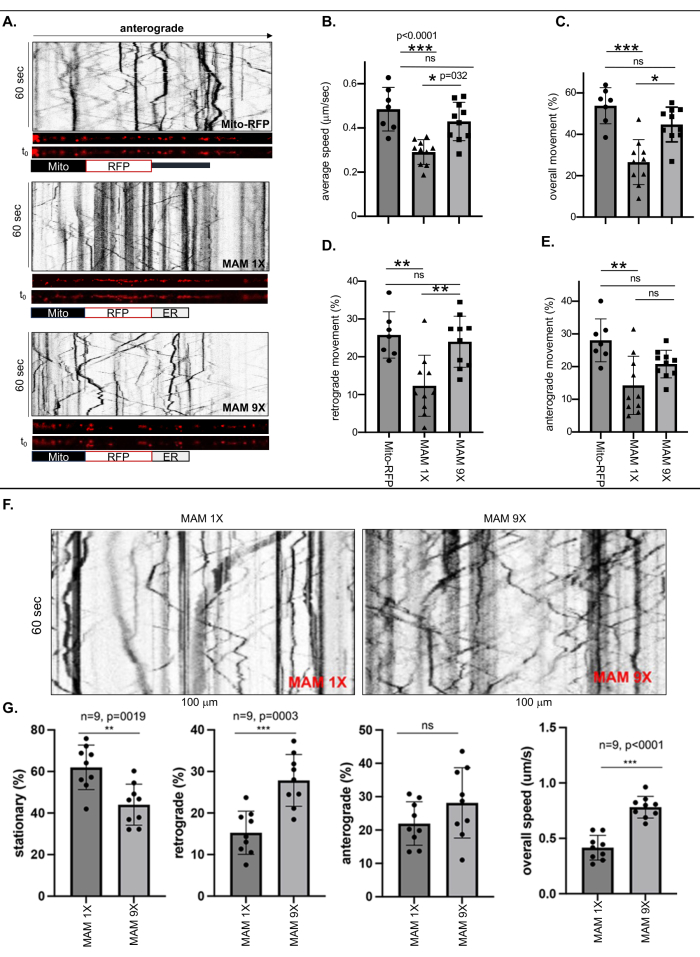
Figura 1: La stabilizzazione dei MAM mediante MAM 1X ha ridotto la velocità media e il movimento (complessivo, retrogrado e anterogrado) dei mitocondri legati all'ER negli assoni delle cellule ReN GA differenziate e delle cellule ReN naive. (A) Chimografi rappresentativi dei puncta marcati con RFP che rappresentano mitocondri liberi (Mito-RFP) o mitocondri legati all'ER stabilizzati da MAM 1X (MAM stretti, 6 nm ± larghezza di contatto 1 nm) o MAM 9X (MAM sciolti, 24 nm ± 3 nm larghezza di contatto) all'interno degli assoni (~100 nm). (B-E) Analisi quantitativa della velocità media (B) e del movimento [(C) complessivo, (D) retrogrado e (E) anterogrado] di Mito-RFP, MAM 1X o MAM 9X all'interno degli assoni delle cellule Ren-GA differenziate a 10 giorni. n>7; È stata eseguita l'ANOVA bidirezionale. *p < 0,05, **p < 0,001. Rappresentativo di tre esperimenti indipendenti. (F) Chimografi rappresentativi del movimento di MAM 1X o MAM 9X all'interno di assoni di cellule ReN naive differenziate a 10 giorni. (G) Analisi quantitativa della percentuale (%) di movimento (stazionario, retrogrado e anterogrado) e della velocità complessiva (micrometro/secondo; μm/s) di MAM stabilizzati con MAM 1X o MAM 9X all'interno degli assoni di cellule ReN naive. n = 9; È stata eseguita l'ANOVA bidirezionale. p < 0,0001. Questa figura è stata adattata con il permesso di Zellmer et al.17. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Immagini di cellule vive di assoni di cellule ReN naive che esprimono MAM 1X o MAM 9X. Immagini video rappresentative di cellule vive che mostrano i movimenti delle MAM stabilizzate da MAM 1X o MAM 9X all'interno di assoni lunghi 100 μm di cellule ReN differenziate che esprimono GFP a 10 giorni. n > 10 immagini da esperimenti duplicati. Le frecce indicano il trasporto anterogrado. Barra di scala: 100 μm. Clicca qui per visualizzare una versione più grande di questa figura.
| Reagente | Concentrazione finale | Importo |
| DMEM con L-glutammina | N/A | 500 ml |
| Eparina | 2 μg/mL | 0,5 ml |
| B27 | 1x | 10 ml |
| bFGF | 20 ng/mL | 0,4 ml |
| FEG | 20 ng/mL | 0,5 ml |
| Penicillina/Streptomicina | 100 unità/mL | 5 ml |
| Totale | 516,4 ml | |
| Filtrare i terreni prima di aggiungere penicillina/streptomicina. Conservare a 4 °C per un massimo di 1 mese. | ||
Tabella 1: Composizione dei mezzi di espansione.
| Reagente | Concentrazione finale | Importo |
| DMEM con L-glutammina | N/A | 500 ml |
| Eparina | 2 μg/mL | 0,5 ml |
| B27 | 1x | 10 ml |
| Peniciilliion/Streptomicina | 100 unità/mL | 5 ml |
| Totale | 515,5 ml | |
| Filtrare i terreni prima di aggiungere penicillina/streptomicina. Conservare a 4 °C per un massimo di 1 mese | ||
Tabella 2: Composizione dei mezzi di differenziazione.
| ReN GA | ReN (ingenuo) | ReN GA (3D) | |||||
| Complessivamente (%) | Retrogrado (%) | Anterogrado (%) | Velocità media (mm/s) | Ab40 (pM) | Ab42 (pM) | ||
| Mito-RFP | 53,82 ± 3,3% | 25,78 ± 2,31% | 28,04 ± 2,48% | 0,66 ± 0,03 | 0,69 ± 0,07 | 241,7 ± 26,74 | 13,77 ± 1,52 |
| MAM 1X | 26,6 ± 3,4% *** | 12,33 ± 2,5% *** | 14,27 ± 2,81% *** | 0,3 ± 0,02*** | 0,43 ± 0,04*** | 377,2 ± 76,87* | 26,62 ± 3,86* |
| MAM 9X | 44,79 ± 2,6% ns | 23,99 ± 2,17%ns | 20,80 ± 1,33% ns | 0,59 ± 0,02 ns | 0,62 ± 0,02 ns | 158,8 ± 3,27* | 17.01 ± 2.02* |
Tabella 3: Analisi quantitativa. Imaging di cellule vive e analisi quantitativa basata sulla chimografia della velocità media (velocità) e dei movimenti assonali (complessivi, retrogradi e anterogradi) di Mito-RFP, MAM 9X e MAM 1X. L'ANOVA bidirezionale è stata eseguita per la velocità assonale o il movimento (%). n = 9. Per Aβ è stata eseguita l'ANOVA ordinaria a una via; n = 3, tre esperimenti indipendenti. La significatività è misurata rispetto alle cellule ReN GA non trasfettate (di controllo). **p < 0,0001; *p < 0,05; non significativo (NS). Questa tabella è stata adattata con il permesso di Zellmer et al.17.
File di codifica supplementare 1: Il codice per la generazione, il tracciamento e la misurazione dei dati del chimografo. Clicca qui per scaricare questo file.
Discussione
L'inibizione del recettore sigma-1 (S1R) ha ridotto la stabilizzazione MAM nei processi neuronali e ha ridotto drasticamente (~90%) la generazione di Aβ dagli assoni ma non dal soma di un sistema di coltura tridimensionale (3D) di cellule progenitrici neurali umane (ReN) che esprimono mutazioni familiari di AD [FAD] nel gene della proteina precursore dell'amiloide [APP] (ReN GA)23,24,25,27. Gli stabilizzatori MAM costitutivi marcati RFP (MAM 1X e MAM 9X) progettati per stabilizzare MAM stretti (6 nm ± 1 nm) e sciolti (24 nm ± 3 nm)15,26 sono strumenti notevoli per misurare quantitativamente la stabilizzazione MAM. Entrambi gli stabilizzatori non solo mostrano un'espressione uguale e stabile nelle cellule ReN GA differenziate in matrice 3D per ~10 giorni, ma hanno anche rilevato MAM in puncta discreti in soma e assoni. Ancora più importante, mentre l'espressione stabile di MAM 1X nel 3D ReN GA arricchito con FACS ha aumentato significativamente la generazione di Aβ, l'espressione di MAM 9X non ha avuto alcun effetto17. Abbiamo anche testato l'effetto di uno stabilizzatore MAM costitutivo contenente 18 linker di aminoacidi (MAM 18X) che rileva e stabilizza i MAM >25 nm. A differenza di MAM 1X o MAM 9X, MAM 18X marcava esclusivamente MAM somali. I neuroni ReN GA che esprimono MAM 18X arricchiti con FACS, hanno ridotto la generazioneAβ 17. Questi risultati hanno suggerito la possibilità di una soglia di stabilità MAM determinata dall'ampiezza del loro gap che varia tra MAM stretti patogeni (aumento della generazione di Aβ) e MAM sciolti non patogeni (mantenimento o riduzione della generazione di Aβ). Trovare un modulatore MAM efficace e la sua concentrazione ottimale in grado di raggiungere la stabilizzazione ottimale del MAM necessaria per attraversare la soglia da MAM patogeni e non patogeni rivelerà una notevole via terapeutica per abbassare l'Aβ assonale o neuronale generazione nel cervello.
Per sviluppare i modulatori MAM sono stati impiegati tre diversi approcci: (1) modulatori che hanno come bersaglio le proteine di legame MAM, (2) modulatori che alterano i livelli di espressione delle proteine residenti in MAM e (3) modulatori delle strutture MAM18. Nonostante questi approcci, l'ostacolo principale alla ricerca di modulatori efficaci della stabilizzazione MAM è la mancanza di metodi per misurare quantitativamente il grado di stabilizzazione MAM. Le tecniche tradizionali come la microscopia elettronica (EM) o la microscopia a super-risoluzione hanno limitazioni nel catturare i cambiamenti in tempo reale o nel fornire dettagli sufficienti per valutare la stabilizzazione della MAM (rivisto in28).
Il metodo qui descritto supererà l'ostacolo e fornirà informazioni chiave sulla relazione tra stabilizzazione MAM e produzione di Aβ. I risultati mostrano che le MAM con uno spessore di 6 nm ± 1 nm, che mostrano un movimento complessivo del 26,6% ± del 3,4% (Tabella 3), sono associate alla generazione di Aβ. Al contrario, le MAM con uno spessore di 24 nm ± 3 nm, che mostrano un movimento complessivo del 44,79% ± del 2,6% (Tabella 3), non influenzano la generazione di Aβ. Il movimento complessivo dei mitocondri (Mito-RFP) è stato del 53,82% ± del 3,3%. Dato che lo spessore del MAM varia tipicamente tra 6 nm e 80 nm, questi risultati delineano i limiti superiore e inferiore della stabilizzazione del MAM in relazione alla produzione di Aβ. Di conseguenza, questo metodo può guidare l'identificazione e l'ottimizzazione di uno o più modulatori per la stabilizzazione MAM. L'obiettivo sarebbe quello di modificare il movimento complessivo dei MAM dal 26,6% ± 3,4% al 53,82% ± 3,3%, o la loro velocità media da  0,4 μm/s a
0,4 μm/s a  0,7 μm/s (Tabella 3), posizionando tali modulatori come potenziali agenti terapeutici contro la produzione di Aβ.
0,7 μm/s (Tabella 3), posizionando tali modulatori come potenziali agenti terapeutici contro la produzione di Aβ.
L'uso di modulatori costitutivi MAM contenenti linker sintetici di lunghezza crescente (0-18 amminoacidi) è un metodo potente per determinare quantitativamente la soglia di stabilizzazione MAM per commutare la stabilizzazione MAM in una che mantiene o possibilmente riduce invece di aumentare la generazione di Aβ. Tuttavia, per valutare l'efficienza o l'efficacia dei modulatori MAM, saranno necessari stabilizzatori MAM inducibili. Sono disponibili stabilizzatori MAM basati su microscopia per imaging FRET/FLIM (FRET/FLIM) con risonanza di risonanza inducibile di Förster che sono plasmidi di espressione che codificano per la sequenza di targeting OMM fusa con YFP di mAKAP1 (34-63) e per la fosfatasi Sac1 mirata all'ER fusa con CFP (521-587). Inoltre, gli stabilizzatori costitutivi potrebbero non rappresentare le MAM fisiologiche, mentre gli stabilizzatori MAM FRET/FLIM, d'altra parte, rileveranno le MAM fisiologiche. Possono essere utilizzate anche le sonde GFP divise, in cui la GFP è scissa in due frammenti non fluorescenti legati all'ER residente o alle proteine mitocondriali ER-GFP (1-10) e Mito-GFP11 che generano la complementazione biomolecolare a fluorescenza (BiFC) dopo la formazione di MAM27. Sebbene i frammenti GFP siano inclini all'assemblaggio spontaneo, BiFC ha la lettura più semplice, il segnale più chiaro e l'analisi meno associata al rumore. Inoltre, l'interazione tra la GFP split è altamente reversibile28, quindi i loro vantaggi superano gli svantaggi e rendono il metodo BiFC adatto per identificare modulatori di stabilizzazione MAM.
Riconoscimenti
Ringraziamo il Dr. György Hajnóczky, Professore della Thomas Jefferson University, Philadelphia per averci generosamente fornito plasmidi di espressione che codificano per RFP-Mito, MAM 1X, MAM 9X e MAM 18X. Un ringraziamento speciale al Dr. Lai Ding, Senior Imaging Scientist, Brigham and Women's Hospital per averci aiutato a scrivere il codice per la generazione, il monitoraggio e la misurazione dei dati del chimografo. Questo studio è stato sostenuto dal Cure Alzheimer's Fund to RB e dalla sovvenzione NIH 5R01NS045860-20 a RET.
Materiali
| Name | Company | Catalog Number | Comments |
| 6 Well Glass Bottom Plate | Cellvis | P06-1.5H-N | |
| B-27 Supplement (50X), serum free | Gibco/Thermo Fisher Scientific | 17504044 | |
| bFGF | R&D System | 233-FB | |
| BSA | Fisher Scientific | 501781532 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10283 | |
| DMEM/F12 with L-glutamine | Gibco/Thermo Fisher Scientific | 11320-033 | |
| EDTA | Life Technologies | 41116134 | |
| EGF | Sigma-Aldrich | 92090408 | |
| Falcon 6 Well Plates | VWR International | 41122107 | |
| GAPDH Polyclonal Antibody | Thermo Fisher Scientific | PA1-988 | |
| Gelatin | VWR International | 9000-70-8 | |
| Graphpad Prism N/A | Prism 9, version 9.5.0 | N/A | |
| Heparin | Sigma-Aldrich | H0200000 | |
| ImageJ Software | ImageJ 1.53a | N/A | |
| Matrigel Basement Membrane Matrix | Corning | 356234 | |
| mCherry Polyclonal Antibody | Invitrogen | PA5-34974 | |
| MS Excel | Microsoft Excel, version 2302 | N/A | |
| Multi-array electrochemiluminescence assay kit | Meso Scale Diagnostics (MSD) | K15200E-2 | V-PLEX Aβ Peptide Panel 1 (6E10) kit |
| NaCl | Fisher Scientific | 7647145 | |
| NuPAGE 4–12% Bis-Tris gel | Invitrogen | NP0321BOX | |
| Penicillin/Streptomycin/Amphotericin B | Lonza | 17-745E | |
| Photoshop | Adobe Photoshop CC 20.0.10 | N/A | |
| Rat Neuron Nucleofector Kit | Lonza | VPG-1003 | |
| StemPro Accutase | Gibco | A1110501 | |
| Tris-HCL, pH 7.6 | Boston BioProducts | 42000000 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Fisher Scientific | 501657287 |
Riferimenti
- Giacomello, M., Pellegrini, L. The coming of age of the mitochondria-ER contact: a matter of thickness. Cell Death Differ. 23 (9), 1417-1427 (2016).
- Degechisa, S. T., Dabi, Y. T., Gizaw, S. T. The mitochondrial associated endoplasmic reticulum membranes: A platform for the pathogenesis of inflammation-mediated metabolic diseases. Immun Inflamm Dis. 10 (7), e647 (2022).
- Schon, E. A., Area-Gomez, E. Mitochondria-associated ER membranes in Alzheimer disease. Mol Cell Neurosci. 55, 26-36 (2013).
- Erpapazoglou, Z., Mouton-Liger, F., Corti, O. From dysfunctional endoplasmic reticulum-mitochondria coupling to neurodegeneration. Neurochem Int. 109, 171-183 (2017).
- Sala-Vila, A., et al. Interplay between hepatic mitochondria-associated membranes, lipid metabolism and caveolin-1 in mice. Sci Rep. 6, 27351 (2016).
- Fujimoto, M., Hayashi, T. New insights into the role of mitochondria-associated endoplasmic reticulum membrane. Int Rev Cell Mol Biol. 292, 73-117 (2011).
- Hung, V., et al. Proteomic mapping of cytosol-facing outer mitochondrial and ER membranes in living human cells by proximity biotinylation. eLife. 6, e24463 (2017).
- Area-Gomez, E., Schon, E. A. On the pathogenesis of Alzheimer's disease: The MAM hypothesis. FASEB J. 31 (3), 864-867 (2017).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Sukhorukov, V. S., et al. Molecular mechanisms of interactions between mitochondria and the endoplasmic reticulum: A new look at how important cell functions are supported. Mol Biol. 56 (1), 59-71 (2022).
- Zhang, P., Konja, D., Zhang, Y., Wang, Y. Communications between Mitochondria and endoplasmic reticulum in the regulation of metabolic homeostasis. Cells. 10 (9), 2195 (2021).
- Ziegler, D. V., Martin, N., Bernard, D. Cellular senescence links mitochondria-ER contacts and aging. Commun Biol. 4 (1), 1323 (2021).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Carpio, M. A., et al. BOK controls apoptosis by Ca(2+) transfer through ER-mitochondrial contact sites. Cell Rep. 34 (10), 108827 (2021).
- Prudent, J., et al. MAPL SUMOylation of Drp1 stabilizes an ER/mitochondrial platform required for cell death. Mol Cell. 59 (6), 941-955 (2015).
- Zellmer, J. C., Tarantino, M. B., et al. Stabilization of mitochondria-associated endoplasmic reticulum membranes regulates Abeta generation in a three-dimensional neural model of Alzheimer’s disease. Alzheimer’s Dement. , 1-20 (2024).
- Magalhaes Rebelo, A. P., et al. Chemical modulation of mitochondria-endoplasmic reticulum contact sites. Cells. 9 (7), 1637 (2020).
- Berger, S. I., Iyengar, R. Role of systems pharmacology in understanding drug adverse events. Wiley Interdiscip Rev Syst Biol Med. 3 (2), 129-135 (2011).
- Friedman, J. R., Webster, B. M., Mastronarde, D. N., Verhey, K. J., Voeltz, G. K. ER sliding dynamics and ER-mitochondrial contacts occur on acetylated microtubules. J Cell Biol. 190 (3), 363-375 (2010).
- Wu, Y., et al. Contacts between the endoplasmic reticulum and other membranes in neurons. Proc Natl Acad Sci U S A. 114 (24), E4859-E4867 (2017).
- Cagin, U., et al. Mitochondrial retrograde signaling regulates neuronal function. Proc Natl Acad Sci U S A. 112 (44), E6000-E6009 (2015).
- Choi, S. H., et al. A three-dimensional human neural cell culture model of Alzheimer's disease. Nature. 515 (7526), 274-278 (2014).
- Kim, Y. H., et al. A 3D human neural cell culture system for modeling Alzheimer's disease. Nat Protoc. 10 (7), 985-1006 (2015).
- Kwak, S. S., et al. Amyloid-beta42/40 ratio drives tau pathology in 3D human neural cell culture models of Alzheimer's disease. Nat Commun. 11 (1), 1377 (2020).
- Csordas, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Bhattacharyya, R., et al. Axonal generation of amyloid-beta from palmitoylated APP in mitochondria-associated endoplasmic reticulum membranes. Cell Rep. 35 (7), 109134 (2021).
- Tebo, A. G., Gautier, A. A split fluorescent reporter with rapid and reversible complementation. Nat Commun. 10 (1), 2822 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon