Method Article
Isolement de cellules stromales mésenchymateuses dérivées du cordon ombilical BAMBIhumain
Dans cet article
Résumé
L’isolementdes CSM BAMBIà hauteMFGE8, l’un des trois principaux sous-groupes constituant les CSM-UC humaines hétérogènes, est utile pour bien comprendre les caractéristiques et les fonctions de ce sous-type en vue de son application future pour améliorer l’efficacité clinique dans des maladies spécifiques. Nous présentons ici une méthode de tri des UC-MSCBAMBI highMFGE8.
Résumé
Les cellules stromales/souches mésenchymateuses dérivées du cordon ombilical (UC-MSCs) présentent une faible immunogénicité et de puissants effets immunomodulateurs pour le traitement de diverses maladies. Les CSM-UC humaines sont une population hétérogène composée de trois sous-populations principales avec des formes cellulaires, des taux de prolifération, des capacités de différenciation et des fonctions de régulation immunitaire différents. Auparavant, les UC-MSCBAMBI highMFGE8 high, le premier sous-groupe isolé avec succès des UC-MSC, n’ont pas réussi à soulager la néphrite lupique. Par conséquent, la fonction et le mécanisme sous-jacent de ce sous-groupe dans le traitement des CSM pour les maladies restent inconnus. Il est nécessaire d’isoler et d’étudier plus en détail les CSM-UCélevées de BAMBIentermes de phénotype, de métabolisme et de fonction pour comprendre complètement la nature de ce sous-groupe de CSM. Dans ce protocole, nous décrivons une méthode détaillée pour isoler la sous-populationBAMBI highMFGE8 high des UC-MSC humaines. La sous-population de CSM-UC est marquée avec deux marqueurs de surface, BAMBI et MFGE8, par tri par cytométrie en flux. Les cellules isolées sont cultivées et vérifiées par analyse par cytométrie en flux. Les gènes spécifiques exprimés dans lesCSM-UC élevées de BAMBIàMFGE8 élevés sont identifiés par RT-qPCR. Ce protocole permet un tri cellulaire très efficace et pur et décrit les profils de marqueurs des UC-MSCBAMBI highMFGE8high.
Introduction
Les cellules stromales/souches mésenchymateuses humaines (CSM) sont des progéniteurs somatiques capables de se différencier en ostéocytes, adipocytes, chondrocytes et autres types de cellules1. Les CSM ont d’abord été isolées dans la moelle osseuse et sont largement dérivées du cordon ombilical, du tissu adipeux et d’autres tissus2. Parce que les UC-MSC sont faciles à obtenir et présentent une faible immunogénicité et des effets immunosuppresseurs, elles sont largement appliquées dans les essais cliniques pour traiter diverses maladies 3,4,5. Bien que la thérapie MSC montre un potentiel prometteur pour le traitement des maladies, les effets thérapeutiques sont incohérents entre les individus6. Cependant, la raison de l’instabilité du traitement par CSM n’est pas encore claire.
Les fluctuations moléculaires, la morphologie, la capacité de différenciation et la fonction thérapeutique constituent l’hétérogénéité des CSM. Certaines études ont également postulé que les CSM constituent des sous-populations ayant des fonctions différentes 7,8 et ont exploré l’hétérogénéité des CSM via le séquençage de l’ARN unicellulaire (scRNA-seq)9,10. Les résultats ont révélé que les CSM-UC humaines ont des sous-populations distinctes avec des caractéristiques transcriptomiques spécifiques, alors que peu d’études ont réussi à isoler ce que l’on appelle les sous-populations de CSM. Nous avons précédemment disséqué les UC-MSC humaines en trois sous-groupes en fonction de leurs signatures via le scRNA-seq et l’analyse bioinformatique, dans laquelle la sous-population BAMBIhighMFGE8high UC-MSC a été purifiée et testée fonctionnellement11. Cependant, ce sous-groupe n’a pas réussi à atténuer la néphrite lupique. Ainsi, il est nécessaire de tester les effets thérapeutiques de BAMBIhighMFGE8high MSCs dans d’autres troubles pour comprendre leurs fonctions authentiques.
Ce protocole décrit les méthodes d’isolementdu sous-groupe BAMBIHighMFGE8 high à partir des UC-MSC humaines par tri cellulaire activé par fluorescence (FACS) par cytométrie en flux et les caractéristiques du sous-groupe BAMBIhighMFGE8high.
Protocole
Cette étude a été menée conformément aux principes énoncés dans la Déclaration d’Helsinki de 1989 et approuvée par le Comité d’éthique de l’hôpital affilié Drum Tower de la faculté de médecine de l’Université de Nanjing (numéro d’approbation : 202019701). Des cordons ombilicaux humains ont été obtenus de mères en bonne santé à l’hôpital Affiliate Drum Tower de la faculté de médecine de l’Université de Nanjing après un travail naturel, qui ont donné leur consentement éclairé pour leur utilisation dans ce travail. Des CSM-UC primaires ont été isolées dans les cordons ombilicaux humains, comme indiqué précédemment11.
1. Culture et identification de l’UC-MSC avant l’isolement
- Une fois que les CSM atteignent 70-80 % de confluence (P0), lavez les cellules une fois avec du PBS et ajoutez 1 mL de trypsine-EDTA à 0,25 % pendant 2 min à 37 °C. Ensuite, ajoutez 9 ml de milieu complet pour neutraliser la trypsine, transférez la suspension cellulaire dans un tube à centrifuger de 15 ml et centrifugez à 300 × g pendant 5 minutes pour recueillir les cellules. Jetez le surnageant et ajoutez le milieu complet approprié pour remettre les cellules en suspension. Transvaser les cellules de chaque boîte dans trois flacons T75 (P1) et les faire cultiver dans un incubateur cellulaire à 37 °C et 5 % de CO2.
- Après avoir passé les UC-MSCs 2x par trypsinisation, à P3, récoltez les cellules en suivant les mêmes étapes que celles décrites à l’étape 1.1. Après la centrifugation, jeter le surnageant. Remettez les cellules en suspension dans le tampon de coloration FACS (1x PBS contenant 2 % de FBS) et comptez-les. Ajouter le tampon de coloration FACS à une concentration finale de 5-10 ×10-6 cellules/mL et maintenir la suspension cellulaire sur de la glace.
- Répartissez 100 μL par tube de cette suspension cellulaire dans différents tubes de 1,5 mL. Ajoutez des contrôles d’isotypes et des anticorps FACS contre CD29, CD73, CD90, CD105, CD14, CD34, CD45, CD79 et HLA-DR (tous à 1:200) dans les cellules à 4 °C pendant 30 min.
- Lavez les cellules 2 fois avec le tampon de coloration FACS et centrifugez-les à 300 × g pendant 5 min. Jeter le surnageant et remettre les précipités en suspension dans 200 μL de tampon de coloration FACS pour une analyse par cytométrie en flux afin d’identifier les marqueurs MSC.
2. Isolement des CSM-UCBAMBI à haute MFGE8élevée par cytométrie en flux
- Cultivez des UC-MSC à une densité d’environ 5-10 × 106 cellules en isolantdes UC-MSCBAMBI à hauteMFGE8. Dissociez les cellules avec 0,5 mM d’EDTA pendant 5 min jusqu’à ce qu’elles commencent à avoir une morphologie ronde, ajoutez le milieu complet pour transférer les cellules dans un tube conique de 15 mL, et pipetez la suspension cellulaire de haut en bas plusieurs fois pour préparer une suspension unicellulaire. Utilisez 10 μL de suspension cellulaire pour compter les cellules et calculer le nombre total de cellules récoltées. Centrifuger le tube conique avec la suspension cellulaire à 300 × g pendant 5 min pour collecter les cellules.
- Remettre les cellules en suspension dans 1 mL de milieu complet jusqu’à une concentration finale de 5 à 10 × 10 à6 cellules/mL et maintenir la suspension cellulaire sur de la glace.
- Divisez les cellules dans quatre tubes de microcentrifugation de 1,5 mL (cellules vides seulement ; cellules marquées BAMBI ; cellules marquées MFGE8 ; et les cellules marquées BAMBI et MFGE8).
- Ajouter des anticorps primaires à des concentrations appropriées (MFGE8 et BAMBI, tous deux à 1:100) dans les tubes, mélanger et incuber les cellules à température ambiante pendant 15 min.
- Lavez une fois les cellules marquées avec 1x PBS et centrifugez à 300×g pendant 5 min. Jetez le surnageant. Remettez les cellules en suspension dans 1 mL de milieu complet, ajoutez des anticorps secondaires fluorescents conjugués (IgG H&L de chèvre anti-lapin Alexa Fluor 488 et Alexa Fluor 647, tous à 1:1 000) aux cellules, mélangez et incubez les cellules à température ambiante pendant 15 min dans l’obscurité.
- Lavez une fois les cellules marquées avec 1x PBS, comme décrit à l’étape 2.5. Remettre les cellules en suspension dans 500 μL de milieu complet, filtrer à travers une crépine cellulaire de 70 μm pour éliminer les amas et les débris, et transférer le filtrat dans un tube de 15 mL pour le tri par cytométrie en flux.
- Faites fonctionner le tube de cellules vierges sans ajouter d’anticorps (contrôle négatif) et ajustez la diffusion vers l’avant (FSC) et la diffusion latérale (SSC) pour bloquer l’échelle de la population négative avec la coloration des anticorps.
- Exécutez les tubes cellulaires marqués d’un seul anticorps (c’est-à-dire MFGE8 + IgG H&L anti-lapin Alexa Fluor 488, ou BAMBI + IgG H&L Alexa Fluor 647 anti-lapin pour chèvre) comme contrôle de contrôle pour déterminer où commence la positivité dans la parcelle.
- Exécutez le(s) tube(s) d’échantillon expérimental pour trier et collecter lapopulation cellulaire BAMBI highMFGE8.
- Plaquez les CSM triées BAMBIhighMFGE8high dans une plaque à 24 puits et cultivez les cellules dans un incubateur cellulaire à 37 °C et 5 % de CO2.
- Lorsqueles CSM triées BAMBIà hauteMFGE8 se sont développées sur deux passages pour obtenir suffisamment de cellules, dissociez les cellules et effectuez une analyse post-tri pour garantir la pureté des populations cellulaires triées par cytométrie en flux, comme décrit aux étapes 2.1 à 2.9. Vous pouvez également examiner la pureté des cellules triées par immunofluorescence conventionnelle par rapport à l’expression humaine de BAMBI et MFGE8 si le nombre de cellules collectées est trop faible.
3. Caractérisation des CSMhautes MFGE8de BAMBI
- Cultivezles CSM BAMBIà hauteMFGE8 triées dans une plaque à 12 puits, examinez leur morphologie cellulaire au microscope et comparez-les à celle des CSM non triées.
REMARQUE : En général,les cellules BAMBIà hauteMFGE8 se développent plus rapidement que les UC-MSC non triées. - Lorsque les cellules sont confluentes à environ 90 % dans la plaque à 12 puits, aspirez le milieu de culture cellulaire, lavez les cellules une fois avec 1x PBS et ajoutez 500 μL de réactif d’extraction d’ARN aux cellules. Agitez lentement la plaque à température ambiante (RT) pendant 5 min.
- Pipetez le mélange et transférez-le dans un tube sans RNase ni DNase. Ajouter 100 μL de chloroforme, boucher le tube et agiter vigoureusement le mélange en tourbillonnant pendant 15 s. Ensuite, laissez le tube reposer à RT pendant 3 min.
- Centrifugez le tube pendant 15 min à 11 000 × g et 4 °C.
- Transférez environ 200 μL de la couche aqueuse supérieure dans un nouveau tube. Ajoutez le même volume d’isopropanol et pipetez soigneusement de haut en bas plusieurs fois. Ensuite, laissez le mélange reposer à RT pendant 10 min.
- Centrifugeuse pendant 15 min à 11 000 × g et 4 °C. Jetez le surnageant.
- Ajoutez 500 μL d’éthanol à 75 % dans le tube et retournez doucement le tube plusieurs fois.
- Centrifugez le tube pendant 5 min à 6 000 × g et 4 °C. Jetez le surnageant. Répétez la centrifugation une fois pendant 10 s à 6 000 × g, aspirez soigneusement le liquide restant à l’aide d’une pipette et séchez le tube à RT.
- Ajoutez 10 μL d’eau exempte de RNase pour dissoudre la pastille d’ARN et mesurez la concentration d’ARN à l’aide d’un spectrophotomètre.
- Suivez les instructions du fabricant pour synthétiser l’ADNc premier brin à partir d’un maximum de 1 μg d’ARN total.
- Utilisez un kit qPCR pour détecter et quantifier l’expression des gènes, selon les instructions du fabricant.
REMARQUE : Les séquences des amorces d’ADN utilisées sont énumérées dans le tableau 1.
Résultats
La figure 1 montre les profils d’expression des marqueurs de surface cellulaire des CSM-UC humaines. Les CSM en culture étaient fortement positives pour l’expression de CD44, CD73, CD90 et CD105 et négatives pour l’expression de CD14, CD34, CD45, CD79 et HLA-DR. LesCSM BAMBI à hauteMFGE8 ont été triées à partir de CSU-UC humaines cultivées, et leur expression de BAMBI et MFGE8 a été réanalysée par cytométrie en flux après expansion pour 3-4 passages (Figure 2). Dans ce processus, la fréquence des CSMBAMBI élevéesen MFGE8 a fluctué en fonction du donneur et de la méthode de dissociation (Figure 3 et Figure 4). La figure 5 montre une série de gènes de signature fortement exprimés dans les CSMélevées de BAMBIà MFGE8 élevées par rapport aux CSM non triées, telles que déterminées par RT-qPCR.

Figure 1 : Immunophénotypage des CSM-UC identifiées par analyse par cytométrie en flux. Les UC-MSC sont positifs pour CD44, CD73, CD90 et CD105 et négatifs pour CD14, CD34, CD45, CD79 et HLA-DR. L’histogramme noir représente le contrôle de l’isotype de l’anticorps, et l’histogramme rouge représente le signal de l’anticorps. Abréviations : UC-MSCs = cellules stromales/souches mésenchymateuses dérivées du cordon ombilical ; HLA-DR = antigène leucocytaire humain-DR. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Pureté des CSMBAMBI à hauteMFGE8 après tri par cytométrie en flux. Les résultats de l’analyse par cytométrie en flux des UC-MSC sans coloration par anticorps (en haut à gauche) et avec coloration MFGE8 et BAMBI avant (en haut au milieu) et après le tri cellulaire (en haut à droite) sont présentés. Les résultats de la coloration simple MFGE8 (en bas à gauche) et BAMBI (en bas à droite) sont également présentés. Abréviations : BAMBI = protéine morphogénique osseuse et inhibiteur membranaire de l’activine ; MFGE8 = globule gras du lait facteur de croissance épidermique 8 ; CSM = cellules stromales/souches mésenchymateuses. Veuillez cliquer ici pour voir une version agrandie de cette figure.
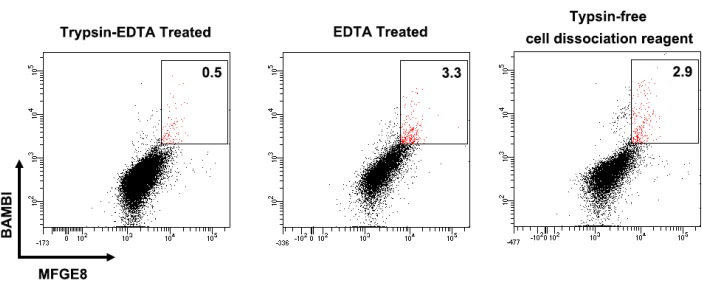
Figure 3 : Différentes fréquences des CSMélevées BAMBIélevéesMFGE8 dissociées par différentes méthodes. Les résultats de l’analyse par cytométrie en flux des CSM-UC du même donneur soumis à la dissociation trypsine-EDTA (à gauche), au traitement EDTA uniquement (au milieu) et au réactif de dissociation cellulaire sans trypsine (à droite) sont présentés. Abréviations : BAMBI = protéine morphogénique osseuse et inhibiteur membranaire de l’activine ; MFGE8 = globule gras du lait facteur de croissance épidermique 8 ; CSM = cellules stromales/souches mésenchymateuses ; TE = trypsine-EDTA. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : La fréquence des CSMBAMBI à forteMFGE8 chez différents donneurs. Les fréquences des CSMBAMBI HighMFGE8High varient entre les échantillons donneurs 1 (à gauche), 2 (au milieu) et 3 (à droite). Abréviations : BAMBI = protéine morphogénique osseuse et inhibiteur membranaire de l’activine ; MFGE8 = globule gras du lait facteur de croissance épidermique 8 ; CSM = cellules stromales/souches mésenchymateuses. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Gènes de signature hautement exprimés dans les CSMBAMBI à hauteMFGE8 examinées par RT-qPCR. Abréviations : BAMBI = protéine morphogénique osseuse et inhibiteur membranaire de l’activine ; MFGE8 = globule gras du lait facteur de croissance épidermique 8 ; CSM = cellules stromales/souches mésenchymateuses ; RT-qPCR = réaction en chaîne de la polymérase quantitative par transcription inverse. Test t de l’élève. *, P<0,05. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Amorce | Séquence avant (5'-3') | Séquence inverse (5'-3') |
| BAMBI | CGCCACTCCAGCTACATCTT | CAGTGGGCAGCATCAGTA |
| COL1A1 | CAAAGAAGGCGGCAAAGGTC | CACGCTGTCCAGCAATACCT |
| COL3A1 | CCTTCGACTTCTCTCCAGCC | TTTCGTGCAACCATCCTCCA |
| DCN | GGCTGGACCGTTTCAACAGA | GATGGCATTGACAGCGGAAG |
| NEAT1 | CACAGGCAGGGGAAATGTCT | TGCTGCGTATGCAAGTCTGA |
| FTH1 | AGCTCTACGCCTCCTACGTT | CCTGAAGGAAGATTCGGCCA |
| IGFBP3 | GCCAGCGCTACAAAGTTGAC | ATGTGTACACCCCTGGGACT |
| IGFBP5 | TCCCCACGTGTGTTCATCTG | AAATGGGATGGACTGAGGCG |
| MALAT1 | TGGGGGAGTTTCGTACTGAG | TCTCCAGGACTTGGCAGTCT |
| MEST | TGGGAGCTCTCTTGCCTCTGTA | AGAATCGACACTGTGGACCG |
| MFGE8 | TGTCTTCCCCTCGTACACCT | AGAAGGTCACACGCACAGAC |
| SERPINE2 | GTCCTCGTCAACGCAGTA | GTCCTCGTCAACGCAGTA |
| NUPR1 | CCTTCCCACCAGCAACCAG | GGTAGGAATGGGCCAGGCTA |
| GAPDH | TCAGTGGTGGACCTGACCTG | TGCTGTAGCCAAATTCGTTG |
Tableau 1 : Séquences des amorces d’ADN utilisées dans ce protocole.
Discussion
Ce protocole décrit comment isoler et enrichir la sous-populationBAMBI highMFGE8 high à partir de UC-MSCs humaines. La méthode est importante pour l’étude plus approfondie de la morphologie, de la croissance et de la fonction de ce sous-groupe MSC. Certaines étapes sont essentielles pour la réussite de l’isolement et le rendement élevédes cellules BAMBIà hauteMFGE8.
Tout d’abord, l’aspect technique le plus critique à prendre en compte est l’utilisation d’une solution de dissociation cellulaire appropriée dans le protocole actuel. Bien que la trypsine-EDTA conventionnelle à 0,25 % soit utilisée pour dissocier les CSM pour le passage, il est préférable d’utiliser une solution EDTA sans enzyme pour trierle sous-groupe BAMBIà hauteMFGE8, car le traitement à la trypsine réduit le rendementdes CSM àhaute MFGE8 de BAMBI lorsqu’elles sont triées (Figure 3). Une raison possible peut être que la trypsine altère la distribution des protéines transmembranaires MFGE8 et BAMBI à la surface de la cellule comme elle le fait pour d’autres protéines12. En revanche, l’utilisation de la solution EDTA a peu d’effet sur l’expression cellulaire de MFGE8 et BAMBI. Deuxièmement, le protocole de tri cellulaire décrit les paramètres de sélectiondes CSM BAMBI HighMFGE8. Il est nécessaire d’établir un échantillon vierge et des échantillons uniques marqués par des anticorps pour contrôler avec précisionles CSM BAMBI élevées BAMBIélevéesMFGE8 élevées pour le tri FACS.
Contrairement à la trypsinisation, le détachement des CSM-UC humaines par EDTA est recommandé pour trier les CSMBAMBI à hauteMFGE8 dans ce protocole. Cependant, d’autres modifications impliquant des méthodes de dissociation cellulaire légères, telles que Dispase et Tryple E13,14, peuvent également être applicables à la récolte de CSMBAMBI à hauteMFGE8, mais doivent être vérifiées. En particulier, la dissociation à long terme des UC-MSC doit être évitée, car une dissociation excessive tend à réduire la viabilité cellulaire. De plus, des inhibiteurs de ROCK (par exemple, Y-27632 à 10 μM), qui empêchent l’apoptose cellulaire15, peuvent être ajoutés au milieu de culture cellulaire pour augmenter la surviedes CSM triées BAMBIà hauteMFGE8 en réduisant leur degré d’apoptose. De plus, il est recommandé de réévaluer la puretédes CSM BAMBI à hauteMFGE8 en expansion régulière après le tri pour s’assurer qu’elles conservent leurs identités primaires, en particulier avant que d’autres tests de dosage fonctionnel ne soient effectués.
Bien qu’il n’y ait pas de corrélation entre le rapport BAMBI élevéMFGE8 et le sexe, les rapports BAMBIélevéMFGE8 élevé UC-MSC ont été acquis auprès de différents donneurs (Figure 4). Notamment, la proportion dela sous-population BAMBIélevéeMFGE8 élevée peut varier en fonction de différents passages ou conditions de culture. Si la proportion de cellules BAMBIà hauttaux de MFGE8 dans la population UC-MSC est extrêmement faible, le protocole actuel ne sera pas bien applicable. D’autres limitations de ce protocole incluent des dommages cellulaires relativement graves causés par la méthode de tri FACS, qui conduisent facilement à la mort cellulaire d’une proportionde CSM BAMBI élevéesen MFGE8 après le tri cellulaire. De plus, les méthodes actuelles de tri des anticorps primaires et secondaires en deux étapes prennent plus de temps et sont moins efficaces pour le tri cellulaire. Les applications futures des anticorps BAMBI et MFGE8 directement conjugués à la fluorescence sont préférables pour augmenter l’efficacité de trides CSM BAMBI à hauteMFGE8.
L’isolement de chaque sous-population de CSM mixtes est indispensable pour révéler leurs fonctions authentiques et leurs mécanismes sous-jacents dans le traitement des maladies. Par conséquent, la présente méthode fournit un outil fondamental et vital pour la mise en commun et l’étude plus approfondiedes CS-UC élevées de BAMBIà MFGE8élevées afin de comprendre complètement leur nature. L’optimisation future des conditions de culture cellulaireélevée de BAMBIHighMFGE8 produira un grand nombre de ces cellules dans l’industrie pour la thérapie par cellules souches pour des patients spécifiques en clinique.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par la Fondation nationale des sciences naturelles de Chine (subvention n° 82271843).
matériels
| Name | Company | Catalog Number | Comments |
| 0.6 mL microcentrifuge tube | Corning | Axygen MCT-060-A | |
| 1.5 mL microcentrifuge tubes | Beijing Labgic Technology | MCT-001-150 | |
| 100 mm cell culture dish | Beijing Labgic Technology | 12311 | |
| 12 well plate | Beijing Labgic Technology | 11210 | |
| 15 mL centrifuge tube | Nanjing Vazyme Material Technology | TCF00115 | |
| 24 well plate | Beijing Labgic Technology | 11310 | |
| 50 mL centrifuge tube | Nanjing Vazyme Material Technology | TCF00150 | |
| 5 mL Round-Bottom Tubes | Corning | FALCON 352003 | |
| 70 μm cell strainer | Falcon | 352350 | Dilution: 1:1000 |
| APC anti-human CD79a (Igα) Antibody | BioLegend | 333505 | 581 Dilution: 1:200 |
| APC/Cyanine7 anti-human CD73 (Ecto-5'-nucleotidase) Antibody | BioLegend | 344022 | G46-6 Dilution: 1:200 |
| APC-Cy7 Mouse IgG1, κ Isotype Control | BD Bioscience | 557873 | MOPC-31C (Isotype Control) Dilution: 1:200 |
| BAMBI antibody | Bioss | bs-12418R | |
| Brilliant Violet 510 anti-mouse/human CD44 Antibody | BioLegend | 103044 | 5E10 Dilution: 1:200 |
| Brilliant Violet 510 Rat IgG2b, κ Isotype Ctrl Antibody | BioLegend | 400646 | MOPC-21 (Isotype Control) Dilution: 1:200 |
| CD105 (Endoglin) Monoclonal Antibody APC | eBioscience | 17-1057-42 | HM47 Dilution: 1:200 |
| Cell Counting Chamber Slides | Shanghai QIUJING | XB-K-25 | |
| Centrifuge | Beijing BAIYANG | BY-320C | |
| ChamQ Universal SYBR qPCR Master Mix | Vazyme | Q711-02 | |
| Chloroform | XILONG Scientific | 13700908 | |
| DMEM/F-12 (1:1) basic (1x) | Gibco | C11330500BT | |
| EDTA (0.5 M), pH 8.0, Rnase free | Invitrogen | AM9260G | Dilution: 1:1000 |
| Ethanol | XILONG Scientific | 12803405 | |
| Fetal Bovine Serum (FBS) | Gibco | 10099-141C | |
| FITC Mouse Anti-Human CD34 | BD Bioscience | 555821 | IM7 Dilution: 1:200 |
| FITC Mouse Anti-Human CD45 | BD Bioscience | 555482 | AD2 Dilution: 1:200 |
| FITC Mouse Anti-Human HLA-DR | BD Bioscience | 555811 | SN6 Dilution: 1:200 |
| Flow Cytometer | BD Bioscience | FACSAria™ III Cell SorterAria | |
| Flowjo | BD Bioscience | V10 | |
| Gentle Cell Dissociation Reagent | STEMCELL Technologies | 100-0485 | |
| Goat Anti-Mouse IgG H&L (Alexa Fluor 488) | abcam | ab150113 | HI30 Dilution: 1:200 |
| Goat Anti-Rabbit IgG H&L (AlexaFluor 594) | abcam | ab150080 | Dilution: 1:1000 |
| HiScript II Q RT SuperMix for qPCR (+gDNA wiper) | Vazyme | R223-01 | |
| Inverted Microscopes | Nikon | ECLIPSE Ts2 | |
| Isopropyl Alcohol | XILONG Scientific | 12802505 | |
| MFGE8 antibody | Biorbyt | orb388429 | Dilution: 1:100 |
| Microcentrifuge | Thermo Fisher Scientific | FRESCO 21 | |
| Mouse IgG1 kappa Isotype Control APC | eBioscience | 17-4714-42 | P3.6.2.8.1 (Isotype Control) Dilution: 1:200 |
| Mouse IgG1 kappa Isotype Control FITC | eBioscience | 11-4714-42 | eBMG2b (Isotype Control) Dilution: 1:200 |
| Mouse IgG2b kappa Isotype Control FITC | eBioscience | 11-4732-42 | RTK4530 (Isotype Control) Dilution: 1:200 |
| PBS (10x) | Sangon Biotech (Shanghai) | E607016-0500 | |
| PE-Cy5 Mouse Anti-Human CD90 | BD Bioscience | 555597 | P3.6.2.8.1 (Isotype Control) Dilution: 1:200 |
| PE-Cy5 Mouse IgG1 κ Isotype Control | BD Bioscience | 550618 | |
| Penicillin-Streptomycin 100x | Cytiva | SV30010 | Dilution: 1:100 |
| Real-Time PCR System | Applied Biosystems byThermo Fisher Scientific | Q6 | |
| RNase-free water | QIAGEN | 129112 | |
| Spectrophotometer | Thermo Fisher Scientific | NanoDrop One(840-317400) | |
| Sterile micropipette tips | Beijing Labgic Technology | Dilution: 1:100 | |
| T75 cell culture flask | Beijing Labgic Technology | 13212A | |
| Thermal Cycler | Applied Biosystems byThermo Fisher Scientific | Veriti | |
| Tri reagent | Sigma Aldrich | T9424 | |
| Typsin-EDTA Solution | Bio-Channel Biotechnology | BC-CE-005 | |
| Water-Jacketed CO2 Incubator | Thermo Fisher Scientific | 3111 |
Références
- Liechty, K. W., et al. Human mesenchymal stem cells engraft and demonstrate site-specific differentiation after in utero transplantation in sheep. Nat Med. 6 (11), 1282-1286 (2000).
- Zhou, T., et al. Challenges and advances in clinical applications of mesenchymal stromal cells. J Hematol Oncol. 14 (1), 24 (2021).
- Xie, Z., et al. Tnf-alpha-mediated m(6)a modification of elmo1 triggers directional migration of mesenchymal stem cell in ankylosing spondylitis. Nat Commun. 12 (1), 5373 (2021).
- Mcguire, J. J., et al. Mesenchymal stem cell-derived interleukin-28 drives the selection of apoptosis resistant bone metastatic prostate cancer. Nat Commun. 12 (1), 723 (2021).
- Yuan, X., et al. Mesenchymal stem cell therapy induces flt3l and cd1c(+) dendritic cells in systemic lupus erythematosus patients. Nat Commun. 10 (1), 2498 (2019).
- Wang, D., et al. Umbilical cord mesenchymal stem cell transplantation in active and refractory systemic lupus erythematosus: A multicenter clinical study. Arthritis Res Ther. 16 (2), R79 (2014).
- Ortiz, L. A., et al. Mesenchymal stem cell engraftment in lung is enhanced in response to bleomycin exposure and ameliorates its fibrotic effects. Proc Natl Acad Sci U S A. 100 (14), 8407-8411 (2003).
- Phinney, D. G. Functional heterogeneity of mesenchymal stem cells: Implications for cell therapy. J Cell Biochem. 113 (9), 2806-2812 (2012).
- Wang, Z., et al. Single-cell transcriptome atlas of human mesenchymal stem cells exploring cellular heterogeneity. Clin Transl Med. 11 (12), e650 (2021).
- Chen, P., et al. Single-cell and spatial transcriptomics decodes wharton's jelly-derived mesenchymal stem cells heterogeneity and a subpopulation with wound repair signatures. Adv Sci (Weinh). 10 (4), e2204786 (2023).
- Chen, H., et al. Dissecting heterogeneity reveals a unique bambi(high) mfge8(high) subpopulation of human uc-mscs. Adv Sci (Weinh). 10 (1), e2202510 (2022).
- Tsuji, K., et al. Effects of different cell-detaching methods on the viability and cell surface antigen expression of synovial mesenchymal stem cells. Cell Transplant. 26 (6), 1089-1102 (2017).
- Lai, T. Y., et al. Different methods of detaching adherent cells and their effects on the cell surface expression of fas receptor and fas ligand. Sci Rep. 12 (1), 5713 (2022).
- Avinash, K., Malaippan, S., Dooraiswamy, J. N. Methods of isolation and characterization of stem cells from different regions of oral cavity using markers: A systematic review. Int J Stem Cells. 10 (1), 12-20 (2017).
- Galvao, I., et al. Rock inhibition drives resolution of acute inflammation by enhancing neutrophil apoptosis. Cells. 8 (9), 964 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon