Method Article
Выделение комар-ассоциированного вируса от комаров, собранных в полевых условиях
В этой статье
Резюме
Многочисленные новые вирусоподобные последовательности были обнаружены у комаров из-за широкого использования технологий секвенирования. Мы предлагаем эффективную процедуру выделения и амплификации вирусов с использованием клеточных линий позвоночных и комаров, которая может послужить основой для будущих исследований вирусов, ассоциированных с комарами, включая переносимые комарами и специфические для комаров вирусы.
Аннотация
Благодаря широкому применению технологий секвенирования у членистоногих, включая комаров, было обнаружено много новых вирусоподобных последовательностей. Двумя основными категориями этих новых вирусов, ассоциированных с комарами, являются «вирусы, переносимые комарами (MBV)» и «специфические для комаров вирусы (MSV)». Эти новые вирусы могут быть патогенными как для позвоночных, так и для комаров, или они могут быть просто симбиотическими с комарами. Сущностные вирусы необходимы для подтверждения биологических характеристик этих вирусов. Таким образом, здесь был описан подробный протокол выделения и амплификации вируса от комаров, собранных в полевых условиях. Во-первых, образцы комаров были подготовлены в качестве надосадочных добавок гомогенатов комаров. После двойного центрифугирования надосадочные жидкости затем инокулировали либо в клеточную линию комаров C6/36, либо в клеточную линию позвоночных BHK-21 для амплификации вируса. Через 7 дней надосадочные жидкости собирали как надосадочные жидкости P1 и хранили при -80 °C. Затем надосадочные жидкости P1 были введены еще дважды в клетки C6/36 или BHK-21, в то время как состояние клеток проверялось ежедневно. Когда было обнаружено цитопатогенное действие (CPE) на клетки, эти надосадочные жидкости были собраны и использованы для идентификации вирусов. Этот протокол служит основой для будущих исследований вирусов, ассоциированных с комарами, включая MBV и MSV.
Введение
Комары представляют собой группу важных патогенных членистоногих переносчиков. В семействе Culicidae 1,2 насчитывается около 3 500 видов комаров. Развитие высокопроизводительных технологий секвенирования привело к открытию множества новых вирусоподобных последовательностей у комаров из разных уголков мира3. Как правило, эти вирусы, ассоциированные с комарами, можно разделить на две основные группы: MBV и MSV.
MBV представляют собой группу разнообразных вирусов, которые являются возбудителями многих болезней человека или животных, таких как вирус желтой лихорадки (YFV), вирус денге (DENV), вирус японского энцефалита (JEV), вирус Западного Нила (WNV) и вирус лихорадки Рифт-Валли (RLVFV)4. Они серьезно угрожают здоровью населения, вызывая тяжелую заболеваемость и смертность как среди людей, так и среди животных во всем мире. MBV естественным образом поддерживают жизненный цикл между различными хозяевами путем передачи от инфицированного комара наивному хозяину, а также от инфицированного вирусом хозяина и кормящемуся комару5. Таким образом, эти вирусы могут инфицировать как клеточные линии комаров, так и клеточные линии позвоночных в лаборатории1.
MSV, которые включают вирус Ичан (YCN), флавивирус Culex flavivirus (CxFV) и вирус Чаоян (CHAOV), представляют собой подгруппу вирусов, специфичных для насекомых 1,6,7. В последние годы наблюдается рост числа открытий новых MSV, и было обнаружено, что некоторые из этих MSV влияют на передачу MBV. Например, CxFV, который может быть персистирующей инфекцией в Culex pipiens, может подавлять репликацию ВЗН на ранней стадии8. Было обнаружено, что другой специфический для насекомых флавивирус, вирус клеточного сливающегося агента (CFAV), ингибирует размножение DENV и вируса Зика (ZIKV) у комаров Aedes aegypti 9. Таким образом, этот протокол является полезным подходом для изоляции вирусов, ассоциированных с комарами, и может помочь в дальнейших исследованиях распространения патогенов, связанных с комарами, и борьбы с болезнями, переносимыми комарами.
протокол
1. Отбор проб и сортировка комаров
- Ловите взрослых комаров с помощью световых ловушек MXA-02 или ловушек для комаров с углекислым газом в полевых условиях.
- Убейте собранных комаров, окунув в жидкий азот10,11. Транспортировка их в лабораторию по логистической системе холодовой цепи12.
ПРИМЕЧАНИЕ: Сухой лед в основном использовался в логистической системе холодовой цепи. - (Необязательно) Если места отбора проб находились рядом с лабораторией, отправьте живых комаров в сетчатых ловушках непосредственно в лабораторию и усыпьте их, заморозив при ≤-20 ° C в течение 30 мин4.
- Проведите морфологическую идентификацию собранных комаров с помощью книгиинструментов 13 для классификации и идентификации комаров.
ПРИМЕЧАНИЕ: При необходимости штрих-кодирование ДНК может быть использовано для классификации и идентификации комаров14,15. - В зависимости от дат и участков отбора проб распределите их по разным пулам, а затем храните в пробирках объемом 2-5 мл.
- Нарисуйте таблицу, чтобы записать каждую пробирку с видами комаров, количеством, кодом выборки, датой и местами.
- Распечатайте этикетки с кодами отбора проб и прикрепите их к пробиркам.
- Храните пробирки с образцами комаров при температуре -80 °C до дальнейшего анализа.
2. Измельчение комаров
- Поместите 20-50 комаров в каждую стерильную пробирку объемом 2 мл с керамическими шариками диаметром 3 мм с 1,5 мл среды Мемориального института Розуэлл-Парк (RPMI), содержащей 2% раствор пенициллина-стрептомицина-амфотерицина B.
ПРИМЕЧАНИЕ: Держите тюбики на подносе со льдом или на подносе. - Гомогенизируйте комаров с помощью низкотемпературного тканевого гомогенизатора путем измельчения в течение 30 с при 70 Гц при 4 °C в течение трех циклов16.
- Центрифугируют смеси при 15 000 × г в течение 30 мин при 4 °C и переносят надосадочные жидкости в новые пробирки.
- Снова центрифугируйте надосадочные жидкости при 15 000 × г в течение 10 мин при 4 °C, чтобы удалить остатки комаров12,17.
ПРИМЕЧАНИЕ: При необходимости надосадочные жидкости можно фильтровать с помощью фильтров 0,22 мкм. - Аликвотируйте надосадочные жидкости (P0) в пробирки для хранения с завинчивающейся крышкой объемом 2 мл (200 мкл на пробирку) и храните их при -80 ° C.
3. Подготовка клеток и среды для содержания клеточных культур
ПРИМЕЧАНИЕ: Для амплификации и выделения вируса использовали клеточную линию комаров C6/36 (дефицит РНК Aedes albopictus ) и клеточную линию позвоночных BHK-21 (почка детеныша хомяка).
- Инокулируют 1 × 10клеток 6 C6/36 в колбу размером 75 см 2 с 10 мл среды RPMI с добавлением 10% эмбриональной бычьей сыворотки (FBS) и 1% пенициллина/стрептомицина (P/S) при 28 °C в 5% увлажненном инкубаторе CO2.
- Инокулируйте 1 × 106 клеток BHK-21 в колбу размером 75 см 2 с 10 мл модифицированной среды Dulbecco Eagle (DMEM) с добавлением 10% FBS и 1% P / S при 37 ° C в увлажненном инкубаторе 5% CO2.
ПРИМЕЧАНИЕ: Клетки проходили два или три раза в неделю17,18. - Засейте клетки, культивируемые в колбе размером75 см 2 (ячейки C6/36 или BHK-21), в 24-луночные планшеты (C6/36 на лунку: 1,4 × 105; БХК-21 на скважину: 0,8 × 105).
- Поместите пластины с 24 лунками при температуре 28 °C или 37 °C на ночь.
- Наблюдайте за клетками под микроскопом и убедитесь, что слияние клеток в каждой лунке составляет 80-90%.
- Подготовьте питательную среду для клеточной культуры (500 мл).
ПРИМЕЧАНИЕ: Для C6/36 средой была среда RPMI с добавлением 2% FBS и 2% раствора пенициллина-стрептомицина-амфотерицина B. Для BHK-21 средой была среда DMEM, дополненная 2% FBS и 2% раствором пенициллина-стрептомицина-амфотерицина B.
4. Изоляция вирусов
ПРИМЕЧАНИЕ: Все этапы были выполнены в лаборатории уровня биобезопасности 2 (BSL-2). Требования к уровню безопасности лаборатории биобезопасности были определены оценкой риска биобезопасности на основе нормативных актов разных стран и регионов. Процесс должен выполняться в шкафу биобезопасности.
- Извлеките питательную среду из 24-луночных планшетов и добавьте 100 мкл среды для поддержания клеточных культур и 100 мкл надосадочной жидкости гомогената комаров (P0) в каждую лунку.
- Инкубируйте пластины при 28 ° C или 37 ° C в течение 60 минут и осторожно встряхивайте их каждые 15 минут, чтобы предотвратить высыхание клеток.
- Удалите надосадочную жидкость и аккуратно промойте каждую лунку 600 мкл среды для поддержания клеточных культур, чтобы полностью удалить мусор.
- Добавьте 800 мкл среды для поддержания клеточных культур в каждую лунку и храните планшеты в увлажненном инкубаторе с 5% CO2 при 28 ° C или 37 ° C в течение 7 дней.
- Ежедневно контролируйте состояние клеток каждой лунки под микроскопом15.
- Собирают надосадочные жидкости клеток (супернатанты Р1) на7-е сутки и хранят надосадочные жидкости при -80 °С.
- Повторите шаги 4.1-4.6 2 раза, чтобы получить надосадочные жидкости P2 и P3.
ПРИМЕЧАНИЕ: На этапе 4.3 для P2 и P3 было необязательным промывание каждой лунки 600 мкл среды для поддержания клеточных культур. - В зависимости от того, какая скважина проявляет цитопатогенное действие (КПЭ) — клетки погибают, пикнозируют и отрываются от поверхности; слияние с соседними клетками с образованием синцитии; или появление ядерных или цитоплазматических тельцов включения — добавьте 300-400 мкл надосадочной жидкости этого CPE в каждую лунку новых 6-луночных пластин, содержащих клетки (слияние клеток составило 80%-90%), чтобы значительно амплифицировать вирусы.
- Соберите надосадочные жидкости и аликвотируйте их в пробирки для хранения с завинчивающейся крышкой объемом 2 мл (500 мкл на пробирку). Храните их при температуре -80 °C.
ПРИМЕЧАНИЕ: После трех поколений последовательного пассажа в клетках образцы, которые не индуцировали CPE, были отброшены. При необходимости поколения последовательного пассажа в клетках могут быть увеличены.
5. Обнаружение вирусных последовательностей методом ОТ-ПЦР
- Извлеките общую РНК из 200 мкл вирусной надосадочной жидкости с помощью мини-наборавирусной РНК 16.
ПРИМЕЧАНИЕ: Здесь использовалась автоматизированная система экстракции нуклеиновых кислот для извлечения общей РНК в соответствии с инструкциями производителя прибора. - Преобразуйте РНК в кДНК в соответствии с инструкциями набора ОТ-ПЦР с помощью прибора ПЦР.
- Добавьте 15 мкл РНК, смешанной с 1 мкл случайных праймеров, в пробирку для ПЦР. Поместите пробирку при температуре 70 °C на 5 минут, а затем поместите ее на лед на 5 минут.
- Добавьте 5 мкл 5-кратного буфера и 25 мкл смеси (10 мМ dNTP, 40 ЕД/мкл ингибитора РНКазы и 200 ЕД/мкл m-mlv) в пробирку и поместите пробирку при 42 ° C на 60 мин и 95 ° C в течение 10 мин.
- Хранить пробирку при температуре -20 °C.
- Используйте набор для ПЦР и универсальные праймеры (табл. 1) для выявления вирусов методом ПЦР.
- Для арбовирусов установите реакционную систему ПЦР (всего 30 мкл) со следующими компонентами: 3 мкл 10-кратного буфера Taq, 3 мкл dNTP (1 мМ), 1 мкл прямого праймера (10 мкм), 1 мкл обратного праймера (10 мкм), 0,3 мкл Taq (10 Ед/мкл), 19,7 мкл ddH 2 O и2мкл кДНК.
- Для обнаружения флавивирусов настройте процесс реакции следующим образом: стадия 1: 35 циклов 94 °C в течение 30 с, 52 °C в течение 30 с и 72 °C в течение 30 с; стадия 2: 72 °C в течение 10 мин.
- Для обнаружения альфавирусов настройте процесс реакции следующим образом: стадия 1: 35 циклов 94 °C в течение 30 с, 50 °C в течение 30 с и 72 °C в течение 30 с; стадия 2: 72 °C в течение 10 мин.
- Для обнаружения буньявирусов настройте процесс реакции следующим образом: стадия 1: 35 циклов 94 °C в течение 30 с, 50 °C в течение 30 с и 72 °C в течение 30 с; стадия 2: 72 °C в течение 10 мин.
- Выявляют продукты амплификации ПЦР с помощью 1% электрофореза в агарозном геле и отправляют их на секвенирование.
ПРИМЕЧАНИЕ: Если позволяют условия, надосадочные жидкости могут быть подвергнуты анализу секвенирования следующего поколения для идентификации новых вирусов и получения целых секвенций вирусного генома.
Результаты
После инокуляции надосадочными продуктами гомогенатов комаров (P0) клетки C6/36 демонстрировали широкое межклеточное пространство, а отслоившиеся клетки наблюдались через 120 ч (рис. 1A) по сравнению с неинокулированными клетками (контроль) в то же время (рис. 1B). После инкубации клеток BHK-21 с надосадочными продуктами P3 в клетках BHK-21 наблюдался видимый CPE через 48 ч (рис. 1C) в отличие от контрольных клеток (рис. 1D). Для определения вида вируса проводили ПЦР. Коммерчески синтезированы универсальные праймеры для обнаружения флавивирусов, альфавирусов и буньявирусов (табл. 1). Продукт ПЦР для вируса буньявируса озера Эбинур был установлен в качестве положительного контроля и добавлен в полосу 1. Универсальные праймеры для буньявирусов, флавивирусов и альфавирусов использовались для получения продуктов ПЦР для вирусного супернатанта, которые затем добавлялись в полосы 2, 3 и 4 соответственно. Предполагаемые размеры продуктов ПЦР для флавивирусов, альфавирусов и буньявирусов составляли 266.н., 434.н. и 251.н. соответственно. Полосы были видны только на полосе 2 и полосе 1 с положительным контролем. В результате вирус в надосадочной жидкости, скорее всего, является буньявирусом (рис. 2).

Рисунок 1: Наблюдение CPE за клетками после инкубации с вирусными надосадочными продуктами. Состояние клеток C6/36 через 120 ч после заражения (CPE) (A) и контрольных клеток в то же время (B). Состояние ячеек BHK-21 при 48 hpi (C) и контрольных ячеек (D). Масштабные линейки = 100 мкм. Сокращения: CPE = цитопатогенный эффект; HPI = количество часов после заражения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
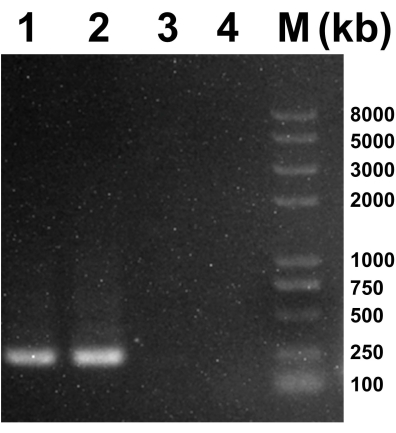
Рисунок 2: Результаты ПЦР для вирусных супернатантов. Полоса 1 представляет собой положительный контроль буньявирусов (вирус озера Эбинур). Полосы 2-4 представляли результаты ПЦР супернатантов с использованием универсальных праймеров для буньявирусов (251.н.), флавивирусов (266.н.) и альфавирусов (434.н.). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| Вирус | Букварь | Олигонуклеотид (5'→3') | Размер продукта ПЦР (.н.) | ||
| Флавивирусы | Формула-1 | TACAACATGATGGGAAAGAGAGAA | 266 | ||
| Ф2 | GTGTCCCAGCCGGCGGTCATCAGC | ||||
| Альфавирусы | M2w | YAGAGCDTTTTCGCAYSTRGCHW | 434 | ||
| cMw3 | ACATRAANKGNGTNGTRTCRAANCCDAYCC | ||||
| Буньявирусы | БКС82С | ATGACTGAGTGGAGTTTCATGATGTCGC | 251 | ||
| БКС332В | TGTTCCTGTTGCCAGGAAAAT | ||||
Таблица 1: Универсальные праймеры для обнаружения арбовирусов.
Обсуждение
Цель этого метода состояла в том, чтобы предложить практический способ выделения вирусов, ассоциированных с комарами, с использованием различных клеточных линий. Крайне важно добавлять антибиотик-антимикотик (пенициллин-стрептомицин-амфотерицин) к надосадочным продуктам гомогенатов комаров, чтобы избежать заражения бактериями или грибками. Комары и вирусные надосадочные жидкости, полученные в полевых условиях, должны храниться в холодильнике при -80 °C, чтобы избежать повторных циклов замораживания-оттаивания.
Еще одним важным шагом в протоколе была шлифовка. Образцы комаров должны быть тщательно измельчены в порошок и храниться на льду. Недостаточно измельченные ткани комаров могут вызвать гибель клеток и могут исказить результаты анализа CPE. Перед выделением вируса необходимо использовать нормальных комаров для подтверждения параметров измельчения. Измельчение должно производиться при низких температурах, чтобы предотвратить неактивность вируса во время процесса.
Этот подход может эффективно изолировать и амплифицировать вирусы, ассоциированные с комарами, но приемлем только для вирусов, которые могут проявлять CPE в клеточных линиях млекопитающих или насекомых. Этот подход не подходит для вирусов, которые не индуцируют CPE. Используя протокол, мы можем получить вирусных кандидатов, которые содержат только один тип вируса или смесь с разными вирусами. Для получения очищенного вируса по-прежнему необходимы дальнейшие исследования — очистка вируса, морфологическая идентификация и анализ бляшек.
Раскрытие информации
У авторов нет конфликтов интересов, которые необходимо раскрывать.
Благодарности
Эта работа была поддержана Уханьским проектом научно-технического плана (2018201261638501).
Материалы
| Name | Company | Catalog Number | Comments |
| 0.22 µm membrane filter | Millipore | SLGP033RB | Polymer films with specific pore ratings.To remove cell debris and bacteria. |
| 24-well plates | CORNING | 3524 | Containers for cell |
| 75 cm2 flasks | CORNING | 430641 | Containers for cell |
| a sterile 2 mL tube with 3 mm ceramic beads | |||
| Antibiotic-Antimycotic | Gibco | 15240-062 | Antibiotic in the medium to prevent contamination from bacteria and fungi |
| Automated nucleic acid extraction system | NanoMagBio | S-48 | |
| BHK-21 cells | National Virus Resource Center, Wuhan Institute of Virology | ||
| C6/36 cells | National Virus Resource Center, Wuhan Institute of Virology | ||
| Centrifugal machine | Himac | CF16RN | Instrument for centrifugation of mosquito samples |
| CO2 | |||
| Dulbecco’s minimal essential medium (DMEM) | Gibco | C11995500BT | medium for vertebrate cell lines |
| Ebinur Lake virus | Cu20-XJ isolation | ||
| Feta Bovine Serum (FBS) | Gibco | 10099141C | Provide nutrition for cells |
| high-speed low-temperature tissue homogenizer | servicebio | KZ-III-F | Instrument for grinding |
| incubator (28 °C) | Panasonic | MCO-18AC | Instrument for cell culture |
| incubator (37 °C) | Panasonic | MCO-18AC | Instrument for cell culture |
| PCR tube | |||
| penicillin-streptomycin | Gibco | 15410-122 | Antibiotic in the medium to prevent contamination from bacteria |
| Penicillin-Streptomycin-Amphotericin B Solution | Gibco | 15240096 | |
| Refrigerator (-80 °C) | sanyo | MDF-U54V | |
| Roswell Park Memorial Institute medium (RPMI) | Gibco | C11875500BT | medium for mosiquto cell lines |
| Screw cap storage tubes (2 mL) | biofil | FCT010005 | |
| sterile pestles | Tiangen | OSE-Y004 | Consumables for grinding |
| TGrinder OSE-Y30 electric tissue grinder | Tiangen | OSE-Y30 | Instrument for grinding |
| The dissecting microscope | ZEISS | stemi508 | |
| the light traps MXA-02 | Maxttrac | ||
| The mosquito absorbing machine | Ningbo Bangning | ||
| The pipette tips | Axygen | TF | |
| The QIAamp viral RNA mini kit | QIAGEN | 52906 | |
| Tweezers | Dumont | 0203-5-PO |
Ссылки
- Xia, H., Wang, Y., Atoni, E., Zhang, B., Yuan, Z. Mosquito-associated viruses in China. Virologica Sinica. 33 (1), 5-20 (2018).
- Atoni, E., et al. A dataset of distribution and diversity of mosquito-associated viruses and their mosquito vectors in China. Scientific Data. 7 (1), 342 (2020).
- Atoni, E., et al. The discovery and global distribution of novel mosquito-associated viruses in the last decade (2007-2017). Reviews in Medical Virology. 29 (6), 2079 (2019).
- Xia, H., et al. Comparative metagenomic profiling of viromes associated with four common mosquito species in China. Virologica Sinica. 33 (1), 59-66 (2018).
- Ong, O. T. W., Skinner, E. B., Johnson, B. J., Old, J. M. Mosquito-borne viruses and non-human vertebrates in Australia: A review. Viruses. 13 (2), 265 (2021).
- Agboli, E., Leggewie, M., Altinli, M., Schnettler, E. Mosquito-specific viruses-transmission and interaction. Viruses. 11 (9), 873 (2019).
- Halbach, R., Junglen, S., van Rij, R. P. Mosquito-specific and mosquito-borne viruses: evolution, infection, and host defense. Current Opinion in Insect Science. 22, 16-27 (2017).
- Bolling, B. G., Olea-Popelka, F. J., Eisen, L., Moore, C. G., Blair, C. D. Transmission dynamics of an insect-specific flavivirus in a naturally infected Culex pipiens laboratory colony and effects of co-infection on vector competence for West Nile virus. Virology. 427 (2), 90-97 (2012).
- Baidaliuk, A., et al. Cell-fusing agent virus reduces arbovirus dissemination in Aedes aegypti mosquitoes in vivo. Journal of Virology. 93 (18), 00715-00719 (2019).
- Atoni, E., et al. Metagenomic virome analysis of Culex mosquitoes from Kenya and China. Viruses. 10 (1), 30 (2018).
- Xia, H., et al. First isolation and characterization of a group C Banna virus (BAV) from Anopheles sinensis mosquitoes in Hubei, China. Viruses. 10 (10), 555 (2018).
- Shi, C., et al. Stability of the virome in lab- and field-collected Aedes albopictus mosquitoes across different developmental stages and possible core viruses in the publicly available virome data of Aedes mosquitoes. mSystems. 5 (5), 00640 (2020).
- Zhou, M., Chu, H. . Handbook for Classification and Identification of Main Vectors. , (2019).
- Wang, G., et al. Identifying the main mosquito species in China based on DNA barcoding. Plos One. 7 (10), (2012).
- Ratnasingham, S., Hebert, P. D. N. Bold: The Barcode of Life Data System (www.barcodinglife.org). Molecular Ecology Notes. 7 (3), 355-364 (2007).
- Huang, Y., et al. In vitro and in vivo characterization of a new strain of mosquito Flavivirus derived from Culicoides. Viruses. 14 (6), 1298 (2022).
- Zhao, L., et al. Characterization of a novel Tanay virus isolated from Anopheles sinensis mosquitoes in Yunnan, China. Frontiers in Microbiology. 10, 1963 (1963).
- Ren, N., et al. Characterization of a novel reassortment Tibet orbivirus isolated from Culicoides spp. in Yunnan, PR China. Journal of General Virology. 102 (9), 001645 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены