Method Article
Construção de vetores adenovirais usando tecnologia de montagem de DNA
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Neste estudo, um clone infeccioso de adenovírus humano tipo 7 (HAdV-7) foi construído, e um sistema vetorial HAdV-7 deletado por E3 foi estabelecido modificando o clone infeccioso. Essa estratégia usada aqui pode ser generalizada para fazer vetores de transferência de genes de outros adenovírus do tipo selvagem.
Resumo
Os vetores adenovirais têm sido usados como ferramenta de transferência de genes na terapia gênica há mais de três décadas. Aqui, apresentamos um protocolo para construir um vetor adenoviral manipulando o DNA genômico do HAdV-7 do tipo selvagem usando um método de montagem de DNA. Primeiro, um clone infeccioso de HAdV-7, pKan-Ad7, foi gerado pela fusão do DNA genômico viral com um produto de PCR da estrutura do plasmídeo, compreendendo o gene resistente à canamicina e a origem da replicação (Kan-Ori), por meio da montagem do DNA. Isso foi feito projetando um par de primers de PCR, que continham ~ 25 nucleotídeos da sequência terminal de HAdV-7 invertida terminal (ITR) na extremidade 5 ', um local de enzima de restrição sem cortador para o genoma HAdV-7 no meio e uma sequência específica do modelo para PCR priming na extremidade 3 '. Em segundo lugar, uma estratégia intermediária baseada em plasmídeo foi empregada para substituir a região E3 por elementos que expressam transgenes no clone infeccioso para gerar um vetor adenoviral. Resumidamente, pKan-Ad7 foi digerido com enzima de restrição de corte duplo Hpa I, e o fragmento contendo a região E3 foi ligado a outro produto de PCR do esqueleto do plasmídeo por montagem de Gibson para construir um plasmídeo intermediário pKan-Ad7HpaI. Por conveniência, a montagem de restrição foi usada para designar o método de clonagem de plasmídeo de digestão e montagem de restrição combinada. Usando montagem de restrição, os genes E3 em pKan-Ad7HpaI foram substituídos por um de expressão de proteína fluorescente verde (GFP), e a região E3 modificada foi liberada do plasmídeo intermediário e restaurada ao clone infeccioso para gerar um plasmídeo adenoviral pKAd7-E3GFP. Finalmente, pKAd7-E3GFP foi linearizado por digestão de Pme I e usado para transfectar células de empacotamento HEK293 para resgatar o vírus HAdV-7 recombinante. Para concluir, uma estratégia baseada em montagem de DNA foi introduzida aqui para a construção de vetores adenovirais em laboratórios gerais de biologia molecular sem a necessidade de materiais e instrumentos especializados.
Introdução
Nas últimas três décadas, os vetores adenovirais recombinantes têm sido amplamente utilizados no desenvolvimento de vacinas e terapia gênica 1,2,3,4 bem como na pesquisa básica devido às suas excelentes propriedades biológicas, como alta eficiência de transdução gênica, não integração ao genoma do hospedeiro, genoma viral manipulativo e facilidade de produção em larga escala.
Atualmente, os vetores adenovirais mais comumente utilizados são construídos com base no adenovírus humano 5 (HAdV-5)5,6. Embora a transdução mediada por vetor HAdV-5 forneça resultados encorajadores, as aplicações pré-clínicas e clínicas revelaram várias desvantagens (por exemplo, alta imunidade antivetorial pré-existente na população humana e baixa eficiência de transdução em células sem o vírus coxsackie e o receptor de adenovírus (CAR)). Para contornar esses problemas, tem havido um grande interesse em construir vetores baseados em outros tipos de adenovírus humanos ou de mamíferos 3,7,8.
Até agora, o método mais popular para construir um vetor adenoviral é a recombinação homóloga em bactérias5. Essas cepas bacterianas devem expressar recombinases, o que pode afetar a estabilidade ou amplificação dos plasmídeos que carregam. Algumas cepas estão até mesmo comercialmente indisponíveis. Recentemente, métodos baseados em outros princípios, incluindo cromossomos artificiais bacterianos, clonagem direta ou montagem direta de DNA, têm sido empregados para gerar clones infecciosos de adenovírus ou vetores adenovirais recombinantes 9,10,11,12. No entanto, esses métodos são um tanto hostis para pesquisadores com pouca experiência neste campo.
Em 2018, o processo de construção de um clone infeccioso de adenovírus é simplificado em laboratório, ligando diretamente o genoma do vírus a um produto de PCR que transporta a estrutura do plasmídeo através da montagem de Gibson13. Depois disso, os métodos de digestão de restrição e montagem de Gibson são combinados para carregar transgenes em plasmídeos adenovirais existentes 14,15,16,17,18. Por uma questão de conveniência, a montagem de restrição é usada a seguir para se referir ao método de digestão combinada da enzima de restrição e montagem de Gibson. Estratégias foram desenvolvidas para construir vetores adenovirais a partir de clones infecciosos usando montagem de restrição19. A essência da montagem de restrição é incluir fragmentos extirpados de plasmídeos tanto quanto possível em uma reação de montagem de DNA, enquanto produtos de PCR curtos servem como ligantes ou patches para modificação de plasmídeo. Ao mesmo tempo, o número de fragmentos incluídos é mantido o mais baixo possível. Tais esforços refletem a recompensa; a possibilidade de mutações indesejadas causadas por PCR ou montagem de DNA pode ser minimizada e a taxa de sucesso pode ser melhorada. Conclusivamente, um pipeline de um adenovírus de tipo selvagem para um vetor adenoviral foi estabelecido em laboratório 13,14,15,16,17,18,19.
Aqui, tentamos apresentar esses métodos fornecendo exemplos de construção de um clone infeccioso HAdV-7 e um vetor HAdV-7 competente para replicação deletado por E3.
Protocolo
NOTA: Os adenovírus são classificados como Nível de Biossegurança 2 (BSL-2); Todas as etapas para o uso dos vírus foram realizadas em um laboratório de nível de biossegurança 2. O HAdV-7 do tipo selvagem foi isolado em 2017 a partir da amostra de aspirado nasofaríngeo de um bebê de 10 meses que foi hospitalizado com infecção aguda do trato respiratório no Hospital Infantil de Pequim20. Os estoques de vírus foram armazenados a -80 °C.
1. Extração do DNA genômico HAdV-7
- Semear 2,0 x 106 das células HEK293 em um frasco T-25 contendo 5 mL de meio de Eagle modificado de Dulbecco (DMEM) mais 10% de soro fetal bovino (FBS) e cultura a 37 °C em atmosfera umidificada suplementada com 5% de CO2. As células devem estar 80% a 90% confluentes e prontas para infecção em 18-24 h.
- Inocular as células com HAdV-7 de tipo selvagem durante 2 h. Remova o meio contendo vírus por aspiração e adicione 5 mL de DMEM fresco contendo 2% de FBS às células.
- Extrair o DNA genômico viral com o método de Hirt modificado21
- Em 2 ou 3 dias após a infecção, quando o efeito citopático (CPE) for observado em 50% a 90% das células, descarte o sobrenadante da cultura e enxágue as células uma vez com solução salina tamponada com fosfato (PBS).
NOTA: O efeito citopático (CPE) é definido como um fenômeno em que as células infectadas pelo vírus se arredondam, incham e se fixam ou se desprendem frouxamente do frasco de cultura em cachos semelhantes a uvas22. - Lise as células no frasco em 1,0 mL de tampão de lise contendo 25 mM de Tris-HCl, 0,5 mM de EDTA, 50 μg/mL de protease K e 0,8% de SDS (pH 7,6) por 5 min em gelo. Transferir o lisado para um tubo de microcentrífuga de 1,5 ml e incubar o tubo em banho-maria a 50 °C durante 30 min.
- Adicione 120 μL de tampão de precipitação ao tubo. Coloque o tubo no gelo por mais 30 minutos depois de misturar suavemente.
NOTA: O tampão de precipitação é composto por 3 M CsCl, 1 M de acetato de potássio e 0.67 M de ácido acético. Isso ajudará a precipitar os detritos celulares e o DNA do cromossomo celular após a centrifugação, mantendo o genoma do vírus no sobrenadante. - Centrifugue o tubo por 25 min a 15.000 x g a 4 ° C e, em seguida, transfira o sobrenadante (cerca de 1,1 mL) para um tubo de polipropileno de 15 mL.
- Adicione 2,2 mL de etanol absoluto gelado ao tubo. Misture delicadamente e deixe o tubo repousar no gelo por 15 min. Centrifugue por 5 min a 2000 x g em temperatura ambiente.
- Aspirar cuidadosamente o sobrenadante e lavar o precipitado (ADN genómico do vírus) uma vez com 800 μL de etanol a 70%. Centrifugue o tubo por 6 min a 15.000 x g em temperatura ambiente. Rejeitar o sobrenadante e secar ao ar o sedimento de ADN na parte inferior. Não seque demais o pellet de DNA, pois pode causar dificuldade em dissolver.
- Dissolva o pellet em 200 μL de tampão TE (10 mM Tris-HCl, pH 8,0, 0,1 mM EDTA), adicione 1 μL de RNase A (10 mg / mL) e incube o tubo a 37 ° C por 15 min. Purifique o DNA dissolvido usando um kit comercial de limpeza e concentrador de DNA genômico (consulte a Tabela de Materiais).
NOTA: Quantifique o DNA genômico viral com o método comumente usado em laboratório. Geralmente, 1-5 μg de DNA genômico podem ser obtidos em um frasco T-25 de células HEK293 infectadas com adenovírus.
- Em 2 ou 3 dias após a infecção, quando o efeito citopático (CPE) for observado em 50% a 90% das células, descarte o sobrenadante da cultura e enxágue as células uma vez com solução salina tamponada com fosfato (PBS).
2. Construção de clone infeccioso de HAdV-7 por montagem de DNA
- Projete primers de PCR para amplificar o esqueleto do plasmídeo (Figura 1). Adicione as sequências de sobreposição necessárias para a montagem do DNA às extremidades 5 'dos primers de PCR. Além disso, inclua locais de restrição sem corte (por exemplo, local Pme I) em ambas as extremidades do genoma do HAdV-7 para ajudar a liberar o genoma viral do clone infeccioso.
NOTA: A estrutura do plasmídeo contendo o gene resistente a antibióticos e a origem da replicação é amplificada por PCR e fundida ao genoma do HAdV-7. Os adenovírus podem ser resgatados de células de empacotamento com maior eficiência quando o genoma do vírus linearizado é usado em vez de um plasmídeo adenoviral circular. Depois de considerar os fatores mencionados acima, os primers são projetados conforme mostrado na Figura 1. - Realize PCR para amplificar um fragmento (Kan-Ori) contendo o gene de resistência à canamicina (Kan) e a origem do pBR322 (Ori) usando pShuttle-CMV como modelo com primers de Ad7KanF1 e Ad7KanR1 (Tabela S1). Defina a reação de PCR da seguinte forma: um ciclo a 98 °C por 30 s, depois cinco ciclos de desnaturação a 98 °C por 8 s, recozimento a 68 °C por 30 s, extensão a 72 °C por 90 s e, finalmente, 25 ciclos de desnaturação a 98 °C por 8 s, recozimento e extensão a 72 °C por 2 min.
- Execute o produto de PCR em um gel de agarose a 1% por eletroforese e purifique o fragmento usando um kit comercial de recuperação de gel de DNA.
- Adicione o Kan-Ori recuperado e o DNA genômico viral do HAdV-7 ao Master Mix de Montagem de DNA comercial. Configure a reação de montagem em um volume total de 20 μL e uma proporção molar de vetor para DNA genômico viral de 1: 2. Incubar a 50 °C durante 1 h.
NOTA: Defina a reação em um volume total de 20 μL com 1-3 μL de vetor, 7-9 μL de DNA genômico viral e 10 μL de DNA Assembly Master Mix. - Transforme as células TOP10 da cepa de E. coli quimicamente competentes com 10 μL do produto de montagem usando choque térmico. Espalhe a mistura de transformação em placas Luria-Bertani (LB-Kan) contendo canamicina (50 μg/mL). Incubar as placas a 37 °C durante, pelo menos, 12 h.
- Inocular pelo menos três clones cada um em 5 ml de meio LB-Kan a 37 °C durante 12 h com agitação suave (200-250 rpm). Extraia o DNA do plasmídeo usando um kit comercial Plasmid Miniprep.
- Digerir o plasmídeo utilizando endonuclease de restrição e, em seguida, verificar o tamanho dos fragmentos de ADN com electroforese em gel de agarose (figura 2A). Além disso, confirme os locais de fusão por sequenciamento. O plasmídeo de montagem correto é denominado pKan-Ad7.
- Cultive uma cultura de 100 mL de bactérias a partir do clone correto. Extraia uma preparação de maxi-plasmídeo usando um kit comercial Plasmid Maxprep.
3. Construção in silico e modificação de um plasmídeo intermediário
NOTA: Geralmente, um plasmídeo intermediário deve ser construído, e como construir um plasmídeo intermediário ideal é o passo principal. Software de análise de DNA com um módulo de enzima de restrição, como o pDRAW32 (um software gratuito, disponível em www.acaclone.com), é necessário para o projeto de um plasmídeo intermediário.
- Importe a sequência de DNA de pKan-Ad7 para pDRAW32 para desenhar um mapa de plasmídeo. Anote as regiões de interesse no mapa. Para pKan-Ad7, as regiões E1, E3 e o backbone do plasmídeo (Kan-Ori) foram anotados.
- Opere o programa pDRAW32. Clique em Configurações > Seleção de enzimas primeiro e, em seguida, clique em Cortes mínimos/máximos. Insira 1 na caixa Min e insira 2 na caixa Max e marque a caixa antes em toda a sequência. Por fim, clique em OK para mostrar os locais de enzimas de restrição de cortador único e duplo no mapa.
NOTA: Geralmente, apenas as enzimas de restrição que reconhecem uma sequência de 6 pb ou mais são selecionadas. Conforme mostrado na Figura 3A, 17 enzimas correspondem aos critérios de seleção enzimática. - Considere o primeiro local de restrição que flanqueia a região E3 desde que a E3 está planejada para ser modificada. Hpa I, Sal I, Avr II-Mlu I e Spe I satisfizeram o requisito.
NOTA: Para este experimento, Hpa I é selecionado porque a digestão de pKan-Ad7 com ele geraria o fragmento mais curto carregando a região E3, e um plasmídeo intermediário menor facilitaria a modificação futura. A seleção de sítios Sal I também tem vantagens - tal plasmídeo intermediário é adequado para a modificação das regiões E1 e E3 (Figura S1). - Crie um arquivo de sequência final exibindo ambas as fitas de DNA do plasmídeo intermediário pKan-Ad7HpaI. Essa sequência virtual pode ser usada como modelo para projetar primers sobrepostos.
- Copie a sequência entre os dois locais de restrição Hpa I, incluindo a região E3. Para montagem subsequente, é necessário copiar cada sequência de ~20 pb (Tm igual ou superior a 48°C) fora do local de restrição de Hpa I (Figura 3C, sobreposição-Final 1 e sobreposição-Final2).
- Adicione a sequência de reconhecimento de Pme I (Figura 3C, gtttaaac) em ambas as extremidades da sequência acima.
- Adicione a sequência de plasmídeo de backbone contendo gene resistente a antibióticos e origem de replicação (Figura 3C, Kan-Ori) fora do local de restrição de Pme I.
- Importe a sequência final para pDRAW32 para desenhar um mapa de plasmídeo. Anote a região E3 no mapa.
- Opere o programa pDRAW32. Clique em Configurações > Seleção de Enzimas primeiro e, em seguida, clique em Cortes Mín/Máximo. Insira 1 na caixa Min e Max e marque a caixa antes em toda a sequência. Clique em OK por último para mostrar os locais exclusivos da enzima de restrição no mapa. Pode-se ver que muitos cortadores exclusivos estão disponíveis para modificação do E3.
NOTA: Para este experimento, BstZ17 I e Mlu I são selecionados para a substituição de E3 parcial. Um cortador duplo também pode ser útil. Por exemplo, o cortador duplo Ssp I junto com o cortador exclusivo BssH II pode ser selecionado para a substituição de toda a região E3 usando PCR de extensão de sobreposição.
- Projete primers usando a sequência virtual como modelo para a construção de plasmídeo intermediário ( Figura 3C ).
NOTA: Para encurtar o comprimento de cada primer, quatro primers foram projetados. O produto de PCR, que é amplificado com primers de Ad7HpaIF1 e Ad7HpaIR1, seria usado como molde para obter o fragmento final de PCR com primers de Ad7HpaIF2 e Ad7HpaIR2 (Figura 3C).
4. Resgate de adenovírus recombinante infeccioso em células HEK293
NOTA: Os três adenovírus gerados neste artigo são todos resgatados de acordo com o protocolo a seguir.
- Digerir 10 μg de plasmídeo adenoviral recombinante com 2 μL de Pme I durante 5 h a 37 °C num volume total de 100 μL para linearizar o plasmídeo. Verifique 1 μL do DNA digerido em um gel de agarose a 0,8%. A digestão deve produzir um fragmento de ~ 34 kb do genoma adenoviral e uma estrutura de plasmídeo de ~ 2,5 kb de Kan-Ori.
- Purifique o plasmídeo adenoviral linearizado remanescente usando o kit genômico DNA Clean and Concentrator.
- Cultive células HEK293 em DMEM mais 10% de FBS. Semeie células HEK293 (2 x 10,6 células) em um frasco T-25 1 dia antes da transfecção, para que atinjam 70% -80% de confluência no dia seguinte. Use 5 μg de plasmídeo adenoviral digerido por Pme I para transfectar células HEK293 usando reagentes de transfecção disponíveis comercialmente.
NOTA: O meio das células transfectadas deve ser substituído a cada 3 dias. - Verifique as células todos os dias, colete as células e o meio de cultura em um tubo de polipropileno de 15 mL quando os focos do cometa se formarem (Figura 2B, indicando replicação do vírus).
- Lise as células por meio de três ciclos de congelamento e descongelamento. Após centrifugação durante 12 min a 1200 x g à temperatura ambiente, recolher o sobrenadante e armazená-lo a -80 °C para posterior propagação do vírus.
NOTA: Quando o adenovírus recombinante expressa GFP ou outras proteínas fluorescentes, é mais fácil identificar se o vírus foi resgatado com sucesso, pois os focos formados por células positivas para proteínas fluorescentes podem ser observados intuitivamente em um microscópio de fluorescência.
5. Construção do plasmídeo do genoma HAdV-7 deletado por E3
NOTA: O genoma do HAdV-7 tem 35.239 pb de comprimento e a região E3 está localizada entre 27.354 pb e 31.057 pb do genoma. Para construir um plasmídeo intermediário, encontre uma endonuclease com dois locais de corte no plasmídeo do clone infeccioso ou duas endonucleases com um único local de corte no plasmídeo. Existem Hpa I, Sal I ou Avr II e Mlu I disponíveis. A sequência entre BstZ17 I (28.312 pb) e Mlu I (30.757 pb) deve ser excluída e substituída pelo CMV-GFP-PA ou CMV-mCherry-PA.
- Digerir 600 ng de pKan-Ad7 com Hpa I. Purificar o fragmento grande pKAd7-HpaI-FL (~30 kb) e o fragmento pequeno pKAd7-HpaI-FS (~7 kb), respectivamente, usando um kit de recuperação de gel de DNA após eletroforese em gel. Conservar o pKAd7-HpaI-FL a -20 °C.
- Amplifique o fragmento contendo Kan e Ori usando pShuttle-CMV como modelo com primers de Ad7HpaIF1 e Ad7HpaIR1 (Tabela S1) por PCR. Recupere e dilua o produto de PCR (2541 pb) a ser usado como modelo para a segunda rodada de reação de PCR para amplificar Kan-Ori2 com primers de Ad7HpaIF2 e Ad7HpaIR2.
- Purifique o produto de PCR (2584 pb) após a resolução em um gel de agarose a 1% por eletroforese. Defina a reação de PCR da seguinte forma: um ciclo a 98 °C por 30 s, depois 30 ciclos de desnaturação a 98 °C por 8 s, recozimento e extensão a 72 °C por 2 min.
NOTA: A parte central de Kan-Ori2 é o fragmento contendo Kan e Ori, seguido pelas sequências de uma endonuclease de restrição única que não existe no pequeno fragmento pKAd7-HpaI-FS (por exemplo, Pme I), e a região sobreposta com o grande fragmento pKAd7-HpaI-FL; as sequências mais externas são a região sobreposta com pequeno fragmento pKAd7-HpaI-FS.
- Purifique o produto de PCR (2584 pb) após a resolução em um gel de agarose a 1% por eletroforese. Defina a reação de PCR da seguinte forma: um ciclo a 98 °C por 30 s, depois 30 ciclos de desnaturação a 98 °C por 8 s, recozimento e extensão a 72 °C por 2 min.
- Adicione o produto de PCR Kan-Ori2 e o pKAd7-HpaI-FS recuperado na etapa 5.1 ao DNA Assembly Master Mix. Incubar a 50 °C durante 1 h. Siga as etapas 2.4 a 2.6. O plasmídeo correto é denominado pKan-Ad7HpaI (~ 10 kb).
- Amplifique o fragmento CMV-GFP-PA (~ 1,7 kb) contendo o promotor CMV, GFP CDS e sinal SV40 polyA (PA) usando pShuttle-GFP23 como modelo com primers de Ad7E3GFPF1 e Ad7E3GFPR1 (Tabela S1). Purifique o produto de PCR (1650 pb) após a resolução em um gel de agarose a 1% por eletroforese.
NOTA: Regiões sobrepostas que podem ser montadas com BstZ17 I e Mlu I digerido pKan-Ad7HpaI devem ser incluídas nos primers, e um local de restrição exclusivo (por exemplo, Sbf I) que não existe no plasmídeo pKan-Ad7 é adicionado aos primers para posterior montagem de restrição. - Digerir 300 ng de pKan-Ad7HpaI com BstZ17 I e Mlu I. Purificar o fragmento de 7,3 kb (a maioria das regiões E3 são deletadas) após a resolução em gel de agarose a 1% por eletroforese. Adicione o fragmento recuperado de 7,3 kb e o fragmento CMV-GFP-PA recuperado (~ 1,6 kb) ao DNA Assembly Master Mix. Siga as etapas 2.4-2.6. O plasmídeo correto é denominado pKAd7-E3GFP (~ 9 kb).
NOTA: Todas as sequências entre BstZ17 I e Mlu I pertencem à região E3. - Digerir 300 ng de pKAd7-HE3GFP com Pme I. Purificar o fragmento grande (~6,4 kb) usando um kit de recuperação de gel de DNA após eletroforese em gel. Adicione o fragmento purificado e o pKAd7-HpaI-FL recuperado na etapa 5.1 ao DNA Assembly Master Mix. Incubar a 50 °C durante 1 h. Siga as etapas 2.4 a 2.6. O plasmídeo correto é denominado pKAd7-E3GFP.
- Transfectar células HEK293 com pKAd7-E3GFP linearizado Pme I, conforme descrito na seção 3. O adenovírus resgatado é denominado Ad7-E3GFP.
- Amplifice o fragmento CMV-mCherry-PA (~ 1,6 kb) contendo o promotor CMV, mCherry CDS e sinal SV40 polyA usando pKFAV4-CX19A como modelo com os primers de Ad7E3CHEF1 e Ad7E3CHER1. Purifique o produto de PCR após a resolução em gel de agarose a 1% por eletroforese.
- Digerir 600 ng de pKAd7-E3GFP com Sbf I e purificar o fragmento de ~35 kb. Adicione o fragmento de 35 kb e o fragmento CMV-mCherry-PA recuperado ao DNA Assembly Master Mix. Incubar a 50 °C durante 1 h. Siga as etapas 2.4-2.6. O plasmídeo correto é denominado pKAd7-E3CHE.
- Transfectar células HEK293 com pKAd7-E3CHE linearizado Pme I, conforme descrito na seção 3. O adenovírus resgatado é denominado Ad7-E3CHE.
6. Amplificação e purificação do adenovírus recombinante em células HEK293
- Inocular uma monocamada confluente de 80% a 90% de células HEK293 cultivadas em oito placas de cultura tratadas com TC de 150 mm de diâmetro com adenovírus. Incubar a 37 °C durante 2 h sob agitação ligeira. Substitua o sobrenadante por 25 mL de DMEM contendo 2% de FBS.
- Incubar as células a 37 °C durante 2-3 dias.
- Rejeitar a maior parte do sobrenadante dos oito pratos de 150 mm de diâmetro quando um ECP afectar a maioria das células. Colha células infectadas usando elevadores de células com um volume final de eluição de ~ 6,0 mL em tubos de polipropileno de 15 mL.
- Interrompa as células por três ciclos de congelamento e descongelamento para liberar o vírus. Limpar o lisado por centrifugação durante 12 min a 1200 x g à temperatura ambiente. Colete o sobrenadante contendo vírus.
- Adicione MgCl2 (0,1 mM) e nuclease benzonase (50 U/mL) ao sobrenadante. Vortex suavemente por 40 min a 37 °C. Adicionar NaCl (600 mM) ao sobrenadante e vórtice suavemente durante mais 40 min a 37 °C. Centrifugue por 12 min a 1200 x g em temperatura ambiente. Colete o sobrenadante contendo vírus para purificação.
- Prepare cloreto de césio (CsCl) com diferentes concentrações de 1,35 g / mL, 1,30 g / mL e 1,25 g / mL com 10 mM de Tris-HCl.
- Adicione 0,8 mL de solução de CsCl a 1,35 g/mL no fundo de um tubo Ultra-Clear de 5,1 mL usando uma pipeta e, em seguida, adicione lentamente 0,8 mL de solução de CsCl a 1,30 g/mL e 0,8 mL de solução de CsCl a 1,25 g/mL em cima da primeira solução sequencialmente.
- Por fim, sobreponha cuidadosamente ~ 2,5 mL do sobrenadante coletado na etapa 6.3 no topo da solução de CsCl de 1,25 g / mL e encha o tubo a 2-3 mm do topo usando 10 mM de Tris-HCl.
- Centrifugue por 2 h a 200.000 x g e 8 °C em um rotor de caçamba oscilante com aceleração e desaceleração lentas para separar as partículas virais intactas das partículas virais defeituosas.
NOTA: Após a centrifugação, várias faixas brancas podem ser vistas claramente na solução de CsCl. - Remova cuidadosamente as bandas das partículas vazias e a camada de detritos celulares usando a pipeta e colete a banda mais baixa contendo o vírus maduro usando uma seringa de 2 mL.
- Transfira a suspensão do vírus coletada para um de diálise MWCO 20 K e dialise em tampão de diálise contendo 10 mM de Tris-HCl, 150 mM de NaCl, 1 mM de MgCl2 e 2% de glicerol (pH 8,0) durante a noite a 4 ° C.
- Troque o tampão de diálise contendo 10 mM de Tris-Cl, 150 mM de NaCl, 1 mM de MgCl2 e 5% de glicerol (pH 8,0) no dia seguinte e dialise 5 h a 4 °C.
- Colete as partículas de vírus dialisadas usando uma seringa de 2 mL. Prepare várias alíquotas de pequenos volumes desejados (20-200 μL) e armazene o vírus purificado a -80 ° C.
NOTA: O título de partículas do vírus purificado pode ser determinado medindo o conteúdo do DNA genômico, e o título de infectividade pode ser determinado em células HEK293 contando células GFP-positivas com o ensaio de diluição limitante.
7. Identificação do genoma do adenovírus recombinante por digestão enzimática de restrição
- Infecte uma monocamada confluente de 80% a 90% de células HEK293 cultivadas em um frasco T-25 com adenovírus recombinante. Incubar durante 2 h a 37 °C. Substitua o meio de cultura por DMEM fresco contendo 2% de FBS.
- Remover o sobrenadante da cultura e extrair o ADN genómico viral com o método de Hirt modificado descrito no passo 1.3 quando o CPE ocorrer no prazo de 2-3 dias.
- Digerir o DNA genômico do adenovírus recombinante com as enzimas indicadas e analisar na eletroforese em gel de agarose. O plasmídeo adenoviral recombinante serviu como controle.
Resultados
A estratégia para a construção de um clone infeccioso do HAdV-7 é mostrada na Figura 1 e na Figura 2. Dois plasmídeos de clones infecciosos foram selecionados aleatoriamente e identificados por BstZ17 I, BamH I e EcoR V, respectivamente. Os resultados mostraram que os fragmentos eram consistentes com o tamanho esperado (Figura 2A), indicando que os plasmídeos foram construídos corretamente. Focos de cometas puderam ser vistos nas células 12 dias após o plasmídeo linearizado Pme I ter sido transfectado em células HEK293 (Figura 2B), indicando que o vírus foi resgatado com sucesso.
Neste estudo, o vetor adenoviral deletado por E3 foi gerado para superexpressar a GFP (Figura 4) ou mCherry como exemplos. Durante a modificação da região E3, um plasmídeo intermediário pKan-Ad7HpaI (Figura 3) foi construído primeiro. Os plasmídeos de três colônias foram analisados com digestão EcoR V e Hind III, os resultados da eletroforese mostraram que todos os plasmídeos estavam corretos (Figura 5A). Além disso, o cromatograma de sequenciamento mostrou que não há mutação nos locais de montagem (Figura 5C, resultados parciais foram mostrados). Depois que a região E3 entre BstZ17 I e Mlu I foi excluída e substituída pelo CMV-GFP-PA, três dos plasmídeos modificados pKAd7-HE3GFPs foram identificados por digestão e sequenciamento de enzimas de restrição (dados não mostrados). Os resultados mostraram que os fragmentos digeridos foram exatamente como esperados, indicando que os plasmídeos foram construídos corretamente (Figura 5B).
Após a modificação, o plasmídeo resultante pKAd7-E3GFP contém o CMV-GFP-PA flanqueado por dois sítios Sbf I (Figura 4), que poderia ser usado para substituir o CMV-GFP-PA por outros genes de forma conveniente. A expressão de GFP pôde ser observada em microscópio de fluorescência 48 h após as células HEK 293 terem sido transfectadas com pKAd7-E3GFP linearizado por Pme I, e focos fluorescentes e CPE puderam ser observados 13 dias após a transfecção (Figura 6). As células e o meio foram colhidos e usados para inocular células HEK293 após três ciclos de congelamento e descongelamento; A expressão de GFP e CPE pôde ser observada 48 h após a infecção (Figura 6). Seguindo o protocolo de purificação descrito, as partículas virais concentradas apareceram como várias bandas de vírion opalescentes dentro do gradiente de CsCl (Figura 7A). A banda mais baixa abaixo de 1,35 g/cm3 CsCl foi coletada e usada para coloração negativa com ácido fosfotúngstico (PTA), e o adenovírus icosaédrico intacto pôde ser observado em microscópio eletrônico (Figura 7B). A banda superior (mais leve) continha partículas vazias e não infecciosas e teve que ser evitada. Neste protocolo, foram utilizadas oito placas de cultura de tecidos T-150 cm2 , para as quais foram produzidos 1,5 mL de estoque de Ad7-E3GFP com título infeccioso de 2,3 x 1011 UI/mL e título de partículas virais de 6,0 x 1012 vp/mL. Para identificar a integridade genômica do Ad7-E3GFP recombinante, o DNA genômico foi digerido com endonuclease de restrição indicada e separado por eletroforese; todos os fragmentos foram consistentes com as expectativas (Figura 7C).
Usando este protocolo, o CMV-GFP-PA em pKAd7-E3GFP pode ser substituído pelo gene de interesse dentro de 2 a 3 dias. Aqui, o CMV-GFP-PA foi substituído por CMV-mCherry-PA usando montagem de restrição, e o novo plasmídeo adenoviral recombinante pKAd7-E3CHE foi usado para transfectar células HEK293 após ser linearizado por Pme I. A expressão de mCherry pôde ser detectada em 24 h pós-transfecção, e os focos fluorescentes foram formados 15 dias após a transfecção (Figura 8A). A expressão de mCherry e CPE pôde ser observada em células HEK293 48 h após a inoculação com o Ad7-E3CHE resgatado. Os resultados da eletroforese após a digestão da endonuclease de restrição mostraram que o genoma de Ad7-E3CHE foi completamente consistente com as expectativas (Figura 8B).

Figura 1: Diagrama esquemático da construção de clones infecciosos do adenovírus humano 7 (HAdV-7) usando montagem de DNA. Os primers (marcados em amarelo) contêm não apenas as sequências emparelhadas com o modelo, mas também as regiões sobrepostas (sobreposição) que podem ser montadas com o genoma do HAdV-7. Além disso, a sequência de um sítio de endonuclease de restrição (por exemplo, Pme I) que está ausente tanto no genoma do HAdV-7 quanto na estrutura do plasmídeo também deve ser incluída nos primers para facilitar a liberação do genoma. A estrutura do plasmídeo amplificado Kan-Ori foi montada com DNA genômico viral de HAdV-7 in vitro para formar o plasmídeo infeccioso do clone pKan-Ad7. Clique aqui para ver uma versão maior desta figura.

Figura 2: Identificação do clone infeccioso de HAdV-7. (A) Dois dos plasmídeos de clones infecciosos pKan-Ad7s foram digeridos com BstZ17 I, BamH I e EcoR V, respectivamente. Os pesos moleculares previstos dos fragmentos digeridos foram 716, 947, 1164, 3460, 4349, 5828, 8116 e 13.138 pb para BstZ17 I; 5713, 6945, 9058 e 16.002 pb para BamH I; 2338, 4048, 4162, 7792, 9306 e 10.072 pb para EcoR V. (B) Resgate de HAdV-7. Pme I-linearizado pKan-Ad7 foi usado para transfectar células HEK293. O ECP ocorreu no dia 12 pós-transfecção, indicando o resgate do HAdV-7 com sucesso. Clique aqui para ver uma versão maior desta figura.

Figura 3: Construção in silico do plasmídeo intermediário. (A) Mapa de plasmídeo do clone infeccioso pKan-Ad7. São mostrados os locais de enzimas de restrição de cortador único e duplo. Hpa I, Sal I e Avr II-Mlu I podem ser selecionados para a construção de um plasmídeo intermediário contendo a região E3. Para construir um menor plasmídeo intermediário, Hpa I foi finalmente escolhido para conduzir os experimentos. (B) Mapa do plasmídeo intermediário pKan-Ad7HpaI. São mostrados os locais de restrição de todos os cortadores exclusivos e alguns duplos. A região E3 entre os locais BstZ17 I e Mlu I foi finalmente substituída por um de expressão GFP controlado pelo promotor CMV. (C) Projeto de primer para amplificar a estrutura do plasmídeo. O produto de PCR do esqueleto do plasmídeo seria fundido ao fragmento curto do pKan-Ad7 digerido por Hpa I para gerar o plasmídeo intermediário pKan-Ad7HpaI pela montagem de Gibson. São mostrados os primers projetados. As sobreposições para os locais de montagem de Gibson e Pme I seriam integradas ao produto por meio de duas rodadas de PCR usando primers de Ad7HpaIF1 / R1 e Ad7HpaIF2 / R2 em sequência. Clique aqui para ver uma versão maior desta figura.

Figura 4: Diagrama esquemático da construção do plasmídeo adenoviral recombinante pKAd7-E3GFP usando montagem de restrição. Clique aqui para ver uma versão maior desta figura.
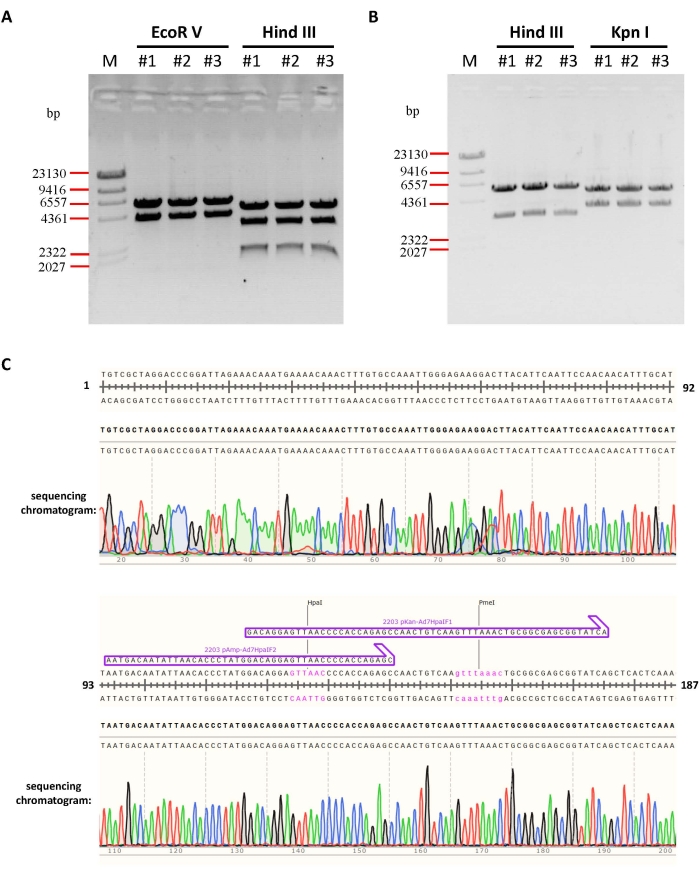
Figura 5: Identificação do plasmídeo intermediário por digestão e sequenciamento. (A) Três dos plasmídeos intermediários pKan-Ad7HpaI foram extraídos e identificados por digestão de endonuclease de restrição indicada. Os pesos moleculares previstos dos fragmentos digeridos foram 4048 e 5713 pb para EcoR V e 1710, 3206 e 4845 pb para Hind III. (B) Três dos plasmídeos pKAd7-HE3GFP foram extraídos e identificados por digestão de endonuclease de restrição. Os pesos moleculares previstos dos fragmentos digeridos foram 3206 e 5709 pb para Hind III e 3725 e 5190 pb para Kpn I. (C) Os sítios de fusão do plasmídeo pKan-Ad7HpaI foram confirmados por sequenciamento. Devido ao espaço limitado, apenas os resultados do sequenciamento de um local de fusão do plasmídeo foram mostrados aqui. Clique aqui para ver uma versão maior desta figura.
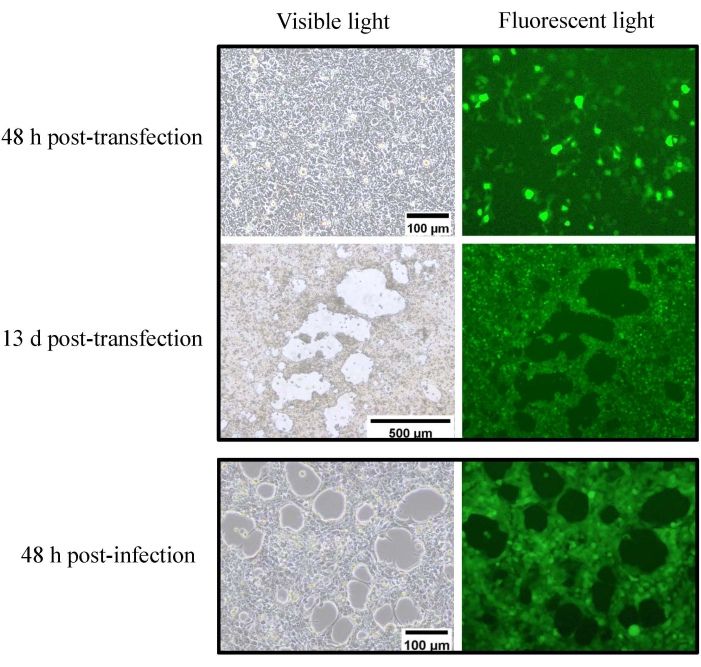
Figura 6: Resgate e amplificação do adenovírus recombinante Ad7-E3GFP. Células HEK293 a 90% de confluência (frasco T-25) foram transfectadas com 5 μg de pKAd7-E3GFP linearizado Pme I usando reagentes de transfecção comerciais. A expressão de GFP e CPE puderam ser observadas em microscópio de fluorescência 48 h e 13 dias após a transfecção, respectivamente. As células e o meio foram coletados e usados para inocular células HEK293; A expressão de CPE e GFP pôde ser observada 48 h após a infecção. Clique aqui para ver uma versão maior desta figura.

Figura 7: Purificação e identificação do adenovírus recombinante Ad7-E3GFP. (A) Purificação do Ad7-E3GFP. Os lisados celulares contendo vírus foram carregados no gradiente de CsCl e as bandas virais puderam ser vistas após a ultracentrifugação. (B) Observação do Ad7-E3GFP purificado sob EM. (C) Identificação do DNA genômico Ad7-E3GFP. O DNA genômico foi digerido com endonuclease de restrição indicada após a extração das células infectadas. O plasmídeo pKAd7-E3GFP foi servido como controle. Os pesos moleculares previstos dos fragmentos digeridos do DNA genômico Ad7-E3GFP foram 530, 1191, 2368, 5310, 10,065 e 14,937 pb para Afl II; 7774, 11.006 e 15.621 pb para Asc I; 531, 2623, 4789, 7413, 9163 e 9882 pb para BsrG I; 342, 2338, 2432, 2860, 4162, 4403, 7792 e 10.072 pb para EcoR V; 1005, 3725, 6084, 9570 e 14.017 pb para Kpn I. Os pesos moleculares previstos dos fragmentos digeridos do plasmídeo pKAd7-E3GFP foram 1191, 5310, 5369, 10,065 e 14,937 pb para Afl II; 7774 e 29.098 pb para Asc I; 531, 2623, 9163, 9882 e 14.673 pb para BsrG I; 342, 2338, 2860, 4162, 7792, 9306 e 10.072 pb para EcoR V; 1005, 3725, 9570 e 22.572 pb para Kpn I. M: marcador de DNA lambda/HindIII. Clique aqui para ver uma versão maior desta figura.

Figura 8: Geração, amplificação e identificação do adenovírus recombinante Ad7-E3CHE. (A) Resgate e amplificação do adenovírus recombinante Ad7-E3CHE. As células HEK293 a 90% de confluência (frasco T-25) foram transfectadas com 5 μg de Pme I-linearizado pKAd7-E3CHE usando reagente de transfecção. Focos fluorescentes e CPE puderam ser observados em microscópio de fluorescência 15 dias após a transfecção. As células e o meio foram coletados e usados para inocular células HEK293. A expressão de CPE e mCherry pôde ser observada 48 h após a infecção. (B) Identificação do DNA genômico Ad7-E3CHE. O DNA genômico foi digerido com endonuclease de restrição indicada após a extração das células infectadas. O plasmídeo pKAd7-E3 CHE serviu como controle. Os pesos moleculares previstos dos fragmentos digeridos do DNA genômico Ad7-E3CHE foram 379, 653, 2619, 3766, 5721, 8335 e 12.939 pb para a Idade I; 716, 750, 947, 1164, 2607, 3460, 4349, 7281 e 13.138 pb para BamH I; 1005, 3725, 6095, 9570 e 14.017 pb para Kpn I; 1635, 4256, 6853 e 21.668 pb para Nde I; 4810, 5920, 6360 e 17.322 pb para Sal I. Os pesos moleculares previstos dos fragmentos digeridos do plasmídeo pKAd7-E3CHE foram 379, 653, 2619, 3766, 5721 e 23.745 pb para a Idade I; 716, 947, 1164, 3460, 4349, 5828, 7281 e 13.138 pb para BamH I; 1005, 3725, 9570 e 22.583 pb para Kpn I; 4256, 10.959 e 21.668 pb para Nde I; 5920, 13.641 e 17.322 pb para Sal I. M: marcador de DNA lambda/HindIII. Clique aqui para ver uma versão maior desta figura.
Tabela S1: Informações resumidas dos primers. Clique aqui para baixar esta tabela.
| Plasmídeo do produto | Tamanho (bp) | Reação de montagem de DNA | Extração de plasmídeo | Análise de restrição | Sequenciamento de Sanger | |
| Fragmento Grande (BP) | Fragmento pequeno (BP) | (colônias colhidas/com tamanho esperado) | (digerido / correto) | (sequenciado/correto) | ||
| pKan-Ad7 | 37718 | 35239 | 2543 | 6/5 | 2/2 | 2/2 |
| pKan-Ad7HpaI | 9761 | 7235 | 2584 | 6/6 | 3/3 | 3/3 |
| pKAd7-HE3GFP | 8915 | 7316 | 1650 | 6/6 | 3/3 | 3/3 |
| pKAd7-E3GFP | 36872 | 30483 | 6444 | 3/2 | 2/2 | 2/2 |
| pKAd7-E3CHE | 36883 | 35277 | 1655 | 3/3 | 2/2 | 2/2 |
Tabela 1: Dados resumidos da construção do plasmídeo. O procedimento de montagem de restrição da construção do plasmídeo incluiu preparação de fragmento, reação de montagem de DNA, transformação bacteriana, extração de plasmídeo, análise de restrição e confirmação de sequenciamento. Geralmente, dezenas ou centenas de colônias podem ser produzidas em uma placa LB após a transformação de células competentes de E. coli TOP10 com produto de montagem de DNA. As colônias foram coletadas e propagadas aleatoriamente, e o DNA do plasmídeo foi extraído e posteriormente digerido com endonuclease e o padrão de bandas do plasmídeo digerido foi fotografado e analisado após eletroforese. Finalmente, todo o plasmídeo ou produtos de PCR que cobrem os locais de fusão ou regiões amplificadas por PCR no plasmídeo foram submetidos ao sequenciamento de Sanger.
Figura S1: Plasmídeo intermediário virtual para modificação das regiões E1 e E3. O longo fragmento de pKan-Ad7 digerido por Sal I pode ser usado para construir o plasmídeo intermediário pKan-Ad7SalI. São mostrados os locais de restrição de todos os cortadores exclusivos e alguns duplos. A região E1, que fica entre os locais SnaB I e MauB I, pode ser modificada com precisão por PCR de extensão de sobreposição. Da mesma forma, Spe I junto com BsrG I podem ajudar a modificar a região E3. Clique aqui para baixar este arquivo.
Discussão
Diferentes adenovírus têm vários tropismos teciduais, e a prevalência de imunidade pré-existente do hospedeiro contra diferentes adenovírus pode flutuar intensamente em seres humanos24, o que atrai o interesse na construção de novos vetores adenovirais para terapia gênica ou desenvolvimento de vacinas. No entanto, o estabelecimento de um novo sistema de vetores adenovirais permanece complicado para laboratórios genéricos de biologia molecular.
Aqui, introduzimos um protocolo para gerar vetores a partir de adenovírus do tipo selvagem usando técnicas de digestão de restrição e montagem de Gibson 13,14,15,16,17,18,19. Em geral, esse processo pode ser dividido em três etapas. Em primeiro lugar, o produto de PCR da estrutura do plasmídeo é misturado com o DNA genômico do adenovírus, e a montagem de Gibson é empregada para gerar um clone infeccioso. O projeto de primers de PCR é o fator chave: as extremidades 3 'dos primers são sequências específicas para amplificar a estrutura do plasmídeo que contém a origem da replicação e o gene resistente a antibióticos, e as extremidades 5' são as sequências de complemento reverso homólogas ao terminal do adenovírus ITR. Além disso, um local de restrição sem cortador do genoma viral é adicionado no meio, que é usado para liberar o genoma viral do clone de DNA infeccioso. Em segundo lugar, uma estratégia intermediária baseada em plasmídeo é realizada para construir um sistema de vetor adenoviral. Um fragmento contendo a região a ser modificada é excisado do clone infeccioso e usado para construir um plasmídeo intermediário. A PCR de extensão de sobreposição é a principal abordagem a ser adotada para modificação direcionada ao local, como deleção de genes não essenciais e inserção do de expressão gênica exógena. O fragmento de vírus modificado é posteriormente excisado do plasmídeo intermediário por digestão por restrição e restaurado na região correspondente no clone de DNA infeccioso para gerar um plasmídeo de vetor adenoviral. Para esta etapa, a análise de sequência é de grande importância para determinar a região a ser deletada e o local de inserção ideal para o gene de interesse. Geralmente, um cortador duplo é selecionado para cortar o clone de DNA infeccioso para gerar um fragmento pequeno e um grande, enquanto a região alvo para modificações adicionais está localizada no pequeno fragmento, com o qual o plasmídeo intermediário é construído. O plasmídeo adenoviral é linearizado e usado para transfectar células de empacotamento para o resgate de vírus recombinantes. Finalmente, a estratégia de carregamento de transgenes já foi considerada antecipadamente e integrada ao sistema vetorial. O de expressão do transgene no plasmídeo adenoviral pode ser facilmente removido e substituído por outros transgenes por montagem de restrição, graças aos locais de corte duplo preservados que flanqueiam essa região.
Um clone infeccioso fornece um ponto de partida sólido para a construção de um novo sistema de vetores adenovirais. Geralmente, o método de recombinação homóloga em bactérias é usado para construir clones infecciosos de adenovírus5. A PCR é realizada para amplificar as sequências em ambas as extremidades do genoma adenoviral como braços homólogos de 500-2000 pb de comprimento, e os produtos da PCR são ligados a uma estrutura de plasmídeo para formar um pequeno plasmídeo. O pequeno plasmídeo é linearizado, misturado com DNA genômico de adenovírus e usado para transformar a cepa E. coli BJ5183 por eletroporação. O produto de recombinação, um plasmídeo de clone infeccioso, precisa ser identificado e transferido para uma cepa de E. coli negativa para recombinase para amplificação de plasmídeo. Todo o procedimento contém pelo menos três rodadas de transformação bacteriana e leva aproximadamente 1 semana, supondo que nenhum evento inesperado ocorra. A montagem de Gibson simplifica substancialmente a construção de clones infecciosos de adenovírus. Comparado com a recombinação homóloga, tem as seguintes vantagens13: Em primeiro lugar, a montagem do DNA é muito mais simples e eficiente, pois apenas uma rodada de transformação bacteriana é necessária. Em segundo lugar, a montagem do DNA preserva o genoma do adenovírus em um clone infeccioso com maior fidelidade porque as bactérias positivas para recombinase, nas quais os plasmídeos podem ser instáveis, são excluídas de todo o procedimento. Finalmente, menos informações de sequência são necessárias para a montagem do DNA, uma vez que sobreposições tão curtas quanto 15-30 pb são suficientes para uma montagem eficiente, o que significa que o genoma pode ser clonado a partir de um adenovírus desconhecido, mesmo que haja apenas informações de sequência no final do ITR.
Uma limitação dessa abordagem é que encontrar a endonuclease adequada costuma ser complicado e complicado. Esse processo percorre todo o protocolo, incluindo a construção de plasmídeo de clone infeccioso, plasmídeo intermediário e modificação da região de interesse (ROI). Às vezes, uma enzima adequada para modificar o ROI não pode ser encontrada no plasmídeo intermediário. Nesse caso, é necessário construir outro plasmídeo intermediário que também dependa do software de análise de sequência de DNA para tornar o plasmídeo menor e facilitar a busca por endonucleases de restrição adequadas. Ocasionalmente, a reação de montagem do DNA pode produzir alguns clones sem a inserção, pois algumas enzimas de restrição não cortam o plasmídeo até a conclusão. Nesse caso, selecione duas endonucleases de restrição diferentes em vez de uma enzima única para digerir o vetor, o que evitaria o auto-recozimento de saliências. Outra limitação da montagem do DNA é que a verificação do sequenciamento é necessária após cada reação, pois existe o potencial de incompatibilidades neste método. No entanto, reações de montagem incorretas não ocorrem com muita frequência. Dezenas ou centenas de colônias podem ser cultivadas em cada placa LB após a transformação de células competentes de E. coli TOP10 com produto de montagem de DNA, e plasmídeos com a montagem errada raramente são encontrados (Tabela 1).
Em resumo, mostramos aqui como gerar um vetor adenoviral deletado por E3 a partir de um HAdV-7 isolado de tipo selvagem, e o conjunto de restrição que introduzimos é um método altamente eficiente, fácil de usar e multiuso para construção e modificação de vetores adenovirais.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Esta pesquisa foi financiada pela Fundação de Ciências Naturais de Pequim (7204258), Fundação Nacional de Ciências Naturais da China (82161138001, 82072266), Fundo de Inovação CAMS para Ciências Médicas (2019-I2M-5-026) e a pesquisa e aplicação sobre rastreamento molecular de patógenos respiratórios essenciais em Pequim, pela Capital Health Development and Research of Special (2021-1G-3012).
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL polypropylene microcentrifuge Tube | Axygen | MCT-150-C | Storage of virus |
| 15 mL polypropylene centrifuge tubes | Corning | 430790 | Storage of virus |
| 150 mm TC-treated culture dishes | Corning | 430599 | Growth of HEK29E cells |
| 20 K MWCO dialysis cassette | ThermoFisher Scientific | 66005 | Dialysis of virus |
| Acetic acid | Amresco | 714 | Extraction of DNA |
| Afl II | NEB | R0520 | Digestion |
| Agarose | Takara | 5260 | Electrophoresis |
| Age I | NEB | R0552 | Digestion |
| Asc I | NEB | R0558 | Digestion |
| BamH I | NEB | R0136 | Digestion |
| Benzonase Nuclease | Sigma | E8263-25KU | Purification of virus |
| BsrG I | NEB | R0575 | Digestion |
| Cell lifter | Corning | 3008 | Scrape off the cells |
| CsCl | Sigma | C3032 | Purification of virus |
| DNA gel recovery kit | Zymo | D4045 | Recovery of DNA |
| Dulbecco’s modified Eagle’s medium (DMEM) | Cytiva | SH30022.01 | HEK293 cells medium |
| E.coli TOP10 competent cells | TIANGEN BIOTECH (BEIJING) CO.,LTD. | CB-104 | Transformation of assembly product |
| EcoR V | NEB | R3195 | Digestion |
| EDTA | Thermo Fisher Scientific | R3104 | Extraction of DNA |
| Fetal bovine serum (FBS) | Cytiva | SV30208.02 | HEK293 cells culture |
| Genomic DNA Clean and concentrator kit | Zymo | D4065 | Purification of DNA |
| Glycerol | Shanghai Macklin Biochemical Co., Ltd | G810575 | Dialysis of virus |
| HEK293 cells | ATCC | CRL-1573 | Amplification of virus |
| High-Fidelity DNA Polymerase | NEB | M0491 | PCR |
| Hind III | NEB | R3104 | Digestion |
| Kanamycin sulfate | Amresco | 408 | Selection of plasmid |
| Kpn I | NEB | R3142 | Digestion |
| Lambda/HindIII DNA marker | Takara | 3403 | Electrophoresis |
| LB broth | BD | 240230 | LB plate for bacteria |
| LB medium | Solarbio Life Science | L1010 | Medium for bacteria |
| MgCl2 | Sigma | 63068 | Dialysis of virus |
| Microcentrifuge | Thermo Fisher Scientific | Sorvall Legend Micro 21R | Extraction of DNA |
| NaCl | Sigma | S5886 | Dialysis of virus |
| Nde I | NEB | R0111 | Digestion |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621 | DNA assembly |
| Nhe I | NEB | R0131 | Digestion |
| Phosphate Buffered Saline | Cytiva | SH30256.01 | Washing of cells |
| Pipette | Thermo Fisher Scientific | Matrix | Aspirate the medium |
| Plasmid Maxprep Kit | Vigorous Biotechnology Beijing Co., Ltd. | N001 | Extraction of DNA |
| Plasmid Miniprep Kit | TIANGEN BIOTECH (BEIJING) CO.,LTD. | DP103 | Extraction of DNA |
| Pme I | NEB | R0560 | Digestion |
| Potassium acetate | Amresco | 698 | Extraction of DNA |
| Protease K | Thermo Fisher Scientific | AM2542 | Extraction of DNA |
| pShuttle-CMV | Stratagene | 240007 | PCR template |
| RNase | Beyotime | D7089 | Extraction of DNA |
| Sal I | NEB | R0138 | Digestion |
| Sbf I | NEB | R3642 | Digestion |
| SDS | Amresco | 227 | Extraction of DNA |
| Swinging-bucket rotor | HITACHI | S52ST | Purification of virus |
| T-25 cell flask | Corning | 430639 | Growth of HEK29E cells |
| T-75 cell flask | Corning | 430641 | Growth of HEK29E cells |
| Transfection reagent | Polyplus-transfection | 114-15 | Transfection |
| Transmission electron microscope | FEI | TECNAI 12 | Obsevation of virus |
| Tris-HCl | Amresco | 234 | Dialysis of virus |
| Ultracentrifuge | HITACHI | Himac CS120GXII | Purification of virus |
Referências
- Lasaro, M. O., Ertl, H. C. New insights on adenovirus as vaccine vectors. Molecular Therapy. 17 (8), 1333-1339 (2009).
- Fuchs, J. D., et al. Safety and immunogenicity of a recombinant adenovirus serotype 35-Vectored HIV-1 vaccine in adenovirus serotype 5 seronegative and seropositive individuals. Journal of AIDS & Clinical Research. 6 (5), (2015).
- Milligan, I. D., et al. Safety and immunogenicity of novel adenovirus type 26- and modified vaccinia ankara-vectored ebola vaccines: a randomized clinical trial. JAMA. 315 (15), 1610-1623 (2016).
- Zhu, F. C., et al. Safety, tolerability, and immunogenicity of a recombinant adenovirus type-5 vectored COVID-19 vaccine: a dose-escalation, open-label, non-randomised, first-in-human trial. Lancet. 395 (10240), 1845-1854 (2020).
- He, T. C., et al. A simplified system for generating recombinant adenoviruses. Proceedings of the National Academy of Sciences of the United States of America. 95 (5), 2509-2514 (1998).
- Danthinne, X., Imperiale, M. J. Production of first generation adenovirus vectors: a review. Gene Therapy. 7 (20), 1707-1714 (2000).
- Guo, X., et al. Systemic and mucosal immunity in mice elicited by a single immunization with human adenovirus type 5 or 41 vector-based vaccines carrying the spike protein of Middle East respiratory syndrome coronavirus. Immunology. 145 (4), 476-484 (2015).
- Folegatti, P. M., et al. Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS-CoV-2: a preliminary report of a phase 1/2, single-blind, randomised controlled trial. Lancet. 396 (10249), 467-478 (2020).
- Jang, Y., Bunz, F. AdenoBuilder: A platform for the modular assembly of recombinant adenoviruses. STAR Protocols. 3 (1), 101123 (2022).
- Zhou, X., Sena-Esteves, M., Gao, G. Construction of recombinant adenovirus genomes by direct cloning. Cold Spring Harbor Protocols. 2019 (5), (2019).
- Ruzsics, Z., Lemnitzer, F., Thirion, C. Engineering adenovirus genome by bacterial artificial chromosome (BAC) technology. Methods in Molecular Biology. 1089, 143-158 (2014).
- Zhou, D., et al. An efficient method of directly cloning chimpanzee adenovirus as a vaccine vector. Nature Protocols. 5 (11), 1775-1785 (2010).
- Zou, X. H., et al. DNA assembly technique simplifies the construction of infectious clone of fowl adenovirus. Journal of Virological Methods. 257, 85-92 (2018).
- Guo, X., et al. Site-directed modification of adenoviral vector with combined DNA assembly and restriction-ligation cloning. Journal of Biotechnology. 307, 193-201 (2020).
- Liu, H., et al. Single plasmid-based, upgradable, and backward-compatible adenoviral vector systems. Human Gene Therapy. 30 (6), 777-791 (2019).
- Yan, B., et al. User-friendly reverse genetics system for modification of the right end of fowl adenovirus 4 genome. Viruses. 12 (3), 301 (2020).
- Zhang, W., et al. Fiber modifications enable fowl adenovirus 4 vectors to transduce human cells. Journal of Gene Medicine. 23 (10), 3368 (2021).
- Zou, X., et al. Fiber1, but not fiber2, is the essential fiber gene for fowl adenovirus 4 (FAdV-4). Journal of General Virology. 102 (3), 001559 (2021).
- Guo, X., et al. Restriction-assembly: a solution to construct novel adenovirus vector. Viruses. 14 (3), 546 (2022).
- Duan, Y., et al. Genetic analysis of human adenovirus type 7 strains circulating in different parts of China. Virologica Sinica. 36 (3), 382-392 (2021).
- Arad, U. Modified Hirt procedure for rapid purification of extrachromosomal DNA from mammalian cells. Biotechniques. 24 (5), 760-762 (1998).
- Knipe, D. M. H. . P. M. Fields' Virology. 2, 1733 (2013).
- Liu, H. Y., Han, B. J., Zhong, Y. X., Lu, Z. Z. A three-plasmid system for construction of armed oncolytic adenovirus. Journal of Virological Methods. 162 (1-2), 8-13 (2009).
- Mennechet, F. J. D., et al. A review of 65 years of human adenovirus seroprevalence. Expert Review of Vaccines. 18 (6), 597-613 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados