Method Article
Construction de vecteurs adénoviraux à l’aide de la technologie d’assemblage de l’ADN
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Dans cette étude, un clone infectieux de l’adénovirus humain de type 7 (HAdV-7) a été construit, et un système de vecteur HAdV-7 délélé E3 a été établi en modifiant le clone infectieux. Cette stratégie utilisée ici peut être généralisée pour fabriquer des vecteurs de transfert de gènes à partir d’autres adénovirus de type sauvage.
Résumé
Les vecteurs adénoviraux sont utilisés comme outil de transfert de gènes en thérapie génique depuis plus de trois décennies. Ici, nous introduisons un protocole pour construire un vecteur adénoviral en manipulant l’ADN génomique de HAdV-7 de type sauvage en utilisant une méthode d’assemblage d’ADN. Tout d’abord, un clone infectieux de HAdV-7, pKan-Ad7, a été généré par fusion de l’ADN génomique viral avec un produit PCR à partir du squelette plasmidique, comprenant le gène résistant à la kanamycine et l’origine de la réplication (Kan-Ori), par assemblage de l’ADN. Cela a été fait en concevant une paire d’amorces PCR, qui contenaient ~25 nucléotides de la séquence terminale de la répétition terminale inversée (ITR) de HAdV-7 à l’extrémité 5', un site d’enzyme de restriction non cutter pour le génome HAdV-7 au milieu et une séquence spécifique à la matrice pour l’amorçage de la PCR à l’extrémité 3'. Deuxièmement, une stratégie intermédiaire basée sur le plasmide a été employée pour remplacer la région E3 par des éléments exprimant le transgène dans le clone infectieux afin de générer un vecteur adénoviral. Brièvement, pKan-Ad7 a été digéré avec l’enzyme de restriction à double coupure Hpa I, et le fragment contenant la région E3 a été ligaturé à un autre produit PCR du squelette plasmidique par assemblage Gibson pour construire un plasmide intermédiaire pKan-Ad7HpaI. Pour plus de commodité, le terme restriction-assemblage a été utilisé pour désigner la méthode de clonage plasmidique de digestion et d’assemblage par restriction combinée. En utilisant l’assemblage-restriction, les gènes E3 de pKan-Ad7HpaI ont été remplacés par une cassette d’expression de protéine fluorescente verte (GFP), et la région E3 modifiée a été libérée du plasmide intermédiaire et restaurée dans le clone infectieux pour générer un plasmide adénoviral pKAd7-E3GFP. Enfin, pKAd7-E3GFP a été linéarisé par digestion Pme I et utilisé pour transfecter les cellules d’emballage HEK293 afin de sauver le virus recombinant HAdV-7. Pour conclure, une stratégie basée sur l’assemblage de l’ADN a été introduite ici pour construire des vecteurs adénoviraux dans les laboratoires généraux de biologie moléculaire sans avoir besoin de matériaux et d’instruments spécialisés.
Introduction
Au cours des trois dernières décennies, les vecteurs adénoviraux recombinants ont été largement utilisés dans le développement de vaccins et la thérapie génique 1,2,3,4 ainsi que dans la recherche fondamentale en raison de leurs propriétés biologiques exceptionnelles, telles que l’efficacité élevée de la transduction génique, la non-intégration au génome de l’hôte, le génome viral manipulateur et la facilité de production à grande échelle.
À l’heure actuelle, les vecteurs adénoviraux les plus couramment utilisés sont construits à partir de l’adénovirus humain 5 (HAdV-5)5,6. Bien que la transduction médiée par le vecteur HAdV-5 donne des résultats encourageants, les applications précliniques et cliniques ont révélé plusieurs inconvénients (par exemple, une immunité antivectorielle préexistante élevée au sein de la population humaine et une faible efficacité de transduction dans les cellules dépourvues du récepteur du virus coxsackie et de l’adénovirus (CAR)). Pour contourner ces problèmes, il y a eu un grand intérêt pour la construction de vecteurs basés sur d’autres types d’adénovirus humains ou mammifères3, 7, 8.
Jusqu’à présent, la méthode la plus populaire pour construire un vecteur adénoviral est la recombinaison homologue chez les bactéries5. Ces souches bactériennes doivent exprimer des recombinases, ce qui peut affecter la stabilité ou l’amplification des plasmides qu’elles portent. Certaines souches sont même indisponibles dans le commerce. Récemment, des méthodes basées sur d’autres principes, notamment les chromosomes artificiels bactériens, le clonage direct ou l’assemblage direct de l’ADN, ont été employées pour générer des clones infectieux d’adénovirus ou des vecteurs adénoviraux recombinants 9,10,11,12. Cependant, ces méthodes sont quelque peu hostiles aux chercheurs peu expérimentés dans ce domaine.
En 2018, le processus de construction d’un clone infectieux d’adénovirus est simplifié en laboratoire en ligaturant directement le génome du virus avec un produit PCR porteur d’un squelette plasmidique via l’assemblage Gibson13. Après cela, les méthodes de digestion de restriction et d’assemblage de Gibson sont combinées pour charger les transgènes sur les plasmides adénoviraux existants 14,15,16,17,18. Pour des raisons de commodité, le terme « assemblage de restriction » est utilisé ci-après pour désigner la méthode de digestion combinée par enzyme de restriction et d’assemblage de Gibson. Des stratégies ont été développées pour construire des vecteurs adénoviraux à partir de clones infectieux en utilisant l’assemblage-restriction19. L’essence de l’assemblage-restriction est d’inclure autant que possible des fragments excisés des plasmides dans une réaction d’assemblage de l’ADN, tandis que les produits PCR courts servent de liants ou de patchs pour la modification du plasmide. Dans le même temps, le nombre de fragments inclus est maintenu aussi bas que possible. De tels efforts reflètent les fruits de l’action ; la possibilité de mutations indésirables causées par la PCR ou l’assemblage de l’ADN peut être minimisée et le taux de réussite peut être amélioré. De manière concluante, un pipeline d’un adénovirus de type sauvage à un vecteur adénoviral a été mis en place dans le laboratoire 13,14,15,16,17,18,19.
Ici, nous tentons d’introduire ces méthodes en fournissant des exemples de construction d’un clone infectieux HAdV-7 et d’un vecteur HAdV-7 compétent pour la réplication supprimée E3.
Protocole
REMARQUE : Les adénovirus sont classés au niveau de biosécurité 2 (BSL-2) ; Toutes les étapes d’utilisation des virus ont été réalisées dans un laboratoire de biosécurité de niveau 2. Le HAdV-7 de type sauvage a été isolé en 2017 à partir de l’échantillon d’aspiration nasopharyngée d’un nourrisson de 10 mois hospitalisé pour une infection aiguë des voies respiratoires à l’hôpital pour enfantsde Pékin 20. Les stocks de virus ont été stockés à -80 °C.
1. Extraction de l’ADN génomique du HAdV-7
- Semez 2,0 x 106 cellules HEK293 dans une fiole T-25 contenant 5 mL de milieu d’Eagle modifié de Dulbecco (DMEM) et 10 % de sérum fœtal bovin (FBS) et cultivez à 37 °C dans une atmosphère humidifiée additionnée de 5 % de CO2. Les cellules doivent être confluentes à 80 % à 90 % et prêtes à être infectées en 18 à 24 heures.
- Inoculer les cellules avec le HAdV-7 de type sauvage pendant 2 h. Retirer le milieu contenant le virus par aspiration et ajouter 5 mL de DMEM frais contenant 2 % de FBS dans les cellules.
- Extraire l’ADN génomique viral avec la méthode de Hirt modifiée21
- Dans les 2 ou 3 jours suivant l’infection, lorsque l’effet cytopathique (EPC) est observé sur 50 à 90 % des cellules, jeter le surnageant de culture et rincer les cellules une fois avec une solution saline tamponnée au phosphate (PBS).
REMARQUE : L’effet cytopathique (EPC) est défini comme un phénomène dans lequel les cellules infectées par le virus se rassemblent, gonflent et se fixent ou se détachent lâchement du flacon de culture en grappes ressemblant à des grappes de raisin22. - Lyser les cellules dans le ballon dans 1,0 mL de tampon de lyse contenant 25 mM de Tris-HCl, 0,5 mM d’EDTA, 50 μg/mL de protéase K et 0,8 % de SDS (pH 7,6) pendant 5 min sur glace. Transférez le lysat dans un tube de microcentrifugation de 1,5 mL et incubez le tube dans un bain-marie à 50 °C pendant 30 min.
- Ajouter 120 μL de tampon de précipitation dans le tube. Placez le tube sur de la glace pendant encore 30 minutes après avoir mélangé délicatement.
REMARQUE : Le tampon de précipitation est composé de 3 M CsCl, 1 M d’acétate de potassium et 0,67 M d’acide acétique. Il aidera à précipiter les débris cellulaires et l’ADN des chromosomes cellulaires après la centrifugation tout en conservant le génome du virus dans le surnageant. - Centrifugez le tube pendant 25 min à 15 000 x g à 4 °C, puis transférez le surnageant (environ 1,1 ml) dans un tube en polypropylène de 15 ml.
- Ajoutez 2,2 ml d’éthanol absolu glacé dans le tube. Mélangez délicatement et laissez reposer le tube sur de la glace pendant 15 min. Centrifugeuse pendant 5 min à 2000 x g à température ambiante.
- Aspirez soigneusement le surnageant et lavez le précipité (ADN génomique du virus) une fois avec 800 μL d’éthanol à 70 %. Centrifugez le tube pendant 6 min à 15 000 x g à température ambiante. Jetez le surnageant et séchez à l’air libre la pastille d’ADN au fond. Ne séchez pas trop la pastille d’ADN car elle pourrait causer des difficultés à se dissoudre.
- Dissoudre la pastille dans 200 μL de tampon TE (10 mM de Tris-HCl, pH 8,0, 0,1 mM d’EDTA), ajouter 1 μL de RNase A (10 mg/mL) et incuber le tube à 37 °C pendant 15 min. Purifiez l’ADN dissous à l’aide d’un kit commercial de nettoyage et de concentration d’ADN génomique (voir le tableau des matériaux).
REMARQUE : Quantifiez l’ADN génomique viral à l’aide de la méthode couramment utilisée en laboratoire. En général, 1 à 5 μg d’ADN génomique peuvent être obtenus dans une fiole T-25 de cellules HEK293 infectées par l’adénovirus.
- Dans les 2 ou 3 jours suivant l’infection, lorsque l’effet cytopathique (EPC) est observé sur 50 à 90 % des cellules, jeter le surnageant de culture et rincer les cellules une fois avec une solution saline tamponnée au phosphate (PBS).
2. Construction d’un clone infectieux de HAdV-7 par assemblage d’ADN
- Concevoir des amorces PCR pour amplifier le squelette plasmidique (Figure 1). Ajoutez les séquences de chevauchement nécessaires à l’assemblage de l’ADN aux extrémités 5' des amorces de PCR. De plus, inclure des sites de restriction non coupants (p. ex., site Pme I) aux deux extrémités du génome HAdV-7 pour aider à libérer le génome viral du clone infectieux.
REMARQUE : Le squelette plasmidique contenant le gène de résistance aux antibiotiques et l’origine de la réplication est amplifié par PCR et fusionné avec le génome HAdV-7. Les adénovirus peuvent être sauvés des cellules d’emballage avec une plus grande efficacité lorsque le génome du virus linéarisé est utilisé au lieu d’un plasmide adénoviral circulaire. Après avoir pris en compte les facteurs mentionnés ci-dessus, les amorces sont conçues comme indiqué sur la figure 1. - Effectuer une PCR pour amplifier un fragment (Kan-Ori) contenant le gène de résistance à la kanamycine (Kan) et l’origine pBR322 (Ori) en utilisant pShuttle-CMV comme matrice avec des amorces d’Ad7KanF1 et d’Ad7KanR1 (tableau S1). Réglez la réaction PCR comme suit : un cycle à 98 °C pendant 30 s, puis cinq cycles de dénaturation à 98 °C pendant 8 s, de recuit à 68 °C pendant 30 s, d’extension à 72 °C pendant 90 s, et enfin de 25 cycles de dénaturation à 98 °C pendant 8 s, de recuit et d’extension à 72 °C pendant 2 min.
- Exécutez le produit PCR dans un gel d’agarose à 1 % par électrophorèse et purifiez le fragment à l’aide d’un kit de récupération de gel d’ADN commercial.
- Ajoutez l’ADN génomique Kan-Ori et viral récupéré de HAdV-7 au Master Mix d’assemblage d’ADN commercial. Mettre en place la réaction d’assemblage dans un volume total de 20 μL et un rapport molaire entre le vecteur et l’ADN génomique viral de 1:2. Incuber à 50 °C pendant 1 h.
REMARQUE : Réglez la réaction dans un volume total de 20 μL avec 1-3 μL de vecteur, 7-9 μL d’ADN génomique viral et 10 μL de DNA Assembly Master Mix. - Transformez les cellules de la souche TOP10 d’E. coli chimiquement compétentes avec 10 μL du produit d’assemblage par choc thermique. Étaler le mélange de transformation sur des plaques de Luria-Bertani (LB-Kan) contenant de la kanamycine (50 μg/mL). Incuber les plaques à 37 °C pendant au moins 12 h.
- Inoculer au moins trois clones chacun dans 5 mL de milieu LB-Kan à 37 °C pendant 12 h en agitant doucement (200-250 tr/min). Extrayez l’ADN plasmidique à l’aide d’un kit de mini-préparation de plasmide commercial.
- Digérer le plasmide à l’aide d’une endonucléase de restriction, puis vérifier la taille des fragments d’ADN à l’aide d’une électrophorèse sur gel d’agarose (Figure 2A). De plus, confirmez les sites de fusion par séquençage. Le plasmide d’assemblage correct est nommé pKan-Ad7.
- Cultivez une culture de 100 ml de bactéries à partir du bon clone. Extrayez une préparation de maxi-plasmide à l’aide d’un kit commercial Plasmid Maxprep.
3. Construction in silico et modification d’un plasmide intermédiaire
REMARQUE : En général, un plasmide intermédiaire doit être construit, et la façon de construire un plasmide intermédiaire idéal est l’étape clé. Un logiciel d’analyse d’ADN doté d’un module d’enzymes de restriction, tel que pDRAW32 (un logiciel gratuit, disponible sur www.acaclone.com), est nécessaire pour la conception d’un plasmide intermédiaire.
- Importez la séquence d’ADN de pKan-Ad7 dans pDRAW32 pour dessiner une carte plasmidique. Annotez les régions d’intérêt sur la carte. Pour pKan-Ad7, les régions E1, E3 et le squelette plasmidique (Kan-Ori) ont été annotés.
- Utiliser le programme pDRAW32. Cliquez d’abord sur Paramètres > Sélection d’enzymes , puis sur Coupes min/max. Entrez 1 dans la zone Min et 2 dans la case Max, puis cochez la case avant dans toute la séquence. Enfin, cliquez sur OK pour afficher les sites uniques et doubles de l’enzyme de restriction de coupe sur la carte.
REMARQUE : Généralement, seules les enzymes de restriction qui reconnaissent une séquence de 6 pb ou plus sont sélectionnées. Comme le montre la figure 3A, 17 enzymes correspondent aux critères de sélection des enzymes. - Prenons l’exemple du premier site de restriction qui borde la région E3 puisque la modification de l’E3 est prévue. Hpa I, Sal I, Avr II-Mlu I et Spe I satisfaisaient à cette exigence.
REMARQUE : Pour cette expérience, Hpa I est sélectionné car la digestion de pKan-Ad7 avec celui-ci générerait le fragment le plus court portant la région E3, et un plasmide intermédiaire plus petit faciliterait la modification future. La sélection des sites Sal I présente également des avantages - un tel plasmide intermédiaire convient à la modification des régions E1 et E3 (Figure S1). - Créez un fichier de séquence final affichant les deux brins d’ADN du plasmide intermédiaire pKan-Ad7HpaI. Cette séquence virtuelle peut être utilisée comme modèle pour concevoir des amorces qui se chevauchent.
- Copiez la séquence entre les deux sites de restriction Hpa I, y compris la région E3. Pour l’assemblage ultérieur, il est nécessaire de copier chaque séquence de ~20 pb (Tm égale ou supérieure à 48 °C) en dehors du site de restriction Hpa I (Figure 3C, chevauchement-Final 1 et chevauchement-Final2).
- Ajoutez la séquence de reconnaissance de Pme I (Figure 3C, gtttaaac) aux deux extrémités de la séquence ci-dessus.
- Ajouter la séquence du plasmide squelaire contenant le gène de résistance aux antibiotiques et l’origine de la réplication (Figure 3C, Kan-Ori) à l’extérieur du site de restriction de Pme I.
- Importez la séquence finale dans pDRAW32 pour dessiner une carte plasmidique. Annotez la région E3 sur la carte.
- Utiliser le programme pDRAW32. Cliquez d’abord sur Paramètres > Sélection d’enzymes , puis sur Coupes Min/Max. Entrez 1 dans les cases Min et Max, et cochez la case avant dans toute la séquence. Cliquez enfin sur OK pour afficher les sites uniques de l’enzyme de restriction sur la carte. On peut voir que de nombreux cutters uniques sont disponibles pour la modification E3.
REMARQUE : Pour cette expérience, BstZ17 I et Mlu I sont sélectionnés pour le remplacement de E3 partiel. Un double cutter peut également être utile. Par exemple, la fraise double Ssp I et la fraise unique BssH II peuvent être sélectionnées pour le remplacement de l’ensemble de la région E3 en utilisant la PCR d’extension de chevauchement.
- Concevez des amorces en utilisant la séquence virtuelle comme modèle pour la construction d’un plasmide intermédiaire (Figure 3C).
REMARQUE : Pour raccourcir la longueur de chaque amorce, quatre amorces ont été conçues. Le produit de PCR, qui est amplifié avec des amorces d’Ad7HpaIF1 et d’Ad7HpaIR1, serait utilisé comme matrice pour obtenir le fragment final de PCR avec des amorces d’Ad7HpaIF2 et d’Ad7HpaIR2 (Figure 3C).
4. Sauvetage d’un adénovirus recombinant infectieux dans des cellules HEK293
REMARQUE : Les trois adénovirus générés dans cet article sont tous sauvés selon le protocole suivant.
- Digérer 10 μg de plasmide adénoviral recombinant avec 2 μL de Pme I pendant 5 h à 37 °C dans un volume total de 100 μL pour linéariser le plasmide. Vérifiez 1 μL de l’ADN digéré sur un gel d’agarose à 0,8 %. La digestion devrait produire un fragment de ~34 kb du génome adénoviral et un squelette plasmidique de ~2,5 kb de Kan-Ori.
- Purifiez le plasmide adénoviral linéarisé restant à l’aide du kit de nettoyage et de concentration d’ADN génomique.
- Cultivez des cellules HEK293 dans du DMEM plus 10 % de FBS. Semez les cellules HEK293 (2 x 106 cellules) dans une fiole T-25 1 jour avant la transfection afin qu’elles atteignent une confluence de 70 à 80 % le lendemain. Utilisez 5 μg de plasmide adénoviral digéré par Pme I pour transfecter les cellules HEK293 en utilisant des réactifs de transfection disponibles dans le commerce.
REMARQUE : Le milieu des cellules transfectées doit être remplacé tous les 3 jours. - Vérifiez les cellules tous les jours, prélevez les cellules et le milieu de culture dans un tube en polypropylène de 15 ml lorsque les foyers de comète se forment (figure 2B, indiquant la réplication du virus).
- Lyser les cellules selon trois cycles de congélation-décongélation. Après une centrifugation de 12 min à 1200 x g à température ambiante, recueillir le surnageant et le stocker à -80 °C pour la propagation ultérieure du virus.
REMARQUE : Lorsque l’adénovirus recombinant exprime la GFP ou d’autres protéines fluorescentes, il est plus facile d’identifier si le virus est sauvé avec succès, car les foyers formés par les cellules positives aux protéines fluorescentes peuvent être observés intuitivement au microscope à fluorescence.
5. Construction du plasmide du génome HAdV-7 délétisé par E3
REMARQUE : Le génome de HAdV-7 a une longueur de 35 239 pb et la région E3 est située entre 27 354 pb et 31 057 pb du génome. Pour construire un plasmide intermédiaire, trouvez une endonucléase avec deux sites de coupe sur le plasmide du clone infectieux, ou deux endonucléases avec un seul site de coupe sur le plasmide. Il existe des Hpa I, Sal I ou Avr II et Mlu I. La séquence entre BstZ17 I (28 312 pb) et Mlu I (30 757 pb) doit être supprimée et remplacée par la cassette CMV-GFP-PA ou la cassette CMV-mCherry-PA.
- Digérer 600 ng de pKan-Ad7 avec Hpa I. Purifier le grand fragment pKAd7-HpaI-FL (~30 kb) et le petit fragment pKAd7-HpaI-FS (~7 kb) respectivement, à l’aide d’un kit de récupération de gel d’ADN après électrophorèse sur gel. Stockez le pKAd7-HpaI-FL à -20 °C.
- Amplifier le fragment contenant Kan et Ori en utilisant pShuttle-CMV comme modèle avec les amorces d’Ad7HpaIF1 et Ad7HpaIR1 (Tableau S1) par PCR. Récupérer et diluer le produit de PCR (2541 pb) à utiliser comme modèle pour le deuxième tour de réaction PCR afin d’amplifier Kan-Ori2 avec des amorces d’Ad7HpaIF2 et Ad7HpaIR2.
- Purifier le produit PCR (2584 pb) après résolution dans un gel d’agarose à 1 % par électrophorèse. Régler la réaction PCR comme suit : un cycle à 98 °C pendant 30 s, puis 30 cycles de dénaturation à 98 °C pendant 8 s, recuit et extension à 72 °C pendant 2 min.
REMARQUE : La partie centrale de Kan-Ori2 est le fragment contenant Kan et Ori, suivi des séquences d’une endonucléase de restriction unique qui n’existe pas dans le petit fragment pKAd7-HpaI-FS (par exemple, Pme I), et la région qui se chevauche avec le grand fragment pKAd7-HpaI-FL ; les séquences les plus externes sont la région qui se chevauche avec le petit fragment pKAd7-HpaI-FS.
- Purifier le produit PCR (2584 pb) après résolution dans un gel d’agarose à 1 % par électrophorèse. Régler la réaction PCR comme suit : un cycle à 98 °C pendant 30 s, puis 30 cycles de dénaturation à 98 °C pendant 8 s, recuit et extension à 72 °C pendant 2 min.
- Ajoutez le produit PCR Kan-Ori2 et le pKAd7-HpaI-FS récupéré à l’étape 5.1 à DNA Assembly Master Mix. Incuber à 50 °C pendant 1 h. Suivez les étapes 2.4 à 2.6. Le plasmide correct est nommé pKan-Ad7HpaI (~10 kb).
- Amplifiez le fragment CMV-GFP-PA (~1,7 kb) contenant le promoteur CMV, le CDS GFP et le signal polyA SV40 (PA) en utilisant pShuttle-GFP23 comme modèle avec les amorces Ad7E3GFPF1 et Ad7E3GFPR1 (Tableau S1). Purifier le produit PCR (1650 pb) après résolution dans un gel d’agarose à 1 % par électrophorèse.
REMARQUE : Les régions qui se chevauchent et qui peuvent être assemblées avec le pKan-Ad7HpaI digéré par BstZ17 I et Mlu I doivent être incluses dans les amorces, et un site de restriction unique (par exemple, Sbf I) qui n’existe pas dans le plasmide pKan-Ad7 est ajouté dans les amorces pour un assemblage de restriction supplémentaire. - Digestion de 300 ng de pKan-Ad7HpaI avec BstZ17 I et Mlu I. Purifier le fragment de 7,3 kb (la plupart des régions E3 sont supprimées) après résolution dans un gel d’agarose à 1 % par électrophorèse. Ajoutez le fragment récupéré de 7,3 kb et le fragment CMV-GFP-PA récupéré (~1,6 kb) à DNA Assembly Master Mix. Suivez les étapes 2.4 à 2.6. Le plasmide correct est nommé pKAd7-E3GFP (~9 kb).
REMARQUE : Toutes les séquences entre BstZ17 I et Mlu I appartiennent à la région E3. - Digérer 300 ng de pKAd7-HE3GFP avec Pme I. Purifier le gros fragment (~6,4 kb) à l’aide d’un kit de récupération de gel d’ADN après électrophorèse sur gel. Ajoutez le fragment purifié et le pKAd7-HpaI-FL récupéré à l’étape 5.1 à l’ADN Assembly Master Mix. Incuber à 50 °C pendant 1 h. Suivez les étapes 2.4 à 2.6. Le plasmide correct est nommé pKAd7-E3GFP.
- Cellules HEK293 transfectrices avec Pme I-linéarisé pKAd7-E3GFP comme décrit dans la section 3. L’adénovirus sauvé est nommé Ad7-E3GFP.
- Amplifiez le fragment CMV-mCherry-PA (~1,6 kb) contenant le promoteur CMV, le mCherry CDS et le signal polyA SV40 en utilisant pKFAV4-CX19A comme modèle avec les amorces de Ad7E3CHEF1 et Ad7E3CHER1. Purifier le produit PCR après résolution dans un gel d’agarose à 1 % par électrophorèse.
- Digérer 600 ng de pKAd7-E3GFP avec Sbf I, et purifier le fragment de ~35 kb. Ajoutez le fragment de 35 kb et le fragment CMV-mCherry-PA récupéré à DNA Assembly Master Mix. Incuber à 50 °C pendant 1 h. Suivez les étapes 2.4 à 2.6. Le plasmide correct est nommé pKAd7-E3CHE.
- Cellules HEK293 transfectées avec Pme I-linéarisé pKAd7-E3CHE comme décrit dans la section 3. L’adénovirus sauvé est nommé Ad7-E3CHE.
6. Amplification et purification de l’adénovirus recombinant dans les cellules HEK293
- Inoculer une monocouche confluente de 80 à 90 % de cellules HEK293 cultivées dans huit boîtes de culture de 150 mm de diamètre traitées par TC avec adénovirus. Incuber à 37 °C pendant 2 h sous une légère agitation. Remplacer le surnageant par 25 mL de DMEM contenant 2 % de FBS.
- Incuber les cellules à 37 °C pendant 2-3 jours.
- Jetez la majeure partie du surnageant des huit paraboles de 150 mm de diamètre lorsqu’une EPC affecte la majorité des cellules. Récoltez les cellules infectées à l’aide d’élévateurs de cellules d’un volume d’élution final de ~6,0 mL dans des tubes en polypropylène de 15 mL.
- Perturbez les cellules par trois cycles de gel-dégel pour libérer le virus. Éclaircir le lysat par centrifugation pendant 12 min à 1200 x g à température ambiante. Prélever le surnageant contenant le virus.
- Ajouter du MgCl2 (0,1 mM) et de la nucléase de benzonase (50 U/mL) au surnageant. Agitez doucement pendant 40 min à 37 °C. Ajouter du NaCl (600 mM) au surnageant et agiter doucement pendant encore 40 min à 37 °C. Centrifuger pendant 12 min à 1200 x g à température ambiante. Prélever le surnageant contenant le virus pour la purification.
- Préparez du chlorure de césium (CsCl) avec différentes concentrations de 1,35 g/mL, 1,30 g/mL et 1,25 g/mL avec 10 mM de Tris-HCl.
- Ajouter 0,8 mL de solution de CsCl de 1,35 g/mL dans le fond d’un tube Ultra-Clear de 5,1 mL à l’aide d’une pipette, puis ajouter lentement 0,8 mL de solution de CsCl de 1,30 g/mL et 0,8 mL de solution de CsCl de 1,25 g/mL sur la première solution de manière séquentielle.
- Enfin, superposer soigneusement ~2,5 mL du surnageant recueilli à l’étape 6.3 sur une solution de CsCl de 1,25 g/mL, et remplir le tube à 2-3 mm du haut avec 10 mM de Tris-HCl.
- Centrifugeuse pendant 2 h à 200 000 x g et 8 °C dans un rotor à godet oscillant avec accélération et décélération lentes pour séparer les particules virales intactes des particules virales défectueuses.
REMARQUE : Après centrifugation, plusieurs bandes blanches sont clairement visibles dans la solution de CsCl. - Retirez avec précaution les bandes des particules vides et la couche de débris cellulaires à l’aide de la pipette, et prélevez la bande la plus basse contenant le virus mature à l’aide d’une seringue de 2 ml.
- Transvasez la suspension virale recueillie dans une cassette de dialyse MWCO de 20 K et dialysez dans un tampon de dialyse contenant 10 mM de Tris-HCl, 150 mM de NaCl, 1 mM de MgCl2 et 2 % de glycérol (pH 8,0) pendant la nuit à 4 °C.
- Remplacez le tampon de dialyse contenant 10 mM de Tris-Cl, 150 mM de NaCl, 1 mM de MgCl2 et 5 % de glycérol (pH 8,0) le lendemain et dialysez 5 h à 4 °C.
- Prélever les particules virales dialysées à l’aide d’une seringue de 2 ml. Préparez plusieurs aliquotes des petits volumes souhaités (20-200 μL) et stockez le virus purifié à -80 °C.
REMARQUE : Le titre de particules du virus purifié peut être déterminé en mesurant le contenu de l’ADN génomique, et le titre d’infectiosité peut être déterminé sur les cellules HEK293 en comptant les cellules GFP positives avec le test de dilution limitative.
7. Identification du génome d’un adénovirus recombinant par digestion enzymatique de restriction
- Infectez une monocouche confluente de 80 à 90 % de cellules HEK293 cultivée dans un flacon T-25 avec un adénovirus recombinant. Incuber pendant 2 h à 37 °C. Remplacer le milieu de culture par du DMEM frais contenant 2 % de FBS.
- Retirer le surnageant de culture et extraire l’ADN génomique viral à l’aide de la méthode de Hirt modifiée décrite à l’étape 1.3 lorsque l’EPC se produit dans les 2 à 3 jours.
- Digérer l’ADN génomique de l’adénovirus recombinant avec les enzymes indiquées, et analyser sur électrophorèse sur gel d’agarose. Le plasmide adénoviral recombinant a servi de témoin.
Résultats
La stratégie de construction d’un clone infectieux de HAdV-7 est illustrée aux figures 1 et 2. Deux plasmides clones infectieux ont été sélectionnés au hasard et identifiés par BstZ17 I, BamH I et EcoR V, respectivement. Les résultats ont montré que les fragments étaient cohérents avec la taille attendue (Figure 2A), indiquant que les plasmides étaient correctement construits. Des foyers de comètes ont pu être observés dans les cellules 12 jours après la transfection du plasmide linéarisé Pme I dans les cellules HEK293 (Figure 2B), ce qui indique que le virus a été sauvé avec succès.
Dans cette étude, le vecteur adénoviral supprimé E3 a été généré pour surexprimer la GFP (Figure 4) ou mCherry à titre d’exemples. Lors de la modification de la région E3, un plasmide intermédiaire pKan-Ad7HpaI (Figure 3) a d’abord été construit. Les plasmides de trois colonies ont été analysés avec digestion EcoR V et Hind III, les résultats de l’électrophorèse ont montré que tous les plasmides étaient corrects (Figure 5A). De plus, le chromatogramme de séquençage a montré qu’il n’y a pas de mutation au niveau des sites d’assemblage (Figure 5C, des résultats partiels ont été présentés). Après que la région E3 entre BstZ17 I et Mlu I a été supprimée et remplacée par la cassette CMV-GFP-PA, trois des plasmides modifiés pKAd7-HE3GFP ont été identifiés par digestion et séquençage par enzyme de restriction (données non présentées). Les résultats ont montré que les fragments digérés étaient exactement comme prévu, ce qui indique que les plasmides ont été construits correctement (Figure 5B).
Après modification, le plasmide pKAd7-E3GFP résultant contient la cassette CMV-GFP-PA flanquée de deux sites Sbf I (Figure 4), qui pourrait être utilisée pour remplacer la cassette CMV-GFP-PA par d’autres gènes de manière opportune. L’expression de la GFP a pu être observée au microscope à fluorescence 48 h après que les cellules HEK 293 aient été transfectées avec pKAd7-E3GFP linéarisée Pme I, et les foyers fluorescents et CPE ont pu être observés 13 jours après la transfection (Figure 6). Les cellules et le milieu ont été récoltés et utilisés pour inoculer des cellules HEK293 après trois cycles de congélation-décongélation ; L’expression de la GFP et de l’EPC a pu être observée 48 h après l’infection (Figure 6). Selon le protocole de purification décrit, les particules virales concentrées sont apparues sous la forme de plusieurs bandes de virion opalescents au sein du gradient CsCl (Figure 7A). La bande la plus basse au-dessous de 1,35 g/cm3 CsCl a été prélevée et utilisée pour la coloration négative à l’acide phosphotungstique (PTA), et l’adénovirus icosaédrique intact a pu être observé au microscope électronique (figure 7B). La bande supérieure (plus légère) contenait des particules vides et non infectieuses et devait être évitée. Dans le cadre de ce protocole, huit boîtes de culture tissulaire T-150 cm2 ont été utilisées, pour lesquelles 1,5 mL de stock Ad7-E3GFP avec un titre infectieux de 2,3 x 1011 UI/mL et un titre de particules virales de 6,0 x 1012 vp/mL ont été produits. Pour identifier l’intégrité génomique de l’Ad7-E3GFP recombinant, l’ADN génomique a été digéré avec une endonucléase de restriction indiquée et séparé par électrophorèse ; tous les fragments étaient conformes aux attentes (figure 7C).
En utilisant ce protocole, le CMV-GFP-PA dans pKAd7-E3GFP pourrait être remplacé par le gène d’intérêt dans un délai de 2 à 3 jours. Ici, le CMV-GFP-PA a été substitué au CMV-mCherry-PA en utilisant l’assemblage-restriction, et le nouveau plasmide adénoviral recombinant pKAd7-E3CHE a été utilisé pour transfecter les cellules HEK293 après avoir été linéarisé par Pme I. L’expression de mCherry a pu être détectée dans les 24 heures suivant la transfection, et les foyers fluorescents se sont formés 15 jours après la transfection (Figure 8A). L’expression de mCherry et de CPE a pu être observée dans les cellules HEK293 48 h après l’inoculation avec l’Ad7-E3CHE sauvé. Les résultats de l’électrophorèse après digestion par endonucléase de restriction ont montré que le génome d’Ad7-E3CHE était tout à fait conforme aux attentes (Figure 8B).

Figure 1 : Schéma de principe de la construction de clones infectieux de l’adénovirus humain 7 (HAdV-7) à l’aide de l’assemblage de l’ADN. Les amorces (marquées en jaune) contiennent non seulement les séquences appariées à la matrice, mais aussi les régions qui se chevauchent (chevauchement) qui peuvent être assemblées avec le génome HAdV-7. De plus, la séquence d’un site d’endonucléase de restriction (par exemple, Pme I) qui est absente à la fois dans le génome de HAdV-7 et dans le squelette plasmidique devrait également être incluse dans les amorces pour faciliter la libération du génome. Le squelette plasmidique amplifié Kan-Ori a été assemblé avec l’ADN génomique viral de HAdV-7 in vitro pour former le plasmide clone infectieux pKan-Ad7. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Identification du clone infectieux de HAdV-7. (A) Deux des plasmides de clones infectieux pKan-Ad7 ont été digérés avec BstZ17 I, BamH I et EcoR V, respectivement. Les poids moléculaires prédits des fragments digérés étaient de 716, 947, 1164, 3460, 4349, 5828, 8116 et 13 138 pb pour BstZ17 I ; 5713, 6945, 9058 et 16 002 pb pour BamH I ; 2338, 4048, 4162, 7792, 9306 et 10 072 pb pour EcoR V. (B) Sauvetage de HAdV-7. Le pKan-Ad7 linéarisé Pme I a été utilisé pour transfecter les cellules HEK293. L’EPC s’est produite au 12e jour après la transfection, ce qui indique que le sauvetage de HAdV-7 a réussi. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Construction in silico du plasmide intermédiaire. (A) Carte plasmidique du clone infectieux pKan-Ad7. Les sites enzymatiques de restriction de coupe uniques et doubles sont illustrés. Hpa I, Sal I et Avr II-Mlu I peuvent être sélectionnés pour la construction d’un plasmide intermédiaire contenant la région E3. Pour construire un plasmide intermédiaire plus petit, Hpa I a finalement été choisi pour mener les expériences. (B) Carte du plasmide intermédiaire pKan-Ad7HpaI. On voit les sites de restriction de tous les couteaux uniques et de certains doubles. La région E3 entre les sites BstZ17 I et Mlu I a finalement été remplacée par une cassette d’expression GFP contrôlée par un promoteur CMV. (C) Conception d’amorce pour amplifier le squelette plasmidique. Le produit PCR du squelette plasmidique serait fusionné au fragment court de pKan-Ad7 digéré par Hpa I pour générer le plasmide intermédiaire pKan-Ad7HpaI par assemblage Gibson. Les apprêts conçus sont illustrés. Les chevauchements pour les sites d’assemblage Gibson et de Pme I seraient intégrés au produit par deux cycles de PCR en utilisant des amorces Ad7HpaIF1/R1 et Ad7HpaIF2/R2 en séquence. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Schéma de principe de la construction du plasmide adénoviral recombinant pKAd7-E3GFP à l’aide de l’assemblage par restriction. Veuillez cliquer ici pour voir une version agrandie de cette figure.
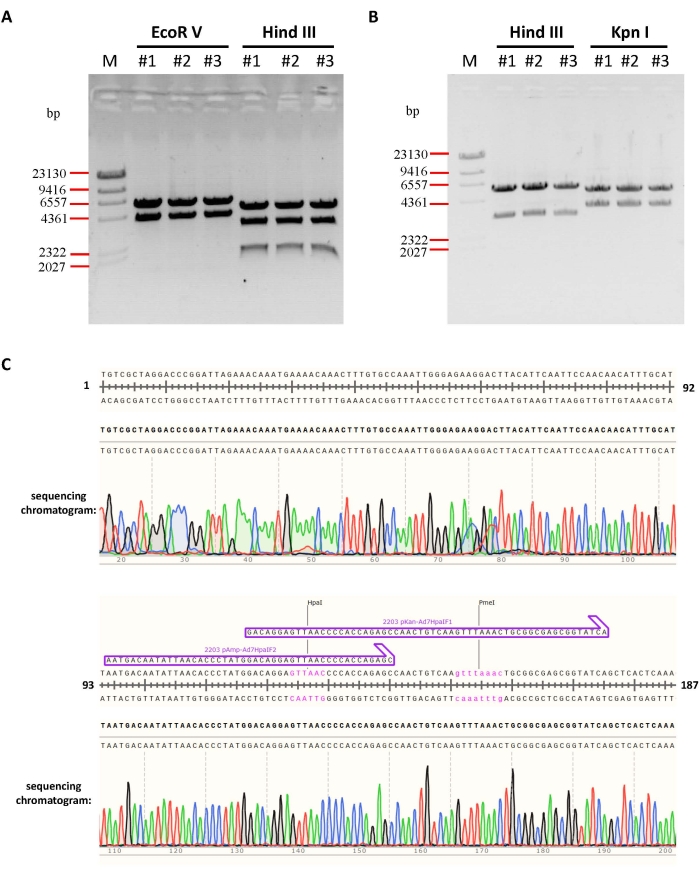
Figure 5 : Identification du plasmide intermédiaire par digestion et séquençage. (A) Trois des plasmides intermédiaires pKan-Ad7HpaI ont été extraits et identifiés par digestion d’endonucléase de restriction indiquée. Les poids moléculaires prédits des fragments digérés étaient de 4048 et 5713 pb pour EcoR V, et de 1710, 3206 et 4845 pb pour Hind III. (B) Trois des plasmides pKAd7-HE3GFP ont été extraits et identifiés par digestion d’endonucléases de restriction. Les poids moléculaires prédits des fragments digérés étaient de 3206 et 5709 pb pour Hind III, et de 3725 et 5190 pb pour Kpn I. (C) Les sites de fusion du plasmide pKan-Ad7HpaI ont été confirmés par séquençage. En raison de l’espace limité, seuls les résultats de séquençage d’un site de fusion du plasmide ont été présentés ici. Veuillez cliquer ici pour voir une version agrandie de cette figure.
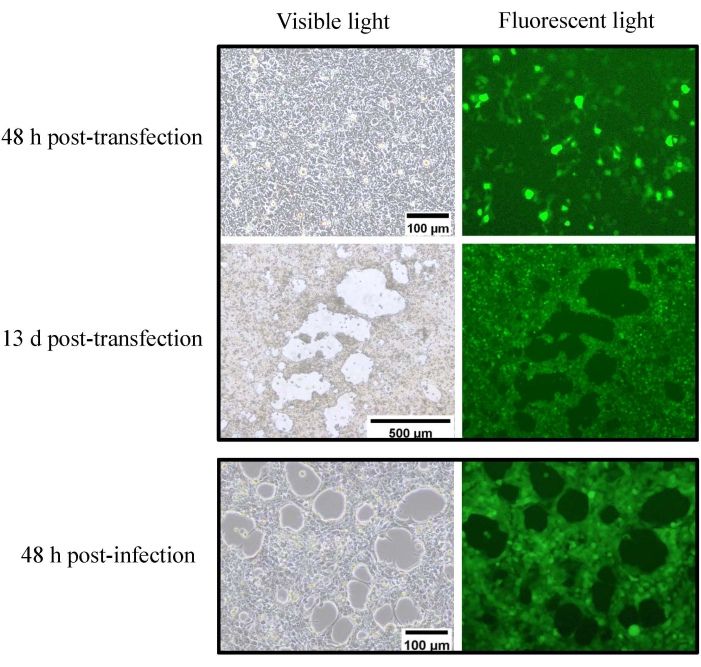
Figure 6 : Sauvetage et amplification de l’adénovirus recombinant Ad7-E3GFP. Les cellules HEK293 à 90 % de confluence (fiole T-25) ont été transfectées avec 5 μg de pKAd7-E3GFP linéarisée Pme I à l’aide de réactifs de transfection commerciaux. L’expression de la GFP et de la CPE a pu être observée au microscope à fluorescence 48 h et 13 jours après la transfection, respectivement. Les cellules et le milieu ont été collectés et utilisés pour inoculer des cellules HEK293 ; L’expression de la CPE et de la GFP a été observée 48 h après l’infection. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 7 : Purification et identification de l’adénovirus recombinant Ad7-E3GFP. (A) Purification de l’Ad7-E3GFP. Les lysats cellulaires contenant des virus ont été chargés sur le gradient CsCl, et des bandes virales ont pu être observées après ultracentrifugation. (B) Observation de l’Ad7-E3GFP purifié sous EM. (C) Identification de l’ADN génomique Ad7-E3GFP. L’ADN génomique a été digéré avec une endonucléase de restriction indiquée après extraction des cellules infectées. Le plasmide pKAd7-E3GFP a servi de témoin. Les poids moléculaires prédits des fragments digérés d’ADN génomique Ad7-E3GFP étaient de 530, 1191, 2368, 5310, 10 065 et 14 937 pb pour Afl II ; 7774, 11 006 et 15 621 pb pour l’Asc I ; 531, 2623, 4789, 7413, 9163 et 9882 pb pour BsrG I ; 342, 2338, 2432, 2860, 4162, 4403, 7792 et 10 072 pb pour EcoR V ; 1005, 3725, 6084, 9570 et 14 017 pb pour Kpn I. Les poids moléculaires prédits des fragments digérés du plasmide pKAd7-E3GFP étaient de 1191, 5310, 5369, 10 065 et 14 937 pb pour Afl II ; 7774 et 29 098 pb pour l’Asc I ; 531, 2623, 9163, 9882 et 14 673 pb pour BsrG I ; 342, 2338, 2860, 4162, 7792, 9306 et 10 072 pb pour EcoR V ; 1005, 3725, 9570 et 22 572 pb pour Kpn I. M : marqueur d’ADN lambda/HindIII. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 8 : Génération, amplification et identification de l’adénovirus recombinant Ad7-E3CHE. (A) Sauvetage et amplification de l’adénovirus recombinant Ad7-E3CHE. Les cellules HEK293 à 90 % de confluence (fiole T-25) ont été transfectées avec 5 de μg de Pme I linéarisé pKAd7-E3CHE à l’aide d’un réactif de transfection. Des foyers fluorescents et des EPC ont pu être observés au microscope à fluorescence 15 jours après la transfection. Les cellules et le milieu ont été collectés et utilisés pour inoculer des cellules HEK293. L’expression de CPE et de mCherry a pu être observée 48 h après l’infection. (B) Identification de l’ADN génomique Ad7-E3CHE. L’ADN génomique a été digéré avec une endonucléase de restriction indiquée après extraction des cellules infectées. Le plasmide pKAd7-E3 CHE a servi de témoin. Les poids moléculaires prédits des fragments digérés d’ADN génomique Ad7-E3CHE étaient de 379, 653, 2619, 3766, 5721, 8335 et 12 939 pb pour l’âge I ; 716, 750, 947, 1164, 2607, 3460, 4349, 7281 et 13 138 pb pour BamH I ; 1005, 3725, 6095, 9570 et 14 017 pb pour Kpn I ; 1635, 4256, 6853 et 21 668 pb pour Nde I ; 4810, 5920, 6360 et 17 322 pb pour Sal I. Les poids moléculaires prédits des fragments digérés du plasmide pKAd7-E3CHE étaient de 379, 653, 2619, 3766, 5721 et 23 745 pb pour l’âge I ; 716, 947, 1164, 3460, 4349, 5828, 7281 et 13 138 pb pour BamH I ; 1005, 3725, 9570 et 22 583 pb pour Kpn I ; 4256, 10 959 et 21 668 pb pour Nde I ; 5920, 13 641 et 17 322 pb pour Sal I. M : marqueur d’ADN lambda/HindIII. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau S1 : Renseignements sommaires sur les amorces. Veuillez cliquer ici pour télécharger ce tableau.
| Plasmide de produit | Taille (bp) | Réaction d’assemblage de l’ADN | Extraction de plasmides | Analyse des restrictions | Séquençage de Sanger | |
| Fragment de grande taille (bp) | Petit fragment (bp) | (colonies cueillies/avec taille prévue) | (digéré/correct) | (séquencé/correct) | ||
| pKan-Ad7 | 37718 | 35239 | 2543 | 6/5 | 2/2 | 2/2 |
| pKan-Ad7HpaI | 9761 | 7235 | 2584 | 6/6 | 3/3 | 3/3 |
| pKAd7-HE3GFP | 8915 | 7316 | 1650 | 6/6 | 3/3 | 3/3 |
| pKAd7-E3GFP | 36872 | 30483 | 6444 | 3/2 | 2/2 | 2/2 |
| pKAd7-E3CHE | 36883 | 35277 | 1655 | 3/3 | 2/2 | 2/2 |
Tableau 1 : Données récapitulatives de la construction des plasmides. La procédure d’assemblage par restriction de la construction plasmidique comprenait la préparation de fragments, la réaction d’assemblage de l’ADN, la transformation bactérienne, l’extraction du plasmide, l’analyse de restriction et la confirmation du séquençage. Généralement, des dizaines ou des centaines de colonies pourraient être produites sur une plaque LB après transformation de cellules compétentes d’E. coli TOP10 avec le produit d’assemblage de l’ADN. Les colonies ont été prélevées et propagées au hasard, et l’ADN du plasmide a été extrait et digéré avec de l’endonucléase et le motif des bandes du plasmide digéré a été photographié et analysé après électrophorèse. Enfin, l’ensemble du plasmide ou des produits de PCR couvrant les sites de fusion ou les régions amplifiées par PCR dans le plasmide a été soumis au séquençage de Sanger.
Figure S1 : Plasmide intermédiaire virtuel pour la modification des régions E1 et E3. Le long fragment de pKan-Ad7 digéré par Sal I peut être utilisé pour construire le plasmide intermédiaire pKan-Ad7SalI. On voit les sites de restriction de tous les couteaux uniques et de certains doubles. La région E1, qui se situe entre les sites SnaB I et MauB I, peut être modifiée précisément par PCR d’extension de chevauchement. De même, Spe I avec BsrG I peut aider à modifier la région E3. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Différents adénovirus ont différents tropismes tissulaires, et la prévalence de l’immunité préexistante de l’hôte contre différents adénovirus peut fluctuer intensément chez les êtres humains24, ce qui suscite l’intérêt pour la construction de nouveaux vecteurs adénoviraux pour la thérapie génique ou le développement de vaccins. Cependant, la mise en place d’un nouveau système de vecteurs adénoviraux reste lourde pour les laboratoires génériques de biologie moléculaire.
Ici, nous avons introduit un protocole pour générer des vecteurs à partir d’adénovirus de type sauvage en utilisant la digestion de restriction et les techniques d’assemblage Gibson 13,14,15,16,17,18,19. En général, ce processus peut être divisé en trois étapes. Tout d’abord, le produit PCR du squelette plasmidique est mélangé à l’ADN génomique de l’adénovirus, et l’assemblage de Gibson est utilisé pour générer un clone infectieux. La conception d’amorces PCR est le facteur clé : les extrémités 3' des amorces sont des séquences spécifiques pour amplifier le squelette plasmidique qui contient l’origine de la réplication et le gène résistant aux antibiotiques, et les extrémités 5' sont les séquences inverses du complément homologues à l’extrémité de l’adénovirus ITR. De plus, un site de restriction non coupeur du génome viral est ajouté au milieu, qui est utilisé pour libérer le génome viral du clone d’ADN infectieux. Deuxièmement, une stratégie intermédiaire basée sur des plasmides est mise en œuvre pour construire un système de vecteurs adénoviraux. Un fragment contenant la région à modifier est excisé du clone infectieux et utilisé pour construire un plasmide intermédiaire. La PCR par extension de chevauchement est la principale approche à adopter pour la modification dirigée par le site, telle que la suppression de gènes non essentiels et l’insertion de la cassette d’expression génique exogène. Le fragment de virus modifié est ensuite excisé du plasmide intermédiaire par digestion de restriction et restauré dans la région correspondante du clone d’ADN infectieux pour générer un plasmide de vecteur adénoviral. Pour cette étape, l’analyse de séquence est d’une grande importance pour déterminer la région à supprimer et le site d’insertion idéal pour le gène d’intérêt. En général, un double cutter est sélectionné pour couper le clone d’ADN infectieux afin de générer un petit et un grand fragment, tandis que la région cible pour une modification ultérieure est située au niveau du petit fragment, avec lequel le plasmide intermédiaire est construit. Le plasmide adénoviral est linéarisé et utilisé pour transfecter les cellules d’emballage afin de sauver les virus recombinants. Enfin, la stratégie de chargement des transgènes a déjà été envisagée en amont et intégrée dans le système vecteur. La cassette d’expression du transgène dans le plasmide adénoviral peut être facilement retirée et remplacée par d’autres transgènes par restriction-assemblage grâce aux sites de coupe doubles préservés qui flanquent cette région.
Un clone infectieux fournit un point de départ solide pour la construction d’un nouveau système de vecteurs adénoviraux. En général, la méthode de recombinaison homologue chez les bactéries est utilisée pour construire des clones infectieux d’adénovirus5. La PCR est effectuée pour amplifier les séquences aux deux extrémités du génome adénoviral en tant que bras homologues de 500 à 2000 pb de longueur, et les produits de PCR sont ligaturés avec un squelette plasmidique pour former un petit plasmide. Le petit plasmide est linéarisé, mélangé à de l’ADN génomique d’adénovirus et utilisé pour transformer la souche d’E. coli BJ5183 par électroporation. Le produit de recombinaison, un plasmide clone infectieux, doit être identifié et transféré dans une souche d’E. coli négative à la recombinase pour l’amplification du plasmide. L’ensemble de la procédure contient au moins trois cycles de transformation bactérienne et dure environ 1 semaine, en supposant qu’aucun événement inattendu ne se produise. L’assemblage Gibson simplifie considérablement la construction de clones infectieux d’adénovirus. Par rapport à la recombinaison homologue, elle présente les avantages suivants13 : Tout d’abord, l’assemblage de l’ADN est beaucoup plus simple et plus efficace car un seul cycle de transformation bactérienne est nécessaire. Deuxièmement, l’assemblage de l’ADN préserve le génome de l’adénovirus dans un clone infectieux avec une plus grande fidélité, car les bactéries positives à la recombinase, chez lesquelles les plasmides peuvent être instables, sont exclues de l’ensemble de la procédure. Enfin, moins d’informations de séquence sont nécessaires pour l’assemblage de l’ADN, car des chevauchements aussi courts que 15 à 30 pb sont suffisants pour un assemblage efficace, ce qui signifie que le génome peut être cloné à partir d’un adénovirus inconnu même s’il n’y a que des informations de séquence à la fin de l’ITR.
L’une des limites de cette approche est qu’il est souvent compliqué et fastidieux de trouver l’endonucléase appropriée. Ce processus traverse l’ensemble du protocole, y compris la construction du plasmide de clone infectieux, du plasmide intermédiaire et de la modification de la région d’intérêt (ROI). Parfois, une enzyme appropriée pour modifier le ROI ne peut pas être trouvée dans le plasmide intermédiaire. Dans ce cas, il est nécessaire de construire un autre plasmide intermédiaire qui dépend également du logiciel d’analyse de séquence d’ADN pour rendre le plasmide plus petit et faciliter la recherche d’endonucléases de restriction appropriées. Parfois, la réaction d’assemblage de l’ADN peut produire quelques clones sans l’insert, car certaines enzymes de restriction ne coupent pas le plasmide jusqu’à son terme. Dans ce cas, sélectionnez deux endonucléases de restriction différentes plutôt qu’une enzyme unique pour digérer le vecteur, ce qui éviterait l’auto-recuit des surplombs. Une autre limitation de l’assemblage de l’ADN est qu’une vérification du séquençage est nécessaire après chaque réaction, car il existe un risque de non-concordance dans cette méthode. Cependant, les mauvaises réactions d’assemblage ne se produisent pas très souvent. Des dizaines ou des centaines de colonies peuvent être cultivées dans chaque plaque LB après avoir transformé des cellules compétentes d’E. coli TOP10 avec un produit d’assemblage d’ADN, et les plasmides avec le mauvais assemblage sont rarement trouvés (Tableau 1).
En résumé, nous avons montré ici comment générer un vecteur adénoviral supprimé E3 à partir d’un HAdV-7 isolé de type sauvage, et l’assemblage de restriction que nous avons introduit est une méthode très efficace, facile à utiliser et polyvalente pour la construction et la modification de vecteurs adénoviraux.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Cette recherche a été financée par la Fondation des sciences naturelles de Pékin (7204258), la Fondation nationale des sciences naturelles de Chine (82161138001, 82072266), le Fonds d’innovation CAMS pour les sciences médicales (2019-I2M-5-026), et la recherche et l’application sur le traçage moléculaire des agents pathogènes respiratoires essentiels à Pékin, par le Capital Health Development and Research of Special (2021-1G-3012).
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL polypropylene microcentrifuge Tube | Axygen | MCT-150-C | Storage of virus |
| 15 mL polypropylene centrifuge tubes | Corning | 430790 | Storage of virus |
| 150 mm TC-treated culture dishes | Corning | 430599 | Growth of HEK29E cells |
| 20 K MWCO dialysis cassette | ThermoFisher Scientific | 66005 | Dialysis of virus |
| Acetic acid | Amresco | 714 | Extraction of DNA |
| Afl II | NEB | R0520 | Digestion |
| Agarose | Takara | 5260 | Electrophoresis |
| Age I | NEB | R0552 | Digestion |
| Asc I | NEB | R0558 | Digestion |
| BamH I | NEB | R0136 | Digestion |
| Benzonase Nuclease | Sigma | E8263-25KU | Purification of virus |
| BsrG I | NEB | R0575 | Digestion |
| Cell lifter | Corning | 3008 | Scrape off the cells |
| CsCl | Sigma | C3032 | Purification of virus |
| DNA gel recovery kit | Zymo | D4045 | Recovery of DNA |
| Dulbecco’s modified Eagle’s medium (DMEM) | Cytiva | SH30022.01 | HEK293 cells medium |
| E.coli TOP10 competent cells | TIANGEN BIOTECH (BEIJING) CO.,LTD. | CB-104 | Transformation of assembly product |
| EcoR V | NEB | R3195 | Digestion |
| EDTA | Thermo Fisher Scientific | R3104 | Extraction of DNA |
| Fetal bovine serum (FBS) | Cytiva | SV30208.02 | HEK293 cells culture |
| Genomic DNA Clean and concentrator kit | Zymo | D4065 | Purification of DNA |
| Glycerol | Shanghai Macklin Biochemical Co., Ltd | G810575 | Dialysis of virus |
| HEK293 cells | ATCC | CRL-1573 | Amplification of virus |
| High-Fidelity DNA Polymerase | NEB | M0491 | PCR |
| Hind III | NEB | R3104 | Digestion |
| Kanamycin sulfate | Amresco | 408 | Selection of plasmid |
| Kpn I | NEB | R3142 | Digestion |
| Lambda/HindIII DNA marker | Takara | 3403 | Electrophoresis |
| LB broth | BD | 240230 | LB plate for bacteria |
| LB medium | Solarbio Life Science | L1010 | Medium for bacteria |
| MgCl2 | Sigma | 63068 | Dialysis of virus |
| Microcentrifuge | Thermo Fisher Scientific | Sorvall Legend Micro 21R | Extraction of DNA |
| NaCl | Sigma | S5886 | Dialysis of virus |
| Nde I | NEB | R0111 | Digestion |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621 | DNA assembly |
| Nhe I | NEB | R0131 | Digestion |
| Phosphate Buffered Saline | Cytiva | SH30256.01 | Washing of cells |
| Pipette | Thermo Fisher Scientific | Matrix | Aspirate the medium |
| Plasmid Maxprep Kit | Vigorous Biotechnology Beijing Co., Ltd. | N001 | Extraction of DNA |
| Plasmid Miniprep Kit | TIANGEN BIOTECH (BEIJING) CO.,LTD. | DP103 | Extraction of DNA |
| Pme I | NEB | R0560 | Digestion |
| Potassium acetate | Amresco | 698 | Extraction of DNA |
| Protease K | Thermo Fisher Scientific | AM2542 | Extraction of DNA |
| pShuttle-CMV | Stratagene | 240007 | PCR template |
| RNase | Beyotime | D7089 | Extraction of DNA |
| Sal I | NEB | R0138 | Digestion |
| Sbf I | NEB | R3642 | Digestion |
| SDS | Amresco | 227 | Extraction of DNA |
| Swinging-bucket rotor | HITACHI | S52ST | Purification of virus |
| T-25 cell flask | Corning | 430639 | Growth of HEK29E cells |
| T-75 cell flask | Corning | 430641 | Growth of HEK29E cells |
| Transfection reagent | Polyplus-transfection | 114-15 | Transfection |
| Transmission electron microscope | FEI | TECNAI 12 | Obsevation of virus |
| Tris-HCl | Amresco | 234 | Dialysis of virus |
| Ultracentrifuge | HITACHI | Himac CS120GXII | Purification of virus |
Références
- Lasaro, M. O., Ertl, H. C. New insights on adenovirus as vaccine vectors. Molecular Therapy. 17 (8), 1333-1339 (2009).
- Fuchs, J. D., et al. Safety and immunogenicity of a recombinant adenovirus serotype 35-Vectored HIV-1 vaccine in adenovirus serotype 5 seronegative and seropositive individuals. Journal of AIDS & Clinical Research. 6 (5), (2015).
- Milligan, I. D., et al. Safety and immunogenicity of novel adenovirus type 26- and modified vaccinia ankara-vectored ebola vaccines: a randomized clinical trial. JAMA. 315 (15), 1610-1623 (2016).
- Zhu, F. C., et al. Safety, tolerability, and immunogenicity of a recombinant adenovirus type-5 vectored COVID-19 vaccine: a dose-escalation, open-label, non-randomised, first-in-human trial. Lancet. 395 (10240), 1845-1854 (2020).
- He, T. C., et al. A simplified system for generating recombinant adenoviruses. Proceedings of the National Academy of Sciences of the United States of America. 95 (5), 2509-2514 (1998).
- Danthinne, X., Imperiale, M. J. Production of first generation adenovirus vectors: a review. Gene Therapy. 7 (20), 1707-1714 (2000).
- Guo, X., et al. Systemic and mucosal immunity in mice elicited by a single immunization with human adenovirus type 5 or 41 vector-based vaccines carrying the spike protein of Middle East respiratory syndrome coronavirus. Immunology. 145 (4), 476-484 (2015).
- Folegatti, P. M., et al. Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS-CoV-2: a preliminary report of a phase 1/2, single-blind, randomised controlled trial. Lancet. 396 (10249), 467-478 (2020).
- Jang, Y., Bunz, F. AdenoBuilder: A platform for the modular assembly of recombinant adenoviruses. STAR Protocols. 3 (1), 101123(2022).
- Zhou, X., Sena-Esteves, M., Gao, G. Construction of recombinant adenovirus genomes by direct cloning. Cold Spring Harbor Protocols. 2019 (5), (2019).
- Ruzsics, Z., Lemnitzer, F., Thirion, C. Engineering adenovirus genome by bacterial artificial chromosome (BAC) technology. Methods in Molecular Biology. 1089, 143-158 (2014).
- Zhou, D., et al. An efficient method of directly cloning chimpanzee adenovirus as a vaccine vector. Nature Protocols. 5 (11), 1775-1785 (2010).
- Zou, X. H., et al. DNA assembly technique simplifies the construction of infectious clone of fowl adenovirus. Journal of Virological Methods. 257, 85-92 (2018).
- Guo, X., et al. Site-directed modification of adenoviral vector with combined DNA assembly and restriction-ligation cloning. Journal of Biotechnology. 307, 193-201 (2020).
- Liu, H., et al. Single plasmid-based, upgradable, and backward-compatible adenoviral vector systems. Human Gene Therapy. 30 (6), 777-791 (2019).
- Yan, B., et al. User-friendly reverse genetics system for modification of the right end of fowl adenovirus 4 genome. Viruses. 12 (3), 301(2020).
- Zhang, W., et al. Fiber modifications enable fowl adenovirus 4 vectors to transduce human cells. Journal of Gene Medicine. 23 (10), 3368(2021).
- Zou, X., et al. Fiber1, but not fiber2, is the essential fiber gene for fowl adenovirus 4 (FAdV-4). Journal of General Virology. 102 (3), 001559(2021).
- Guo, X., et al. Restriction-assembly: a solution to construct novel adenovirus vector. Viruses. 14 (3), 546(2022).
- Duan, Y., et al. Genetic analysis of human adenovirus type 7 strains circulating in different parts of China. Virologica Sinica. 36 (3), 382-392 (2021).
- Arad, U. Modified Hirt procedure for rapid purification of extrachromosomal DNA from mammalian cells. Biotechniques. 24 (5), 760-762 (1998).
- Knipe, D. M. H. P. M. Fields' Virology. 2, 1733(2013).
- Liu, H. Y., Han, B. J., Zhong, Y. X., Lu, Z. Z. A three-plasmid system for construction of armed oncolytic adenovirus. Journal of Virological Methods. 162 (1-2), 8-13 (2009).
- Mennechet, F. J. D., et al. A review of 65 years of human adenovirus seroprevalence. Expert Review of Vaccines. 18 (6), 597-613 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon