JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
토끼 근육에서 육-소포체 Ca2+-ATPase의 정제
요약
이 프로토콜은 최종 원심분리 단계에서 이당류 트레할로스를 포함하는 개선된 SERCA 정제 방법을 설명합니다. 이 탄수화물은 가혹한 조건에서 단백질을 안정화시킵니다. 정제된 SERCA는 촉매 활성을 보였고 순도가 높았기 때문에 구조 및 기능 연구에 적합했습니다.
초록
SARCO/endoplasmic reticulum Ca2+-ATPase(SERCA)와 같은 일부 P형 ATPase는 본질적으로 불안정한 막 단백질로, 높은 순도 및 구조적 품질로 촉매 활성 형태로 얻기 위해 정제 중에 특정 물리화학적 조건이 필요합니다. 이당류 트레할로스(trehalose)는 막과 단백질을 안정화하기 위해 효모 세포질에서 고농도로 합성되고 축적되는 호환 가능한 용질입니다. 원형질막 H+-ATPase의 정제를 위한 프로토콜에서 첨가제로 트레할로스를 사용하면 고품질 제제가 생성되며, 그 헥사메릭 구조는 생화학적 분석 방법으로 표시됩니다. 따라서 트레할로스는 막 단백질(P-ATPase)의 정제를 위한 안정화 첨가제로 사용할 수 있습니다. 이 프로토콜은 트레할로스 농도 구배에서 SERCA를 원심분리하여 SERCA 정제를 위한 기존 프로토콜의 수정을 설명합니다. 이 탄수화물의 포함은 SERCA의 순도가 높은 촉매 활성 형태로, 그리고 중요하게는 안정적인 형태로 정제되었습니다. 정제된 SERCA의 부분적인 생화학적 특성화(SDS-PAGE, 효소 반응속도학, FITC 라벨링, 원형 이색성 분광법)는 효소가 기능 및 구조 연구에 적합하다는 것을 보여주었습니다. P형 ATPase 및 기타 불안정한 막(및 세포소) 단백질의 정제 프로토콜에서 트레할로스의 사용이 제안됩니다.
서문
막 단백질/효소는 다양한 과정에서 중요한 역할을 하기 때문에 세포의 필수적인 생물학적 구성 요소입니다 1,2,3. 일부 기능에는 세포/내부 구획(능동/수동) 안팎으로 이온 및 분자의 이동, 세포-세포 인식, 세포 간 결합, 고정/부착 및 정상적이고 가혹한 물리적 및 화학적 조건(고염, 저수, 고온, 약물 내성, 등)3 그러므로, 막 단백질 및/또는 효소의 3차원(3D) 구조를 결정하는 것은 기초 및 응용 연구 4,7,6 모두에서 매우 중요해졌습니다. 중요한 것은 멤브레인 단백질/효소가 약물 발견(천연 또는 설계)의 표적으로 광범위하게 사용되어 왔다는 것입니다7,8,9. 즉, 막 단백질은 건강에 본질적으로 중요합니다 9,10,11.
막 단백질/효소의 소수성 특성은 실험 실험실에서 기술적으로 가장 까다로운 물리화학적 특성이며, 12,13,14, 올리고머 및/또는 매우 불안정한 일체형 막 단백질15,16으로 작업할 때 더욱 그렇습니다. 기능적 실험 분석 및 구조 연구를 위해 가능한 최고 품질의 막 단백질/효소를 적절한 양으로 분리하는 것이 매우 바람직하다17. 멤브레인 단백질은 본질적으로 소수성이기 때문에 정제하기가 매우 어렵고 세제의 선택은 일반적으로 고려해야 할 가장 중요한 문제 중 하나입니다 17,18,19,20. 이와 관련하여, 막 단백질을 분리하기 위해 사용되는 실험실 방법은 전형적으로 단백질(21)의 3D 구조 배열에 어느 정도의 손상을 일으킨다. 이러한 방법 중 일부에는 (a) 초음파 처리 (고주파 / 에너지 초음파), (b) 단백질 가용화 세제 (가혹, 중간 또는 부드러운) 22, (c) 컬럼 크로마토 그래피 및 고속 초 원심 분리에서 상대적으로 높은 압력23, (d) 침전 분자, (e) 소화 효소 및 기타21. 이러한 모든 과정은 정제 중 단백질 불안정화에 기여하거나 주요 원인이 될 수 있습니다21. 이러한 점에서, 일부 프로토콜은 주어진 막 단백질에 대해 상대적으로 잘 작동하는 것으로 보입니다. 그러나 만족스러운 결과를 얻기 위해 더 높은 품질의 막 단백질 전처리가 필요한 새로운 최신 분석 또는 방법을 사용할 때 최적화는 항상 환영받습니다24. 최적화 단계에는 구조 설계 개선, 막 단백질 발현을 위한 최적의 조건 찾기, 더 나은 취급 조건(즉, pH, 온도 등) 설정, 가장 적합한 세제 찾기, 초음파 처리 시간, 원심분리 속도와 같은 정제 단계 조정, 안정화제 첨가를 통한 완충액 재형성 등이 포함될 수 있지만 이에 국한되지 않습니다.25,26,27. 따라서 정제된 멤브레인 단백질/효소의 품질(순도)과 활성을 증가시키는 정제 방법의 변경이 중요합니다.
P형 ATPase 계열에서 효모 원형질막 H+-ATPase는 가장 불안정한 구성원 중 하나로 보입니다28,29. H+-ATPase는 탈수(동결 건조), 열 충격 등에 따라 ATP 가수분해 활성을 잃습니다.28,29. 단백질 안정제로서의 트레할로스의 사용은 효모 K. lactis30,31로부터 원형질막 H+-ATPase의 분리에서 시험되었다. 얻어진 H+-ATPase 제제는 순도가 높고 촉매 활성이었습니다. 중요한 것은, 이것은 효소의 올리고머 상태가 생화학적 방법으로 분해되어 헥사머로 밝혀지고 나중에 초저온 전자 현미경 31,32,33,34,35로 확인되었다는 것입니다. 그러므로, 막 단백질의 섬세한 3차원(3D) 배열은 정제36 동안 상대적으로 가혹한 조건 하에서 손실될 수 있는 것으로 보인다. 열 매개 불활성화 동안 H+-ATPase에 대해 Biphasic inactivation kinetic이 관찰됩니다29. 다른 많은 유기체에서와 마찬가지로 효모 세포에서 이당류 트레할로스는 환경 스트레스 조건 하에서 고농도로 축적됩니다 37,38,39. Trehalose는 단백질(막 내장 및 세포소 모두)과 세포막을 안정화하여 막 무결성 및 수송 기능을 유지합니다40,41. 트레할로스의 안정화 메커니즘은 여러 그룹과 본 연구실에서 광범위하게 연구되었습니다 42,43,44,45. H+-ATPase 및 다른 효소를 가진 실험은 trehalose가 mono- 및 disaccharides28,29 중 가장 효과적인 단백질 안정제임을 보여주었다. 이로 인해 H+-ATPase 정제 프로토콜30에 포함되었습니다. 최근에는 토끼 속경근에서 근망상 Ca2+-ATPase(SERCA)를 정화하는 데에도 트레할로스가 사용되어 단백질 순도와 활성이 우수한 결과를 보였다46. 그러므로, trehalose는 P형 ATPases 및 아마 다른 막 및 cytosolic 단백질의 정화를 위한 좋고 적당한 첨가물인 것 같습니다.
P-ATPase의 경우, 구조적 세포질 도메인의 존재는 특히 기질/리간드 상호 작용 연구에서 실험적 이점입니다47; ATP 결합은 고순도 재조합 N 도메인 47,48,49에서 연구되어 왔으며, 따라서 전막 효소 47,48,50 등의 정제에 대한 기술적 고려 사항을 제거했습니다51. 불행히도 일부 기능적(촉매/에너지 변환) 및 구조적(소단위 배열 및 다른 단백질과의 상호 작용) 연구에서는 여전히 전체 P-ATPase52,53을 필요로 합니다. 이와 관련하여, SERCA의 정화는 여러 연구 그룹에 의해 달성되었습니다 54,55,56,57,58. 그러나, 개선은 여전히 구현될 수 있습니다46, 예를 들어, 정제된 ATPase59의 온전함을 증가시키고, 단백질 복합체의 변성/파괴를 피하고(25), 막 단백질의 변성 없이 가용화를 증가시키고(즉, 고분자 응집체의 형성을 피함) 60, 분석 및 기타 다운스트림 분석 방법과의 더 나은 호환성을 제공합니다61 더욱이, 새로운 실험 전략, 첨가제, 효소 억제제 등이 과학 문헌 61,62,63,64,65,66에 나타나 있기 때문에 때때로 전체 P-ATPase로 테스트해야 합니다. 이 연구는 SERCA의 정제 및 단백질 구조 및 ATPase 활성을 안정화하기 위한 첨가제로 트레할로스를 사용하기 위한 프로토콜에 대해 설명합니다. 즉, 효소(구조적) 품질을 높이는 것 외에도 트레할로스는 효소 분리 및 저장 중 효소 구조 및 활성의 손실을 방지하는 데 도움이 되며, 이는 생물학적 물질을 저장하여 효소 정제 횟수를 줄이는 데 도움이 됩니다.
프로토콜
모든 동물 절차는 실험 실험실에서의 동물 취급에 대한 국제 및 지역(NORMA Oficial Mexicana NOM-062-ZOO-1999) 지침 51,67,68에 따라 수행되었습니다. 근육 조직은 지역 동물 취급 장치(INE/CITES/DGVS-ZOO-E0055-SLP-98)46의 야생형 Oryctolagus cuniculus에서 얻었습니다. 실험실 동물 관리에 대한 전문 지식을 갖춘 수의사가 초기 근육 절제 및 처리를 수행했습니다. 이 연구에 사용된 시약 및 장비의 세부 사항은 재료 표에 나열되어 있습니다.
1. 빠르게 움직이는 토끼 근육에서 근체망체의 분리(타이밍, 7 - 9시간)
참고: 절차에 대한 자세한 내용은 Champeil et al.69를 참조하십시오.
- 야생형 Oryctolagus cuniculus에서 속경 근육 조직(동물당 양쪽 뒷다리에서 ~70g)을 얻습니다.
참고: 절개할 근육에 대한 사전 육안 식별이 필요합니다70,71. - 빠르게 움직이는 근육을 믹서기에 100mM KCl의 3 볼륨으로 매달아 1 분 및 1 분 얼음처럼 차가운 휴식으로 3 회 반복72.
- 4°C에서 5분 동안 원심분리(1500 × g)로 조직 파편을 제거합니다. 필요한 경우 두 번째 원심분리(4200 × g)를 수행합니다.
- 상등액을 수집하고 조직 분쇄기(저속, 즉 500-1,500rpm에서 10회 스트로크)를 사용하여 균질화합니다.72,73. 균질액(10,000 × g)을 4°C에서 15분 동안 원심분리합니다.
알림: 상대적으로 높은 속도를 사용하면 시스템과 작업자가 손상될 수 있으므로 균질화할 때 주의해야 합니다. - 상층액(근소포체 함유)(33,000 × g)을 4°C에서 120분 동안 원심분리합니다.
- 펠릿을 40mL의 수크로오스 0.5mL와 원심분리기(12,000× g)에 4°C에서 15분 동안 현탁합니다. 상층액(0.6 M KCl 및 0.15 M 자당)을 희석하고 현탁액(34,000 × g)을 4°C에서 165분 동안 원심분리한다.
- 근질망 소포체(SRV) 시트를 포함하는 펠릿을 0.3M의 자당, 0.1M의 KCl 및 5mM의 Tris-HCl, pH 7.0에 현탁합니다. Lowry assay74 및 인간 혈청 알부민을 단백질 표준물질로 사용하여 단백질 농도를 측정합니다.
- SRV(30-32 mg/mL)를 1 mL 용량으로 분주합니다. 추가 처리를 위해 SRV 샘플을 -72°C에서 보관하십시오.
참고: 단백질 수율은 동물당 100-110mg SRV입니다.
2. SERCA 정화 (타이밍, 18 - 20 시간)
참고: 절차에 대한 자세한 내용은 Rivera-Morán et al.46을 참조하십시오.
- 얼음처럼 차가운 75mM의 Tris-HCl, pH 7.2, 0.6M의 KCl, 6mM의 EDTA, 1mM의 EGTA 및 0.1%(w/v) 데옥시콜레이트(DOC)를 사용하여 SRV 현탁액을 2mg protein/mL로 희석합니다. SRV 현탁액을 얼음 위에서 10분 동안 배양하고 부드럽게 교반합니다. SRV 현탁액(100,000 × g)을 4°C에서 60분 동안 원심분리기합니다.
참고: DOC는 한 방울씩 추가해야 합니다. - 25mM의 Tris-HCl, pH 7.5, 0.3M의 KCl, 45% 글리세롤(v/v) 및 2mM EDTA <3mL에 펠릿을 현탁시킵니다. 현탁액을 균질화하고 최종 부피 10mL로 조정합니다. 단백질 농도를 측정한 다음 6.5mg/mL로 조정합니다. 5 mg/mL 및 0.85 (w/w) Zwittergent 3-14에서 아졸렉틴을 추가합니다.
참고: Azolectin과 Zwittergent 3-14는 한 방울씩 추가해야 합니다. - 현탁액과 원심분리기(100,000 × g)를 4°C에서 60분 동안 균질화합니다. 상층액을 모아 2mM의 EGTA(pH 7.2)로 1:2로 희석합니다.
- 10mM의 Tris-HCl, pH 7.0, 1mM의 EDTA, 0.1% 데옥시콜레이트 및 1mg/mL의 아졸렉틴에서 불연속 트레할로스 농도 구배(45, 40, 35 및 30% w/v)에 현탁액을 부드럽게 붓습니다. 원심분리기(100,000 × g) 4°C에서 14시간 동안. 튜브 바닥에 형성된 투명하고 약간 황색을 띤 펠릿을 수집합니다.
참고: 트레할로스 구배의 형성과 샘플 현탁액의 추가는 모두 적절한 기술을 사용하여 수행해야 합니다. 하룻밤 동안 원심분리하는 것이 좋습니다. - Tris-HCl 25mM, pH 7.5, M KCl 0.3, 글리세롤 45% 및 EDTA 2mM의 소량(<1.5mL)으로 펠릿을 부드럽게 현탁시킵니다. 단백질 농도를 측정합니다. 단백질 농도를 2mg/mL로 희석하여 조정합니다. 현탁액의 부분 표본(~50 μL)을 취하고 정제된 SERCA를 사용할 때까지 -72 °C에서 보관합니다.
- 정제된 SERCA72 및 다양한 정제 단계에서 채취한 단백질 샘플의 SDS-PAGE를 수행합니다. Coomassie Blue72로 젤을 염색합니다.
참고: 현탁액에 글리세롤 대신 0.6M 트레할로스를 사용하면 저장 및 동결-해동 주기 동안 효소의 안정성이 증가합니다28,29.
3. ATPase 활성에 대한 분석 (타이밍, 1 - 2 시간)
- 효소 결합 분석법: ATPase-Piruvate kinase-lactate dehydrogenase
- 반응 혼합물 (1 mL)을 준비합니다 : 50 mM MOPS (pH 7.0), 1 mM EGTA, 80 mM KCl, 5 mM MgCl2, 3 mM CaCl2, 5 mM 포스포에놀피루베이트, 250 μM NADH. 다양한 ATP 농도(0.001 - 0.25mM)를 포함합니다.
- 와류(vortexing)에 의한 반응 분석을 조심스럽게 균질화합니다. 반응 분석을 37°C에서 10분 동안 배양하고 0.9U L-젖산 탈수소효소(LDH) 및 1.5U 피루브산 키나아제(PK)를 추가합니다.
- SERCA 10μg을 첨가하여 ATPase 반응을 시작합니다.
- NADH의 형성을 결정합니다. 온도 조절 셀 홀더가 있는 분광 광도계를 사용하여 시간이 지남에 따라 340nm의 파장(λ)에서 600초 동안 초당 흡광도 강도의 변화를 따릅니다.
참고: 흡광도 측정을 위한 시간 간격은 다를 수 있지만 ATPase 반응의 초기 직선을 그리려면 가능한 한 많은 데이터 포인트를 기록하십시오.
- ATP 가수분해 속도 측정
참고: 절차에 대한 자세한 내용은 Rivera-Morán et al.46을 참조하십시오.- 형성된 각 곡선에서 선형 부분의 기울기 값(Δabs/min)을 계산합니다. NADH(λ = 6,220 M−1·cm−1)의 몰 흡광 계수(ε)를 사용하여 ATPase 활성(μmoles ATP 가수분해/min.mg prot)을 측정합니다.
- 동역학 매개변수 K, m 및 Vmax 결정
- 데이터 분석 및 그래프 작성 소프트웨어를 사용하여 속도 대 ATP 농도를 플롯합니다.
- 비선형 회귀를 통해 데이터를 Michaelis-Menten 방정식(식 1)46에 맞춥니다.
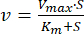
여기서 v 는 속도, Vmax 는 최대 속도, [S]는 ATP 농도, Km 은 Michaelis-Menten 상수입니다. - 반복을 통해 Km 및 Vmax 를 결정합니다.
참고: DOC를 포함하면 ATPase 속도 반응이 증가할 수 있습니다. 최적을 찾기 위해 DOC의 다양한 농도를 테스트하는 것이 좋습니다.
4. FITC(Fluorescein Isothiocyanate)를 사용한 SERCA의 라벨링 분석(타이밍, 30 - 45분, FITC 라벨링에만 해당)
참고: 절차에 대한 자세한 내용은 54,75,76을 참조하십시오.
- 1mM의 FITC를 포함하는 50mM의 라벨링 버퍼(100mM KCl, 5mM MgCl2 및 30mM Tris-HCl, pH 8.9)의 50μL(최종 부피)에 SERCA(20μg)를 현탁합니다. 소용돌이에 의하여 섞으십시오.
- 어두운 곳과 실온에서 서로 다른 시간(15분, 10분, 7분, 5분, 2분)에 샘플을 배양합니다.
- ATP(5mM)를 포함하는 1부피의 얼음-저온 정지 완충액(480mM의 자당 및 48mM의 MOPS, pH 7.0)을 첨가하여 라벨링 반응을 중지합니다. 어둠 속에서 5분 동안 얼음 위에서 배양합니다.
- FITC 라벨이 붙은 SERCA에 SDS-PAGE72를 적용한다. 투명 젤을 자외선(λ = 302nm)에 노출시킵니다. 결과를 사진으로 문서화합니다.
- 쿠마시 블루로 젤을 염색합니다. 결과를 사진으로 문서화합니다.
참고: 어두운 방에서 FITC 라벨링을 수행하면 형광 신호의 품질이 향상될 수 있습니다.
5. 원형 이색성(CD) 스펙트럼(타이밍, 30분)
참고: 절차에 대한 자세한 내용은11,77을 참조하십시오.
- 25°C에서 400μL의 10mM 인산염 완충액(pH 7.0)에 SERCA(3μM)를 현탁합니다.
- 0.1cm 경로 길이 셀에 샘플을 로드합니다. 원자외선 스펙트럼 λ 범위를 190-260nm 사이로 설정합니다. 내부 해상도와 대역폭을 1nm로 설정합니다.
- 25°C에서 50nm/min으로 CD 신호를 기록합니다.
결과
다양한 정제 단계에서 SERCA의 SDS-PAGE(그림 1). Coomassie 파란색 염색 겔은 정제 프로토콜이 진행됨에 따라 SERCA 단백질 밴드(겉보기 분자량 >100kDa)가 풍부해지는 것을 보여줍니다. SERCA에 해당하는 단백질 밴드는 트레할로스 농도 구배에서 원심분리 후 >90%의 순도를 보여줍니다. ATP 가수분해 속도 대 ATP 농도의 플롯(그림 2A). ?...
토론
대부분의 분자와 이온은 세포막을 자유롭게 통과 할 수 없으며, 예를 들어 양성자 (H +)는 미토콘드리아83 , 84와 같은 다양한 유기체 및 소기관의 원형질막에서 막 수송체를 필요로합니다. 세포막은 선택적이며 세포막을 가로지르는 분자와 이온이 다양하기 때문에 세포에서는 (a) ABC 수송체, (b) 이온 채널, (c) 막 결합 ATPas...
공개
저자는 자신은 경쟁하는 재정적 이해관계가 없다고 선언한다.
감사의 말
저자는 비디오 편집에 Edmundo Mata-Morales, VM, SRV 정제에 Valentín de la Cruz-Torres, SERCA 정제 및 분석에 Miguel A. Rivera-Moran, 비디오 제작에 Juan C. Gonzalez-Castro, Franco E. Juarez, Alejandra Nevarez, Nicolas Rocha-Vizuet 및 Jocelin I. Ramírez-Alonso의 도움을 인정합니다. 이 연구를 수행하기 위한 자금, 보조금 또는 기타 지원을 받지 못했습니다.
자료
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
참고문헌
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유