需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
从兔肌肉中纯化肌内质网 Ca2+-ATP 酶
摘要
该方案描述了一种改进的 SERCA 纯化方法,其中包括最终离心步骤中的二糖海藻糖。这种碳水化合物在恶劣的条件下稳定蛋白质。纯化的 SERCA 具有催化活性且纯度高,使其适用于结构和功能研究。
摘要
一些 P 型 ATP 酶,如肌瘤/内质网 Ca2+-ATP 酶 (SERCA),本质上是不稳定的膜蛋白,在纯化过程中需要特定的物理化学条件才能获得具有高纯度和结构质量的催化活性形式。二糖海藻糖是一种相容的溶质,在酵母细胞质中以高浓度合成和积累,以稳定细胞膜和蛋白质。在纯化质膜 H+-ATP 酶的方案中使用海藻糖作为添加剂可产生高质量的制剂,其六聚体结构通过生化分析方法显示。因此,海藻糖可用作纯化膜蛋白 (P-ATP 酶) 的稳定添加剂。该方案描述了通过在海藻糖浓度梯度上离心 SERCA 来修改 SERCA 纯化的经典方案。这种碳水化合物的加入导致 SERCA 以高纯度的催化活性形式纯化,重要的是,以稳定形式纯化。纯化的 SERCA 的部分生化表征 (SDS-PAGE、酶动力学、FITC 标记、圆二色谱光谱) 表明该酶适用于功能和结构研究。建议在 P 型 ATP 酶和其他不稳定的膜(和胞质)蛋白的纯化方案中使用海藻糖。
引言
膜蛋白/酶是细胞的重要生物成分,因为它们在各种过程中起着关键作用 1,2,3。一些功能可能包括离子和分子进出细胞/内部隔室(主动/被动)、细胞间识别、细胞间结合、锚定/附着,以及在正常和恶劣的物理化学条件(高盐、低水、高温、耐药、 等)3 因此,膜蛋白和/或酶的三维 (3D) 结构的测定对于基础和应用研究都变得非常重要 4,7,6。重要的是,膜蛋白/酶已被广泛用作药物发现的靶标(无论是天然的还是设计性的)7,8,9。也就是说,膜蛋白在健康中具有内在的重要性 9,10,11。
膜蛋白/酶的疏水特性是实验实验室最具技术挑战性的物理化学性质 12,13,14,在处理寡聚和/或高度不稳定的整合膜蛋白时更是如此15,16。对于功能实验测定和结构研究,非常需要以尽可能高的质量分离适量的膜蛋白/酶17。膜蛋白本质上具有疏水性,非常难以纯化,去污剂的选择通常是要考虑的最重要的问题之一 17,18,19,20。在这方面,用于分离膜蛋白的实验室方法通常会对蛋白质的 3D 结构排列造成一定程度的损害21。其中一些方法包括使用 (a) 超声处理(高频/高能超声波),(b) 蛋白质溶解去污剂(刺激性、中等或温和)22,(c) 柱色谱和高速超速离心中相对较高的压力23,(d) 沉淀分子,(e) 消化酶等21。所有这些过程都可能导致或成为纯化过程中蛋白质不稳定的主要原因21。在这方面,一些方案似乎对给定的膜蛋白效果相对较好。然而,当使用新的现代检测或方法时,需要更高质量的膜蛋白制备以获得满意的结果,优化总是受欢迎的24。优化步骤可能包括但不限于改进构建体设计、寻找膜蛋白表达的最佳条件、建立更好的处理条件(即 pH 值、温度等)、找到最佳相容的去污剂、调整纯化步骤(如超声处理时间、离心速度)、通过添加稳定剂重新配制缓冲溶液等25,26,27.因此,纯化方法中任何导致纯化膜蛋白/酶的质量(纯度)和活性提高的变化都很重要。
在 P 型 ATP 酶家族中,酵母质膜 H+-ATP 酶似乎是最不稳定的成员之一28,29。H+-ATP 酶在脱水(冷冻干燥)、热休克等过程中失去其 ATP 水解活性28,29。在从酵母乳酸克雷伯菌中分离质膜 H+-ATP 酶 30,31 中测试了海藻糖作为蛋白质稳定剂的使用;所得 H+-ATP 酶制备物纯度高且具有催化活性。重要的是,这使得酶的寡聚状态可以通过生化方法解决,显示它是六聚体,后来通过冷冻电子显微镜证实 31,32,33,34,35。因此,在纯化过程中,在相对恶劣的条件下,膜蛋白的精细三维 (3D) 排列似乎可能会丢失36。在热介导的失活过程中观察到 H+-ATP 酶的双相失活动力学29。在酵母细胞中,与许多其他生物体一样,二糖海藻糖在环境应激条件下以高浓度积累 37,38,39。海藻糖通过稳定蛋白质(膜包埋和胞质)和细胞膜来维持膜完整性和转运功能40,41。海藻糖的稳定机制已被多个小组和我们的实验室 42,43,44,45 广泛研究。H+-ATP 酶和其他酶的实验表明,海藻糖是单糖和双糖中最有效的蛋白质稳定剂28,29。这导致其被纳入 H+-ATP 酶纯化方案30。最近,海藻糖还被用于从兔快肌中纯化肌浆网 Ca2+-ATP 酶 (SERCA),在蛋白质纯度和活性方面具有良好的效果46。因此,海藻糖似乎是纯化 P 型 ATP 酶以及可能的其他膜和胞质蛋白的良好且合适的添加剂。
对于 P-ATP 酶,结构胞质结构域的存在是一个实验优势,特别是对于底物/配体相互作用研究47;已在高纯度重组 N 结构域 47,48,49 中研究了 ATP 结合,从而消除了纯化全膜酶 47,48,50 等的技术考虑 41。不幸的是,一些功能(催化/能量转换)和结构(亚基排列和与其他蛋白质的相互作用)研究仍然需要整个 P-ATP 酶52,53。在这方面,几个研究小组已经实现了 SERCA 的纯化 54,55,56,57,58。然而,仍然可以实施改进46,例如,增加纯化的 ATP 酶59 的完整性,避免蛋白质复合物的蛋白质变性/破坏25,增加溶解而不使膜蛋白变性(即,避免形成大分子聚集体)60,从而与测定和其他下游分析方法具有更好的兼容性61此外,随着新的实验策略、添加剂、酶抑制剂等出现在科学文献61、62、63、64、65、66 中,它们有时需要用整个 P-ATP 酶进行测试。这项工作描述了纯化 SERCA 的方案以及使用海藻糖作为添加剂来稳定蛋白质结构和 ATP 酶活性;即,除了提高酶(结构)质量外,海藻糖还有助于防止酶分离和储存过程中酶结构和活性的损失,这有助于节省生物材料,从而减少酶纯化的次数。
研究方案
所有动物程序均按照国际和当地 (NORMA Oficial Mexicana NOM-062-ZOO-1999) 实验实验室处理动物指南进行 51,67,68。肌肉组织来自当地动物处理单位 (INE/CITES/DGVS-ZOO-E0055-SLP-98)46 的野生型 Oryctolagus cuniculus。具有实验动物管理专业知识的兽医进行了初步的肌肉解剖和处理。本研究中使用的试剂和设备的详细信息列在材料表中。
1. 从快肌兔肌肉中分离肌浆网(计时,7 - 9 小时)
注:有关该程序的详细信息,请参阅 Champeil 等人 69。
- 从野生型 Oryctolagus cuniculus 获得快肌组织(每只动物双后腿 ~70 克)。
注意:需要事先目视识别要解剖的肌肉70,71。 - 在搅拌机中浸渍快肌,将其悬浮在三个体积的 100 mM KCl 中,开启 1 分钟,冰冷休息 1 分钟,重复 3 次72。
- 通过在 4 °C 下离心 (1500 × g) 5 分钟去除组织碎片;如有必要,进行第二次离心 (4200 × g)。
- 收集上清液并使用组织研磨机(以低速冲程 10 次,即 500-1,500 rpm)进行匀浆72,73。将匀浆(10,000 × g)在 4 °C 下离心 15 分钟。
注意:均质化时必须小心,因为使用相对较高的速度可能会损坏系统和作员。 - 将上清液(含有肌浆网)(33,000 × g)在 4 °C 下离心 120 分钟。
- 将沉淀悬浮在 40 mL 的 0.5 M 蔗糖中,并在 4 °C 下离心 (12,000 × g) 15 分钟。稀释上清液(0.6 M KCl 和 0.15 M 蔗糖)并将悬浮液(34,000 × g)在 4 °C 下离心 165 分钟。
- 将含有肌浆网囊泡 (SRV) 片的沉淀悬浮在 0.3 M 蔗糖、0.1 M KCl 和 5 mM Tris-HCl(pH 7.0)中。使用 Lowry 测定74 和人血清白蛋白作为蛋白质标准品测定蛋白质浓度。
- 将 SRV (30-32 mg/mL) 分装在 1 mL 体积中。将 SRV 样品储存在 -72 °C 以供进一步处理。
注:蛋白质产量为每只动物 100-110 mg SRV。
2. SERCA 纯化(计时,18 - 20 小时)
注:有关该程序的详细信息,请参阅 Rivera-Morán 等人 46。
- 使用冰冷的 75 mM Tris-HCl(pH 7.2)、0.6 M KCl、6 mM EDTA、1 mM EGTA 和 0.1% (w/v) 脱氧胆酸盐 (DOC) 将 SRV 悬浮液稀释至 2 mg 蛋白质/mL。将 SRVs 悬浮液在冰上孵育 10 分钟并轻轻搅拌。将 SRVs 悬浮液(100,000 × g)在 4 °C 下离心 60 分钟。
注意:DOC 应逐滴添加。 - 将沉淀悬浮在 <3 mL 的 25 mM Tris-HCl、pH 7.5、0.3 M KCl、45% 甘油 (v/v) 和 2 mM EDTA 中。均质化悬浮液并调整至最终体积为 10 mL。测定蛋白质浓度,然后将其调节至 6.5 mg/mL。添加 5 mg/mL 和 0.85 (w/w) Zwittergent 3-14 的偶氮凝集素。
注意:偶氮凝集素和两性晶 3-14 应逐滴添加。 - 将悬浮液均质化,并在 4 °C 下离心 (100,000 × g) 60 分钟。收集上清液并用 2 mM EGTA (pH 7.2) 以 1:2 稀释。
- 将悬浮液轻轻倒入 10 mM Tris-HCl(pH 7.0)、1 mM EDTA、0.1% 脱氧胆酸盐和 1 mg/mL 偶氮凝集素中不连续的海藻糖浓度梯度(45、40、35 和 30% w/v)上。在 4 °C 下离心 (100,000 × g) 14 小时。收集在管底部形成的透明、略带黄色的颗粒。
注:海藻糖梯度的形成和样品悬浮液的添加都必须使用适当的技术进行。建议过夜离心。 - 将沉淀轻轻悬浮在小体积 (<1.5 mL) 的 25 mM Tris-HCl、pH 7.5、0.3 M KCl、45% 甘油和 2 mM EDTA 中。测定蛋白质浓度。通过稀释至 2 mg/mL 来调节蛋白质浓度。取等分试样 (~50 μL) 的悬浮液,并将纯化的 SERCA 储存在 -72 °C 直至使用。
- 对纯化的 SERCA72 和在不同纯化步骤中采集的蛋白质样品进行 SDS-PAGE。用考马斯蓝72 对凝胶进行染色。
注:在悬浮缓冲液中使用 0.6 M 海藻糖代替甘油可增加酶在储存和冻融循环期间的稳定性28,29。
3. ATP 酶活性测定(时间,1 - 2 小时)
- 酶偶联试验:ATP 酶-吡鲁酸激酶-乳酸脱氢酶
- 制备反应混合物 (1 mL):50 mM MOPS (pH 7.0)、1 mM EGTA、80 mM KCl、5 mM MgCl2、3 mM CaCl2、5 mM 磷酸烯醇式丙酮酸、250 μM NADH。包括可变的 ATP 浓度(0.001 至 0.25 mM)。
- 通过涡旋仔细匀浆反应测定。将反应测定在 37 ◦C 下孵育 10 分钟。加入 0.9 U L-乳酸脱氢酶 (LDH) 和 1.5 U 丙酮酸激酶 (PK)。
- 通过添加 10 μg SERCA 引发 ATP 酶反应。
- 确定 NADH 的形成。使用带有恒温比色池支架的分光光度计,在 340 nm 的波长 (λ) 下每秒跟踪吸光度强度的变化 600 秒。
注:吸光度测量的时间间隔可能会有所不同,但请尝试记录尽可能多的数据点,以绘制 ATP 酶反应的初始直线。
- 确定 ATP 水解速率
注:有关该程序的详细信息,请参阅 Rivera-Morán 等人 46。- 计算每条形成的曲线中线性部分的斜率值 (Δabs/min)。使用 NADH 的摩尔消光系数 (ε) (λ = 6,220 M-1·cm-1) 来确定 ATP 酶活性(μmoles ATP 水解/min.mg prot)。
- 确定动力学参数 Km 和 Vmax
- 使用数据分析和绘图软件绘制速度数据与 ATP 浓度的关系图。
- 通过非线性回归到 Michaelis-Menten 方程(方程 1)46 来拟合数据:
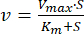
其中 v 是速度, Vmax 是最大速度,[S] 是 ATP 浓度, Km 是 Michaelis-Menten 常数。 - 通过迭代确定 Km 和 Vmax 。
注意:包含 DOC 可能会增加 ATP 酶速率响应;建议测试不同浓度的 DOC 以找到最佳浓度。
4. 用异硫氰酸荧光素 (FITC) 对 SERCA 进行标记测定(定时,30 - 45 分钟,仅用于 FITC 标记)
注意:有关该过程的详细信息,请参阅 54,75,76。
- 将 SERCA (20 μg) 悬浮在 50 μL(终体积)含有 1 mM FITC 的标记缓冲液(100 mM KCl、5 mM MgCl2 和 30 mM Tris-HCl,pH 8.9)中。涡旋混合。
- 在不同时间(15 分钟、10 分钟、7 分钟、5 分钟和 2 分钟)在黑暗和室温下孵育样品。
- 通过加入 1 体积的冰冷终止缓冲液(480 mM 蔗糖和 48 mM MOPS,pH 7.0),终止标记反应,含有 ATP (5 mM)。在黑暗中在冰上孵育 5 分钟。
- 将 FITC 标记的 SERCA 置于 SDS-PAGE72 中。将透明凝胶暴露在紫外光 (λ = 302 nm) 下。照片记录结果。
- 用考马斯蓝对凝胶进行染色。照片记录结果。
注意:在暗室中进行 FITC 标记可能会提高荧光信号的质量。
5. 圆二色性 (CD) 光谱(定时,30 分钟)
注意:有关该过程的详细信息,请参阅11,77。
- 在 25 °C 下将 SERCA (3 μM) 悬浮在 400 μL 的 10 mM 磷酸盐缓冲液 (pH 7.0) 中。
- 将样品加载到 0.1 cm 光程的比色皿中。将远紫外光谱 λ 范围设置在 190-260 nm 之间。将内部分辨率和带宽设置为 1 nm。
- 在 25 °C 下以 50 nm/min 的速度记录 CD 信号。
结果
不同纯化阶段 SERCA 的 SDS-PAGE(图 1)。考马斯蓝染色凝胶显示随着纯化方案的进行,SERCA 蛋白条带(表观分子量 >100 kDa))的富集。对应于 SERCA 的蛋白质条带在海藻糖浓度梯度上离心后显示纯度为 >90%。ATP 水解速率与 ATP 浓度的关系图(图 2A)。观察到纯化的 SERCA 的 Michaelis-Menten ATP 酶动力学(双曲线模式)。
讨论
大多数分子和离子不能自由穿过细胞膜,例如,质子 (H+) 需要各种生物体和细胞器(如线粒体)质膜中的膜转运蛋白83,84。细胞膜是选择性的,穿过细胞膜的分子和离子是多种多样的,因此可以在细胞中找到几种类型的膜蛋白,例如 (a) ABC 转运蛋白,(b) 离子通道,(c) 膜结合的 ATP 酶,(d) SLC 转运蛋白,以?...
披露声明
提交人声明他没有相互竞争的经济利益。
致谢
作者感谢 Edmundo Mata-Morales 在视频编辑、VM、Valentín de la Cruz-Torres 在纯化 SRV 方面的帮助,Miguel A. Rivera-Moran 在 SERCA 纯化和分析方面的帮助,以及 Juan C. Gonzalez-Castro、Franco E. Juarez、Alejandra Nevarez、Nicolas Rocha-Vizuet 和 Jocelin I. Ramírez-Alonso 在视频制作中的帮助。没有收到任何资金、赠款或其他支持来进行这项研究。
材料
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
参考文献
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。