Method Article
Regolazione chemiogenetica nelle cellule precursori derivate da cellule staminali riprogrammate nel trattamento delle malattie neurodegenerative
In questo articolo
Riepilogo
Qui, descriviamo un protocollo per l'ingegnerizzazione di cellule staminali riprogrammate chimicamente per ottenere una modulazione neuronale precisa differenziando queste cellule in cellule precursori dopaminergiche, trapiantandole in modelli murini della malattia di Parkinson e valutando i risultati comportamentali ed elettrofisiologici per confermare il successo dell'integrazione e l'efficacia funzionale delle cellule trapiantate.
Abstract
L'integrazione di recettori progettati attivati esclusivamente da farmaci progettati (DREADD) con terapie basate su cellule staminali presenta una strategia avanzata per una modulazione neuronale precisa. Qui, abbiamo utilizzato cellule staminali umane riprogrammate ingegnerizzate con CRISPR che esprimono recettori DREADD eccitatori (hM3Dq) o inibitori (hM4Di) per valutare l'integrazione funzionale e la modulazione dei precursori dopaminergici trapiantati in un modello murino di malattia di Parkinson (PD). I passaggi chiave includevano la generazione di linee di cellule staminali che esprimono DREADD senza fusione, la loro differenziazione in precursori dopaminergici del mesencefalo e il trapianto di queste cellule nello striato di topi con lesione da 6-idrossidopamina (6-OHDA). Abbiamo condotto valutazioni comportamentali e registrazioni elettrofisiologiche per analizzare gli effetti delle cellule trapiantate. I test comportamentali, come il test del cilindro, hanno dimostrato una modulazione significativa della funzione motoria dopo la somministrazione di clozapina-N-ossido (CNO). In particolare, l'attivazione di hM4Di ha ridotto il movimento controlaterale degli arti anteriori, mentre l'attivazione di hM3Dq è stata associata a un miglioramento del comportamento motorio. Le registrazioni elettrofisiologiche hanno rivelato risposte sinaptiche distinte. L'attivazione di hM4Di ha aumentato gli intervalli tra gli eventi e diminuito le ampiezze di picco delle correnti postsinaptiche eccitatorie spontanee (sEPSC), mentre l'attivazione di hM3Dq ha diminuito gli intervalli tra gli eventi e ha aumentato le ampiezze di picco, riflettendo un aumento della segnalazione eccitatoria. In sintesi, l'integrazione di valutazioni comportamentali ed elettrofisiologiche convalida l'esatta incorporazione funzionale di cellule staminali ingegnerizzate chimicamente riprogrammate nei circuiti neurali dell'ospite.
Introduzione
I recettori progettati attivati esclusivamente da farmaci progettati (DREADD) sono recettori accoppiati a proteine G ingegnerizzate che possono essere attivati selettivamente da ligandi sintetici altrimenti inerti1. L'approccio chemiogenetico è diventato uno strumento essenziale nelle neuroscienze, consentendo ai ricercatori di studiare la connettività dei circuiti neurali con elevata precisione e migliorando la nostra comprensione delle funzioni cellulari sia in vivo che in vitro attraverso l'attivazione selettiva o l'inibizione di specifiche regioni cerebrali o tipi di cellule 2,3.
La terapia basata sulle cellule staminali rappresenta una strategia promettente per il trattamento delle malattie neurodegenerative. L'efficacia delle cellule del trapianto si basa sulla corretta integrazione, sopravvivenza e contributo funzionale ai tessuti ospiti. Un'attività cellulare incontrollata può portare a conseguenze negative, tra cui la tumorigenesi4; che richiede un controllo preciso di queste cellule dopo il trapianto. L'utilizzo della tecnologia DREADD nelle cellule staminali umane riprogrammate e nei neuroni derivati fornisce un mezzo per controllare con precisione l'attività neuronale attraverso la somministrazione del farmaco di progettazione CNO 2,5. Nel contesto della malattia di Parkinson (PD), che è caratterizzata dalla perdita di neuroni dopaminergici, la manipolazione dell'attività dei neuroni dopaminergici derivati da cellule staminali è fondamentale per studiare i loro input sinaptici e i modelli di proiezione nei modelli di roditori 6,7,8,9,10. L'incorporazione di recettori eccitatori hM3Dq e inibitori hM4Di in questi modelli consente una modulazione precisa dell'attività neuronale11,12.
La combinazione di valutazioni comportamentali animali e registrazioni elettrofisiologiche consente una valutazione completa degli effetti della modulazione chemiogenetica sulle cellule trapiantate in vivo13. Le valutazioni comportamentali, tra cui la rotazione indotta dall'apomorfina, il test del cilindro e il test della rotarod, valutano la coordinazione motoria e forniscono informazioni sui cambiamenti nella funzione motoria associati ai modelli sperimentali di PD14. Le tecniche elettrofisiologiche, come le registrazioni patch-clamp, consentono il monitoraggio in tempo reale delle risposte sinaptiche e dei potenziali d'azione, fornendo una visione completa del modo in cui le cellule trapiantate si integrano nelle reti neurali esistenti15. Combinando valutazioni comportamentali con valutazioni elettrofisiologiche, possiamo studiare come la modulazione chemiogenetica influenzi l'integrazione e la funzionalità di queste cellule all'interno dei circuiti neurali dell'ospite16. I risultati preliminari suggeriscono che la somministrazione di CNO modula efficacemente l'attività neuronale nelle cellule trapiantate, con conseguente miglioramento dei risultati funzionali nei modelli animali.
In questo protocollo, le cellule staminali umane riprogrammate sono state ingegnerizzate per esprimere i recettori hM3Dq o hM4Di utilizzando la tecnologia CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats). Dopo aver differenziato le cellule staminali riprogrammate modificate in cellule precursori dopaminergiche del mesencefalo, queste cellule sono state trapiantate in modelli murini di PD per valutare la loro integrazione e regolazione funzionale all'interno dei circuiti neurali dell'ospite utilizzando valutazioni comportamentali e registrazioni elettrofisiologiche.
Protocollo
Tutti gli esperimenti sugli animali sono stati eseguiti in conformità con le linee guida stabilite dall'Associazione di Pechino per la scienza degli animali da laboratorio e dal National Institutes of Health for the Care and Use of Laboratory Animals. Le cellule mononucleate del sangue periferico umano (PBMC) sono state ottenute da un donatore sano con consenso informato scritto, come descritto in uno studio precedente17.
1. Costruzione di linee cellulari basate su staminali DREADD non fuse
- Costruisci il plasmide donatore DREADD.
NOTA: I siti di digestione devono essere progettati secondo la sequenza vettoriale. I frammenti devono essere sintetizzati e amplificati con sovrapposizioni di 20-30 bp corrispondenti ai bracci del frammento per migliorare l'efficienza dell'assemblaggio.- Progettare e sintetizzare il frammento T2A-ZsGreen (Frammento I).
- Amplificare il frammento hM3Dq/hM4Di-T2A dal plasmide originale utilizzando la PCR (Frammento II). Impostare la reazione PCR (50 μL di volume totale) in modo che contenga 1 tampone PCR, 200 μM di ciascuna miscela dNTP, 0,5 μM di primer diretti e inversi, 10-50 ng di DNA plasmidico e 1,25 U di DNA polimerasi ad alta fedeltà. Eseguire il termociclo nelle seguenti condizioni: denaturazione iniziale a 98 °C per 2,75 minuti per denaturare completamente il modello; 32 cicli di amplificazione composti da 98 °C per 15 s (denaturazione), 60 °C per 30 s (ricottura di primer) e 72 °C per 60 s (estensione); allungamento finale a 72 °C per 7 min.
- Digerire il plasmide vettore con l'enzima appropriato per ottenere il vettore linearizzato. Purificare tutti i frammenti mediante elettroforesi su gel di agarosio e quantificarli utilizzando uno spettrofotometro.
NOTA: Qui, il plasmide originale è stato digerito con MluI e SalI.
- Mescolare delicatamente 50-100 ng del vettore linearizzato, del Frammento I e del Frammento II in un rapporto molare 1:3:3, insieme a 2x Cloning Mix. Incubare la miscela a 55 °C per 1 ora per facilitare la ricombinazione omologa attraverso il Gibson Assembly.
- Inserire l'RNA guida che ha come bersaglio il sito AAVS1 nel sito BbsI del vettore pX458.
NOTA: Eseguire la verifica della sequenza su tutti i plasmidi costruiti utilizzando il sequenziamento Sanger. - Mantenere le cellule staminali riprogrammate nel terreno, puntando a una densità cellulare di 2-5 × 106 cellule/mL.
NOTA: Le cellule staminali riprogrammate qui utilizzate sono state indotte da PBMC umane come descritto in uno studio precedente17. Il monitoraggio dello stato delle celle è essenziale, poiché lo stato delle cellule influisce in modo significativo sull'efficienza dell'elettroporazione. Vedere la tabella dei materiali per un confronto dei supporti. - Miscelare 2-5 × 106 cellule, 2 μg di ciascun plasmide donatore (hM4Di-T2A-ZsGreen o hM3Dq-T2A-ZsGreen) e 2 μg di plasmide gRNA in 100 μL di tampone per elettroporazione e trasferire in una cuvetta per elettroporazione.
NOTA: Assicurarsi che la miscela sia ben amalgamata e priva di bolle prima di procedere all'elettroporazione. Ottimizza i parametri di elettroporazione in base al tipo di cella per massimizzare l'efficienza della trasfezione, poiché dispositivi diversi possono richiedere regolazioni specifiche. - Impostare il Nucleofector sul programma B16 e avviare l'elettroporazione per facilitare la trasfezione efficiente delle iNSC target.
- Aggiungere 1 mL di poli-D-lisina (PDL) da 50 μg/mL a ciascun pozzetto di una piastra a 6 pozzetti e incubare a temperatura ambiente per almeno 2 ore. Rimuovere tutto il PDL dai pozzetti e aggiungere 1,5 mL di 5 μg/mL di laminina in ciascun pozzetto e incubare a 37 °C per 2-4 ore. Utilizzare immediatamente la piastra rivestita o conservare a 2-8 °C per 1 settimana.
- Trasferire le celle elettroporate in piastre a sei pozzetti rivestite con PDL e laminina. Distribuire circa 500.000 cellule per pozzetto in un volume di 2 mL di terreno di coltura per pozzetto.
- Dopo 72 ore di elettroporazione, utilizzare un citometro a flusso per selezionare le cellule fluorescenti positive.
- Risospendere le cellule dissociate in DPBS e filtrarle attraverso un filtro cellulare da 70 μm.
- Configurare lo smistatore con il canale FITC per il rilevamento ZsGreen. Includere le iNSC non trattate come controlli negativi per definire la fluorescenza basale.
- Calibrare lo strumento utilizzando ugelli da 100 μm a una pressione della guaina di 20 psi. Implementare una modalità di ordinamento della purezza a quattro vie con conferma a cella singola.
- Piastra singole cellule ZsGreen+ in piastre da 96 pozzetti prerivestite con PDL e laminina e aggiungi 200 μl di terreno di coltura per pozzetto per supportare la crescita iniziale. Conservare in terreno di coltura per 7-14 giorni
NOTA: Monitorare quotidianamente i pozzetti per segni di crescita e fluorescenza che indichino un'integrazione riuscita ed etichettare i pozzetti positivi. - Isolare singoli cloni che presentano fluorescenza utilizzando un microscopio a fluorescenza invertito dotato di un set di filtri GFP. Cloni dello schermo con ingrandimento 10x. Contrassegnare i pozzetti contenenti colonie monoclonali con segnale ZsGreen sostenuto e uniforme. Acquisisci immagini in campo chiaro e a fluorescenza per documentare la morfologia e l'intensità della fluorescenza.
NOTA: Prima dell'imaging, sostituire il terreno fresco per ridurre la fluorescenza di fondo. Escludere le colonie con morfologia irregolare o autofluorescenza. - Espandi le celle positive su piastre rivestite a 48 pozzetti. Quindi diviso in due parti, una per la coltura e l'altra per la genotipizzazione.
- Estrarre il DNA genomico da cloni candidati. Preparare le reazioni di PCR con due coppie di primer: una coppia per confermare l'inserimento del gene di interesse, l'altra per distinguere i cloni omozigoti ed eterozigoti. Eseguire il termociclo nelle seguenti condizioni: denaturazione iniziale a 95 °C per 5 min; 38 cicli a 95 °C per 30 s, 60 °C per 30 s e 72 °C per 45 s; allungamento finale a 72 °C per 7 min. Risolvere i prodotti PCR su un gel di agarosio al 2%.
NOTA: Vedere la tabella dei materiali per i primer. Confermare l'inserimento del gene di interesse mediante una singola banda di 650 bp (primer 1). Identificare i cloni eterozigoti per la presenza di una singola banda di 630 bp e i cloni omozigoti per l'assenza di questa banda (primer 2). - Espandi le celle positive su piastre rivestite a sei pozzetti.
- Digerire le cellule e lavarle in soluzione salina tamponata con fosfato (PBS) mediante centrifugazione a 250 × g per 3 minuti. Congelare le cellule in terreno di crioconservazione con il 10% di DMSO a una densità di 2,5 × 106 cellule per fiala.
2. Trapiantate cellule precursori derivate da cellule staminali DREADD in topi modello PD
- Somministrare un'iniezione intraperitoneale di desipramina a una concentrazione di 5 mg/mL (10 mg/kg) a topi immunodeficienti 30 minuti prima dell'intervento chirurgico. Anestetizzare i topi con isoflurano al 2%.
- Condurre iniezioni stereotassiche unilaterali di 3 μL di 6-idrossidopamina (6-OHDA, 5 μg/μL). Individua il corpo striato destro utilizzando le seguenti coordinate: anteroposteriore = 0,5 mm, laterale = 2,1 mm e verticale = -3,2 mm.
NOTA: Il 6-OHDA deve essere sciolto in soluzione salina contenente lo 0,2% di acido ascorbico. Garantire un targeting preciso per ridurre al minimo la variabilità della gravità della lesione.
ATTENZIONE: Il 6-OHDA è citotossico e produce specie reattive dell'ossigeno che inducono stress cellulare. Maneggiare con cura. - Somministrare un'iniezione intraperitoneale di apomorfina (1 mg/kg, 0,5 mg/mL disciolto in soluzione fisiologica) per convalidare le lesioni 4 settimane dopo l'intervento. Selezionare topi lesionati che mostrano più di 100 rotazioni controlaterali entro un periodo di osservazione di 30 minuti per gli esperimenti successivi.
NOTA: L'apomorfina è un agonista del recettore della dopamina, che attiva direttamente i recettori postsinaptici supersensibili nello striato denervato, sbilanciando l'attività bilaterale del circuito nigrostriatale. La rotazione indotta dall'apomorfina serve a valutare la gravità delle lesioni dopaminergiche unilaterali da 6-OHDA. - Cellule di semina e coltura in piastre rivestite a sei pozzetti (150.000 cellule per pozzetto). Incubare nel terreno di differenziazione di Fase 1 per 10 giorni, rinfrescando il terreno ogni 2 giorni. L'11° giorno, passare al terreno di differenziazione di Fase 2 e continuare l'incubazione fino al 13° giorno, cambiando il terreno ogni giorno.
NOTA: Vedere la tabella dei materiali per un confronto del mezzo di differenziazione. - Raccogli le cellule nei giorni 10 e 13 di differenziazione, quindi mescolale con un rapporto di 1:7. Sospendere la miscela in un tampone di trapianto a una concentrazione di 100.000 cellule/μL.
NOTA: Vedere la Tabella dei materiali per un confronto del tampone di trapianto. - Somministrare un'iniezione stereotassica di un volume totale di 4 μl della sospensione cellulare preparata in un topo PD.
- Eseguire il test del cilindro in vari momenti dopo il trapianto di cellule. Posizionare un bicchiere di vetro (diametro: 20 cm, altezza: 30 cm) su una superficie piana e non riflettente. Posiziona una telecamera dall'alto sopra il cilindro per catturare l'intero diametro.
NOTA: Racchiudere il cilindro con tende nere per eliminare gli stimoli visivi esterni. - Posiziona ogni mouse al centro del cilindro e avvia la registrazione video simultanea (3 min/sessione). Dopo la sessione, rimetti il mouse nella sua gabbia di casa.
NOTA: Acclimatare i topi alla sala prove per 30 minuti in condizioni di luce. Pulisci il cilindro con etanolo al 70% tra una prova e l'altra per eliminare i segnali olfattivi. - Valutare la capacità di movimento degli arti superiori dei topi per 3 minuti. Calcolare il numero di contatti con la parete effettuati dall'arto anteriore danneggiato rispetto al numero totale di contatti effettuati da entrambi gli arti anteriori.
- Somministrare soluzione fisiologica o CNO alla dose di 1,2 mg/kg tramite iniezione intraperitoneale. Condurre il test del cilindro per valutare la modulazione comportamentale nel modello murino PD dopo la somministrazione di CNO
NOTA: Attendere un periodo di recupero di circa 40 minuti prima di procedere con la valutazione.
3. Profilazione elettrofisiologica in vivo di cellule modulate chemiogeneticamente
- Anestetizzare i topi mediante un'iniezione intraperitoneale di 250 mg/kg di pentobarbital. Estrarre il cervello e metterlo immediatamente nel liquido cerebrospinale artificiale a base di saccarosio ghiacciato (s-ACSF) per mantenere l'integrità cellulare. Usando un vibratomo, taglia il cervello in sezioni di 300-400 μm di spessore.
NOTA: Lavora rapidamente per mantenere il cervello a basse temperature per preservare la funzione cellulare. Vedere la tabella dei materiali per l's-ACSF utilizzato nella preparazione di sezioni cerebrali fresche. - Trasferire i campioni in una camera riempita con ACSF bilanciata con il 95% di O2 e il 5% di CO2 a 34 °C, assicurandosi che questa temperatura sia mantenuta per tutta la durata dell'esperimento.
NOTA: Vedere la tabella dei materiali per ACSF. - Caricare le pipette di vetro con soluzione intracellulare ghiacciata, con resistenza dell'elettrodo compresa tra 7 e 10 MΩ.
NOTA: Vedere la Tabella dei materiali per la soluzione intracellulare. Le pipette di vetro sono ricavate da capillari di vetro mediante un estrattore per micropipette18. Prima di iniziare gli esperimenti, verificare la presenza di eventuali perdite nelle pipette di vetro, assicurandosi che le punte siano sufficientemente affilate da penetrare nella membrana cellulare. Preparare rapidamente le pipette per ridurre al minimo le fluttuazioni di temperatura nella soluzione intracellulare. - Montare le fette in una camera di registrazione sommersa superfusa (2 mL/min) con ACSF ossigenato a 34 °C. Posizionamento di pipette mediante micromanipolatori motorizzati al microscopio a contrasto interferenzo-interferenza differenziale a infrarossi. Stabilire la configurazione dell'intera cellula applicando un'aspirazione delicata (resistenza > 1 GΩ) ai neuroni rattoppati all'interno delle regioni di innesto. Clamp celle a -70 mV utilizzando un amplificatore patch-clamp; acquisire sEPSC di base per 8 minuti a una frequenza di campionamento di 10 kHz (filtrata a 2 kHz) e monitorare continuamente la resistenza di accesso (target: <20 MΩ; scartare se >25 MΩ o fluttuante >10%).
NOTA: Utilizzare una pressione appropriata quando si applica l'aspirazione per evitare spostamenti o rotture indesiderate delle cellule e regolare attentamente la posizione della pipetta per ottenere una buona tenuta. Se la qualità della sigillatura è scarsa, gettare la pipetta e riprovare. - Aggiungere 50 μM di CNO alla camera di registrazione subito dopo aver completato la misurazione di base e continuare a registrare i dati per altri 16 minuti osservando le variazioni di frequenza o ampiezza di sEPSC.
- Eseguire una procedura di lavaggio sostituendo la soluzione di CNO con ACSF fresco per rimuovere efficacemente il CNO dalla camera di registrazione. Continuare a registrare i dati per il resto dell'esperimento per valutare l'eventuale recupero dell'attività sinaptica.
Risultati
La Figura 1 mostra le fasi chiave di questo approccio metodologico per cellule staminali umane riprogrammate ingegnerizzate che esprimono recettori DREADD eccitatori (hM3Dq) o inibitori (hM4Di) per valutare l'integrazione funzionale e la modulazione di precursori dopaminergici derivati in un modello murino di malattia di Parkinson (PD). La Figura 2 delinea la strategia di knock-in genico mediata da CRISPR/Cas9 per l'introduzione di costrutti non di fusione di hM4Di-T2A-ZsGreen e hM3Dq-T2A-ZsGreen in cellule staminali riprogrammate e convalidate mediante genotipizzazione.
La Figura 3 caratterizza gli effetti funzionali e sinaptici della modulazione chemiogenetica nelle cellule trapiantate. Abbiamo condotto valutazioni comportamentali e registrazioni elettrofisiologiche per analizzare gli effetti delle cellule DREADD innestate. La Figura 3A mostra l'analisi statistica dei risultati del movimento controlaterale degli arti anteriori in un test del cilindro tra i gruppi di controllo, hM4Di- e hM3Dq in condizioni di washout salino, CNO e postCNO. La somministrazione di CNO ha ridotto l'impegno controlaterale degli arti anteriori negli animali trapiantati con hM4Di, ma ha migliorato le prestazioni motorie nel gruppo hM3Dq rispetto ai controlli salini. La Figura 3B-D mostra l'analisi elettrofisiologica delle correnti postsinaptiche eccitatorie spontanee (sEPSC). Il gruppo trapiantato con hM4Di ha mostrato intervalli intereventi prolungati e ampiezze di picco ridotte, indicative di silenziamento sinaptico, mentre le cellule trapiantate con hM3Dq hanno dimostrato intervalli più brevi e ampiezze aumentate, coerenti con una maggiore neurotrasmissione eccitatoria. Figura 3E,F caratterizzano le cellule trapiantate in vivo mediante immunofluorescenza.

Figura 1: Regolazione chemiogenetica in cellule precursori derivate da cellule staminali riprogrammate per il trattamento di malattie neurodegenerative in un modello murino. Un diagramma di flusso che illustra l'approccio metodologico per stabilire un modello murino di PD e valutare i risultati comportamentali attraverso la regolazione chemiogenetica nelle cellule precursori derivate da cellule staminali riprogrammate. Fase 1: Costruzione di linee di cellule staminali DREADD non di fusione utilizzando la tecnologia CRISPR. Fase 2: Definizione del modello murino PD, seguita dal trapianto di cellule precursori derivate da cellule staminali DREADD nel cervello. Fase 3: Valutazione degli esiti comportamentali ed elettrofisiologici per verificare il successo dell'integrazione e della modulazione funzionale delle cellule trapiantate. Abbreviazioni: CRISPR = breve ripetizione palindromotica raggruppata regolarmente intervallata; DREADD = recettori progettati attivati esclusivamente da farmaci sintetici; PD = morbo di Parkinson; 6-OHDA = 6-idrossidopammina; CNO = clozapina N-ossido. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Ingegnerizzazione mediata da CRISPR/Cas9 di cellule staminali che esprimono DREADD senza fusione. (A) Diagramma di flusso che illustra la strategia di knock-in genico mediata da CRISPR/Cas9 per l'integrazione di costrutti hM4Di-T2A-ZsGreen o hM3Dq-T2A-ZsGreen in cellule staminali riprogrammate. (B) Il plasmide donatore di non fusione hM4Di-T2A-ZsGreen o hM3Dq-T2A-ZsGreen è stato modificato attraverso una strategia di clonazione a più fasi. La rappresentazione dell'allele bersaglio illustra il knock-in genico di hM4Di-T2A-ZsGreen o hM3Dq-T2A-ZsGreen non di fusione nelle cellule staminali riprogrammate. Le immagini a fluorescenza confermano che le cellule ingegnerizzate mostrano una fluorescenza verde omogenea, indicando un'espressione transgenica riuscita. Barra della scala = 200 μm. (C) I risultati della genotipizzazione confermano il successo dell'integrazione dei costrutti DREADD nel genoma delle linee di cellule staminali bersaglio. Questa cifra è stata modificata da Wang et al.19. Abbreviazioni: gRNA = RNA guida; FACS = smistamento cellulare attivato dalla fluorescenza; DREADD = recettori progettati attivati esclusivamente da droghe sintetiche. Clicca qui per visualizzare una versione più grande di questa figura.
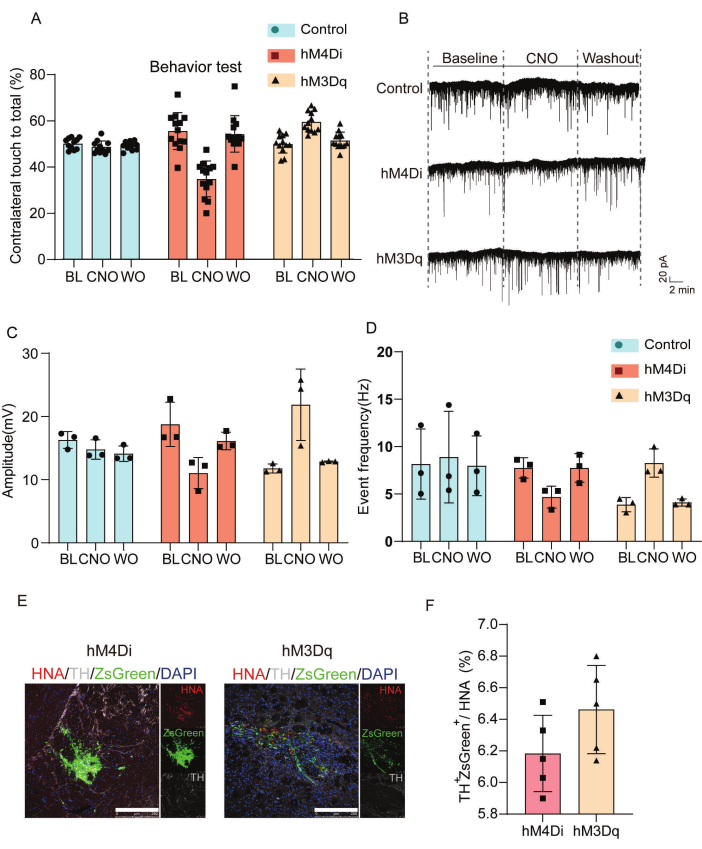
Figura 3: Valutazione degli esiti comportamentali ed elettrofisiologici per la conferma dell'integrazione e della modulazione funzionale delle cellule trapiantate. (A) Valutazione della modulazione comportamentale nel modello murino PD dopo somministrazione di CNO; analisi statistica dettagliata che confronta i risultati del movimento controlaterale degli arti anteriori nel test del cilindro tra i gruppi hM4Di, hM3Dq e di controllo al basale e 8 settimane dopo il trapianto, con valutazioni condotte dopo il trattamento con soluzione salina e CNO, nonché dopo il washout postCNO. (B) Registrazioni di patch-clamp su cellule intere che rivelano sEPSC dagli innesti dei gruppi di controllo, hM4Di o hM3Dq analizzati al basale e durante il trattamento con CNO (50 μM), insieme alle fasi di washout del CNO. (C) Analisi statistica delle misure di ampiezza di picco per le sEPSC attraverso il basale, il CNO e il washout del CNO. (D) Statistiche sulla frequenza degli eventi delle sEPSC registrate dagli innesti dei gruppi di controllo, hM4Di e hM3Dq. (E)Caratterizzazione di cellule trapiantate in vivo mediante immunofluorescenza. Barre della scala = 250 μm. (F) Analisi quantitativa dell'espressione della tirosina idrossilasi e di ZsGreen nei gruppi hM4Di e hM3Dq, rispetto all'espressione dell'antigene nucleare umano. Questa cifra è stata modificata da Wang et al.19. Abbreviazioni: BL = linea di base; CNO = trattamento con clozapina N-ossido (CNO, 50 μM); WO = CNO di lavaggio; PD = morbo di Parkinson; sEPSC = correnti postsinaptiche eccitatorie spontanee; TH = tirosina idrossilasi; HNA = antigene nucleare umano. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Questo protocollo ha utilizzato la tecnologia CRISPR per ingegnerizzare cellule staminali umane riprogrammate per esprimere recettori eccitatori hM3Dq e inibitori hM4Di. La modulazione dell'attività neuronale da parte di CNO è stata valutata attraverso il trapianto di cellule in modelli murini di malattia di Parkinson, accompagnata da valutazioni comportamentali e registrazioni elettrofisiologiche.
Il primo passo critico nella generazione di costrutti DREADD stabilmente espressi e non fusi in cellule staminali riprogrammate chemiogeneticamente coinvolge la cassetta bicistronica T2A-ZsGreen. Tuttavia, i plasmidi che esprimono proteine di fusione, come hM3Dq-mCherry e hM4Di-mCherry7, sono inefficaci per questa linea di cellule staminali neurali indotte. Per aggirare questo problema, abbiamo ingegnerizzato un plasmide donatore tramite l'assemblaggio di Gibson, come descritto nelle fasi del protocollo 1.1-1.2.
Stabilire un modello di PD somministrando unilateralmente un'iniezione stereotassica di 6-OHDA nel corpo striato di topi immunodeficienti è fondamentale per una precisa modulazione in vivo. Durante l'intero periodo di studio, compreso il trapianto di cellule e i trattamenti farmacologici, alcuni animali possono sperimentare una mortalità inaspettata. Pertanto, la dimensione iniziale del campione dovrebbe essere attentamente pianificata per garantire numeri sufficienti per i gruppi sperimentali15,17.
Diversi test comportamentali relativi alla funzione motoria potrebbero essere utilizzati per valutare le risposte comportamentali degli animali alla modulazione DREADD, tra cui il test di rotazione indotta da apomorfina, il test del cilindro e il test del rotarod. Qui abbiamo impiegato il test del cilindro, che misura quantitativamente il contatto dell'arto anteriore con le pareti di un cilindro20. Valutando le alterazioni nell'uso degli arti superiori derivanti da danni neurali, il test del cilindro fornisce preziose informazioni su come il CNO può migliorare la funzione motoria.
L'integrazione funzionale e la connettività sinaptica delle cellule del donatore riprogrammate chemiogeneticamente all'interno dei circuiti neurali dell'ospite sono determinanti critici dell'efficacia terapeutica per le malattie neurodegenerative come la PD16. Le registrazioni di patch-clamp su cellule intere condotte su fette di cervello dopo il trapianto forniscono informazioni su come queste cellule ingegnerizzate interagiscono con i circuiti neuronali nativi. I neuroni spinosi medi (MSN) nello striato sono bersagli a valle della segnalazione dopaminergica.
Utilizzando gli MSN di registrazione patch-clamp, possiamo valutare con precisione come le cellule modificate con DREADD si integrano nei circuiti neurali e influenzano la loro funzione21. La composizione del liquido cerebrospinale artificiale (ACSF) per fasi procedurali distinte è fondamentale per il patch-clamp. Durante la preparazione della fetta, si raccomanda vivamente un liquido cerebrospinale artificiale a base di saccarosio (s-ACSF). Le proprietà iperosmotiche del saccarosio mitigano il gonfiore neuronale durante il sezionamento, mantenendo i gradienti ionici essenziali per la vitalità cellulare. La fabbricazione degli elettrodi e il controllo qualità determinano direttamente la stabilità della registrazione. Le pipette in vetro borosilicato tirate a resistenze del puntale di 7-10 MΩ rappresentano un prerequisito non negoziabile per registrazioni sEPSC prolungate. Pipette non ottimali con resistenze inferiori a 5 MΩ mostrano uno scambio diffusionale accelerato tra la soluzione della pipetta e il citoplasma, che porta a un esaurimento progressivo della corrente entro 3-5 minuti.
Tuttavia, questo studio dimostra la modulazione chemiogenetica del comportamento motorio e dell'attività sinaptica nei topi PD attraverso iNSC-DAP ingegnerizzati, il sostanziale attrito procedurale correlato agli interventi dual-stereotassici e la somministrazione cronica di CNO sottolineano la necessità di raffinati paradigmi di xenotrapianto in modelli immunocompromessi, in particolare per quanto riguarda la standardizzazione chirurgica. Questa strategia, che combina l'ingegneria del recettore DREADD mediata da CRISPR/Cas9 con l'interrogazione a livello di circuito tramite saggi comportamentali (test del cilindro) ed elettrofisiologia patch-clamp su cellule intere, fornisce un modello di neuromodulazione specifico per il sottotipo di neurone e offre un toolkit versatile per far progredire le terapie cellulari di precisione per i disturbi neurodegenerativi.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalla Beijing Natural Science Foundation (7242068), dalla National Natural Science Foundation of China (82171250), dal Beijing Municipal Health Commission Fund (PXM2020_026283_000005) e dal Progetto per lo sviluppo tecnologico degli istituti di ricerca medica affiliati a Pechino (11000023T000002036310).
Materiali
| Name | Company | Catalog Number | Comments |
| 2 x Rapid Taq Master Mix | Vazyme | P222-01 | used for genotyping analysis |
| 2×Seamless Cloning Mix | Biomed Gene Tech. | CL117-01 | used for plasmid construction |
| 6-OHDA | Sigma-Aldrich | H4381 | used for establishing PD mice model |
| AAVS1-Pur-CAG-EGFP | Addgene | 80945 | used as control |
| AAVS1-Pur-CAG-hM4Di-mCherry | Addgene | 80947 | original plasmid for construction of hM4Di-T2A-ZsGreen |
| AAVS1-Pur-CAG-hM3Dq-mCherry | Addgene | 80948 | original plasmid for construction of hM3Dq-T2A-ZsGreen |
| AAVS1-CAG-hM4Di-T2A-ZsGreen | N/A | N/A | Constructed donor plasmid based on #80947 |
| AAVS1-CAG-hM3Dq-T2A-ZsGreen | N/A | N/A | Constructed donor plasmid based on #80948 |
| Accutase | Invitrogen | A11105-01 | Used for digesting cells |
| AMAXA Nucleofector | Lonza | AAD-1001S | |
| Apomorphine | Sigma-Aldrich | A4393 | |
| Artificial cerebrospinal fluid (ACSF) | N/A | N/A | 125 mM NaCl, 2.5 mM KCl, 2 mM CaCl2, 1.25 mM NaH2PO4, 1 mM MgSO4, 25 mM glucose, and 26 mM NaHCO3 |
| Ascorbic acid | Sigma-Aldrich | 1043003 | |
| B-27 Supplement | Gibco | 17504044 | |
| BDNF | Peprotech | 450-02 | |
| cAMP | Sigma-Aldrich | D0627 | |
| CHIR99021 | Yeasen | 53003ES10 | |
| Clozapine-N-oxide | Enzo | BML-NS105 | |
| DAPT | Sigma-Aldrich | D5942 | |
| Desipramine | Sigma-Aldrich | D3900 | |
| D-glucose | Sigma-Aldrich | G5767 | |
| DMEM/F12 | Gibco | 11330-032 | |
| DMEM/F12 | Gibco | 11320-033 | |
| DMSO | Sigma-Aldrich | D2650 | |
| FGF8 | Peprotech | 100-25 | |
| GDNF | Peprotech | 450-10 | |
| GlutaMax | Gibco | 35050-061 | |
| Hank’s Balanced Salt Solution (HBSS) | Gibco | 14175095 | |
| Human leukemia inhibitory factor (hrLIF) | Millipore | LIF1010 | |
| Iced intracellular fluid | N/A | N/A | 130 mM K-gluconate, 16 mM KCl, 0.2 mM EGTA, 2 mM MgCl2, 10 mM HEPES, 4 mM Na2-ATP, 0.4 mM Na3-GTP, 0.3% of neurobiotin |
| KnockOut Serum Replacement | Gibco | 10828028 | |
| Laminin | Roche | 11243217001 | |
| Micropipette Puller | Sutter Instrument Company | P-1000 | |
| N-2 Supplement | Thermo Fisher | 17502048 | |
| Neurobasal-A Medium | Gibco | 10888-022 | |
| Non-essential amino acids (NEAA) | Gibco | 11140-050 | |
| PBS | Gibco | 10010023 | |
| pCLAMP 11 software suite | Molecular Devices | N/A | Patch-clamp electrophysiology data acquisition and analysis software |
| Phase 1 differentiation medium | N/A | N/A | 96% DMEM/F12 (Gibco, 11320-033), 1% B-27 Supplement, 1% N-2 Supplement, 1% NEAA, 1% GlutaMax, 1 µM SAG1, and 100 ng/mL FGF8 |
| Phase 2 differentiation medium | N/A | N/A | 96% DMEM/F12 (Gibco, 11320-033), 1% B-27 Supplement, 1% N-2 Supplement, 1% NEAA, and 1% GlutaMax, 10 ng/mL BDNF, 10 ng/mL GDNF, 1 ng/mL TGF-βIII, 10 µM DAPT, 0.2 mM ascorbic acid, and 0.5 mM cAMP. |
| Poly-D-lysine hydrobromide (PDL) | Sigma-Aldrich | P7886 | |
| Primers for genotyping | N/A | N/A | Insertion Foward: TCTTCACTCGCTGGGTTCCCTT; Insertion Reverse: CCTGTGGGAGGAAGAGAAGAGGT; Homozygosity Foward:CGTCTCCCTGGCTTTAGCCA; Homozygosity Reverse: GATCCTCTCTGGCTCCATCG |
| pX458 | Addgene | 152199 | |
| SAG1 | Enzo | ALX-270-426-M01 | |
| SB431542 | Yeasen | 53004ES50 | |
| sucrose-based artificial cerebrospinal fluid (s-ACSF) | 234 mM sucrose, 2.5 mM KCl, 26 mM NaHCO3, 1.25 mM NaH2PO4, 11 mM Dglucose, 0.5 mM CaCl2, and 10 mM MgSO4 | ||
| Stem cell culture media | N/A | N/A | 48% DMEM/F12 (Gibco, 11330-032) and 48% Neurobasal, with the addition of 1% B27, 1% N2, 1% NEAA, 1% GlutaMax, 10 ng/mL hrLIF, 2 µM SB431542, and 3 µM CHIR99021 |
| TGF-βIII | Peprotech | 100-36E | |
| Transplantation buffer | N/A | N/A | HBSS buffer with 5 g/L D-glucose, 100 ng/mL BDNF, 100 ng/mL GDNF, and 0.2 mM ascorbic Acid |
| Vibratome | Leica | VT1000 S | |
| Whole-cell patch-clamp | Molecular Devices | MultiClamp700B |
Riferimenti
- Thiel, G. Designer receptors exclusively activated by designer drugs. Humana Press (2015).
- Roth, B. L. Dreadds for neuroscientists. Neuron. 89 (4), 683-694 (2016).
- Gomez, J. L. et al. Chemogenetics revealed: DREADD occupancy and activation via converted clozapine. Science. 357 (6350), 503-507 (2017).
- Chehelgerdi, M. et al. Exploring the promising potential of induced pluripotent stem cells in cancer research and therapy. Mol Cancer. 22 (1), 189 (2023).
- Song, J., Patel, R. V., Sharif, M., Ashokan, A., Michaelides, M. Chemogenetics as a neuromodulatory approach to treating neuropsychiatric diseases and disorders. Mol Ther. 30 (3), 990-1005 (2022).
- Parmar, M., Grealish, S., Henchcliffe, C. The future of stem cell therapies for Parkinson disease. Nat Rev Neurosci. 21 (2), 103-115 (2020).
- Dell'anno, M. T. et al. Remote control of induced dopaminergic neurons in Parkinsonian rats. J Clin Invest. 124 (7), 3215-3229 (2014).
- Chen, Y. et al. Chemical control of grafted human PSC-derived neurons in a mouse model of Parkinson's disease. Cell Stem Cell. 18 (6), 817-826 (2016).
- Bjorklund, A. Parmar, M. Dopamine cell therapy: From cell replacement to circuitry repair. J Parkinsons Dis. 11 (S2), S159-S165 (2021).
- Alcacer, C. et al. Chemogenetic stimulation of striatal projection neurons modulates responses to Parkinson's disease therapy. J Clin Invest. 127 (2), 720-734 (2017).
- Alexander, G. M. et al. Remote control of neuronal activity in transgenic mice expressing evolved G protein-coupled receptors. Neuron. 63 (1), 27-39 (2009).
- Stachniak, T. J., Ghosh, A., Sternson, S. M. Chemogenetic synaptic silencing of neural circuits localizes a hypothalamus midbrain pathway for feeding behavior. Neuron. 82 (4), 797-808 (2014).
- Kang, H. J., Minamimoto, T., Wess, J., Roth, B. L. Chemogenetics for cell-type-specific modulation of signalling and neuronal activity. Nat Rev Methods Primers. 3 (1), 93 (2023).
- Miyanishi, K. et al. Behavioral tests predicting striatal dopamine level in a rat hemi-Parkinson's disease model. Neurochem Int. 122, 38-46 (2019).
- Zheng, X. et al. Human IPSC-derived midbrain organoids functionally integrate into striatum circuits and restore motor function in a mouse model of Parkinson's disease. Theranostics. 13 (8), 2673-2692 (2023).
- Xiong, M. et al. Human stem cell-derived neurons repair circuits and restore neural function. Cell Stem Cell. 28 (1), 112-126.e116 (2021).
- Yuan, Y. et al. Dopaminergic precursors differentiated from human blood-derived induced neural stem cells improve symptoms of a mouse Parkinson's disease model. Theranostics. 8 (17), 4679 (2018).
- Zhu, M. et al. Preparation of acute spinal cord slices for whole-cell patch-clamp recording in substantia gelatinosa neurons. J Vis Exp. (143) (2019).
- Wang, X., Han, D., Zheng, T., Ma, J., Chen, Z. Modulation of human induced neural stem cell-derived dopaminergic neurons by DREADD reveals therapeutic effects on a mouse model of Parkinson's disease. Stem Cell Res Ther. 15 (1), 297 (2024).
- Iancu, R., Mohapel, P., Brundin, P., Paul, G. Behavioral characterization of a unilateral 6-OHDA-lesion model of Parkinson's disease in mice. Behav Brain Res. 162 (1), 1-10 (2005).
- Surmeier, D. J. et al. The role of dopamine in modulating the structure and function of striatal circuits. Prog Brain Res. 183, 148-167 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon