Method Article
Criblage de composés de médecine traditionnelle chinoise pour inhiber l’activité d’UCHL3 sur la base de la technologie des sondes enzymatiques d’amarrage moléculaire et de désubiquitination
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
L’expérience utilisée ici montre une méthode d’amarrage moléculaire combinée à des technologies de sonde pour prédire et valider l’interaction entre de petites molécules de la médecine traditionnelle chinoise et des cibles protéiques.
Résumé
Les enzymes désubiquitinantes (DUB) jouent un rôle central dans la modulation de l’homéostasie de l’ubiquitination, UCHL3 étant un archétype de la cystéine DUB intimement impliquée dans une myriade de processus physiologiques et pathologiques. Par conséquent, le développement de petites molécules inhibitrices ciblant l’ubiquitine C-terminale Hydrolase L3 (UCHL3) est d’une grande importance. Ce protocole vise à établir un processus de criblage virtuel et de validation in vitro de petites molécules inhibitrices de la cystéine DUB représentée par UCHL3. Tout d’abord, les inhibiteurs potentiels d’UCHL3 sont virtuellement criblés à l’aide de la technologie d’amarrage moléculaire, et l’interaction entre les médicaments et les cibles protéiques est visualisée. Par la suite, l’efficacité du médicament testé, Danshensu, est vérifiée par des essais d’inhibition de l’activité in vitro . L’ubiquitine-7-amino-4-méthylcoumarine (Ub-AMC) et l’hémagglutinine-ubiquitine-vinyl sulfone (HA-Ub-VS) sont utilisées comme sondes pour les tests d’activité in vitro , car elles peuvent se lier de manière compétitive à DUB avec des inhibiteurs de petites molécules pour évaluer l’activité d’UCHL3. Les résultats indiquent que Danshensu a une bonne affinité de liaison avec UCHL3 dans l’amarrage moléculaire, et qu’il peut inhiber de manière compétitive l’activité d’UCHL3 avec HA-Ub-VS. Ces résultats constituent des références importantes pour la recherche et le développement de médicaments thérapeutiques ciblant UCHL3.
Introduction
L’ubiquitination est une modification post-traductionnelle des protéines, un processus par lequel les enzymes activatrices de l’ubiquitine E1, les enzymes conjuguées à l’ubiquitine E2 et les ubiquitines ligases E3 attachent l’ubiquitine à la protéine cible, et l’ensemble du processus d’ubiquitination peut être inversé par les enzymes de désubiquitination (DUBs)1,2,3,4. En raison de leur rôle physiologique et pathologique important, les DUB sont considérés comme des cibles importantes pour la découverte de médicaments 5,6.
Plus de 100 DUBs ont été identifiés chez l’homme 7,8. Ils fonctionnent généralement comme une isopeptidase responsable du clivage de la liaison isopeptide entre l’extrémité C de l’ubiquitine et un résidu de lysine dans un substrat ou une autre molécule d’ubiquitine 9,10. À l’heure actuelle, ils sont principalement classés en sept grandes familles, à savoir : les peptidases spécifiques de l’ubiquitine (USP), les protéases tumorales ovariennes (UTO), les protéases N-terminales + domaines Jab1/Mov34/Mpr1 Pad 1 (JAMMO), le motif interagissant avec les nouvelles protéases de la famille DUB contenant de l’ubiquitine (MINDY), les hydroxylases C-terminales de l’ubiquitine (UCH), les protéases à domaine Machado-Josephin (MJD) et l’ubiquitine peptidase 1 contenant du doigt de zinc (ZUP1)9. En plus des JUM, qui appartiennent à la famille des métalloprotéases de zinc11, les autres DUBs sont des protéases cystéine caractérisées par une triade catalytique composée de cystéine catalytique, d’histidine et d’un troisième résidu acide12,13. Cette spécificité ouvre la voie au développement d’inhibiteurs à petites molécules ciblant le site actif de l’enzyme ou les poches allostériques voisines.
Dans le domaine de la recherche sur les enzymes désubiquitinantes, il existe un défi important dans la caractérisation de leur activité14,15. La caractérisation à l’aide de sondes actives constitue une approche cruciale pour l’étude des inhibiteurs de DUB16. En effectuant des tests compétitifs avec des sondes et des inhibiteurs basés sur l’activité dans des lysats cellulaires ou des protéines recombinantes, l’activité des DUB peut être caractérisée, facilitant ainsi le développement de petites molécules inhibitrices ciblant ces enzymes. Ub-AMC est une sonde précoce utilisée pour détecter l’activité DUB, qui a un groupe fluorescent attaché à l’extrémité C-terminale de l’ubiquitine17,18. Lorsque les DUBs exercent leur activité catalytique, l’AMC est libérée en grande quantité et l’intensité fluorescente de sa lumière détectée est augmentée en conséquence. Cette sonde a été largement utilisée dans le criblage à haut débit des inhibiteurs de DUB19,20. La sonde HA-Ub-VS est également utilisée pour mesurer l’activitéDUB 21. Il a un groupe sulfone de vinyle à l’extrémité C-terminale de l’ubiquitine, ce qui en fait un substrat suicidaire pour les DUBs. Après la séparation par électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide (SDS-PAGE), les DUB actifs peuvent être détectés à l’aide du western blot 22,23,24.
La médecine traditionnelle chinoise (MTC) utilise les plantes médicinales depuis plus de 2000 ans. Le développement de nouveaux médicaments à partir de produits naturels revêt une grande importance médicale, avec un accent clé sur l’identification des ingrédients actifs et l’élucidation de leurs mécanismes25. Salvia miltiorrhiza Bge est une plante largement utilisée dans le traitement d’une variété de maladies, notamment le cancer, les maladies cardiovasculaires, hépatiques etneurologiques. Actuellement, il contient de petites molécules connues telles que la tanshinone27, le Danshensu28, l’acide tanshinique29, etc. Ces composés présentent diverses activités biologiques telles que des effets anti-thrombotiques, antioxydants et antitumoraux, ce qui les rend très précieux pour la recherche26. Des études récentes ont identifié Danshensu comme un inhibiteur covalent de la protéase de type 3-chymotrypsine (3CLpro) du SRAS-CoV-230. Il a été démontré qu’il forme une liaison covalente avec le résidu du site actif C145 de 3CLpro, indiquant la présence d’inhibiteurs potentiels de la protéase à petites molécules dans Salvia miltiorrhiza Bge.
L’ubiquitine C-Terminal Hydrolase L3 (UCHL3) appartient aux protéases cystéine de la famille des DUBs UCH. Il s’appuie sur des résidus conservés comme la cystéine95, l’histidine169 et l’acide aspartique184 pour catalyser efficacement ses fonctions31,32. Il joue un rôle crucial dans de multiples voies moléculaires, notamment le cycle cellulaire, la recombinaison homologue et la réparation des cassures de l’ADN liées aux protéines33. De plus, il est régulé à la hausse dans divers cancers tels que les cancers de l’ovaire, de la prostate, du pancréas, colorectal et du poumon non à petites cellules34. Sur la base de ces études, UCHL3 semble être une cible prometteuse pour le traitement des maladies. Plusieurs petites molécules inhibitrices d’UCHL3 ont été identifiées et progressent vers une utilisation clinique35,36.
Dans cette étude, l’amarrage moléculaire a été effectué pour étudier les interactions entre les petites molécules de Salvia miltiorrhiza Bge et UCHL3. Par la suite, une expérience in vitro utilisant des sondes spécifiques de DUB Ub-AMC et HA-Ub-VS a identifié Danshensu comme une petite molécule inhibitrice d’UCHL3. L’amarrage moléculaire a également prédit des sites de liaison potentiels pour Danshensu, suggérant son mécanisme d’action.
Protocole
1. Téléchargement des structures des petites molécules de Salvia miltiorrhiza Bge et de l’UCHL3
- Téléchargez le fichier des petites molécules.
- Ouvrez la base de données TCMSP (https://old.tcmsp-e.com), entrez danshen (nom de l’herbe), puis appuyez sur rechercher et cliquez sur le Radix Salviae dans la liste des résultats.
- Cliquez sur télécharger un par un des éléments et enregistrez la structure 2D au format .mol2.
- Téléchargez la fiche protéine.
- Ouvrez la base de données PDB (https://www.rcsb.org/), entrez UCHL3, puis appuyez sur rechercher. Cliquez sur Homo sapiens, puis appuyez sur Rechercher dans les raffinements.
- Sélectionnez la structure 1XD3 avec co-cristallisation petites molécules-protéines, puis cliquez sur télécharger les fichiers et sélectionnez le format PDB .
2. Amarrage moléculaire
- Enregistrez le fichier.
- Créez un nouveau dossier nommé danshen_UCHL3 docking sur le bureau et enregistrez les composés des structures Salvia miltiorrhiza Bge et UCHL3 dans ce dossier. Nommez le dossier en anglais ; Sinon, le fichier ne pourra pas être importé.
- Définissez le chemin d’accès.
- Ouvrez le logiciel Maestro, cliquez sur Fichier, sélectionnez Modifier le répertoire de travail, cliquez sur Bureau, double-cliquez pour sélectionner le dossier d’ancrage danshen_CUHL3, puis cliquez sur Choisir les options.
- Traitement de petites molécules
- Importez des structures de petites molécules.
- Cliquez sur les options Fichier et Importer des structures. Cliquez sur Bureau, double-cliquez sur le dossier d’ancrage, puis cliquez sur le fichier de structure Salvia miltiorrhiza Bge. Ensuite, cliquez sur Ouvrir pour importer toutes les petites molécules.
- Préparez la petite molécule.
- Cliquez sur Tâches et sélectionnez l’option LigPrep . Dans la fenêtre LigPrep affichée, cliquez sur la table du projet dans l’option Utiliser la structure de , cochez l’option Déterminer les chiralités à partir de la structure 3D sous Calcul et laissez tous les autres paramètres du logiciel par défaut.
- Remplacez le nom du travail par danshen_ligprep1, puis cliquez sur Exécuter pour exécuter le traitement des petites molécules.
- Importez des structures de petites molécules.
- Traitement de la structure des protéines
- Importez la structure protéique.
- Cliquez sur les options Fichier et Importer des structures , cliquez sur Bureau et double-cliquez sur le dossier d’ancrage danshen_CUHL3.
- Cliquez sur le fichier de structure de la protéine UCHL3 , puis cliquez sur Ouvrir pour importer le fichier de structure de la protéine.
- Préparez les protéines.
- Sélectionnez les deux liaisons covalentes reliant UCHL3 et la petite molécule, et supprimez-les à l’aide du bouton Supprimer. Ensuite, sélectionnez les deux résidus protéiques qui sont incomplets après la suppression. Cliquez sur le bouton Compiler, sélectionnez Autres modifications, puis pour Gly75, cliquez sur le bouton C, pour Cys95, choisissez Mutate Residue et CYS.
- Cliquez sur Tâches et sélectionnez l’option Flux de préparation des protéines . Laissez tous les autres paramètres du logiciel par défaut. Remplacez le nom de la tâche par UCHL3_protein pre, puis cliquez sur Exécuter pour effectuer le traitement des protéines.
- Configurez la boîte d’accueil.
- Cliquez sur Tâches, choisissez Génération de grille de récepteurs, puis sélectionnez Choisir pour identifier la molécule de ligand . Sélectionnez la petite molécule dans le workpac e, et une boîte d’ancrage rose centrée sur les coordonnées de la petite molécule apparaîtra.
- Conservez les paramètres par défaut, nommez le travail comme 1XD3_danshen_glide_grid, puis cliquez sur Exécuter.
REMARQUE : Dans 1XD3, la petite molécule forme une liaison covalente avec la protéine. Il est nécessaire de supprimer cette liaison covalente pour séparer la petite molécule de la protéine. Sinon, pendant le processus de configuration de la grille réceptrice, la petite molécule ne peut pas être sélectionnée.
- Importez la structure protéique.
- Effectuer l’amarrage moléculaire.
- Cliquez sur Tâches et sélectionnez l’option Ligand Docking . Sélectionnez la grille réceptrice, cliquez sur À partir d’un fichier, puis cliquez sur Parcourir. Sélectionnez 1XD3_danshen_glide_grid.zip fichier, puis cliquez sur Ouvrir.
- Cliquez sur Utiliser les ligands de fichiers en tant que fichiers, puis cliquez sur Parcourir. Cliquez sur danshen_ligprep1 fichier, sélectionnez danshen_ligprep1-out. maegz file, puis cliquez sur Ouvrir.
- Cliquez sur Paramètres et sélectionnez l’option Précision en tant que SP , remplacez le nom de la tâche par danshen_UCHL3_ _SP glidedock, puis cliquez sur Exécuter.
- Affichez les résultats de la station d’accueil.
- Cliquez sur les options Fichier et Importer des structures . Cliquez sur Bureau et double-cliquez sur le dossier d’ancrage danshen_CUHL3.
- Double-cliquez sur le fichier _SP danshen_UCHL3_ glidedock , cliquez sur le fichier danshen_UCHL3_ glidedock _SP_pv.maezz , puis cliquez sur Ouvrir.
- Cliquez sur l’option Tableau , puis affichez le score sous Score d’ancrage.
- Visualisez les interactions danshensu et UCHL3.
- Double-cliquez sur le fichier danshen_UCHL3_glidedock _SP_pv.maegz et ouvrez-le dans Maestro. Dans la liste des entrées, maintenez la touche Maj enfoncée et sélectionnez simultanément danshensu et la protéine.
- Cliquez avec le bouton droit de la souris et sélectionnez Fusionner pour créer une nouvelle structure. Sélectionnez la nouvelle structure, cliquez avec le bouton droit de la souris, choisissez Exporter, puis cliquez sur Structures, nommez le fichier danshensu_UCHL3 et exportez-le au format .pdb.
- Sélectionnez la structure de fusion dans le panneau, cliquez sur Tâches, sélectionnez Esquisse 2D et obtenez une image de structure 2D de l’interaction entre danshensu et UCHL3.
- Importez le fichier danshensu_UCHL3.pdb dans le logiciel pymol et visualisez-le sur la base de la structure 2D obtenue à partir de Maestro.
3. Purification de la protéine UCHL3
- Construction du plasmide d’expression procaryote pHUE-UCHL3
- Obtenez la séquence codante du gène de la protéine recombinante UCHL3 à partir de NCBI (isform2). Intégrer le fragment de gène obtenu dans le vecteur pHUE-10HIS par recombinaison homologue et le transformer en cellules compétentes DH5α. Extrayez l’ADN plasmidique pour obtenir la construction souhaitée.
- Induction et purification de protéines recombinantes
- Culture bactérienne :
- Transformer les plasmides recombinants en cellules compétentes BL21 (DE3) et les étaler sur des plaques de gélose Luria-Bertani (LB) contenant de l’ampicilline. Incuber les plaques à 37 °C pendant la nuit.
- Prélever des colonies individuelles, les inoculer dans 12 mL de milieu LB liquide complété par 50 μg/mL d’ampicilline, et les faire cultiver pendant une nuit à 37 °C.
- Transformation et induction
- Diluer la culture bactérienne de nuit à 2 % à l’aide d’un milieu frais. Lorsque la DO 600 a atteint 0,4-0,6, ajouter l’isopropyl-bêta-D-thiogalactopyranoside (IPTG) à une concentration finale de 0,4 mM pour une induction à basse température à 16 °C pendant 12 h.
- Collecte de cultures bactériennes
- Transférez la culture bactérienne dans des tubes à centrifuger stériles et centrifugez à 2200 x g pendant 10 min. Retirez le surnageant et conservez les souches à -80 °C pour la conservation.
- Sonication
- Remettre la souche en suspension dans 25 mL (1/20 de la culture bactérienne recueillie) du tampon 1 (50 mM HEPES pH 7,5, 200 mM NaCl, 1 mM EDTA). Effectuez la sonication bactérienne dans les conditions suivantes : allumé pendant 4 s, éteint pendant 6 s, énergie réglée à 60 %, pendant 15 min. Ajouter 1 % de Triton X-100 et laisser reposer à 4 °C pendant 30 à 60 min.
- Séparation du surnageant et du granulé
- Centrifuger l’échantillon à 9000 x g à 4 °C pendant 30 min. Récupérez le surnageant et filtrez-le à travers un filtre à membrane de 0,45 μm. Réserver 50 μL du surnageant comme entrée.
- Remettez la pastille en suspension dans le tampon 1, ajoutez une quantité appropriée de tampon d’échantillon 5x (25 % de Tris-HCl 1M [pH 6,8], 10 % de dodécylsulfate de sodium, 0,5 % de bleu de bromophénol, 41,67 % de glycérol et 10 % de DL-dithiothréitol) et faites bouillir à 100 °C pendant 10 min.
- Culture bactérienne :
- Purification des protéines
- Équilibrage des colonnes
- Chargez 1 mL de résine de nickel NiNTA dans la colonne, laissez-la reposer pendant 10 min, puis équilibrez séquentiellement la colonne avec 5 volumes de colonne de tampon 2 (50 mM HEPES pH 7,5, 50 mM NaCl) contenant 10 mM, 500 mM et 10 mM d’imidazole.
- À l’aide d’une pipette, ajouter 5 mL (environ 5 volumes de colonne) de la solution d’imidazole de 10 mM le long de la paroi de la colonne. Faites ce processus sans contrôler le débit.
- Une fois que le liquide recouvre à peine le matériau d’emballage et est presque égoutté, ajoutez séquentiellement 5 volumes de colonne de la solution d’imidazole de 500 mM et de la solution d’imidazole de 10 mM pour l’équilibration. Lorsque la solution d’imidazole restante de 10 mM ne couvre que le matériau d’emballage, fermez le régulateur de débit pour arrêter l’équilibre.
- Purification des protéines
- Faire passer le surnageant protéique filtré dans la colonne à un débit de 0,5 mL/min (<15 s par goutte), en recueillant la fraction d’écoulement. Préparez un gradient de concentration de 10 mM, 30 mM, 50 mM et 100 mM d’imidazole à l’aide du tampon 2 et de 0,5 M de NaCl.
- Éluer séquentiellement de faibles à élevées concentrations d’imidazole, à l’aide d’une pipette pour ajouter environ 5 volumes de solution le long de la paroi de la colonne sans contrôler le débit. Prélever l’éluat dans des tubes de microcentrifugation propres de 1,5 ml, en prenant 1 ml par tube.
- Après avoir collecté cinq tubes, prélever 1 μL de flux continu de chaque tube pour réagir avec 1 μL de tampon Bradford. Si la réaction reste bleue, continuez les étapes ; S’il devient transparent, arrêtez l’élution à cette concentration et passez à la suivante.
- Après l’élution avec 100 mM d’imidazole, laver avec 10 volumes de colonne de 0,5 M NaCl sans contrôler le débit ni recueillir l’éluat.
- Enfin, éluer avec 500 mM d’imidazole (<15 s par goutte). À l’aide d’une pipette, ajoutez 1 mL de la solution dans la colonne, en maintenant le taux d’élution lent susmentionné. Prélever 1 mL de l’éluat à l’aide d’un tube de microcentrifugation de 1,5 mL et répéter ce processus 10 fois.
- Évaluez la protéine purifiée à l’aide de la coloration Coomassie Brilliant Blue.
- Quantifiez la pastille précédemment obtenue, le surnageant de pré-purification et les 10 tubes protéiques collectés en prélevant 10 μL de chaque groupe et en ajoutant le tampon d’échantillon 5x approprié, puis en chauffant à 100 °C pendant 5 min.
- Préparer la solution étalon d’albumine sérique bovine (BSA) à 1 mg/mL, 500 μg/mL et 100 μg/mL. Ajoutez ensuite le tampon d’échantillon 5x approprié et chauffez à 100 °C pendant 5 min.
- Effectuez une électrophorèse sur gel SDS-PAGE sur les échantillons, puis retirez le gel et incuberez-le à température ambiante (RT) dans une solution de Coomassie Brilliant Blue sur un agitateur à basse vitesse pendant la nuit.
- Le lendemain, transférez le gel dans une solution décolorante (40 % d’éthanol, 10 % d’acide acétique, 50 % de H2O) pour la décoloration, en appliquant une chaleur douce si nécessaire. Une fois que le gel est transparent, observez à l’aide d’un instrument de développement pour quantifier la protéine purifiée en fonction des niveaux de BSA et évaluer la pureté en vérifiant la présence d’une seule bande.
- Stockage des protéines : Aliquote les protéines dans des tubes de microcentrifugation à raison de 50 μL par tube, congélation instantanée dans de l’azote liquide et stockage à -80 °C.
- Équilibrage des colonnes
4. Dosage de l’activité UCHL3 (dosage Ub-AMC)
- Préparation protéinée
- Décongelez la protéine sur de la glace, centrifugez l’échantillon à 1000 x g à RT pendant 3 min et déterminez la concentration en protéines à l’aide de la méthode BCA. Diluez la protéine à une concentration de 40 nM à l’aide du tampon 1.
- Configuration des groupes
- Désignez le tampon 1 comme groupe témoin et UCHL3 comme groupe expérimental. Ajouter 200 μL de chaque échantillon par puits dans une plaque de 96 puits, en créant 3 répétitions par groupe.
- Détection
- Juste avant la mesure, ajoutez rapidement Ub-AMC dans chaque puits et agitez vigoureusement pendant 2 à 5 s. La concentration d’Ub-AMC utilisée est de 250 nM. Mesurez les valeurs de diamètre extérieur à une longueur d’onde d’excitation de 380 nm et à une longueur d’onde d’émission de 460 nm dans des conditions de 37 °C, en prenant des mesures toutes les 30 s pendant 10 minutes maximum.
5. Dosage de l’inhibition de l’activité d’UCHL3 par HA-Ub-VS (dosage HA-Ub-VS)
- Préparation protéinée
- Décongelez la protéine sur de la glace, centrifugez l’échantillon à 1000 x g à RT pendant 3 min et déterminez la concentration en protéines à l’aide de la méthode BCA. Diluer la protéine à une concentration de 10 μg/mL.
- Préparation de petites molécules
- Pesez Danshensu et diluez-le en gradient avec de l’eau de haute pureté à 5 mM, 1 mM, 100 μM, 10 μM et 1 μM.
- Configuration des groupes
- Établissez le groupe des protéines pures et le groupe sans médicaments à petites molécules en tant que groupes témoins négatifs. Utilisez PR619 (inhibiteur de DUB) comme groupe témoin positif, et les autres groupes comme groupes de traitement médicamenteux à petites molécules.
- Préparation des échantillons
- Selon le regroupement, ajouter séquentiellement 9 μL d’UCHL3 et 1 μL de Dansensu aux concentrations correspondantes à chaque groupe traité par le médicament. Ajouter 1 μL de PR619 (50 μM en utilisation) au groupe témoin positif. Utilisez de l’eau de haute pureté et le tampon 1 pour compenser le groupe témoin négatif.
- Bien mélanger par vortex et incuber à 37 °C pendant 30 min. Placez les échantillons de réaction sur de la glace, ajoutez HA-Ub-VS jusqu’à une concentration finale de 1 μM, mélangez soigneusement par vortex et incubez à 37 °C pendant 30 min.
- Ajouter une quantité appropriée de tampon d’échantillon 5x et chauffer dans un bain métallique à 100 °C pendant 10 min. Mettez les échantillons à RT pendant 5 minutes et chargez-les sur le gel.
6. Transfert Western
- Préparation du gel SDS-PAGE
- Rincez un ensemble de panneaux de verre avec de l’eau ultra-pure. Placez les plaques dans la fente de serrage, puis sur une planche transparente. Ajoutez de l’eau ultra-pure et vérifiez l’étanchéité pendant 10 min.
- Préparez la colle de séparation selon la table de distribution de colle.
- Versez l’eau de haute pureté de la cassette de gel et absorbez l’eau restante avec du papier filtre. Ajoutez rapidement et uniformément le gel séparateur à 12 % préparé. Ajoutez ensuite 1 ml d’isopropanol et attendez environ 30 minutes jusqu’à ce que le gel se solidifie.
- Préparez la couche supérieure de colle concentrée selon la table de distribution de colle.
- Retirez l’isopropanol du haut du gel de séparation, lavez-le 3 fois avec de l’eau de haute pureté et séchez-le avec du papier filtre. Ajoutez rapidement le gel d’empilage à 3 % préparé, insérez un peigne à 15 trous de 1,0 mm et attendez environ 30 min.
REMARQUE : Gardez le peigne perpendiculaire à la surface de la colle et assurez-vous qu’aucune bulle d’air n’apparaît.
- Électrophorèse
- Retirez verticalement le peigne et versez une quantité suffisante de tampon coulant dans la chambre intérieure de l’appareil d’électrophorèse, en vous assurant que le liquide recouvre les puits de l’échantillon.
- Placez le marqueur protéique et les échantillons expérimentaux préparés à RT, faites un tour complet et centrifugez brièvement. Ajouter 3 μL de marqueur protéique dans le premier puits d’échantillon de gauche, puis 10 μL d’échantillon expérimental dans chaque puits subséquent.
- Ajoutez une quantité appropriée de tampon de coulée dans la chambre extérieure du réservoir d’électrophorèse, couvrez le réservoir avec son couvercle et connectez l’alimentation électrique. Assurez-vous que l’alimentation est correctement connectée.
- Allumez l’alimentation électrique et réglez la tension sur 80 V jusqu’à ce que les échantillons se concentrent en lignes, puis augmentez la tension à 120 V une fois la séparation commencée dans le gel de séparation.
REMARQUE : Pendant le processus, assurez-vous qu’aucune bulle ne se forme au fond. Si des bulles apparaissent, inclinez l’appareil d’électrophorèse d’un côté pour les expulser.
- Transfert sur membrane
- À l’aide d’un couteau en plastique, ouvrez la cassette de gel. Découpez un coin en haut à gauche où le chargement de l’échantillon a commencé pour marquer l’orientation. Faites tremper le gel et le papier filtre requis dans le tampon de transfert pendant 1 à 2 min.
- Mesurez et coupez la membrane de difluorure de polyvinylidène (PVDF) en fonction de la taille nécessaire pour la protéine cible. Activez la membrane PVDF en la trempant dans du méthanol pendant 20 s à 1 min, puis plongez-la avec le gel dans le tampon de transfert.
- Versez une petite quantité de tampon de transfert dans l’appareil de transfert et utilisez un rouleau pour mouiller l’unité de transfert semi-sec. Disposez les composants dans l’ordre suivant de bas en haut : trois couches de papier filtre, de gel, de membrane PVDF et trois autres couches de papier filtre.
- Après avoir placé chaque couche, utilisez le rouleau pour éliminer les bulles. Mouillez le couvercle avec un tampon de transfert, puis couvrez l’appareil.
REMARQUE : Assurez-vous qu’une quantité adéquate de tampon de transfert est présente, en ajoutant une petite quantité après avoir terminé chaque couche. Préparez à l’avance des marquages, en distinguant les côtés gauche et droit du gel où les échantillons ont été chargés, ainsi qu’en identifiant l’avant et l’arrière de la membrane. Une fois chaque couche placée, roulez doucement pour éliminer les bulles. - Allumez l’alimentation en veillant à ce que la polarité soit correcte. Réglez le courant à 190 mA et réglez la durée sur 45 min.
- Bloquant
- Préparez une solution de lait écrémé en poudre à 5 % dans du TBST (1 % de Tween20) comme tampon de blocage. Placez la membrane PVDF avec la face avant vers le haut dans une boîte d’hybridation, versez 10 ml de tampon de blocage et secouez doucement sur un agitateur à basse vitesse à RT pendant 1 h pour bloquer.
- Incubation de l’anticorps primaire
- Préparez l’anticorps primaire avec la solution d’anticorps1 à 1:1000 et ajoutez 10 ml d’anticorps primaire dans la nouvelle boîte d’incubation. Placez la boîte d’incubation dans une chambre froide à 4 °C et conservez-la sur un agitateur pendant la nuit.
- Incubation de l’anticorps secondaire
- Recueillez l’anticorps primaire dans la boîte d’incubation le lendemain, puis ajoutez la solution de TBST appropriée et placez-la sur un agitateur pour laver pendant 5 min, répétez l’opération 3 fois.
- Préparez l’anticorps secondaire avec une solution de lait à 5 % à 1:5000. Déposer 1 mL d’anticorps secondaires au fond d’une boîte d’hybridation humidifiée. Placez la membrane PVDF avec le côté protéique vers le bas sur l’anticorps secondaire, puis incubez à RT sur un agitateur à basse vitesse pendant 1,5 h.
- Ajoutez la solution TBST appropriée et placez-la sur un shaker pour laver pendant 5 min ; Répétez 3 fois.
- Exposition des bandes
- Prenez des volumes égaux de réactifs chimiluminescents A et B, agitez et mélangez bien. Protégez la solution de la lumière.
- Appliquez la solution de développement uniformément sur la membrane PVDF, en la secouant doucement pour vous assurer que la membrane est uniformément recouverte de la solution.
- Placez la membrane dans le système d’imagerie sur gel et réglez le temps d’exposition, le nombre d’expositions et d’autres paramètres pertinents.
REMARQUE : Le développeur doit couvrir complètement la bande et le développement doit se faire dans un environnement sombre.
Résultats
Pour éliminer les petites molécules de Salvia miltiorrhiza Bge qui peuvent inhiber efficacement UCHL3, nous avons effectué un amarrage moléculaire entre les petites molécules obtenues sur le site Web TCMSP avec UCHL3. Les 30 premières petites molécules dans les résultats d’amarrage et leurs scores sont présentés dans le tableau 1. Les résultats d’amarrage pour toutes les petites molécules sont présentés dans le tableau supplémentaire 1. Nous avons sélectionné Danshensu comme petite molécule représentative pour la recherche. Comme le montre la figure 1, Danshensu et UCHL3 ont une liaison hydrogène avec ARG-186, HIS-169, VAL-166 et ASN-93, respectivement, et les scores d’amarrage entre eux sont de -4,75 kcal/mol avec une activité de liaison favorable.
Pour valider l’activité inhibitrice de Danshensu sur UCHL3, nous avons procédé en purifiant la protéine recombinante d’UCHL3 in vitro, comme le montre la figure 2A. Ensuite, des tests d’hydrolyse Ub-AMC ont été effectués pour évaluer l’activité enzymatique de la protéine UCHL3 purifiée. Comme le montre la figure 2B, par rapport au groupe témoin, la valeur de fluorescence du groupe UCHL3 était plus élevée dans des conditions de lumière d’excitation de 380 nm et de lumière d’émission de 460 nm. De plus, en 2 minutes, les valeurs de fluorescence du groupe UCHL3 ont montré une relation linéaire avec le temps de réaction et ont atteint un plateau après 2 minutes. Il a démontré que l’UCHL3 purifié conserve une activité enzymatique significative in vitro, répondant ainsi aux exigences expérimentales.
Ici, nous rapportons une autre sonde, HA-Ub-VS, qui peut se lier de manière covalente avec UCHL3. L’effet inhibiteur de Danshensu sur l’activité d’UCHL3 peut être démontré par une liaison compétitive avec HA-Ub-VS. Comme le montre la figure 3A, lorsque la protéine UCHL3 est ajoutée seule, elle migre à environ 25 kDa. Cependant, lors de l’ajout de la sonde d’un poids moléculaire d’environ 10 kDa, la bande se déplace vers environ 35 kDa, indiquant une UCHL3 inhibée. La capacité d’UCHL3 à se lier à HA-Ub-VS diminue progressivement avec l’augmentation des concentrations de Danshensu. Comme le montre la figure 3B, la valeur IC50 de Danshensu pour inhiber de manière compétitive la liaison d’UCHL3 à HA-Ub-VS est de 12,91 μM.
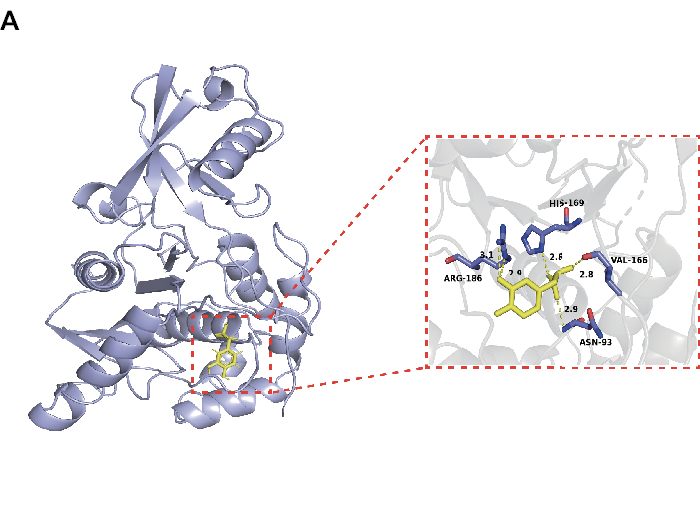
Figure 1 : Interaction entre Danshensu et la protéine UCHL3 (PDB : 1XD3). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Essai d’hydrolyse Ub-AMC catalysé par UCHL3. (A) L’image SDS-PAGE de la protéine cible UCHL3 purifiée in vitro. (B) Le résultat de l’essai d’hydrolyse Ub-AMC catalysé par UCHL3, les données sont représentées sous forme de moyenne ± ET. Veuillez cliquer ici pour voir une version agrandie de cette figure.
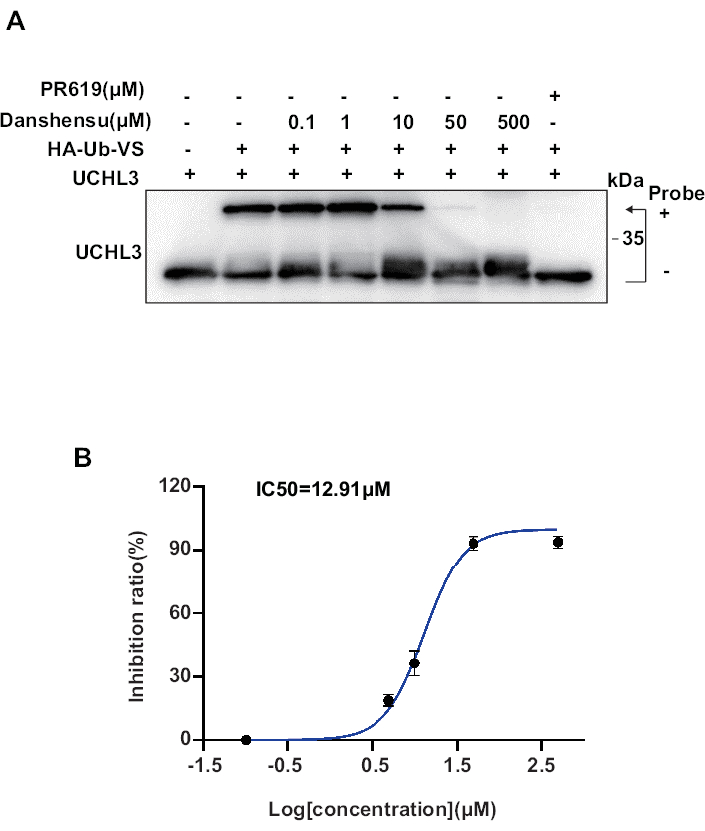
Figure 3 : Utilisation de HA-Ub-VS pour détecter l’activité d’inhibition de Danshensu contre UCHL3. (A) L’effet inhibiteur de Danshensu sur UCHL3 à différentes concentrations. (B) La courbe de quantification de Danshensu inhibant l’activité UCHL3, avec Danshensu inhibant de manière compétitive HA-Ub-VS, a donné une valeur IC50 de 12,91 μM. Les données sont représentées sous forme de moyenne ± ET. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Les noms et les scores des 30 principales petites molécules d’amarrage moléculaire. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 1 : Les résultats d’amarrage de toutes les petites molécules de Salvia miltiorrhiza Bge avec UCHL3.> Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Les DUBs jouent un rôle crucial dans la régulation de l’homéostasie de l’ensemble du système ubiquitine en éliminant l’ubiquitine des substrats ou des chaînes de polyubiquitine37. Ces dernières années, ces enzymes ont également attiré beaucoup d’attention en tant que cibles pour le développement de médicaments13. Cependant, le processus de développement de médicaments à petites molécules présente des défis. Par exemple, le criblage à haut débit impliquant des dizaines de milliers de petites bibliothèques de molécules entraîne des coûts élevés et une charge de travail importante38. La MTC a une histoire de milliers d’années d’utilisation clinique et une base théorique solide en médecine chinoise, et ses produits naturels jouent un rôle irremplaçable dans la prévention et le traitement des maladies humaines, ainsi que dans l’ouverture etla conception de médicaments.
Dans cet article, nous rapportons une méthode qui combine l’amarrage moléculaire avec la technologie de sonde de protéase spécifique de l’ubiquitine pour identifier les produits naturels de la médecine traditionnelle chinoise qui inhibent des cibles DUB spécifiques. Les résultats ont montré que Danshensu avait une activité d’amarrage élevée contre UCHL3 dans la bibliothèque de petites molécules de la médecine traditionnelle chinoise Salvia miltiorrhiza Bge. Des expériences in vitro ont démontré que Danshensu pouvait inhiber de manière compétitive l’activité d’UCHL3 avec HA-Ub-VS. La méthode actuelle, qui combine la bio-informatique et les expériences moléculaires, peut être appliquée à la prédiction d’inhibiteurs ou d’agonistes de cibles.
Dans l’expérience, plusieurs points clés nécessitent une attention particulière. Tout d’abord, avant d’effectuer un amarrage moléculaire, il est crucial de bien comprendre les caractéristiques structurelles du site actif catalytique de la protéine cible. Privilégiez la sélection de structures présentant une co-cristallisation de protéines avec de petites molécules qui inhibent l’activité de cette protéine. Deuxièmement, lors de la purification des protéines, il est essentiel de préserver leurs régions critiques d’activité enzymatique. Si la protéine est trop grosse et difficile à exprimer ou à purifier en quantité suffisante, une option consiste à tronquer sa séquence pour préserver ses régions actives. Lors de l’utilisation de protéines, des efforts doivent être faits pour éviter les cycles répétés de gel-dégel afin d’éviter la perte d’activité. Troisièmement, pour les expériences d’inhibition compétitive entre les petites molécules et HA-Ub-VS, assurez-vous que l’expérience est menée à 37 °C pour faciliter une interaction suffisante entre la petite molécule et le DUB. De plus, assurez-vous que le solvant de la petite molécule ne provoque pas d’inactivation de la protéine pour éviter de générer des résultats faussement positifs.
Cependant, au cours du processus de criblage de médicaments, cette technique peut rencontrer des résultats faussement négatifs en raison d’une sensibilité insuffisante de la sonde et de concentrations excessives de protéines. Pour répondre à ces problèmes potentiels, nous proposons plusieurs solutions. Nous visons à augmenter la concentration des sondes pour assurer une interaction adéquate avec les protéines tout en maintenant les niveaux de sous-saturation des protéines. Cette approche permet d’éviter les faux négatifs causés par la liaison compétitive entre le médicament et la sonde. Dans des applications pratiques, nous avons observé que la sonde capte efficacement les différences significatives d’activité protéique induites par les médicaments. Cependant, il reste moins efficace pour détecter les différences subtiles. Par conséquent, lors du criblage de médicaments, nous intégrerons des techniques plus raffinées, telles que la résonance plasmonique de surface (SPR)39 et le test de décalage thermique cellulaire (CETSA)40, pour une évaluation complète. Néanmoins, en tant que premier outil de criblage des composés actifs DUB, la technologie de la sonde présente toujours un excellent rapport coût-performance.
Les technologies d’amarrage moléculaire et de sonde d’ubiquitine sont deux méthodes importantes dans l’étude des inhibiteurs de DUB. L’amarrage moléculaire repose sur des prédictions virtuelles basées sur la structure cible, en utilisant des algorithmes spécifiques pour simuler les interactions entre les petites molécules et les protéines41. Les petites molécules rapportées dans la MTC sont classées en fonction de leurs scores d’amarrage, guidant les expériences ultérieures de validation de l’activité in vitro et servant de méthode de criblage initial pour les produits naturels en médecine chinoise42,43. L’application des sondes d’activité d’ubiquitination est un outil important dans le domaine de la recherche sur l’ubiquitination, et le développement de la technologie des sondes d’activité DUB est continuellement itéré et mis à jour23,24. Ub-AMC est une sonde précoce utilisée pour détecter l’activité DUB. Il est largement utilisé dans le criblage à haut débit des inhibiteurs de DUB en raison de ses méthodes de détection pratiques17. Cependant, à l’instar d’autres sondes comme Ub-TAMRA, son domaine d’application est limité par la plage de détection de longueur d’onde d’excitationrestreinte 44. Alors que ces dernières années, le test Ub-Rho a émergé avec une gamme de longueurs d’onde d’excitation plus large, élargissant son application dans la découverte de médicaments, la cinétique des composés et les expériences moléculaires45,46.
En plus de l’application de sondes pour détecter l’activité des protéines purifiées in vitro, les DUB peuvent également être utilisés dans divers autres contextes. HA-Ub-VS, une sonde où une tête nucléophile est liée à l’extrémité C-terminale de l’ubiquitine, comme d’autres conceptions similaires comme Ub-VME et Ub-PA, détecte non seulement l’activité de DUB, mais forme également une liaison covalente avec DUB par l’attaque nucléophile de la tête23. Dans des conditions où l’on utilise des anticorps endogènes, l’activité de la DUB peut être caractérisée à l’aide du transfert Western. Lorsque l’Ub est attaché à un marqueur ou marqué à la biotine, le DUB qui subit une liaison covalente peut être enrichi et caractérisé par spectrométrie de masse. Cette approche permet la détection simultanée de plusieurs activités DUB, ce qui améliore considérablement l’efficacité de la détection46. Les DUBs sont spécifiques à la chaîne d’ubiquitine, et la limitation des sondes ci-dessus est que le facteur de spécificité de la chaîne est ignoré7. Mais ces dernières années, des sondes spécifiques à la chaîne ont également été appliquées avec succès dans plusieurs études47,48. L’application de sondes sélectives DUB sera la clé du développement d’inhibiteurs de DUB. À l’heure actuelle, il est difficile de cibler les DUB de métalloprotéase avec des sondes d’ubiquitination. Ce domaine offre des possibilités de recherche plus poussée dans la mise au point d’inhibiteurs de DUB23,24.
En résumé, les technologies d’amarrage moléculaire et de sonde d’ubiquitine jouent un rôle crucial dans la recherche sur les inhibiteurs de DUB, fournissant des méthodes et des outils importants pour le développement de médicaments naturels issus de la médecine traditionnelle chinoise.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par la Fondation nationale des sciences naturelles de Pékin [numéro de subvention 7244498].
matériels
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide | Beijing Lablead Biotech Co., Ltd | A3291 | |
| Ammonium persulfate | China National Medicines Corporation Ltd | 10002616 | |
| Anti-rabbit IgG, HRP-linked Antibody #7074 | Cell Signaling Technology | 7074P2 | |
| BeyoECL Plus | Beyotime | P0018S | |
| Bradford Protein Assay Kit | Beyotime | P0006 | |
| ClonExpress Ultra One Step Cloning Kit | Vazyme | C115-01 | |
| Danshensu | Shanghai yuanye Bio-Technology Co., Ltd | B20254 | |
| DMSO | Ameresco, Inc. | 21K2356571 | |
| Electrophoresis System | Liuyi Biotechnology | 112-0630 | |
| HEPES | Sigma | H3375 | |
| His-tagged protein purification kit (NTA-Ni agarose magnetic beads) | Beyotime | P2247S | |
| Immun-Blot PVDF Membrane, Roll, 26 cm x 3.3 m | Bio-Rad Laboratories (Shanghai) Co., Ltd | 1620177 | |
| Isopropyl alcohol | Macklin | I811925 | |
| M5 Prestained Protein Ladder | Mei5 Biotechnology Co.Ltd | MF-212-01 | |
| Maestro | Schrödinger’s | https://www.schrodinger.com/platform/products/maestro/ | |
| Methyl alcohol | China National Medicines Corporation Ltd | 10014108 | |
| MF-Millipore | Millipore | HAWP04700 | |
| MyFug mini centrifuge | Sigma | Z764183 | |
| Pierce Dilution-Free Rapid Gold BCA Protein Assay | Thermo Scientific | A55860 | |
| PR-619 | Cell Signaling Technology | 26065S | |
| Primary Antibody Dilution Buffer for Western Blot | Macklin | P917820 | |
| Recombinant Human HA-Ubiquitin Vinyl Sulfone Protein, CF | R&D Systems | U-212-025 | |
| Recombinant Human Ubiquitin AMC Protein, CF | R&D Systems | U-550-050 | |
| Skim Milk | Becton,Dickinson and Company | 232100 | |
| Sodium Dodecyl Sulfate (SDS) | Ameresco, Inc. | 205-788-1 | |
| TEMED | Ameresco, Inc. | 2545C134 | |
| Tween 20 | Beijing Lablead Biotech Co., Ltd | 0777-1 | |
| UCHL3 (D25E6) Rabbit mAb | Cell Signaling Technology | 8141T |
Références
- Ciechanover, A. The ubiquitin proteolytic system and pathogenesis of human diseases: A novel platform for mechanism-based drug targeting. Biochem Soc Trans. 31 (2), 474-481 (2003).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by e1 enzymes: The apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10 (5), 319-331 (2009).
- Ye, Y., Rape, M. Building ubiquitin chains: E2 enzymes at work. Nat Rev Mol Cell Biol. 10 (11), 755-764 (2009).
- Buetow, L., Huang, D. T. Structural insights into the catalysis and regulation of E3 ubiquitin ligases. Nat Rev Mol Cell Biol. 17 (10), 626-642 (2016).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: Structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Clague, M. J., et al. Deubiquitylases from genes to organism. Physiol Rev. 93 (3), 1289-1315 (2013).
- Clague, M. J., Urbé, S., Komander, D. Breaking the chains: Deubiquitylating enzyme specificity begets function. Nat Rev Mol Cell Biol. 20 (6), 338-352 (2019).
- Harrigan, J. A., Jacq, X., Martin, N. M., Jackson, S. P. Deubiquitylating enzymes and drug discovery: Emerging opportunities. Nat Rev Drug Discov. 17 (1), 57-78 (2018).
- Mevissen, T. E. T., Komander, D. Mechanisms of deubiquitinase specificity and regulation. Annu Rev Biochem. 86, 159-192 (2017).
- Hershko, A., Ciechanover, A., Heller, H., Haas, A. L., Rose, I. A. Proposed role of ATP in protein breakdown: Conjugation of protein with multiple chains of the polypeptide of ATP-dependent proteolysis. Proc Natl Acad Sci U S A. 77 (4), 1783-1786 (1980).
- Abdul Rehman, S. A., et al. Mindy-1 is a member of an evolutionarily conserved and structurally distinct new family of deubiquitinating enzymes. Mol Cell. 63 (1), 146-155 (2016).
- Storer, A. C., Ménard, R. Catalytic mechanism in papain family of cysteine peptidases. Methods Enzymol. 244, 486-500 (1994).
- Lange, S. M., Armstrong, L. A., Kulathu, Y. Deubiquitinases: From mechanisms to their inhibition by small molecules. Mol Cell. 82 (1), 15-29 (2022).
- Ndubaku, C., Tsui, V. Inhibiting the deubiquitinating enzymes (dubs). J Med Chem. 58 (4), 1581-1595 (2015).
- Schauer, N. J., Magin, R. S., Liu, X., Doherty, L. M., Buhrlage, S. J. Advances in discovering deubiquitinating enzyme (dub) inhibitors. J Med Chem. 63 (6), 2731-2750 (2020).
- Magin, R. S., et al. Small molecules as tools for functional assessment of deubiquitinating enzyme function. Cell Chem Biol. 28 (7), 1090-1100 (2021).
- Dang, L. C., Melandri, F. D., Stein, R. L. Kinetic and mechanistic studies on the hydrolysis of ubiquitin c-terminal 7-amido-4-methylcoumarin by deubiquitinating enzymes. Biochemistry. 37 (7), 1868-1879 (1998).
- Li, Y. T., et al. New semi-synthesis of ubiquitin c-terminal conjugate with 7-amino-4-methylcoumarin. J Pept Sci. 20 (2), 102-107 (2014).
- Feng, X., et al. Ubiquitination of UVRAG by SMURF1 promotes autophagosome maturation and inhibits hepatocellular carcinoma growth. Autophagy. 15 (7), 1130-1149 (2019).
- Qin, X., et al. Identification of an autoinhibitory, mitophagy-inducing peptide derived from the transmembrane domain of USP30. Autophagy. 18 (9), 2178-2197 (2022).
- Borodovsky, A., et al. Chemistry-based functional proteomics reveals novel members of the deubiquitinating enzyme family. Chem Biol. 9 (10), 1149-1159 (2002).
- Ovaa, H. Active-site directed probes to report enzymatic action in the ubiquitin proteasome system. Nat Rev Cancer. 7 (8), 613-620 (2007).
- Ekkebus, R., Flierman, D., Geurink, P. P., Ovaa, H. Catching a dub in the act: Novel ubiquitin-based active site-directed probes. Curr Opin Chem Biol. 23, 63-70 (2014).
- D'arcy, P., et al. Inhibition of proteasome deubiquitinating activity as a new cancer therapy. Nat Med. 17 (12), 1636-1640 (2011).
- Sucher, N. J. The application of Chinese medicine to novel drug discovery. Expert Opin DrugDiscov. 8 (1), 21-34 (2013).
- Jung, I., Kim, H., Moon, S., Lee, H., Kim, B. Overview of salvia miltiorrhiza as a potential therapeutic agent for various diseases: An update on efficacy and mechanisms of action. Antioxidants (Basel). 9 (9), 857 (2020).
- Wang, Z., Peters, R. J. Tanshinones: Leading the way into lamiaceae labdane-related diterpenoid biosynthesis. Curr Opin Plant Biol. 66, 102189 (2022).
- Bai, M., et al. Astrocytes and microglia-targeted danshensu liposomes enhance the therapeutic effects on cerebral ischemia-reperfusion injury. J Control Release. 364, 473-489 (2023).
- Zhang, H., et al. Salvianolic acid a protects RPE cells against oxidative stress through activation of Nrf2/HO-1 signaling. Free Radic Biol Med. 69, 219-228 (2014).
- Wang, R., et al. Danshensu inhibits sars-cov-2 by targeting its main protease as a specific covalent inhibitor and discovery of bifunctional compounds eliciting antiviral and anti-inflammatory activity. Int J Biol Macromol. 257 (Pt 2), 128623 (2024).
- Misaghi, S., et al. Structure of the ubiquitin hydrolase UCH-L3 complexed with a suicide substrate. J Biol Chem. 280 (2), 1512-1520 (2005).
- Wing, S. S. Deubiquitinating enzymes--the importance of driving in reverse along the ubiquitin-proteasome pathway. Int J Biochem Cell Biol. 35 (5), 590-605 (2003).
- Samy, M. A., Abd El Fatah, N. M., Yahia, S. E., Arafa, R. K. Friend or foe: UCHL3 mediated carcinogenesis and current approaches in small molecule inhibitors' development. Curr Med Chem. 28 (42), 8782-8799 (2021).
- Hafez, N., Modather El-Awadly, Z., Arafa, R. K. UCH-L3 structure and function: Insights about a promising drug target. Eur J Med Chem. 227, 113970 (2022).
- Song, Z., et al. A novel UCHL(3) inhibitor, perifosine, enhances PARP inhibitor cytotoxicity through inhibition of homologous recombination-mediated DNA double strand break repair. Cell Death Dis. 10 (3), 398 (2019).
- Hirayama, K., Aoki, S., Nishikawa, K., Matsumoto, T., Wada, K. Identification of novel chemical inhibitors for ubiquitin c-terminal hydrolase-L3 by virtual screening. Bioorg Med Chem. 15 (21), 6810-6818 (2007).
- Nijman, S. M., et al. A genomic and functional inventory of deubiquitinating enzymes. Cell. 123 (5), 773-786 (2005).
- Guo, M., et al. High-throughput screening for amyloid-β binding natural small-molecules based on the combinational use of biolayer interferometry and UHPLC-DAD-Q/TOF-MS/MS. Acta Pharm Sin B. 12 (4), 1723-1739 (2022).
- Chavanieu, A., Pugnière, M. Developments in spr fragment screening. Expert Opin Drug Discov. 11 (5), 489-499 (2016).
- Dai, L., et al. Horizontal cell biology: Monitoring global changes of protein interaction states with the proteome-wide cellular thermal shift assay (CETSA). Annu Rev Biochem. 88, 383-408 (2019).
- Pinzi, L., Rastelli, G. Molecular docking: Shifting paradigms in drug discovery. Int J Mol Sci. 20 (18), 4331 (2019).
- Shang, L., et al. Mechanism of Sijunzi decoction in the treatment of colorectal cancer based on network pharmacology and experimental validation. J Ethnopharmacol. 302 (Pt A), 115876 (2023).
- Fan, Z., Wang, S., Xu, C., Yang, J., Cui, B. Mechanisms of action of fu fang gang liu liquid in treating condyloma acuminatum by network pharmacology and experimental validation. BMC Complement Med Ther. 23 (1), 128 (2023).
- Tirat, A., et al. Synthesis and characterization of fluorescent ubiquitin derivatives as highly sensitive substrates for the deubiquitinating enzymes UCH-L3 and USP-2. Anal Biochem. 343 (2), 244-255 (2005).
- Hassiepen, U., et al. A sensitive fluorescence intensity assay for deubiquitinating proteases using ubiquitin-rhodamine110-glycine as substrate. Anal Biochem. 371 (2), 201-207 (2007).
- Chan, W. C., et al. Accelerating inhibitor discovery for deubiquitinating enzymes. Nat Commun. 14 (1), 686 (2023).
- Haj-Yahya, N., et al. Dehydroalanine-based diubiquitin activity probes. Org Lett. 16 (2), 540-543 (2014).
- Li, G., Liang, Q., Gong, P., Tencer, A. H., Zhuang, Z. Activity-based diubiquitin probes for elucidating the linkage specificity of deubiquitinating enzymes. Chem Commun (Camb). 50 (2), 216-218 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon