Method Article
Detección de compuestos de la medicina tradicional china para inhibir la actividad de UCHL3 basada en la tecnología de sonda de enzimas de acoplamiento molecular y desubiquitinación
* Estos autores han contribuido por igual
En este artículo
Resumen
El experimento utilizado aquí muestra un método de acoplamiento molecular combinado con tecnologías de sonda para predecir y validar la interacción entre pequeñas moléculas de la medicina tradicional china y objetivos proteicos.
Resumen
Las enzimas desubiquitinantes (DUB) desempeñan un papel fundamental en la modulación de la homeostasis de la ubiquitinación, siendo UCHL3 una cisteína DUB arquetípica intrincadamente involucrada en una miríada de procesos fisiológicos y patológicos. Por lo tanto, el desarrollo de inhibidores de moléculas pequeñas dirigidos a la ubiquitina C-terminal hidrolasa L3 (UCHL3) es de gran importancia. Este protocolo tiene como objetivo establecer un proceso de cribado virtual y validación in vitro de inhibidores de moléculas pequeñas de cisteína DUB representadas por UCHL3. En primer lugar, los posibles inhibidores de UCHL3 se criban virtualmente mediante tecnología de acoplamiento molecular, y se visualiza la interacción entre los fármacos y las proteínas diana. Posteriormente, se verifica la eficacia del fármaco seleccionado, Danshensu, mediante ensayos de inhibición de la actividad in vitro . La ubiquitina-7-amino-4-metilcumarina (Ub-AMC) y la hemaglutinina-ubiquitina-vinil sulfona (HA-Ub-VS) se utilizan como sondas para las pruebas de actividad in vitro , ya que pueden unirse competitivamente a DUB con inhibidores de moléculas pequeñas para evaluar la actividad de UCHL3. Los resultados indican que Danshensu tiene una buena afinidad de unión con UCHL3 en el acoplamiento molecular, y puede inhibir competitivamente la actividad de UCHL3 con HA-Ub-VS. Estos hallazgos proporcionan referencias importantes para futuras investigaciones y desarrollos de fármacos terapéuticos dirigidos a UCHL3.
Introducción
La ubiquitinación es una modificación postraduccional de las proteínas, un proceso mediante el cual las enzimas activadoras de ubiquitina E1, las enzimas conjugadoras de ubiquitina E2 y las ligasas de ubiquitina E3 se unen a la proteína objetivo, y todo el proceso de ubiquitinación puede revertirse mediante enzimas desubiquitinantes (DUB)1,2,3,4. Debido a su importante papel fisiológico y patológico, los DUB se consideran objetivos importantes para el descubrimiento de fármacos 5,6.
Se han identificado más de 100 DUBs en humanos 7,8. Por lo general, funcionan como una isopeptidasa responsable de romper el enlace isopeptídico entre el extremo C-terminal de la ubiquitina y un residuo de lisina en un sustrato u otra molécula de ubiquitina 9,10. En la actualidad, se clasifican principalmente en siete familias principales, a saber: peptidasas específicas de ubiquitina (USP), proteasas de tumores de ovario (OTU), proteasas Jab1/Mov34/Mpr1 Pad 1 N-terminal + dominio proteasas (JAMM), motivo que interactúa con nuevas proteasas de la familia DUB que contienen ubiquitina (MINDY), hidroxilasas C-terminales de ubiquitina (UCH), proteasas de dominio Machado-Josephin (MJD) y peptidasa de ubiquitina 1 que contiene dedos de zinc (ZUP1)9. Además de las JAMMs, que pertenecen a la familia de las metaloproteasas de zinc11, las otras DUBs son proteasas de cisteína caracterizadas por una tríada catalítica formada por cisteína catalítica, histidina y un tercer residuo ácido12,13. Esta especificidad abre vías para el desarrollo de inhibidores de moléculas pequeñas dirigidos al sitio activo de la enzima o a las bolsas alostéricas cercanas.
En el campo de la investigación de las enzimas deubiquitinantes, existe un desafío significativo en la caracterización de su actividad14,15. La caracterización basada en sondas activas sirve como un enfoque crucial para el estudio de los inhibidores de DUB16. Mediante la realización de ensayos competitivos con sondas e inhibidores basados en la actividad en lisados celulares o proteínas recombinantes, se puede caracterizar la actividad de los DUB, lo que facilita el desarrollo de inhibidores de moléculas pequeñas dirigidos a estas enzimas. Ub-AMC es una sonda temprana utilizada para detectar la actividad de DUB, que tiene un grupo fluorescente unido al extremo C-terminal de la ubiquitina17,18. Cuando los DUB ejercen su actividad catalítica, AMC se libera en grandes cantidades y la intensidad fluorescente de su luz detectada aumenta en consecuencia. Esta sonda ha sido ampliamente utilizada en el cribado de alto rendimiento de inhibidores de DUB19,20. La sonda HA-Ub-VS también se utiliza para medir la actividad DUB21. Tiene un grupo vinil sulfona en el extremo C de la ubiquitina, lo que lo convierte en un sustrato suicida para los DUB. Después de la separación por electroforesis en gel de dodecil sulfato de sodio-poliacrilamida (SDS-PAGE), los DUB activos se pueden detectar mediante Western blot 22,23,24.
La medicina tradicional china (MTC) utiliza plantas medicinales desde hace más de 2000 años. El desarrollo de nuevos fármacos a partir de productos naturales es de gran importancia médica, con un enfoque clave en la identificación de ingredientes activos y la elucidación de sus mecanismos25. Salvia miltiorrhiza El bge es una hierba ampliamente utilizada en el tratamiento de una variedad de enfermedades, incluyendo el cáncer, cardiovasculares, hepáticas y neurológicas26. Actualmente, contiene moléculas pequeñas conocidas como tanshinona27, Danshensu28, ácido tanshinic29, etc. Estos compuestos exhiben diversas actividades biológicas como efectos antitrombóticos, antioxidantes y antitumorales, lo que los hace muy valiosos para la investigación26. Estudios recientes han identificado a Danshensu como un inhibidor covalente de la proteasa similar a la 3-quimotripsina (3CLpro) del SARS-CoV-230. Se ha demostrado que forma un enlace covalente con el residuo del sitio activo C145 de 3CLpro, lo que indica la presencia de posibles inhibidores de la proteasa de moléculas pequeñas en Salvia miltiorrhiza Bge.
La ubiquitina C-terminal hidrolasa L3 (UCHL3) pertenece a las cisteína proteasas dentro de la familia de DUBs de la UCH. Se basa en residuos conservados como la cisteína95, la histidina169 y el ácido aspártico184 para catalizar sus funciones de manera efectiva31,32. Desempeña un papel crucial en múltiples vías moleculares, incluido el ciclo celular, la recombinación homóloga y la reparación de roturas de ADN vinculadas a proteínas33. Además, está regulado al alza en varios tipos de cáncer, como el cáncer de ovario, próstata, páncreas, colorrectal y pulmón de células no pequeñas34. Sobre la base de estos estudios, UCHL3 parece ser un objetivo prometedor para el tratamiento de enfermedades. Se han identificado varios inhibidores de moléculas pequeñas de UCHL3 que están progresando hacia su uso clínico35,36.
En este estudio, se realizó un acoplamiento molecular para investigar las interacciones entre moléculas pequeñas de Salvia miltiorrhiza Bge y UCHL3. Posteriormente, un experimento in vitro utilizando las sondas específicas de DUB Ub-AMC y HA-Ub-VS identificó a Danshensu como un inhibidor de molécula pequeña de UCHL3. El acoplamiento molecular también predijo posibles sitios de unión para Danshensu, lo que sugiere su mecanismo de acción.
Protocolo
1. Descarga de las estructuras de pequeñas moléculas de Salvia miltiorrhiza Bge y UCHL3
- Descargue el archivo de la molécula pequeña.
- Abra la base de datos TCMSP (https://old.tcmsp-e.com), ingrese danshen (nombre de la hierba), luego presione buscar y haga clic en Radix Salviae en la lista de resultados.
- Haga clic en descargar uno por uno de los elementos y guarde la estructura 2D en formato .mol2.
- Descarga el archivo de proteínas.
- Abra la base de datos PDB (https://www.rcsb.org/), escriba UCHL3 y, a continuación, pulse buscar. Haz clic en Homo sapiens y, a continuación, pulsa Buscar en refinamientos.
- Seleccione la estructura 1XD3 con cocristalización de molécula pequeña y proteína, luego haga clic en descargar archivos y seleccione el formato PDB .
2. Acoplamiento molecular
- Guarde el archivo.
- Cree una nueva carpeta llamada danshen_UCHL3 docking en el escritorio y guarde los compuestos de las estructuras Salvia miltiorrhiza Bge y UCHL3 en esta carpeta. Asigne un nombre a la carpeta en inglés; de lo contrario, el archivo no se podrá importar.
- Establezca la ruta.
- Abra el software Maestro, haga clic en Archivo, seleccione Cambiar directorio de trabajo, haga clic en Escritorio, haga doble clic para seleccionar la carpeta de acoplamiento danshen_CUHL3 y haga clic en Elegir opciones.
- Procesamiento de moléculas pequeñas
- Importar estructuras de moléculas pequeñas.
- Haga clic en las opciones Archivo e Importar estructuras. Haga clic en Escritorio, haga doble clic en la carpeta de acoplamiento y haga clic en el archivo de estructura Salvia miltiorrhiza Bge. A continuación, haga clic en Abrir para importar todas las moléculas pequeñas.
- Prepara la molécula pequeña.
- Haga clic en Tareas y seleccione la opción LigPrep . En la ventana de LigPrep que se muestra, haga clic en la tabla del proyecto en la opción Usar estructura desde , marque la opción Determinar quiralidades a partir de estructura 3D en cálculo y deje todas las demás configuraciones de software como predeterminadas.
- Cambie el nombre del trabajo a danshen_ligprep1 y, a continuación, haga clic en Ejecutar para ejecutar el procesamiento de moléculas pequeñas.
- Importar estructuras de moléculas pequeñas.
- Procesamiento de la estructura de las proteínas
- Importar la estructura de la proteína.
- Haga clic en las opciones Archivo e Importar estructuras , haga clic en Escritorio y haga doble clic en la carpeta de acoplamiento danshen_CUHL3.
- Haga clic en el archivo de estructura de proteínas UCHL3 y, a continuación, haga clic en Abrir para importar el archivo de estructura de proteínas.
- Prepara la proteína.
- Seleccione los dos enlaces covalentes que conectan UCHL3 y la molécula pequeña, y elimínelos con el botón Eliminar. A continuación, seleccione los dos residuos de proteínas que están incompletos después de la eliminación. Haga clic en el botón Compilar, seleccione Otras ediciones y, a continuación, para Gly75 haga clic en el botón C, para Cys95 elija Mutar residuo y CYS.
- Haga clic en Tareas y seleccione la opción Flujo de trabajo de preparación de proteínas . Deje todas las demás configuraciones de software como predeterminadas. Cambie el nombre del trabajo a UCHL3_protein pre y, a continuación, haga clic en Ejecutar para realizar el procesamiento de proteínas.
- Configure la caja de acoplamiento.
- Haga clic en Tareas, elija Generación de cuadrícula de receptores y seleccione Seleccionar para identificar la molécula del ligando . Seleccione la molécula pequeña en el workspac e y aparecerá un cuadro de acoplamiento rosa centrado en las coordenadas de la molécula pequeña.
- Mantenga la configuración predeterminada, asigne al trabajo el nombre 1XD3_danshen_glide_grid y, a continuación, haga clic en Ejecutar.
NOTA: En 1XD3, la pequeña molécula forma un enlace covalente con la proteína. Es necesario eliminar este enlace covalente para separar la pequeña molécula de la proteína. De lo contrario, durante el proceso de configuración de la rejilla del receptor, no se puede seleccionar la molécula pequeña.
- Importar la estructura de la proteína.
- Realizar acoplamiento molecular.
- Haga clic en Tareas y seleccione la opción Acoplamiento de ligandos . Seleccione la cuadrícula del receptor, haga clic en Desde archivo y, a continuación, haga clic en Examinar. Seleccione 1XD3_danshen_glide_grid.zip archivo y, a continuación, haga clic en Abrir.
- Haga clic en Usar ligandos como archivos y, a continuación, haga clic en Examinar. Haga clic en danshen_ligprep1 archivo, seleccione danshen_ligprep1-out. maegz file y, a continuación, haga clic en Abrir.
- Haga clic en Configuración y seleccione la opción Precisión como SP , cambie el nombre del trabajo a danshen_UCHL3_ _SP de deslizamiento y haga clic en Ejecutar.
- Ver los resultados del acoplamiento.
- Haga clic en las opciones Archivo e Importar estructuras . Haga clic en Escritorio y haga doble clic en la carpeta de acoplamiento danshen_CUHL3.
- Haga doble clic en el archivo de _SP de deslizamiento danshen_UCHL3_, haga clic en el archivo danshen_UCHL3_ de _SP_pv.maegz de deslizamiento y, a continuación, haga clic en Abrir.
- Haga clic en la opción Tabla y, a continuación, vea la puntuación en Puntuación de acoplamiento.
- Visualice las interacciones de danshensu y UCHL3.
- Haga doble clic en el archivo danshen_UCHL3_glidedock _SP_pv.maegz y ábralo en Maestro. En la lista de entradas, mantenga presionada la tecla Mayús y seleccione simultáneamente danshensu y la proteína.
- Haga clic con el botón derecho y seleccione Combinar para crear una nueva estructura. Seleccione la nueva estructura, haga clic con el botón derecho, elija Exportar y, a continuación, haga clic en Estructuras, asigne un nombre danshensu_UCHL3 al archivo y expórtelo en formato .pdb.
- Seleccione la estructura de combinación en el panel, haga clic en Tareas, seleccione Boceto 2D y obtenga una imagen de la estructura 2D de la interacción entre danshensu y UCHL3.
- Importe el archivo danshensu_UCHL3.pdb en el software pymol y visualícelo en base a la estructura 2D obtenida de Maestro.
3. Purificación de la proteína UCHL3
- Construcción del plásmido de expresión procariota pHUE-UCHL3
- Obtener la secuencia codificante del gen UCHL3 de la proteína recombinante del NCBI (isform2). Integrar el fragmento de gen obtenido en el vector pHUE-10HIS mediante recombinación homóloga y transformarlo en células competentes para DH5α. Extraiga el ADN plasmídico para obtener la construcción deseada.
- Inducción y purificación de proteínas recombinantes
- Cultivo bacteriano:
- Transformar los plásmidos recombinantes en células competentes BL21 (DE3) y esparcirlos en placas de agar Luria-Bertani (LB) que contienen ampicilina. Incubar las placas a 37 °C durante la noche.
- Recoger colonias individuales, inocularlas en 12 mL de medio LB líquido suplementado con 50 μg/mL de ampicilina y cultivar durante la noche a 37 °C.
- Transformación e inducción
- Diluya el cultivo bacteriano durante la noche al 2% utilizando un medio fresco. Cuando OD600 alcance 0,4-0,6, agregue isopropil-beta-D-tiogalactopiranósido (IPTG) hasta una concentración final de 0,4 mM para la inducción a baja temperatura a 16 °C durante 12 h.
- Colección de cultivo bacteriano
- Transfiera el cultivo bacteriano a tubos de centrífuga estériles y centrifugue a 2200 x g durante 10 min. Retire el sobrenadante y almacene las cepas a -80 °C para su conservación.
- Sonicación
- Vuelva a suspender la cepa en 25 mL (1/20 del cultivo bacteriano recogido) de Tampón 1 (50 mM HEPES pH 7,5, 200 mM NaCl, 1 mM EDTA). Realizar la sonicación bacteriana en las siguientes condiciones: encendido durante 4 s, apagado durante 6 s, energía ajustada al 60%, durante 15 min. Añadir un 1% de Triton X-100 y lisar a 4 °C durante 30-60 min.
- Separación de sobrenadante y pellet
- Centrifugar la muestra a 9000 x g a 4 °C durante 30 min. Recoja el sobrenadante y fíltrelo a través de un filtro de membrana de 0,45 μm. Reserve 50 μL del sobrenadante como entrada.
- Vuelva a suspender el pellet en el tampón 1, añada una cantidad adecuada de tampón de muestra 5x (25 % de Tris-HCl 1M [pH 6,8], 10 % de dodecil sulfato de sodio, 0,5 % de azul de bromofenol, 41,67 % de glicerol y 10 % de DL-ditiotreitol) y hervir a 100 °C durante 10 min.
- Cultivo bacteriano:
- Purificación de proteínas
- Equilibrio de columnas
- Cargue 1 mL de resina de níquel NiNTA en la columna, deje que se asiente durante 10 minutos, luego equilibre secuencialmente la columna con 5 volúmenes de columna de tampón 2 (50 mM HEPES pH 7.5, 50 mM NaCl) que contienen 10 mM, 500 mM y 10 mM de imidazol.
- Use una pipeta para agregar 5 mL (aproximadamente 5 volúmenes de columna) de la solución de imidazol de 10 mM a lo largo de la pared de la columna. Realice este proceso sin controlar el caudal.
- Una vez que el líquido cubra el material de empaque y esté casi drenado, agregue secuencialmente 5 volúmenes de columna de la solución de imidazol de 500 mM y la solución de imidazol de 10 mM para equilibrar. Cuando los 10 mM restantes de la solución de imidazol solo cubran el material de empaque, cierre el controlador de flujo para detener el equilibrio.
- Purificación de proteínas
- Pase el sobrenadante de proteína filtrada a través de la columna a un caudal de 0,5 mL/min (<15 s por gota), recogiendo la fracción de flujo. Prepare un gradiente de concentración de 10 mM, 30 mM, 50 mM y 100 mM de imidazol utilizando el tampón 2, junto con 0,5 M de NaCl.
- Eluir secuencialmente de concentraciones bajas a altas de imidazol, utilizando una pipeta para agregar aproximadamente 5 volúmenes de columna de solución a lo largo de la pared de la columna sin controlar el caudal. Recoja el eluido en tubos de microcentrífuga limpios de 1,5 mL, con un flujo de 1 mL por tubo.
- Después de recolectar cinco tubos, tome 1 μL de flujo de cada tubo para que reaccione con 1 μL de tampón Bradford. Si la reacción sigue siendo azul, continúe con los pasos; Si se vuelve transparente, detenga la elución en esa concentración y cambie a la siguiente.
- Después de eluir con 100 mM de imidazol, lavar con 10 volúmenes de columna de 0,5 M de NaCl sin controlar el caudal ni recoger el eluido.
- Finalmente, eluir con 500 mM de imidazol (<15 s por gota). Con ayuda de una pipeta, añadir 1 mL de la solución a la columna, manteniendo la tasa de elución lenta antes mencionada. Recoja 1 mL del eluido utilizando un tubo de microcentrífuga de 1,5 mL y repita este proceso 10 veces.
- Evalúe la proteína purificada utilizando la tinción Coomassie Brilliant Blue.
- Cuantifique el pellet previamente obtenido, el sobrenadante de prepurificación y los 10 tubos de proteínas recolectados tomando 10 μL de cada grupo y agregando el tampón de muestra 5x apropiado, luego calentando a 100 °C durante 5 min.
- Prepare la solución patrón de albúmina sérica bovina (BSA) a 1 mg/mL, 500 μg/mL y 100 μg/mL. A continuación, añada el tampón de muestra 5x adecuado y caliente a 100 °C durante 5 min.
- Realice la electroforesis en gel SDS-PAGE en las muestras, luego retire el gel e incube a temperatura ambiente (RT) en una solución de Coomassie Brilliant Blue en un agitador de baja velocidad durante la noche.
- Al día siguiente, transfiera el gel a una solución decolorante (40% de etanol, 10% de ácido acético, 50% de H2O) para la decoloración, aplicando calor suave según sea necesario. Una vez que el gel es transparente, observe bajo un instrumento de desarrollo para cuantificar la proteína purificada en función de los niveles de BSA y evaluar la pureza mediante la verificación de una sola banda.
- Almacenamiento de proteínas: Aliquotar la proteína en tubos de microcentrífuga a 50 μL por tubo, congelar rápidamente en nitrógeno líquido y almacenar a -80 °C.
- Equilibrio de columnas
4. Ensayo de actividad UCHL3 (ensayo Ub-AMC)
- Preparación de proteínas
- Descongele la proteína en hielo, centrifugue la muestra a 1000 x g a RT durante 3 minutos y determine la concentración de proteína utilizando el método BCA. Diluya la proteína a una concentración de 40 nM utilizando el tampón 1.
- Configuración de grupos
- Designe el Tampón 1 como el grupo de control y UCHL3 como el grupo experimental. Agregue 200 μL de cada muestra por pocillo en una placa de 96 pocillos, configurando 3 réplicas por grupo.
- Detección
- Justo antes de la medición, agregue rápidamente Ub-AMC a cada pocillo y agite vigorosamente durante 2-5 s. La concentración de Ub-AMC utilizada es de 250 nM. Mida los valores de diámetro exterior a una longitud de onda de excitación de 380 nm y una longitud de onda de emisión de 460 nm en condiciones de 37 °C, tomando lecturas cada 30 s durante un máximo de 10 minutos.
5. Ensayo de inhibición de la actividad de UCHL3 por HA-Ub-VS (ensayo HA-Ub-VS)
- Preparación de proteínas
- Descongele la proteína en hielo, centrifugue la muestra a 1000 x g a RT durante 3 minutos y determine la concentración de proteína utilizando el método BCA. Diluir la proteína hasta una concentración de 10 μg/mL.
- Preparación de moléculas pequeñas
- Pesar Danshensu y diluirlo en un gradiente con agua de alta pureza a 5 mM, 1 mM, 100 μM, 10 μM y 1 μM.
- Configuración de grupos
- Configure el grupo de proteína pura y el grupo sin fármacos de molécula pequeña como grupos de control negativos. Utilice PR619 (inhibidor de DUB) como grupo de control positivo y los otros grupos como grupos de tratamiento con fármacos de moléculas pequeñas.
- Preparación de la muestra
- De acuerdo con el agrupamiento, agregue secuencialmente 9 μL de UCHL3 y 1 μL de Dansensu a las concentraciones correspondientes a cada grupo tratado con medicamentos. Agregue 1 μL de PR619 (50 μM en uso) al grupo de control positivo. Use agua de alta pureza y tampón 1 para compensar el grupo de control negativo.
- Mezclar bien por vórtice e incubar a 37 °C durante 30 min. Colocar las muestras de reacción en hielo, añadir HA-Ub-VS hasta una concentración final de 1 μM, mezclar bien por vórtice e incubar a 37 °C durante 30 min.
- Añadir una cantidad adecuada de tampón de muestra 5x y calentar en un baño metálico a 100 °C durante 10 min. Coloque las muestras en RT durante 5 minutos y cárguelas en el gel.
6. Western blot
- Preparación del gel SDS-PAGE
- Enjuague un conjunto de paneles de vidrio con agua ultrapura. Coloque las placas en la ranura de sujeción, luego en una tabla transparente. Agregue agua ultrapura y verifique las fugas durante 10 minutos.
- Prepare el pegamento de separación de acuerdo con la mesa de dispensación de pegamento.
- Vierta el agua de alta pureza del casete de gel y absorba el agua restante con papel de filtro. Agregue rápida y uniformemente el gel separador al 12% preparado. Luego agregue 1 mL de isopropanol y espere aproximadamente 30 minutos hasta que el gel se solidifique.
- Prepare la capa superior de pegamento concentrado de acuerdo con la tabla de dispensación de pegamento.
- Retira el isopropanol de la parte superior del gel separador, lávalo 3 veces con agua de alta pureza y sécalo con papel de filtro. Agregue rápidamente el gel de apilamiento al 3% preparado, inserte un peine de 1,0 mm y 15 orificios y espere aproximadamente 30 minutos.
NOTA: Mantenga el peine perpendicular a la superficie del pegamento y asegúrese de que no aparezcan burbujas de aire.
- Electroforesis
- Retire verticalmente el peine y vierta una cantidad suficiente de tampón corriente en la cámara interna del aparato de electroforesis, asegurándose de que el líquido cubra los pocillos de muestra.
- Coloque el marcador de proteína y las muestras experimentales preparadas en RT, vórtice a fondo y centrifugar brevemente. Añadir 3 μL de marcador de proteína en el primer pocillo de muestra de la izquierda, seguido de 10 μL de muestra experimental en cada pocillo subsiguiente.
- Agregue una cantidad adecuada de tampón de funcionamiento en la cámara exterior del tanque de electroforesis, cubra el tanque con su tapa y conecte la fuente de alimentación. Asegúrese de que la fuente de alimentación esté conectada correctamente.
- Encienda la fuente de alimentación y ajuste el voltaje a 80 V hasta que las muestras se concentren en líneas, luego aumente el voltaje a 120 V una vez que comience la separación en el gel separador.
NOTA: Durante el proceso, asegúrese de que no se formen burbujas en la parte inferior. Si aparecen burbujas, incline el aparato de electroforesis hacia un lado para expulsarlas.
- Transferencia a membrana
- Use un cuchillo de plástico para abrir el casete de gel. Corte una esquina en la parte superior izquierda donde comenzó la carga de la muestra para marcar la orientación. Remoje el gel y el papel de filtro requerido en el tampón de transferencia durante 1-2 minutos.
- Mida y corte la membrana de difluoruro de polivinilideno (PVDF) de acuerdo con el tamaño necesario para la proteína objetivo. Active la membrana de PVDF sumergiéndola en metanol durante 20 s a 1 min, luego sumérjala con el gel en el tampón de transferencia.
- Vierta una pequeña cantidad de tampón de transferencia en el aparato de transferencia y use un rodillo para humedecer la unidad de transferencia semiseca. Coloque los componentes en el siguiente orden de abajo hacia arriba: tres capas de papel de filtro, gel, membrana de PVDF y tres capas más de papel de filtro.
- Después de colocar cada capa, use el rodillo para eliminar las burbujas. Humedece la tapa con tampón de transferencia y luego cubre el aparato.
NOTA: Asegúrese de que haya una cantidad adecuada de búfer de transferencia, agregando una pequeña cantidad después de completar cada capa. Prepare las marcas con anticipación, distinguiendo entre los lados izquierdo y derecho del gel donde se cargaron las muestras, así como identificando la parte delantera y trasera de la membrana. Después de colocar cada capa, enrolle suavemente para eliminar las burbujas. - Encienda la fuente de alimentación, asegurándose de la polaridad correcta. Ajuste la corriente a 190 mA y ajuste el tiempo a 45 min.
- Bloqueante
- Prepare una solución de leche en polvo descremada al 5% en TBST (1% Tween20) como tampón de bloqueo. Coloque la membrana de PVDF con la parte delantera hacia arriba en una caja de hibridación, vierta 10 ml de tampón de bloqueo y agite suavemente un agitador de baja velocidad a RT durante 1 h para bloquear.
- Incubación de anticuerpo primario
- Prepare el anticuerpo primario con la solución de anticuerpos1 a 1:1000 y agregue 10 ml de anticuerpo primario a la nueva caja de incubación. Coloque la caja de incubación en una cámara frigorífica a 4 °C y manténgala en un agitador durante la noche.
- Incubación de anticuerpos secundarios
- Recoja el anticuerpo primario en la caja de incubación al día siguiente, luego agregue la solución TBST adecuada y colóquela en un agitador para lavar durante 5 minutos, repita 3 veces.
- Prepare el anticuerpo secundario con una solución de leche al 5% a 1:5000. Coloque 1 ml de anticuerpo secundario en el fondo de una caja de hibridación humedecida. Coloque la membrana de PVDF con el lado de la proteína hacia abajo sobre el anticuerpo secundario y, a continuación, incube a RT en un agitador de baja velocidad durante 1,5 h.
- Agregue la solución TBST adecuada y colóquela en una coctelera para lavar durante 5 minutos; Repita 3 veces.
- Exposición de las tiras
- Tome volúmenes iguales de los reactivos quimioluminiscentes A y B, agite y mezcle bien. Protege la solución de la luz.
- Aplique la solución reveladora de manera uniforme sobre la membrana de PVDF, agitándola suavemente para asegurarse de que la membrana esté uniformemente cubierta con la solución.
- Coloque la membrana en el sistema de imágenes de gel y establezca el tiempo de exposición, el número de exposiciones y otros parámetros relevantes.
NOTA: El desarrollador debe cubrir completamente la tira y el desarrollo debe estar en un entorno oscuro.
Resultados
Para descartar las moléculas pequeñas en la Salvia miltiorrhiza Bge que pueden inhibir eficazmente UCHL3, realizamos un acoplamiento molecular entre las moléculas pequeñas obtenidas del sitio web de TCMSP con UCHL3. Las 30 moléculas pequeñas principales en los resultados de acoplamiento y sus puntuaciones se muestran en la Tabla 1. Los resultados de acoplamiento para todas las moléculas pequeñas se presentan en la Tabla Suplementaria 1. Seleccionamos Danshensu como una molécula pequeña representativa para la investigación. Como se muestra en la Figura 1, Danshensu y UCHL3 tienen enlaces de hidrógeno con ARG-186, HIS-169, VAL-166 y ASN-93, respectivamente, y las puntuaciones de acoplamiento entre ellos son de -4,75 kcal/mol con actividad de unión favorable.
Para validar la actividad inhibitoria de Danshensu en UCHL3, procedimos a purificar la proteína recombinante de UCHL3 in vitro, como se muestra en la Figura 2A. A continuación, se realizaron ensayos de hidrólisis de Ub-AMC para evaluar la actividad enzimática de la proteína UCHL3 purificada. Como se muestra en la Figura 2B, en comparación con el grupo de control, el valor de fluorescencia del grupo UCHL3 fue mayor en condiciones de luz de excitación de 380 nm y luz de emisión de 460 nm. Además, en 2 min los valores de fluorescencia del grupo UCHL3 mostraron una relación lineal con el tiempo de reacción y alcanzaron una meseta después de 2 min. Demostró que el UCHL3 purificado conserva una actividad enzimática significativa in vitro, cumpliendo con los requisitos experimentales.
Aquí, reportamos otra sonda, HA-Ub-VS, que puede unirse covalentemente con UCHL3. El efecto inhibidor de Danshensu sobre la actividad de UCHL3 puede demostrarse mediante la unión competitiva con HA-Ub-VS. Como se muestra en la Figura 3A, cuando la proteína UCHL3 se agrega sola, migra a alrededor de 25 kDa. Sin embargo, tras la adición de la sonda con un peso molecular de aproximadamente 10 kDa, la banda cambia a alrededor de 35 kDa, lo que indica UCHL3 inhibido. La capacidad de UCHL3 para unirse a HA-Ub-VS disminuye gradualmente con el aumento de las concentraciones de Danshensu. Como se muestra en la Figura 3B, el valor IC50 de Danshensu para inhibir competitivamente la unión de UCHL3 a HA-Ub-VS es de 12,91 μM.
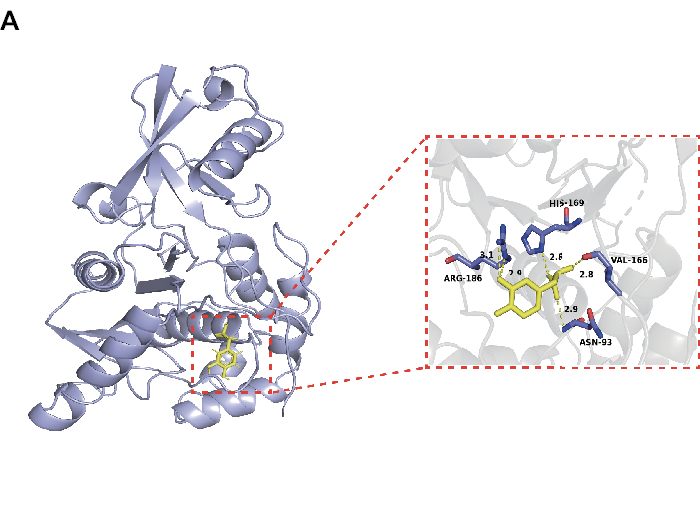
Figura 1: Interacción entre Danshensu y la proteína UCHL3 (PDB: 1XD3). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Ensayo de hidrólisis de Ub-AMC catalizado por UCHL3. (A) La imagen SDS-PAGE de la proteína diana UCHL3 purificada in vitro. (B) El resultado del ensayo de hidrólisis Ub-AMC catalizado por UCHL3, los datos se representan como media ± SD. Haga clic aquí para ver una versión más grande de esta figura.
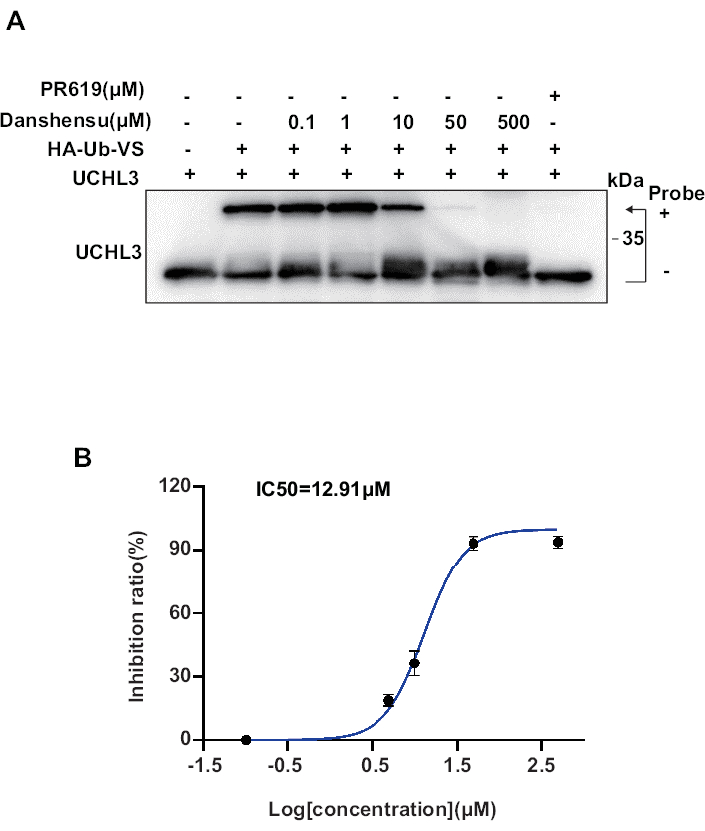
Figura 3: Uso de HA-Ub-VS para detectar la actividad de inhibición de Danshensu frente a UCHL3. (A) El efecto inhibidor de Danshensu sobre UCHL3 a diferentes concentraciones. (B) La curva de cuantificación de Danshensu inhibiendo la actividad de UCHL3, con Danshensu inhibiendo competitivamente HA-Ub-VS, arrojó un valor de IC50 de 12,91 μM. Los datos se representan como media ± SD. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Los nombres y puntuaciones de las 30 moléculas pequeñas más importantes en acoplamiento molecular. Haga clic aquí para descargar esta tabla.
Tabla suplementaria 1: Los resultados de acoplamiento de todas las moléculas pequeñas de Salvia miltiorrhiza Bge con UCHL3.> Haga clic aquí para descargar este archivo.
Discusión
Los DUB juegan un papel crucial en la regulación de la homeostasis de todo el sistema de ubiquitina al eliminar la ubiquitina de los sustratos o cadenas de poliubiquitina37. En los últimos años, estas enzimas también han atraído mucha atención como objetivos para el desarrollo de fármacos13. Sin embargo, existen desafíos en el proceso de desarrollo de fármacos de moléculas pequeñas. Por ejemplo, el cribado de alto rendimiento en el que participan decenas de miles de bibliotecas de moléculas pequeñas genera costes elevados y una importante carga de trabajo38. La medicina tradicional china tiene una historia de miles de años de uso clínico y una sólida base teórica en la medicina china, y sus productos naturales desempeñan un papel insustituible en la prevención y el tratamiento de enfermedades humanas, así como en la apertura y el diseño de medicamentos25.
En este artículo, informamos sobre un método que combina el acoplamiento molecular con la tecnología de sonda de proteasa específica de ubiquitina para identificar productos naturales de la medicina tradicional china que inhiben objetivos específicos de DUB. Los resultados mostraron que Danshensu tenía una alta actividad de acoplamiento contra UCHL3 en la biblioteca de moléculas pequeñas de la medicina tradicional china Salvia miltiorrhiza Bge. Los experimentos in vitro demostraron que Danshensu podía inhibir competitivamente la actividad de UCHL3 con HA-Ub-VS. El presente método, que combina bioinformática y experimentos moleculares, puede aplicarse a la predicción de inhibidores o agonistas diana.
En el experimento, varios puntos clave requieren una atención especial. En primer lugar, antes de realizar el acoplamiento molecular, es crucial comprender a fondo las características estructurales del sitio activo catalítico de la proteína objetivo. Priorizar la selección de estructuras que presenten co-cristalización de proteínas con moléculas pequeñas que se ha reportado que inhiben la actividad de esta proteína. En segundo lugar, durante la purificación de proteínas, es esencial preservar sus regiones críticas de actividad enzimática. Si la proteína es demasiado grande y difícil de expresar o purificar en cantidades suficientes, una opción es truncar su secuencia para preservar sus regiones activas. Durante el uso de proteínas, se deben hacer esfuerzos para evitar ciclos repetidos de congelación y descongelación para evitar la pérdida de actividad. En tercer lugar, para los experimentos de inhibición competitiva entre moléculas pequeñas y HA-Ub-VS, asegúrese de que el experimento se realice a 37 °C para facilitar una interacción suficiente entre la molécula pequeña y DUB. Además, asegúrese de que el solvente de la molécula pequeña no cause inactivación de proteínas para evitar generar resultados falsos positivos.
Sin embargo, durante el proceso de cribado de fármacos, esta técnica puede encontrar resultados falsos negativos debido a la insuficiente sensibilidad de la sonda y a las concentraciones excesivas de proteínas. Para abordar estos posibles problemas, proponemos varias soluciones. Nuestro objetivo es aumentar la concentración de las sondas para asegurar una interacción adecuada con las proteínas mientras se mantienen los niveles de subsaturación de las proteínas. Este enfoque ayuda a prevenir los falsos negativos causados por la unión competitiva entre el fármaco y la sonda. En aplicaciones prácticas, hemos observado que la sonda captura eficazmente diferencias significativas en la actividad proteica inducida por los fármacos. Sin embargo, sigue siendo menos eficaz para detectar diferencias sutiles. Por lo tanto, durante el cribado de fármacos, incorporaremos técnicas más refinadas, como la Resonancia de Plasmón de Superficie (SPR)39 y el Ensayo de Desplazamiento Térmico Celular (CETSA)40, para una evaluación integral. Sin embargo, como herramienta inicial para el cribado de compuestos activos en DUB, la tecnología de sonda sigue demostrando una sólida relación coste-rendimiento.
Las tecnologías de acoplamiento molecular y sonda de ubiquitina son métodos importantes en el estudio de los inhibidores de DUB. El acoplamiento molecular se basa en predicciones virtuales basadas en la estructura del objetivo, utilizando algoritmos específicos para simular interacciones entre moléculas pequeñas y proteínas41. Las moléculas pequeñas reportadas en la medicina tradicional china se clasifican en función de sus puntuaciones de acoplamiento, guiando los experimentos posteriores de validación de la actividad in vitro y sirviendo como método de detección inicial para productos naturales en la medicina china42,43. La aplicación de sondas de actividad de ubiquitinación es una herramienta importante en el campo de la investigación de la ubiquitinación, y el desarrollo de tecnología para sondas de actividad DUB se itera y actualiza continuamente23,24. Ub-AMC es una sonda temprana utilizada para detectar la actividad de DUB. Es ampliamente utilizado en el cribado de alto rendimiento de inhibidores de DUB debido a sus convenientes métodos de detección17. Sin embargo, al igual que otras sondas como Ub-TAMRA, su rango de aplicación está limitado por el rango de detección de longitud de onda de excitación restringida44. Mientras que en los últimos años, el ensayo Ub-Rho ha surgido con un rango de longitud de onda de excitación más amplio, ampliando su aplicación en el descubrimiento de fármacos, cinética de compuestos y experimentos moleculares45,46.
Además de la aplicación de sondas para detectar la actividad de proteínas purificadas in vitro, los DUB también se pueden utilizar en otros contextos. HA-Ub-VS, una sonda en la que una cabeza nucleofílica está unida al extremo C-terminal de la ubiquitina, al igual que otros diseños similares como Ub-VME y Ub-PA, no solo detecta la actividad de DUB sino que también forma un enlace covalente con DUB a través del ataque nucleofílico de la cabeza23. En condiciones con anticuerpos endógenos, la actividad de DUB se puede caracterizar mediante Western blot. Cuando el Ub se adhiere a una etiqueta o se marca con biotina, el DUB que se une a la covalente puede enriquecerse y caracterizarse mediante espectrometría de masas. Este enfoque permite la detección simultánea de múltiples actividades DUB, mejorando significativamente la eficiencia de detección46. Los DUB son específicos de la cadena de ubiquitina, y la limitación de las sondas anteriores es que se ignora el factor de especificidad de la cadena7. Pero en los últimos años, las sondas específicas de cadena también se han aplicado con éxito en varios estudios47,48. La aplicación de sondas selectivas de DUB será la clave para el desarrollo de inhibidores de DUB. En la actualidad, es un reto dirigirse a los DUB de metaloproteasas con sondas de ubiquitinación. Esta área presenta oportunidades para futuras investigaciones en el desarrollo de inhibidores de DUB23,24.
En resumen, las tecnologías de acoplamiento molecular y sonda de ubiquitina desempeñan un papel crucial en la investigación de inhibidores de DUB, proporcionando métodos y herramientas importantes para el desarrollo de medicamentos de productos naturales a partir de la medicina tradicional china.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por la Fundación Nacional de Ciencias Naturales de Beijing [subvención número 7244498].
Materiales
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide | Beijing Lablead Biotech Co., Ltd | A3291 | |
| Ammonium persulfate | China National Medicines Corporation Ltd | 10002616 | |
| Anti-rabbit IgG, HRP-linked Antibody #7074 | Cell Signaling Technology | 7074P2 | |
| BeyoECL Plus | Beyotime | P0018S | |
| Bradford Protein Assay Kit | Beyotime | P0006 | |
| ClonExpress Ultra One Step Cloning Kit | Vazyme | C115-01 | |
| Danshensu | Shanghai yuanye Bio-Technology Co., Ltd | B20254 | |
| DMSO | Ameresco, Inc. | 21K2356571 | |
| Electrophoresis System | Liuyi Biotechnology | 112-0630 | |
| HEPES | Sigma | H3375 | |
| His-tagged protein purification kit (NTA-Ni agarose magnetic beads) | Beyotime | P2247S | |
| Immun-Blot PVDF Membrane, Roll, 26 cm x 3.3 m | Bio-Rad Laboratories (Shanghai) Co., Ltd | 1620177 | |
| Isopropyl alcohol | Macklin | I811925 | |
| M5 Prestained Protein Ladder | Mei5 Biotechnology Co.Ltd | MF-212-01 | |
| Maestro | Schrödinger’s | https://www.schrodinger.com/platform/products/maestro/ | |
| Methyl alcohol | China National Medicines Corporation Ltd | 10014108 | |
| MF-Millipore | Millipore | HAWP04700 | |
| MyFug mini centrifuge | Sigma | Z764183 | |
| Pierce Dilution-Free Rapid Gold BCA Protein Assay | Thermo Scientific | A55860 | |
| PR-619 | Cell Signaling Technology | 26065S | |
| Primary Antibody Dilution Buffer for Western Blot | Macklin | P917820 | |
| Recombinant Human HA-Ubiquitin Vinyl Sulfone Protein, CF | R&D Systems | U-212-025 | |
| Recombinant Human Ubiquitin AMC Protein, CF | R&D Systems | U-550-050 | |
| Skim Milk | Becton,Dickinson and Company | 232100 | |
| Sodium Dodecyl Sulfate (SDS) | Ameresco, Inc. | 205-788-1 | |
| TEMED | Ameresco, Inc. | 2545C134 | |
| Tween 20 | Beijing Lablead Biotech Co., Ltd | 0777-1 | |
| UCHL3 (D25E6) Rabbit mAb | Cell Signaling Technology | 8141T |
Referencias
- Ciechanover, A. The ubiquitin proteolytic system and pathogenesis of human diseases: A novel platform for mechanism-based drug targeting. Biochem Soc Trans. 31 (2), 474-481 (2003).
- Schulman, B. A., Harper, J. W. Ubiquitin-like protein activation by e1 enzymes: The apex for downstream signalling pathways. Nat Rev Mol Cell Biol. 10 (5), 319-331 (2009).
- Ye, Y., Rape, M. Building ubiquitin chains: E2 enzymes at work. Nat Rev Mol Cell Biol. 10 (11), 755-764 (2009).
- Buetow, L., Huang, D. T. Structural insights into the catalysis and regulation of E3 ubiquitin ligases. Nat Rev Mol Cell Biol. 17 (10), 626-642 (2016).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: Structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Clague, M. J., et al. Deubiquitylases from genes to organism. Physiol Rev. 93 (3), 1289-1315 (2013).
- Clague, M. J., Urbé, S., Komander, D. Breaking the chains: Deubiquitylating enzyme specificity begets function. Nat Rev Mol Cell Biol. 20 (6), 338-352 (2019).
- Harrigan, J. A., Jacq, X., Martin, N. M., Jackson, S. P. Deubiquitylating enzymes and drug discovery: Emerging opportunities. Nat Rev Drug Discov. 17 (1), 57-78 (2018).
- Mevissen, T. E. T., Komander, D. Mechanisms of deubiquitinase specificity and regulation. Annu Rev Biochem. 86, 159-192 (2017).
- Hershko, A., Ciechanover, A., Heller, H., Haas, A. L., Rose, I. A. Proposed role of ATP in protein breakdown: Conjugation of protein with multiple chains of the polypeptide of ATP-dependent proteolysis. Proc Natl Acad Sci U S A. 77 (4), 1783-1786 (1980).
- Abdul Rehman, S. A., et al. Mindy-1 is a member of an evolutionarily conserved and structurally distinct new family of deubiquitinating enzymes. Mol Cell. 63 (1), 146-155 (2016).
- Storer, A. C., Ménard, R. Catalytic mechanism in papain family of cysteine peptidases. Methods Enzymol. 244, 486-500 (1994).
- Lange, S. M., Armstrong, L. A., Kulathu, Y. Deubiquitinases: From mechanisms to their inhibition by small molecules. Mol Cell. 82 (1), 15-29 (2022).
- Ndubaku, C., Tsui, V. Inhibiting the deubiquitinating enzymes (dubs). J Med Chem. 58 (4), 1581-1595 (2015).
- Schauer, N. J., Magin, R. S., Liu, X., Doherty, L. M., Buhrlage, S. J. Advances in discovering deubiquitinating enzyme (dub) inhibitors. J Med Chem. 63 (6), 2731-2750 (2020).
- Magin, R. S., et al. Small molecules as tools for functional assessment of deubiquitinating enzyme function. Cell Chem Biol. 28 (7), 1090-1100 (2021).
- Dang, L. C., Melandri, F. D., Stein, R. L. Kinetic and mechanistic studies on the hydrolysis of ubiquitin c-terminal 7-amido-4-methylcoumarin by deubiquitinating enzymes. Biochemistry. 37 (7), 1868-1879 (1998).
- Li, Y. T., et al. New semi-synthesis of ubiquitin c-terminal conjugate with 7-amino-4-methylcoumarin. J Pept Sci. 20 (2), 102-107 (2014).
- Feng, X., et al. Ubiquitination of UVRAG by SMURF1 promotes autophagosome maturation and inhibits hepatocellular carcinoma growth. Autophagy. 15 (7), 1130-1149 (2019).
- Qin, X., et al. Identification of an autoinhibitory, mitophagy-inducing peptide derived from the transmembrane domain of USP30. Autophagy. 18 (9), 2178-2197 (2022).
- Borodovsky, A., et al. Chemistry-based functional proteomics reveals novel members of the deubiquitinating enzyme family. Chem Biol. 9 (10), 1149-1159 (2002).
- Ovaa, H. Active-site directed probes to report enzymatic action in the ubiquitin proteasome system. Nat Rev Cancer. 7 (8), 613-620 (2007).
- Ekkebus, R., Flierman, D., Geurink, P. P., Ovaa, H. Catching a dub in the act: Novel ubiquitin-based active site-directed probes. Curr Opin Chem Biol. 23, 63-70 (2014).
- D'arcy, P., et al. Inhibition of proteasome deubiquitinating activity as a new cancer therapy. Nat Med. 17 (12), 1636-1640 (2011).
- Sucher, N. J. The application of Chinese medicine to novel drug discovery. Expert Opin DrugDiscov. 8 (1), 21-34 (2013).
- Jung, I., Kim, H., Moon, S., Lee, H., Kim, B. Overview of salvia miltiorrhiza as a potential therapeutic agent for various diseases: An update on efficacy and mechanisms of action. Antioxidants (Basel). 9 (9), 857(2020).
- Wang, Z., Peters, R. J. Tanshinones: Leading the way into lamiaceae labdane-related diterpenoid biosynthesis. Curr Opin Plant Biol. 66, 102189(2022).
- Bai, M., et al. Astrocytes and microglia-targeted danshensu liposomes enhance the therapeutic effects on cerebral ischemia-reperfusion injury. J Control Release. 364, 473-489 (2023).
- Zhang, H., et al. Salvianolic acid a protects RPE cells against oxidative stress through activation of Nrf2/HO-1 signaling. Free Radic Biol Med. 69, 219-228 (2014).
- Wang, R., et al. Danshensu inhibits sars-cov-2 by targeting its main protease as a specific covalent inhibitor and discovery of bifunctional compounds eliciting antiviral and anti-inflammatory activity. Int J Biol Macromol. 257 (Pt 2), 128623(2024).
- Misaghi, S., et al. Structure of the ubiquitin hydrolase UCH-L3 complexed with a suicide substrate. J Biol Chem. 280 (2), 1512-1520 (2005).
- Wing, S. S. Deubiquitinating enzymes--the importance of driving in reverse along the ubiquitin-proteasome pathway. Int J Biochem Cell Biol. 35 (5), 590-605 (2003).
- Samy, M. A., Abd El Fatah, N. M., Yahia, S. E., Arafa, R. K. Friend or foe: UCHL3 mediated carcinogenesis and current approaches in small molecule inhibitors' development. Curr Med Chem. 28 (42), 8782-8799 (2021).
- Hafez, N., Modather El-Awadly, Z., Arafa, R. K. UCH-L3 structure and function: Insights about a promising drug target. Eur J Med Chem. 227, 113970(2022).
- Song, Z., et al. A novel UCHL(3) inhibitor, perifosine, enhances PARP inhibitor cytotoxicity through inhibition of homologous recombination-mediated DNA double strand break repair. Cell Death Dis. 10 (3), 398(2019).
- Hirayama, K., Aoki, S., Nishikawa, K., Matsumoto, T., Wada, K. Identification of novel chemical inhibitors for ubiquitin c-terminal hydrolase-L3 by virtual screening. Bioorg Med Chem. 15 (21), 6810-6818 (2007).
- Nijman, S. M., et al. A genomic and functional inventory of deubiquitinating enzymes. Cell. 123 (5), 773-786 (2005).
- Guo, M., et al. High-throughput screening for amyloid-β binding natural small-molecules based on the combinational use of biolayer interferometry and UHPLC-DAD-Q/TOF-MS/MS. Acta Pharm Sin B. 12 (4), 1723-1739 (2022).
- Chavanieu, A., Pugnière, M. Developments in spr fragment screening. Expert Opin Drug Discov. 11 (5), 489-499 (2016).
- Dai, L., et al. Horizontal cell biology: Monitoring global changes of protein interaction states with the proteome-wide cellular thermal shift assay (CETSA). Annu Rev Biochem. 88, 383-408 (2019).
- Pinzi, L., Rastelli, G. Molecular docking: Shifting paradigms in drug discovery. Int J Mol Sci. 20 (18), 4331(2019).
- Shang, L., et al. Mechanism of Sijunzi decoction in the treatment of colorectal cancer based on network pharmacology and experimental validation. J Ethnopharmacol. 302 (Pt A), 115876(2023).
- Fan, Z., Wang, S., Xu, C., Yang, J., Cui, B. Mechanisms of action of fu fang gang liu liquid in treating condyloma acuminatum by network pharmacology and experimental validation. BMC Complement Med Ther. 23 (1), 128(2023).
- Tirat, A., et al. Synthesis and characterization of fluorescent ubiquitin derivatives as highly sensitive substrates for the deubiquitinating enzymes UCH-L3 and USP-2. Anal Biochem. 343 (2), 244-255 (2005).
- Hassiepen, U., et al. A sensitive fluorescence intensity assay for deubiquitinating proteases using ubiquitin-rhodamine110-glycine as substrate. Anal Biochem. 371 (2), 201-207 (2007).
- Chan, W. C., et al. Accelerating inhibitor discovery for deubiquitinating enzymes. Nat Commun. 14 (1), 686(2023).
- Haj-Yahya, N., et al. Dehydroalanine-based diubiquitin activity probes. Org Lett. 16 (2), 540-543 (2014).
- Li, G., Liang, Q., Gong, P., Tencer, A. H., Zhuang, Z. Activity-based diubiquitin probes for elucidating the linkage specificity of deubiquitinating enzymes. Chem Commun (Camb). 50 (2), 216-218 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados