Research Article
Identificación de genes relacionados con la ferroptosis y la hipoxia en células precursoras de oligodendrocitos derivadas de iPSC de pacientes con esclerosis múltiple
En este artículo
Resumen
Este estudio proporciona nuevos conocimientos sobre las interacciones entre la hipoxia, la ferroptosis y la infiltración inmunitaria en la patogénesis de la esclerosis múltiple (EM) a través del análisis bioinformático. Mediante el empleo del análisis de red de coexpresión génica ponderada (WGCNA) y el análisis de interacción proteína-proteína (PPI), identificamos tres genes centrales fundamentales (ITGB1, ITGB8 y VIM).
Resumen
La esclerosis múltiple (EM) es un trastorno inflamatorio crónico caracterizado por la desmielinización, con una remielinización fallida que conduce a la pérdida progresiva de axones en etapas crónicas. Las células precursoras de oligodendrocitos (OPC) son fundamentales para la remielinización. Estudios recientes sugieren que tanto la hipoxia como la ferroptosis juegan un papel crucial en la diferenciación disfuncional de las OPC. Esta investigación busca identificar genes clave relacionados con la hipoxia y la ferroptosis y las características de infiltración inmune en OPC derivadas de células madre pluripotentes inducidas (iPSC) de pacientes con EM y construir un modelo de diagnóstico centrado en estos genes fundamentales.
Analizamos los datos de expresión génica de los conjuntos de datos de GSE196575 y GSE147315 y comparamos a los pacientes con EM con individuos sanos. Mediante el análisis de redes de coexpresión génica ponderada (WGCNA), identificamos los genes del módulo primario y los genes esenciales asociados con la hipoxia, la ferroptosis y la EM. La puntuación Z de ferroptosis y la puntuación Z de hipoxia calculadas mediante el análisis de variación del conjunto de genes (GSVA) fueron mayores en las OPC derivadas de iPSC de los pacientes con EM que en las del grupo control. Los genes implicados están predominantemente vinculados a la vía PI3K/Akt/mTOR, identificada a través de los análisis de enriquecimiento de la vía Gene Ontology (GO) y Kyoto Encyclopedia of Genes and Genomes (KEGG).
Una red de interacción proteína-proteína (PPI) de genes cruciales reveló 10 genes de eje central (COL4A1, COL4A2, ITGB5, ITGB1, ITGB8, ITGAV, VIM, FLNA, VCL y SPARC). La expresión robusta de ITGB1, ITGB8 y VIM se validó en el conjunto de datos GSE151306, lo que respalda su papel como genes centrales clave. Además, se estableció una red de interacción entre los factores de transcripción (TF) y los genes hub a través de las Relaciones Reguladoras Transcripcionales Desentrañadas por Texto Basado en Oraciones (TRRUST), que identificó cinco TF clave. Los resultados de este estudio podrían ayudar a dilucidar nuevos biomarcadores u dianas terapéuticas para la EM.
Introducción
La esclerosis múltiple (EM) es una enfermedad inflamatoria crónica caracterizada por la desmielinización, que afecta a aproximadamente 2,5 millones de personas en todo el mundo. La mayoría de las personas diagnosticadas con EM presentan un curso de enfermedad remitente-recurrente (RR). Durante la fase recurrente, la inflamación aguda conduce a la pérdida inevitable de mielina y axones. Por el contrario, durante la remisión, las lesiones de desmielinización pueden repararse mediante remielinización, proporcionando soporte trófico a los axones y evitando la pérdida progresiva de los axones1. El fallo de la remielinización se produce en las fases crónicas de la EM y conduce a una degeneración axonal progresiva2.
El proceso de remielinización está críticamente correlacionado con las células precursoras de oligodendrocitos (OPC), lo que implica la proliferación y migración de OPC para diferenciarse en oligodendrocitos maduros (OL), que son las células formadoras de mielina en el sistema nervioso central (SNC)3. En las etapas iniciales de la enfermedad, el número de nuevas OL generadas por las OPC alrededor de las lesiones desmielinizadas está relativamente preservado y puede promover con éxito la remielinización4. Sin embargo, durante los estadios avanzados de la EM, la migración inadecuada y la diferenciación de las OPC conducen a una reducción de las nuevas OL y a una alteración de la remielinización5, lo que conduce a la degeneración del nervio y a la acumulación de discapacidad.
Se han propuesto dos hipótesis para explicar la neurodegeneración en la EM. La hipótesis extrínseca sugiere que la respuesta inmune iniciada por las células T activadas causa desmielinización, así como neurodegeneración6. El modelo intrínseco, sin embargo, sugiere que las anomalías intrínsecas en las OPC7, OL8 y otras células del SNC pueden contribuir a la neurodegeneración. Anteriormente, se consideraba que el modelo intrínseco solo era aplicable en las etapas más avanzadas de la EM, como la EM primaria o secundaria progresiva (EMPP y EMSP). Sin embargo, recientemente se ha observado neurodegeneración independiente de la inflamación o recaída en la EMRR 9,10, lo que sugiere que las anomalías celulares intrínsecas pueden estar involucradas en todas las etapas de la enfermedad, incluida la EMRR.
Además, la ferroptosis, una vía distintiva de muerte celular relacionada con las alteraciones metabólicas de los lípidos mediadas por el hierro, desempeña un papel fundamental en la neurodegeneración. Esta vía implica un desequilibrio de los estados redox intracelulares impulsados por el exceso de hierro, lo que conduce a la acumulación de peróxido lipídico y a la producción de especies reactivas de oxígeno (ROS), lo que en última instancia resulta en la muerte celular oxidativa11. Las enfermedades neurodegenerativas como la enfermedad de Alzheimer, la enfermedad de Parkinson y la enfermedad de Huntington a menudo se originan por el daño oxidativo de las células neuronales, que con frecuencia se desencadena por concentraciones inusualmente altas de hierro dentro de las lesiones. En la EM, el aumento de la vulnerabilidad al daño oxidativo, combinado con la disfunción mitocondrial debido al alto contenido de lípidos y al consumo de oxígeno de las células del SNC, promueve la peroxidación lipídica, un factor crítico en la ferroptosis. Los OL son sensibles a la peroxidación lipídica, una característica esencial de la ferroptosis12. El depósito de hierro cerca de las lesiones inflamatorias espinales13 y la vulnerabilidad de los OL a la peroxidación lipídica14 y a los radicales libres15 ponen de manifiesto la susceptibilidad de la EM a la ferroptosis.
La hipoxia es otro factor crítico en la patogénesis de la EM que contribuye a la pérdida de oligodendrocitos. La evidencia de daño similar a la hipoxia y la generación de ROS y óxido de nitrógeno (NO) en las lesiones agudas de EM indica que tales factores estresantes pueden precipitar la disfunción mitocondrial y los posteriores déficits energéticos16. Este estrés metabólico no solo afecta a los OL, sino que también perjudica a los axones vecinos a través de la interrupción de la transferencia de energía17, ya que los canales mielínicos transmiten energía entre la vaina de mielina y los espacios periaxonales.
Las OPC y las OL humanas primarias del SNC son extremadamente difíciles de acceder en los pacientes con EM. Por lo tanto, las OPC y OL humanas derivadas de células madre pluripotentes inducidas (iPSC) se han convertido en herramientas prometedoras para estudiar los trastornos intrínsecos de la EM. A la luz de los roles cruciales de la ferroptosis y la hipoxia en la patogénesis de la EM y su impacto en el linaje de oligodendrocitos, este estudio empleó el análisis de redes de coexpresión génica ponderada (WGCNA) para extraer información del módulo18 y dilucidar los patrones de expresión génica asociados con estos fenómenos en la EM. Al analizar los coeficientes de correlación entre genes, podemos identificar redes o módulos de coexpresión iguales o similares, lo que arroja luz sobre nuevos biomarcadores o posibles dianas terapéuticas para la EM. Además, al centrarse en los factores de transcripción (TF) que regulan los genes críticos, este estudio proporciona una base para una mayor exploración de los mecanismos y las posibles estrategias de intervención para la EM.
Protocolo
1. Descarga y preprocesamiento de datos
- Descargue los conjuntos de datos GSE196575 y GSE147315 del Gene Expression Omnibus (GEO).
- Combine los dos conjuntos de datos y elimine los efectos de lote, utilizando una plataforma de análisis en línea (consulte la tabla de materiales).
- Elija el módulo Expresión | módulo de combinación de datos. Cargue los dos archivos de matriz de expresión como archivos de entrada y haga clic en Ejecutar para generar automáticamente la matriz combinada.
- Cree un archivo de anotación de muestra como una hoja de cálculo.
- Elija el módulo Expresión | Eliminar el módulo de efecto por lotes. Ingrese el archivo de anotación y la matriz combinada y haga clic en Ejecutar para crear la matriz combinada con el efecto de lote eliminado (matriz combinada 2).
- Normalice los datos de expresión mediante las características de combinación de conjuntos de datos, efecto por lotes y normalización disponibles en una plataforma de análisis en línea. Elija módulo de expresión y módulo de normalización. Entrada de la matriz combinada 2 creada en el paso 1.2.3. Haga clic en el botón Ejecutar . (ver la Tabla de Materiales).
- Genere el archivo de matriz de expresión después de ejecutar todo el proceso descrito anteriormente. Extraiga la información de agrupación de acuerdo con la intervención proporcionada en el sitio web de GEO. Introduzca la información anterior en un documento con formato ".txt" para crear el archivo de información de agrupación para el conjunto de datos combinado.
2. Análisis de genes expresados diferenciales
- Realice un análisis de expresión génica diferencial utilizando el paquete limma en R. Ingrese el archivo de matriz de expresión y el archivo de información de agrupación. Establezca los criterios de filtrado en |logFC| = 1 y p < 0,05 para identificar genes expresados diferencialmente (DEG) relacionados con la esclerosis múltiple.
3. Análisis de enriquecimiento funcional (GO y KEGG)
- Realizar análisis de enriquecimiento funcional para los DEG a través de los módulos GO y KEGG en plataformas en línea.
- El conjunto del organismo modelo se estableció en Homo sapiens, con la versión Ensemble_109 como archivo genético de fondo y genes como tipo de datos.
- Evite los términos amplios de GO; refinar el análisis con ToppFun. Aplique filtros para limitar los términos a 500 y 1.000 genes asociados.
NOTA: Los resultados del filtro de 500 genes se presentan en el manuscrito principal. Los resultados completos de GO están disponibles en la Figura Suplementaria S2. La plataforma KEGG en línea se muestra en la Figura Complementaria S3.
4. Análisis de variación del conjunto de genes para ferroptosis e hipoxia (GSVA)
- Recuperar un total de 538 genes relacionados con la ferroptosis de FerrDb y 200 genes relacionados con la hipoxia de MsigDB.
- Cree archivos en formato GMT para ambos conjuntos de genes.
- Utilice el módulo GSVA para calcular la puntuación Z de ferroptosis y la puntuación Z de hipoxia para cada muestra del conjunto de datos combinado, y utilice el archivo de matriz de expresión, el archivo de información de agrupación y los archivos GMT como entradas.
- Compare las puntuaciones Z de diferentes grupos (Figura complementaria S4).
5. Análisis de red de coexpresión génica ponderada (WGCNA)
- Extraiga la puntuación Z de ferroptosis e hipoxia de cada muestra calculada en el paso 4.4. Utilice las puntuaciones Z anteriores de ferroptosis e hipoxia como archivos de rasgos junto con los archivos de la matriz de expresión para identificar módulos genéticos clave relacionados con la ferroptosis y la hipoxia.
- Utilice el módulo WGCNA y establezca R2 = 0,9 y el umbral suave β = 18 para construir una red de coexpresión génica ponderada. Concéntrese en los módulos significativamente asociados tanto con la ferroptosis como con la hipoxia (Figura complementaria S4).
6. Identificación de genes diferencialmente expresados relacionados con ferroptosis e hipoxia en pacientes con EM
- Intersección de los DEG relacionados con la enfermedad con módulos genéticos relacionados con la ferroptosis y la hipoxia a través de una herramienta de diagrama de Venn en una plataforma de análisis en línea (Figura suplementaria S3). Identificar los genes asociados tanto con la ferroptosis como con la hipoxia en la EM.
7. Análisis de redes de interacción proteína-proteína (PPI)
- Introduzca los genes intersectados en el módulo de proteínas múltiples de la base de datos STRING.
- Establezca el organismo en Homo sapiens y haga clic en Buscar.
- Utilice la opción CONTINUAR en el centro de la página web para generar la red PPI.
- Exporte la información de la red de interacción en formato TSV e impórtela en el software de análisis de redes bioinformáticas (Figura complementaria S5).
- Utilice el complemento Cytohubba para identificar los 10 genes principales del centro sobre la base del algoritmo MCC.
8. Validación con el conjunto de datos GSE151306
- Descargue el conjunto de datos GSE151306 de GEO.
- Use el paquete limma en el software R studio (los códigos de lenguaje R se proporcionan en GitHub: https://github.com/Drxiazhang/Identification-of-Ferroptosis--and-Hypoxia-Related-Genes-in-iPSC-Derived-OPCs-from-MS) para validar la expresión de los genes hub en diferentes grupos e introduzca el archivo de matriz de expresión, el archivo de agrupación del conjunto de validación y la lista de genes hub.
9. Trazado de la curva ROC para los genes hub
- Utilice el paquete pROC en R para trazar curvas ROC para los genes hub expresados diferencialmente.
- Valide el valor de diagnóstico de estos genes hub en el conjunto de datos de validación (los códigos de lenguaje R se proporcionan en GitHub: https://github.com/Drxiazhang/Identification-of-Ferroptosis--and-Hypoxia-Related-Genes-in-iPSC-Derived-OPCs-from-MS).
10. Predicción de redes reguladoras de genes de factor de transcripción-hub
- Acceda a la base de datos de TRRUST v2 en https://www.grnpedia.org/trrust/.
- Utilice el módulo Buscar reguladores clave para consultar genes para identificar los factores de transcripción que regulan los 10 genes centrales.
- Conjunto Humano para la especie. Introduzca los factores de transcripción y los genes hub en el módulo de proteínas múltiples de STRING, configure el Homo sapiens para los organismos y haga clic en CONTINUAR para generar una red reguladora.
Resultados
El conjunto de datos combinado, que consta de cuatro individuos sanos como controles y nueve personas con EM (PwMS), se analizó y luego se validó en otro conjunto de datos de cuatro PwMS y cuatro controles sanos. El protocolo de análisis se muestra en la Figura 1, y la información detallada de todas las muestras se enumera en la Tabla Complementaria S1. A través del análisis, se identificaron 706 genes expresados diferencialmente (DEGs, p < 0,01), de los cuales 378 genes estaban regulados al alza y 328 genes a la baja en los pacientes con EM en comparación con los controles (Figura 2). El análisis de enriquecimiento funcional reveló la participación significativa de estos DEG en diversos procesos biológicos. En particular, los términos GO más enriquecidos refinados por 500 genes incluyeron la adhesión focal, la unión célula-sustrato, la vía de señalización mediada por integrinas, la unión de integrinas y la unión de cadherina (Figura 3A-C). El análisis de la vía KEGG reveló además que estos genes están predominantemente involucrados en la vía de señalización PI3K-Akt y la vía de señalización Hippo (Figura 3D), que son críticas en los procesos celulares relevantes para la patogénesis de la EM.
Las puntuaciones Z de ferroptosis e hipoxia, calculadas a través de GSVA, fueron significativamente mayores en las células precursoras de oligodendrocitos (OPC) derivadas de iPSC de pacientes con EM que en las de controles sanos (p = 0,0028 y 0,075, respectivamente, Figura 4), lo que indica el papel fundamental de estas vías en esta enfermedad. Este hallazgo fue respaldado por el WGCNA, que identificó dos módulos genéticos clave (Figura 5A). Entre estos, el módulo turquesa, que comprende 3.671 genes, se asoció significativamente tanto con la ferroptosis como con la hipoxia (Figura 5B). Es importante destacar que se identificaron 71 genes en la intersección de los genes relacionados con la enfermedad expresados diferencialmente y los del módulo WGCNA, lo que sugiere una fuerte correlación con la ferroptosis (beta=0,94, p=2e-06) y la hipoxia (beta= 0,92, p=7e-06) en la EM (Figura 6).
Una investigación más profunda en la red PPI reveló 10 genes de eje central, a saber, COL4A1, COL4A2, ITGB5, ITGB1, ITGB8, ITGAV, VIM, FLNA, VCL y SPARC (Figura 7). Estos genes centrales se destacaron por su papel crucial en la mediación de procesos celulares relacionados con la ferroptosis y la hipoxia.
Además, validamos el conjunto de datos de validación (GSE151306). Los resultados sugirieron que tres genes hub (ITGAV, ITGB8 y VIM) estaban regulados al alza en PwMS (Figura 8), lo que era consistente con los hallazgos en el conjunto de datos fusionado (Figura suplementaria S1). Entre ellos, ITGB8 fue el gen más significativamente regulado al alza. Estos hallazgos sugieren que ITGB8, un miembro de la familia de las integrinas, puede estar críticamente involucrado en la patogénesis de la EM, particularmente en relación con los mecanismos de ferroptosis e hipoxia. Además, el análisis de la curva de características operativas del receptor (ROC) confirmó que ITGAV, ITGB8 y VIM tienen buena sensibilidad y especificidad para el diagnóstico de enfermedades (AUC=58,3%, 100% y 91,7%, respectivamente; Figura 9A-C), con ITGB8 mostrando un AUC del 100%, lo que demuestra su potencial como un biomarcador robusto para la EM.
Finalmente, se estableció una red reguladora de genes de factor de transcripción y se identificaron tres factores de transcripción clave: HOXD3, SP1 y VHL, como reguladores de los genes hub (Figura 10). Estos factores de transcripción pueden desempeñar un papel esencial en la regulación de la expresión génica en respuesta a la hipoxia y la ferroptosis, lo que pone de manifiesto su posible implicación en la progresión de la EM.
Video 1: Vista coronal obtenida con un transductor de baja frecuencia centrado en el hemidiafragma derecho que muestra una excursión diafragmática cualitativamente normal durante la respiración en capacidad vital. Haga clic aquí para descargar este video.

Figura 1: Diagrama de flujo que muestra el diseño del estudio. Abreviaturas: EM = esclerosis múltiple; GSVA = Análisis de Variación del Conjunto de Genes; WGCNA = análisis de redes de coexpresión génica ponderada; KEGG = Enciclopedia de Genes y Genomas de Kioto; GO = Ontología de genes; PPI = interacción proteína-proteína; ROC = Característica de funcionamiento del receptor. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Gráfico volcánico que ilustra los genes expresados diferencialmente entre el grupo de control sano y el grupo de EM. Abreviaturas: FC = criterios de filtrado; PEG3 = gen 3 expresado por el padre; NEBL = nebuleta; CRABP1 = proteína celular de unión al ácido retinoico 1. Haga clic aquí para ver una versión más grande de esta figura.
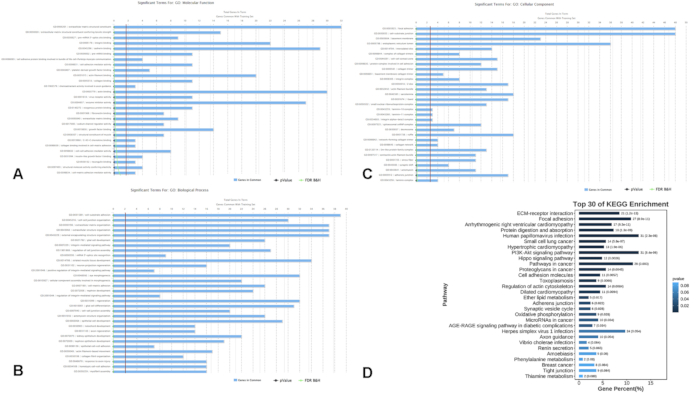
Figura 3: Análisis de enriquecimiento funcional de genes expresados diferencialmente. (A-C) Categorías de ontología génica refinadas mediante el filtrado de 500 genes, que incluyen (A) función molecular, (B) proceso biológico y (C) componente celular. (D) Análisis de enriquecimiento de la vía KEGG que indica vías enriquecidas entre los DEG. Abreviaturas: DEGs = genes expresados diferencialmente; GO = Ontología de genes; MF = función molecular; BP = proceso biológico; CC = componente celular; KEGG = Enciclopedia de Genes y Genomas de Kioto. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Diagrama de caja que compara las puntuaciones Z de ferroptosis e hipoxia entre el grupo de control sano y el grupo de EM. Las puntuaciones Z de ferroptosis e hipoxia se calcularon mediante GSVA. El grupo de control se muestra en azul y el grupo de EM se muestra en rojo. Las comparaciones estadísticas se realizaron mediante la prueba de Wilcoxon. ** indica valores de p entre 0 y 0,01. Abreviaturas: EM = esclerosis múltiple; HC = control sano; GSVA = Análisis de Variación del Conjunto de Genes. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Resultados del análisis de redes de coexpresión génica ponderada. (A) Dendrograma de genes categorizados en módulos. (B) Correlaciones entre módulos y rasgos relacionados con la hipoxia y la ferroptosis. Abreviatura: ADJ = ajustado. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Diagrama de Venn que revela 71 genes que se cruzan. El diagrama ilustra la superposición de los DEG de los pacientes con esclerosis múltiple con los genes de los módulos relacionados con la hipoxia y la ferroptosis. Abreviaturas: WGCNA = análisis de red de coexpresión génica ponderada; DEGs = genes expresados diferencialmente. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Diagrama de red de interacciones proteína-proteína entre los 10 genes hub identificados. Abreviaturas: VCL = vinculina; FLNA = filamina A; ITGAV = Subunidad de Integrina Alfa V; GOL4A2 = Cadena de colágeno tipo IV alfa 2; COL4A1 = Cadena de colágeno tipo IV alfa 1; ITGB5 = subunidad de integrina beta 5; VIM = Vimentina; SPARC = proteína secretada ácida y rica en cisteína; ITGB8 = subunidad de integrina beta 8; ITGB1 = Subunidad de integrina beta 1. Haga clic aquí para ver una versión más grande de esta figura.

Figura 8: Gráfico de barras que muestra los niveles de expresión de los genes hub en el conjunto de datos de validación (GSE151306). * indica valores de p entre 0,01 y 0,05, y # indica valores de p entre 0,05 y 0,2. ns indica valores de p entre 0,2 y 1. Abreviaturas: HC: controles sanos; EM: esclerosis múltiple. Haga clic aquí para ver una versión más grande de esta figura.
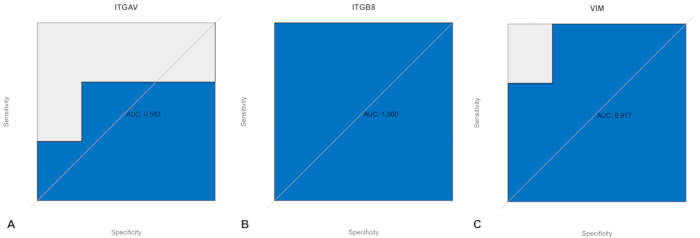
Figura 9: Curvas ROC para evaluar la sensibilidad diagnóstica y la especificidad de los genes hub expresados diferencialmente en GSE151306. (A) Curva ROC para ITGAV. (B) Curva ROC para ITGB8. (C) Curva ROC para VIM. Abreviaturas: ROC = Receiver Operating Characteristic; ITGAV = subunidad de integrina alfa V; ITGB8 = subunidad de integrina beta 8; VIM = vimentina. Haga clic aquí para ver una versión más grande de esta figura.

Figura 10: Diagrama de la red reguladora de genes del factor de transcripción -hub. Los triángulos representan factores de transcripción; Los cuadrados representan los genes centrales. Abreviaturas: ITGB1 = integrina subunidad beta 1; ITGAV = subunidad de integrina alfa V; VIM = Vimentina; SPARC = proteína secretada ácida y rica en cisteína; COL4A2 = colágeno tipo IV cadena alfa 2; HOXD3 = homeobox D3; VHL = supresor tumoral de von Hippel-Lindau; SP1 = Proteína de especificidad 1. Haga clic aquí para ver una versión más grande de esta figura.

Figura 11: El impacto de la integrina β1 en la vía PI3K-Akt. La forma de extensión con una integrina β1 de cabeza abierta (estado activo, alta afinidad) podría formar "adhesión focal" y "complejos focales" e interactuar con la quinasa FAK y Scr. El complejo FAK/Src activo recluta moléculas adaptadoras para activar la vía PI3K-Akt. Esta cifra fue adaptada de Yan y Cui37. Abreviaturas: PI3K = fosfatidilinositida-3-quinasa; FAK = quinasa de adhesión focal. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria S1: Las tendencias de expresión de los genes clave en el conjunto de datos fusionado. Para las pruebas estadísticas se utilizó la función Wilcox.test del paquete R "limma". "*" indica valores p entre 0,01 y 0,05, y "#" indica valores p entre 0,05 y 0,2. Haga clic aquí para descargar esta figura.
Figura suplementaria S2: Diseño original del análisis de Gene Ontology de genes expresados diferencialmente. Haga clic aquí para descargar esta figura.
Figura complementaria S3: Página de inicio de la plataforma de análisis en línea Omicshare. Este sitio web es un centro bioinformático chino en el que se realizó KEGG. Haga clic aquí para descargar esta figura.
Figura complementaria S4: Página de inicio de la plataforma de análisis en línea de Biowinford en la que se realizó WGCNA. Haga clic aquí para descargar esta figura.
Figura suplementaria S5: Página de inicio de la base de datos STRING en la que se realizó el PPI. Haga clic aquí para descargar esta figura.
Tabla complementaria S1: Características de las muestras y conjuntos de datos agrupados y analizados. Haga clic aquí para descargar esta tabla.
Discusión
Dado su papel fundamental en el proceso de remielinización, se ha determinado desde hace mucho tiempo que la migración, la diferenciación y la muerte de las OPC son factores cruciales en la patogénesis de la EM y en las dianas terapéuticas de la EM. Se ha observado una degeneración nerviosa progresiva independiente de la inflamación en los tres tipos de EM19, y se ha observado una pérdida pronunciada de oligodendrocitos en el centro de las lesiones desmielinizadas20, lo que sugiere que los trastornos primarios de las células del linaje de oligodendrocitos podrían acelerar la progresión de la EM.
En este estudio, realizamos un análisis bioinformático de secuencias de ARNm dirigidas centrándonos en genes asociados con ferroptosis e hipoxia en OPC inducidas por iPSC de PwMS y controles sanos a través de conjuntos de datos combinados de cuatro controles sanos y nueve PwMS. Descubrimos 706 DEGs, 378 de los cuales estaban regulados al alza y 328 de los cuales estaban regulados a la baja. Además, identificamos los genes relacionados con la ferroptosis y la hipoxia y comparamos su expresión en las OPC inducidas por iPSC de pacientes con EM y controles sanos mediante el cálculo de la puntuación Z de cada muestra. Las puntuaciones Z de ferroptosis e hipoxia fueron significativamente mayores en las PwMS que en los controles sanos.
La hipoxia desempeña un papel importante en la oligodendrogliopatía en la EM. Los estudios anatomopatológicos han demostrado la oligodendrogliopatía distal en las lesiones de EM tipo III que se asemejan al ictus isquémico de la sustancia blanca16 , así como las lesiones hipóxicas junto con niveles elevados de ROS y NO en las lesiones agudas de EM21. La interrupción del complejo IV de la cadena respiratoria mitocondrial perjudica la formación y viabilidad de las OPC y los oligodendrocitos maduros15, aunque los mecanismos exactos que subyacen a este deterioro aún no se han dilucidado.
Además, se ha descubierto que la ferroptosis, una forma de muerte celular programada dependiente del hierro, media la pérdida de oligodendrocitos y la progresión de la desmielinización en modelos animales de EM inducida por cuprizona22. La secuenciación de una sola célula ha revelado una disminución en la expresión génica de antiferroptosis en OPC y OL en el tejido cerebral de PwMS23. En la fase crónica de la EM, la deficiencia de hierro en los oligodendrocitos puede interrumpir la remielinización y comprometer la integridad axonal en la sustancia blanca, lo que resulta en la progresión de la enfermedad24. Además, la secuenciación de snRNA de las lesiones de EM en la sustancia blanca sugiere que la ferroptosis es principalmente activa en las lesiones de la sustancia blanca de los pacientes con EM y se asocia con la activación del sistema de fagocitos25. En ratones experimentales con encefalomielitis autoinmune (EAE), los niveles de GPX4, un importante regulador de la ferroptosis, se reducen26. La progresión de la enfermedad puede acelerarse mediante la ferroptosis inducida por ACSL4, que mejora la señalización del receptor de células T (TCR). El bloqueo de la ferroptosis y la ACSL4 ralentiza significativamente la progresión de la discapacidad en EAE27.
La ferroptosis está involucrada en el deterioro cognitivo causado por la hipoxia en modelos de rata28. La hipoxia y el factor 1α inducible por hipoxia promueven la proliferación y migración de los ganglios de la raíz dorsal29. Por lo tanto, la ferroptosis y la hipoxia pueden actuar sinérgicamente en la patogénesis de la EM. A través de WGCNA, nuestro estudio identificó dos módulos significativos; el módulo turquesa, en particular, se correlacionó fuertemente con el estado de EM, hipoxia y ferroptosis. Sobre la base de los resultados de los WGCNA y DEGs, identificamos 71 genes clave que se correlacionaron simultáneamente con ferroptosis, hipoxia y EM.
Los análisis de enriquecimiento funcional de GO y KEGG de los 706 DEG revelaron que la vía de señalización PI3K-Akt es una vía crucial en el desarrollo de la EM. La inhibición de CXCR2 mejora la diferenciación de OPC y promueve la remielinización en modelos de ratón con EM mediante la activación de la señalización PI3K/AKT/mTOR30. Un desequilibrio en el suministro de oxígeno podría afectar a las vías de señalización MAPK/PI3K-Akt, desencadenando mecanismos clave implicados en el estrés oxidativo31.
El análisis de PPI reveló que 10 genes centrales, a saber, COL4A1, COL4A2, ITGB5, ITGB1, ITGB8, ITGAV, VIM, FLNA, VCL y SPARC, se correlacionaron simultáneamente con hipoxia, ferroptosis y EM, con una sobreexpresión notable en los pacientes con EM en comparación con los controles. Estos genes se validaron aún más en GSE151306, una base de datos de líneas OPC inducidas por iPSC derivadas de pacientes con SPMS. Descubrimos que ITGAV, ITGB8 y VIM también estaban regulados al alza en el conjunto de validación. En particular, ITGB8, parte de la familia de integrinas, se sobreexpresó significativamente en MS, lo que fue consistente con los conjuntos de prueba combinados GSE147315 y GSE196575.
El gen ITGB8 es un miembro de la familia de las integrinas (ITG) y codifica la integrina beta 8, que se ha descrito previamente como un regulador del crecimiento tumoral y la metástasis32,33. La integrina beta 8 está fuertemente expresada por las OPC y los oligodendrocitos maduros. Un estudio demostró que la osteopontina protege a las OPC de la apoptosis inducida por H2O2 mediante la regulación positiva de las integrinas, incluida la ITGB834. Estos hallazgos indican que ITGB8 desempeña un papel en la supervivencia celular en condiciones de estrés oxidativo. En la esclerosis múltiple, donde el estrés oxidativo es prevalente, ITGB8 puede ayudar a las OPC a sobrevivir y diferenciarse en oligodendrocitos para aumentar la remielinización.
ITGAV codifica la subunidad de integrina alfa V, que regula la angiogénesis y la progresión del cáncer. ITGAV, en combinación con beta 5 (formando la integrina αvβ5), media la activación microglial en respuesta a la deposición de fibronectina y vitronectina, que están elevadas en las lesiones de EM. En el modelo de rata EM de EAE, la integrina avβ5 activó la microglía, lo que condujo a un aumento de la expresión de la metaloproteinasa-9 de la matriz (MMP-9), una enzima que degrada la matriz extracelular y las proteínas de mielina, contribuyendo a la desmielinización35.
En particular, se descubrió que uno de los genes centrales que se expresó diferencialmente en nuestro conjunto combinado, ITGB1, estaba regulado al alza en las células T CD4+ extraídas del líquido cefalorraquídeo de los pacientes con EM36. La integrina β1 tuvo un efecto notable en la vía de señalización PI3K-Akt, lo que es consistente con nuestros hallazgos (Figura 11)37. Además, se detectó un aumento de la expresión de β1-integrina en las células Th17 en pacientes con EM en comparación con los controles sanos. La activación de la β1-integrina en las células Th17 puede aumentar significativamente los niveles de glutamato y promover la neuroinflamación38.
Realizamos un análisis de la curva ROC para evaluar el potencial de la expresión de ITGAV, ITGB8 y VIM en OPC como biomarcadores para la predicción del riesgo de EM. El AUC de ITGB8 fue del 100%, seguido del 91,7% para VIM y el 58,3% para ITGAV. Actualmente, no existen estudios que reporten la asociación de la expresión de ITGB8 en oligodendrocitos con la EM. Se deben realizar más estudios para explorar el uso de ITGB8 como biomarcador para la predicción de la EM.
Finalmente, construimos una red de genes de factor de transcripción e identificamos tres factores de transcripción clave: HOXD3, SP1 y VHL. SP1 es un factor de transcripción que se une a muchos promotores. Se ha demostrado que SP1, conocida por su amplia unión a promotores, regula la expresión de Prdx6, inhibe las ROS y elimina la ferroptosis en entornos con alto contenido de glucosa39. La expresión de SP1 también se ha relacionado con la actividad del factor de necrosis tumoral (TNF), que desempeña un papel crucial en el desarrollo de RRMS40. El receptor de andrógenos (AR), otro factor de transcripción identificado, es activado por hormonas esteroides y se detecta en los astrocitos dentro de las zonas de remielinización, destacando su potencial implicación en la función de los oligodendrocitos41.
Las vías inmunitarias y las células desempeñan un papel importante en la patogénesis y la neurodegeneración de la EM y posiblemente se asocian con la ferroptosis, la hipoxia y las OPC. Las observaciones de ratones intoxicados por cuprizona, así como las lesiones progresivas de EM, han mostrado altas tasas de prevalencia y proliferación de linfocitos T CD8+ con sobreexpresión de gránulos citotóxicos42. Los linfocitos T suficientemente activados son capaces de interactuar con los oligodendrocitos presentadores de antígenos, lo que puede provocar la destrucción de oligodendrocitos mediada por linfocitos T CD8+ . En el microambiente tumoral, el interferón-γ secretado por las células T CD8+ reduce la expresión del intercambiador de cisteína y glutamato (xCT), lo que mejora la ferroptosis43. Además, se ha sugerido que la ferroptosis activa las células T a través de la señalización del receptor de células T en modelos EAE24.
La investigación actual sobre las OPC es significativamente limitada debido a la escasez de estas células. Esta limitación se puede abordar parcialmente mediante el uso de OPC inducidas por iPSC. Sin embargo, la mayoría de los estudios sobre las OPC inducidas por iPSC involucran tamaños de muestra pequeños, generalmente menos de 10 donantes, debido al proceso largo y costoso de la inducción celular. Nuestro estudio proporciona resultados más robustos al integrar muestras de múltiples estudios.
Refinamos aún más la canalización de bioinformática dividiendo el conjunto de datos disponible en conjuntos de entrenamiento y validación. Este enfoque minimiza los posibles sesgos inducidos por las diferencias en los métodos de inducción celular y las técnicas de secuenciación entre los laboratorios. La mayoría de los estudios existentes sobre la esclerosis múltiple se centran en la neurodegeneración provocada por la inflamación o emplean análisis aislados de la ferroptosis. Los análisis tradicionales de expresión génica son útiles para identificar genes individuales expresados diferencialmente, pero a menudo no revelan las complejas redes de interacción y los patrones de coexpresión asociados con la progresión de la EM. Por el contrario, nuestra metodología aprovecha WGCNA para construir una red de coexpresión génica, lo que permite la identificación de módulos génicos altamente correlacionados tanto con la ferroptosis como con la hipoxia. Esto proporciona una comprensión a nivel de sistema de cómo interactúan estas vías dentro de la patología de la EM, que no se puede lograr solo con los métodos estándar de expresión diferencial.
Este método se puede aplicar a nuevos tratamientos para la EM mediante la identificación de dianas génicas para terapias destinadas a reducir la neurodegeneración. También podría utilizarse en otras enfermedades neurodegenerativas que implican ferroptosis e hipoxia, como la enfermedad de Parkinson o la enfermedad de Alzheimer, para desarrollar intervenciones personalizadas y predecir respuestas terapéuticas.
Nuestra investigación tiene varias limitaciones. En primer lugar, es necesaria una mayor validación in vivo e in vitro . En segundo lugar, nuestros resultados provienen de dos bases de datos y se validaron en una base de datos que cubre un número limitado de pacientes. Se deben recolectar cohortes más grandes para una mayor exploración. En tercer lugar, teniendo en cuenta la dificultad de acceso a las OPC humanas, la base de datos que adoptamos se basó en OPC inducidas por iPSC, que pueden no parecerse completamente al tejido humano. En el futuro se deben llevar a cabo más estudios centrados en investigar el impacto de la hipoxia y la ferroptosis en la diferenciación y muerte celular de las células del linaje de oligodendrocitos en la EM.
Otra limitación de este estudio es la imposibilidad de analizar las interacciones de las células inmunitarias debido al uso de OPCs derivadas de iPSCs, que carecen de las células inmunitarias presentes en las lesiones de EM. Si bien las células inmunitarias son cruciales en la patogénesis de la EM, esta limitación refleja los desafíos del uso de cultivos derivados de iPSC. Los estudios futuros deberían centrarse en la diafonía entre OPC y células inmunitarias a través de sistemas de cocultivo u organoides, que pueden modelar mejor las interacciones celulares en el entorno de la EM.
Divulgaciones
Los autores no tienen intereses contrapuestos que declarar que sean relevantes para el contenido de este artículo.
Agradecimientos
Este estudio fue apoyado y financiado por el Fondo Nacional de Investigación Clínica de Hospitales de Alto Nivel (2022-PUMCH-B-103). Los autores desean agradecer a la Dra. Shuang Song, del Departamento de Bioestadística de la Escuela de Salud Pública T. H. Chan de la Universidad de Harvard, por sus valiosos consejos y orientación en la etapa de revisión de este artículo.
Materiales
| Name | Company | Catalog Number | Comments |
| BioInfoTools | / | online analysis website http://biowinford.site:3838/patrick_wang87/ | |
| Cytoscape | / | bioinformatics network analysis software | |
| GSE196575,GSE147315 and GSE151306 | / | RNA-seq from GEO dataset | |
| Omicshare | GENE DENOVO | online analysis tools https://www.omicshare.com/tools/Home/Soft/getsoft | |
| R-studio | RStudio, Inc | R integrated development environment software |
Referencias
- Fünfschilling, U., et al. Glycolytic oligodendrocytes maintain myelin and long-term axonal integrity. Nature. 485 (7399), 517-521 (2012).
- Nishiyama, A., et al. Polydendrocytes (NG2 cells): multifunctional cells with lineage plasticity. Nat Rev Neurosci. 10 (1), 9-22 (2009).
- Franklin, R. J. M., Ffrench-Constant, C. Remyelination in the CNS: from biology to therapy. Nat Rev Neurosci. 9 (11), 839-855 (2008).
- Lucchinetti, C., et al. A quantitative analysis of oligodendrocytes in multiple sclerosis lesions. A study of 113 cases. Brain. 122 (Pt 12), 2279-2295 (1999).
- Patrikios, P., et al. Remyelination is extensive in a subset of multiple sclerosis patients. Brain. 129 (Pt 12), 3165-3172 (2006).
- Starost, L., et al. Extrinsic immune cell-derived, but not intrinsic oligodendroglial factors contribute to oligodendroglial differentiation block in multiple sclerosis. Acta Neuropathol. 140 (5), 715-736 (2020).
- Niu, J., et al. Aberrant oligodendroglial-vascular interactions disrupt the blood-brain barrier, triggering CNS inflammation. Nat Neurosci. 22 (5), 709-718 (2019).
- Plastini, M. J., et al. Transcriptional abnormalities in induced pluripotent stem cell-derived oligodendrocytes of individuals with primary progressive multiple sclerosis. Front Cell Neurosci. 16, 972144(2022).
- Pisa, M., et al. Subclinical anterior optic pathway involvement in early multiple sclerosis and clinically isolated syndromes. Brain. 144 (3), 848-862 (2021).
- Vidal-Jordana, A., et al. Optical coherence tomography measures correlate with brain and spinal cord atrophy and multiple sclerosis disease-related disability. Eur J Neurol. 27 (11), 2225-2232 (2020).
- Stockwell, B. R. Ferroptosis turns 10: Emerging mechanisms, physiological functions, and therapeutic applications. Cell. 185 (14), 2401-2421 (2022).
- Thamizhoviya, G., Vanisree, A. J. Enriched environment enhances the myelin regulatory factor by mTOR signaling and protects the myelin membrane against oxidative damage in rats exposed to chronic immobilization stress. Neurochem Res. 46 (12), 3314-3324 (2021).
- Williams, R., et al. Pathogenic implications of iron accumulation in multiple sclerosis. J Neurochem. 120 (1), 7-25 (2012).
- Stojkovic, L., et al. Targeted RNAseq revealed the gene expression signature of ferroptosis-related processes associated with disease severity in patients with multiple sclerosis. Int J Mol Sci. 25 (5), 3016(2024).
- Ziabreva, I., et al. Injury and differentiation following inhibition of mitochondrial respiratory chain complex IV in rat oligodendrocytes. Glia. 58 (15), 1827-1837 (2010).
- Aboul-Enein, F., et al. Preferential loss of myelin-associated glycoprotein reflects hypoxia-like white matter damage in stroke and inflammatory brain diseases. J Neuropathol Exp Neurol. 62 (1), 25-33 (2003).
- Halder, S. K., Milner, R. Hypoxia in multiple sclerosis; is it the chicken or the egg. Brain. 144 (2), 402-410 (2021).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Pisa, M., et al. Subclinical neurodegeneration in multiple sclerosis and neuromyelitis optica spectrum disorder revealed by optical coherence tomography. Mult Scler. 26 (10), 1197-1206 (2020).
- Lucchinetti, C., et al. Heterogeneity of multiple sclerosis lesions: implications for the pathogenesis of demyelination. Ann Neurol. 47 (6), 707-717 (2000).
- Aboul-Enein, F., Lassmann, H. Mitochondrial damage and histotoxic hypoxia: a pathway of tissue injury in inflammatory brain disease. Acta Neuropathol. 109 (1), 49-55 (2005).
- Jhelum, P., et al. Ferroptosis mediates cuprizone-induced loss of oligodendrocytes and demyelination. J Neurosci. 40 (48), 9327-9341 (2020).
- Zhang, D., et al. Evidence of pyroptosis and ferroptosis extensively involved in autoimmune diseases at the single-cell transcriptome level. J Transl Med. 20 (1), 363(2022).
- Luoqian, J., et al. Ferroptosis promotes T-cell activation-induced neurodegeneration in multiple sclerosis. Cell Mol Immunol. 19 (8), 913-924 (2022).
- Wu, T., et al. Role of ferroptosis in neuroimmunity and neurodegeneration in multiple sclerosis revealed by multi-omics data. J Cell Mol Med. 28 (10), e18396(2024).
- Zeng, L., et al. Advances in research on immunocyte iron metabolism, ferroptosis, and their regulatory roles in autoimmune and autoinflammatory diseases. Cell Death Dis. 15 (7), 481(2024).
- Neel, D. V., et al. Catching a killer: Mechanisms of programmed cell death and immune activation in Amyotrophic Lateral Sclerosis. Immunol Rev. 311 (1), 130-150 (2022).
- Liu, L., et al. Ferritinophagy-mediated hippocampus ferroptosis is involved in cognitive impairment in immature rats induced by hypoxia combined with propofol. Neurochem Res. 49 (7), 1703-1719 (2024).
- An, S., et al. HIF-1α induced by hypoxia promotes peripheral nerve injury recovery through regulating ferroptosis in DRG neuron. Mol Neurobiol. 61 (9), 6300-6311 (2024).
- Wang, L., et al. CXCR2 antagonism promotes oligodendrocyte precursor cell differentiation and enhances remyelination in a mouse model of multiple sclerosis. Neurobiol Dis. 134, 104630(2020).
- Merelli, A., et al. Understanding the role of hypoxia inducible factor during neurodegeneration for new therapeutics opportunities. Curr Neuropharmacol. 16 (10), 1484-1498 (2018).
- Huang, L., et al. miR19b-3p promotes the growth and metastasis of colorectal cancer via directly targeting ITGB8. Am J Cancer Res. 7 (10), 1996-2008 (2017).
- Li, J., et al. Exosomal circDNER enhances paclitaxel resistance and tumorigenicity of lung cancer via targeting miR-139-5p/ITGB8. Thorac Cancer. 13 (9), 1381-1390 (2022).
- Mazaheri, N., et al. Ameliorating effect of osteopontin on H2O2-induced apoptosis of human oligodendrocyte progenitor cells. Cell Mol Neurobiol. 38 (4), 891-899 (2018).
- Milner, R., et al. Fibronectin- and vitronectin-induced microglial activation and matrix metalloproteinase-9 expression is mediated by integrins alpha5beta1 and alphavbeta5. J Immunol. 178 (12), 8158-8167 (2007).
- Hrastelj, J., et al. CSF-resident CD4+ T-cells display a distinct gene expression profile with relevance to immune surveillance and multiple sclerosis. Brain Commun. 3 (3), fcab155(2021).
- Yan, L., Cui, Z. Integrin β1 and the repair after nervous system injury. Eur Neurol. 86 (1), 2-12 (2023).
- Birkner, K., et al. β1-Integrin- and KV1.3 channel-dependent signaling stimulates glutamate release from Th17 cells. J Clin Invest. 130 (2), 715-732 (2020).
- Zhang, Q., et al. Sp1-mediated upregulation of Prdx6 expression prevents podocyte injury in diabetic nephropathy via mitigation of oxidative stress and ferroptosis. Life Sci. 278, 119529(2021).
- Hadi, N., et al. Study of the correlation between miR-106a, miR-125b, and miR-330 on multiple sclerosis patients by targeting TNFSF4 and SP1 in NF-кb/TNF-α pathway: A case-control study. Cell J. 24 (7), 403-409 (2022).
- Asbelaoui, N., et al. Interplay between androgen and CXCR4 chemokine signaling in myelin repair. Acta Neuropathol Commun. 12 (1), 18(2024).
- Kaddatz, H., et al. Cuprizone-induced demyelination triggers a CD8-pronounced T cell recruitment. Glia. 69 (4), 925-942 (2021).
- Wang, W., et al. CD8+ T cells regulate tumour ferroptosis during cancer immunotherapy. Nature. 569 (7755), 270-274 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados