Method Article
Zellfreier Dot-Blot als praktische und anpassungsfähige Immunoassay-Plattform zum Nachweis der Antikörperantwort in humanen und tierischen Seren
In diesem Artikel
Zusammenfassung
Wir beschreiben eine kürzlich entwickelte Immunoassay-Plattform, die auf den Prinzipien der zellfreien synthetischen Biologie und der Dot-Blot-Technik zum individualisierbaren Nachweis der Antikörperantwort in menschlichen und tierischen Seren basiert.
Zusammenfassung
Die Reihe von weltweiten Ausbrüchen von Krankheitserregern in den letzten zwei Jahrzehnten hat die Bedeutung von Serosurveillance Strategien deutlich gemacht. Immunoassay-Plattformen, die zum Nachweis krankheitsspezifischer Antikörper in den Seren von Patienten dienen, sind das Herzstück der Seroüberwachung. Gängige Beispiele sind enzymgebundene Immunsorbent-Assays und Lateral-Flow-Assays; Obwohl es sich hierbei um Goldstandardmethoden handelt, erfordern sie erregerspezifische Verbrauchsmaterialien und Spezialgeräte, was ihren Einsatz außerhalb gut ausgestatteter Labore einschränkt.
Vor kurzem haben wir eine neuartige Immunoassay-Plattform namens Cell-Free Dot-Blot (CFDB) entwickelt und anhand von menschlichen und tierischen Seren gegen SARS-CoV-2 validiert. Im Gegensatz zu herkömmlichen Immunoassays werden CFDB-Patientenserumproben in einer festen Phase (Nitrozellulosemembran) immobilisiert, während das Zielantigen in der mobilen Phase des Assays suspendiert wird. Um den Zugang zu Serosurveillance zu verbessern, werden CFDB-Antigene bei Bedarf und mit einer Infrastruktur mit geringem Aufwand unter Verwendung von In-vitro-Proteinexpression hergestellt. Hier wird das Antigen mit einem Peptid-Tag fusioniert, der mit einem einzigen universellen Reporterprotein für jeden CFDB-Assay nachgewiesen werden kann. Das Ergebnis ist, dass der CFDB keinen Zugang zu einem Multiwell-Plattenleser oder gereinigten kommerziellen molekularen Assay-Komponenten benötigt. Mit diesen Designüberlegungen adressiert CFDB die Unzulänglichkeiten bestehender Immunoassay-Plattformen, indem es den Zugang zu nicht zentralisierten Laboren, die Anpassungsfähigkeit für neu auftretende Krankheitserreger und die Erschwinglichkeit für einkommensschwache Gemeinden ermöglicht.
Im aktuellen Artikel stellen wir Ihnen ein Schritt-für-Schritt-Protokoll zur Vorbereitung und Durchführung eines CFDB-Immunoassays zur Verfügung. Am Beispiel unserer jüngsten Arbeit an SARS-CoV-2 CFDB werden wir das Antigen-DNA-Design für die zellfreie On-Demand-Produktion behandeln, gefolgt von der Vorbereitung des CFDB-Reporterproteins, der Immobilisierung von Serumproben in der festen Phase und schließlich den Antigenbindungs- und Nachweisschritten des Assays. Wir gehen davon aus, dass die Forscher durch die Befolgung dieser Anweisungen in der Lage sein werden, den CFDB-Assay so anzupassen, dass er Immunreaktionen in menschlichen und tierischen Seren auf einen bestimmten Krankheitserreger nachweist.
Einleitung
Die COVID-19-Pandemie hat den dringenden Bedarf an erschwinglichen, skalierbaren Diagnoseinstrumenten gezeigt, insbesondere für ressourcenarme Umgebungen1. Konventionelle Immunoassays wie Enzyme-linked Immunosorbent Assays (ELISAs) haben sich als unverzichtbar für den Nachweis von Immunantworten erwiesen 2,3. Ihre hohen Kosten, die Abhängigkeit von komplexen Reagenzien und die Abhängigkeit von Spezialgeräten schränken jedoch ihre Zugänglichkeit ein, insbesondere während globaler Gesundheitskrisen. Als Antwort auf diese Herausforderungen haben wir den Cell-Free Dot Blot (CFDB) entwickelt, eine kostengünstige, anpassungsfähige Immunoassay-Plattform, die für den Nachweis von Anti-SARS-CoV-2-Antikörpern in menschlichen und tierischen Seren entwickelt wurde.
CFDB nutzt die zellfreie synthetische Biologie für die schnelle On-Demand-Produktion viraler Antigene unter Verwendung linearer DNA-Templates 4,5. Dadurch entfallen herkömmliche zellbasierte Klonierungs-, Expressions- und Aufreinigungsprozesse, wodurch die Antigenproduktion erheblich beschleunigt und gleichzeitig die Kosten gesenkt werden. Die CFDB-Methode vereinfacht den Antikörpernachweis durch die Verwendung eines Dot-Blot-Formats, bei dem Seren direkt auf Nitrozellulosemembranen aufgetümmelt werden. Dieses System macht teure Multiwell-Platten und spezielle Laborgeräte überflüssig und ermöglicht einen einfachen "Tauch"-Arbeitsablauf für Inkubations- und Waschschritte. Die Plattform verwendet auch ein SpyCatcher-SpyTag-System, bei dem eine SpyCatcher2-Apex2-Peroxidase-Chimäre als universelles sekundäres Nachweisreagenzfungiert 5,6. Dies wird unter Verwendung der standardmäßigen Escherichia coli-basierten Expression hergestellt, wodurch die Abhängigkeit von teuren kommerziellen Antikörperkonjugaten entfällt. Infolgedessen kann das CFDB-System serologische Assays mit einer vergleichbaren Leistung wie ELISAs zu deutlich geringeren Kosten durchführen – etwa 3 USD pro 96 Proben im Vergleich zu über 300 USD für ein kommerzielles ELISA-Kit5.
Um die Wirksamkeit von CFDB zu demonstrieren, haben wir seine Fähigkeit zum Nachweis von Antikörpern in vorcharakterisierten menschlichen und tierischen Seren getestet. Unsere Ergebnisse korrelierten eng mit dem ELISA bei der Identifizierung von COVID-19-positiven und -negativen Proben. Zusätzlich zur Humandiagnostik untersuchten wir den Nutzen von CFDB in Tiermodellen, indem wir Seren von SARS-CoV-2-infizierten Hamstern und solchen, die mit rekombinantem Nukleokapsidprotein geimpft wurden, testeten. Diese Tests bestätigten das Potenzial von CFDB für den Einsatz sowohl in der Human- als auch in der Veterinärdiagnostik und machen es zu einem vielseitigen Instrument zur Überwachung von Immunreaktionen über Spezies hinweg. Einer der wichtigsten Vorteile von CFDB ist seine Flexibilität. Durch einfaches Modifizieren des DNA-Templates, das für das interessierende Antigen kodiert, kann die Plattform schnell angepasst werden, um Antikörper gegen verschiedene Krankheitserreger nachzuweisen, was sie für die zukünftige Pandemievorsorge wertvoll macht. Aufgrund seiner geringen Kosten, seines einfachen Arbeitsablaufs und seiner minimalen Infrastrukturanforderungen eignet es sich besonders für dezentrale Labore und ressourcenarme Umgebungen, in denen der Zugang zu kommerzieller Diagnostik begrenzt ist.
In dieser Arbeit geben wir eine Schritt-für-Schritt-Anleitung für die Vorbereitung und Durchführung eines CFDB-Assays. Zunächst befassen wir uns mit dem Design und der Synthese von linearen DNA-Templates für die zellfreie Produktion von Antigenen, die die primären Nachweisreagenzien des Assays sind. Anschließend beschreiben wir die Schritte zur Vorbereitung des sekundären Nachweisreagenzes des Assays, SpyCatcher2-Apex2. Danach geben wir Anleitungen für die zellfreie Herstellung und Qualitätskontrolle der Antigene selbst. Abschließend beschreiben wir detailliert den Prozess zur Durchführung eines CFDB-Assays an menschlichen oder tierischen Serumproben.
Protokoll
Alle Hamsterversuche wurden am National Microbiology Laboratory (NML) der Public Health Agency of Canada durchgeführt, vom Canadian Science Centre for Human and Animal Health genehmigt und nach den Richtlinien des Canadian Council on Animal Care durchgeführt. Alle menschlichen Serum-/Plasmaproben wurden kommerziell für interne Tests gewonnen oder von klinischen Mitarbeitern dem NML für unabhängige Tests am NML zur Verfügung gestellt.
1. Design und Erstellung von Antigen Linear Expression Templates (LETs)
- Entwerfen Sie die zellfreien Expressions-LETs für das Zielantigen basierend auf den Anweisungen in Norouzi et al.7 und mit einem N/C-terminalen SpyTag, wie in Norouzi et al.5 beschrieben.
HINWEIS: Hier finden Sie Details zur Herstellung von SARS-CoV-2 Nukleokapsid Protein (NP, Aminosäuren 2-419).- Es ist sicherzustellen, dass das LET für den SARS-CoV-2-NP (Abbildung 1 und ergänzende Datei 1) 5'- und 3'-Ter-Stellen mit einer Puffersequenz von 50 Basenpaaren vor dem T7-Promotor und nach dem Stoppcodon enthält.
- Markieren Sie das NP-Protein am N-Terminus mit His 6-SpyTag-TEV.
- Beziehen Sie die Proteinsequenz aus der UniProt-Datenbank (Accession Code P0DTC9) und optimieren Sie sie für die E . coli-basierte Expression mit dem IDT-Codon-Optimierungstool8. Bestellen Sie diese Sequenz für die kommerzielle Synthese als einzelsträngiges DNA-Fragment, das in Wasser bei 10 ng/μL resuspendiert wird.
HINWEIS: Die TEV-Protease-Spaltstelle und das His6-Tag sind keine wesentlichen Merkmale im LET-Design.
- PCR-Amplifikation des DNA-Fragments mit dem universellen Ter FW (GGCTCCGAATAAGTATGTTGTAACTAAAGTGCGGCC
ACGATGCGTCCGGCGTAGAGGATCG) und Ter RV (CCGAGGCAATAAGTATGTTGTAACTAAAGTGCTCAG
CTTCCTTTCGGGCTTTGTTAGCAGCC) unter Verwendung des High-Fidelity-DNA-Polymerase-Kits (siehe Materialtabelle)- Eine 100-μl-Reaktion wird wie folgt aufgebaut: 75 μl nukleasefreies Wasser, 20 μl 5x DNA-Polymerase-Puffer, 2 μl 10 mM dNTPs (Endkonzentration 200 μM), 0,5 μl 100 μM Ter-FW-Primer, 0,5 μl 100 μM Ter-RV-Primer, 1 μl 10 ng/μl einzelsträngiges LET, 1 μl(2U) DNA-Polymerase.
- Verwenden Sie die folgenden PCR-Einstellungen: Erstdenaturierung bei 98 °C für 30 s; 35 Zyklen: 98 °C für 6 s, 60 °C für 15 s, 72 °C für 90 s; Bei 4 °C halten.
- Reinigen Sie das PCR-Produkt mit einem kommerziellen PCR-Aufreinigungskit, überprüfen Sie seine Qualität, indem Sie eine Probe auf ein 1%iges Agarosegel laufen lassen, und messen Sie seine Konzentration auf einem UV-Vis-Spektralphotometer.
HINWEIS: Rohe PCR-Produkte können auch direkt für die zellfreie Expression verwendet werden. Die Reinigung ermöglicht jedoch ein standardisierteres Verfahren.

Abbildung 1: Lineare Expressionsvorlage für SARS-CoV-2-NP. Ein Schema, das die Merkmale des linearen DNA-Templates His-SpyTag-SARS-CoV-2 NP darstellt. Wichtige DNA-Vorlagenelemente sind beschriftet. Die kodierende Sequenz für das interessierende Protein, hier das NP, wird für eine effiziente Expression unter die transkriptionelle Kontrolle eines T7-Promotors gestellt. Am N-Terminus wird das NP-Protein mit einem SpyTag für den spezifischen Nachweis unter Verwendung des SpyCatcher2-Apex2-Detektionsreagenzes angehängt. Die x6His-Tag- und TEV-Proteasestellen sind, obwohl sie als Teil des allgemeinen LET-Designs enthalten sind, für CFDB-Zwecke entbehrlich. An den Enden des linearen DNA-Templates sind "Upstream"- und "Downstream"-Ter-Stellen, denen jeweils jeweils 50 Basenpaar-Puffersequenzen vorangestellt sind, für den Tus-vermittelten Schutz vor dem Abbau exonukleolytischer DNA im zellfreien Lysat enthalten. Abkürzungen: NP = Nukleokapsidprotein; LET = lineare Ausdrucksvorlage; TEV = Tabakätzvirus; CFDB = zellfreier Dot-Blot. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Aufreinigung des Reporterproteins SpyCatcher2-Apex2
- Verwenden Sie das pET24b-SpyCatcher2-Apex2-Plasmid, das ursprünglich in Norouzi et al.5 konstruiert wurde (vollständige Sequenz in Supplemental File 2 und Plasmidkarte in Supplemental Abbildung S1), um das SpyCatcher2-Apex-2-Reporterprotein herzustellen.
- Bereiten Sie Agarplatten und Lysogenie-Bouillon (LB) mit 50 μg/ml Kanamycin vor. Transformieren Sie E . coli BL21 (DE3)-Zellen mit dem Plasmid pET24b-SpyCatcher2-Apex2. Impfen Sie eine einzelne Kolonie in eine 15-ml-Starter-LB-Kultur und züchten Sie sie über Nacht bei 37 °C und Schütteln bei 250 U/min.
- Am nächsten Tag geben Sie 10 ml der Starterkultur in 500 ml frisches LB-Medium mit 50 μg/ml Kanamycin. Inkubieren Sie bei 37 °C und schütteln Sie bei 250 U/min, bis die Kultur bei 600 nm eine optische Dichte von 0,6-0,8 (~3 h) erreicht.
- Induzieren Sie die Expression von SpyCatcher2-Apex2, indem Sie die Kultur mit 0,5 mM Isopropyl-β-D-1-thiogalactopyranosid und 1 mM 5-Aminolävulinsäurehydrochlorid ergänzen. Reduzieren Sie die Wachstumstemperatur auf 30 °C und lassen Sie die Kultur weitere 4 Stunden inkubieren.
- Ernten Sie die Bakterien durch Zentrifugation bei 8.000 × g für 15 min. Fahren Sie mit der Zelllyse fort oder lagern Sie das Pellet bis zur Verwendung bei -80 °C.
- Resuspendieren Sie das Pellet in 20 ml Lysepuffer, der 50 mM Tris-HCl (pH 7,8), 300 mM NaCl, 1 mg/ml Lysozym, EDTA-freie Proteasehemmer-Tablette und 1 mM Dithiothreitol (DTT) enthält. Die Zellen werden durch Beschallung bei 50 % Amplitude in Intervallen von 5 s EIN und 10 s AUS für eine Gesamteinschaltzeit von 3 min lysiert.
- Das Lysat wird durch Zentrifugation bei 20.000 × g für 1 h bei 4 °C geklärt. Der Überstand wird durch einen 0,2 μm Spritzenvorsatzfilter geleitet.
- Häminchlorid in einer Endkonzentration von 250 μM zum geklärten Lysat geben und über Nacht bei 4 °C inkubieren. Fahren Sie mit der Proteinreinigung fort.
HINWEIS: Die Inkubation mit Häminchlorid maximiert den Häm-Einbau in die Apex-2-Peroxidase für eine optimale Enzymaktivität. - Geben Sie 2,5 mL Ni-Harz zum geklärten Lysat und inkubieren Sie bei 4 °C unter leichtem Schütteln für 45 Minuten. Tragen Sie die Mischung auf eine Schwerkraft-Flow-Säule auf und waschen Sie das Harz mit 50 mL Tris-Puffer (50 mM Tris-HCl (pH 7,8), 300 mM NaCl und 1 mM DTT).
- Eluieren Sie das SpyCatcher2-Apex2-Protein in 25 ml Tris-Puffer, der 400 mM Imidazol enthält. Konzentrat und Puffer tauschen das Eluat mit einer Zentrifugalfiltereinheit in Tris-Puffer aus, wobei ein Endvolumen von 0,5 bis 1,0 ml angestrebt wird.
- Verwenden Sie den molaren Extinktionskoeffizienten von SpyCatcher2-Apex2 (27.390 M-1 cm-1), um die Proteinkonzentration auf einem UV-Vis-Spektralphotometer zu bestimmen. Glycerin bis zu einer Endkonzentration von 40 % zugeben und die Aliquote bei -20 °C lagern.
HINWEIS: Es wird erwartet, dass dieses Protokoll etwa 40 mg hochreines und aktives SpyCatcher2-Apex2 liefert, ausreichend für 400 CFDB-Läufe mit einer Standard-Blot-Größe von 96 Proben. - Für den Nachweis von SpyTagged Proteinen mit herkömmlichem Western Blot9 und für CFDB-Experimente blockieren Sie die Membranen immer in 5 % fettfreier Trockenmilch in 1x Tris-gepufferter Kochsalzlösung mit 0,05 % Tween-20 (TBST). Verwenden Sie das SpyCatcher2-Apex2-Protein in einer Endkonzentration von 10 μg/ml in Blockierungslösung.
HINWEIS: Der SpyCatcher2-Apex2-Reporter erfordert eine ECL-Lösung (Enhanced Chemilumineszenz) für die Signalentwicklung. Die ECL-Lösung kann kommerziell bezogen oder im eigenen Haus gemäß den Anweisungen in Mruk et al.10 hergestellt werden. Die endgültige ECL-Lösung besteht hier aus 0,4 mM p-Cumarsäure, 2,5 mM Luminol und 0,015 % H2O2 in 100 mM Tris-HCl (pH 8,6).
3. Zellfreie Produktion und Qualitätskontrolle von Antigenen
- Bereiten Sie ein zellfreies Lysat und Reaktionskomponenten von E. coli BL21 vor und fügen Sie es zusammen, indem Sie den Anweisungen in Levine et al.11 und Norouzi et al.5 folgen. Ergänzen Sie das endgültige Reaktionsgemisch mit 5 μM Tus-Protein und 1,2 μM T7-RNA-Polymerase wie folgt für eine repräsentative 100-μl-Reaktion: 14,6 μl Lösung A, 14 μl Lösung B, 33,3 μl E. coli BL21-Lysat, 2,5 μl 200 mM Tus-Protein, 1,2 μl 100 mM T7-RNA-Polymerase, 1 μl 1,5 μM lineares DNA-Template, 33,4 μl nukleasefreies Wasser.
HINWEIS: In der ergänzenden Datei 3 finden Sie wichtige Anweisungen zur Herstellung eines zellfreien Lysats BL21 für E . coli und in der ergänzenden Tabelle S1 für eine detaillierte Rezeptur der Lösungen A und B. Das zellfreie Expressionssystem für E. coli kann auch unter Verwendung alternativer Protokolle hergestellt oder kommerziell erworben werden, solange die Reaktion mit Tus und T7-RNA-Polymerase ergänzt wird. - Führen Sie einen ersten Expressionstest im 5-μl-Maßstab durch, indem Sie der zellfreien Reaktion in einem PCR-Röhrchen 10 % (v/v) Roh-PCR-Produkt zusetzen. Ohne Schütteln bei 30 °C für 15 h inkubieren.
- Überprüfen Sie die Expressionsqualität des NP-Antigens, indem Sie 1 μl der zellfreien Reaktion auf ein 12%iges Natriumdodecylsulfat-Polyacrylamid-Gel (SDS-PAGE) laden und anschließend auf eine Nitrozellulosemembran für Western Blot übertragen, wie in Schritt 2.12 beschrieben. Verwenden Sie das Reporterprotein SpyCatcher2-Apex2 für die Blot-Markierung über den SpyTag und optional einen spezifischen kommerziellen Antikörper (hier einen Anti-SARS-CoV-2-NP) gegen das Zielantigen.
- Assemblieren Sie eine zellfreie Reaktion im 1-ml-Maßstab mit 15 nM gereinigtem LET-Produkt, das für den CFDB-Assay verwendet wird. Die Expressionsmischung wird in einem konischen 15-ml-Röhrchen unter Schütteln bei 80 U/min für 15 Stunden bei 30 °C inkubiert. Überprüfen Sie die Expressionsqualität mit Western Blot und lagern Sie 50 μL Aliquots bei -20 °C.
4. Serum-Proben
- Beziehen Sie Serumproben (oder Plasmaproben für Menschen/Tiere) aus kommerziellen Quellen oder von klinischen Mitarbeitern oder intern unter Verwendung von Standardverfahren und gemäß den Richtlinien der institutionellen Ethikkommission für die Humanforschung oder der Kommission für Tierpflege und -verwendung. Stellen Sie sicher, dass geeignete Maßnahmen zur Probenvorbehandlung ergriffen wurden, um das Kontaminationsrisiko zu mindern.
HINWEIS: Vorbehandlungsmaßnahmen können eine Behandlung mit Lösungsmittel und Reinigungsmitteln, eine Hitzeinaktivierung und Tests auf durch Blut übertragbare Virusmarker umfassen. Ein nützliches Protokoll für die Serum- und Plasmaisolierung aus Vollblut steht zur Verfügung12. Wie bei anderen Immunoassay-Plattformen ist nicht zu erwarten, dass Serumproben, die früh (<3 Wochen) nach der Erstinfektion oder dem Ausbruch der Krankheit entnommen werden, ausreichende Mengen einer Antikörperreaktion enthalten, und ihre Ergebnisse sollten mit Vorsicht behandelt werden. - Für die Qualitätsüberprüfung der SARS-CoV-2 NP CFDB-Reagenzien verwenden Sie das internationale Referenzgremium der Weltgesundheitsorganisation (WHO) des National Institute for Biological Standards and Control (NIBSC) für Anti-SARS-CoV-2-Immunglobulin, das x1 prä-COVID-19 und x4 SARS-CoV-2-positive Proben mit unterschiedlichen Gehalten an Anti-NP-Immunglobulinenenthält 13. Alternativ können Sie vorcharakterisierte gesunde und positive Serumproben aus anderen Quellen verwenden.
- Für CFDB-Experimente können Sie eine negative Kontrollprobe gewinnen oder vorbereiten, indem Sie mehrere (>3) gesunde Serumproben zusammenfassen, um die Genauigkeit zu erhöhen und die Analyse und Interpretation der Ergebnisse zu erleichtern.
5. Verfahren des zellfreien Dot-Blots (CFDB)
- Laden Sie die Master-Grid-Bilddatei herunter und drucken Sie sie aus (Ergänzende Abbildung S2). Das Master-Gitter ist ein 6 x 6 cm großes Muster mit 12 x 12 Kreisen mit je 2 mm Durchmesser und bietet eine ähnliche Fleckenkapazität wie eine 96-Well-Platte.
- Schieben Sie das Gitter fest zwischen zwei Lagen selbstklebender PCR-Platten-Siegelfolie ein und schneiden Sie es entlang der äußeren Gitterränder zu. Verwenden Sie einen 2-mm-Biopsiestanzer, um jeden markierten Kreis auszuhöhlen.
HINWEIS: Dieser Master-Rost kann nach dem Abwischen mit 70% Ethanol mehrmals wiederverwendet werden. - Schneiden Sie ein 6,5 x 6,5 cm großes Stück Nitrozellulosemembran ab und positionieren Sie es unter dem Master-Gitter auf einer sauberen Oberfläche, wobei Sie das Setup mit Klebeband sichern, wie in Abbildung 2 und Ergänzende Abbildung S3 gezeigt. Markieren Sie mit einem Markierungsstift die äußersten Kreispositionen auf der Nitrozellulosemembran, die als Leitfaden für das Schneiden der Membran nach der Probenaufschluss dienen sollen.
- Verdünnen Sie die Serumproben zu 1/10 in 1x phosphatgepufferter Kochsalzlösung (pH 7,4). Verwenden Sie eine Mikropipette, um ein dreifaches Volumen von 0,4 μl jeder Probe an vorgegebenen Gitterpositionen auf die Nitrozellulosemembran zu dispensieren. Die Abgabe dauert ca. 15 s pro Stelle.
HINWEIS: Achten Sie darauf, dass Sie jedem Assay sowohl Negativ- als auch Positivkontrollproben beifügen, da dies für die Analyse der Ergebnisse erforderlich ist. - Lassen Sie die gefleckten Proben 10 Minuten bei Umgebungstemperatur binden und trocknen. Ziehen Sie die Nitrozellulosemembran vorsichtig mit einer Pinzette heraus und schneiden Sie entlang der markierten äußeren Kreise.
- Blockieren Sie die Membran in 10 ml Blockierungslösung (5 % fettfreie Trockenmilch in TBST) in einer 10 cm Petrischale für 30 Minuten bei Raumtemperatur und schütteln Sie sie leicht bei 100 U/min.
- Tauen Sie auf und geben Sie ein 50 μl Aliquot des zellfreien Antigenexpressionsgemisches zu 5 ml Blockierungslösung in einer 10 cm Petrischale. Die Membran wird direkt in diese antigenhaltige Lösung überführt und 1 h bei Raumtemperatur inkubiert, wobei bei 100 U/min geschüttelt wird.
HINWEIS: Dies ist der primäre Nachweisschritt, bei dem das SpyTagged-Antigen an antikörperhaltige Spot-Positionen bindet, falls vorhanden. - Spülen Sie die Membran aus, waschen Sie sie 5 Minuten lang und spülen Sie sie erneut in TBST, bevor Sie mit dem sekundären Nachweisschritt fortfahren.
- Inkubieren Sie die Membran in 10 mL Blockierungspuffer mit 10 μg/ml gereinigtem SpyCatcher2-Apex2-Protein für 1 h unter Schütteln bei 100 U/min.
- Spülen Sie die Membran aus und waschen Sie sie 2 x 5 min in TBST, mit einer abschließenden Spülung in TBS.
- Kleben Sie ein Stück Parafilm auf eine saubere Arbeitsfläche in der Nähe des Blot-Imaging-Instruments und befestigen Sie es. Entfernen Sie überschüssige Flüssigkeit, indem Sie mit einer Pinzette auf die Membran klopfen und die Membran auf den Parafilm legen.
- Geben Sie sofort 3 mL (100 μL/cm2) ECL-Lösung auf die Membran und inkubieren Sie sie genau 90 s lang bei Raumtemperatur.
- Trocknen Sie die Membran sofort ab und übertragen Sie sie zur Visualisierung der Ergebnisse auf ein Chemilumineszenz-kompatibles Bildgebungsinstrument.
HINWEIS: Die optimale Bildgebungszeit kann bei verschiedenen Instrumenten variieren. Es wird empfohlen, die Standardeinstellungen für die automatische Erfassungszeit des Geräts zu verwenden und, falls nicht vorhanden, vorcharakterisierte Serumproben zu verwenden, um die Bildaufnahmezeit zu optimieren. - Verwenden Sie die Bildanalysefunktion des Geräts, um Punktintensitäten zu erhalten, einschließlich für drei leere (Hintergrund-)Positionen auf der Nitrozellulosemembran; Halten Sie ein konstantes Messvolumen pro Spot aufrecht. Exportieren Sie die Daten in eine Tabelle und denken Sie daran, jede Punktposition korrekt zu beschriften.
- Berechnen Sie den Mittelwert und die Standardabweichung (SD) der dreifachen Punktintensitäten. Subtrahieren Sie dann den Hintergrund der Nitrozellulosemembran von allen Proben.
- Verwenden Sie die folgende Gleichung, um einen Grenzwert für die Interpretation der -/+-Ergebnisse zu erhalten:
(Mittelwert der Negativkontrollen) + (3 x SD der Negativkontrollen). Betrachten Sie Proben als positiv, wenn ihre durchschnittliche Intensität über den Grenzwert fällt, und als negativ, wenn ihre durchschnittliche Intensität unter den Grenzwert fällt.
HINWEIS: Ein Schema des CFDB-Verfahrens ist in Abbildung 3 dargestellt.

Abbildung 2: CFDB-Baugruppe. Ein Schema des Aufbaus des CFDB-Mastergitters und der NC-Membranbaugruppe. Das Master-Gitter wird auf der NC-Membran überlagert, um ein regelmäßiges, adressierbares Muster für die Spotting und Immobilisierung von Serumproben zu erhalten. Abkürzungen: CFDB = zellfreier Dot-Blot; NC = Nitrocellulose. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Eine schematische Darstellung des CFDB-Arbeitsablaufs. Bei einem CFDB-Assay wird eine kleine Menge (<0,4 μl) von 10x verdünnten Serumproben manuell auf eine vorgeschnittene Nitrozellulosemembran (linkes Bild) an diskreten, adressierbaren Stellen (mittleres Bild) aufgetragen. Ablage einer Serumprobe pro Spot in dreifachen Spots und Immobilisierung des Proteingehalts, einschließlich des gesamten Antikörperreservoirs der Seren, auf dem festen NC-Substrat (beige Flecken im mittleren Feld). In diesem Beispiel können die in den Serumproben enthaltenen Anti-NP-Antikörper zunächst durch das CFDB-Primärdetektionsreagenz SpyTag-NP gebunden und schließlich durch das CFDB-Sekundärdetektionsreagenz SpyCatcher2-Apex2 (vergrößerte Blase auf dem rechten Feld) nachgewiesen werden. Diese Abbildung stammt von Norouzi et al.5. Abkürzungen: CFDB = zellfreier Dot-Blot; NP = Nukleokapsidprotein; LET = lineare Ausdrucksvorlage. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
PCR-Amplifikation des linearen Expressions-Templates für das Zielantigen
Zur PCR-Amplifikation des SARS-CoV-2 NP LET wurden universelle Ter-Forward- und Reverse-Primer verwendet, wie in Protokollabschnitt 1.2 beschrieben, und 1 μl des Produkts wurde auf einem Agarosegel überprüft (Abbildung 4), bevor mit der Aufreinigung des PCR-Produkts fortgefahren wurde.

Abbildung 4: PCR einer linearen Expressionsvorlage. Ein 1%iges Agarose-Gel, das in Lane 1: 1 kb DNA-Leiter und Lane 2: das PCR-amplifizierte lineare DNA-Template für den His-SpyTag-SARS-CoV-2 NP (1.630 bp) gezeigt wird. Abkürzungen: PCR = Polymerase-Kettenreaktion; NP = Nukleokapsid-Protein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Aufreinigung des Reporterproteins SpyCatcher2-Apex2
Das SpyCatcher2-Apex2-Protein wurde wie in Protokollabschnitt 2 beschrieben hergestellt. Um einen adäquaten Häm-Einbau in die Apex2-Peroxidase zu gewährleisten, wurden 5-Aminolävulinsäurehydrochlorid (ein Häm-Vorläufer) und Häminchlorid während der Proteinsynthese bzw. nach der Zelllyse zugesetzt. Proben aus allen Reinigungsschritten wurden auf ein SDS-PAGE-Gel geladen, um die Qualität des Endprodukts sicherzustellen (Abbildung 5).

Abbildung 5: SpyCatcher2-Apex2-Reinigung. Ein 12%iges SDS-PAGE-Gel, das Fraktionen aus den Reinigungsschritten des SpyCatcher2-Apex2-Proteins zeigt. L: Lysat (2 μL), FT: Durchfluss (2 μL), W: Wash (10 μL), E1-3: Elution je 1-3 (10 μL). Das erwartete Band bei 42 kDa ist markiert. Abkürzung: SDS-PAGE = Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Zellfreie Expression und Qualitätsverifizierung des Zielantigens
Nach Durchführung einer Pilotexpression des NP-Proteins im 5-μL-Maßstab und einer Qualitätsprüfung mittels SDS-PAGE wurde eine Expression im 1-ml-Maßstab durchgeführt, wie in Protokollabschnitt 3 beschrieben. Eine 1-μl-Probe des endgültigen zellfreien Expressionsgemisches wurde separat einem Western Blot unterzogen, um mit SpyCatcher2-Apex2 und einem kommerziellen Anti-NP-Antikörper nachgewiesen zu werden (Abbildung 6). Dieser Schritt stellt sicher, dass das Antigen erfolgreich exprimiert wird und spezifisch sowohl an Anti-NP-Immunglobuline als auch an das Reporterprotein SpyCatcher2-Apex2 binden kann.

Abbildung 6: Qualitätskontrolle von zellfrei produzierten Antigenen. Western-Blot-Nachweis des zellfrei produzierten NP-Proteins mit SpyCatcher2-Apex2 (linkes Bild) und einem polyklonalen Anti-NP-Kaninchen-Antikörper (rechtes Bild). Das erwartete Produkt in voller Länge mit 50 kDa ist markiert. Der Größenmarker in beiden Gelen ist der Color Pre-stained Protein Standard, Broad Range. Diese Abbildung wurde von Norouzi et al.5 übernommen. Abkürzung: NP = Nukleokapsidprotein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Verifizierung von CFDB-Reagenzien mit vorcharakterisierten humanen Seren
Um zu bestätigen, dass die CFDB-Reagenzien von hoher Qualität sind und die CFDB-Protokolle erfolgreich implementiert werden, verwendeten wir ein vorcharakterisiertes Serumpanel, das vom NISBC-dem internationalen Referenzpanel der WHO für Anti-SARS-CoV-2-Immunglobulin 20/26813 zur Verfügung gestellt wurde. Dieses Gremium besteht aus x1 prä-COVID-19 (negativ) und x4 SARS-CoV-2-positiven Proben mit unterschiedlicher Antikörperantwort (Ergänzende Tabelle S2). Darüber hinaus erwarben wir ein Serumpanel von RayBiotech (CoV-PosSet), das 10 Prä-COVID-19 (negativ) und 20 mutmaßlich SARS-CoV-2-positive Proben enthält. Wir haben gleiche Fraktionen aus allen 10 negativen Seren gepoolt, um sie als Negativkontrolle für alle in unserem Labor durchgeführten Experimente zu verwenden. Diese Pooling-Strategie erhöht die Genauigkeit und Robustheit der Ergebnisse, da einzelne negative Stichproben subtile Hintergrundsignale aufweisen können, die die Interpretation der Ergebnisse erschweren können.
Ein CFDB-Assay wurde auf dem vorcharakterisierten WHO-Referenzpanel und dem CoV-PosSet von RayBiotech gemäß Protokollabschnitt 5 durchgeführt und der Blot abgebildet. Das Lautstärkewerkzeug in der Image Lab Software wurde verwendet, um einheitliche Kreise auf jede Punktposition zu zeichnen und individuelle Signalintensitäten zu exportieren. Nach Subtraktion der Hintergrundintensität der NC-Membran von allen Proben wurde auch der Cut-off-Wert ((Mittelwert der Negativkontrollen) + (3 x SD der Negativkontrollen)) von den durchschnittlichen Signalintensitäten subtrahiert. Proben gelten als anti-NP-positiv, wenn ihre Signalintensität über den Grenzwert fällt. Ein Bild des Blots und das entsprechende Signalintensitätsdiagramm sind in Abbildung 7 dargestellt. Die Ergebnisse zeigen, dass die CFDB-Technik unter Verwendung eines zellfrei produzierten SARS-CoV-2-NP-Proteins alle negativen und alle positiven Proben korrekt identifizieren kann, mit Ausnahme von zwei Proben aus dem RayBiotech-Set (RB P5-gesammelt 34 Tage nach Symptombeginn (dpso) und RB P10-gesammelt 4 dpso). Eine weitere Durchsicht des Datenblatts des Lieferanten bestätigte, dass diese beiden Proben auch negativ für anti-S1RBD (Spike Receptor Binding Domain) IgG mittels ELISA und anti-N IgG/M durch Lateral-Flow-Assay erscheinen (Ergänzende Tabelle S3), was auf einen Mangel an nachweisbaren Antikörperspiegeln in diesen Proben hindeutet.

Abbildung 7: Verifizierung von CFDB anhand von vorcharakterisierten kommerziellen humanen Seren. (A) Blot-Bild, das einen Anti-SARS-CoV-2 NP CFDB auf dem internationalen Referenzpanel der WHO und RayBiotech PosSet-Serumproben darstellt. Die Proben wurden in dreifacher Ausfertigung säulenweise gesichtet und durch Beschriftungen gekennzeichnet. (B) Diagramm, das die quantifizierten Intensitätswerte des Chemilumineszenzsignals für den Blot in Panel A darstellt. Zur Einstellung des Cutoff-Wertes wurde die Negativkontrolle verwendet, und alle Messungen sind Mittel von dreifachen Spots ± Standardabweichungen (SD). Diese Abbildung stammt von Norouzi et al.5. Abkürzungen: Neg Cont = Negativkontrollprobe, die durch Pooling aller 10 PräCOVID-19-Proben von RayBiotech gewonnen wird; RB N1 bis N10 = RayBiotech vermutlich negative Proben 1 bis 10; RB P1 bis P20 = RayBiotech vermutete positive Proben 1 bis 20; CFDB = zellfreier Dot-Blot; NP = Nukleokapsidprotein; WHO = Weltgesundheitsorganisation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Unabhängige Testung des CFDB-Assays an humanen Patientenseren
Um die Anwendbarkeit des CFDB-Assays zu demonstrieren, stellten wir CFDB-Materialien und -Anweisungen einem unabhängigen Labor an einem anderen Standort (National Microbiology Laboratory (NML), Winnipeg) zur Verfügung. Ziel war es, die Plattform unabhängig an menschlichen Patientenproben zu testen, die in der Klinik von der Feldstudiengruppe am NML gesammelt wurden. Insgesamt wurden 24 Proben (x12 negativ und x12 positiv-bestätigte ELISA für NP-Antikörper (Ergänzende Tabelle S4) einer CFDB unterzogen und die Ergebnisse wie oben beschrieben analysiert. Wie in Abbildung 8 zu sehen ist, identifizierte CFDB alle x12 negativen und x12 positiven Proben korrekt, was die Robustheit und Praktikabilität des Assays in einem unabhängigen Test bestätigt.

Abbildung 8: Unabhängige Testung von CFDB mit humanen Patientenseren am NML. (A) Blot-Bild, das einen Anti-SARS-CoV-2 NP CFDB an 12 COVID-19-negativen (N1 bis N12) oder 12 positiven (P1 bis P12) Patientenproben darstellt. Die Seren wurden in dreifacher Ausfertigung spaltenweise gesichtet, wie beschriftet. (B) Quantifizierte Intensitätswerte des Chemilumineszenz-Signals aus dem Blot in Feld A. Alle Messungen sind Mittel von dreifachen Spots ± Standardabweichungen (SD). Diese Abbildung stammt von Norouzi et al.5. Abkürzungen: CFDB = zellfreier Dot-Blot; NML = nationales mikrobiologisches Laboratorium; NP = Nukleokapsid-Protein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Unabhängige Testung des CFDB-Assays anhand von SARS-CoV-2-infizierten Hamsterseren
Unter Verwendung der gleichen CFDB-Reagenzien und -Anweisungen führte das NML-Team dann einen CFDB-Assay an SARS-CoV-2-infizierten Hamsterseren durch, um zu testen, ob dieser Assay auch zum Nachweis einer Antikörperreaktion in Tierproben verwendet werden kann. Insgesamt wurden 36 Proben getestet, x12 präinfektionierte und 24 postinfektionierte Seren. Die vermutlich positiven Seren stammten von Tieren, die mit 105 50 % infektiösen Gewebekulturdosen (TCID50) von SARS-CoV-2 infiziert waren und 140 Tage nach der Infektion beprobt wurden (dpi 140, n = 3); oder mit dpi 140 erneut infiziert und 5 Tage später beprobt (dpi 5, n = 21). Wie in Abbildung 9 gezeigt, wiesen alle Seren vor der Infektion Signalintensitäten unterhalb des Cut-off-Wertes auf, und 18 von 24 Seren nach der Infektion (alle ab 5 Tagen nach der Reinfektion) lösten diskrete positive Signalintensitäten aus. Interessanterweise waren von den sechs vermeintlich positiven Proben, die von CFDB übersehen wurden, drei Proben (alle aus dem ursprünglichen dpi 140) auch mit einem Standard-Anti-NP-ELISA nicht nachweisbar (Supplemental Table S5), was auf einen Verlust oder eine Abnahme der Antikörperspiegel im Laufe der Zeit hinweist.
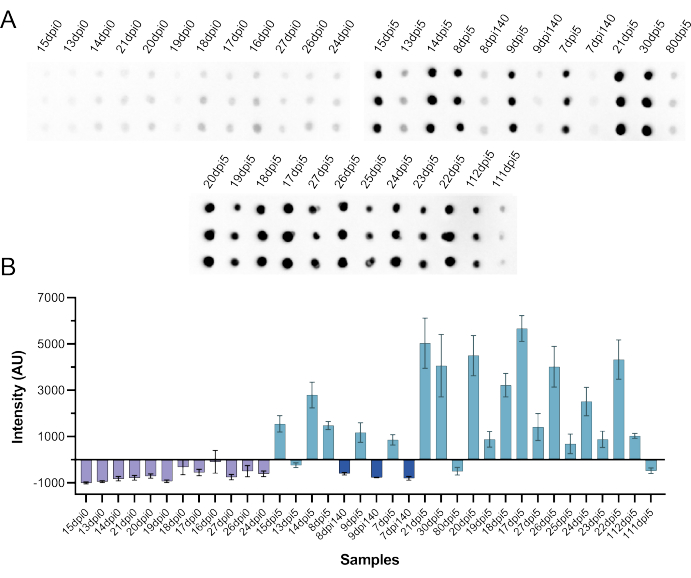
Abbildung 9: Unabhängige Testung von CFDB anhand von SARS-CoV-2-infizierten Hamsterseren am NML. (A) Blot-Bild, das eine Anti-SARS-CoV-2 NP CFDB auf Hamsterseren vor der Infektion (dpi 0, n = 12), 140 Tage nach der Infektion (dpi 140, n = 3) und 5 Tage nach der Reinfektion (dpi 5, n = 21) mit 105 TCID50 SARS-CoV-2 zeigt. Jede Probe wurde als dreifache Flecken säulenweise deponiert und nach dem Schema der ursprünglichen NML-Studie beschriftet. (B) Quantifizierte Intensitätswerte des Chemilumineszenzsignals aus dem Blot in Feld A. Alle Messungen sind Mittelwerte von dreifachen Spots ± Standardabweichungen (SD). Diese Abbildung stammt von Norouzi et al.5. Abkürzungen: CFDB = zellfreier Dot-Blot; NML = nationales mikrobiologisches Laboratorium; NP = Nukleokapsidprotein; dpi = Tage nach der Infektion; TCID50 = 50 % infektiöse Dosis der Gewebekultur. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Nukleotidsequenz für die lineare Expressionsvorlage His-SpyTag-SARS-CoV-2 NP. Abkürzung: NP = Nukleokapsidprotein. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: Vollständige Nukleotidsequenz des pET24b SpyCatcher2-Apex2-Plasmids. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: Ein Überblick über die wichtigsten Schritte bei der Vorbereitung eines zellfreien E. coli BL21-Proteinexpressionslysats. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S1: Konstruktkarte für das Plasmid pET24b SpyCatcher2-Apex2. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S2: Eine druckbare Kopie des CFDB-Master-Gitters. Abkürzung: CFDB = zellfreier Dot-Blot. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S3: Ein Bild des Aufbaus der CFDB NC-Membran und der Master-Grid-Baugruppe. Die NC-Membran wird zwischen einer Glasplatte auf der Unterseite und dem Master-Gittermuster auf der Oberseite eingeklemmt und sichert die Baugruppe mit stationärem Klebeband. Diese Abbildung stammt von Norouzi et al.5. Abkürzungen: CFDB = zellfreier Dot-Blot; NC = Nitrocellulose Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S1: Vollständige Rezepturen für die Komponenten Lösung A und Lösung B des zellfreien Reaktionsgemisches. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S2: Beispielinformationen für das NIBSC WHO-Referenzpanel 20/268 (siehe auch Abbildung 7). Abkürzungen: NISBC = The National Institute for Biological Standards and Control; WHO = Weltgesundheitsorganisation. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S3: Probeninformationen RayBiotech CoV-PosSet (siehe auch Abbildung 7). Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S4: NP-ELISA-Ergebnisse für Serumproben von menschlichen Patienten (durchgeführt bei NML). Siehe auch Abbildung 8. Abkürzungen: NP = Nukleokapsidprotein; ELISA = Enzyme-linked Immunosorbent Assay; NML = nationales mikrobiologisches Laboratorium. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S5: NP-ELISA-Ergebnisse für SARS-CoV-2-infizierte Hamsterseren (durchgeführt bei NML). Siehe auch Abbildung 9. Abkürzungen: NP = Nukleokapsidprotein; ELISA = Enzyme-linked Immunosorbent Assay; NML = nationales mikrobiologisches Laboratorium. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
COVID-19 hat deutlich gemacht, wie wichtig zugängliche und robuste Diagnostika für die Kontrolle von Infektionsausbrüchen und die Optimierung globaler Gesundheitsstrategien sind. Serologische Tests zum Nachweis schützender Antikörper erwiesen sich als unerlässlich für die Verfolgung von Übertragbarkeitsmustern neuer Varianten, die Identifizierung von Hotspots, die Steuerung der Impfstoffentwicklung, die Triage von Verdachtsfällen und den Schutz gefährdeter Bevölkerungsgruppen14. Die Pandemie hat auch Ungerechtigkeiten bei der Zugänglichkeit von Tests aufgedeckt, die durch Rückstände und Anforderungen an Einrichtungen mit hochentwickelter Ausrüstung noch verschärft wurden15. Der Fokus liegt daher nun darauf, unsere globale Reaktion auf zukünftige Ausbrüche zu stärken, was neue Innovationen erfordert, die Testbarrieren abbauen und schnell an unvorhersehbare Bedrohungen angepasst werden können.
CFDB ist ein papierbasierter Immunoassay, der serologische Tests, die herkömmlich mit ELISAs oder Lateral-Flow-Assays durchgeführt werden, vereinfacht, indem er Patientenproben in der festen Phase immobilisiert und zellfrei produzierte Antigene in der mobilen Phase zum Nachweis von antiviralen Antikörpern liefert5. Der Assay kann in einfachen Laborumgebungen mit Western-Blot-Funktionen durchgeführt werden. In der Zwischenzeit können Reagenzien für CFDB vor Ort in der Grundlagenforschung hergestellt und leicht ausgetauscht werden, um den Assay für neue Krankheitserreger umzufunktionieren. Das chimäre SpyCatcher2-Apex2 Universal-Detektionsreagenz ist eine grundlegende Funktion, die die Beschaffung kommerzieller Sekundärantikörperkonjugate überflüssig macht und effizient in einem E. coli-Expressionssystem hergestellt wird. CFDB bietet auch Hochdurchsatzfähigkeiten, die parallele klinische Assays zu einem Bruchteil der Kosten ermöglichen, und zwar zu einem Preis von nur 3 USD pro Assay-Kit im Vergleich zu etwa 300 USD für einen kommerziellen 96-Well-ELISA.
Die Konfiguration des CFDB für serologische COVID-19-Tests wurde durch die Entwicklung linearer DNA-Templates erreicht, die das zellfreie Transkriptions-Translationssystem anwiesen, das Zielantigen SARS-CoV-2 NP zu produzieren. Menschliche und tierische Seren wurden dann auf Nitrozellulose-Membrangitter aufgespießt und Inkubationen mit der zellfreien Antigenexpressionsmischung durchgeführt, gefolgt von dem Reporterprotein SpyCatcher2-Apex2. In dieser Studie wurde die CFDB an Seren von COVID-19-negativen und -positiven Probanden durchgeführt, sowohl in unserem Labor als auch in einem unabhängigen Setting (NML), und insgesamt wurde eine gute Korrelation mit dem ELISA zur Unterscheidung von positiven und negativen Proben erreicht. Durch die Verwendung von gepoolten gesunden Seren als CFDB-Negativkontrolle und die Berechnung eines Assay-Cutoff-Wertes auf der Grundlage dieser Probe wurde das Risiko einer falsch negativen/positiven Identifizierung minimiert.
Die hohe Sensitivität des CFDB-Assays zeigt sich in seiner Fähigkeit, alle Proben des umfassend vorcharakterisierten NIBSC-Referenzgremiums 20/26813 eindeutig zu unterscheiden, insbesondere die Probe mit dem niedrigsten Antikörpertiter (Probe 20/140 niedrig, siehe ergänzende Tabelle S2 und Abbildung 7). In ähnlicher Weise wird die hohe Spezifität von CFDB durch das Fehlen einer falsch-positiven Identifizierung für eine der 35 in dieser Arbeit getesteten gesunden Seren demonstriert. Wichtig ist, dass die klinische Validierung an mehreren Standorten bestätigte, dass das CFDB-Protokoll für einen breiten Einsatz ausreichend einfach ist und eine hohe Leistung beibehält, die mit Standard-ELISAs vergleichbar ist, wenn es von verschiedenen Benutzern ausgeführt wird. Nach unserer Erfahrung und wie andere16 gezeigt haben, sind die in E. coli-Lysaten produzierten zellfreien Antigene mindestens 3 Monate bei -20 °C stabil, und mehrere zellfreie Antigen-Chargenpräparate funktionieren nicht voneinander zu unterscheiden.
Vor der breiten Einführung der CFDB-Plattform und der Umstellung des Systemdesigns auf neue Krankheitserreger sollten mehrere Überlegungen berücksichtigt werden. Die CFDB-Plattform erfordert jedoch, dass das virale Antigen oder Antigenfragment durch ein zellfreies Expressionssystem ordnungsgemäß synthetisiert wird, so dass antigene Einheiten vorhanden und für die Antikörperbindung verfügbar sind. So hatten wir ursprünglich vor, auch die SARS-CoV-2-Spike-Rezeptor-Bindungsdomäne (RBD) in unsere CFDB-Assays aufzunehmen. Trotz einer hohen Ausbeute an Expression und aufgrund seines Bedarfs an komplexen Disulfidbindungen (im Gegensatz zum NP-Antigen, dem Cystein-Aminosäuren fehlen) aggregierte das RBD-Produkt jedoch leicht und schien im E. coli-Extrakt unlöslich zu sein, was seine Verwendung als zuverlässige CFDB-Sonde ausschloss. In solchen Szenarien, in denen komplexe posttranslationale Modifikationen für eine korrekte Faltung und antigene Funktion erforderlich sind, wird der Einsatz von eukaryotischen zellfreien Systemen empfohlen. Wie bei anderen Peroxidase-basierten Immunoassays ist es auch hier zwingend erforderlich, dass Serumproben mit sichtbarer Hämolyse ausgeschlossen werden, da die Hämoglobinperoxidase-Aktivität die Ergebnisse verfälschen kann. Schließlich ist das CFDB so konzipiert, dass es die gesamte Antikörperantwort nachweist und berichtet nicht über die spezifische Immunglobulinklasse, die an Antikörper-Antigen-Wechselwirkungen beteiligt ist.
Zusammenfassend lässt sich sagen, dass es sich bei der CFDB um eine neuartige Diagnosetechnik handelt, die Elemente der zellfreien synthetischen Biologie und des Dot-Blottings zusammenführt, um serologische Tests zu rationalisieren. Der CFDB bietet eine kostengünstige Alternative zu herkömmlichen ELISAs, die problemlos in Forschungslaboren weltweit eingesetzt werden können, wenn zusätzliche Testkapazitäten benötigt werden, wie es bei einer Pandemie oft der Fall ist. Das CFDB kann somit als Frontinstrument bei der nächsten globalen Pandemiebekämpfung dienen, das schnell an neue Bedrohungen angepasst werden kann und die Testkapazitäten von Ländern mit niedrigem und mittlerem Einkommen stärken wird.
Offenlegungen
M.N. und K.P. sind Miterfinder des zellfreien Dot-Blot-Verfahrens. Eine vorläufige Patentanmeldung für diese Arbeit wurde eingereicht (PCT/CA2024/050097, eingereicht im Januar 2024).
Danksagungen
S.S. und R.Z. werden durch Mittel der Defense Advanced Research Projects Agency (DARPA), Vertragsnummer N66001-23-2-4042, unterstützt. Die geäußerten Ansichten, Meinungen und/oder Ergebnisse sind die der Autoren und sollten nicht so interpretiert werden, dass sie die offiziellen Ansichten oder Richtlinien des Verteidigungsministeriums oder der US-Regierung repräsentieren. Diese Arbeit wurde mit Mitteln an K.P. aus dem CIHR Foundation Grant Program (201610FDN-375469), dem CIHR Canada Research Chair Program (950-231075 und 950-233107), der Medicine by Design Initiative der University of Toronto, die vom Canada First Research Excellence Fund finanziert wird, und Mitteln an K.P. aus dem Canadian Safety and Security Program von Defense Research and Development Canada (Vertrag 39903-200137) unterstützt. Abbildung 1 und ergänzende Abbildung S1 wurden mit SnapGene Viewer erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 kb DNA ladder | NEB | N3232 | Used as a size marker for agarose gels |
| 20 Amino acids | Sigma-Aldrich | LAA21-1KT | A component of the cell-free reaction Solution B |
| 2 mm biopsy punch | Integra Miltex | 33-31-P/25 | For preparation of CFDB master grid |
| 5-aminolevulinic acid hydrochloride | Sigma-Aldrich | A3785 | Heme precursor for induction of SpyCatcher2-Apex2 |
| Agarose powder | BioShop | AGA002 | For electrophoretic analysis of DNA |
| Anti-SARS-CoV2-Nucleocapsid antibody | Sinobiological | 40588-T62 | For WB detection of cell-free produced NP antigen |
| Bio-Rad ChemiDoc XRS+ | Bio-Rad | N/A | Gel imager instrument |
| Centrifugal concentrators | Cytiva | 28-9323-60 | For concentration and buffer exchange of proteins |
| Centrifuge | Eppendorf | EP022628257 | For harvesting bacterial culture |
| Coenzyme A sodium salt hydrate (CoA) | Sigma-Aldrich | C3144 | A component of the cell-free reaction Solution A |
| Color pre-stained protein standard (broad range) | NEB | P7719 | Used as a size marker for SDS-PAGE gels |
| Covid-19 serum panel | RayBiotech | CoV-PosSet | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| D-(−)-3-Phosphoglyceric acid disodium salt (3-PGA) | Sigma-Aldrich | P8877 | A component of the cell-free reaction Solution B |
| Dithiothreitol (DTT) | Sigma-Aldrich | 10197777001 | Component of buffers |
| E. coli 5-alpha | NEB | C2987 | for plasmid preparation |
| E. coli BL21 | NEB | C2530 | For preparation of cell-free lysates |
| E. coli BL21 (DE3) | NEB | C2527 | For expression of SpyCatcher2-Apex2 |
| EDTA-free protease inhibitor tablet | Sigma-Aldrich | 11836153001 | A component of E. coli lysis buffer |
| Eppendorf New Brunswick Innova 43/43R Incubator Shaker | Eppendorf | EPM1320 | Incubator for growing E. coli cells for protein expression and cell-free lysate preparation |
| Folinic acid | Sigma-Aldrich | 47612 | A component of the cell-free reaction Solution A |
| Glycerol | Sigma-Aldrich | G9012 | Component of SpyCatcher2-Apex2 storage buffer |
| Hemin chloride | Sigma-Aldrich | H9039 | For Heme supplementation of SpyCatcher2-Apex2 |
| Hydrogen peroxide 30% | Sigma-Aldrich | H1009 | For making ECL reagent |
| Image Lab Software | Bio-Rad | 1709690 | Software for ChemiDoc gel imaging instrument |
| Isopropyl-b-D-1-thiogalactopyranoside | Bioshop | IPT001 | For induction of SpyCatcher2-Apex2 expressikon |
| Kanamycin Sulfate | Sigma-Aldrich | 60615 | For preparation of SpyCatcher2-Apex2 bacterial culture |
| LB agar | BioShop | LBL406 | For E. coli growth |
| LB broth | BioShop | LBL407 | For E. coli growth |
| luminol | Sigma-Aldrich | A4685 | For making ECL reagent |
| Lysozyme | Sigma-Aldrich | L6876 | For lysis of bacterial cells |
| Magnesium Acetate | Sigma-Aldrich | M5661 | A component of the cell-free reaction Solution B |
| NEBExpress Ni resin | NEB | S1428S | For purification of SpyCatcher2-Apex2 |
| Non-fat dry milk | Bioshop | SKI400 | For blocking of WB and CFDB membranes |
| Parafilm | Bemis | 2099-1337410 | For ECL-incubation of CFDB blots |
| p-coumaric acid | Sigma-Aldrich | C9008 | For making ECL reagent |
| pET24b-SpyCatcher2-Apex2 plasmid | Pardee Laboratory | N/A | Used for the expression of SpyCatcher2-Apex2 protein |
| Petri dish | Fisherbrand | FB0875712 | Container for WB and CFDB membrane incubation |
| Phosphate buffered saline 10% | BioShop | PBS405 | Component of buffers |
| Potassium Glutamate | Sigma-Aldrich | G1501 | A component of the cell-free reaction Solution B |
| Potassium Oxalate Monohydrate (Oxalic acid) | Sigma-Aldrich | 223425 | A component of the cell-free reaction Solution A |
| Precast SDS-PAGE gel | BioRad | 4561036EDU | For electrophoretic analysis of protein |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification of LETs |
| QIAquick PCR purification kit | Qiagen | 28106 | For purification of LETs |
| Ribonucleotide Solution Set | NEB | N0450S | A component of the cell-free reaction Solution A |
| Skim milk powder | BioShop | SKI400 | Used as a blocking agent for western blot and CFDB |
| Sodium Chloride | Sigma-Aldrich | S9625 | A component of protein purification buffers |
| Sonicator | Qsonica | Q500 | For lysis of bacterial cells |
| Spermidine | Sigma-Aldrich | S2626 | A component of the cell-free reaction Solution A |
| Syringe filter | Sigma-Aldrich | SLGSR33SS | For sterilisation of buffers and solutions |
| Thermocycler | BioRad | T100 | Instrument for incubating PCR reactions |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8759 | A component of the cell-free reaction Solution A |
| Tris buffered saline | Thermo Scientific | J60764.K2 | Component of WB and CFDB wash buffer |
| Trizma base | Sigma-Aldrich | T1503 | For making tris buffer |
| Tunair SS-5012 Half-Baffle Shake Flask, 2.5 L | Cole-Parmer | RK-01835-39 | Vessel for growing E. coli cells for protein expression and cell-free lysate preparation |
| Tween-20 | BioShop | TWN510 | Component of WB and CFDB wash buffer |
| Tweezer | Almedic | 7728-A10-100 | For handling CFDB NC membrane |
| UV-Vis spectrophotometer | Thermo Scientific | 13400518 | Used for the measurement of nucleic acid and DNA concentration and bacterial culture OD |
| WHO International Reference Panel for anti-SARS-CoV-2 immunoglubulin | NISBC | 20/268 | Pre-characterised sera for verification and optimisation of CFDB reagents/conditions |
| β-Nicotinamide adenine dinucleotide hydrate (NAD) | Sigma-Aldrich | 10127965001 | A component of the cell-free reaction Solution A |
Referenzen
- Carter, L. J., et al. Assay techniques and test development for COVID-19 diagnosis. ACS Cent Sci. 6, 591-605 (2020).
- Amanat, F., et al. A serological assay to detect SARS-CoV-2 seroconversion in humans. Nat Med. 26 (7), 1033-1036 (2020).
- Stadlbauer, D., et al. SARS-CoV-2 seroconversion in humans: A detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol. 57 (1), e100(2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259.e12 (2016).
- Norouzi, M., et al. Cell-free dot blot: an ultra-low-cost and practical immunoassay platform for detection of anti-SARS-CoV-2 antibodies in human and animal sera. Microbiol Spectr. 11 (2), e0245722(2023).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Norouzi, M., Panfilov, S., Pardee, K. High-efficiency protection of linear DNA in cell-free extracts from Escherichia coli and Vibrio natriegens. ACS Synth Biol. 10 (7), 1615-1624 (2021).
- IDT codon optimization tool. , Integrated DNA Technologies. https://www.idtdna.com/pages/tools/codon-optimization-tool (2025).
- General protocol for western blotting. , Bio-Rad. https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6376.pdf (2025).
- Mruk, D. D., Cheng, C. Y. Enhanced chemiluminescence (ECL) for routine immunoblotting. Spermatogenesis. 1, 121-122 (2011).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-based cell-free protein synthesis: Protocols for a robust, flexible, and accessible platform technology. J Vis Exp. (144), (2019).
- Plasma and serum preparation. , ThermoFisher Scientific. https://www.thermofisher.com/ca/en/home/references/protocols/cell-and-tissue-analysis/elisa-protocol/elisa-sample-preparation-protocols/plasma-and-serum-preparation.html (2025).
- Mattiuzzo, G., et al. WHO/BS.2020.2403 Establishment of the WHO International Standard and Reference Panel for anti-SARS-CoV-2 antibody. , (2020).
- Gong, F., Wei, H., Li, Q., Liu, L., Li, B. Evaluation and comparison of serological methods for COVID-19 diagnosis. Front Mol Biosci. 8, 682405(2021).
- Peeling, R. W., et al. Serology testing in the COVID-19 pandemic response. Lancet Infect Dis. 20 (9), e245-e249 (2020).
- Smith, M. T., Berkheimer, S. D., Werner, C. J., Bundy, B. C. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. Biotechniques. 56 (4), 186-193 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten