Method Article
Investigando a função do gene alvo em um modelo de colite induzida por anticorpos agonísticos CD40 usando tecnologias baseadas em CRISPR/Cas9
Neste Artigo
Resumo
Aqui, descrevemos a metodologia para eliminar um gene de interesse no sistema imunológico usando tecnologias baseadas em repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR) / endonuclease associada a CRISPR (Cas9) e a avaliação desses camundongos em um modelo de colite induzida por anticorpos agonísticos de cluster de diferenciação 40 (CD40).
Resumo
O sistema imunológico funciona para defender os humanos contra invasores estranhos, como bactérias e vírus. No entanto, distúrbios do sistema imunológico podem levar à autoimunidade, doenças inflamatórias e câncer. As doenças inflamatórias intestinais (DII) - doença de Crohn (DC) e colite ulcerativa (UC) - são doenças crônicas marcadas por inflamação intestinal recorrente. Embora a DII seja mais prevalente nos países ocidentais (1 em 1.000), as taxas de incidência estão aumentando em todo o mundo. Por meio de estudos de associação, os pesquisadores ligaram centenas de genes à patologia da DII. No entanto, a patologia elaborada por trás da DII e o alto número de genes potenciais representam desafios significativos para encontrar os melhores alvos terapêuticos. Além disso, as ferramentas necessárias para caracterizar funcionalmente cada associação genética introduzem muitos fatores limitantes de taxa, como a geração de camundongos geneticamente modificados para cada gene. Para investigar o potencial terapêutico dos genes-alvo, um sistema modelo foi desenvolvido usando tecnologias baseadas em repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR) / endonuclease associada a CRISPR (Cas9) e um anticorpo agonístico de diferenciação 40 (CD40). O presente estudo mostra que a edição mediada por CRISPR/Cas9 no sistema imunológico pode ser usada para investigar o impacto de genes in vivo. Limitada ao compartimento hematopoiético, essa abordagem edita de forma confiável o sistema imunológico reconstituído resultante. Os camundongos editados por CRISPR/Cas9 são gerados mais rapidamente e são muito mais baratos do que os camundongos geneticamente modificados tradicionais. Além disso, a edição CRISPR/Cas9 de camundongos tem vantagens científicas significativas em comparação com a geração e criação de camundongos geneticamente modificados, como a capacidade de avaliar alvos embrionários letais. Usando CD40 como alvo modelo no modelo de colite induzida por anticorpos agonísticos CD40, este estudo demonstra a viabilidade dessa abordagem.
Introdução
As doenças autoimunes referem-se a condições nas quais o sistema imunológico de um paciente ataca suas próprias células e órgãos, resultando em inflamação crônica e danos aos tecidos. Quase 100 tipos diferentes de doenças autoimunes foram descritos até o momento, afetando 3-5% da população humana1. Muitas das condições autoimunes, incluindo lúpus eritematoso sistêmico e DII, carecem de tratamentos eficazes e apresentam necessidades médicas significativas não atendidas. Atualmente afetando cerca de 1,5 milhão de pessoas apenas nos EUA, a DII é uma doença devastadora marcada por inflamação intestinal progressiva, persistente e recidivante, sem cura disponível. Desvendar a patogênese e a fisiopatologia subjacentes é necessário para fornecer as novas estratégias de tratamento e prevenção que os pacientes com DII exigem 2,3.
Mais de 230 loci diferentes de DII foram identificados por meio de análises de associação genômica ampla (GWAS)4. Embora essas associações tenham elucidado novos genes que são potencialmente importantes nos principais mecanismos e vias da DII, apenas alguns genes desses loci foram estudados. Alguns genes foram implicados em vias específicas. Por exemplo, a via de detecção de micróbios tem sido associada à proteína 2 contendo domínio de oligomerização de ligação a nucleotídeos (NOD2); a via da autofagia tem sido associada à autofagia relacionada à 16 como 1 (ATG16L1), GTPase família M relacionada à imunidade (IRGM) e membro da família do domínio de recrutamento de caspase 9 (CARD9); e a via pró-inflamatória tem sido associada a respostas de células T impulsionadas por interleucina (IL)-234. Vários modelos de camundongos in vivo foram usados para caracterizar funcionalmente genes identificados por meio de GWAS 5,6.
Um dos principais modelos usados para estudar a patogênese da DII 7,8 é o modelo CD40 de colite, que induz inflamação intestinal imune inata após a injeção de um anticorpo agonístico CD40 em camundongos imunodeficientes (células T e B). Usado principalmente para examinar a contribuição da imunidade inata para o desenvolvimento da DII, principalmente macrófagos e células dendríticas9, não está claro se a doença pode ser induzida em camundongos do tipo selvagem (WT) totalmente imunocompetentes. Além dos modelos animais, ferramentas específicas de genes também são necessárias para a caracterização funcional de um gene, incluindo compostos químicos e biológicos. Mais importante, os animais geneticamente modificados são essenciais para revelar a função de um gene específico. No entanto, as estratégias normalmente usadas para fazer injeção e reprodução de embriões de camundongos geneticamente modificados geralmente levam mais de um ano e incorrem em um custo financeiro significativo. Esse processo de limitação de taxa apresenta um desafio significativo na busca para elucidar as funções dos genes relacionados à DII identificados pelo GWAS.
O protocolo aqui apresentado fornece uma alternativa viável à criação de camundongos geneticamente modificados. Primeiro, conforme mostrado no esquema da Figura 1 , as células de linhagem negativa, positivas para antígeno de células-tronco 1, positivas para receptor de tirosina quinase Kit (linhagem-Sca1 + c-Kit + ou LSK) são isoladas da medula óssea de camundongos Cas9 knockin (KI) portadores de um alelo específico (CD45.2) para permitir o rastreamento de células imunes do doador. Em seguida, essas células são expostas a lentivírus com diferentes RNAs guia (gRNAs) e um marcador fluorescente, proteína fluorescente verde excitada por violeta (VexGFP), para permitir o rastreamento de células transduzidas. Dois dias depois, as células VexGFP+ são classificadas e injetadas em camundongos Pep Boy receptores letalmente irradiados, que são camundongos C57Bl/6 portadores do alelo CD45.1 para permitir o rastreamento de células imunes do receptor. Doze semanas depois, o sistema imunológico é totalmente reconstituído e os camundongos podem ser inscritos em modelos in vivo.
Além do benefício de economia de custos e tempo de geração mais rápido em comparação com a geração e criação de animais geneticamente modificados, essa metodologia é ideal para alvos embrionários letais, pois visa especificamente o compartimento hematopoiético. Além disso, para alvos onde não há ferramentas disponíveis, como um anticorpo, este sistema fornece uma abordagem viável. Em resumo, para enfrentar os desafios descritos até agora, uma plataforma de edição de genoma baseada em CRISPR/Cas9 in vivo foi desenvolvida para gerar rapidamente modelos animais geneticamente modificados 10,11,12,13,14. Este estudo demonstra que a inflamação intestinal em camundongos WT C57Bl / 6 pode ser induzida por um anticorpo agonístico CD40. O CD40 é um regulador chave da doença neste modelo e, portanto, foi usado como um alvo modelo para validar o nocaute baseado em CRISPR / Cas9 e a perda da função do gene.
Protocolo
Todos os experimentos com animais realizados seguindo este protocolo devem ser aprovados pelo respectivo Comitê Institucional de Cuidados e Uso de Animais (IACUC). Todos os procedimentos descritos aqui foram aprovados pela AbbVie IACUC.
1. Geração de lentivírus necessários e colheita de animais doadores e receptores
NOTA: A Tabela de Materiais inclui detalhes de origem e número de pedido para todos os animais, instrumentos e reagentes usados neste protocolo.
- Construa os plasmídeos usando um vetor lentiGuide-puro modificado para VexGFP ou mCherry.
- Clone o gRNA não direcionado embaralhado (SgNone) ou o gRNA direcionado a CD40 no vetor modificado.
NOTA: Neste estudo, as sequências para cada gRNA foram as seguintes: SgNone, CTATGATTGCAACTGTGCAG; SgCD40.1, AGCGAATCTCCCTGTTCCAC; SgCD40.2, GACAAACAGTACCTCCACGA; e SgCD40.3, ACGTAACACACTGCCCTAGA. - Produzir as partículas lentivirais conforme descrito anteriormente15.
- Co-transfectar os plasmídeos codificadores de gRNA em células 293T com plasmídeos de empacotamento VSV-G e pLEX.
- Troque o meio de cultura após 18 h de transfecção e colete os sobrenadantes contendo o vírus após 24-48 h após a troca do meio.
- Para avaliar a eficiência do gRNA CD40, gere uma linhagem celular 293T estável de CD40 por transfecção de um plasmídeo pcDNA3.1 que codifica para CD40.
- Transfectar as células usando os lentivírus descritos e avaliá-las por classificação de células ativadas por fluorescência (FACS) duas semanas depois.
NOTA: Camundongos Cas9 KI foram usados como camundongos doadores. Certifique-se de que os camundongos Cas9 KI estejam em um fundo C57Bl/6 (e expressando o alelo CD45.2) ao usar camundongos C57Bl/6-Ly5.1 Pep Boy (expressando o alelo CD45.1) como receptores, para que a GvHD seja evitada e rastreie as células doadoras e receptoras.
- Clone o gRNA não direcionado embaralhado (SgNone) ou o gRNA direcionado a CD40 no vetor modificado.
- Projete o experimento. Para nocaute genético (KO), use 3-6 gRNAs por gene. Para cada gRNA, prepare 20% de camundongos receptores extras para contabilizar a perda de animais durante e após o transplante.
NOTA: O modelo de colite induzida por anticorpos agonísticos CD40 requer um mínimo de 8 camundongos por grupo para alimentação estatística. Assim, 10 camundongos por gRNA foram preparados neste estudo. O número de mouses necessários para outros modelos in vivo varia de acordo com o modelo usado. - Use camundongos doadores e receptores do mesmo sexo com 8 a 12 semanas de idade.
NOTA: Os camundongos foram sacrificados de acordo com as diretrizes aprovadas pela IACUC usando um mínimo de 5% de isoflurano e confirmados com luxação cervical como método secundário.
2. Colheita de medula óssea e preparação para triagem celular
- Usando fórceps e tesouras, colha ossos de cada camundongo doador (fêmur, tíbia, úmero e ulna), removendo cuidadosamente o máximo de músculo / tecido possível.
- Perfure o fundo de um tubo de microcentrífuga de 0,6 mL com uma agulha de 23 G e coloque o tubo dentro de um tubo de centrífuga de 1,5 mL. Faça 2 séries por animal.
- Corte uma extremidade dos ossos e coloque 4-8 ossos dentro do tubo de microcentrífuga com as extremidades abertas voltadas para o orifício da agulha de 23 G.
NOTA: O número de ossos por tubo depende do osso, por exemplo, 8 tíbias, úmeros e ulnas ou 4 fêmures cabem. - Centrifugue os tubos (com o tubo de microcentrífuga de 0,6 mL contendo os ossos dentro do tubo de centrífuga de 1,5 mL) a 300 × g por 2 min à temperatura ambiente (RT).
- Descarte o tubo de microcentrífuga de 0,6 mL, agora contendo ossos sem medula, deixando apenas o tubo de centrífuga de 1,5 mL agora cheio de medula. Adicione 1 mL de tampão de lise de glóbulos vermelhos (contendo cloreto de amônio) a cada tubo de centrífuga e ressuspenda o pellet celular por pipetagem. Incubar em RT por 1 min. Repita a etapa de lise, se necessário.
- Transfira a suspensão celular para um tubo cônico de 50 mL e adicione solução salina tamponada com fosfato de Dulbecco (DPBS) sem cálcio e magnésio (pelo menos o dobro do volume da suspensão celular) para neutralizar o tampão de lise. Células de pellets a 300 × g por 5 min. Aspirar completamente o sobrenadante.
- Carregue um filtro de 70 μm em um tubo cônico de 50 mL e filtre as células, usando DPBS para lavar o tubo e o filtro. Descarte o filtro e conte as células.
NOTA: O uso de 30 camundongos doadores (12 semanas de idade) produzirá 200-300 × 106 células em média.
3. Classificação de células para isolar células LSK para transdução
- Ressuspenda as células contadas na etapa 2.7 em tampão de classificação magnética de células (MACS) (DPBS, ácido etilenodiamina tetracético 2 mM, albumina sérica bovina a 0,5%, 10 μg / mL de penicilina-estreptomicina) no volume desejado (90 μL por 107 células).
- Adicione 10 μL de grânulos CD117+ por 107 células. Misture bem, proteja da luz e incube a 4 °C por 15 min.
- Prepare a coluna MACS LS colocando-a no campo magnético e enxaguando-a com tampão MACS.
- Lave as células da etapa 3.2 com um volume apropriado de tampão MACS (pelo menos o dobro do seu volume) e centrifugue a 300 × g por 10 min.
- Aspirar completamente o sobrenadante. Ressuspenda o pellet de modo que a concentração final seja de 10a 8 células em 500 μL de tampão MACS.
- Aplique a suspensão da célula na coluna LS. Lavar com 3 vezes com 3 ml de tampão MACS.
NOTA: Uma coluna LS pode ocupar até 108 células rotuladas e 2 × 109 células no total. - Remova a coluna do separador magnético e coloque-a em um tubo de coleta adequado. Adicione 5 mL de tampão MACS e lave imediatamente as células marcadas magneticamente empurrando firmemente o êmbolo para dentro da coluna.
- Pulverize as células a 300 × g por 10 min. Aspirar completamente o sobrenadante.
- Ressuspenda as células em 100 μL de tampão MACS por 107 células e adicione os anticorpos de coloração para linhagem (CD3, B220, Ter119, Gr-1, CD11b) e Sca-1.
NOTA: O uso de 30 camundongos doadores (12 semanas de idade) produzirá 100-180 × 106 células em média. - Misture bem, proteja da luz e incube a 4 °C por 20 min.
- Lave as células adicionando 5-10 mL de tampão MACS e centrifugue a 300 × g por 10 min. Aspirar completamente o sobrenadante.
- Ressuspenda até 108 células em 500 μL de tampão MACS
- Filtre as células com um filtro de células de 70 μm e execute FACS para a população de linhagem Sca1+.
NOTA: Aproximadamente 1,8-2,6% da população deve ser linhagem-Sca1+. - Colete as células em meio de cultura LSK (100 μL por 10.000 células): meio de expansão sem soro, 100 ng/mL de trombopoietina, fator de células-tronco de camundongo, ligante tirosina quinase 3 relacionado a Fms e IL-7 com 100 μg/mL de penicilina-estreptomicina.
- Células de pellets a 300 × g por 10 min. Aspirar completamente o sobrenadante.
4. Transdução e cultura de LSK para gerar células de controle e nocaute
- Ressuspenda as células em meio de cultura LSK.
- Semeie 10.000 células por poço em placas de cultura de tecidos (TC) de fundo plano de 96 poços em meio de cultura LSK.
- Incubar durante a noite a 37 °C com 5% de CO2 e 95% de humidade em condições assépticas.
- Prepare 50 μg / mL de solução de retronectina em DPBS e adicione 300 μL a cada poço de uma placa de poliestireno de 24 poços não tratada com TC. Incubar durante a noite a 4 °C.
- No dia seguinte, descarte a solução de retronectina, enxágue a placa revestida com 300 μL de DPBS e repita.
- Transfira 50.000 células LSK em 500 μL de meio (5 poços da placa de 96 poços na etapa 4.2) para cada poço revestido com retronectina da placa de poliestireno de 24 poços da etapa 4.4.Use 100 μL adicionais de meio LSK por 5 poços para enxaguar e coletar quaisquer células extras restantes na placa de 96 poços. Certifique-se de que 600 μL é o volume por alvéolo na placa de 24 poços após esta etapa (5 × 100 μL de poços da placa de 96 poços + 100 μL usados para lavar esses 5 poços).
NOTA: Após a transferência, olhe ao microscópio para a placa de 24 poços agora vazia para confirmar se todas as células foram coletadas. Use o meio LSK para coletar células que não foram transferidas para minimizar a perda celular dessa pequena população. - Adicione 300 μL de sobrenadante de vírus a cada poço e agite a placa a um valor de ajuste de 500 por 5 min. Girar a placa a 600 × g e 37 °C durante 20 min. Use pelo menos 5 × 106 partículas virais por mL.
NOTA: Neste estudo, foi utilizado um vetor modificado do pLentiPuro, no qual o elemento de resistência à puromicina foi trocado por VexGFP. O vírus foi adicionado a uma multiplicidade de infecção de 50-100. - Incubar por 1 h. Adicione 500 μL de meio LSK pré-aquecido.
- Incubar durante 2 dias a 37 °C com 5% de CO2 e 95% de humidade em condições assépticas.
5. Irradiação animal para se preparar para o enxerto de células-tronco do doador
- Após 2 dias de incubação, irradiar os animais receptores com 475 cGy duas vezes em um intervalo de 4 h. Após a segunda rodada de irradiação, coloque os animais em gaiolas autoclavadas e trate-os como animais imunodeficientes por 12 semanas. Os camundongos receberam enriquecimento, hidrogel e monitoramento lateral da gaiola para fornecer cuidados de suporte e intervenção conforme necessário. Os camundongos podem receber alimentos antibióticos ou água, além de cuidados de suporte e métodos de intervenção.
NOTA. As dosagens de irradiação podem diferir com diferentes irradiadores. Realize uma titulação da dose do irradiador para identificar a melhor dosagem. Doses entre 700 e 1300 cGy para camundongos C57Bl/6 são consideradas eficazes em vários exemplos da literatura17. Selecione 3-4 doses neste intervalo e avalie 5 camundongos por dose para sobrevivência e enxerto. Se o enxerto não for bem-sucedido ou a dose de radiação for muito alta, os camundongos não viverão mais de 3 semanas após o enxerto. Às 4 semanas após o enxerto, sangrar os camundongos sobreviventes para avaliar o enxerto por FACS. Realizamos coletas de sangue retro-orbital (RO) de ~ 50-100 μL por camundongo para avaliar o enxerto. Os camundongos estavam sob anestesia (2-3% de isoflurano) durante a coleta e receberam lubrificante ocular e 1,0 mL de solução salina subcutânea como cuidados de suporte após as coletas de sangue de RO.
6. Preparação e injecção de células em animais receptores irradiados
- Pipetar as células de cada poço a partir da etapa 4.9, mantendo os grupos separados agora que as células foram transduzidas com diferentes gRNAs, e pellet-as a 300 × g por 10 min.
- Ressuspenda as células no tampão MACS, filtre com um filtro de 70 μm e classifique a população VexGFP+.
NOTA: Aproximadamente 10-15% das células devem ser VexGFP+. - Pulverize as células a 300 × g por 10 min. Ressuspenda-os na solução salina balanceada de Hank: 5.000 ou mais células por camundongo em 200 μL.
NOTA: O HBSS é de grau não farmacêutico. Você também pode considerar o uso de uma solução salina estéril de grau farmacêutico para injeção. - Injete as células por via intravenosa 3 h após a última dose de irradiação.
NOTA: Doze semanas depois, os camundongos terão um sistema imunológico totalmente enxertado e poderão ser inscritos em modelos in vivo.
7. Modelo de colite induzida por anticorpos agonísticos CD40 em camundongos selvagens
NOTA: Pese e avalie os animais diariamente. Forneça cuidados de suporte conforme necessário: 1,0 mL de solução subcutânea de cloreto de sódio com perda de peso a 10% ou se estiverem desidratados. O controle positivo para este modelo é o anti-p40 administrado por via intraperitoneal a 25 mg / kg duas vezes por semana, começando no dia -1.
NOTA: Neste estudo, os grupos experimentais incluíram controle ingênuo, controle veicular (negativo) e controle anti-p40 (positivo). Juntos, esses grupos controlam o comportamento normal do modelo de colite induzida por anticorpos agonísticos CD40. Grupos vetorial, SgNone e SgRNA: Os controles vetoriais para o vetor lentiviral comum, SgNone é um controle guia não direcionado embaralhado, e os grupos SgRNA são os grupos de "tratamento" com expressão reduzida do gRNA alvo.
- Dia 0: Injetar anticorpo agonístico CD40 a 10 mg/kg por via intraperitoneal na sapb em todos os animais, exceto no controlo sem tratamento.
- Dias 3 e 6: Realizar videoendoscopia para avaliar a progressão da doença conforme descrito anteriormente16.
- Anestesiar os camundongos com 2-3% de isoflurano. Forneça suporte térmico por meio do mecanismo interno da máquina de anestesia e/ou almofada térmica externa durante a recuperação.
- Assim que os camundongos estiverem sob anestesia, administre um enema de DPBS (sem cálcio e magnésio) para preparar o cólon para endoscopia.
NOTA: Aproximadamente 1-2 mL são necessários para realizar um enema. - Após um enema e sob anestesia, mova o mouse para o cone do nariz da máquina de anestesia.
- Insira suavemente o endoscópio no cólon, avance lentamente para o cólon proximal enquanto mantém a câmera centralizada e o cólon inflado com ar (usando a bomba de ar incluída ou conecte um tubo/seringa para inflar manualmente) e, em seguida, retire lentamente o endoscópio e colete um vídeo e imagens a 3 cm, 2 cm e 1 cm do ânus.
NOTA: Para minimizar a irritação do cólon, lubrificante estéril pode ser adicionado à ponta do endoscópio. - Pontuar cada imagem individualmente com base no padrão vascular e espessamento da mucosa, conforme descrito na Tabela 1. Combine as pontuações das 3 imagens por rato para atribuir uma pontuação de soma a cada animal.
- Dia 7: Eutanásia de todos os camundongos por overdose de isoflurano ou câmara de CO2 e colete o máximo de sangue possível por punção cardíaca para soro. Pesar o baço para medir a esplenomegalia e coletar para citometria de fluxo e coletar o cólon para histopatologia.
NOTA: Seções de tecido fixadas em formalina e embebidas em parafina do cólon de camundongos foram preparadas e usadas para imuno-histoquímica (IHQ), conforme descrito anteriormente por Wang et al19.
Resultados
Seguindo o procedimento descrito acima, camundongos expressando gRNA direcionado a CD40 foram gerados. Na semana 2, células B, macrófagos CD11b+ e células dendríticas CD11c+ (DCs) foram enxertadas (Figura 2). As células T, no entanto, como esperado com base na literatura anterior18, levaram mais tempo para enxertar completamente e exigiram 12 semanas após o enxerto para atingir ~ 90% (Figura 2). Órgãos imunológicos, como o baço e os gânglios linfáticos, tinham a população mais notável de células derivadas de doadores; no entanto, outros órgãos, incluindo fígado, pulmão e intestino, também mostraram a presença de células doadoras (Figura 3). Uma forte redução na expressão de CD40 foi observada apenas em camundongos que expressam gRNA direcionado a CD40 (Figura 4).
Semelhante aos camundongos imunodeficientes, após a injeção de anticorpo agonístico CD40, os camundongos WT C57Bl / 6J exibiram perda de peso corporal, perda vascular e espessamento da mucosa observados por colonoscopia e infiltração de células mieloides determinada por IHQ com molécula adaptadora de ligação ao cálcio ionizado 1 (IBA1) (Figura 5). Além das leituras típicas para o modelo de colite induzida por anticorpos agonísticos CD40, também foi observada uma resposta imune adaptativa, conforme mostrado pela IHQ CD3 revelando infiltração de células T no cólon e pelas análises FACS revelando a ativação de células T e B por meio da regulação positiva de CD86 em esplenócitos (Figura 5). É importante ressaltar que um anticorpo monoclonal anti-p40 inibiu a indução da doença (Figura 5), o que foi consistente com os achados do modelo usando cepas imunodeficientes9.
Uma redução na expressão de CD40 por gRNA direcionado protegeu os camundongos da colite induzida por anticorpos agonísticos CD40. O grau de proteção correlacionou-se com a eficiência de edição de cada gRNA (Figura 6). Mais especificamente, SgCD40.1 foi o gRNA mais eficiente (Figura 4), o que levou à inibição mais poderosa da doença, conforme indicado pela regulação positiva de CD86 e infiltrado intestinal de células imunes (Figura 6D, E). Por outro lado, SgCD40.2 teve a menor eficiência de edição de todos os três gRNAs usados in vitro, o que resultou na menor proteção in vivo; SgCD40.3 exibiu edição e proteção intermediárias (Figura 6D, E). Em conjunto, os dados mostrados aqui revelam a viabilidade de usar CRISPR / Cas9 para reduzir a expressão de um alvo, que pode proteger com sucesso da indução de colite. Mais importante ainda, esses resultados demonstram que esta plataforma baseada em CRISPR/Cas9 in vivo pode ser usada para investigar a função do gene na patogênese da inflamação intestinal.
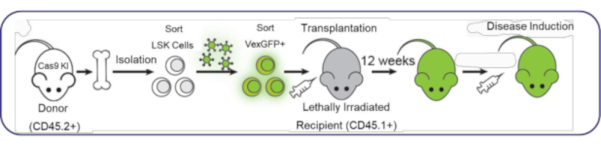
Figura 1: Estratégia de colheita, transdução e transplante de células-tronco usada para gerar camundongos editados por CRISPR/Cas9. Os ossos são colhidos de camundongos Cas9 KI, com o alelo CD45.2 como marcador de células doadoras. A medula óssea é isolada e as células são classificadas para células-tronco Lin-Sca1+c-Kit+ (LSK). Essas células são então expostas a vários lentivírus (controle vetorial, controle SgNone, SgCD40.1), todos com VexGFP como um marcador fluorescente para indicar transduzido. As células-tronco são então classificadas para células VexGFP + para injetar uma população pura de células-tronco editadas em camundongos receptores letalmente irradiados. Os camundongos receptores são camundongos Ly5.1 Pep Boy, que são camundongos C57Bl / 6 WT com o alelo CD45.1 como marcador de célula receptora. Abreviaturas: KI = knockin; VexGFP = proteína fluorescente verde excitada violeta; WT = tipo selvagem. Clique aqui para ver uma versão maior desta figura.

Figura 2: Diferenciação de subconjuntos de células imunes após transplante de LSK. Camundongos doadores CD45.2+ foram eutanasiados e suas células LSK foram isoladas e transplantadas em animais C57Bl/6 congênicos CD45.1 letalmente irradiados. A cada duas semanas após o transplante, uma pequena coorte de camundongos (n = 5) foi sacrificada e as taxas de enxerto no baço, medula óssea e sangue foram avaliadas por FACS. A porcentagem de subconjuntos de células doadoras em diferentes momentos pós-transplante é mostrada. Cada ponto representa um ponto de dados de um único animal. Abreviaturas: LSK = Lin-SCA1+C-Kit+; FACS = classificação de células ativadas por fluorescência; DC = célula dendrítica; EPM = erro padrão da média. Clique aqui para ver uma versão maior desta figura.

Figura 3: Distribuição de células diferenciadas por LSK do doador em animais reconstituídos. Os animais receptores foram reconstituídos usando células LSK infectadas com o vírus que expressa mCherry. Os tecidos foram colhidos na semana 12 pós-transplante e a expressão de mCherry (marrom) foi avaliada por IHQ: (A) baço, (B) linfonodos mesentéricos, (C) pulmão, (D) fígado, (E) intestino delgado, (F) intestino grosso, (G) rim, (H) pele. Essa figura foi modificada a partir de Wang et al.19. Barra de escala = 100 μm. Abreviaturas: LSK = Lin-SCA1+C-Kit+; IHQ = imuno-histoquímica. Clique aqui para ver uma versão maior desta figura.
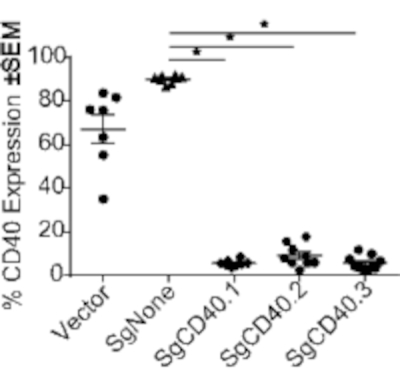
Figura 4: Redução mediada por CRISPR/Cas9 na expressão de CD40 em células B. Redução da expressão de CD40 em camundongos reconstituídos, modulada usando uma plataforma baseada em CRISPR/Cas9. Os esplenócitos foram avaliados 8 semanas após o transplante por FACS quanto à expressão de CD40 em células B. Cada ponto representa um animal individual. *p<0,005 Os dados mostrados são representativos de dois experimentos independentes. Abreviaturas: FACS = classificação de células ativadas por fluorescência; EPM = erro padrão da média; Sg = RNA guia único. Clique aqui para ver uma versão maior desta figura.

Figura 5: Inflamação intestinal induzida por anticorpos agonísticos CD40 em camundongos C57Bl / 6. O anticorpo agonístico CD40 foi injetado em camundongos C57Bl/6 para induzir inflamação, e a indução da doença foi avaliada com base em (A) alteração do peso corporal, (B) colonoscopia no dia 3 e no dia 6 após a injeção de anticorpo agonístico CD40, (C) porcentagem de áreas IBA1+ e CD3+ da área total da mucosa, espessura da mucosa, bem como porcentagem da área de células caliciformes da área total da mucosa, e (D) regulação positiva da expressão de CD86 em células B e T esplênicas. Em (B), são mostradas imagens representativas para a colonoscopia do dia 6. Em (C), são mostradas imagens representativas para a histologia do dia 7. Barra de escala = 100 μm. *p<0,001 Os dados são resultados representativos de dois experimentos independentes. Esta figura foi modificada de Wang et al19. Abreviaturas: IBA1 = molécula adaptadora de ligação ao cálcio ionizado 1; EPM = erro padrão da média. Clique aqui para ver uma versão maior desta figura.
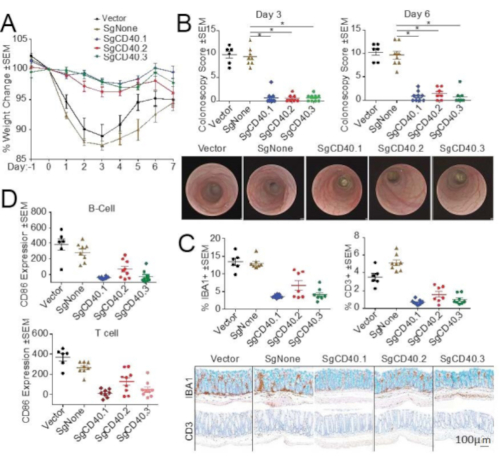
Figura 6: Nocaute baseado em CRISPR/Cas9 de CD40 melhora a patogênese da doença em um modelo de colite induzida por anticorpos agonísticos CD40. As células LSK foram infectadas com controle que expressa lentivírus ou SgCD40 e classificadas para VexGFP. As células VexGFP+ foram usadas para transplantar camundongos receptores CD45.1+ letalmente irradiados (n=10). Doze semanas após o transplante, a colite foi induzida pela injeção de anticorpo agonístico CD40. (A-D) A indução da doença foi avaliada por (A) alteração do peso corporal, (B) colonoscopia no dia 3 e no dia 6 após a injeção de anticorpos agonísticos CD40, (C) porcentagem de áreas de células IBA1+ e CD3+ da área total da mucosa, espessura da mucosa, bem como porcentagem da área de células caliciformes da área total da mucosa e (D) regulação positiva da expressão de CD86 em células B e T esplênicas. Imagens representativas de colonoscopia do dia 6 e (C) imagens histológicas do dia 7 estão incluídas. Cada ponto representa um animal individual. Barra de escala = 100 μm. *p<0,001. Os dados mostrados são representativos de dois experimentos. Esta figura foi modificada de Wang et al19. Abreviaturas: IBA1 = molécula adaptadora de ligação ao cálcio ionizado 1; LSK = Lin-Sca1+c-Kit+; VexGFP = proteína fluorescente verde excitada violeta; Sg = RNA guia único; EPM = erro padrão da média. Clique aqui para ver uma versão maior desta figura.
| Pontuação | Vasculatura | Engrossamento |
| 0 | Os vasos sanguíneos pequenos e grandes são brilhantes, nítidos e têm um padrão contínuo, | A superfície do cólon é lisa e brilhante. |
| 1 | Os vasos sanguíneos pequenos e grandes são visíveis, mas não se conectam e estão fora de foco. | A parede mucosa é menos transparente e ligeiramente irregular com uma camada mucosa brilhante. |
| 2 | Os grandes vasos sanguíneos ainda são visíveis, mas descontínuos, e vários pequenos vasos parecem ter estourado. | Há uma camada branca clara, brilhante e irregular cobrindo a maior parte da circunferência. |
| 3 | Nenhum vaso sanguíneo é visível e a superfície do cólon é muito irregular. | Há uma superfície opaca, branca e irregular cobrindo a circunferência. |
Tabela 1: Escala de pontuação de endoscopia de colite induzida por anticorpos agonísticos CD40. As imagens do cólon foram coletadas a 3 cm, 2 cm e 1 cm do ânus. Cada imagem foi então avaliada quanto à vascularização e espessamento, pontuando cada parâmetro de 0 a 3, conforme indicado. A pontuação total de todas as 3 imagens por animal foi então combinada para atribuir uma pontuação de soma endoscópica a cada animal.
Discussão
Os resultados mostrados aqui apresentam uma nova plataforma de edição de genoma baseada em CRISPR / Cas9 capaz de investigar a função do gene neste modelo de colite induzida por anticorpos agonísticos CD40. A classificação de células enriqueceu o conjunto de células LSK geneticamente modificadas, resultando em mais de 90% de redução na expressão de CD40 nos animais reconstituídos - em apenas 4 meses. Além disso, a expressão reduzida de CD40 no sistema imunológico teve um efeito profundo no modelo de colite induzida por anticorpos agonísticos CD40, reduzindo significativamente os desfechos da doença. Com base nesses resultados, foi estabelecida uma plataforma in vivo baseada em CRISPR/Cas9, que fornece aos pesquisadores uma ferramenta poderosa para estudar a função de um gene no sistema imunológico. Esse avanço tecnológico acelerará o processo de validação de novas biologias-alvo e, em última análise, a capacidade de fornecer terapias transformadoras para pacientes com DII e autoimunes necessitados.
A plataforma baseada em CRISPR/Cas9 apresentada aqui permite a modulação gênica eficiente e eficaz em camundongos WT C57Bl/6. Como a plataforma utiliza irradiação letal e transplantes de medula óssea, espera-se que os animais sejam perdidos antes do enxerto completo. Portanto, alimentar os grupos com 20% de ratos extras ajudará a contabilizar essas perdas. Como as células LSK isoladas após o tratamento com 5-fluorouracil reduziram a expressão do c-kit, recomenda-se a classificação celular para isolar LSKs. Além disso, o vírus de alto título deve ser usado após a concentração por ultracentrifugação. No entanto, apesar do alto título, os vírus não infectaram eficientemente as células LSK neste estudo. Talvez a ultracentrifugação tenha concentrado os fatores inibitórios, necessitando do uso de um gradiente de sacarose para melhorar a eficiência.
Normalmente, o modelo de colite induzida por anticorpos agonísticos CD40 é usado com camundongos imunodeficientes com gene ativador de recombinação (RAG) e imunodeficiência combinada grave (SCID), pois o valor primário no modelo é avaliar a imunidade inata em vez da imunidade adaptativa. Camundongos induzidos exibem perda de peso corporal, esplenomegalia, inflamação intestinal e infiltração de células mieloides no cólon. Demonstrado aqui, camundongos C57Bl / 6 do tipo selvagem respondem de forma semelhante ao anticorpo agonístico CD40 e ao controle positivo, anti-p40, que inibiu a doença. As duas principais diferenças entre os camundongos WT C57Bl / 6 e os camundongos imunodeficientes são 1) os camundongos WT C57Bl / 6 requerem o dobro da dose do anticorpo agonístico CD40 e 2) os camundongos WT C57Bl / 6 podem ter níveis reduzidos da doença no dia 6 em comparação com o dia 3, conforme medido por endoscopia. Geralmente, os camundongos imunodeficientes mantêm um nível semelhante de doença do dia 3 ao dia 6, às vezes mostrando exacerbação. Mais experimentos são necessários para elucidar definitivamente a causa dessa diferença, mas a hipótese aponta para células T reguladoras, que não estão presentes em camundongos imunodeficientes, limitando e/ou revertendo a doença uma semana após o agonismo de CD40.
Em conclusão, esta plataforma in vivo baseada em CRISPR/Cas9 utiliza e combina o transplante de LSK com a edição de genes CRISPR para reduzir com eficiência a expressão de genes-alvo no sistema imunológico. Com o potencial de editar vários genes em células LSK via CRISPR/Cas9 em conjunto, esta plataforma pode fornecer a oportunidade de avaliar fenótipos digênicos ou poligênicos observados em pacientes com DII. Fundamental para avaliar de forma eficiente e eficaz os genes ligados a pacientes com DII, esta plataforma acelera a capacidade de avaliar com precisão a função dos genes dentro do sistema imunológico, o que reduzirá o tempo desde a descoberta até o desenvolvimento de terapias que mudam a vida.
Divulgações
O desenho, a condução do estudo e o apoio financeiro para esta pesquisa foram fornecidos pela AbbVie. A AbbVie participou da interpretação dos dados, revisão e aprovação da publicação. Os autores declaram não haver conflito de interesses.
Agradecimentos
Obrigado a Ruoqi Peng, Donna McCarthy, Jamie Erikson, Liz O'Connor, Robert Dunstan, Susan Westmoreland e Tariq Ghayur por seus esforços em apoio a este trabalho. Obrigado aos líderes de farmacologia, incluindo Rajesh Kamath e outros, por sua liderança no estabelecimento do modelo de colite induzida por anticorpos agonísticos CD40 em camundongos WT C57Bl / 6. Além disso, obrigado a todos aqueles do AbbVie Bioresearch Center e do Cambridge Research Center no Departamento de Medicina Comparativa do Leste que apoiam experimentos in vivo.
Gostaríamos de agradecer ao laboratório Zhang do Broad Institute e ao McGovern Institute of Brain Research do Massachusetts Institute of Technology por fornecer reagentes CRISPR [multiplex Genome Engineering Using CRISPR/Cas Systems. Cong, L, Ran, FA, Cox, D, Lin S, Barretto, R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F Science. 3 de janeiro de 2013].
Materiais
| Name | Company | Catalog Number | Comments |
| 6-well tissue culture plates | Corning/Costar | #3506 | |
| TransIT-LT1 | Mirus Bio | MIR 2300/5/6 | |
| MACS Buffer (autoMACS Running Buffer) | Miltenyi Biotec | 130-091-221 | |
| 0.45 µm filter unit | Millipore | #SLHV013SL | |
| 0.6 mL microcentrifuge Tube | Axygen | MCT-060-C-S | |
| 1.5 mL Eppendorf Tube | Axygen | MCT-150-C-S | |
| 15mL Conical | VWR | 21008-918 | |
| 23 G Needle | VWR | #305145 | |
| 24 Well Non-TC Plates | Falcon | #351147 | |
| 24-Well TC Plates | Falcon | #353047 | |
| 50 mL Conical tube | VWR | 21008-951 | |
| 5 mL Syringe | BD Biosciences | #309647 | |
| 70 µm Filter | Miltenyi | #130-098-462 | |
| 96-Well Flat Bottom Plates | Corning | #3599 | |
| 96-Well U-Bottom Plates | Corning/Costar | #3365 | |
| Anesthesia Machine | VetEquip - COMPAC5 | #901812 | |
| Anti-CD40 Agonist monoclonal antibody | BioXcell | BE-0016 | |
| Anti-p40 monoclonal antibody | BioXcell | BE-0051 | |
| B220 PE Antibody | BioLegend | #103208 | |
| Bovine serum albumin | Sigma Aldrich | A7906-100G | |
| Cas9 Knock-in Mice | Jackson Labs | #026179 | C57Bl/6 background |
| CD117+ Beads | Miltenyi | #130-091-224 | |
| CD11b PE Antibody | BioLegend | #101208 | |
| CD3 PE Antibody | BD Biosciences | #553240 | |
| Centrifuge | Beckman Coulter | Allegra 6KR Centrifuge | |
| Countertop Centrifuge | Eppendorf | Centrifuge 5424 | |
| DPBS | ThermoFisher | #14190136 | |
| Dulbecco’s Modified Eagle Medium | Mediatech | #10-013-CV | |
| Ethylenediamine tetraacetic acid (EDTA) | Invitrogen | AM9260G | |
| Endoscope | Karl Storz | N/A | Custom Coloview Tower |
| Flow cytometer | BD Biosciences | FACS Aria II | |
| Fms-related tyrosine kinase 3 ligand (Flt-L) | PeproTech | #250-31L | |
| Gr-1 PE Antibody | BD Biosciences | #553128 | |
| Hank's balanced salt solution (HBSS) | ThermoFisher | #14170120 | |
| Heat-Inactivated Fetal Bovine Serum | HyClone | #SH30071.03 | |
| IL-7 | PeproTech | #217-17 | |
| Incubator | Binder | #9040-0116 | |
| Isoflurane | HenrySchein | #6679401710 | |
| LS Column | Miltenyi | #130-042-041 | |
| Ly5.1 Pepboy Mice | Jackson Labs | #002014 | C57Bl/6 background |
| mouse stem cell factor (mSCF) | PeproTech | #250-03 | |
| Sodium chloride (NaCl) | Hospira | #00409488850 | |
| OPTI-MEM serum-free media | Invitrogen | #31985-070 | |
| Penicillin-streptomycin (PenStrep) | ThermoFisher | #15140-122 | |
| Plate Shaker | ThermoFisher | #88880023 | |
| pLentiPuro | Addgene | #52963 | |
| Polybrene (10 µg/µL) | Sigma Aldrich | #TR-1003-G | |
| Red Blood Cell Lysis Buffer | eBioscience | #00-4333 | |
| Retronectin | Takarbio | #T100B | |
| Sca-1 APC Antibody | BioLegend | #108112 | |
| StemSpan | StemCell Technologies | #09600 | |
| Ter119 PE Antibody | eBioscience | #12-5921 | |
| Thrombopoietin (TPO) | PeproTech | #315-14 | |
| X-ray Irradiator | Precision X-Ray | X-Rad 320 |
Referências
- Wang, L., Wang, F. S., Gershwin, M. E. Human autoimmune diseases: a comprehensive update. Journal of Internal Medicine. 278 (4), 369-395 (2015).
- Uhlig, H. H., Powrie, F. Translating immunology into therapeutic concepts for inflammatory bowel disease. Annual Review of Immunology. 36, 755-781 (2018).
- Gajendran, M., Loganathan, P., Catinella, A. P., Hashash, J. G. A comprehensive review and update on Crohn's disease. Disease-a-Month. 64 (2), 20-57 (2018).
- Uhlig, H. H., Muise, A. M. Clinical genomics in inflammatory bowel disease. Trends in Genetics. 33 (9), 629-641 (2017).
- Sollid, L. M., Johansen, F. E. Animal models of inflammatory bowel disease at the dawn of the new genetics era. PLoS Medicine. 5 (9), 198 (2008).
- Jiminez, J. A., Uwiera, T. C., Douglas Inglis, G., Uwiera, R. R. Animal models to study acute and chronic intestinal inflammation in mammals. Gut Pathogens. 7, 29 (2015).
- Kiesler, P., Fuss, I. J., Strober, W. Experimental models of inflammatory bowel diseases. Cell Molecular Gastroenterology and Hepatology. 1 (2), 154-170 (2015).
- Mizoguchi, A. Animal models of inflammatory bowel disease. Progress in Molecular Biology and Translational Science. 105, 263-320 (2012).
- Uhlig, H. H., et al. Differential activity of IL-12 and IL-23 in mucosal and systemic innate immune pathology. Immunity. 25 (2), 309-318 (2006).
- Dow, L. E. Modeling disease in vivo With CRISPR/Cas9. Trends in Molecular Medicine. 21 (10), 609-621 (2015).
- Hochheiser, K., Kueh, A. J., Gebhardt, T., Herold, M. J. CRISPR/Cas9: A tool for immunological research. European Journal of Immunology. 48 (4), 576-583 (2018).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Fellmann, C., Gowen, B. G., Lin, P. C., Doudna, J. A., Corn, J. E. Cornerstones of CRISPR-Cas in drug discovery and therapy. Nature Reviews Drug Discovery. 16 (2), 89-100 (2017).
- Yin, H., et al. Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype. Nature Biotechnology. 32 (6), 551-553 (2014).
- Bagley, J., Tian, C., Iacomini, J. Prevention of type 1 diabetes in NOD mice by genetic engineering of hematopoietic stem cells. Methods in Molecular Biology. 2008 (433), 277-285 (2008).
- Becker, C., Fantini, M. C., Neurath, M. F. High resolution colonoscopy in live mice. Nature Protocols. 1 (6), 2900-2904 (2006).
- Duran-Struuck, R., Dysko, R. C. Principles of bone marrow transplantation (BMT): providing optimal veterinary and husbandry care to irradiated mice in BMT studies. Journal of the American Association for Laboratory Animal Science. 48 (1), 11-22 (2009).
- Haynes, B. F., Martin, M. E., Kay, H. H., Kurtzberg, J. Early events in human T cell ontogeny. Phenotypic characterization and immunohistologic localization of T cell precursors in early human fetal tissues. Journal of Experimental Medicine. 168 (3), 1061-1080 (1988).
- Wang, R., et al. CRISPR/Cas9-targeting of CD40 in hematopoietic stem cells limits immune activation mediated by anti-CD40. PLoS One. 15 (3), 0228221 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados