Method Article
Plugin open-source Micro-Manager pour l’imagerie en direct de dipôles fluorescents
Dans cet article
Résumé
Nous avons développé un plug-in open-source Micro-Manager, qui permet d’observer en direct les dipôles fluorescents sur un microscope à éclairage structuré. Le plugin prend en charge l’observation de l’orientation des dipôles 2D et 3D.
Résumé
La microscopie à polarisation de fluorescence (FPM) permet d’imager la position et l’orientation dipolaire des fluorophores. Malgré les réalisations de la microscopie à polarisation de fluorescence à super-résolution, leur dépendance à l’égard de la post-acquisition entrave l’observation en temps réel. La microscopie à éclairage structuré polarisé (pSIM) offre une imagerie à super-résolution des dipôles fluorescents avec une vitesse d’imagerie rapide et est bien adaptée aux applications de cellules vivantes. Nous avons développé une implémentation open-source pour la reconstruction en temps réel des images de polarisation et l’affichage des dipôles fluorescents. De plus, nous avons étendu la méthode pour obtenir une cartographie d’orientation 3D (3DOM), élargissant ainsi son utilité pour les études biologiques complexes. De plus, nous avons présenté une introduction approfondie à l’extension d’un microscope SIM existant sur l’imagerie polarisée et fourni un guide de configuration détaillé de Micro-Manager 2.0 pour contrôler le microscope, permettant une prévisualisation en temps réel de l’imagerie polarisée. De plus, nous avons fourni le code MATLAB pour une reconstruction complète, englobant à la fois la pSIM et la 3DOM. Ce guide complet vise à aider les débutants à maîtriser rapidement et à démarrer facilement les opérations.
Introduction
La microscopie à polarisation de fluorescence (FPM) est apparue comme une technique puissante pour imager simultanément la position et l’orientation dipolaire des fluorophores, offrant des informations approfondies sur l’imagerie biologique 1,2. En facilitant l’observation de la direction de l’orientation des biomolécules, le FPM dévoile l’arrangement complexe de macromolécules telles que l’actine 3,4,5, le microtubule5, la septine6, le filament d’ADN 7-9, le complexe de pores nucléaires10 et les protéines membranaires11. Ses capacités à grande vitesse, non invasives et compatibles avec les cellules vivantes permettent de suivre la dynamique de la rotation moléculaire avec une résolution temporelle élevée11,12. Lorsqu’il est intégré aux sondes de bioforce, le FPM cartographie non seulement les amplitudes de force à la résolution subcellulaire, mais mesure également les directions de force, faisant ainsi progresser notre compréhension des processus biomécaniques en révélant la direction des forces13.
Au cours des dernières décennies, la microscopie à polarisation de fluorescence à super-résolution a connu une évolution rapide. Une avancée notable dans ce domaine est la microscopie de localisation d’orientation d’une molécule unique, également appelée SMOLM, qui peut localiser à la fois la position et l’orientation des fluorophores, permettant ainsi une localisation multidimensionnelle. La mesure de polarisation dans SMOLM peut être effectuée à l’aide de la modulation d’excitation de polarisation7, de la détection polarisée multicanal3 ou de la fonction d’étalement du point sensible à la polarisation (PSF)14. Bien que SMOLM atteigne une résolution spatiale de l’ordre de quelques dizaines de nanomètres et mesure la polarisation de molécules uniques, il souffre d’un temps d’imagerie prolongé. Cela est dû aux cycles répétitifs de clignotement et de localisation des fluorophores, qui posent un défi pour l’imagerie à débit vidéo et les applications de cellules vivantes.
En revanche, la microscopie à éclairage structuré polarisé (pSIM) offre une résolution spatiale d’environ 100 nm, associée à l’acquisition d’informations de polarisation avec le même ensemble de données SIM. Notamment, la pSIM peut atteindre des vitesses d’imagerie à débit vidéo et est hautement compatible avec l’imagerie de cellules vivantes, sans exigences strictes sur les molécules fluorescentes. Récemment, la pSIM a réussi à révéler la structure du cycle d’actine dans le squelette périodique associé à la membrane (MPS)5 et a permis une cartographie à super-résolution des forces biologiques13.
Cependant, la pSIM nécessite une reconstruction d’image post-acquisition, ce qui empêche la visualisation en temps réel des résultats de polarisation. Ce retard entrave l’observation immédiate des phénomènes biologiques, empêchant les chercheurs de capturer rapidement les phénomènes biologiques d’intérêt et d’apporter des ajustements en temps réel aux échantillons et aux conditions d’imagerie. Pour remédier à cette limitation, nous avons développé une implémentation open-source qui facilite l’acquisition d’images et la reconstruction en temps réel et l’affichage des résultats de polarisation, basée sur la plate-forme ImageJ et Micro-Manager (https://github.com/KarlZhanghao/live-pol-imaging).
De plus, alors que la pSIM a été limitée à fournir des informations de polarisation 2D dans le plan, nous avons récemment étendu ses capacités pour réaliser une cartographie d’orientation 3D en utilisant presque le même équipement15, appelé cartographie d’orientation 3D (3DOM). Ce logiciel open-source permet également le contrôle, la reconstruction et la visualisation de 3DOM. Les modules de reconstruction et de visualisation sont également compatibles avec l’application de suivi de l’orientation d’une molécule unique. Toutes ces fonctionnalités renforcent l’utilité de l’imagerie de polarisation dans les études biologiques complexes.
Protocole
1. Extension d’un microscope à éclairage structuré existant pour l’imagerie de polarisation
- Préparez un microscope SIM.
REMARQUE : Nous supposons que les lecteurs ont une certaine base dans la configuration du microscope et possèdent déjà un microscope à lumière structurée. Si vous manquez d’expérience pertinente, reportez-vous à ce document, qui fournit une description détaillée de la façon de construire un microscope SIM16. L’unité de contrôle de polarisation de « HWP+WQP+LCVR » peut être remplacée par une plaque d’onde vortex pizza dans notre pSIM work5 ou une plaque demi-onde pizza17. - Mesurez les rapports d’extinction de polarisation de trois directions de bande en chargeant différents motifs sur le modulateur spatial de lumière (SLM). Placez et faites pivoter un polariseur juste après l’objectif et utilisez un wattmètre pour mesurer la puissance du laser. Pour la pSIM, faites pivoter la plaque d’onde pour maintenir la polarisation s des trois directions avec un rapport d’extinction > 10.
- Pour 3DOM, tournez le HWP2 pour maintenir la polarisation p de six directions. Remplacez le masque spatial SIM par le masque spatial 3DOM pour permettre le passage de faisceaux d’ordre 1 de six directions (voir Figure 1B).
REMARQUE : la polarisation s est requise dans la pSIM tandis que la polarisation p est requise dans le 3DOM. La configuration détaillée est incluse ailleurs pour la pSIM5 et la 3DOM15.
2. Configuration du Micro-Manager
- Téléchargez la version 2.0 de Micro-Manager depuis le site officiel, installez le logiciel en suivant les instructions.
- Préparez les pilotes de périphériques et les logiciels correspondants pour vous connecter avec succès au système Micro-Manager. Dans ce système de microscope, utilisez Micro-Manager pour contrôler la caméra, la platine de translation, le laser, la carte DAQ et le microscope.
REMARQUE : Les instruments pris en charge sont répertoriés dans https://micro-manager.org/Device_Support. Voir le tableau des matériaux pour plus de détails sur les instruments à connecter. Installez des pilotes ou des logiciels officiels/d’assistance et connectez l’instrument à l’ordinateur à l’aide d’un câble USB. - Ajoutez les instruments à Micro-Manager à l’aide de l’assistant de configuration matérielle.
- Ouvrez le logiciel et sélectionnez Aucun dans la liste déroulante initiale.
- Sélectionnez Appareils | Assistant de configuration matérielle... | Créez une nouvelle configuration et cliquez sur Suivant pour accéder à la page de configuration.
- Localisez le plug-in Appareil photo/Laser/Platine/DAQ/microscope dans la liste déroulante du matériel et cliquez sur Ajouter | D’accord | Ensuite.
- Allez dans Sélectionner les appareils par défaut et choisissez le réglage de l’obturateur automatique. Définissez l’appareil photo par défaut, l’obturateur par défaut et l’étape de mise au point par défaut. Cliquez sur Suivant jusqu’à ce que l’option Enregistrer la configuration et quitter soit atteinte, puis enregistrez le nom du fichier de configuration. Cliquez sur Terminer.
- Paramètres de modèle courants
- Enregistrez un modèle pour le mode en direct et le mode d’accrochage SIM.
- Réglez le temps d’exposition et le mode de déclenchement comme suit : choisissez le groupe ' + ' dans les paramètres de configuration, nommez le nom du groupe comme 'Mode', sélectionnez Exposition et mode de déclenchement, cliquez sur OK.
- Choisissez le préréglage « + » pour ajouter différents préréglages pour le groupe « Mode ». Nommez un préréglage « en direct » pour les paramètres d’exposition en mode en direct, et un autre « SIM » pour le mode SIM. Par exemple, en mode « live », réglez le temps d’exposition sur 15 ms, tandis que pour le mode SIM snap, réglez 10 ms pour le temps d’exposition et le mode de déclenchement est Standard (chevauchement).
REMARQUE : Un fichier de configuration (MMConfig_psim_demo.cfg) est inclus dans notre dépôt de code, qui peut être chargé directement par Micro-Manager 2.0.
3. Calibrage du système avec des billes fluorescentes
- Plongez les lamelles dans de l’éthanol à 75 %, rincez-les 3 fois avec de l’eau désionisée (ddH2O) et séchez-les.
- Vortex les billes fluorescentes de 100 nm (diluées à 1:1 000 avec une solution saline tamponnée au phosphate [PBS]) et pipetez la suspension directement sur les lamelles. Incuber pendant 5 à 10 minutes dans un environnement sombre, puis laver doucement avec du PBS.
- Ajoutez 20 μL de support de montage sur la lame de microscope et placez la lamelle par-dessus, en veillant à éviter toute bulle. Maintenez la température à 4 °C et gardez la lame dans l’obscurité, de sorte que la lame avec des billes fluorescentes de 100 nm soit préparée pour l’étalonnage du système.
- Pour la pSIM, acquérez trois images des trois phases différentes des billes fluorescentes. Un code MATLAB personnalisé (Bead_calib.m) prend les images en entrée et en émet deux images d’étalonnage (calib1.tif, calib2.tif) pour une analyse plus approfondie.
REMARQUE : Comme démontré précédemment5, l’expérience pSIM peut être effectuée sur la plupart des systèmes commerciaux. Sur les microscopes SIM fabriqués à la maison ou commerciaux, l’étalonnage du système est nécessaire pour éliminer l’erreur de mesure des dipôles fluorescents, causée par l’éclairage non uniforme pendant trois directions de motif. L’expérience d’étalonnage suppose la polarisation isotrope des billes fluorescentes et ne nécessite que les images brutes lors de l’acquisition de la carte SIM. La procédure d’étalonnage détaillée est incluse dans notre travail original5. La sortie de deux images d’étalonnage calib1.tif et calib2.tif est nécessaire pour la reconstruction pSIM, qui indique l’énergie d’éclairage au pixel près de la direction du motif 2 ; 3 fait référence à la direction du motif 1.
4. Préparation de l’échantillon : actine dans les cellules fixées
- Immergez les lamelles dans de l’éthanol à 75 %, rincez les lamelles 3 fois avec du ddH2O. Placez les lamelles dans une plaque de culture cellulaire à six puits.
- Cultiver des cellules U2OS (lignée cellulaire ATCC HTB-96) dans le milieu modifié de Dulbecco (DMEM) avec 10 % (v/v) de sérum fœtal bovin (FBS) à 37 °C et 5 % de CO2 sur des lamelles de recouvrement jusqu’à ce qu’elles atteignent une confluence d’environ 75 %.
- Lavez doucement les cellules 3x avec du PBS. Pour la fixation cellulaire, appliquer 4 % de paraformaldéhyde pendant 15 min à température ambiante. Après la fixation, lavez à nouveau les cellules 3 fois avec du PBS.
- Pour améliorer la perméabilité de la membrane, traitez les cellules avec 0,1 % de Triton X-100 dans du PBS pendant 5 min, puis lavez les cellules 3x avec du PBS.
- Dissoudre la phalloïdine fluorescente avec 150 μL de DMSO pour solution mère (66 μM). Préparez la solution de travail du colorant phalloïdine en diluant 5 μL de la solution mère dans 995 μL de PBS. Incuber les cellules dans l’obscurité avec une solution de travail de colorant Phalloïdine à température ambiante pendant ~1 h. Lavez les cellules pendant 3 x 3 min avec du PBS.
- Ajoutez 20 μL de support de montage sur la lame de microscope. Placez soigneusement la lamelle sur la glissière sans aucune bulle. Rangez la lame à 4 °C et conservez-la dans un endroit sombre.
5. Préparation de l’échantillon : λ-ADN in vitro
- Dissoudre 0,5 g de polyméthacrylate de méthyle dans 10 mL de matériaux de base de prothèse. Après une dissolution complète, appliquez uniformément la solution sur la surface de la lame, puis attendez qu’elle s’évapore pour former un film.
- Diluez 0,3 μL d’ADN-λ et 32 μL de colorant d’acide nucléique orange 1 000 fois SYTOX dans 968 μL de PBS. Ajoutez 1 μL de la solution préparée à la lame et laissez-la sécher.
- Scellez la lamelle sur la lame avec le collant pour protéger et préserver l’échantillon. Stockez la lame à 4 °C dans un environnement sombre.
6. Imagerie
- Placez la lamelle sur le porte-échantillon et ajustez-la sur le plan focal en mode direct. Sélectionnez un retour d’intérêt de 512 x 512 pixels et ajustez le temps d’exposition et l’intensité du laser.
- Ouvrez le logiciel de contrôle indépendant du modulateur de lumière spatial et choisissez le chemin de séquence de motif d’éclairage approprié pour pSIM / 3DOM ou le mode en direct.
REMARQUE : Pour pSIM, le motif d’éclairage comprend trois angles de polarisation différents et trois phases différentes. Pour 3DOM, le motif d’éclairage comprend six angles de polarisation différents. - Sélectionnez le mode de capture SIM , sélectionnez Multi-D Acq., choisissez Points temporels et réglez le nombre (9 ou 6 images pour pSIM ou 3DOM) et l’intervalle (0 ms) dans la fenêtre ouverte. Cliquez sur Acquérir !
- Cliquez sur TriggerSLM.bsh (un fichier de configuration est inclus dans notre référentiel de code), la carte d’acquisition MyDaq active le modulateur de lumière spatial pour prendre les photos.
7. Plugin de visualisation en direct de pSIM et 3DOM
- Configurer le chemin d’accès à la FakeCamera et à l’image : chargez le certificat (MMConfig_FakeCam_demo.cfg) en cliquant sur Périphériques | Charger la configuration matérielle.... Configurer le répertoire local des images dans Périphériques | Périphériques Navigateur de propriétés... | Masque de trajectoire de caméra (Figure 2A-C).
REMARQUE : La partie acquisition du script dépend de la configuration matérielle, nous avons donc fourni une version (MMConfig_FakeCam_demo.cfg) utilisant le périphérique FakeCamera pour lire les images locales sur le disque dur à des fins de démonstration. Les utilisateurs peuvent apporter des modifications dans notre version. N’oubliez pas de modifier le chemin de lecture de l’image (masque Camera-Path). - Configurez le panneau d’accès rapide en cliquant sur Outils | Panneau d’accès rapide | Créez un nouveau panneau. Cliquez sur l’icône Paramètres dans le coin inférieur gauche, puis sur « Run Script » dans la partie supérieure, ce qui permet de charger les fichiers « psim.bsh » et « 3DOM.bsh » (Figure 2D).
REMARQUE : Le système de panneau d’accès rapide permet aux utilisateurs de créer des fenêtres personnalisées et d’accéder facilement aux commandes de Micro-Manager, ce qui est le plus fréquemment nécessaire. Il est également possible d’ajouter des canaux « Live », « Snap » ou d’autres canaux, comme le montre la figure 2D. - En prenant l’exemple de la pSIM, effectuez une reconstruction d’image de polarisation en temps réel en cliquant sur 'psim.bsh' ; observer les images reconstruites en temps réel avec les résultats de polarisation (Figure 2E). N’oubliez pas de modifier le chemin d’accès aux images d’étalonnage dans le fichier 'psim.bsh' (calibPath = 'chemin d’accès au fichier d’images d’étalonnage'").
REMARQUE : Le plugin de vue en direct est un script beanshell (psim.bsh et 3DOM.bsh), qui peut être exécuté dans le panneau de script. Le plugin acquiert neuf images pour la pSIM ou six images pour le 3DOM et calcule les résultats de la cartographie d’orientation sur la base des images à grand champ. Les images de cartographie d’orientation sont affichées pour fournir des informations de polarisation en temps réel, ce qui facilite l’inspection immédiate, malgré sa faible résolution.
8. Analyse des données avec reconstruction en super-résolution
- Installez MATLAB R2019b à partir du site Web (voir la table des matériaux).
- Ouvrez le logiciel ; pour la reconstruction pSIM, obtenir neuf images, composées de trois phases différentes pour chacun des trois angles d’orientation. Assurez-vous que les noms des neuf images sont compris entre 1.tif et 9.tif, placez-les dans le fichier nommé « Input » et exécutez le programme nommé « PSIM.m ». Observez l’image grand champ reconstruite, l’image SIM et l’image pSIM avec la roue chromatique.
REMARQUE : La reconstruction de la pSIM nécessite deux étapes : une étape SIM et une étape PM. Les données sont analysées par un programme MATLAB personnalisé pour faciliter le débogage. Obtenez le code de reconstruction à partir du dossier 'reconsr' dans GitHub. - Pour 3DOM, capturez six figures de directions de polarisation différentes. Fusionnez les six images, nommez le fichier résultant 'Raw_data.tif' et exécutez le programme nommé 'Recon_3DOM.m'. Observez l’image grand champ reconstruite et l’image 3DOM avec les résultats azimutaux et les résultats de l’angle polaire.
REMARQUE : Pendant le processus de reconstruction, une erreur peut se produire'--'la fonction ou la variable 'bfopen' n’est pas reconnue'. 'bfopen' est une fonction de la bibliothèque Bio-formats et est couramment utilisée pour l’ouverture et la lecture de fichiers image. Entrez addpath ('path_to_bioformats_toolbox') dans la fenêtre de commande MATLAB. Remplacez 'path_to_bioformats_toolbox' par le chemin d’accès réel du dossier de la bibliothèque. Nous avons également ajouté la dépendance Bio-Formats au référentiel GitHub.
Résultats
La méthode pSIM peut être réalisée sur des microscopes SIM en fonction de l’interférence à l’aide de faisceaux laser polarisés s. Les interférences de polarisation S sont le type de carte SIM le plus largement utilisé et génèrent des bandes d’éclairage à contraste élevé. Le prototype académique d’une configuration de microscope est inclus dans le travail original de pSIM5. Brièvement présenté dans la figure 1, un modulateur spatial de lumière (SLM) génère l’ordre ±1 des faisceaux diffractifs et une plaque demi-onde à pizza maintient la polarisation s de trois directions. Alors que d’autres alternatives SIM peuvent utiliser des réseaux pour générer des faisceaux diffractifs ou utiliser d’autres méthodes de modulation de polarisation pour maintenir la polarisation s, ces microscopes peuvent également être étendus pour l’imagerie de polarisation.
Les microscopes 3DOM utilisent un système d’imagerie optique similaire (Figure 1A) avec deux différences. Tout d’abord, le 3DOM ne nécessite pas l’interférence de deux faisceaux laser. Au lieu de cela, il n’utilise que les faisceaux +1 pour contrôler la polarisation de l’excitation. Comme démontré dans le travail original15, 3DOM nécessite un minimum de six directions pour calculer l’orientation du dipôle 3D. Par conséquent, le masque spatial 3DOM contient six directions, chacune avec un sténopé (voir Figure 1B). Une autre différence est que le 3DOM utilise des faisceaux laser polarisés p, ce qui peut être réalisé en faisant pivoter le HWP2. Le SLM génère une lumière diffractée à divers angles obliques pour modifier la partie de la polarisation p.
Dans le système pSIM, la lumière sortante est modulée en lumière polarisée s en tournant l’angle des plaques demi-onde. Le masque de la pSIM est symétrique, ce qui permet à la lumière d’ordre ±1 de passer simultanément. À l’inverse, en 3DOM, en ajustant l’angle de la plaque demi-onde, le laser est transformé en lumière polarisée p. Le masque de 3DOM n’est pas asymétrique et ne laisse passer que +1 ordre de lumière. En ajustant la fréquence de bande du SLM, il modifie l’angle de diffraction des faisceaux laser. Ainsi, la partie de polarisation hors plan est modulée.
En analysant deux dipôles fluorescents adjacents qui partagent le même angle azimutal mais des angles polaires différents, la pSIM et la 3DOM peuvent distinguer les dipôles proches dans la limite de diffraction. De plus, 3DOM peut distinguer efficacement différents dipôles avec une orientation 3D dans le domaine de polarisation (Figure 1C,D). En effet, la technologie 3DOM peut obtenir plus d’informations spatiales en modifiant l’état de polarisation de la lumière excitée pour obtenir une mesure précise de l’orientation du dipôle dans l’espace 3D. En revanche, la technologie pSIM repose principalement sur les informations de polarisation 2D et ne peut pas tirer pleinement parti des caractéristiques d’orientation 3D ; Il ne fournit donc pas d’informations sur l’orientation des dipôles dans le sens vertical.
Comme les résultats de la cartographie d’orientation nécessitent un post-traitement, nous avons développé et présenté ici le 'Live-view plugin' ('psim.bsh' et '3DOM.bsh') pour Micro-Manager 2.0. Pour permettre aux utilisateurs d’ordinateurs sans caméra réelle connectée d’apprendre à utiliser le plugin, nous configurons d’abord Micro-Manager avec FakeCamera.In figure 2, nous avons détaillé le processus opérationnel pour que la pSIM obtienne des effets de polarisation en temps réel, et les étapes opérationnelles pour 3DOM sont similaires. La figure 2A présente l’interface logicielle de Micro-Manager 2.0, ainsi que les étapes de configuration nécessaires à la configuration de notre plugin. Dans la figure 2B, nous montrons la configuration matérielle à l’aide des appareils de démonstration de FakeCamera et DStage. La configuration matérielle de FakeCamera est capable de simuler divers comportements d’un appareil photo réel, tels que l’exposition, la mise au point et la balance des blancs, permettant ainsi aux développeurs d’effectuer des développements et des tests de logiciels sans avoir besoin d’un appareil photo réel. Les utilisateurs doivent modifier le chemin de lecture de l’image en fonction des circonstances réelles, comme le montre la figure 2C. La figure 2D affiche un panneau d’accès rapide, qui peut contenir des raccourcis vers une série de fonctions ou de paramètres couramment utilisés, permettant aux utilisateurs d’accéder à ces fonctions et de les utiliser plus rapidement. Les plugins de prévisualisation en temps réel, 'psim.bsh' et '3DOM.bsh', peuvent être chargés à partir de ce panneau. Les étapes opérationnelles spécifiques sont décrites en détail à la section 7 du protocole. Les scripts 'psim.bsh' et '3DOM.bsh' sont utilisés pour générer instantanément les images de cartographie d’orientation. Ce plugin 'psim.bsh' permet une visualisation immédiate des effets de polarisation pendant l’étape de capture d’image pSIM. La figure 2E illustre un résultat typique de l’imagerie de polarisation de la pSIM. La roue chromatique indique la relation entre l’orientation du dipôle et la pseudo-couleur, ce qui permet aux chercheurs de prendre des décisions immédiates sur les images à capturer en fonction des données de polarisation affichées.
Les cellules U2OS ont été marquées à la phalloïdine et le processus de reconstruction d’image de la pSIM a été réalisé à l’aide de MATLAB. Neuf photos ont été capturées à partir de trois états de polarisation différents et de trois phases différentes. Les doubles flèches vertes marquent la direction de la lumière polarisée sur la figure 3A. À partir de l’intensité de la lumière et de l’obscurité de l’image, l’orientation de polarisation des dipôles fluorescents peut être déterminée.
Dans la figure 3B, la figure de gauche présente les résultats de super-résolution de la carte SIM, tandis que la figure de droite présente les résultats de la pSIM. Notamment, la pSIM atteint la même capacité de résolution spatiale que la SIM et offre une résolution spatiale deux fois supérieure à celle de la microscopie à grand champ. En cartographiant des couleurs spécifiques à des directions distinctes des filaments d’actine, la roue chromatique fournit une représentation visuelle qui nous permet d’interpréter l’alignement des dipôles fluorescents dans les filaments d’actine de la cellule. La couleur du filament d’actine montre que les dipôles sont approximativement alignés avec l’orientation du filament.
La figure 4A montre la distribution de l’intensité d’émission sous polarisation, modulation d’excitation ; 3DOM peut produire six modulations de polarisation différentes. Dans cette configuration, la lumière excitée de la molécule fluorescente est modulée en modifiant l’état de polarisation, affectant ainsi l’intensité d’émission de la molécule. Cela permet aux chercheurs de déduire l’orientation des molécules en mesurant l’intensité de la fluorescence dans différents états de polarisation. La figure 4B montre l’intensité de fluorescence de l’ADN-λ sous différents états de polarisation dans la figure 4A. Les molécules d’ADN-λ présentent des intensités de fluorescence différentes dans différents états de polarisation, ce qui indique que l’orientation du dipôle de fluorescence et de l’environnement a une influence significative sur l’intensité du signal de fluorescence. La figure 4C présente une imagerie à super-résolution de l’ADN-λ réalisée par 3DOM, présentant à la fois les résultats azimutaux et les résultats de l’angle polaire. Nos observations révèlent que les molécules de colorant étaient alignées presque perpendiculairement aux brins d’ADN. Comme l’axe de l’ADN se plie, l’orientation moyenne des molécules de colorant tournera en conséquence. Les résultats de nos mesures d’orientation valident l’insertion de fluorophores STOYOX Orange entre les bases d’ADN adjacentes, et l’alignement du moment dipolaire d’absorption est perpendiculaire à l’axe de l’ADN.
En 3DOM, une lumière d’excitation inclinée est donc nécessaire pour obtenir les informations de l’axe z de la molécule cible. Dans notre système, SLM est utilisé pour réaliser le réglage de la lumière d’éclairage. Combiné avec les caractéristiques du SLM, si le SLM est chargé avec un motif rayé, la lumière sortant du SLM sera composée de 0 et ±1 ordres de lumière (et les ordres de lumière +1 et -1 sont symétriques). Par conséquent, nous utilisons la lumière d’ordre +1 comme lumière d’excitation et utilisons le masque asymétrique pour filtrer la lumière d’ordre 0 et -1. De plus, la lumière d’excitation oblique peut être réalisée non seulement en utilisant le SLM, mais aussi en utilisant un galvanomètre à balayage ou en déplaçant latéralement la lentille devant la lentille de l’objectif.
L’utilisation de six états de polarisation d’excitation différents dans 3DOM est dictée par son principe15. En ajustant le modèle d’imagerie avant, le modèle obtenu se compose de six parties. Par conséquent, il n’est pas possible d’utiliser moins de six ajustements des états de polarisation, et un excès de six entraînerait une redondance de l’information. Le principe spécifique du 3DOM est présenté ci-dessous.
En coordonnées sphériques, nous caractérisons la molécule fluorescente à l’aide de l’ange azimutal ρ et de l’angle polaire η comme suit :

ρ est l’angle dans le sens des aiguilles d’une montre entre la projection de la molécule sur le plan x-y et l’axe x, et η est l’angle entre la molécule et l’axe z.
La lumière d’excitation polarisée p est caractérisée comme suit :
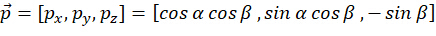
Où α et β représentent respectivement l’angle azimutal et l’angle d’incidence oblique de la lumière d’excitation. L’efficacité d’absorption des photons est 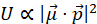 , et en supposant que l’émission de w dipôles dans le plan focal est capturée dans le nième pixel, alors le photon fluorescent émis par le dipôle est noté :
, et en supposant que l’émission de w dipôles dans le plan focal est capturée dans le nième pixel, alors le photon fluorescent émis par le dipôle est noté :


Où C représente l’irradiance de l’échantillon et  représente l’état de polarisation de la lumière d’excitation comme décrit comme suit :
représente l’état de polarisation de la lumière d’excitation comme décrit comme suit :
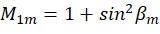


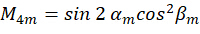


En combinant le coefficient 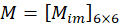 , l’orientation
, l’orientation  du nième pixel peut être résolue en prenant les images
du nième pixel peut être résolue en prenant les images  acquises comme entrée comme suit :
acquises comme entrée comme suit :
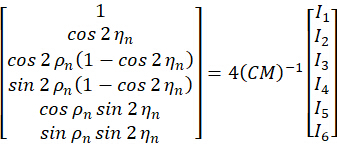
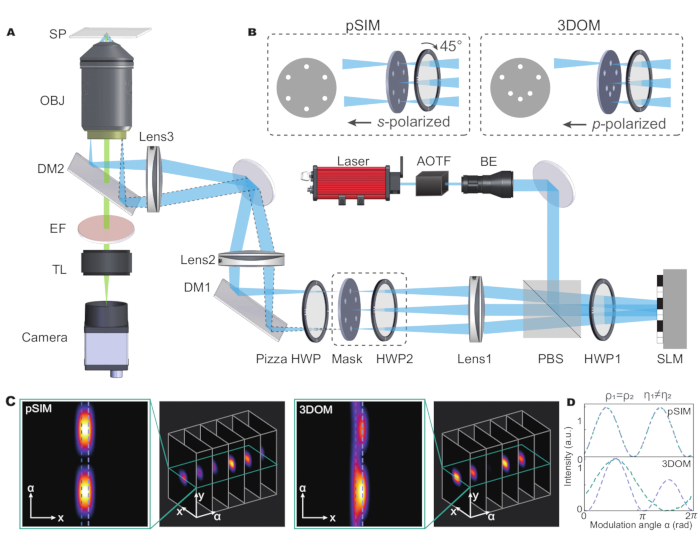
Figure 1 : Illustration schématique des microscopes pSIM et 3DOM. (A) Montage expérimental pour pSIM et 3DOM. (B)Les principales différences entre les systèmes pSIM et 3DOM sont le masque et HWP2. Le masque de la pSIM garantit que la lumière d’ordre ±1 passe simultanément, et que la lumière sortante est polarisée en S en tournant le HWP2. Le masque de 3DOM ne passe que par la lumière d’ordre +1, et la lumière sortante est polarisée p. (C, D) Comparaison des variations d’intensité de fluorescence d’émission entre pSIM et 3DOM. Abréviations : pSIM = microscopie à éclairage structuré polarisé ; 3DOM = cartographie d’orientation 3D ; AOTF = filtre accordable acousto-optique ; BE = expanseur de faisceau ; PBS = séparateur de faisceau de polarisation ; HWP = plaque demi-onde ; SLM = modulateur spatial de la lumière ; DM = miroir dichroïque ; OBJ = objectif ; SP = plan d’échantillonnage ; EF = filtre d’émission ; TL = lentille du tube. Cette figure a été modifiée à partir de Zhanghao et al.5 et Zhong et al.15. Veuillez cliquer ici pour voir une version agrandie de cette figure.
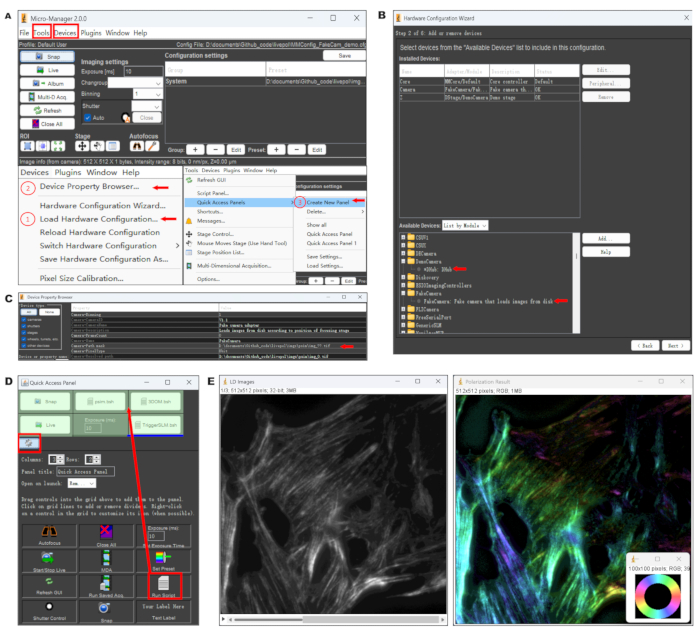
Figure 2 : Capture d’écran du logiciel et résultats représentatifs de la pSIM. (A)L’interface de Micro-Manager 2.0. Certaines options peuvent être utilisées pour configurer les fichiers. (B) Configuration du plugin de visualisation en direct avec FakeCamera et DStage. (C)Interface de sélection du chemin de lecture d’image (masque de chemin). (D) Configuration du panneau d’accès rapide et chargement de 'Run Script' avec 'psim.bsh' et '3DOM.bsh'. (E) La reconstruction d’image en temps réel démontre les effets de polarisation. Les orientations dipolaires sont pseudocolores. La roue chromatique indique la relation entre l’orientation du dipôle et la pseudo-couleur. Abréviations : pSIM = microscopie à éclairage structuré polarisé ; 3DOM = cartographie d’orientation 3D. Veuillez cliquer ici pour voir une version agrandie de cette figure.
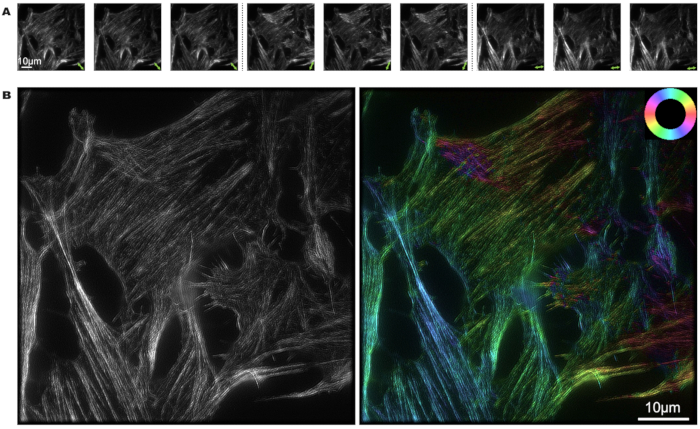
Figure 3 : Imagerie pSIM des filaments d’actine dans une cellule U2OS marquée à la phalloïdine. (A) Modulation de l’état de polarisation de la lumière d’excitation 3x dans la pSIM, capturant trois phases distinctes pour chaque état de polarisation. Les doubles flèches indiquent la direction de la lumière polarisée. (B) Image SIM et pSIM d’un filament d’actine en A, où l’image de gauche montre les résultats de la SIM et l’image de droite montre les résultats de la pSIM avec l’angle de polarisation. La roue chromatique positionnée en haut à droite illustre la relation entre la pseudo-couleur et l’orientation dipolaire. Barres d’échelle = 10 μm. Abréviation : pSIM = microscopie à éclairage structuré polarisé. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : SYTOX Marqué en orange Adn-λ marqué en orange imagé par 3DOM. (A) Modulation de l’état de polarisation de la lumière d’excitation 6x dans 3DOM. (B) La distribution de l’intensité d’émission sous modulation d’excitation de polarisation de A. (C) Image 3DOM d’un brin d’ADN-λ, où l’image de gauche montre les résultats azimutaux et l’image de droite montre les résultats de l’angle polaire. Barres d’échelle = 10 μm. Abréviation : 3DOM = cartographie d’orientation 3D. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Dans notre étude, nous avons développé un plugin qui permet de prévisualiser en temps réel deux techniques d’imagerie de polarisation, pSIM et 3DOM. Les deux technologies peuvent être réalisées dans un système SIM existant avec de légères modifications. Nous avons fourni les étapes détaillées pour installer le microscope pSIM et 3DOM et configurer Micro-Manager pour contrôler le microscope et montrer comment obtenir les résultats de polarisation en direct. Les résultats expérimentaux incluent le filament d’actine imagé par pSIM et l’ADN-λ imagé par 3DOM. L’orientation des dipôles de la phalloïdine est à peu près parallèle à la direction des filaments d’actine, tandis que l’orientation des fluorophores STOYOX Orange est perpendiculaire à l’axe de l’ADN. La microscopie à illumination structurée peut également réaliser une imagerie à haute résolution spatio-temporelle de l’appareil de Golgi, du réseau de microtubules18 et du réticulum endoplasmique19.
Nous avons développé des plugins personnalisés pour pSIM et 3DOM ; Les utilisateurs peuvent choisir le programme de prévisualisation approprié pour leur mode de prise de vue. Au cours du processus d’exécution du plug-in de prévisualisation en temps réel, si le plug-in ne démarre pas correctement, les utilisateurs doivent d’abord vérifier le paramètre de chemin dans le fichier de configuration concernant la lecture des images de la caméra virtuelle, en s’assurant que le chemin a été modifié avec précision à l’emplacement réel où les images sont stockées. Par la suite, il est essentiel de s’assurer que le chemin de lecture des images d’étalonnage dans le plugin de prévisualisation en temps réel a également été mis à jour en conséquence. Différentes versions de Micro-Manager peuvent introduire de nouvelles fonctionnalités, mais elles peuvent également entraîner des problèmes de compatibilité. Par conséquent, des opérations spécifiques doivent être ajustées en fonction de la version réelle utilisée.
La limite de la technologie réside dans le fait que, bien que les plugins de prévisualisation en temps réel actuels nous permettent d’observer la direction de polarisation de fluorescence des dipôles à la fois dans la pSIM et le 3ODM, la résolution des images est encore insuffisante, étant à peine suffisante pour des observations approximatives. Cependant, les images à haute résolution sont particulièrement cruciales lors du processus d’analyse et de prise de décision. Pour obtenir des détails plus fins, nous devons toujours nous appuyer sur la plateforme MATLAB et utiliser des algorithmes de traitement d’image avancés tels que des techniques de reconstruction à super-résolution pour un traitement en profondeur. L’absence de reconstruction complète avec notre plugin est due aux faibles performances du script beanshell utilisé. La reconstruction complète à l’aide de beanshell prendra beaucoup plus de temps que l’acquisition d’images. Les travaux futurs comprennent la mise en œuvre de la reconstruction complète avec Java et le calcul parallèle et l’inclusion de la fonctionnalité dans l’aperçu en direct.
Nous espérons que ce plugin améliorera l’efficacité de l’expérience en observant l’état de polarisation en temps réel. L’observation instantanée permet aux chercheurs d’observer immédiatement les orientations des dipôles dans les spécimens afin de localiser immédiatement la cible d’intérêt ou un événement rare lors du suivi en temps réel. Le protocole fournit également la configuration matérielle et le contrôle logiciel, ce qui rend pSIM et 3DOM plus conviviaux.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par le Programme national de recherche et de développement clé de la Chine (2022YFC3401100).
matériels
| Name | Company | Catalog Number | Comments |
| 100 nm Fluorescent beads | Invitrogen | F8801 | |
| 4% Formaldehyde solution | Invitrogen | R37814 | |
| Camera | Tucsen | Dhyana 400BSI V3 | https://www.tucsen.com/download-software/ |
| Denture base materials (Type I Thermally setting type, liquid) | New Century Dental | N/A | |
| Dulbecco’s Modified Eagle’s Medium | Gibco | C11995500BT | |
| Eclipse TE2000 Inverted Microscope | Nikon | TE2000 E | |
| Fetal Bovine Serum | Gibco | 10099141C | |
| MATLAB R2019b | MathWorks | Version R2019b | https://ww2.mathworks.cn/downloads/ |
| MetroCon V4.0 | Kopin | Version 4.0 | Software of Spatial light modulator |
| Micro-Manager 2.0 | μΜanager | Version 2.0 | Download Micro-Manager Latest Release |
| MS-2000 XYZ Automated Stage | Applied Scientific Instrumentation | MIM3 | https://www.asiimaging.com/support/downloads/usb-support-on-ms-2000-wk-controllers/ |
| myDAQ | National Instruments | 781325-01 | Software and Driver Downloads - NI |
| OBIS 561 nm LS 20 mW Laser | Coherent | 1325777 | |
| Phalloidin-AF568 | Invitrogen | A12380 | |
| Phosphate buffered saline | Corning | 21-040-CV | |
| Poly Methyl Methacrylate | Solarbio | M9810 | |
| ProLong Diamond | Invitrogen | P36980 | |
| Spatial light modulator | Kopin | SXGA-12 | |
| SYTOX orange nucleic acid stain | Invitrogen | S11368 | |
| Triton X-100 | Invitrogen | HFH10 | |
| Trypsin | Gibco | 25200056 | |
| λ-DNA | Invitrogen | S11368 |
Références
- Zhanghao, K., Gao, J., Jin, D., Zhang, X., Xi, P. Super-resolution fluorescence polarization microscopy. J Innov Opt Health Sci. 11 (01), 1730002(2018).
- Alonso, M. A., Brasselet, S. Polarization microscopy: from ensemble structural imaging to single-molecule 3D orientation and localization microscopy. Optica. 10 (11), 1486-1510 (2023).
- Valades Cruz, C. A., et al. Quantitative nanoscale imaging of orientational order in biological filaments by polarized superresolution microscopy. Pro Natl Acad Sci USA. 113 (7), E820-E828 (2016).
- Zhanghao, K., et al. Super-resolution dipole orientation mapping via polarization demodulation. Light Sci Appli. 5 (10), e16166(2016).
- Zhanghao, K., et al. Super-resolution imaging of fluorescent dipoles via polarized structured illumination microscopy. Nat Commun. 10 (1), 4694(2019).
- Vrabioiu, A. M., Mitchison, T. J. Structural insights into yeast septin organization from polarized fluorescence microscopy. Nature. 443 (7110), 466-469 (2006).
- Backer, A. S., Lee, M. Y., Moerner, W. E. Enhanced DNA imaging using super-resolution microscopy and simultaneous single-molecule orientation measurements. Optica. 3 (6), 659-666 (2016).
- Backer, A. S., et al. Single-molecule polarization microscopy of DNA intercalators sheds light on the structure of S-DNA. Sci Adv. 5 (3), eaav1083(2019).
- Hulleman, C. N., et al. Simultaneous orientation and 3D localization microscopy with a Vortex point spread function. Nat Commun. 12 (1), 5934(2021).
- Kampmann, M., Atkinson, C. E., Mattheyses, A. L., Simon, S. M. Mapping the orientation of nuclear pore proteins in living cells with polarized fluorescence microscopy. Nat Struct Mol Biol. 18 (6), 643-649 (2011).
- Lazar, J., Bondar, A., Timr, S., Firestein, S. J. Two-photon polarization microscopy reveals protein structure and function. Nat Methods. 8 (8), 684-690 (2011).
- Dong, B., et al. Parallel Three-Dimensional Tracking of Quantum Rods Using Polarization-Sensitive Spectroscopic Photon Localization Microscopy. ACS Photonics. 4 (7), 1747-1752 (2017).
- Blanchard, A., et al. Turn-key mapping of cell receptor force orientation and magnitude using a commercial structured illumination microscope. Nat Commun. 12 (1), 4693(2021).
- Toprak, E., et al. Defocused orientation and position imaging (DOPI) of myosin V. Proc Natl Acad Sci USA. 103 (17), 6495-6499 (2006).
- Zhong, S., et al. Three-dimensional dipole orientation mapping with high temporal-spatial resolution using polarization modulation. PhotoniX. 5 (1), 12(2024).
- Young, L. J., Ströhl, F., Kaminski, C. F. A Guide to Structured Illumination TIRF Microscopy at High Speed with Multiple Colors. J Vis Exp. (111), e53988(2016).
- Huang, X., et al. long-term, super-resolution imaging with Hessian structured illumination microscopy. Nat Biotechnol. 36 (5), 451-459 (2018).
- Ando, R., et al. StayGold variants for molecular fusion and membrane-targeting applications. Nat Methods. 21 (4), 648-656 (2024).
- Hirano, M., et al. A highly photostable and bright green fluorescent protein. Nat Biotechnol. 40 (7), 1132-1142 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon